このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
脂質混合アッセイ用リポソームへの組換えショウジョウバエアラストシンの洗剤支援再構成
要約
生体膜融合は、特殊な融合タンパク質によって触媒されます。タンパク質のフソジェニック特性の測定は、脂質混合アッセイによって達成することができる。ERの同質融合を媒実化するタンパク質である組換えショウジョウバエを精製し、あらかじめ形成されたリポソームに再構成し、融合能力を試験する方法を提示する。
要約
膜融合は真核細胞における重要なプロセスです。融合を触媒するには特殊なタンパク質が必要です。アラストリンは、ERの同質性融合に関与する小プラズム網膜(ER)常駐タンパク質である。ここでは、グルタチオンS-トランスフェラーゼ(GST)とポリヒスチジンタグドロソフィラアラストシンを2ラウンドの親和性クロマトグラフィーで精製する方法を詳述する。インビトロで融合反応を研究するには、精製された融合タンパク質を脂質二重層に挿入する必要があります。リポソームは、脂質組成物およびサイズが調整されてもよいとして、理想的なモデル膜である。そのために、予め形成されたリポソームにショウジョウバエのアラストリンに対する洗剤除去による再構成法について述べた。いくつかの再構成方法が利用可能であるが、洗剤除去による再構成は、アラストシンおよび他の同様のタンパク質に適しているいくつかの利点を有する。この方法の利点は、高い再構成収率および再構成されたタンパク質の正しい向きを含む。この方法は、他の膜タンパク質およびプロテオリポソームを必要とする他のアプリケーションに拡張することができる。さらに、膜融合の測定として用いられるプロテオリポソームのFRETベース脂質混合アッセイについて述べる。
概要
膜融合は、多くの生物学的反応において重要なプロセスです。生物学的条件下では、膜融合は自発的ではなく、そのような反応を触媒するために特殊な融合タンパク質を必要とする1.ERホモティピック膜融合は、ダイナミン関連GTPaseアラストシン2によって動物に媒介される。同質融合におけるアラストシンの役割は、末梢ERの三方接合部の基本であり、細胞全体に広がる管状の大規模な相互接続ネットワークを構成する。アラストニンは、大きなGTPase、3つのらせん束中ドメイン、疎水性膜アンカー、および短い細胞質C末端尾3からなる保存されたドメイン形態を有する。組換えショウジョウバエアラトミンを伴うインビトロ研究では、リポソームに再構成すると、そのフソジェニック特性を維持することが示されている。ヒトホモロを含む他のアラストチンは、インビトロでの融合を要約することができませんでした。ここでは、GSTとポリヒスチジンを精製する方法論について、アラストシンの組換えショウジョウバエをタグ付けし、リポソームに再構成し、融合をアッセイする方法論を説明する。
インビトロでの膜融合の研究は、通常、フソジェニックタンパク質が膜アンカーを持っているため、課題を提示します。それらを研究するためには、モデル脂質二重層にそれらを再構成する必要があります。大きなユニラメラ小胞(LUV)は、脂質タンパク質相互作用を研究するのに有用なツールです。ここでは、タンパク質再構成および融合アッセイ用の異なる脂質組成物のLuVを作るシステムを提示する。LUVへの一体タンパク質の再構成は、有機溶媒媒介性再構成、機械的メカニズム、または洗剤支援再構成4を含む様々な方法によって達成することができる。ここでは、洗剤除去により予め形成されたリポソームにショウジョウバエのアラストシンを再構成する方法を提示する。この再構成法の利点は、高い再構成収率と脂質二重層におけるアラストシンの適切な配向を含む。さらに、この方法を通じて、タンパク質は乾燥したり有機溶媒にさらされたりせず、構造や機能を維持します。その欠点の中で、洗剤の存在はすべてのタンパク質に理想的ではない可能性があり、最終的なプロテオリポソームは、脂質二重層に何らかの洗剤を組み込む可能性があります。さらに透析は、より多くの洗剤を排除するために使用されてもよい。しかし、透析には時間がかかるため、タンパク質活性が失われる可能性があります。
アラストシンの融合活性を評価することは、前述の2として脂質混合アッセイによって決定することができる。ここでは、N-(7-ニトロベンツ-2-オキサ-1,3-ジアゾル-4-yl(NBD)/リッサミンローダミンBスルホニル(ローダミン)標識脂質を介したアラストイン媒介融合を測定する方法を用いて述記する。このアッセイは、ドナー(標識された)プロテオリポソームと受諾器(ラベルなし)プロテオリポソームの融合を必要とする。FRET放出は、膜融合中の脂質混合の結果として、「標識された」リポソームから「標識されていない」リポソームへのドナー-受諾ペアの希釈として反応中に測定することができる(図1)5。このアッセイは膜融合の代理として機能するが、膜融合とヘミフュージョン(外側のリーフレットのみが混在する状態)を区別することに制限がある。この問題に対処するために、代替案は、ジチオニテによるNBDの外側のリーフレットクエンチングである。NBD/ロダミン脂質混合アッセイと同様の方法論に続いて、融合による任意のNBD FRET放出を外側のリーフレットを消光すると、内部リーフレット混合8に起因する。
内部水性含有量混合による代替融合アッセイアドレスフルフュージョンのみ5.その例としては、テルビウム(Tb)/ジピコリン酸(DPA)アッセイおよびアミノナフタレントリスルホン酸(ANTS)/p-キシレンビス(ピリジニウム)臭化物(DPX)アッセイがあります。Tb/DPAアッセイでは、カプセル化されたTbを持つリポソームのプールが混合され、封入されたDPAとリポソームと融合されます。融合時に、蛍光は[Tb(DPA)3]3-キレート複合体6内のDPAからTbへの内部エネルギー伝達を介して増加する。対照的に、ANTS/DPXアッセイの場合、ANTS蛍光はDPX7によって消光されます。これらのシステムは内部含有量の混合に対処する一方で、非カプセル化試薬の除去、ならびに蛍煙素の意図しない相互作用のために、リポソームのより詳細な調製が必要である。
プロトコル
1. GST-DAtl-His8の精製
-
タンパク質発現とリサート製剤
- pGEX4-T32のGST-DAtl-His8コンストラクトでBL21(DE3)大腸菌を変換し、アンピシリンプレート上で選択します。
- 14 mL培養チューブでLB+アンピシリン(100mg/mLアンピシリンの5μL)を単一の形質転換体を選択し、6-8時間の200rpmで振盪して25°Cでインキュベートします。
注:漏れ発現のため、より高い温度でインキュベートすることはお勧めしません。25 °Cでの成長は、この成長期間中のタンパク質の凝集を減少させます。 - LB+アンピシリンの200mLを5mL培養の1mLで接種し、25°Cで一晩(〜15〜18時間)インキュベートします。
- 翌朝、遠心分離(2,000 x g~10分)で細胞を収穫し、LBの5mLで再中断する。
- LB+アンピシリンの4Lを接種し、OD 600(0.05~0.15)を測定します。振りながら25°Cでインキュベートします。
注:細菌を追加する前に、OD600測定のブランクとして機能するいくつかのメディアを予約します。 - ODは0.4 ~ 0.5 の間の OD に達するまで、1 時間ごとに OD 600 を測定します。この時点で、温度を16°Cに下げます。
- 0.2 mM IPTG(1Mストックの800 μL)で誘導し、インキュベーターが16°Cに達してから10分後に誘導する。16°Cで一晩(~15~18時間)インキュベートします。
注:温度が低いほど、アラストシンの凝集を減らすことによって機能性タンパク質の収率が向上します。 - 翌朝、4°Cで7,500 x gで遠心分離して細胞を収穫する。
- A200(25 mM HEPES(pH 7.4)および200 mM KCl)の200 mLで細胞を再中断します。
- 4°Cで5分間11,000 x gで細胞を遠心分離します。
- 破断バッファーの40 mLでペレットを再中断する(A200プラス10%グリセロール、2 mM 2-メルカプトエタノール、4%トリトンX-100(TX100)、40mMイミダゾール、および1 EDTAフリープロテアーゼ阻害剤カクテル錠剤)。
注:気泡や泡の発生を避けるために、再中断後にTX-100を追加します。 - 18 G の針を通過し、約 10,000 psi で細胞破壊を 3 回実行します。
- 1時間125,000 x gで抽出物を遠心分離します。
注:必要に応じてペレット1:2(w:v)を8M尿素に溶解し、室温で一晩ナットします。脱窒剤としての尿素は、SDS-PAGE分析のために不溶性ペレットタンパク質をゆっくりと溶解します。SDS-PAGEおよびクーマッシー染色分析のために1 μLを節約する。 - 0.45 μmのセルロース硝酸塩生殖不能膜フィルターを通して抽出物を濾過し、細菌や大きな細菌の破片を除去します。
注:必要に応じて、SDS PAGEおよびクーマッシー染色分析用に1 μLを保存します。
-
親和性クロマトグラフィーによるタンパク質精製
- 濾過されたライサテを固定化された金属親和性クロマトグラフィー(IMAC)樹脂カラムにロードし、Ni2+を帯電し、低イミダゾールバッファー(A100プラス10%グリセロール、2mM 2-メルカプトエタノール、1%TX-100、40mM imidazole)で予め平衡化4 °Cで分る。
- A100プラス10%グリセロール、2 mM 2-メルカプトエタノール、0.1%アナポーX-100、40 mMイミダゾールの25 mLでカラムを洗い、4°Cで1mL/minの割合で洗います。40 mMから500mMのイミダゾールの30 mL線形勾配でタンパク質を溶出し、500mMで最終的な5mL洗浄を行います。
- ピーク画分を一緒にプールし、4°Cで1時間のインキュベートを4°Cで膨らませ、以前は水に腫れ、10%グリセロール、2mM 2-メルカプトエタノール、0.1%アナポエX-100、および1mM EDTAでA100で平衡化した。
注:GSH-アガロースビーズは、前日に50mLの水で膨らませ、4°Cで一晩、またはRTで1時間インキュベートすることができます。水と平衡バッファーを除去するには、ブレーキなしで1分間500 x gのスイングバケツローターで遠心分離機を使用します。26 G針で上清を吸引します。 - ペレットGSH-アガロースビーズは、ブレーキなしで500 x gのスイングバケツローターで遠心分離し、26Gの針で吸引によってリサート上清を除去します。
- ビーズを10mLポリプレップカラムに移し、5mLの平衡バッファー(10%グリセロール付きA100、2mM 2-メルカプトエタノール、0.1%アナポーX-100、および1mM EDTA)を500x gで遠心分離することにより5回洗浄します。
- 10 mM減少グルタチオンを補充した平衡バッファーの1-1.5 mLでタンパク質を溶出させる。アリコトは液体窒素中のタンパク質とフラッシュ凍結を放流した。タンパク質は-80°Cで無期限に保存できます。
注:溶出バッファーの pH を 7.4 に調整します。 - アミドブラックタンパク質アッセイ9によるタンパク質濃度を定量し、SDS-PAGEおよびクマシー染色により純度を評価する。
2. 組換えアラストシンをリポソームに再構成
- 押し出し法によるリポソーム製造10歳
- クロロホルム(10mM総脂質)で脂質ミックスストックを作ります。必要な脂質は、1-パルミトイル-2-オレオイルグリセロ-3-ホスホコリン(POPC)、1,2-ジオレオイル-sn-グリセロ-3-ホスホ-セリン(DOPS)、、 1,2-ジオレオイル-sn-グリセロ-3-ホスホエタノールアミン-N-(7-ニトロ-2-1,3-ベンゾキサジアゾロール-4-yl)(NBD-DPPE)、および1,2-ジパルミトイル-sn-グリセロ-3-ホスホエタノールアミン-N-(リッサミンロダミンBスルホニル)(Rh-DPPE)。受け入れリポソームは、POPC:DOPS(85:15モル比)とPOPC:DOPS:Rh-PE:NBD-PE(83:15:1.5:1.5モル比)のドナーリポソームで構成されています。
注:POPC:DOPS脂質ミックスは、伝統的にインビトロ脂質混合アッセイに使用されてきましたが、代替脂質組成物は、異なる実験目的に適合してもよい。POPC:DOPSリポソームは非常に安定しており、融合しにくいため、融合には非常に厳しいシステムです。 - L-α-ジパルミトイル-ホスファチジルコリン(コリンメチル-3H)の1 μCi/mLを脂質混合に加え、液体シンチレーション計数により後工程で脂質濃度を決定する。この在庫の少なくとも 8 μL を予約します。
- フリントガラスチューブに脂質ミックスの所望の量を転送します。
- クロロホルムが見えなくなるまで、N2ガスの穏やかな流れの下で脂質ミックスを10分間乾燥させます。
- 30分間真空でデシケータでさらに脂質フィルムを乾燥させます。
- 脂質フィルムに10%グリセロール、2 mM 2-メルカプトエタノール、および1 mM EDTAを加え、濃度を10mMに戻す。室温で15分間軽く渦を塗って脂質フィルムを再濁させる。フリントガラスチューブを収容できるボルテキサーをお勧めします。
- 液体窒素中の水和脂質を10回凍結解凍する。液体窒素中で凍結した後、溶液を室温で30sに座らせてリポソームを解凍し、その後、より速く解凍するために水に移します。この凍結融解サイクリングは、マルチラメラ小胞を最小限に抑えます。
注意:チューブに0.5mLを超える体積が含まれ、液体窒素を水に直接移すと割れる場合があります。 - ミニ押出機を使用して100 nmの細孔サイズのポリカーボネートフィルターを通して脂質を渡します。
- シンチレーションカウントによるリポソームの総脂質濃度の決定
注:脂質の一部は、ガラス管やミニ押出機に残されることがあります。- シンチレーションカクテルの3 mLに脂質ストックとリポソームの4 μLを追加します。
- ストックおよびリポソームの 1 分あたりの平均数 (CPMA) を測定します。そして、次の式でリポソーム濃度を計算します。
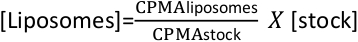
- リポソームを4°Cで1週間保存します。リポソームは時間とともに集約され、再構成効率が低下する可能性があるため、保存時間を長くすることはお勧めしません。
- クロロホルム(10mM総脂質)で脂質ミックスストックを作ります。必要な脂質は、1-パルミトイル-2-オレオイルグリセロ-3-ホスホコリン(POPC)、1,2-ジオレオイル-sn-グリセロ-3-ホスホ-セリン(DOPS)、、 1,2-ジオレオイル-sn-グリセロ-3-ホスホエタノールアミン-N-(7-ニトロ-2-1,3-ベンゾキサジアゾロール-4-yl)(NBD-DPPE)、および1,2-ジパルミトイル-sn-グリセロ-3-ホスホエタノールアミン-N-(リッサミンロダミンBスルホニル)(Rh-DPPE)。受け入れリポソームは、POPC:DOPS(85:15モル比)とPOPC:DOPS:Rh-PE:NBD-PE(83:15:1.5:1.5モル比)のドナーリポソームで構成されています。
- あらかじめ形成されたリポソームへの洗剤による再構成
- 一緒に混合するバッファー、タンパク質、リポソームと余分な洗剤の量を計算します。
- 必要な合計ボリュームを決定します。この容積は10%のグリセロール、2 mM 2-メルカプトエタノールおよび1 mM EDTAの緩衝A100から成っている。250 μL 未満の容積は、0.5 mL マイクロ遠心分離管ではうまく混合されません。
- 約1mMの最終的な脂質濃度を与えるために必要なリポソームの体積を計算します。バッファー・ボリュームからボリュームを減算します。
- 所望のタンパク質を脂質モル比に与えるために必要なタンパク質の体積を計算します(通常は1:400)。それに応じてバッファボリュームを減らす。
- リポソームを飽和させるために追加する必要がある追加の洗剤の量を決定します 0.64 - 0.8 の間の有効な洗剤の脂質比 (Reff)を目指します。タンパク質に添加される洗剤を考慮することを忘れないでください。(Reff)は、総洗剤
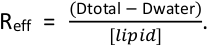 濃度とD水量の合計が単体洗剤濃度(TX-100及びアナポエX-100の存在下で0.18mM)である式Dによって決定される4、 11.
濃度とD水量の合計が単体洗剤濃度(TX-100及びアナポエX-100の存在下で0.18mM)である式Dによって決定される4、 11.
- バッファー、洗剤、タンパク質、リポソーム:0.5 mLチューブで溶液を一緒に混ぜます。リポソームを迅速に加え、5s用にすぐに渦を加えて混合物を均質化する。
- 4°Cで1時間のヌタターで反応をインキュベートします。
- 0.2 g/mLの非極性ポリスチレン吸着ビーズ「スラリー」を水中に入れます。
注:ステップ2.2.3で1時間インキュベーションの前の間にポリスチレン吸着ビード「スラリー」を作ります。 - ポリスチレン吸着ビーズの0.2gを計量し、マイクロ遠心管に移します。
- チューブにメタノールの1 mLを追加することにより、ビーズをドガと1分間混合し、ドガセドビーズが沈みます。
- メタノールを吸引し、ビーズに水を加えます。ビーズを5分間水と混ぜ、水を吸引します。4回繰り返し、ポリスチレン吸着ビード「スラリー」を水で1mLの体積にし、最終的な濃度を0.2g/mLにします。
注:ビーズはまだチューブの底に落ち着く必要があります。そうでない場合は、再びドガ。21G以上の針を使用して、ビーズからメタノールと水を吸引します。 - 各サンプルのすべての洗剤を吸収するために必要なポリスチレン吸着ビーズの量を計算します。ポリスチレン吸着ビーズの1gは、TX-100の70mgを吸収します。各反応に必要なポリスチレン吸着ビードスラリーの体積を計算するには、反応中の全洗剤(ステップ2.2.1.4)を70mgで割り、次いでビーズスラリーの濃度(0.2 g/mL(ステップ2.2.7)で割ります。
- 計算されたポリスチレン吸着ビーズスラリーの量を0.5mLチューブに移し、水を吸引する。
注:ビーズを転送するために20〜200 μLの先端の端をカットし、落ち着いたビーズを再中断するためにピペッティングの直前にチューブを渦。 - ポリスチレン吸着ビーズを含む0.5 mLチューブにサンプルを追加し、4°Cで1時間のナタチングサンプルをインキュベートします。
- 古いビーズを残して、新鮮なビーズにサンプルを転送する2回繰り返します。
- 新鮮なビーズで4番目のチューブにサンプルを追加し、4°Cで一晩(〜15〜18時間)インキュベートします。
- 一緒に混合するバッファー、タンパク質、リポソームと余分な洗剤の量を計算します。
- 午前中は、4°Cで16,000 x gで10分間遠心分離することにより、ポリスチレン吸着ビーズおよびペレット不溶性タンパク質凝集体からサンプルを除去する。
- 上清を回収し、液体シンチレーション計数で最終的な脂質濃度を決定します(ステップ2.1.9.2を参照)。任意に、タンパク質濃度は、アミド黒タンパク質アッセイ9によって決定されてもよい。
注:アラストシンプロテオリポソームの場合、酵素アッセイは新鮮なリポソームで行われるべきである。4°Cまたは凍結での長い貯蔵は、アラストシン活性の著しい損失につながる。
3. 脂質混合アッセイ
- ドナーおよびアクセプタープロテオリポソームをA100にそれぞれ0.15mMの濃度に持ち込み、10%のグリセロール、2 mM β-メルカプトエタノール、1 mM EDTAおよび5 mM MgCl2を含む。各反応(50μL)は、蛍光測定値に適した平らな白色96ウェルプレートでウェルに添加する必要があります。三つ三つ、および非GTP陰性制御を含む少なくとも4つの反応を準備する。
- プレートを予熱プレートリーダーに37°Cに置きます。NBD蛍光(励起460nmおよび発光535nm)を毎分5分間測定します。
- 5 mM GTP (50 mM GTP の 5 μL) を加えて融合を誘導します。
- 毎分1時間毎分NBD蛍光を測定します。
- 2.5%w/v n-ドデシルβ-D-マルトシドの5 μLを加えてプロテオリポソームを溶解し、最大NBD蛍光量を測定する。毎分15分間NBD蛍光を読んでください。
4. イオエキソール不連続勾配上のリポソーム浮動12
- (オプション)浮動アッセイ13による再構成の効率を分析する。
- 10%グリセロール、2 mM 2-メルカプトエタノール、および1 mM EDTAでA100に80%および30%w/v iohexolを調製します。イオエクソールは容易に溶解しないため、4°Cで一晩nutatingすることにより、前日に準備する必要があります。
- 80%イオエキソールストックの150 μLを150μLのプロテオリポソームサンプルと完全に混合し、40%のイオエキソールに持ち込みます。5 x 41 mm2超クリアチューブに追加して気泡を避けます。
- 30%イオエキソールストックの層250μLをサンプルの上にゆっくりと中間層を作る。任意の気泡を避け、下の層を乱します。中間層の上に、2 mM 2-メルカプトエタノールと1 mM EDTAでA100の50 μLをゆっくりと追加します。
- スイングバケットローターの勾配を220,000 x gで4°Cで4°Cで遠心分離し、ゆっくりと加速し、ブレークなし。
- グラデーションの層を収穫し、SDS-PAGEおよびクーマッシー染色物によって分析し、定量化は密度メトリーによって行われてもよい。再構成されたタンパク質は最上層に浮かぶ必要があり、タンパク質および脂質凝集体は底部または中間層に堆積する。
5. トロンビンプロテオリシスによる再構成タンパク質の配向の解析
注:ここで報告されたアラストイン構造体は、N末端GSTタグの終端とアラストシンの始まりとの間にトロンビン切断部位を有する。正しい向きでアラストシンは、この切断部位がプロテアーゼにアクセス可能になり、間違った方向のタンパク質は脂質二重層によって保護される。
- アラストシンプロテオリポソームの向きをアッセイするには、新鮮な再構成されたプロテオリポソームの少なくとも8 μLを予約する。
- プロテオリポソームの8 μLと1 ΜLの1 μLを加えます。37 °Cで1時間インキュベートします。
- EDTAフリープロテアーゼ阻害剤カクテルの5mg/mLの1 μLでプロテアーゼをクエンチし、37°Cで30分間インキュベートします。
- SDS-PAGEとクーマッシー染色でサンプルを分析します。クチビと不潔なタンパク質の割合は、デンシトメトリーによって決定されてもよい。
結果
アラストシン再構成の効率を図2に示す。再構成されたプロテオリポソームは、イオエクソール不連続勾配で浮動させた。未組み込みタンパク質は、底層(B)または中間層(M)に堆積させた。再構成されたタンパク質は、最上層(T)に浮動するであろう。グラデーションのサンプルをSDS-PAGEおよびクーマシー染色によって採取し、分析した。デンシトメトリーによるゲルの定量?...
ディスカッション
ここでの方法は、アラストシン組換えの核融合活性を精製、再構成、測定するための効率的な方法を示す。機能的なアラストインの高い収率を確保するためにいくつかの重要なステップを考慮する必要があります。.アラストシンの発現は、凝集を避けるために低温(16°C)で行う必要があり、1つは0.4〜1.5 mg/mLの間の最終的な濃度を目指す必要があります。非常に希薄なタンパク質は、脂質比に?...
開示事項
著者は何も開示していない。
謝辞
私たちは、マイケル・スターン博士と彼の研究室に対して、アラストイン関連プロジェクトに関する洞察とフィードバックに感謝します。この研究は、国立総合医学研究所[R01GM101377]と国立神経障害・脳卒中研究所[R01NS102676]によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 10 mL poly-prep chromatography columns | Biored | 731-1550 | |
| 10 x 75 mm Flint glass tubes | VWR | 608225-402 | |
| 47 mm diameter, 0.45um pore whatman sterile membrane filters | Whatman | 7141 104 | |
| 96 well white plate | NUNC | 437796 | |
| Anapoe X-100 | Anatrace | 9002-931-1 | |
| Cell disrupter | Avestin | Avestin Emulsiflex C3 | |
| DOPS (1,2-dioleoyl-sn-glycero-3-phospho-L-serine (sodium salt)) | Avanti | 840035P-10mg | DOPS |
| EDTA | Research organics inc. | 6381-92-6 | Ethylenediaminetetraacetic acid |
| EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Complete protease inhibitor |
| Extruder | Sigma Aldrich | Z373400 | Liposofast Basic Extruder |
| GE Akta Prime liquid chromatography system | GE Pharmacia | 8149-30-0006 | |
| Glutathione agarose beads | Sigma aldrich | G4510-50ml | |
| Glycerol | EMD | GX0185-5 | |
| GTP | Sigma Aldrich | 36051-31-7 | Guanosine 5' triphosphate sodium salt hydrate |
| HEPES, acid free | Omnipur | 5330 | |
| Imidazole | fluka | 5670 | |
| Immobilized metal affinity chromatography (IMAC) resin column | GE Healthcare | 17040801 | 1 mL HiTrap Chelating HP immobilized metal affinity chromatography columns |
| Iohexol | Accurate chemical and scientific corporation | AN 7050 BLK | Accudenz/Nycodenz |
| IPTG | Research products international corp. | I56000-100.0 | IPTG, dioxane free |
| L-Glutathione reduced | Sigma-Aldrich | G4251-5g | |
| Magnesium chloride | Fisher | 7791-18-6 | |
| Methanol | Omnisolv | MX0488-1 | |
| NBD-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl) (ammonium salt)) | Avanti | 236495 | NBD-DPPE |
| n-Dodecyl β-D-maltoside | Chem-Impex International | 21950 | |
| Nonpolar polystyrene adsorbent beads | BioRad | 152-3920 | SM2 Biobeads |
| Nuclepore track-etch polycarbonate 19 mm 0.1 um pore membrane | Whatman | 800309 | |
| Optima LE80K Ultra centrifuge | Beckman Coulter | ||
| Phosphatidylcholine, L-α-dipalmitoyl [choline methyl-3H] | ARC | ART0284 | Titriated lipids |
| Plate reader | TECAN | TECAN infinite M200 plate reader | |
| POPC (1-palmitoyl-2-oleoyl-glycero-3-phosphocholine) | Avanti | 850457C-25mg | POPC |
| Potassium chloride | MP | 151944 | |
| Rh-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(lissamine rhodamine B sulfonyl) (ammonium salt)) | Avanti | 236495 | Rh-DPPE |
| Scintillation Cocktail | National Diagnostics | LS-272 | Ecoscint XR Scintillation solution for aqueous or non-aqueous samples |
| Scintillation vials | Beckman | 592928 | Fast turn cap Mini Poly-Q Vial |
| Thrombin | Sigma | T1063-1kU | Thrombin from human plasma |
| Triton X-100 | Fisher | BP151-500 | |
| Ultra-clear centrifuge tubes 5 x 41 mm | Beckman | 344090 | |
| Vortex 9 to 13mm Tube Holder | VWR | 58816-138 | Insert for vortexing flint glass tubes |
| Vortex Insert Retainer | VWR | 58816-132 | Retainer needed for vortex tube holder |
| Vortexer | VWR | 2.235074 | Vortex Genie 2 model G560 |
| β-mercaptoethanol molecular biology grade | Calbiochem | 444203 |
参考文献
- Chernomordik, L. V., Kozlov, M. M. Mechanics of membrane fusion. Nature Structural and Molecular Biology. 15, 675-683 (2008).
- Orso, G., et al. Homotypic fusion of ER membranes requires the dynamin-like GTPase Atlastin. Nature. 460, 978-983 (2009).
- McNew, J. A., Sondermann, H., Lee, T., Stern, M., Brandizzi, F. GTP-dependent membrane fusion. Annual Review of Cell and Developmental Biology. 29, 529-550 (2013).
- Rigaud, J. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1231, 223-246 (1995).
- Düzgüneş, N. Fluorescence Assays for Liposome Fusion. Methods in Enzymology. 372, 260-274 (2003).
- Wilschut, J., Duzgunes, N., Fraley, R., Papahadjopoulos, D. Studies on the mechanism of membrane fusion: kinetics of calcium ion induced fusion of phosphatidylserine vesicles followed by a new assay for mixing of aqueous vesicle contents. Biochemistry. 19, 6011-6021 (1980).
- Ellens, H., Bentz, J., Szoka, F. C. Proton- and calcium-induced fusion and destabilization of liposomes. Biochemistry. 24, 3099-3106 (1985).
- Meers, P., Ali, S., Erukulla, R., Janoff, A. S. Novel inner monolayer fusion assays reveal differential monolayer mixing associated with cation-dependent membrane fusion. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1467, 277-243 (2000).
- Schaffner, W., Weissmann, C. A rapid, sensitive, and specific method for the determination of protein in dilute solution. Analytical Biochemistry. 56, 502-514 (1973).
- MacDonald, R. C., et al. Small-volume extrusion apparatus for preparation of large, unilamellar vesicles. Biochimica Et Biophysica Acta. 1061, 297-303 (1991).
- Paternostre, M. T., Roux, M., Rigaud, J. L. Mechanisms of membrane protein insertion into liposomes during reconstitution procedures involving the use of detergents. 1. Solubilization of large unilamellar liposomes (prepared by reverse-phase evaporation) by Triton X-100, octyl glucoside, and sodium cholate. Biochemistry. 27, 2668-2677 (1988).
- Scott, B. L., et al. Liposome Fusion Assay to Monitor Intracellular Membrane Fusion Machines. Methods in Enzymology. 372, 274-300 (2003).
- Zhang, W., et al. Crystal structure of an orthomyxovirus matrix protein reveals mechanisms for self-polymerization and membrane association. PNAS. 114, 8550-8555 (2017).
- Parlati, F., et al. Rapid and efficient fusion of phospholipid vesicles by the α-helical core of a SNARE complex in the absence of an N-terminal regulatory domain. PNAS. 96, 12565-12570 (1999).
- Sugiura, S., Mima, J. Physiological lipid composition is vital for homotypic ER membrane fusion mediated by the dynamin-related GTPase Sey1p. Scientific Reports. 6, 20407 (2016).
- Powers, R. E., Wang, S., Liu, T. Y., Rapoport, T. A. Reconstitution of the tubular endoplasmic reticulum network with purified components. Nature. 543, 257-260 (2017).
- Betancourt-Solis, M. A., Desai, T., McNew, J. A. The atlastin membrane anchor forms an intramembrane hairpin that does not span the phospholipid bilayer. Journal of Biological Chemistry. 293, 18514-18524 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved