É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Reconstituição detergente-assistida da Drosophila atlastin recombinante em lipossomas para ensaios de mistura de lipídios
Neste Artigo
Resumo
A fusão biológica da membrana é catalisada por proteínas especializadas da fusão. Medir as propriedades fusogénicas das proteínas pode ser conseguida por ensaios de mistura lipídico. Nós apresentamos um método para purificante o atlastin de recombinação da Drosophila, uma proteína que medese a fusão neutralizantes do er, reconstituindo o aos lipossomas pré-formados, e testando para a capacidade da fusão.
Resumo
A fusão da membrana é um processo crucial na pilha eucarióticas. As proteínas especializadas são necessárias para catalisar a fusão. As atlastinas são proteínas residentes do retículo endoplasmático (er) implicadas na fusão neutralizantes do er. Nós detalhar aqui um método para purificar uma Glutationa S-transferase (GST) e poli-histidina Tagged Drosophila atlastin por duas rodadas de cromatografia de afinidade. Estudar reações de fusão in vitro requer que as proteínas de fusão purificadas sejam inseridas em uma bicamada lipídica. Os lipossomas são membranas modelo ideais, pois a composição lipídica e o tamanho podem ser ajustados. Para este fim, nós descrevemos um método da reconstituição pela remoção detergente para o atlastin de Drosophila em lipossomas pré-formados. Enquanto vários métodos de reconstituição estão disponíveis, a reconstituição por remoção de detergente tem várias vantagens que o tornam adequado para atlastinas e outras proteínas semelhantes. A vantagem deste método inclui um alto rendimento de reconstituição e orientação correta da proteína reconstituída. Este método pode ser estendido para outras proteínas de membrana e para outras aplicações que necessitam de proteoliposomas. Adicionalmente, nós descrevemos um ensaio de mistura baseado fret do lipido dos sobreviventes usados como uma medida da fusão da membrana.
Introdução
A fusão da membrana é um processo crítico em muitas reações biológicas. condições biológicas, a fusão da membrana não é espontânea e requer proteínas de fusão especializadas para catalisar tais reações1. A fusão neutralizantes da membrana do er é negociada nos animais pelo dynamin relacionou o atlastin do GTPase2. O papel da atlastin na fusão homotípica é fundamental para junções de três vias no ER periférico, que constitui uma grande rede interconectada de túbulos que se estendem por toda a célula. Atlastins têm uma morfologia conservada do domínio que consiste em um GTPase grande, em um domínio médio do pacote de três hélice, em uma escora hydrofóbica da membrana, e em um curto C-terminal cytoplasmic cauda3. Estudos in vitro com a Drosophila atlastin recombinante demonstraram que, quando reconstituídos a lipossomas, mantém as suas propriedades fusogênicas. Outros atlastins, incluindo homólogos humanos não puderam recapitular a fusão in vitro. Nós descrevemos aqui uma metodologia para purificante um GST e um poli-histidine etiquetaram o atlastin de recombinação da Drosophila, reconstituindo o aos lipossomas, e ensaio a fusão.
Estudar a fusão da membrana in vitro apresenta um desafio porque as proteínas atividade têm geralmente uma escora da membrana. A fim de estudá-los, é necessário reconstituí-los em bilayers lipídico modelo. As grandes vesículas unilamelares (LUV) são uma ferramenta útil para estudar as interações protéicas lipídicas. Apresentamos aqui um sistema para fazer LUVs de diferentes composições lipídicas para reconstituição de proteínas e ensaios de fusão. A reconstituição de proteínas integrais em LUVs pode ser alcançada por uma variedade de métodos, incluindo a reconstituição orgânica mediada por solventes, mecanismos mecânicos ou reconstituição assistida por detergente4. Nós apresentamos aqui um método para reconstituir o atlastin de Drosophila em lipossomas pré-formados pela remoção detergente. As vantagens deste método da reconstituição incluem rendimentos elevados da reconstituição e orientação apropriada do atlastin no bilayer do lipido. Adicionalmente, com este método, a proteína não é secada ou expor aos solventes orgânicos que mantêm desse modo a estrutura e a função. Entre suas desvantagens, a presença de detergentes pode não ser ideal para todas as proteínas e os proteoliposomas finais podem ter algum detergente incorporado na bicamada lipídica. Uma diálise adicional pode ser usada para eliminar mais do detergente. No entanto, a diálise pode demorar muito tempo e, portanto, pode levar à perda de atividade protéica.
A avaliação da atividade de fusão da atlastin pode ser determinada por ensaios de mistura lipídico como descrito anteriormente2. Aqui, Delineamos um método para medir a fusão mediada por atlastina através de lipídios rotulados de N-(7-nitrobenz-2-Oxa-1,3-diazol-4-YL (NBD)/lissamina RHODAMINE-B sulfonila (RHODAMINE). Este ensaio exige a fusão dos sobreviventes (etiquetados) doadores (rotulados) e do aceitador (unlabeled). Uma liberação de FRET pode ser medida durante a reação como a diluição de um par doador-aceitador de lipossomas "rotulados" para lipossomas "sem rótulo" como resultado da mistura lipídica durante a fusão da membrana (Figura 1)5. Quando este ensaio sere como um proxy para a fusão da membrana, é limitado em distinguir entre a fusão da membrana e o hemifusion, um estado onde somente os folhetos exteriores misturam. Para abordar este problema, uma alternativa é a têmpera de folheto externo de NBD por ditiononite. Seguindo a mesma metodologia que os ensaios de mistura de lipídeos de NBD/rhodamina, ao extinguir o folheto externo, qualquer liberação de NBD FRET por fusão será devida ao folheto interno que mistura8.
Ensaios alternativos de fusão por conteúdo aquoso interno misturando o endereço de fusão completa apenas5. Exemplos disso são os ensaios de ácido térbio (TB)/dipicolinic (DPA) e os ensaios de ácido aminonaphtaleno trisulfonico (formigas)/p-xylene bis (piridinium) (DPX). Em ensaios de TB/DPA, um conjunto de lipossomas com TB encapsulada é misturado e fundido com lipossomas com DPA encapsulado; após a fusão, a fluorescência é aumentada através da transferência de energia interna da DPA para a TB dentro do complexo de3 quelação de [TB (DPA)3]6. Em contraste, para os ensaios ANTS/DPX, a fluorescência da ANTS é apagada pelo DPX7. Quando estes sistemas endereçam o conteúdo interno que mistura, uma preparação mais detalhada dos lipossomas é exigida para a remoção de reagentes não-encapsulados, assim como a interação não intencional dos Fluorophores.
Protocolo
1. purificação de GST-DAtl-His8
-
Expressão protéica e preparação de lisados
- Transforme BL21 (DE3) e. coli com a construção de GST-Datl-His8 em PGEX4-T32 e selecione em uma placa de ampicilina.
- Selecione um único transformante e inocular 5 mL de LB + ampicilina (5 μL de 100 mg/mL de ampicilina) num tubo de cultura de 14 mL e incubar a 25 ° c com agitação a 200 rpm por 6-8 h.
Nota: Devido à expressão de vazamento, não é recomendável incubar em temperaturas mais elevadas. O crescimento a 25 ° c reduz a agregação de proteínas durante este período de crescimento. - Inocular 200 mL de LB + ampicilina com 1 mL da cultura de 5 mL e incubar durante a noite (~ 15 – 18 h) a 25 ° c.
- Na manhã seguinte, colher as células por centrifugação (2.000 x g por 10 min) e ressuscitava em 5 ml de lb.
- Inocular 4 L de LB + ampicilina e medir OD600 (0,05 – 0,15). Incubar a 25 ° c com agitação.
Nota: Reserve alguns meios de comunicação para servir como um espaço em branco para o OD600 medições antes de adicionar bactérias. - Medida OD600 cada hora até que atinja um OD entre 0.4 – 0.5. Neste ponto, reduza a temperatura a 16 ° c.
- Induzir com 0,2 mM IPTG (800 μL de 1 M de estoque), 10 min após a incubadora atinge 16 ° c. Incubar durante a noite (~ 15 – 18 h) a 16 ° c.
Nota: A temperatura mais baixa melhora o rendimento da proteína funcional reduzindo a agregação de atlastin. - Na manhã seguinte, colher as células por centrifugação em 7.500 x g a 4 ° c.
- Ressuscitem as células em 200 mL de A200 (25 mM HEPES (pH 7,4) e 200 mM KCl).
- Centrifugue as células a 11.000 x g durante 5 min a 4 ° c.
- Ressuscite o pellet em 40 mL de quebra tampão (A200 mais 10% glicerol, 2 mM 2-mercaptoethanol, 4% Triton X-100 (TX100), 40mM imidazol, e um EDTA-livre inibidor da protease cocktail comprimido).
Nota: Adicionar TX-100 depois de ressuscitarem para evitar a geração de bolhas e espuma. - Passe por uma agulha de 18 G e execute as células através de um disruptor de células três vezes em ~ 10.000 psi.
- Centrifugue o extrato em 125.000 x g por 1 h.
Nota: Opcionalmente dissolver a pelota 1:2 (w: v) em 8 M de ureia e nutate à temperatura ambiente durante a noite. A ureia como um desnaturante dissolverá lentamente qualquer proteína peletizada insolúvel para análise de SDS-PAGE. Excepto 1 μL para a análise da mancha de SDS-PAGE e de Coomassie. - Filtre o extrato através de um filtro de membrana estéril de nitrato de celulose de 0,45 μm para remover bactérias e grandes detritos bacterianos.
Nota: opcionalmente, excepto 1 μL para a análise da mancha do SDS PAGE e do Coomassie.
-
Purificação de proteínas por cromatografia de afinidade
- Carregue o lisado filtrado em uma coluna de resina de cromatografia de afinidade metálica imobilizada (IMAC) carregada com Ni2 +, pré-balanceada com baixo Tampão Imidazol (A100 mais 10% de glicerina, 2 mm 2-Mercaptoetanol, 1% TX-100, 40 mm imidazol) a uma taxa de 1 ml/ min a 4 ° c.
- Lave a coluna com 25 mL de A100 mais 10% de glicerol, 2 mM 2-Mercaptoetanol, 0,1% Anapoe X-100, 40 mM imidazol com uma taxa de 1 mL/min a 4 ° c. Elute a proteína com um gradiente linear de 30 mL de imidazol de 40 mM a 500 mM, e uma lavagem final de 5 mL a 500 mM.
- Piscina juntas de pico e incubar por 1 h a 4 ° c com grânulos inchados de GSH-agarose, previamente inchado em água e equilibrado em A100 com 10% de glicerol, 2 mM 2-Mercaptoetanol, 0,1% Anapoe X-100, e 1 mM EDTA.
Nota: Os grânulos de GSH-agarose podem ser inchados em 50 ml da água o dia antes e incubadas durante a noite em 4 ° c, ou para 1 h no RT. Para remover a água e o amortecedor do equilíbrio, centrifugue em um rotor de balanço da cubeta em 500 x g por 1 minuto sem freio. Aspirar o sobrenadante com uma agulha de 26 G. - Grânulos do GSH-agarose da pelota centrifugação em um rotor de balanço da cubeta em 500 x g sem freio e removem o sobrenadante do lisado pela aspiração com uma agulha de 26 g.
- Transfira as contas para uma coluna polyprep de 10 mL e lave cinco vezes com 5 mL de tampão de equilíbrio (A100 com 10% de glicerol, 2 mM 2-Mercaptoetanol, 0,1% de Anapoe X-100 e 1 mM de EDTA) por centrifugação a 500 X g.
- Elute a proteína com 1 – 1.5 ml de tampão do equilíbrio suplementado com a glutationa reduzida de 10 milímetros. Proteína aliquot eluída e congelamento de flash em nitrogênio líquido. A proteína pode ser armazenada a-80 ° c indefinidamente.
Nota: Ajuste o pH da reserva de eluição para 7,4. - Quantificar a concentração proteica pelo ensaio de proteína preta de amido9 e avaliar a pureza por SDS-PAGE e coloração de Coomassie.
2. reconstituição da Atlastina recombinante em lipossomas
- Liposome produção por método de extrusão 10 de
- Faça estoques de mistura lipídica em clorofórmio (10 mM de lipídio total). Os lipídios necessários são 1-Palmitoyl-2-oleoyl-glycero-3-fosfocolina (POPC), 1,2-dioleoyl-SN-glicero-3-fosho-L-serina (DOPS), 1,2-dioleoilo-SN-glicero-3-Fosfoetanolamina-N-(7-Nitro-2-1,3-benzoxadiazol-4-YL) (NBD-DPPE), e 1,2- dipalmitoyl-SN-glicero-3-Fosfoetanolamina-N-(lissamina rhodamina B sulfonyl) (RH-DPPE). Os lipossomas do Acceptor consistem em popc: DOPS (85:15 relação do molar) e lipossomas do doador de popc: DOPS: RH-PE: NBD-PE (83:15:1.5: relação do molar 1.5).
Nota: Embora as misturas lipídicas do POPC: DOPS tenham sido tradicionalmente usadas para ensaios de mistura lipídica in vitro, composições lipídicas alternativas podem ser adaptadas para diferentes finalidades experimentais. POPC: os lipossomas de DOPS são muito estáveis e mais duros de fundir, é conseqüentemente um sistema muito estrito para a fusão. - Adicione 1 μCi/mL de L-α-dipalmitoyl-Phosphatidylcholine (colina metil-3H) a misturas lipídicas, a fim de determinar as concentrações lipídicas em etapas posteriores por contagem de cintilação líquida. Reserve pelo menos 8 μL deste estoque.
- Transfira a quantidade desejada de misturas lipídicas para tubos de vidro de sílex.
- Seque as misturas lipídicas um fluxo suave de N2 gás para ~ 10 min até que não mais clorofórmio é visível.
- Seque o filme lipídico ainda mais em um dessecador aspirando por 30 min.
- Adicione o suficiente A100 aquosa com 10% de glicerol, 2 mM 2-Mercaptoetanol, e 1 mM de EDTA para o filme lipídico e trazer de volta a concentração para 10 mM. Resuspend o filme lipídico por vortexing levemente por 15 min à temperatura ambiente. Um vortexer que possa acomodar os tubos de vidro do sílex é recomendado.
- Congelar-degelo os lipídios hidratados em nitrogênio líquido dez vezes. Após o congelamento em nitrogênio líquido, descongelar os lipossomas, deixando a solução sentar-se à temperatura ambiente por 30 s, em seguida, transferir para a água para descongelamento mais rápido. Este ciclagem do Freeze-Thaw minimizará vesículas multilamelares.
Atenção: Os tubos podem rachar se contêm volumes maiores de 0,5 mL e se são transferidos diretamente dão forma ao nitrogênio líquido à água. - Passe o lipídio através de filtros de policarbonato com 100 nm poros tamanho 19 vezes usando mini-extrusora.
- Determinação da concentração lipídica total de lipossomas por contagem de cintilação
Nota: Alguns dos lipídios podem ser deixados para trás em tubos de vidro e em mini-extrusora.- Adicionar 4 μL de lipídeos e lipossomas em 3 mL de coquetel de cintilação.
- Medir contagens médias por minuto (CPMA) para estoque e lipossomas. E calcule a concentração dos lipossomas com a seguinte fórmula:
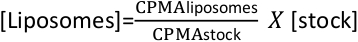
- Armazene os lipossomas a 4 ° c por até uma semana. Armazenamento mais longo não é recomendado como lipossomas podem agregar com o tempo, diminuindo a eficiência de reconstituição.
- Faça estoques de mistura lipídica em clorofórmio (10 mM de lipídio total). Os lipídios necessários são 1-Palmitoyl-2-oleoyl-glycero-3-fosfocolina (POPC), 1,2-dioleoyl-SN-glicero-3-fosho-L-serina (DOPS), 1,2-dioleoilo-SN-glicero-3-Fosfoetanolamina-N-(7-Nitro-2-1,3-benzoxadiazol-4-YL) (NBD-DPPE), e 1,2- dipalmitoyl-SN-glicero-3-Fosfoetanolamina-N-(lissamina rhodamina B sulfonyl) (RH-DPPE). Os lipossomas do Acceptor consistem em popc: DOPS (85:15 relação do molar) e lipossomas do doador de popc: DOPS: RH-PE: NBD-PE (83:15:1.5: relação do molar 1.5).
- Reconstituição por incorporação assistida por detergente em lipossomas pré-formados
- Calcule a quantidade de tampão, proteína, lipossomas e detergente extra a ser misturado junto:
- Determine o volume total desejado. Este volume é constituído por tampão A100 com glicerol a 10%, 2 mM 2-Mercaptoetanol e 1 mM de EDTA. Os volumes inferiores a 250 μL não se misturam bem em tubos de microcentrífuga de 0,5 mL.
- Calcule o volume de lipossomas necessários para dar uma concentração lipídica final de cerca de 1 mM. Subtrair o volume do volume do buffer.
- Calcule o volume de proteína necessária para dar a proteína desejada para a relação molar lipídico (geralmente 1:400). Diminua o volume do buffer de acordo.
- Determine quanto detergente extra precisa ser adicionado para saturar os lipossomas visando um detergente efetivo para a razão lipídica (REFF) entre 0,64 – 0,8. Lembre-se de considerar o detergente que é adicionado com a proteína. O (REFF) é determinado pela
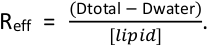 equação dtotal é a concentração de detergente total e dágua é a concentração de detergente monomérico (0,18 mm para TX-100 e anapoe X-100 na presença de detergente)4, o 11.
equação dtotal é a concentração de detergente total e dágua é a concentração de detergente monomérico (0,18 mm para TX-100 e anapoe X-100 na presença de detergente)4, o 11.
- Misture as soluções juntas em um tubo de 0,5 mL na seguinte ordem: tampão, detergente, proteína e lipossomas. Adicione os lipossomas rapidamente e vórtice imediatamente para 5 s para homogeneizar a mistura.
- Incubar a reacção num pulverizador durante 1 h a 4 ° c.
- Fazer 0,2 g/mL poliestireno não polar adsorvente talão "chorume" na água.
Nota: Faça o grânulo adsorvente do poliestireno "pasta" durante precedente a incubação de 1 h na etapa 2.2.3. - Pesar 0,2 g de grânulos adsorvente de poliestireno e transferir para um tubo de microcentrífuga.
- Degas os grânulos adicionando 1 mL de metanol ao tubo e misture por 1 min. os grânulos desgaseificados afundarão.
- Aspirar o metanol e adicionar água aos grânulos. Deixe os grânulos misturar com água por 5 min, em seguida, aspirar a água. Repita quatro vezes, em seguida, trazer o poliestireno adsorvente talão "chorume" para 1 mL de volume com água e uma concentração final de 0,2 g/mL.
Nota: Os grânulos devem ainda assentar à parte inferior do tubo. Se não o fizerem, Degas novamente. Use uma agulha de 21 G ou superior para aspirar metanol e água a partir de grânulos. - Calcule a quantidade de grânulos adsorvente do poliestireno necessários para absorver todo o detergente em cada amostra. 1 g de grânulos adsorvente de poliestireno absorve 70 mg de TX-100. Para calcular o volume de polpa de grânulo adsorvente de poliestireno necessário para cada reação, divida o detergente total na reação (Step 2.2.1.4) por 70 mg e, em seguida, pela concentração da polpa do cordão (0,2 g/mL (passo 2.2.7).
- Transfira a quantidade calculada de pasta de grânulo adsorvente de poliestireno para um tubo de 0,5 mL e aspirar a água.
Nota: Corte a extremidade de uma ponta de 20 – 200 μL para transferir os grânulos, vortex o tubo apenas antes da pipetagem para ressuscitar grânulos liquidados. - Adicione as amostras ao tubo de 0,5 mL contendo grânulos adsorvente de poliestireno e incubar a amostra de nutating por 1 h a 4 ° c.
- Repita duas vezes deixando os grânulos velhos atrás e transferindo a amostra para grânulos frescos.
- Adicione a amostra a um quarto tubo com grânulos frescos e incubar durante a noite (~ 15 – 18 h) a 4 ° c.
- Calcule a quantidade de tampão, proteína, lipossomas e detergente extra a ser misturado junto:
- De manhã, retire a amostra de grânulos adsorvente de poliestireno e granulados de proteína insolúveis por centrifugação por 10 min a 16.000 x g a 4 ° c.
- Recupere o sobrenadante e determine a concentração lipídica final por contagem de cintilação líquida (ver passo 2.1.9.2). Opcionalmente, a concentração proteica pode ser determinada pelo ensaio de proteína preta de amido9.
Nota: Para os proteoliposomas atlastin, os ensaios enzimáticos devem ser realizados com lipossomas frescos. O armazenamento longo a 4 ° c ou a congelação conduzem à perda significativa na atividade do atlastin.
3. ensaios de mistura de lipídios
- Traga os proteoliposomas do doador e do aceitador a uma concentração de 0,15 milímetros cada em A100 com o glicerol de 10%, o β-mercaptoethanol de 2 milímetros, o EDTA de 1 milímetro e o MgCl2de 5 milímetros. Cada reação (50 μL) deve ser adicionada a um poço em uma placa branca lisa do poço 96 apropriada para leituras da fluorescência. Prepare pelo menos 4 reações, incluindo um triplicado, e um controle negativo no-GTP.
- Coloque a placa num leitor de chapa pré-aquecido a 37 ° c. Medida de fluorescência NBD (excitação 460 nm e emissão 535 Nm) para 5 min a cada minuto.
- Induzir a fusão adicionando 5 mM GTP (5 μL de 50 mM GTP).
- Medir a fluorescência de NBD a cada minuto durante 1 h.
- Adicionar 5 μL de 2,5% w/v n-Dodecyl β-D-maltoside para dissolver os proteoliposomas e medir a fluorescência máxima de NBD. Leia a fluorescência de NBD por 15 minutos cada minuto.
4. liposome FLOATATION em gradiente descontínuo de Iohexol12
- opcional Analisar a eficiência da reconstituição por ensaios de floatação13.
- Prepare um 80% e um iohexol de 30% w/v em A100 com 10% de glicerol, 2 mM 2-Mercaptoetanol e 1 mM de EDTA. Iohexol não se dissolve prontamente, então isso deve ser preparado um dia antes por nutating durante a noite a 4 ° c.
- Misture completamente 150 μL de 80% de estoque de iohexol com 150 μL de amostra de proteoliposomas para trazê-lo para um iohexol de 40%. Adicione isto a um tubo de 5 x 41 mm2 Ultra-Clear evitando bolhas.
- Camada 250 μL de estoque de iohexol a 30% lentamente em cima da amostra para fazer uma camada intermediária. Evite quaisquer bolhas e perturbar a camada inferior. No topo da camada média, adicione lentamente 50 μL de A100 com 2 mM 2-Mercaptoetanol e 1 mM de EDTA.
- Centrifugue o gradiente em um rotor de balde oscilante em 220.000 x g por 4 h a 4 ° c com aceleração lenta e sem ruptura.
- Colher as camadas do gradiente e analisar pela mancha SDS-PAGE e Coomassie, a quantificação pode ser feita por densitometria. A proteína reconstituída deve flutuar para a camada superior, enquanto agregados de proteínas e lipídios vão sedimento na parte inferior ou na camada intermediária.
5. análise da orientação da proteína reconstituída por proteólise de trombina
Nota: O construto atlastin relatado aqui tem um local de corte de trombina entre o final da marca N-terminal GST e o início da atlastin. Atlastin na orientação correta terá este local de corte acessível à protease, enquanto a proteína na orientação errada será protegida pela bicamada lipídica.
- Para o ensaio de orientação de atlastin, Reserve pelo menos 8 μL de proteoliposomas reconstituídos frescos.
- Adicionar 8 μL de proteoliposomas e 1 μL de 1 U/μL de trombina. Incubar a 37 ° c por 1 h.
- Saciar a protease com 1 μL de 5 mg/mL de coquetel inibidor de protease livre de EDTA e incubar por 30 min a 37 ° c.
- Analise a amostra pela mancha de SDS-PAGE e de Coomassie. A proporção de proteína clivada e uncleaved pode ser determinada pela densitometria.
Resultados
A eficiência da reconstituição da atlastina é apresentada na Figura 2. Os sobreviventes reconstituídos foram flutuados em um inclinação descontínuo do iohexol. A proteína não incorporada foi sedimentado na camada inferior (B) ou na camada média (M). A proteína reconstituída flutuaria para a camada superior (T). Amostras do gradiente foram colhidas e analisadas pela coloração de SDS-PAGE e Coomassie. A quantificação do gel pela densitometria mostra uma eficiência muito eleva...
Discussão
Os métodos aqui delineiam um método eficiente para purificar, reconstituir e medir a atividade de fusão da atlastina recombinante. Para assegurar rendimentos elevados do atlastin funcional algumas etapas críticas devem ser consideradas. A expressão da atlastina deve ser feita a baixas temperaturas (16 ° c) para evitar a agregação e uma deve apontar para uma concentração final entre 0,4 – 1,5 mg/mL. A proteína muito diluida não será reconstituída de forma otimizada numa proporção de proteína 1:400 a lip...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos ao Dr. Michael Stern e seu laboratório por seus insights e feedback sobre os projetos relacionados à atlastin. Este trabalho foi apoiado pelo Instituto Nacional de ciências médicas gerais [R01GM101377] e pelo Instituto Nacional de distúrbios neurológicos e AVC [R01NS102676].
Materiais
| Name | Company | Catalog Number | Comments |
| 10 mL poly-prep chromatography columns | Biored | 731-1550 | |
| 10 x 75 mm Flint glass tubes | VWR | 608225-402 | |
| 47 mm diameter, 0.45um pore whatman sterile membrane filters | Whatman | 7141 104 | |
| 96 well white plate | NUNC | 437796 | |
| Anapoe X-100 | Anatrace | 9002-931-1 | |
| Cell disrupter | Avestin | Avestin Emulsiflex C3 | |
| DOPS (1,2-dioleoyl-sn-glycero-3-phospho-L-serine (sodium salt)) | Avanti | 840035P-10mg | DOPS |
| EDTA | Research organics inc. | 6381-92-6 | Ethylenediaminetetraacetic acid |
| EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Complete protease inhibitor |
| Extruder | Sigma Aldrich | Z373400 | Liposofast Basic Extruder |
| GE Akta Prime liquid chromatography system | GE Pharmacia | 8149-30-0006 | |
| Glutathione agarose beads | Sigma aldrich | G4510-50ml | |
| Glycerol | EMD | GX0185-5 | |
| GTP | Sigma Aldrich | 36051-31-7 | Guanosine 5' triphosphate sodium salt hydrate |
| HEPES, acid free | Omnipur | 5330 | |
| Imidazole | fluka | 5670 | |
| Immobilized metal affinity chromatography (IMAC) resin column | GE Healthcare | 17040801 | 1 mL HiTrap Chelating HP immobilized metal affinity chromatography columns |
| Iohexol | Accurate chemical and scientific corporation | AN 7050 BLK | Accudenz/Nycodenz |
| IPTG | Research products international corp. | I56000-100.0 | IPTG, dioxane free |
| L-Glutathione reduced | Sigma-Aldrich | G4251-5g | |
| Magnesium chloride | Fisher | 7791-18-6 | |
| Methanol | Omnisolv | MX0488-1 | |
| NBD-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl) (ammonium salt)) | Avanti | 236495 | NBD-DPPE |
| n-Dodecyl β-D-maltoside | Chem-Impex International | 21950 | |
| Nonpolar polystyrene adsorbent beads | BioRad | 152-3920 | SM2 Biobeads |
| Nuclepore track-etch polycarbonate 19 mm 0.1 um pore membrane | Whatman | 800309 | |
| Optima LE80K Ultra centrifuge | Beckman Coulter | ||
| Phosphatidylcholine, L-α-dipalmitoyl [choline methyl-3H] | ARC | ART0284 | Titriated lipids |
| Plate reader | TECAN | TECAN infinite M200 plate reader | |
| POPC (1-palmitoyl-2-oleoyl-glycero-3-phosphocholine) | Avanti | 850457C-25mg | POPC |
| Potassium chloride | MP | 151944 | |
| Rh-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(lissamine rhodamine B sulfonyl) (ammonium salt)) | Avanti | 236495 | Rh-DPPE |
| Scintillation Cocktail | National Diagnostics | LS-272 | Ecoscint XR Scintillation solution for aqueous or non-aqueous samples |
| Scintillation vials | Beckman | 592928 | Fast turn cap Mini Poly-Q Vial |
| Thrombin | Sigma | T1063-1kU | Thrombin from human plasma |
| Triton X-100 | Fisher | BP151-500 | |
| Ultra-clear centrifuge tubes 5 x 41 mm | Beckman | 344090 | |
| Vortex 9 to 13mm Tube Holder | VWR | 58816-138 | Insert for vortexing flint glass tubes |
| Vortex Insert Retainer | VWR | 58816-132 | Retainer needed for vortex tube holder |
| Vortexer | VWR | 2.235074 | Vortex Genie 2 model G560 |
| β-mercaptoethanol molecular biology grade | Calbiochem | 444203 |
Referências
- Chernomordik, L. V., Kozlov, M. M. Mechanics of membrane fusion. Nature Structural and Molecular Biology. 15, 675-683 (2008).
- Orso, G., et al. Homotypic fusion of ER membranes requires the dynamin-like GTPase Atlastin. Nature. 460, 978-983 (2009).
- McNew, J. A., Sondermann, H., Lee, T., Stern, M., Brandizzi, F. GTP-dependent membrane fusion. Annual Review of Cell and Developmental Biology. 29, 529-550 (2013).
- Rigaud, J. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1231, 223-246 (1995).
- Düzgüneş, N. Fluorescence Assays for Liposome Fusion. Methods in Enzymology. 372, 260-274 (2003).
- Wilschut, J., Duzgunes, N., Fraley, R., Papahadjopoulos, D. Studies on the mechanism of membrane fusion: kinetics of calcium ion induced fusion of phosphatidylserine vesicles followed by a new assay for mixing of aqueous vesicle contents. Biochemistry. 19, 6011-6021 (1980).
- Ellens, H., Bentz, J., Szoka, F. C. Proton- and calcium-induced fusion and destabilization of liposomes. Biochemistry. 24, 3099-3106 (1985).
- Meers, P., Ali, S., Erukulla, R., Janoff, A. S. Novel inner monolayer fusion assays reveal differential monolayer mixing associated with cation-dependent membrane fusion. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1467, 277-243 (2000).
- Schaffner, W., Weissmann, C. A rapid, sensitive, and specific method for the determination of protein in dilute solution. Analytical Biochemistry. 56, 502-514 (1973).
- MacDonald, R. C., et al. Small-volume extrusion apparatus for preparation of large, unilamellar vesicles. Biochimica Et Biophysica Acta. 1061, 297-303 (1991).
- Paternostre, M. T., Roux, M., Rigaud, J. L. Mechanisms of membrane protein insertion into liposomes during reconstitution procedures involving the use of detergents. 1. Solubilization of large unilamellar liposomes (prepared by reverse-phase evaporation) by Triton X-100, octyl glucoside, and sodium cholate. Biochemistry. 27, 2668-2677 (1988).
- Scott, B. L., et al. Liposome Fusion Assay to Monitor Intracellular Membrane Fusion Machines. Methods in Enzymology. 372, 274-300 (2003).
- Zhang, W., et al. Crystal structure of an orthomyxovirus matrix protein reveals mechanisms for self-polymerization and membrane association. PNAS. 114, 8550-8555 (2017).
- Parlati, F., et al. Rapid and efficient fusion of phospholipid vesicles by the α-helical core of a SNARE complex in the absence of an N-terminal regulatory domain. PNAS. 96, 12565-12570 (1999).
- Sugiura, S., Mima, J. Physiological lipid composition is vital for homotypic ER membrane fusion mediated by the dynamin-related GTPase Sey1p. Scientific Reports. 6, 20407 (2016).
- Powers, R. E., Wang, S., Liu, T. Y., Rapoport, T. A. Reconstitution of the tubular endoplasmic reticulum network with purified components. Nature. 543, 257-260 (2017).
- Betancourt-Solis, M. A., Desai, T., McNew, J. A. The atlastin membrane anchor forms an intramembrane hairpin that does not span the phospholipid bilayer. Journal of Biological Chemistry. 293, 18514-18524 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados