Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Reconstitution assistée par détergent de Recombinant Drosophila Atlastin en Liposomes pour des essais de mélange de lipides
Dans cet article
Résumé
La fusion des membranes biologiques est catalysée par des protéines de fusion spécialisées. La mesure des propriétés fusogéniques des protéines peut être obtenue par des essais de mélange de lipides. Nous présentons une méthode pour purifier l'atlastine recombinante de Drosophila, une protéine qui médiatise la fusion homotypique de l'ER, la reconstituant aux liposomes préformés, et testant la capacité de fusion.
Résumé
La fusion de membrane est un processus crucial dans la cellule eucaryotique. Des protéines spécialisées sont nécessaires pour catalyser la fusion. Les atlastines sont des protéines réticulum-endoplasmiques (ER) impliquées dans la fusion homotypique de l'ER. Nous détaillons ici une méthode pour purifier un glutathion S-transferase (GST) et poly-histidine marqué Drosophila atlastin par deux séries de chromatographie d'affinité. L'étude des réactions de fusion in vitro nécessite l'insertion de protéines de fusion purifiées dans une couche lipidique. Les liposomes sont des membranes modèles idéales, car la composition et la taille des lipides peuvent être ajustées. À cette fin, nous décrivons une méthode de reconstitution par l'enlèvement de détergent pour drosophila atlastin dans les liposomes préformés. Tandis que plusieurs méthodes de reconstitution sont disponibles, la reconstitution par l'enlèvement de détergent a plusieurs avantages qui le rendent approprié pour des atlastins et d'autres protéines semblables. L'avantage de cette méthode comprend un rendement de reconstitution élevé et une orientation correcte de la protéine reconstituée. Cette méthode peut être étendue à d'autres protéines membranaires et à d'autres applications qui nécessitent des protéoliposomes. En outre, nous décrivons un test de mélange de lipides basé sur FRET des protéoliposomes utilisés comme mesure de la fusion de membrane.
Introduction
La fusion de membrane est un processus critique dans beaucoup de réactions biologiques. Dans des conditions biologiques, la fusion de la membrane n'est pas spontanée et nécessite des protéines de fusion spécialisées pour catalyser ces réactions1. ER fusion de membrane homotypic est médiée chez les animaux par le dynamin liés GTPase atlastin2. Le rôle d'Atlastin dans la fusion homotypique est fondamental pour les jonctions à trois voies dans les urgences périphériques, qui constituent un grand réseau interconnecté de tubules qui s'étendent dans toute la cellule. Les atlastines ont une morphologie de domaine conservée composée d'un grand GTPase, d'un domaine intermédiaire de faisceau detrois hélices, d'une ancre hydrophobe de membrane, et d'une courte queue Cytoplasmique C-terminal 3. Des études in vitro avec recombinant Drosophila atlastin ont montré que lorsqu'il est reconstitué en liposomes, il maintient ses propriétés fusogéniques. D'autres atlastines, y compris les homologs humains n'ont pas été en mesure de récapituler la fusion in vitro. Nous décrivons ici une méthodologie pour purifier une TPS et poly-histidine étiquetée recombinante Drosophila atlastin, la reconstituer en liposomes, et d'essayer la fusion.
L'étude de la fusion de membrane in vitro présente un défi car les protéines fusogéniques ont habituellement une ancre de membrane. Pour les étudier, il est nécessaire de les reconstituer en bicouches lipidiques modèles. Les grandes vésicules unilamellaires (LUV) sont un outil utile pour étudier les interactions entre protéines lipidiques. Nous présentons ici un système pour faire des VUS de différentes compositions lipidiques pour la reconstitution des protéines et les analyses de fusion. La reconstitution des protéines intégrales en JV peut être réalisée par une variété de méthodes, y compris,la reconstitution organique de solvant-négocié, les mécanismes mécaniques, ou la reconstitution assistée de détergent 4. Nous présentons ici une méthode pour reconstituer d'atlastin de Drosophila en liposomes préformés par l'enlèvement de détergent. Les avantages de cette méthode de reconstitution incluent des rendements élevés de reconstitution et l'orientation appropriée de l'atlastine dans la bicouche de lipide. En outre, grâce à cette méthode, la protéine n'est pas séchée ou exposée à des solvants organiques, maintenant ainsi la structure et la fonction. Parmi ses inconvénients, la présence de détergents peut ne pas être idéal pour toutes les protéines et les protéoliposomes finaux peuvent avoir un certain détergent incorporé dans la bicouche lipidique. D'autres dialyses peuvent être utilisées pour éliminer davantage le détergent. Cependant, la dialyse peut prendre beaucoup de temps et peut donc entraîner une perte d'activité protéique.
L'évaluation de l'activité de fusion de l'atlastine peut être déterminée par des essais de mélange de lipides comme décrit précédemment2. Ici, nous délinons une méthode pour mesurer la fusion médiée par N-(7-nitrobenz-2-oxa-1,3-diazol-4-yl (NBD)/Lissamine rhodamine-B sulfonyl (rhodamine) lipides étiquetés. Cet test nécessite la fusion de protéoliposomes donneurs (étiquetés) et de protéoliposomes accepteurs (non étiquetés). Une libération de FRET peut être mesurée au cours de la réaction comme la dilution d'une paire donneur-acceptant de liposomes « étiquetés » à des liposomes « non étiquetés » à la suite du mélange de lipides pendant la fusion de membrane (figure 1)5. Bien que cet assay serve de proxy pour la fusion de membrane, il est limité dans la distinction entre la fusion de membrane et l'hémifusion, un état où seuls les folioles extérieures se mélangent. Pour résoudre ce problème, une alternative est l'étanchéité externe de la DNB par la dithionite. Suivant la même méthodologie que NBD/rhodamine lipides mélangeant des essais, lors de l'étanchéité du dépliant externe toute libération NBD FRET par fusion sera due à la foliole interne mélange8.
Des essais alternatifs de fusion par le contenu aqueux intérieur mélangeant l'adresse pleine fusion seulement5. Des exemples de ceci sont le terbium (Tb)/acide dipicolinique (DPA) essais et l'acide trisulfonic aminonaphthalene (ANTS)/p-xylène bis (pyridinium) bromure (DPX) essais. Dans tb/DPA, un pool de liposomes avec tb encapsulé sont mélangés et fusionnés avec des liposomes avec dPA encapsulé; lors de la fusion, la fluorescence est augmentée par transfert d'énergie interne de DPA à Tb dans le [Tb(DPA)3]3- chelation complexe6. En revanche, pour les essais ANTS/DPX, la fluorescence ANTS est étanchée par DPX7. Bien que ces systèmes traitent du mélange de contenu interne, une préparation plus approfondie des liposomes est nécessaire pour l'élimination des réactifs non encapsulés, ainsi que l'interaction involontaire des fluorophores.
Protocole
1. Purification de la TPS-DAtl-His8
-
Expression protéique et préparation du lysate
- Transformez BL21 (DE3) E. coli avec la construction GST-DAtl-His8 en pGEX4-T32 et sélectionnez sur une plaque d'ampicilline.
- Sélectionnez un seul transformateur et inoculez 5 ml d'ampicilline LB (5 l d'ampicilline de 100 mg/mL) dans un tube de culture de 14 ml et incubez à 25 oC en secouant à 200 tr/min pendant 6 à 8 h.
REMARQUE: En raison de l'expression qui fuit, il n'est pas recommandé d'incuber à des températures plus élevées. La croissance à 25 oC réduit l'agrégation des protéines pendant cette période de croissance. - Inoculer 200 ml d'ampicilline lb-est avec 1 ml de culture de 5 ml et incuber pendant la nuit (15 à 18 h) à 25 oC.
- Le lendemain matin, récolter les cellules par centrifugation (2 000 x g pendant 10 min) et resuspendre en 5 ml de LB.
- Inoculer 4 L de LB et de l'ampicilline et mesurer OD600 (0,05 à 0,15). Incuber à 25 oC en secouant.
REMARQUE: Réservez quelques supports pour servir de blanc pour les mesures OD600 avant d'ajouter des bactéries. - Mesurez600 OD toutes les heures jusqu'à ce qu'il atteigne une OD entre 0.4-0.5. À ce stade, réduire la température à 16 oC.
- Induire avec 0,2 mM d'IPTG (800 oL de 1 M de stock), 10 min après que l'incubateur a atteint 16 oC. Incuber toute la nuit (15 à 18 h) à 16 oC.
REMARQUE: La température inférieure améliore le rendement des protéines fonctionnelles en réduisant l'agrégation de l'atlastine. - Le lendemain matin, récoltez les cellules en centrifugeant à 7 500 x g à 4 oC.
- Resuspendre les cellules dans 200 ml d'A200 (25 mM HEPES (pH 7,4) et 200 mM KCl).
- Centrifuger les cellules à 11 000 x g pendant 5 min à 4 oC.
- Resuspendre la pastille en 40 ml de tampon de rupture (A200 plus 10% de glycérol, 2 mM 2-mercaptoethanol, 4% Triton X-100 (TX100), 40mM imidazole, et un comprimé de cocktail inhibiteur de protéase sans EDTA).
REMARQUE: Ajouter le TX-100 après le ressurcage pour éviter de générer des bulles et de la mousse. - Passer à travers une aiguille de 18 G et faire passer les cellules à travers un perturbateur cellulaire trois fois à 10 000 psi.
- Centrifuger l'extrait à 125 000 x g pour 1 h.
REMARQUE: Dissoudre le granule 1:2 (w:v) dans 8 M Urea et nutate à température ambiante pendant la nuit. L'urée en tant que dénaturant dissoudra lentement toute protéine pastille insoluble pour l'analyse SDS-PAGE. Enregistrer 1 L pour l'analyse des taches SDS-PAGE et Coomassie. - Filtrer l'extrait à travers un filtre à membrane stérile de nitrate de cellulose de 0,45 m pour éliminer les bactéries et les gros débris bactériens.
REMARQUE : En option, enregistrez 1 L pour l'analyse des taches SDS PAGE et Coomassie.
-
Purification des protéines par chromatographie d'affinité
- Chargez le lysate filtré sur une chromatographie d'affinité métallique immobilisée (IMAC) chargée de Ni2,pré-équilibré avec un tampon faible en imidazole (A100 plus 10% de glycérol, 2 mM 2-mercaptoethanol, 1% TX-100, 40 mM imidazole) à un taux de 1 mL/ min à 4 oC.
- Laver la colonne avec 25 mL d'A100 plus 10% de glycérol, 2 mM 2-mercaptoethanol, 0,1% Anapoe X-100, 40 mM imidazole avec un taux de 1 ml/min à 4 oC. Élichez la protéine avec un gradient linéaire de 30 ml d'imidazole de 40 mm à 500 mM, et un lavage final de 5 ml à 500 mm.
- Mettre en commun les fractions de pointe et incuber pendant 1 h à 4 oC avec des perles gonflées de GSH-Agarose, auparavant gonflées dans l'eau et liblibées en A100 avec 10 % de glycérol, 2 mM 2-mercaptoéthanol, 0,1 % Anapoe X-100 et 1 mM EDTA.
REMARQUE: Les perles de GSH-Agarose peuvent être gonflées dans 50 ml d'eau la veille et incubées pendant la nuit à 4 oC, ou pour 1 h à RT. Pour enlever l'eau et le tampon d'équilibre, centrifugeuse dans un rotor de seau oscillant à 500 x g pendant 1 min sans frein. Aspirez le supernatant avec une aiguille de 26 G. - Pellet GSH-Agarose perles en centrifuge dans un rotor de seau oscillant à 500 x g sans frein et enlever le lysate supernatant par aspiration avec une aiguille de 26 G.
- Transférer les perles dans une colonne polypréparatoire de 10 ml et laver cinq fois avec 5 mL de tampon d'équilibre (A100 avec 10 % de glycérol, 2 mM 2-mercaptoéthanol, 0,1 % Anapoe X-100 et 1 mM EDTA) en centrifuant à 500 x g.
- Éluter la protéine avec 1-1.5 ml de tampon d'équilibre complété avec 10 mM de glutathion réduit. Aliquot a éludé les protéines et le gel éclair dans l'azote liquide. Les protéines peuvent être stockées à -80 oC indéfiniment.
REMARQUE: Ajuster le pH du tampon d'élution à 7,4. - Quantifiez la concentration de protéines par l'amido test de protéines noires9 et évaluez la pureté par la coloration SDS-PAGE et Coomassie.
2. Reconstitution de recombinant Atlastin en Liposomes
- Production de Liposome par méthode d'extrusion 10 Ans et plus
- Faire des stocks de mélange lipidique dans le chloroforme (10 mM lipidique total). Les lipides nécessaires sont 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC), 1,2-dioleoyl-sn-glycero-3-phospho-L-serine (DOPS), 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl) (NBD-DPPE), et 1,2- dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(lissamine rhodamine B sulfonyl) (Rh-DPPE). Les liposomes d'accepteur se composent de POPC : DOPS (85:15 molaire ratio) et liposomes de donneur de POPC:DOPS:RH-PE:NBD-PE (83:15:1.5:1.5 molaire ratio).
REMARQUE: Tandis que les mélanges de lipides de POPC:DOPS ont été traditionnellement employés pour des essais in vitro de mélange de lipide, les compositions alternatives de lipide peuvent être adaptées pour différentes fins expérimentales. POPC:DOPS liposomes sont très stables et plus difficiles à fusionner, est donc un système très strict pour la fusion. - Ajoutez 1 'Ci/mL de L-dipalmitoyl-phosphatidylcholine (méthylle choline-3H) aux mélanges lipidiques afin de déterminer les concentrations lipidiques à des étapes ultérieures par comptage de scintillation liquide. Réserver au moins 8 ll de ce stock.
- Transférer la quantité désirée de mélanges lipidiques dans des tubes en verre de silex.
- Séchez les mélanges lipidiques sous un flux doux de gaz N2 pendant 10 min jusqu'à ce qu'aucun chloroforme ne soit visible.
- Séchez le film lipidique plus loin dans un dessiccateur en passant l'aspirateur pendant 30 min.
- Ajoutez suffisamment d'A100 aqueuse avec 10 % de glycérol, 2 mM 2-mercaptoethanol et 1 mM EDTA au film lipidique et ramenez la concentration à 10 mM. Resuspendre le film lipidique en vortexant légèrement pendant 15 min à température ambiante. Un vortexer qui peut accueillir des tubes en verre de silex est recommandé.
- Congeler-dégel les lipides hydratés dans l'azote liquide dix fois. Après congélation dans l'azote liquide, décongeler les liposomes en laissant la solution s'asseoir à température ambiante pendant 30 s, puis transférer à l'eau pour une décongélation plus rapide. Ce cycle de gel-dégel minimisera les vésicules multilamellar.
MISE EN GARDE: Les tubes peuvent se fissurer s'ils contiennent des volumes supérieurs à 0,5 ml et s'ils sont transférés directement sous forme d'azote liquide dans l'eau. - Passer le lipide à travers des filtres en polycarbonate avec 100 nm pore taille 19 fois en utilisant mini-extrudeur.
- Déterminer la concentration totale de lipides des liposomes par comptage de scintillation
REMARQUE: Une partie du lipide peut être laissée dans des tubes de verre et dans le mini-extrudeur.- Ajouter 4 ll de stock lipidique et de liposomes dans 3 ml de cocktail de scintillation.
- Mesurer le nombre moyen par minute (CPMA) pour les stocks et les liposomes. Et calculer la concentration de liposomes avec la formule suivante:
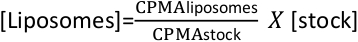
- Conserver les liposomes à 4 oC jusqu'à une semaine. Le stockage plus long n'est pas recommandé car les liposomes peuvent s'agréger avec le temps, diminuant ainsi l'efficacité de la reconstitution.
- Faire des stocks de mélange lipidique dans le chloroforme (10 mM lipidique total). Les lipides nécessaires sont 1-palmitoyl-2-oleoyl-glycero-3-phosphocholine (POPC), 1,2-dioleoyl-sn-glycero-3-phospho-L-serine (DOPS), 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl) (NBD-DPPE), et 1,2- dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(lissamine rhodamine B sulfonyl) (Rh-DPPE). Les liposomes d'accepteur se composent de POPC : DOPS (85:15 molaire ratio) et liposomes de donneur de POPC:DOPS:RH-PE:NBD-PE (83:15:1.5:1.5 molaire ratio).
- Reconstitution par détergent assisté incorporation en liposomes préformés
- Calculez la quantité de tampon, de protéines, de liposomes et de détergent supplémentaire à mélanger :
- Déterminer le volume total souhaité. Ce volume est composé de tampon A100 avec 10% de glycérol, 2 mM 2-mercaptoethanol, et 1 mM EDTA. Les volumes de moins de 250 l ne se mélangent pas bien dans des tubes microcentrifuges de 0,5 ml.
- Calculer le volume de liposomes nécessaires pour donner une concentration lipidique finale d'environ 1 mM. Soustrayez le volume du volume tampon.
- Calculer le volume de protéines nécessaires pour donner le rapport protéine/molaire lipidique souhaité (habituellement 1:400). Diminuez le volume de mémoire tampon en conséquence.
- Déterminer la quantité de détergent supplémentaire à ajouter pour saturer les liposomes visant un ratio détergent/lipide efficace (Reff) entre 0,64 et 0,8. N'oubliez pas de considérer le détergent qui est ajouté avec la protéine. Le (Reff) est
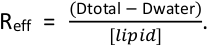 déterminé par l'équation Dtotal est la concentration totale de détergent et l'eau D est la concentration de détergent monomeric (0,18 mM pour TX-100 et Anapoe X-100 en présence de détergent)4 , 11.
déterminé par l'équation Dtotal est la concentration totale de détergent et l'eau D est la concentration de détergent monomeric (0,18 mM pour TX-100 et Anapoe X-100 en présence de détergent)4 , 11.
- Mélanger les solutions dans un tube de 0,5 ml dans l'ordre suivant : tampon, détergent, protéines et liposomes. Ajouter les liposomes rapidement et vortex immédiatement pour 5 s pour homogénéiser le mélange.
- Incuber la réaction dans un nutator pendant 1 h à 4 oC.
- Faire 0,2 g/mL de lédre en polystyrène non polaire dans l'eau.
REMARQUE: Faire la perle adsorbent en polystyrène " loue" au cours de l'incubation précédente de 1 h à l'étape 2.2.3. - Peser 0,2 g de perles adsorbent en polystyrène et transférer dans un tube de microcentrifuge.
- Dégazer les perles en ajoutant 1 ml de méthanol au tube et mélanger pendant 1 min. Les perles de degassed couleront.
- Aspirer le méthanol et ajouter de l'eau aux perles. Laisser les perles se mélanger avec de l'eau pendant 5 min, puis aspirer l'eau. Répétez quatre fois, puis apportez la perle adsorbent en polystyrène "slurry" à 1 ml de volume avec de l'eau et une concentration finale de 0,2 g/mL.
REMARQUE: Les perles doivent encore s'installer au fond du tube. S'ils ne le font pas, dégazez à nouveau. Utilisez une aiguille de 21 G ou plus pour aspirer le méthanol et l'eau des perles. - Calculez la quantité de perles adsorbent en polystyrène nécessaires pour absorber tous les détergents de chaque échantillon. 1 g de perles adsorbent en polystyrène absorbe 70 mg de TX-100. Pour calculer le volume de boue de perles adsorbent en polystyrène nécessaire pour chaque réaction, divisez le détergent total dans la réaction (étape 2.2.1.4) par 70 mg, puis par la concentration de la boue de perle (0.2 g/mL (étape 2.2.7).
- Transférer la quantité calculée de lisière de perles en polystyrène dans un tube de 0,5 ml et aspirer l'eau.
REMARQUE: Couper l'extrémité d'une pointe de 20 à 200 l pour transférer les perles, vortex le tube juste avant le tuyauterie pour resuspendre les perles réglées. - Ajouter les échantillons au tube de 0,5 ml contenant des perles adsorbent en polystyrène et incuber l'échantillon en écroupendant pendant 1 h à 4 oC.
- Répétez deux fois en laissant de vieilles perles derrière et en transférant l'échantillon à des perles fraîches.
- Ajouter l'échantillon à un quatrième tube avec des perles fraîches et incuber toute la nuit (15 à 18 h) à 4 oC.
- Calculez la quantité de tampon, de protéines, de liposomes et de détergent supplémentaire à mélanger :
- Le matin, retirer l'échantillon des perles d'adsorbent en polystyrène et des agrégats de protéines insolubles en centrifuge pendant 10 min à 16 000 x g à 4 oC.
- Récupérer le supernatant et déterminer la concentration lipidique finale par comptage de scintillation liquide (voir l'étape 2.1.9.2). Optionnellement, la concentration de protéine peutêtre déterminée par l'assay noir de protéine amido 9.
REMARQUE: Pour les protéoliposomes atlastin, les essais enzymatiques doivent être exécutés avec des liposomes frais. Un stockage long à 4 oC ou le gel entraîne une perte importante d'activité d'atlastine.
3. Tests de mélange de lipides
- Amener les protéoliposomes donneurs et accepteurs à une concentration de 0,15 mM chacun en A100 avec 10 % de glycérol, 2 mM d'éthanol, 1 mM EDTA et 5 mM MgCl2. Chaque réaction (50 l) doit être ajoutée à un puits dans une plaque de puits blanche plate de 96 propice aux lectures de fluorescence. Préparer au moins 4 réactions, y compris un triplicate, et un contrôle négatif sans GTP.
- Placer la plaque dans un lecteur de plaque préchauffé à 37 oC. Mesurer la fluorescence DENB (excitation 460 nm et émission 535 nm) pendant 5 min par min.
- Induire la fusion en ajoutant 5 mM GTP (5 l de 50 mM GTP).
- Mesurer la fluorescence de la DNB chaque minute pendant 1 h.
- Ajouter 5 'L de 2,5% w/v n-Dodecyl '-D-maltoside pour dissoudre les protéoliposomes et mesurer la fluorescence maximale de la DNB. Lisez la fluorescence NBD pendant 15 min chaque min.
4. Flottation liposome sur le gradient discontinu d'Iohexol12
- (facultatif) Analyser l'efficacité de la reconstitution par des essais de flottaison13.
- Préparer un iohexol de 80 % et 30 % w/v en A100 avec 10 % de glycérol, 2 mM 2-mercaptoethanol et 1 mM EDTA. L'iohexol ne se dissout pas facilement, donc cela doit être préparé la veille en ditant pendant la nuit à 4 oC.
- Mélanger soigneusement 150 l de 80 % de stock d'iohexol avec 150 ll d'échantillon de protéoliposomes pour l'amener à un iohexol de 40 %. Ajoutez ceci à un tube ultra-clair de 5 x 41 mm2 évitant les bulles.
- Couche 250 'L de 30% de stock d'iohexol lentement sur le dessus de l'échantillon pour faire une couche moyenne. Évitez les bulles et perturbant la couche inférieure. Au-dessus de la couche médiane, ajoutez lentement 50 oL d'A100 avec 2 mM 2-mercaptoethanol et 1 mM EDTA.
- Centrifuger le gradient dans un rotor de seau oscillant à 220 000 x g pour 4 h à 4 oC avec une accélération lente et sans rupture.
- Récoltez les couches du gradient et analysez par sDS-PAGE et la tache de Coomassie, la quantification peut être faite par densitométrie. Les protéines reconstituées devraient flotter vers la couche supérieure, tandis que les agrégats protéiques et lipidiques se sédimenteront au fond ou dans la couche moyenne.
5. Analyse de l'orientation des protéines reconstituées par thrombin proteolyse
REMARQUE: La construction d'atlastine rapportée ici a un site de coupe de thrombine entre la fin de l'étiquette de TPS N-terminal e et le début de l'atlastine. Atlastin dans la bonne orientation aura ce site de coupe accessible à la protéase, tandis que la protéine dans la mauvaise orientation sera protégée par la bicouche lipidique.
- Pour essayer d'essayer l'orientation protéoliposome atlastine, réservez au moins 8 'L de protéoliposomes reconstitués frais.
- Ajouter 8 ll de protéoliposomes et 1 l de 1 U/L de thrombine. Incuber à 37 oC pendant 1 h.
- Désinguer la protéase avec 1 l de 5 mg/mL de cocktail inhibiteur de protéase sans EDTA et incuber pendant 30 min à 37 oC.
- Analyser l'échantillon par SDS-PAGE et Coomassie tache. La proportion de protéines clivées et non clivées peut être déterminée par densitométrie.
Résultats
L'efficacité de la reconstitution de l'atlastine est présentée à la figure 2. Des protéoliposomes reconstitués ont été flottés dans un gradient discontinu d'iohexol. Les protéines non incorporées ont été sédimentées dans la couche inférieure (B) ou dans la couche moyenne (M). La protéine reconstituée flotterait vers la couche supérieure (T). Des échantillons du gradient ont été récoltés et analysés par SDS-PAGE et Coomassie coloration. La quantification du gel par de...
Discussion
Les méthodes ici délimiter une méthode efficace pour purifier, reconstituer, et mesurer l'activité de fusion de l'atlastine recombinante. Pour assurer des rendements élevés de l'atlastine fonctionnelle, certaines étapes critiques doivent être prises en considération. L'expression de l'atlastine doit être faite à basse température (16 oC) pour éviter l'agrégation et il faut viser une concentration finale entre 0,4 et 1,5 mg/mL. Les protéines très diluées ne seront pas reconstituées de manière optimale ?...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions le Dr Michael Stern et son laboratoire pour leurs idées et leurs commentaires sur les projets liés à l'atlastine. Ces travaux ont été soutenus par l'Institut national des sciences médicales générales [R01GM101377] et l'Institut national des troubles neurologiques et des accidents vasculaires cérébraux [R01NS102676].
matériels
| Name | Company | Catalog Number | Comments |
| 10 mL poly-prep chromatography columns | Biored | 731-1550 | |
| 10 x 75 mm Flint glass tubes | VWR | 608225-402 | |
| 47 mm diameter, 0.45um pore whatman sterile membrane filters | Whatman | 7141 104 | |
| 96 well white plate | NUNC | 437796 | |
| Anapoe X-100 | Anatrace | 9002-931-1 | |
| Cell disrupter | Avestin | Avestin Emulsiflex C3 | |
| DOPS (1,2-dioleoyl-sn-glycero-3-phospho-L-serine (sodium salt)) | Avanti | 840035P-10mg | DOPS |
| EDTA | Research organics inc. | 6381-92-6 | Ethylenediaminetetraacetic acid |
| EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Complete protease inhibitor |
| Extruder | Sigma Aldrich | Z373400 | Liposofast Basic Extruder |
| GE Akta Prime liquid chromatography system | GE Pharmacia | 8149-30-0006 | |
| Glutathione agarose beads | Sigma aldrich | G4510-50ml | |
| Glycerol | EMD | GX0185-5 | |
| GTP | Sigma Aldrich | 36051-31-7 | Guanosine 5' triphosphate sodium salt hydrate |
| HEPES, acid free | Omnipur | 5330 | |
| Imidazole | fluka | 5670 | |
| Immobilized metal affinity chromatography (IMAC) resin column | GE Healthcare | 17040801 | 1 mL HiTrap Chelating HP immobilized metal affinity chromatography columns |
| Iohexol | Accurate chemical and scientific corporation | AN 7050 BLK | Accudenz/Nycodenz |
| IPTG | Research products international corp. | I56000-100.0 | IPTG, dioxane free |
| L-Glutathione reduced | Sigma-Aldrich | G4251-5g | |
| Magnesium chloride | Fisher | 7791-18-6 | |
| Methanol | Omnisolv | MX0488-1 | |
| NBD-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl) (ammonium salt)) | Avanti | 236495 | NBD-DPPE |
| n-Dodecyl β-D-maltoside | Chem-Impex International | 21950 | |
| Nonpolar polystyrene adsorbent beads | BioRad | 152-3920 | SM2 Biobeads |
| Nuclepore track-etch polycarbonate 19 mm 0.1 um pore membrane | Whatman | 800309 | |
| Optima LE80K Ultra centrifuge | Beckman Coulter | ||
| Phosphatidylcholine, L-α-dipalmitoyl [choline methyl-3H] | ARC | ART0284 | Titriated lipids |
| Plate reader | TECAN | TECAN infinite M200 plate reader | |
| POPC (1-palmitoyl-2-oleoyl-glycero-3-phosphocholine) | Avanti | 850457C-25mg | POPC |
| Potassium chloride | MP | 151944 | |
| Rh-DPPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(lissamine rhodamine B sulfonyl) (ammonium salt)) | Avanti | 236495 | Rh-DPPE |
| Scintillation Cocktail | National Diagnostics | LS-272 | Ecoscint XR Scintillation solution for aqueous or non-aqueous samples |
| Scintillation vials | Beckman | 592928 | Fast turn cap Mini Poly-Q Vial |
| Thrombin | Sigma | T1063-1kU | Thrombin from human plasma |
| Triton X-100 | Fisher | BP151-500 | |
| Ultra-clear centrifuge tubes 5 x 41 mm | Beckman | 344090 | |
| Vortex 9 to 13mm Tube Holder | VWR | 58816-138 | Insert for vortexing flint glass tubes |
| Vortex Insert Retainer | VWR | 58816-132 | Retainer needed for vortex tube holder |
| Vortexer | VWR | 2.235074 | Vortex Genie 2 model G560 |
| β-mercaptoethanol molecular biology grade | Calbiochem | 444203 |
Références
- Chernomordik, L. V., Kozlov, M. M. Mechanics of membrane fusion. Nature Structural and Molecular Biology. 15, 675-683 (2008).
- Orso, G., et al. Homotypic fusion of ER membranes requires the dynamin-like GTPase Atlastin. Nature. 460, 978-983 (2009).
- McNew, J. A., Sondermann, H., Lee, T., Stern, M., Brandizzi, F. GTP-dependent membrane fusion. Annual Review of Cell and Developmental Biology. 29, 529-550 (2013).
- Rigaud, J. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1231, 223-246 (1995).
- Düzgüneş, N. Fluorescence Assays for Liposome Fusion. Methods in Enzymology. 372, 260-274 (2003).
- Wilschut, J., Duzgunes, N., Fraley, R., Papahadjopoulos, D. Studies on the mechanism of membrane fusion: kinetics of calcium ion induced fusion of phosphatidylserine vesicles followed by a new assay for mixing of aqueous vesicle contents. Biochemistry. 19, 6011-6021 (1980).
- Ellens, H., Bentz, J., Szoka, F. C. Proton- and calcium-induced fusion and destabilization of liposomes. Biochemistry. 24, 3099-3106 (1985).
- Meers, P., Ali, S., Erukulla, R., Janoff, A. S. Novel inner monolayer fusion assays reveal differential monolayer mixing associated with cation-dependent membrane fusion. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1467, 277-243 (2000).
- Schaffner, W., Weissmann, C. A rapid, sensitive, and specific method for the determination of protein in dilute solution. Analytical Biochemistry. 56, 502-514 (1973).
- MacDonald, R. C., et al. Small-volume extrusion apparatus for preparation of large, unilamellar vesicles. Biochimica Et Biophysica Acta. 1061, 297-303 (1991).
- Paternostre, M. T., Roux, M., Rigaud, J. L. Mechanisms of membrane protein insertion into liposomes during reconstitution procedures involving the use of detergents. 1. Solubilization of large unilamellar liposomes (prepared by reverse-phase evaporation) by Triton X-100, octyl glucoside, and sodium cholate. Biochemistry. 27, 2668-2677 (1988).
- Scott, B. L., et al. Liposome Fusion Assay to Monitor Intracellular Membrane Fusion Machines. Methods in Enzymology. 372, 274-300 (2003).
- Zhang, W., et al. Crystal structure of an orthomyxovirus matrix protein reveals mechanisms for self-polymerization and membrane association. PNAS. 114, 8550-8555 (2017).
- Parlati, F., et al. Rapid and efficient fusion of phospholipid vesicles by the α-helical core of a SNARE complex in the absence of an N-terminal regulatory domain. PNAS. 96, 12565-12570 (1999).
- Sugiura, S., Mima, J. Physiological lipid composition is vital for homotypic ER membrane fusion mediated by the dynamin-related GTPase Sey1p. Scientific Reports. 6, 20407 (2016).
- Powers, R. E., Wang, S., Liu, T. Y., Rapoport, T. A. Reconstitution of the tubular endoplasmic reticulum network with purified components. Nature. 543, 257-260 (2017).
- Betancourt-Solis, M. A., Desai, T., McNew, J. A. The atlastin membrane anchor forms an intramembrane hairpin that does not span the phospholipid bilayer. Journal of Biological Chemistry. 293, 18514-18524 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon