Method Article
GEN1によるホリデイ接合分解能のリアルタイム調査のための単分子フェルスター共鳴エネルギー伝達法
要約
ここで提示されるのは、HJ分解能を研究するために単分子フェルスター共鳴エネルギー伝達を行うためのプロトコルである。解離定数を決定するために、2 色交互励起が使用されます。シングルカラータイムラプスsmFRETは、リアルタイム切断アッセイに適用され、HJ分解能の前にドウェル時間分布を得ます。
要約
バルク法は分子のアンサンブル挙動を測定し、基礎となるステップの個々の反応速度が母集団全体で平均化される。単分子フェルスター共鳴エネルギー伝達(smFRET)は、個々の分子によって起こる立体構造変化をリアルタイムで記録します。したがって、smFRETは結合および触媒の間に酵素または基板の構造変化を測定する上で強力である。本研究では、サイトソール同義語組換え酵素である4ウェイホリデイ接合部(HJ)とギャップエンドヌクレアーゼI(GEN1)の相互作用を単分子イメージングするためのプロトコルを提示する。また、GEN1によるHJの分解能をリアルタイムで踏襲する単色および2色交互励起(ALEX)smFRET実験プロトコルも提示される。GEN1二量化の運動学は、HJの解決に重要な役割を果たすることが示唆され、これまで解明されていないHJで決定される。ここで説明する技術は、多くの酵素DNA系の貴重な機械的洞察を得るために広く適用することができる。
概要
蛍光検出に基づく単分子法は、高いシグナル対ノイズ比1を提供する。FRETは、1〜10nmの範囲で距離を測定できる分光技術であり、ナノメートル範囲2、3の距離を測定するための分子定規としてこの技術をレンダリングします。受け入れ器の吸収スペクトルは、より短い波長端におけるドナーの発光スペクトルと部分的なスペクトルが重なっている。FRETは、ドナーと受入者ペア間の放射線のないエネルギー伝達によって媒介されるが、エネルギー伝達の効率は、インセプタ4の距離および向きに依存する。
背景を最小限に抑え、蛍光シグナル5,6の検出効率を向上させるためにいくつかのアプローチが実装されている。1つのアプローチは、ピンホールが回折限界7以下のサイズに励起スポットを制限する共焦点顕微鏡です。もう1つのアプローチは、光が臨界角度8の上に軸を外に向ける広視野照明技術である全内部反射蛍光(TIRF)である。その後、光はガラスと水溶液の間の界面に完全に内部的に反射され、ガラス表面に取り付けられた蛍光色素のみを照らし、残りの部分の蛍光色素からの背景を防ぐエバネッセント波を生成します。解決策。
共焦点顕微鏡検査では、分子は自由に拡散または表面固定化することができる。達成された時間分解能は、マイクロ秒から数ミリ秒9までです。単一分子の共焦点検出は、単一光子雪崩ダイオード(SPAD)および対象領域10のポイントバイポイントスキャンによって行われる。TIRFでは、表面に固定された数百の分子の時系列が位置感受性2次元電荷結合検出器(CCD)によって記録される。CCDは、蛍光信号を蛍光信号を増強蛍光スクリーンおよびマイクロチャネルプレートまたは光電子(EMCCD)のオンチップ増殖によって増幅します。時間分解能は、CCD の読み出し速度と量子効率に依存し、通常は数十ミリ秒6の順序で行われます。
HJはDNA修復および組み換え11、12、13、14の中枢中間体である。HJには、互いに交差することなく連続ストランド間を接続する2本の連続鎖と2本の交差ストランドがあります。HJはX積層コンフォーマとして溶液中に存在し、連続鎖が交差し、交差ストランドが他のコンフォーマ15で連続的になることによって連続的な逆性化を受ける。HJの異性愛好はコア配列およびイオン環境に依存し、FRET16、17、18、19によって広範囲に研究されている。
GEN120は、溶液21中の単量体タンパク質であり、HJを切り離すために二量体化を必要とし、したがって再結合鎖22、23の適切な分離を可能にする。HJのスタッキングコンフォーマの好みは、HJ解決24による分解能の向きを設定することによって遺伝的組み換えの結果に影響を与える。GEN1がどのようにHJを結合するかを理解し、2つの切開部を調整し、その完全な解像度がすべて集中的な研究の下にあることを保証する21、22、23、25、26 ,27,28,29,30.
本研究では、前述の31として、目的ベースのTIRFセットアップを使用する。2色交流励起(ALEX)は、GEN1と標識されたHJとの相互作用に基づく立体構造変化を決定するために適用される。ALEXは、ドナー受入者距離依存である2つのレシオメトリックパラメータFRET効率Eと、ドナー受症体検血32を測定するストイチオメトリーパラメータSに基づいて2Dヒストグラムを生成する。ALEXは、ドナーのみ、受け入れ者のみ、混合亜集団を含む蛍光色素のストイチオメトリーに基づいて蛍光種の選別を可能にする。ALEXは、FRETの使用を全範囲に拡張することができ、蛍光素明るさとオリゴマー化の違いを検出できるだけでなく、高分子リガンド相互作用33を監視することができます。
GEN1複合体の寿命内で一貫してHJの解決に成功していることがわかった。時間依存的な立体構造の変化は、個々の分子の時間トレースに由来し、ヒストグラムは基礎となる集団の分布を表します。タイムラプスシングルカラーFRETを使用して、GEN1ダイマーの高速オンレートとスローオフレートが実証され、最初の切開製品で組み立てられたGEN1ダイマーの親和性が向上します。
プロトコル
1. 表面機能カバースリップの準備
-
クリーニング
- コプリンの瓶の中にエタノールに5つのカバーリップ(24 mm x 60 mm)を入れます。エタノールで30分間水酸化カリウムを1Mで30分間ソニケートする。アセトン3xで洗浄し、その後デカント。
-
サイラ化
- アセトン中に2.8%3-アミノプロピルトリエトキシシラン(APTES)の溶液を調製する。APTESボトルをパラフィンフィルムで密封し、4°Cに保管してください。
注:安全ゴーグルを使用し、ヒュームフードの下で作業します。シラン溶液の容器は、シラン溶液を瓶に注ぐ直前と直後にアセトンで完全に乾燥し、すすいでください。 - カバースリップを含むCoplinの瓶に2.8%のAPTES溶液の70 mLを注ぎます。軌道シェーカーで4分間瓶を振ります。
- 瓶を5分間ベンチに置き、1分間超音波処理し、最後にシランがガラス表面のヒドロキシル基と反応するために、さらに10分間ベンチに瓶を置きます。
- 迅速な溶媒交換のために瓶に直接水を注ぐことにより、脱イオン水の1Lを添加して反応をクエンチする。平らな表面に瓶を横に振ることによって水中でスライド3倍をすすいでください。
- カバーを瓶から取り出し、アルミホイルトレイの上に置きます。カバースリップを110°Cのオーブンで30分間焼き、カバースリップを乾燥させ、シランを治します。カバーが室温まで冷却するために、トレイをベンチに置いておきます。
- アセトン中に2.8%3-アミノプロピルトリエトキシシラン(APTES)の溶液を調製する。APTESボトルをパラフィンフィルムで密封し、4°Cに保管してください。
-
ペギレーション
- ビオチニル化PEGおよびPEGを温め、-20°Cで室温(RT)に保存し、容器を開けた時の水分の凝縮を防ぎます。
- 5つのカバーリップを箱の上に向けたシラン化された表面で置きます。2つのカバーガラススリップ(22 mm x 22 mm)を、シラミ化されたカバースリップの端に沿ってスペーサーとして配置します。
- 温めたら、1mLの新鮮な0.1M重炭酸ナトリウム溶液で約1:100の割合でビオチン化PEGおよびPEG溶液を作り、1.5 mLチューブに1.5mgのバイオチン化PEGと150mgのPEGを加えます。
- PEGを溶解し、気泡を除去するためにスピンダウンするチューブを渦。
注:PEGは、分のタイムスケール内の溶液中で加水分解するので、このステップから進みます。 - 各カバースリップに100 μLのPEG溶液を素早く適用します。別の焼きカバースリップを取り、PEGソリューションでカバースリップの上にその上のシラン化された表面を下向きに置き、したがって、22ミリメートルx22ミリメートルの非シラン化カバーリップを可能にするガラス溶液ガラスサンドイッチを形成し、2つの機能化されたカバースリップを可能にします簡単に分離される。
- 暗闇の中で一晩(16時間)カバーリップをインキュベートします。インキュベーションが完了したら、カバースリップを分解し、ホヤボトルで側面から洗浄することにより、脱イオン水を使用して10倍をすすいでください。
- 乾燥窒素の流れの下でカバースリップを乾燥させます。ドライカバーを真空下に保管してください。
注:スライドは、品質の低下することなく、1ヶ月間使用することができます。
2. フローセルの調製
-
単一チャネルフローセル
- 石英スライド(50 mm x 20 mm)の中央部分に直径 1.22 mm の 2 つの穴を開け、中心はスライドの端から 37 mm 離れ、スライドの端から 6.5 mm 離れています(図 1A)。
- 電子カッターを使用して、41 mm x 2.25 mm チャネルを 50 mm x 20 mm の二重粘着シートに切り取ります。
- 保護カバーのプラスチック側を剥がし、石英スライドのエッジにピースの端を合わせます。ポリテトラフルオロエチレンピンセットで穏やかに押すことによって、閉じ込められた気泡を取り除きます。
- 粘着部分の紙側をはがします。カバースリップの機能面にピースを取り付けます。
- ポリエチレンチューブ(I.D.1.22 mm)を入口用に11cm、出口に25cmの長さにカットします。フローセルの入口と出口として、以前に掘削された穴にチューブを挿入します。
- 5分のエポキシ接着剤を使用して、石英カバースリップインターフェイスの端と入口と出口のチューブの周りをシールします。
- 乾燥した状態になったら直ちにフローセルを使用するか、乾燥真空下に保管して後で使用してください。
- 0.03 mg/mLの濃度にPBSでアビジンを溶解します。0.2 μmの注射器フィルターを通してフィルターを出す。
- 1 mL注射器を用いて流れセルにアビジンを流します。余分なアビジンを洗い流すためにバッファで満たされた別の注射器を使用してください。注射器を交換する間、気泡を導入しないように注意してください。
-
マルチチャネルフローセル
- 石英スライド(76 mm x 25 mm)の各側に直径 1.22 mm の 6 つの穴をドリルします (図 1B)。スライドの端から穴を4.5mm、9.3mm離します。各穴ペアの中心間の距離が15mmであることを確認します。
- 電子カッターを使用して、6つのチャンネル(20 mm x 2.25 mm)を 76 mm x 25 mm の二重粘着テープに切り取ります。
- 保護カバーのプラスチック側を剥がし、接着剤片の端を石英スライドの端に合わせます。ポリテトラフルオロエチレンピンセットを使用して穏やかに押すことによって、任意の閉じ込められた気泡を削除します。
- 接着剤片の紙側を剥がし、カバースリップの機能化面に取り付けます。
注:紙側を剥がして石英スライドに取り付けることは、マルチチャンネルフローセルでうまく機能する場合があります。 - 6チャンネルの入口管(11cm)と出口管(25cm)を切ります。手順 2.1.6 ~ 2.1.9 で説明するようにフロー セルを準備します。
- 最初のチャンネルの出口をポンプに接続します。入り込みはOSSで0.5 mLチューブに入れます。
注:流入管の長さは、酵素の流入時間をフローセルに同期し、イメージングの開始によって連続的な流れの下で行われる切断実験の事象の数を最大化するために選択され、したがって、早期の光漂白を減少させる蛍煙管。 - 使用するチャンネルのコンセントを切断して、新しいチャネルに移動します。プラスチック部分に接着剤で密封された注射器針から作られたプラグでコンセントを閉じます。使用するチャネルの入り込み情報を閉じます。
3. 酸素清掃システム(OSS)の準備
- メタノールの0.2g(±)-6-ヒドロキシ-2,5,7,8-テトラメチルクロマン-2-カルボキシリン酸(フッ素の点滅を最小限に抑えるトリプレット状態クエンチャー)を800μLのメタノールで溶解します。
- 脱イオンH2Oの6 mLを追加し、溶解するまで1 N NaOHを滴下に加えます。注射器フィルターを通してフィルターを通し、1 mLのアリコートにし、-80°Cで保存します。ストック濃度は~100μMです。
- ddH2Oの4 mLに61mgのPCA粉末を溶解することにより、3,4-ジロキシ安息香酸(PCA)の新鮮な溶液を調製する。ストック濃度は~100nMです。
- 10 N NaOHの58 μLを滴下に加え、PCAが完全に溶解するまで各滴の後に渦を作るようにします(pH = 9)。
- PCD貯蔵バッファーの7 mLにプロトカテチュエート3,4-ジオキシゲナーゼ(3,4-PCD)の5.3mgを溶解する(表1)。3,4-PCDは、プロトカテチュ酸34の酸化を触媒することにより結合/切断バッファーから酸素を除去する。
- PCD溶液を1mLのアリコートに分割します。ストック濃度は~1 μM. 液体窒素中のアリコートをスナップフリーズし、長期保存のために-80°C、または-20°Cで長期保存します。
- 新しいバインディング バッファーを準備します (表 1)。smFRET切断実験のために2 mMMgCl 2で2 mM CaCl2を置き換えます。
- イメージングバッファの1 mLを準備する(表1)。酸素清掃システムの活性を維持するために、フローセルに導入されるまで、イメージングバッファを氷上に保持します。
4. 蛍光標識HJの調製
- トリス-EDTAバッファー(表1)の凍結乾燥オリゴ(表2)を100μMの濃度に再構成する。
- 表1に記載されているX0オリゴの各々の等化部分〜3μLを混合して合成接合部を調製する。
- 95°Cで5分間加熱し、その後1°C/分の速度でRTに遅い冷却を行い、熱ブロックまたはPCRサーモサイクラーを使用して、所望の冷却速度を達成します。
- 10%トリスボレート-EDTAポリアクリルアミドゲルの8 cm x 8 cmに混合物をロードします。100Vを適用し、〜2時間ゲルを実行します。バンドは目ではっきりと見え、その色は紫色です。
- きれいな刃とアニール基板のバンドを物品格上。ゲル片をオートクレーブされた1.5 mLチューブに移します。
- きれいなプランジャーでチューブ内のゲル片を粉砕し、TE100バッファーの100 μLを追加します(表1)。
- サーモミキサーで1,500rpmでチューブを20°Cで振り、HJを抽出するか、4°Cで一晩インキュベートします。
- 基板35を含む溶液にエタノール沈殿を行う。
- TE100 バッファーの 20 μL で基板を再中断します (表 1)。最終的な濃度は、各チューブで1-3 μM. アリコット2 μLであり、-20 °Cで保存します。
5. GEN1のタンパク質発現と精製
- 切り捨てられたヒトGEN11、2、3、4、5の発現のためのプラスミドを、エントリベクターのPCRによりC終端20でヘキサヒスチジンタグで構築する。
注: N 端末のタグ付けにより、GEN1 が非アクティブになります。非構造化Cテールは、全長GEN1の精製を大幅に困難にします。また、全長GEN1は、切り捨てられたバージョン23よりも少ない活性を示す報告された。 - 発現ベクトルを大腸菌BL21-コドンプラス(DE3)-RIPL株に変換します。
- 変換した細胞を2つの6Lフラスコに接種し、0.8のOD600に達するまで180rpmで振盪を起こす37°Cで2Lのルリアブロス培中を含む。
- 培養物を16°Cに冷却し、0.1mMイソプロピルβ-d-チオガラクピラノシド(IPTG)でGEN1発現を48時間誘導する。
- 遠心分離機で1000 x gで4°Cで回して細胞を収穫します。培養の各リットルは、ペレットの5-6グラムを得る。
- 上清を廃棄し、4 mL/gの細胞を用いて、リシスバッファー(表1)でペレット化した細胞を再懸濁する。
- 30 kPsiで細胞破壊器を使用して細胞分解を行い、4°Cで1時間10,000 x gでスピンダウンします。上清を回収し、0.45 μmフィルターを使用して氷の上に濾過します。
- バッファーA(表1)を用いて5mL Ni-NTAカラムを2.5ml/分の流量で濾液を通過させることにより、FPLCを用いてタンパク質精製を行う。
- 15カラムボリューム(CV)で洗浄します。バッファAと500mMイミダゾールの線形勾配を5mL画分で20CV以上のElute。GEN1は、約100mMイミダゾールでカラムから溶出する。
- 収集された分画からピペット10 μLアリコートは、各アリコートに2x SDS負荷染料の等しい体積を追加します。90°Cで5分間加熱し、冷却し、サンプルをスピンダウンしてサンプルを変性させます。
- サンプルを10%のビストリスゲルにロードします。クーマッシーブリリアントブルーを使用して200 V.ステインで30~45分間ゲルを実行し、その後デステインします。精製されたGEN1を含む分数を収集します。
- 緩衝C(表1)を用いて希釈することにより、結合分画の塩濃度を100mMに減らす。
- 低塩タンパク質をバッファーB(表1)を用いて3mL/minの流量で5mLヘパリンカラムを通す。
- バッファBと1 M NaClで20 CVのグラデーションを使用して10 CV.Eluteで洗浄します。GEN1が約360 mM NaClを溶出する5mL分画を収集します。
- 手順 5.8 で説明されているように、精製された GEN1 分画の eluted 分数を確認します。これらの分数を組み合わせ、バッファ C を使用して 100 mM NaCl に希釈します。
- バッファーBを使用して、低塩タンパク質を1mL/min流量で陽イオン交換カラムにロードします。
- バッファBと1 M NaClを使用して40 CVのグラデーションで溶出します。GEN1が約300mM NaClを溶出する1.7mL分画を収集します。
- ステップ 5.8 で説明されているように、ELuted 画分の GEN1 の純度を確認します。
- 最も純粋な分画を組み合わせ、ストレージバッファに対して4°Cで透析を行います(表1)。透析中にバッファーの少なくとも1つの交換を実行します。
- タンパク質濃度~0.5–1 mg/mLを測定します。小管中の10~15μLのダイアライズタンパク質をアリコートし、液体窒素中のフラッシュフリーズを行い、-80°Cで保存します。
6. 単分子FRET実験
注: smFRET 実験は、前述の31に基づくカスタム構築された目的ベースの TIRF セットアップ (図 1C)に基づいて実行されます。
- 単色FRET実験
- 100x TIRFの目的に浸漬オイルの1滴を適用します。EMCCDを適切なゲインに設定して、信号をバックグラウンドに最適化し、飽和を防ぎます。
注:レーザーを直接見ないで、レーザーを合わせるときに保護ゴーグルを着用しないでください。 - フローセルをサンプルホルダーに慎重に置きます。オイルがカバースリップに触れるまで、粗い調整を使用して徐々に目的を上げます。
- 緑色のレーザー(532 nm)をオンにします。目的の微調整モードに切り替えます。カメラポートに放出を指示し、モニター上の画像を観察します。
- カバースリップの機能化された表面がフォーカスに持ち込まれ、モニターで観察されるまで、目的の高さを調整します。
注:EMCCDによる画像取得により、アコスト光学調合フィルタ(AOTF)を介してレーザー励起がトリガされ、画像が取得されていないときにサンプル光漂白を防止します。 - カバースリップの機能化された表面からの背景が蛍光標識HJに流れる前に少数のスポットを超えないことを確認してください。
- TE100バッファー(表1)で約1000回ストック基板を1~5nMの最終濃度に希釈します。希釈基板のピペット 0.2~0.5 μL をOSSを用いるイメージングバッファの120 μLを0.5mLチューブに入れます。
- フローセルの出口をシリンジポンプに接続します。フローセルの入り込み管を1.5mLチューブに挿入し、シリンジポンプを30~50μL/minの流量で操作し、チューブから溶液を引き出します。
- 緑色のレーザーで簡単に画像化することにより、表面の良好なカバレッジ(均質に分布し、十分に間隔をあけた基板の100~300)を頻繁に確認してください。
- 表面カバレッジがまだ十分でない場合は、溶液から蛍光標識されたHJが表面に沈降するか、流れるステップを繰り返すのに数分間待ちます。
- 30~50 μL/minで別の120 μLのイメージングバッファー(表1)を流し、無結合に標識されたHJを洗浄します。その後、OSSが溶存酸素を枯渇させるのを可能にするために、フローセルを5分間座らせます。蛍光素の光漂白は、イメージングの開始時に最小限にする必要があります。
- 露光時間(~60ミリ秒)を設定し、データ転送速度(~104ミリ秒)に基づいてソフトウェアによってサイクル時間が自動的に設定され、必要なサイクル数またはフレーム数(~400)を指定します。ドナー(Cy3)および受入者(Alexa Fluor 647)からの放出は、画像スプリッタデバイスによって2つのカラーチャンネルに分割される。
- サーフェス上の適切な領域を見つけ、目的と記録の高さを調整して画像に焦点を合わせ、ムービーを 16 ビット TIFF 形式で保存します。
- 新しい領域に移動します。
注: 同じ領域を 2 回イメージングしないように、常に一方向にのみ移動します(出口から入口まで)。 - 120 μL のイメージング バッファーに 1、2、5、10、25、50、75、および 100 nM GEN1 を一度に 1 つずつ準備します。30~50 μL/minの流量で溶液を流します。
注:GEN1によるHJの結合またはフリーHJの異性化のように、必要な測定が定常状態で行われる場合は、流れが止まってから3~5分待って映画を録画します。GEN1濃度ごとに新しい領域から3~4本の映画を獲得。 - GEN1によるHJの切断のように連続的な流れの下で測定が行われる場合は、GEN1の流入口の前に5~10sの記録を開始する。6チャンネルフローセルの新しいチャンネルに移動して測定を繰り返します。
- 最後に、固定蛍光ビーズスライドを使用して、画像分割装置内のドナーと受諾粒子を互いにマッピングする。
- 1 μm 径蛍光ビーズの 0.2 μL を 1 M トリスの 500 μL (pH = 8.0) に加えて、ビーズが表面に付き付けられるようにします。
- 両面粘着シールの 22 mm x 22 mm の部分の中に正方形 (18 mm x 18 mm) を切ります。76 mm x 25 mm クォーツ スライドの中央にピースをはがし、貼り付けします。
- 希釈ビーズ溶液の50 μLを置き、5-10分間放置して落ち着きます。正方形の部分の上に22 mm x 22 mmカバースリップを取り付けます。組織で余分なビーズ溶液を乾燥させ、次いでエポキシ接着剤によってチャンバーを密封する。
- 60ミリ秒の露出時間でビーズスライドの100フレームを取得します。
注意: 検出器の飽和を避けるために、レーザーパワーとEMCCDゲインを最小限に抑えます。 - ソフトウェアパッケージ(例えば、TwoTones)をインストールし、ユーザーマニュアル36に示されているように、その中のムービーを開きます。ドナーおよびインセプターチャネル内の個々のビーズの位置を選択します。マニュアルで説明されているように、変換行列を生成します。
注:このソフトウェアは、ドナーとインセプターチャネル内の粒子の位置を一致させ、画像分割デバイスのわずかなミスアライメントを修正するために変換マトリックスを使用しています。 - [ファイル]に移動し、[ムービーの読み込み]を押し、ムービー ファイルを選択して[開く]を押します。ファイルメニューで、TFORMを押して、ビーズスライドから生成された変換マトリックスを選択します。偽陽性が含まれなくなるまで、ドナーチャネルと受諾チャネルのしきい値を調整します。
- [チャンネル フィルタ]メニューで、[D&A] オプションを選択して、ドナーとアパニタの両方でラベル付けされたパーティクルを選択します。 最も近い近傍の制限フィールドをチェックして、互いに非常に近い分子を除外します。非常に偏心分子を除外し、非常に広いまたは非常に狭い分子を除外するために幅の制限をチェックするには、最大楕円性をチェックしてください。
- ヒストグラムを構築するためにツートーンマニュアルで指示されているようにプロットヒストCWを入力します。
注:「明らかな」FRET効率は、受け入れ者の放出をドナーおよび受入者からの総排出量で割ることによってプログラムによって計算される。ツートーンは、FRET効率に対する分子の状態の分布をビン化するために100の間隔を使用します。 - 各分子のタイムトレースを生成するために、Twotonesマニュアルで指示されているようにプロットTimetraceCWを入力します。
注: 時間トレースは vbFRET37でさらに分析して、異なる FRET 状態、それぞれの所因時間、および異なる状態間の遷移率を識別できます。
- 100x TIRFの目的に浸漬オイルの1滴を適用します。EMCCDを適切なゲインに設定して、信号をバックグラウンドに最適化し、飽和を防ぎます。
- 2色交流励起FRET(ALEX)実験
- ドナーと受容者の連続したフレームから成るムービーを、緑と赤のレーザーで直接励起し、それぞれ約80ミリ秒の持続時間で記録します。
- 取得した ALEX ムービーをツートーンで開きます。ドナー励起(DexDem)によるドナー放出:3つのチャネルに対して、適切な検出閾値を~300として設定します。ドナー励起による受容者放出(DexAem);直接励起(AexAem)による受容者の放出。
- チャネルフィルタDexDem&DexAem&AexAemを適用して、ドナーとアセプターの両方を持つパーティクルを選択します。パーティクルを 3 つのチャネルで ~200 ~300 にリンクします。
- プロットヒスタレックス MATLAB コードを使用して、ALEX ヒストグラムを生成します。ヒストグラムの異なるピークをガウス関数に合わせ、Originソフトウェア38を使用して曲線の下の領域の各母集団の割合を決定します。
注:結合アッセイのピークは結合されたGEN1-HJ複合体に対応し、自由HJではピークは相互作用する異性体を表す。 - plotTimetraceALEX MATLABコードを使用して、直接励起によるドナー放出、およびFRETおよび直接励起による受容放出を示す各分子のタイムトレースを生成します。
注:ALEXタイムトレースは、ドナーとアセプターの両方の放出を示すために独立してトレースしますが、単色FRETよりも低い時間分解能で。単色 FRET と同様に、ALEX タイム トレースを vbFRET でさらに分析して、異なる FRET 状態とそれぞれの所持ち時間を識別できます。 - 結合母集団とGEN1濃度のパーセンテージを双曲線関数に合わせて解離定数を決定します。
- タイムラプスシングルカラーFRET
- 観測されたダイナミクスの速度に応じて、緑色のレーザーの露出時間を 60 ミリ秒、サイクル時間を 624 ミリ秒以下に設定します。
- 6チャンネルフローセルの1チャンネルで流量を110μL/minに設定します。フローセル内のGEN1の入り口の前に、少し録音を開始します。
注:連続的な流れは、蛍光素の急速な光漂白につながるので、イメージングとタンパク質のエントリの開始を同期させることで、キャプチャされたイベントの数を最大化します。最適なシリンジポンプの読み取りは、デッドボリュームとフローセルを組み立てるために使用される正確なチューブに依存します。私たちの場合は~25 μLです。 - 合計取得時間 78 秒で約 125 フレームのムービーを取得します。記録の最後に、サンプルを赤いレーザーにそれぞれ50フレーム、25ミリ秒の露出時間で赤いレーザーにさらして、受け入れをプローブします。
注: この方法は、励起サイクルの数を減らすことによって切断実験の観察ウィンドウを延長します。偏光のkオンおよびkオフとしての運動パラメータは、分布をバイ指数モデル30、38に適合させることによって導出される。
7. 電気泳動シフトアッセイ(EMSA)
- 50 μLの総体積において、EMSA結合バッファーで30分間RTで50 pM Cy5標識HJを用いてGEN1の所望の濃度をインキュベートする(表1)。
- 6%トリスボレートEDTAゲルの8 cm x 8 cmのサンプルをロードします。RTで1x TBEバッファで1時間+20分の100Vを使用してゲルを実行します。
- 各レーンの総蛍光強度に対する相対的な寄与からGEN1濃度での結合基板のパーセンテージを決定する。
注:GEN1モノマー-HJ(バンドI)は、その大きさの合意によって同定され、その大きさは、ニックされたHJ21、30に対するGEN1モノマーのピコモル結合と同定される。GEN1-dimer-HJは、Gen1モノマーをHJ21,23に段階的に結合するため、バンドIIに割り当てられます。 - 方程式を使用して、見かけの結合定数Kd-モノマー アプリ EMSAとKd-ダイマー アプリ-EMSAを計算します。

どこ:最大は、それぞれの種がその最大結合に達した濃度(モノマーまたはダイマー);。n はヒル係数です。Kd-app-EMSAは、各種の明らかな結合定数であり、モノマーまたはダイマーの半分の最大値が存在するGEN1の濃度を示す。
結果
HJのコンフォーマーバイアスと偏同化
HJの逆化は、ジャンクション17、18、39の2つの隣接する腕の標識を通じてFRETによって広範囲に調査されている。ドナー(Cy3)およびアクセプタ(Alexa Fluor 647)は、それぞれ2つの隣接するアーム、R(ストランド2)およびX(ストランド3)に位置する(図2A)。積み上げXの同数体は、2本の連続鎖(すなわち、Iso(1,3)またはIso(2,4))によって割り当てられた。隣接するラベルX0のALEX FRETヒストグラムは、より豊富なIso(1,3)(E~0.75)とそれほど豊富でないIso(E~0.40)(図2B)の交換に対応する2つのピークを示しています。
単色FRETは、EMCCD2カメラの使用領域を小さくすることにより、高い時間分解能で自由なHJの急速な立体構造変化を記録するためのタイムトレースを取得するために使用されます。X0ジャンクションの代表的な単色 FRET タイム トレースは、高 FRET と低 FRET の間の遷移を示します (図 2B)。異性化率kIso(1,3)-Iso(2,4)およびkIso(2,4)-Iso(1,3)は、Iso(1,3)およびIso(2,4)(図2C)の在入時間ヒストグラムから得られたものと一致している。
SMFRETはGEN1によるHJのアクティブな歪みを示す
HJはGEN122に拘束すると構造的な再編成を受ける。したがって、ドナーとアセプターの間隔は、Iso(1,3)とIso(2,4)の両方で類似しています(図3A)。SmFRET結合アッセイは、HJの切断を防止するためにCa2+の存在下で行った。異なるGEN1濃度における隣接する標識X0接合部のFRETヒストグラムは、ALEX(図3B)によって取得された。ヒストグラムは、低FRETピークからIso(2,4)の寄与を引いた後、自由高FRET Iso(1,3)に対応する2つのガウス関数に適合し、もう1つは結合されたGEN1-HJ母集団に対応する。
飽和GEN1濃度では、X0のFRETヒストグラムは、モデル22によって予測されるHJのいずれかの異性ソーマーに結合されたGEN1に対応する単一の低FRETピークしか有しない。見かけのモノマー解離定数(Kd-モノマーアプリ)は、GEN1濃度の関数としてのGEN1結合集団の割合の双曲線適合から決定される(図3C)。隣接するラベルnk-X0は、最初の切開反応後に製品を模倣する、ひっかき傷を付けたバージョンHJを表す。シミュレートされたニックによる積み重ね歪みの緩和のため、nk-X 0はX 0(図3D対図3B)とは異なり、E~0.40の単一基板ピークから明らかな非異性化構造体40である。GEN1-nk-X0複合体の構造はGEN1-X0複合体の構造と類似しており、FRET効率の類似性(nk-X 0の場合はE~0.25、X0の場合は0.32)(図3D対図)。 図 3B)。GEN1モノマーとnk-X0の強い結合は、X0(図3E対図3C)よりも40倍低いKd-モノマーアプリ値によって実証される。このタイトな結合は、GEN1ダイマーまたはそのモノマーの解離の可能性が低い場合にHJの不完全な分解に対する保護メカニズムとして作用し得る。
Gen1モノマーをHJに段階的に結合
GEN1モノマーをHJに結合し、ダイマー形成に続いてダイマー形成は、溶液21、23、41のダイメリック形態に存在する原核生物リソルバゼと比較して真核生物HJリソルバゼGEN1に対するユニークな特徴である。50 pM X0におけるGEN1のEMSAは、上部パネルのローマ数字によって示される高次複合体へのGEN1の段階的な関連を示す(図4A)。EMSA(Kd-モノマー-EMSA)によって決定されたGEN1モノマーの解離定数は、smFRET結合アッセイKd-モノマーアプリ(図4Aおよび図3C)からの解離定数と一致する、それぞれ)。バンドIIの定量化は、GEN1ダイマー(Kd-dimer-EMSA)の平衡解離定数を計算するために使用される。50 pM nk-X0のGEN1のEMSAは、非常に低いKd-モノマーアプリEMSAがX 0よりも30倍低いのに示すように顕著なモノマー結合を示し、K d-dimer-EMSAはX0 (図4B)に匹敵します。
GEN1モノマーがHJに結合し歪むさらなる証拠は、低GEN1濃度でMg2+の存在下で安定した低FRET状態(図4C)を有する汚れた粒子のかなりの数の痕跡の観察である。これらのトレースの数は、GEN1濃度の増加に伴って減少した。HJの分解能は、ダイマー形成をサポートするGEN1モノマーのタイトな結合によって駆動されます。モノマー結合はMg2+における不潔なnk-X0の時間トレースで観察され、ナノモル濃度が少ないまで延びる(図4D)。GEN1モノマーはnk-X 0を保護するためにしっかりと結合し、最終的にはダイマー形成によって完全な決断を保障する。
HJのSMFRET分解能アッセイ
smFRETアッセイにおける「切断」という用語は、HJの「分解能」と同じ意味で使用され、このアッセイでは第2の切断事象に続く製品リリースのみが検出される。イベントは、約1.3分の獲得時間にわたって光感受性アクセプターの光漂白を最小限に抑えるために、タイムラプス単色励起によって記録されます。
図5Aの回路図は、機能化ガラスに取り付けられたX0のGEN1による結合および歪み後のX0 Iso(1,3)の鎖1および3の切開を示す。ドナーと受入者の両方が、HJ決議後に信号を失う結果、溶液に入ります。第1および第2の切開はnk-X 0で分離され、部分的に解決されたHJのプロトタイプを例示する。GEN1の結合時に、nk-X0はX0と同様の構造を採用する。図5Bに示すように、解像は鎖1の単一切開部によって進行する。
中間FRET(E=~0.40)の出現なしに解決されたX0のトレースで安定した低FRET状態の後のドナーとアクセプターの同時出発は、GEN1-HJの寿命内に完全な解決が起こることを示します。複雑な (図 5C)したがって、これらの結果は、HJ分解能がGEN1-HJ複合体寿命内に起こることを示唆している。nk-X0の分解も構造的な再配置後に進み、X0と同様の2つの蛍煙素(図5D)を運ぶ二重の出発によって終了する。
GEN1モノマー結合HJにおけるGEN1ダイ精化の運動学
タイムラプスsmFRETは、主にGEN1モノマーによる歪み後のHJのダイマー形成と分解に要する時間を含む切断前の測定を測定します。この技術を適用すると、前切断の分布はGEN1濃度依存性であるため、X0およびnk-Xの両方の分解にダイマー形成が必要であるという主張を支持する直接的な証拠が提供される。
HJ分解能(kアプリ)の明らかな速度は、それぞれのGEN1濃度における切断前の平均の逆数として定義される。「明らか」という用語は、HJ分解能の後にGEN1が製品に拘束されたままである可能性を排除できないため、HJ分解能のレートを記述するために使用される。
X0(図6A)の切断前分布の確率密度関数(PDF)は、低GEN1濃度で長いダイマー形成の時間を反映し、その後、より高いGEN1濃度で短くなります。ダイマー、kオンダイマーおよびkオフダイマーの関連および解離率は、それぞれ、バイ指数モデル30から決定される。また、nk-X0(図6B)のPDFは、ダイマー形成の要件を示すX0と同様の分布を示す。
kアプリ対GEN1濃度のプロットを双曲線関数に適合させた。X0および nk-X0の明らかな触媒レート定数(kMax-app)は、それぞれ 0.107 ± 0.011 s-1および 0.231 ± 0.036 s-1です (図 6C)。X0およびnk-X0ジャンクションのためのkアプリのプロットは、より速いkMax-appとより遅いkオンダイマーのためにGEN1濃度〜5.6 nMで交差する無傷のジャンクション。
要約すると、比較的速いkオンダイマーと遅いkオフダイマーは、ダイマーが形成されるとHJ分解能に向かって前方反応の進行につながります。GEN1モノマーをnk-X0接合部に強く結合することは、中止される可能性が低い第2の切断に対するフェイルセーフ機構を構成するか、細胞内の一次分解経路によって残された不完全に未解決のHJを拾うのに役立ちます。
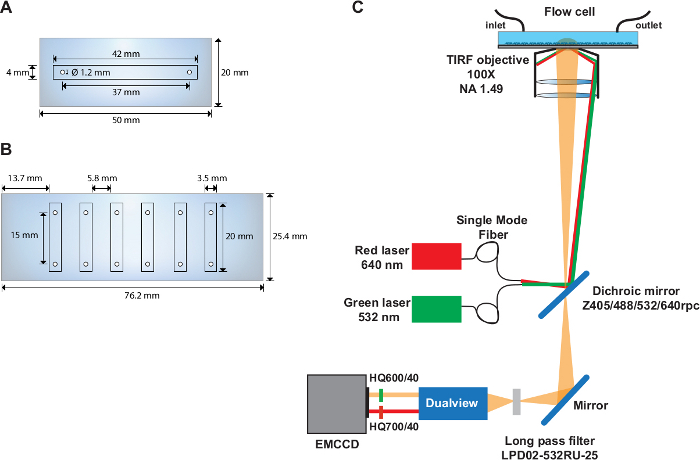
図1:単一および複数チャンネルのフローセルと光学セットアップのレイアウト。
(A)単一チャネルフローセルの回路図。(B)6チャネルフローセルの回路図。(C)励起源を描写した光学セットアップのレイアウト、TIRF目的、フィルタキューブ内に設置されたダイクロイックミラー、および画像スプリッタデバイスで使用される発光フィルタ。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図2:FRETによって観察されたHJのコンフォーマーバイアスと偏同化。
(A)隣接するラベルX積み上げHJ適合子の対称化は、2本の連続鎖にちなんで名付けられた。ストランドには番号が付けられており、腕は文字で示されます。切開部位は矢印で示されます。ドナー(緑色)と受入者(赤)の位置と、対方化時のFRETの変化を示す。(B) 右パネル: FRET タイム トレース (黒) と理想化された FRET トレース (赤) は、50 mM Mg2+で X0です。左パネル:50 mM Mg2+でX0のFRETヒストグラム。ドナー(緑色)及び受理器(赤色)の蛍光強度を以下に示す。(C)隣接するラベルX0 Iso(1,3)とIso(2,4)のドウェルタイムヒストグラムを単一指数関数に取り付け、対方化率を決定した。不確実性は、適合の95%の信頼区間を示します。この図は、以前に公開された文献30から変更されています。
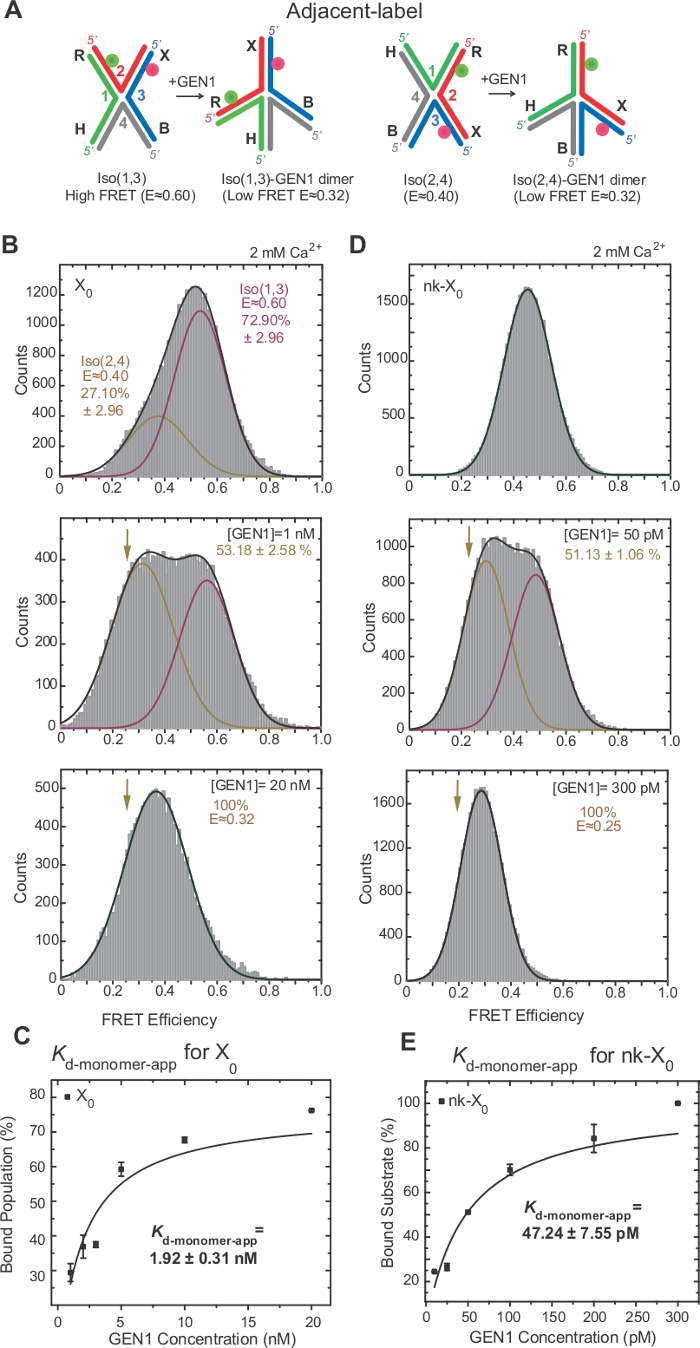
図3:GEN1によるHJのアクティブ歪み。
(A)提案されたモデル22に基づく隣接する標識HJの構造改変。(B)隣接するラベルX0のALEX FRETヒストグラムは、Iso(1,3)に対応する主要な高FRETピーク(E=~0.6)と、Iso(2,4)の低いFRETピーク(E=~0.4)に対応しています。ヒストグラム全体は、自由高 FRET Iso(1,3)に対応する 1 つと結合母集団に対応する 1 つは、総母集団に対する Iso(2,4) の初期寄与度を引いた 2 つのガウス関数に適合します。(C)見かけのモノマー解離定数(Kd-モノマーアプリ)は、GEN1濃度の関数としてのGEN1結合集団のパーセンテージの双曲線適合から決定される。(D)異なるGEN1濃度で隣接する標識nk-XのFRETヒストグラム。低 FRET (E = ~0.25) ガウスの下の領域は、結合された母集団のパーセンテージに対応します。(E)nk-X0のKd-モノマーアプリは、GEN1結合集団の双曲線適合から決定される。誤差バーは、2つ以上の実験からの標準偏差を表します。この図は、以前に公開された文献30から変更されています。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図4:GEN1をHJに段階的に結合する。
(A)50 pM X0でGEN1の電気泳動シフトアッセイ(EMSA)。上部パネル:ローマ数字は、複合体内のGEN1モノ素の数を示す。下部パネル:GEN1モノマーをX0に結合する。明らかな解離定数は、それぞれの種のシグモイドフィットから得られ、2つの実験の平均を表す。(B)50 pM nk-X0におけるGEN1のEMSAは、非常に低いKd-モノマーアプリ-EMSAによって示される顕著なモノマー結合を示す。(C) MG2+でバインドされたが、隣接するラベル X0の FRET タイム トレース 。ドナー励起は〜1.3分間行われ、続いて直接受容性興奮(シェードピンク領域)が続いた。(D) MG2+でバインドされたが、隣接するラベル nk-X0のバインドされた FRET タイム トレース 。この図は、以前に公開された文献30から変更されています。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図5:HJのSMFRET分解能アッセイ。
(A)GEN1による歪み後の隣接するラベルX0 Iso(1,3)の概略図。基板は、ビオチン/アビジンリンケージを介して機能化表面に取り付けられています。2つの切開後のGEN1の解離は、溶液に入るドナーおよび受理者の両方の損失をもたらす。(B)鎖1を断断として隣接する標識nk-X0の分解能の概略図。(C)Iso(1,3)の切断の2mM Mg2+でのタイムトレース(黒)。GEN1結合の発症は、FRET信号が切断によって突然失われるまで安定した低FRET状態を形成する。それに応じて、GEN1結合時のドナーの増加および受理者蛍光強度の低下は、切断時に両方の色素からの蛍光の同時消失に続く。(D)同様に、nk-X0のタイムトレースは、FRET信号の突然の損失によって結論付けられたGEN1結合時の安定した低FRET状態を示す。この図は、以前に公開された文献30から変更されています。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図6:GEN1モノマー結合HJ上のGEN1ダイ精化の運動学。
(A)X0の前切断分布の確率密度関数(PDF)プロットは、GEN1濃度への依存性を示す。それぞれのGEN1濃度における低FRET状態(切断前)のウェル回数は、2回以上の実験から得られ、平均レート(kアプリ)を得るために用いられた。記載されたkアプリレートは、それぞれのGEN1濃度における平均τ前切断の逆から決定される。ダイマー形成のアソシエーション(kオンダイマー)と解離(kオフダイマー)率は、バイ指数モデル38から計算されます。エラーはkアプリの SEM を表します。(B) nk-X0の切断前分布とそれぞれのkアプリレートのPDFプロット。(C)見かけの触媒速度(kMax-app)を決定するために双曲線関数にフィットしたkアプリ対GEN1濃度のプロット。X0と nk-X0のkアプリのプロットは、X0の高速初期kアプリを示しており、その後、nk-X0を超える [GEN1] ~5.6 nM を超えています。この図は、以前に公開された文献30から変更されています。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| バッファー | 堆肥化 | ||
| バインディング バッファ | 40 mM トリス-HCl pH 7.5, 40 mM NaCl, 2 mM CaCl2,1 mM DTT, 0.1% BSA および 5% (v/v) グリセロール | ||
| バッファ A | 20 mM トリス-HCl pH 8.0、1 mM DTT および 300 mM NaCl | ||
| バッファ B | 20 mM トリス-HCl pH 8.0、1 mM DTT および 100 mM NaCl | ||
| バッファ C | 20 mM トリス-HCl pH 8.0 および 1 mM DTT | ||
| 切断バッファー | 40 mM トリス-HCl pH 7.5, 40 mM NaCl, 2 mM MgCl2,1 mM DTT, 0.1% BSA および 5% (v/v) グリセロール | ||
| EMSA バインディング バッファ | 40 mM トリス-HCl pH 7.5, 40 mM NaCl, 1 mM DTT, 2 mM CaCl2,0.1 mg/ml BSA, 5% (v/v) グリセロールおよび 5 ng/μl ポリdI-dC | ||
| イメージング バッファ (バインディング) | 40 μL (±)-6-ヒドロキシ-2,5,7,8-テトラメチルチロマン-2-カルボキシリン酸(4 μM)、60 μL PCA(6 nM)、60 μL PCD(60 nM)および840 μLの結合バッファー | ||
| イメージングバッファ(切断) | 40 μL (±)-6-ヒドロキシ-2,5,7,8-テトラメチルチロマン-2-カルボキシリン酸(4 μM)、60 μL PCA(6 nM)、60 μL PCD(60 nM)および840 μLの切断バッファー | ||
| リシスバッファ | 20 mM トリス-HCl pH 8.0, 10 mM β-メルカプトエタノール, 300 mM NaCl および 2 mM PMSF | ||
| PCD ストレージ バッファ | 100 mM トリス-HCl pH 7.5, 1 mM EDTA, 50 mM KCl および 50% グリセロール | ||
| ストレージ バッファ | 20 mM トリス-HCl pH 8.0, 1 mM DTT, 0.1 mM EDTA, 100 mM NaCl および 10% グリセロール | ||
| TBE バッファ | 89 mM トリス-HCl, 89 mM ボリック酸および 2 mM EDTA | ||
| TE100 バッファ | 10 mM トリス.HCl pH 8.0 および 100 mM NaCl | ||
| トリス-EDTA バッファ | 50 mM トリス-HCl pH 8.0 および 1 mM EDTA pH 8.0 | ||
表 1: 本研究で使用したバッファーとその構成のリスト。
| オリゴ | シーケンス | ||
| X0-st1 | ACGCTCGAATCTATAGCGCGCGCGCGTGTGTGTGTGTGTGTCTCTCTCTTCTCTCTCTGCGCGCTGCTCTTCTTCTTCTTCTTCTTCACCC | ||
| X0-st2 | GGGTGAACCTGCAGGGGG/iCy3/AAAGATGTCCCTGTTTTAATCGTCAAGTTTTGCCGT | ||
| X0-st3 | ACGGAAAAGCTTGACGA/iAF647-dT/タカアカガッタガットガットガットガッカトガッカト | ||
| X0-st4 | 5'ビオチンチャガタグツッカッカッカッカグスチャッタッカッカクタクタクタクタクタトCGGCGCGT | ||
| X0-Adj | X0-st1, X0-st2, X0-st3 および X0-st4 | ||
| X0イン・st2 | GGGTGAACCTAGGAGGGGAAGAAGAAGATGTGTGTTGTAATCTTAATCTTCTAATCTCAAGCTTTGCCGT | ||
| X0イン_st4 | 5'ビオチンガットックカッチャッカッカグスタッカグスチャッカッカグ/iCy3/TGGTAGAATCGGCGCGC | ||
| Nk-X0 | X0-st1, X0-st2, X0-nk3a, X0-nk3b および X0-st4 | ||
| X0-nk3a | アッグカタアグットガCGA/iAF647-dT/タカアカガトク | ||
| X0-nk3b | アッガググトグタガガチャッチャク | ||
表 2: SMFRET および EMSA HJ 基板。smFRETおよびEMSAに蛍光標識されたHJの調製に使用されるオリゴヌクレオチドのリスト。オリゴは商業的に得られた。蛍光標識オリゴはHPLC精製され、可能であれば≥60bpのオリゴをPAGE精製した。
ディスカッション
本研究では、GEN130によるHJ分解能の運動性を決定するために、異なるsmFRET技術を実施した。同様のsmFRETアプローチは、DNA複製および修復フラップエンドヌクレアーゼ142、43、44によるダブルフラップDNA立体構造要件および切断に従うために使用された。ここでは、このプロトコルの重要な手順について説明します。サイレン化反応は、湿度の任意の痕跡から自由でなければなりません。PEGを溶解すると、加水分解を避けるために、ペグレーション溶液をシラン化ガラスに迅速に適用する必要があります。マルチチャネルフローセルでは、隣接するチャネル間の漏れを避けるために、粘着シートに閉じ込められた空気を除去する必要があります。PCA溶液は、時間の経過とともに酸化するので、新鮮に調製する必要があります。10 N NaOH の添加は、その間に渦を伴って、ドロップワイズする必要があります。カバースリップ中の蛍光背景は、蛍光標識HJを流す前に最小限にする必要があります。フローセル内のイメージングは、漂白領域のイメージングを避けるために一方向に行われるべきである。ALEX実験では、アクセプターの急速な漂白を避けるために、赤色レーザーのパワーを低減する必要があります。タイムラプス実験では、サイクルタイムは最速のイベントよりも短くする必要があります。
smFRETは、生体分子反応に貴重なリアルタイムの洞察を提供できる敏感な技術です。しかし、この方法にはいくつかの技術的な課題があり、その中で生化学反応中のFRETの測定可能な変化を達成しています。これは、ヒストグラムとタイム トレースで区別可能な状態で十分に分離されたフィーチャを取得するために必要です。多くの場合、smFRETは基板の慎重な設計、蛍敵対とその位置の選択、および基板45の構造変化が小さいためDNA基板におけるFRET変化の増幅を必要とする。FRETを実行するためのもう一つのアプローチは、標識タンパク質46を使用することです。FRETの観察ウィンドウは、ドナーよりも急速に漂白する傾向があるCy5またはAlexa Fluor 647などのアクセプタの安定性によって制限されます(この場合はCy3)。したがって、FRETは、実験期間を延長し、蛍光信号を延長し、信号対雑音比を最大化するための酸素清掃システムの開発に取り組むために、安定した蛍光素を継続的に検索する必要があります47,48.
smFRETのトラブルシューティングのヒントの中には、レーザーパワー、露光時間、サイクル時間、サイクル回数などのイメージングに関連するいくつかのパラメータのバランスをとって、蛍光放出を最大化し、実験期間を延長し、達成することです。酵素ダイナミクスのための適切なサンプリング間隔。酵素ダイナミクスを表す高忠実度の在り時間分布を得るためには、観察時間が長く、光漂白による影響を最小限に抑える必要があります。ALEXは、この方法は、単色FRETと比較して光漂白粒子からの低い寄与を受けるので、より良いヒストグラムを生成します。ただし、ALEX の時間分解能は、単色 FRET の場合よりも低くなります。
最後に、個々の分子の立体構造変化をリアルタイムで検出することに重点を置き、高分解能構造技術(X線結晶学、核磁気共鳴、電子顕微鏡検査)のギャップを埋め合わせ、これは、測定可能なプロパティのアンサンブル平均を生み出す静的条件とバルクメソッドの下で原子分解構造の詳細を提供します。多くの面で、smFRETは、リアルタイムで生物学的システムを研究するための強力な技術であることが証明されています。
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
この研究は、中核的な資金と競争研究賞(CRG3)を通じて、科学技術のキング・アブドゥラ大学によってS.M.H.に支援されました。
資料
| Name | Company | Catalog Number | Comments |
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox) | Sigma-Aldrich | 238813 | |
| 0.1 M sodium bicarbonate buffer | Fisher | 144-55-8 | |
| 10 % Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6275BOX | |
| 100 X TIRF objective | Olympus | NAPO 1.49 | |
| 3,4-dihroxybenzoic acid (PCA) | Sigma-Aldrich | P5630 | |
| 3-aminopropyltriethoxysilane (APTES) | Sigma-Aldrich | 741442 | |
| 6% Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6265BOX | |
| Adhesive sheet | Grace bio-labs | SA-S-1L | |
| Benchtop refrigerated centrifuge | Eppendorf | Z605212 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA 5000 | |
| Bovine Serum Albumin (BSA) | New England Biolabs | B9001S | |
| Calcium Chloride Dihydrate | Sigma-Aldrich | 31307 | |
| cation exchange column | GE healthcare | MonoS (4.6/100) | |
| Cell distruptor | Constant Cell Disruption System | TS5/40/CE/GA | |
| Coomassie Brilliant Blue | MP Biomedicals | 808274 | |
| Cy3 emission filter | Chroma | HQ600/40M-25 | |
| Cy5/Alexa Fluor 647 emission filter | Chroma | HQ700/40M-25 | |
| Dichroic for DV2 filter cube | Photometrics | 630dcxr-18x26 | |
| Dithiothreitol (DTT) | Thermo Scientific | R0861 | |
| Drill | Dremel | 200-1/21 | |
| Electronic cutter | Copam | CP-2500 | |
| EMCCD camera | Hamamatsu | C9100-13 | |
| Epoxy glue | Devcon | 14250 | |
| FPLC Aktapurifier UPC 10 | GE Healthcare | 28406268 | |
| GelQuant.NET software | biochemlabsolutions.com | Version 1.8.2 | |
| GEN1 entry vector | Harvard plasmid repository | HSCD00399935 | |
| Glycerol | Sigma Life Science | G5516 | |
| green laser (emission 532 nm) | Coherent | Compass 315M-100 | |
| Heparin column | GE healthcare | HiTrap Heparin column | |
| HEPES | BDH | BDH4162 | |
| Image splitter | Photometrics | Dualview (DV2) | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Inverted microscope | Olympus | IX81 | |
| Isopropyl-ß-D-thiogalactoside (IPTG) | Goldbio. | 12481C100 | |
| Laser scanner | GE healthcare | Typhoon Trio | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Long pass 532nm filter | Semrock | LPD02-532RU-25 | |
| Magnesium Chloride | Sigma Life Science | M8266 | |
| mPEG | Laysan Bio | mPEG-SVA 5000 | |
| Neutravidin | Pierce | 31000 | |
| Ni-NTA column | GE healthcare | HisTrap FF | |
| NuPAGE 10% Bis-Tris gels | Novex Life technologies | NP0301BOX | |
| NuPAGE 10% Bis-Tris Protein Gels | Thermo Fisher Scientific | NP0302PK2 | |
| Origin software | OriginLab Corporation | Version 8.5 | |
| Phenylmethylsulfonyl fluoride (PMSF) | Alexis Biochemicals | 270-184-G025 | |
| Phosphate-buffered saline | GIBCO | 14190 | |
| Polyethylene Tubing (I.D. 0.76 mm O.D. 1.22mm) | Fisher (Becton Dickinson) | 427416 | |
| Protocatechuate 3,4-dioxygenase (3,4-PCD) | Sigma-Aldrich | P8279-25UN | |
| Quad-band dichroic | Chroma Inc | Z405/488/532/640rpc | |
| red laser (emission 640 nm) | Coherent | Cube 640 100C | |
| Sodium Chloride | Fisher Chemical | S271 | |
| Sorvall RC-6 plus centrifuge | Thermo Fisher Scientific | 46910 | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000 | |
| Syringe pump | Harvard Apparatus | 70-3007 | |
| Teflon tweezers | Rubis | K35A | |
| Tris Base | Promega | H5135 | |
| Ultracentrifuge | Beckman Coulter | Optima L-90K | |
| Ultrafiltration membrane | Millipore | UFC90300 |
参考文献
- Moerner, W. E., Fromm, D. P. Methods of single-molecule fluorescence spectroscopy and microscopy. Review of Scientific Instruments. 74 (8), 3597-3619 (2003).
- Ha, T. Single-molecule fluorescence resonance energy transfer. Methods. 25 (1), 78-86 (2001).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nature Methods. 5 (6), 507-516 (2008).
- Walter, N. G., Huang, C. Y., Manzo, A. J., Sobhy, M. A. Do-it-yourself guide: how to use the modern single-molecule toolkit. Nature Methods. 5 (6), 475-489 (2008).
- Conchello, J. A., Lichtman, J. W. Optical sectioning microscopy. Nature Methods. 2 (12), 920-931 (2005).
- Axelrod, D. Total internal reflection fluorescence microscopy in cell biology. Methods in Enzymology. 361, 1-33 (2003).
- Kim, H. D., et al. Mg2+-dependent conformational change of RNA studied by fluorescence correlation and FRET on immobilized single molecules. Proceedings of the National Academy of Sciences of the United States of America. 99 (7), 4284-4289 (2002).
- Lee, T. H., et al. Measuring the folding transition time of single RNA molecules. Biophysical Journal. 92 (9), 3275-3283 (2007).
- Holliday, R. Mechanism for Gene Conversion in Fungi. Genetical Research. 5 (2), 282-304 (1964).
- West, S. C., et al. The Formation and Resolution of Holliday Junctions during the Recombinational Repair of DNA Damages. Journal of Cellular Biochemistry. , 269-269 (1995).
- Cox, M. M., et al. The importance of repairing stalled replication forks. Nature. 404 (6773), 37-41 (2000).
- West, S. C. Molecular views of recombination proteins and their control. Nature Reviews: Molecular Cell Biology. 4 (6), 435-445 (2003).
- Duckett, D. R., et al. The structure of the Holliday junction, and its resolution. Cell. 55 (1), 79-89 (1988).
- Clegg, R. M., et al. Fluorescence resonance energy transfer analysis of the structure of the four-way DNA junction. Biochemistry. 31 (20), 4846-4856 (1992).
- McKinney, S. A., Declais, A. C., Lilley, D. M., Ha, T. Structural dynamics of individual Holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2003).
- Joo, C., McKinney, S. A., Lilley, D. M., Ha, T. Exploring rare conformational species and ionic effects in DNA Holliday junctions using single-molecule spectroscopy. Journal of Molecular Biology. 341 (3), 739-751 (2004).
- Hyeon, C., Lee, J., Yoon, J., Hohng, S., Thirumalai, D. Hidden complexity in the isomerization dynamics of Holliday junctions. Nature Chemistry. 4 (11), 907-914 (2012).
- Ip, S. C., et al. Identification of Holliday junction resolvases from humans and yeast. Nature. 456 (7220), 357-361 (2008).
- Rass, U., et al. Mechanism of Holliday junction resolution by the human GEN1 protein. Genes & Development. 24 (14), 1559-1569 (2010).
- Liu, Y., et al. Crystal Structure of a Eukaryotic GEN1 Resolving Enzyme Bound to DNA. Cell Reports. 13 (11), 2565-2575 (2015).
- Chan, Y. W., West, S. GEN1 promotes Holliday junction resolution by a coordinated nick and counter-nick mechanism. Nucleic Acids Research. 43 (22), 10882-10892 (2015).
- van Gool, A. J., Hajibagheri, N. M., Stasiak, A., West, S. C. Assembly of the Escherichia coli RuvABC resolvasome directs the orientation of holliday junction resolution. Genes & Development. 13 (14), 1861-1870 (1999).
- Lee, S. H., et al. Human Holliday junction resolvase GEN1 uses a chromodomain for efficient DNA recognition and cleavage. eLife. 4, (2015).
- Chan, Y. W., West, S. C. Spatial control of the GEN1 Holliday junction resolvase ensures genome stability. Nature Communications. 5, 4844(2014).
- Liu, Y., Freeman, A. D., Declais, A. C., Lilley, D. M. J. A monovalent ion in the DNA binding interface of the eukaryotic junction-resolving enzyme GEN1. Nucleic Acids Research. 46 (20), 11089-11098 (2018).
- Zhou, R., et al. Junction resolving enzymes use multivalency to keep the Holliday junction dynamic. Nature Chemical Biology. 15 (3), 269-275 (2019).
- Bellendir, S. P., et al. Substrate preference of Gen endonucleases highlights the importance of branched structures as DNA damage repair intermediates. Nucleic Acids Research. 45 (9), 5333-5348 (2017).
- Sobhy, M. A., et al. Resolution of the Holliday junction recombination intermediate by human GEN1 at the single-molecule level. Nucleic Acids Research. 47 (4), 1935-1949 (2019).
- Sobhy, M. A., et al. Versatile single-molecule multi-color excitation and detection fluorescence set-up for studying biomolecular dynamics. Review of Scientific Instruments. 82 (11), 113702(2011).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Rashid, F., et al. Initial state of DNA-Dye complex sets the stage for protein induced fluorescence modulation. Nature Communications. 10 (1), 2104(2019).
- Sambrook, J., Russell, D. W. Standard ethanol precipitation of DNA in microcentrifuge tubes. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Holden, S. J., et al. Defining the limits of single-molecule FRET resolution in TIRF microscopy. Biophysical Journal. 99 (9), 3102-3111 (2010).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L. Jr, Wiggins, C. H. Learning rates and states from biophysical time series: a Bayesian approach to model selection and single-molecule FRET data. Biophysical Journal. 97 (12), 3196-3205 (2009).
- Kou, S. C., Cherayil, B. J., Min, W., English, B. P., Xie, X. S. Single-molecule Michaelis-Menten equations. Journal of Physical Chemistry B. 109 (41), 19068-19081 (2005).
- Clegg, R. M., Murchie, A. I., Lilley, D. M. The solution structure of the four-way DNA junction at low-salt conditions: a fluorescence resonance energy transfer analysis. Biophysical Journal. 66 (1), 99-109 (1994).
- Pohler, J. R., Duckett, D. R., Lilley, D. M. Structure of four-way DNA junctions containing a nick in one strand. Journal of Molecular Biology. 238 (1), 62-74 (1994).
- Fogg, J. M., Lilley, D. M. Ensuring productive resolution by the junction-resolving enzyme RuvC: large enhancement of the second-strand cleavage rate. Biochemistry. 39 (51), 16125-16134 (2000).
- Sobhy, M. A., Joudeh, L. I., Huang, X., Takahashi, M., Hamdan, S. M. Sequential and multistep substrate interrogation provides the scaffold for specificity in human flap endonuclease 1. Cell Reports. 3 (6), 1785-1794 (2013).
- Rashid, F., et al. Single-molecule FRET unveils induced-fit mechanism for substrate selectivity in flap endonuclease 1. eLife. 6, e21884(2017).
- Zaher, M. S., et al. Missed cleavage opportunities by FEN1 lead to Okazaki fragment maturation via the long-flap pathway. Nucleic Acids Research. 46 (6), 2956-2974 (2018).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31 (5), 1106-1116 (2001).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved