Method Article
Metodi di trasferimento di energia di risonanza a singola molecola per l'analisi in tempo reale della risoluzione dello svincolo di Holliday da gen1
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentato qui è un protocollo per l'esecuzione di trasferimento di energia di risonanza monomolecola di Fàrster per studiare la risoluzione HJ. L'eccitazione alternata a due colori viene utilizzata per determinare le costanti di dissociazione. Il time lapse a colori singolo smFRET viene quindi applicato nei saggi di scissione in tempo reale per ottenere la distribuzione del tempo di dimora prima della risoluzione HJ.
Abstract
I metodi sfusi misurano il comportamento di insieme delle molecole, in cui i tassi di reazione individuali dei passi sottostanti sono mediati in tutta la popolazione. Il trasferimento di energia di risonanza a singola molecola (smFRET) fornisce una registrazione dei cambiamenti conformazionali che avvengono da singole molecole in tempo reale. Pertanto, smFRET è potente nel misurare i cambiamenti strutturali nell'enzima o nel substrato durante il legame e la catalisi. Questo lavoro presenta un protocollo per l'imaging a singola molecola dell'interazione di una giunzione ostilia a quattro vie (HJ) e di endonuclease I (GEN1), un enzima citosolico omologa di ricombinazione. Sono stati presentati anche protocolli sperimentali smFRET a colori e a due colori per seguire la risoluzione dell'HJ di GEN1 in tempo reale. La cinetica della dimerizzazione GEN1 è determinata presso l'HJ, che è stato suggerito per svolgere un ruolo chiave nella risoluzione dell'HJ ed è rimasto sfuggente fino ad ora. Le tecniche qui descritte possono essere ampiamente applicate per ottenere preziose intuizioni meccanicistiche di molti sistemi enzimatico-DNA.
Introduzione
I metodi a singola molecola basati sul rilevamento della fluorescenza forniscono elevati rapporti segnale-rumore1. FRET è una tecnica spettroscopica in grado di misurare le distanze nell'intervallo di 1-10 nm, rendendo questa tecnica come righello molecolare per misurare le distanze nell'intervallo nanometrico2,3. Lo spettro di assorbimento dell'accettatore presenta una sovrapposizione spettrale parziale con lo spettro di emissione del donatore all'estremità della lunghezza d'onda più corta. FRET è mediato dal trasferimento di energia senza radiazioni tra una coppia di donatori e accettanti, mentre l'efficienza del trasferimento di energia dipende dalla distanza e dall'orientamento dell'accettatore4 .
Sono stati implementati diversi approcci per ridurre al minimo lo sfondo e migliorare l'efficienza di rilevamento del segnale di fluorescenza5,6. Un approccio è la microscopia confocale, in cui un foro stenopeico limita il punto di eccitazione a una dimensione inferiore al limite di diffrazione7. Un altro approccio è la fluorescenza totale di riflessione interna (TIRF), che è una tecnica di illuminazione a campo largo in cui la luce è diretta fuori asse sopra un angolo critico8. La luce viene quindi totalmente riflessa internamente all'interfaccia tra il vetro e la soluzione acquosa, generando un'onda evanescente che illumina solo i fluorofori attaccati alla superficie del vetro e impedisce lo sfondo dai fluorofori nel resto la soluzione.
Nella microscopia confocale, le molecole possono essere liberamente diffuso o immobilizzate superficiali. La risoluzione temporale raggiunta può essere entro microsecondi a diversi millisecondi9. Il rilevamento confocale per una singola molecola viene eseguito mediante diodo da valanga a singolo fotone (SPAD) e la scansione punto per punto della regione di interesse10. In TIRF, una serie temporale di poche centinaia di molecole immobilizzate sulla superficie è registrata da un rilevatore bidimensionale a carica bidimensionale (CCD) sensibile alla posizione. Il CCD amplifica il segnale di fluorescenza mediante schermo di fosforo intensificato e piastra a microcanale o moltiplicazione su chip di fotoelettroni (EMCCD). La risoluzione temporale dipende dalla velocità di lettura e dall'efficienza quantistica del CCD e di solito dall'ordine di poche decine di millisecondi6.
HJ è un intermedio centrale nella riparazione e ricombinazione del DNA11,12,13,14. HJ ha due fili continui e due di incrocio che si collegano tra i fili continui senza intersecarsi l'uno con l'altro. HJ esiste in soluzione come conformatori X-stacked, che subiscono la continua isomerizzazione da parte dei fili continui che diventano incrociando e i fili di attraversamento diventando continuo nell'altro conformer15. Isomer preferenza del HJ dipende dalla sequenza di base e ambiente ionico ed è stato ampiamente studiato da FRET16,17,18,19.
GEN120 è una proteina monomerica nella soluzione21 e richiede la dimerizzazione per fendere l'HJ, consentendo così una corretta separazione dei fili ricombinati22,23. La preferenza di accatastamento conformer dell'HJ influenza l'esito della ricombinazione genetica impostando l'orientamento della risoluzione da parte dei risolvi HJ24. Capire come GEN1 lega l'HJ, coordina le due incisioni e assicura che la sua risoluzione completa sia stata tutte oggetto di studio intensivo21,22,23,25,26 ,27,28,29,30.
In questo studio, viene utilizzato un allestimento TIRF basato su obiettivo come descritto in precedenza31. L'eccitazione alternata a due colori (ALEX) viene applicata per determinare i cambiamenti conformazionali al momento dell'interazione di GEN1 con fluoroforo etichettato HJ. ALEX produce istogrammi 2D basati su due parametri ratiometrici di efficienza FRET E, che è donatore-accettatore a seconda della distanza, e il parametro stoichiometria S, che misura la stoichiometria donatore-acceleratore32. ALEX consente lo smistamento di specie fluorescenti in base alle stoichiometrie dei fluorofori, tra cui sola donazione, solo accetto e sottopopolazioni miste. ALEX può estendere l'uso di FRET all'intera gamma e può rilevare le differenze nella luminosità e nell'oligomerizzazione del fluoroforo, nonché monitorare le interazioni macromolecola-ligando33.
Si è scoperto che GEN1 riesce costantemente a risolvere l'HJ entro la durata del complesso GEN1-HJ. I cambiamenti conformazionali dipendenti dal tempo derivano dalle tracce temporali delle singole molecole, mentre gli istogrammi rappresentano la distribuzione delle popolazioni sottostanti. Utilizzando FRET monocolore time-lapse, sono dimostrati rapidi on-rate e tempi di off-rate lenti per il dimero GEN1, che aumentano l'affinità del dimero GEN1 assemblato al primo prodotto di incisione.
Protocollo
1. Preparazione di copricopredi funzionalizzati per la superficie
-
pulizia
- Posizionare cinque copriletti (24 mm x 60 mm) in etanolo all'interno di un barattolo Coplin. Sonicare in etanolo poi in idrossido di potassio da 1 M per 30 min per 3x. Lavare in acetone 3x quindi decant.
-
Silanizzazione
- Preparare una soluzione del 2,8% 3-aminopropyltriethoxysilane (APTES) in acetone. Sigillare la bottiglia APTES con una pellicola di paraffina e conservarla a 4 gradi centigradi.
NOTA: Utilizzare gli occhiali di sicurezza e lavorare sotto un cofano di fumi. Il contenitore della soluzione silano deve essere completamente asciutto e risciacquato da acetone immediatamente prima e dopo aver versato la soluzione silano nel barattolo. - Versare 70 mL della soluzione APTES del 2,8% nel barattolo Coplin contenente i copricapi. Agitare il barattolo per 4 min in uno shaker orbitale.
- Lasciare riposare il barattolo sulla panchina per 5 min, sonicare per 1 min, e infine tenere il barattolo sulla panchina per altri 10 min per la silarzia a reagire con i gruppi idrossili sulla superficie di vetro.
- Spegnire la reazione con l'aggiunta di 1 L di acqua deionizzata versando acqua direttamente nel barattolo per un rapido scambio di solventi. Sciacquare gli scivoli 3x in acqua agitando lateralmente il vaso su una superficie piana.
- Togliere i coperchi dal barattolo e metterli su un vassoio di lamina di alluminio. Cuocere i copricopro in forno a 110 gradi centigradi per 30 min per asciugare i coperchi e curare il silano. Lasciare il vassoio sul banco per le coperturelips per raffreddare a temperatura ambiente.
- Preparare una soluzione del 2,8% 3-aminopropyltriethoxysilane (APTES) in acetone. Sigillare la bottiglia APTES con una pellicola di paraffina e conservarla a 4 gradi centigradi.
-
PEGylation
- Riscaldare il PEG biotinylato e il PEG, conservati a -20 gradi centigradi a temperatura ambiente (RT) per evitare la condensazione dell'umidità all'apertura del contenitore.
- Posizionare cinque copriletti con la superficie silanizzata rivolta verso l'alto su una scatola. Posizionare due vetrini di vetro di copertura (22 mm x 22 mm) come distanziali lungo i bordi delle parentesi insabbiate.
- Una volta riscaldato, fare soluzioni PEG e PEG biotinylati ad un rapporto di 1:100 dollari in 1 mL di soluzione fresca di bicarbonato di sodio da 0,1 M aggiungendo 1,5 mg di PEG biotinylated e 150 mg di PEG in un tubo da 1,5 mL.
- Vorticare il tubo per sciogliere PEG e girare verso il basso per rimuovere le bolle d'aria.
NOTA: Andando avanti da questo passaggio, essere veloce, perché PEG idrolizza in soluzione entro una scala temporale di min. - Applicare rapidamente 100 l della soluzione PEG ad ogni coverslip. Prendi un altro coverslip al forno e posiziona la sua superficie silanizzata superiore a faccia in giù sopra la coverslip con la soluzione PEG, formando così un sandwich in vetro-soluzione-vetro in cui i copricapi non silanizzati da 22 mm x 22 mm consentono ai due coperchi funzionalizzati di essere separati facilmente.
- Incubare i copriletti notturni (16 h) al buio e a RT. Una volta completata l'incubazione, smontare le coverlips, quindi risciacquare 10x utilizzando acqua dionizzata lavando dal lato con una bottiglia di spruzzo.
- Asciugare i copricopertine sotto un flusso di azoto secco. Conservare i copriletti asciutti sotto vuoto.
NOTA: Le diapositive possono essere utilizzate per 1 mese senza degradazione della qualità.
2. Preparazione della cella di flusso
-
Cella di flusso a canale singolo
- Forare due fori con un diametro di 1,22 mm nella parte centrale di una velina di quarzo (50 mm x 20 mm) con i centri situati a 37 mm di distanza e 6,5 mm dal bordo del vetrino (Figura 1A).
- Ritagliare un canale di 41 mm x 2,25 mm in un pezzo da 50 mm x 20 mm di un foglio adesivo doppio utilizzando una fresa elettronica.
- Sbucciare il lato di plastica del coperchio protettivo e allineare i bordi del pezzo con i bordi del vetrino al quarzo. Rimuovere le bolle d'aria intrappolate premendo delicatamente con un paio di pinzette in politetrafluoretilene.
- Sbucciare il lato della carta del pezzo adesivo. Montare il pezzo sulla superficie funzionalizzata della vessilla.
- Tagliare il tubo in polietilene (I.D. 1.22 mm) in una lunghezza di 11 cm per l'ingresso e 25 cm per l'uscita. Inserire il tubo nei fori precedentemente forati come ingresso e presa per la cella di flusso.
- Utilizzare 5 min colla epossidica per sigillare intorno ai bordi dell'interfaccia quarzo-coverslip e intorno ai tubi per l'ingresso e la presa.
- Utilizzare la cella di flusso immediatamente una volta che si asciuga o conservare sotto vuoto asciutto per un uso successivo.
- Sciogliere avidin in PBS ad una concentrazione di 0,03 mg/mL. Filtrare attraverso il filtro di 0,2 m .
- Flusso avidin nella cella di flusso utilizzando una siringa da 1 mL. Utilizzare un'altra siringa riempita con tampone per lavare l'avidino in eccesso. Fare attenzione a non introdurre bolle d'aria durante lo scambio delle siringhe.
-
Cella di flusso a più canali
- Forare sei fori con un diametro di 1,22 mm su ciascuno dei lati lunghi di un vetrino di quarzo (76 mm x 25 mm) (Figura 1B). Rendere i fori a 4,5 mm dal bordo del vetrino e a 9,3 mm di distanza. Assicurarsi che la distanza tra i centri di ogni coppia di fori sia di 15 mm.
- Ritaglia sei canali (20 mm x 2,25 mm) in un pezzo di nastro adesivo doppio da 76 mm x 25 mm utilizzando la fresa elettronica.
- Sbucciare il lato di plastica del coperchio protettivo e allineare i bordi del pezzo adesivo con i bordi del vetrino di quarzo. Rimuovere eventuali bolle d'aria intrappolate premendo delicatamente utilizzando un paio di pinzette in politetrafluoretilene.
- Sbucciare il lato della carta del pezzo adesivo e montare sulla superficie funzionalizzata del coperchio.
NOTA: A volte staccare il lato della carta e attaccarsi al vetrino al quarzo funziona bene nella cella di flusso multicanale. - Tagliare i tubi di uscita (11 cm) e i tubi di uscita (25 cm) per i sei canali. Preparare la cella di flusso come descritto nei passaggi 2.1.6–2.1.9.
- Collegare la presa del primo canale alla pompa. Posizionare l'ingresso nel tubo da 0,5 mL con OSS.
NOTA: La lunghezza del tubo di ingresso viene scelta per massimizzare il numero di eventi negli esperimenti di scissione eseguiti durante il flusso continuo sincronizzando il tempo di ingresso dell'enzima nella cella di flusso e l'inizio dell'imaging diminuendo così il fotobleaching prematuro fluorofori. - Passare a un nuovo canale scollegando la presa del canale utilizzato. Chiudere la presa con una spina fatta da un ago di siringa sigillato con colla nella parte di plastica. Chiudere l'ingresso del canale utilizzato.
3. Preparazione del sistema di scavenging dell'ossigeno (OSS)
- Sciogliere l'acido di 2,5,7,8-tetramethylchromane-2-carboxylic (quencher dello stato tripletta che riduce al minimo le palpebre dei fluorofori) in 800 l.
- Aggiungere 6 mL di deionizzato H2O e aggiungere 1 N NaOH dropwise fino a quando non si scioglie. Filtrare attraverso un filtro di siringa, trasformare in 1 mL aliquote, e conservare a -80 gradi centigradi. La concentrazione di stock è di 100 dollari.
- Preparare una soluzione fresca di 3,4-dihroxybenzoic acid (PCA) sciogliendo 61 mg di polvere DI PCA in 4 mL di ddH2O. La concentrazione di stock è di 100 nM.
- Aggiungete 58 luna di 10 N NaOH, assicurandosi di vortice dopo ogni goccia fino a quando la PCA non sarà completamente dissolta (pH - 9).
- Sciogliere 5,3 mg di protocatechuate 3,4-diossigenasi (3,4-PCD) in 7 mL di buffer di archiviazione PCD (tabella 1). 3,4-PCD rimuove l'ossigeno dai buffer di legame/scissione catalmente l'ossidazione dell'acido protocatechuic34.
- Dividere la soluzione PCD in 1 mL. La concentrazione di stock è di 1 M. Blocca le aliquote in azoto liquido e conserva a -80 gradi centigradi per lo stoccaggio a lungo termine o a -20 gradi centigradi per lo stoccaggio a breve termine.
- Preparare un buffer di rilegatura aggiornato (Tabella 1). Sostituire 2 mM CaCl2 con 2 mM MgCl2 per esperimenti di scissione smFRET.
- Preparare 1 mL del buffer di imaging (Tabella 1). Mantenere il buffer di imaging sul ghiaccio fino a quando non viene introdotto nella cella di flusso per mantenere l'attività del sistema di scavenging dell'ossigeno.
4. Preparazione di HJ etichettati fluorescentmente
- Ricostituire gli oligos lofili (tabella 2) nel buffer Tris-EDTA (Tabella 1) in una concentrazione di 100 M.
- Preparare la giunzione sintetica mescolando porzioni di equimolar 3 dollari l- di ciascuno degli oligo X0 elencati nella tabella 1.
- Anneal da riscaldamento a 95 s per 5 min seguita da raffreddamento lento a RT ad una velocità di 1 C/min. Utilizzare un blocco di calore o un termociclista PCR per raggiungere la velocità di raffreddamento desiderata.
- Caricare la miscela su 8 cm x 8 cm di 10% gel di poliacrilammide Tris-borate-EDTA. Applicare 100 V ed eseguire il gel per 2 h. Le bande sono chiaramente viste a occhio, e il loro colore è viola.
- Acciglia la fascia del substrato annesso con una lama pulita. Trasferire il pezzo di gel in un tubo autoclaved da 1,5 mL.
- Schiacciare il pezzo di gel all'interno del tubo con uno stantuffo pulito, quindi aggiungere 100 L di te100 buffer (Tabella 1).
- Estrarre l'HJ agitando il tubo a 20 gradi centigradi a 1.500 giri/min in un termomixer per 2 h o incubare durante la notte a 4 gradi centigradi.
- Eseguire precipitazioni etanolo sulla soluzione contenente il substrato35.
- Risospendere il substrato in 20 : L di TE100 buffer (tabella 1). La concentrazione finale è di 1–3 M. 2 - l in ogni tubo e conservare a -20 gradi centigradi.
5. Espressione proteica e purificazione di GEN1
- Costruire il plasmide per l'espressione di GEN11,2,3,4,4 con tag issidina esadico al C-terminus20 dalla PCR del vettore di ingresso.
NOTA: l'etichettatura a TerminalE comporterebbe l'inattivazione di GEN1. La coda C non strutturata rende la purificazione dell'intera lunghezza GEN1 significativamente più difficile. Inoltre, è stato segnalato che l'intera lunghezza GEN1 mostra meno attività rispetto alla versione23troncata. - Trasformare il vettore di espressione in ceppo E. coli BL21-CodonPlus (DE3)-RIPL.
- Inoculare le cellule trasformate in due flaconi da 6 L, ciascuno contenente 2 L di mezzi di brodo Luria a 37 gradi centigradi con agitazione a 180 giri/min fino a raggiungere un OD600 di 0,8.
- Raffreddare la coltura a 16 gradi centigradi e indurre l'espressione GEN1 con 0,1 mM di isopropile-z-d-thiogalactopyranoside (IPTG) per 48 h.
- Raccogliere le cellule facendole girare a 4 gradi centigradi a 1000 x g in una centrifuga. Ogni litro di cultura produce 5–6 g di pellet.
- Eliminare il supernatante e sospendere nuovamente le celle pellettate nel buffer di lisi (Tabella 1) utilizzando 4 mL/g di celle.
- Eseguire la lisi cellulare utilizzando un disgregatore di cellule a 30 kPsi poi girare verso il basso a 10.000 x g per 1 h a 4 gradi centigradi. Raccogliere il supernatante e filtrarlo sul ghiaccio utilizzando filtri da 0,45 m.
- Eseguire la purificazione delle proteine utilizzando FPLC passando il filtrato attraverso una colonna Ni-NTA da 5 mL a una velocità di flusso di 2,5 ml/min utilizzando il Buffer A(tabella 1).
- Lavare con 15 volumi di colonne (CV). Eluire con una sfumatura lineare di buffer A e 500 mMIdazolo oltre 20 CV in 5 mL frazioni. GEN1 eluisce dalla colonna a circa 100 mM Imidazole.
- Pipetta 10 -L aliquote dalle frazioni raccolte, aggiungere lo stesso volume di 2x sdS di carico ad ogni aliquota. Denaturare i campioni riscaldando a 90 gradi centigradi per 5 min, raffreddare e far girare verso il basso i campioni.
- Caricare i campioni su gel 10% Bis-Tris. Eseguire il gel per 30–45 min a 200 V. Stain utilizzando Coomassie Brilliant Blue, quindi destain. Raccogliere le frazioni che contengono GEN1 purificato.
- Ridurre la concentrazione di sale delle frazioni combinate a 100 mM mediante diluizione utilizzando il buffer C (Tabella 1).
- Passare la proteina di sale basso attraverso una colonna di eparina di 5 mL ad una portata di 3 mL/min utilizzando il buffer B(Tabella 1).
- Lavare con 10 CV. Elute utilizzando un gradiente di 20 CV con buffer B e 1 M NaCl. Raccogliere 5 frazioni mL in cui GEN1 eluisce circa 360 mM NaCl.
- Controllare le frazioni eluite per le frazioni GEN1 purificate come descritto nel passaggio 5.8. Combinare queste frazioni e diluire a 100 mM NaCl utilizzando buffer C.
- Caricare la proteina salina più bassa su una colonna di scambio di cation a 1 mL/min flow rate utilizzando buffer B.
- Elute con un gradiente di 40 CV utilizzando buffer B e 1 M NaCl. Raccogliere 1,7 mL di frazioni in cui GEN1 si eluisce circa 300 mM NaCl.
- Verificare la purezza di GEN1 nelle frazioni eluite come descritto nel passaggio 5.8.
- Unire le frazioni più pure e dialyze a 4 gradi centigradi rispetto al buffer di archiviazione(tabella 1). Eseguire almeno uno scambio del buffer durante la dialisi.
- Misurare la concentrazione di proteine 0,5–1 mg/mL. La proteina dialisica in 10–15 volumi l in piccoli tubi, flash-freeze in azoto liquido, e conservare a -80 gradi centigradi.
6. Esperimenti FRET a singola molecola
NOTA: gli esperimenti smFRET vengono eseguiti su un set-up TIRF basato su obiettivo personalizzato (Figura 1C) descritto in precedenza31.
- Esperimenti FRET monocolore
- Applicare una goccia di olio per immersione sull'obiettivo TIRF 100x. Impostare l'EMCCD su un guadagno adatto per ottimizzare il segnale sullo sfondo ed evitare la saturazione.
NOTA: Non guardare direttamente nel raggio laser e indossare occhiali protettivi quando si allinea il laser. - Posizionare la cella di flusso con attenzione sul supporto del campione. Sollevare gradualmente l'obiettivo utilizzando la regolazione grossolana fino a quando l'olio tocca il coperchio.
- Accendere il laser verde (532 nm). Passare alla modalità di regolazione fine dell'obiettivo. Dirigere l'emissione alla porta della fotocamera per osservare l'immagine sul monitor.
- Regolare l'altezza dell'obiettivo fino a quando la superficie funzionalizzata del coperchio viene messa a fuoco e può essere osservata sul monitor.
NOTA: l'acquisizione di immagini da parte di EMCCD attiva l'eccitazione laser tramite il filtro regolabile acousto-ottico (AOTF) per evitare il fotosbleaching campione quando le immagini non vengono acquisite. - Verificare che lo sfondo dalla superficie funzionalizzata dei coperchiri non superi pochi punti prima di fluire nell'HJ fluorescente.
- Diluire il substrato di magazzino circa 1000 volte nel buffer di TE100 (tabella 1) per una concentrazione finale di 1-5 nM. Pipetta 0,2–0,5 - L del substrato diluito in 120 - L del buffer di imaging con OSS in un tubo da 0,5 mL.
- Collegare la presa della cella di flusso alla pompa della siringa. Inserire il tubo di ingresso della cella di flusso nel tubo da 1,5 mL e azionare la pompa della siringa ad una portata di 30-50 l/min per ritirare la soluzione dal tubo.
- Controllare spesso la superficie per una buona copertura (100-300 di substrato omogeneamente distribuito e ben distanziato) immaginando brevemente con il laser verde.
- Se la copertura superficiale non è ancora sufficiente, attendere alcuni minuti che l'HJ fluorescente etichettato dalla soluzione si stabilizzi sulla superficie o ripetere il passo che scorre.
- Flusso di altri 120 l di buffer di imaging (tabella 1) a 30-50 l/min per lavare fluorescente senza slegatura etichettato HJ. Quindi lasciare che la cella di flusso si esitano per 5 min per consentire all'OSS di esaurire l'ossigeno disciolto. Il fotobleaching dei fluorofori deve essere minimo all'inizio dell'imaging.
- Impostare il tempo di esposizione (60 ms), il tempo di ciclo verrà impostato automaticamente dal software in base alla velocità di trasferimento dei dati (104 ms) e specificare il numero desiderato di cicli o fotogrammi (400 USD). L'emissione da donatore (Cy3) e accettante (Alexa Fluor 647) è suddivisa in due canali di colore da un dispositivo di divisione dell'immagine.
- Trovare un'area adatta sulla superficie, mettere a fuoco l'immagine regolando l'altezza dell'obiettivo e registrare e salvare il filmato in formato TIFF a 16 bit.
- Spostarsi in una nuova area.
NOTA: Spostarsi sempre in una sola direzione (ad esempio, dalla presa all'uscita) per evitare di imaging della stessa area due volte. - Preparare 1, 2, 5, 10, 25, 50, 75 e 100 nM GEN1 in 120 buffer di imaging uno alla volta. Scorrere la soluzione ad una portata di 30-50 l/min.
NOTA: Se la misurazione richiesta viene eseguita in stato stabile come nell'associazione di HJ da GEN1 o nell'isomerizzazione dell'HJ libero, attendere 3-5 minuti dopo che il flusso si ferma per registrare il filmato. Acquisire da tre a quattro film da nuove aree per ogni concentrazione DI GEN1. - Se la misurazione viene eseguita a flusso continuo come nella scissione di HJ da GEN1, iniziare a registrare 5-10 s prima dell'ingresso di GEN1 nella cella di flusso. Ripetere la misurazione passando a un nuovo canale nella cella di flusso a sei canali.
- Alla fine, utilizzare una diapositiva di perline fluorescente fissa per mappare le particelle donatore e accettatore l'una all'altra nel dispositivo di divisione dell'immagine.
- Aggiungete le albe fluorescenti di 1,l di 1 m di diametro in 500 -L di 1 M Tris (pH - 8,0) per consentire alle perline di aderire alla superficie.
- Tagliare un quadrato (18 mm x 18 mm) all'interno di un pezzo di 22 mm x 22 mm di una guarizione adesiva a doppio lato. Sbucciare e attaccare il pezzo al centro di uno scivolo di quarzo da 76 mm x 25 mm.
- Mettere 50 l della soluzione di luitto perline e lasciare riposare per 5-10 min. Fissare un coperchio di 22 mm x 22 mm sopra il pezzo quadrato. Soluzione di perline in eccesso secche con tessuto, quindi sigillare la camera con colla epossidica.
- Acquisire 100 fotogrammi delle perline scivolare a un tempo di esposizione 60 ms.
AVVISO: Abbassare al minimo la potenza del laser e l'EMCCD per evitare la saturazione del rilevatore. - Installare il pacchetto software (ad esempio, TwoTones) e aprire i filmati in questo, come indicato nel manuale dell'utente36. Selezionare le posizioni delle singole perline nei canali donatore e accettatore. Generare una matrice di trasformazione come descritto nel manuale.
NOTA: Questo software utilizza la matrice di trasformazione per abbinare le posizioni delle particelle nei canali donatore e accettatore e correggere qualsiasi lieve disallineamento nel dispositivo di divisione delle immagini. - Vai a File, premi Carica filmato, quindi seleziona il file del filmato e premi Apri. Nel menu File, premere Carica TFORM e selezionare la matrice di trasformazione generata dalla diapositiva perline. Regolare la soglia per i canali donatore e accettatore fino a quando non sono inclusi falsi positivi.
- Nel menu Filtro canale, scegliete d&&a per selezionare le particelle etichettate sia con il donatore che con l'accettatore. Controllare il campo Limite vicino più vicino per escludere le molecole che sono molto vicine l'una all'altra. Controllare l'ellittica Max per escludere molecole molto eccentriche e controllare i limiti di larghezza per escludere molecole molto larghe o molto strette.
- Digitare plotHistCW come indicato nel manuale Twotones per costruire gli istogrammi.
NOTA: l'efficienza FRET "apparente" viene calcolata per il programma dividendo l'emissione dell'accettatore per le emissioni totali del donatore e dell'accettatore. Twotones utilizza 100 intervalli per collocare la distribuzione degli stati delle molecole contro l'efficienza FRET. - Digitare plotTimetraceCW come indicato nel manuale Twotones per generare le tracce temporali per ogni molecola.
NOTA: le tracce temporali possono essere ulteriormente analizzate da vbFRET37 per identificare diversi stati FRET, i rispettivi tempi di pergolato e i tassi di transizione tra i diversi stati.
- Applicare una goccia di olio per immersione sull'obiettivo TIRF 100x. Impostare l'EMCCD su un guadagno adatto per ottimizzare il segnale sullo sfondo ed evitare la saturazione.
- Esperimenti FRET (ALEX) di eccitazione alternata a due colori
- Registrare un film composto da fotogrammi consecutivi di emissioni di donatori ed accettanti mediante eccitazioni dirette con i laser verdi e rossi, rispettivamente, di durata di 80 ms.
- Aprire i film ALEX acquisiti in Twotones. Impostare la soglia di rilevazione idonea come 300 USD per i tre canali: emissione di donatori dovuta all'eccitazione del donatore (DexDem); emissione di donatori dovuta all'eccitazione del donatore (DexAem); e l'emissione dell'accettatore a causa dell'eccitazione diretta (AexAem).
- Applicare il filtro canale DexDem&&DexAem&&AexAem per selezionare le particelle che hanno sia donatore che accettatore. Collegate le particelle da 200 a 300 dollari nei tre canali.
- Utilizzare il codice plotHistALEX MATLAB per generare gli istogrammi ALEX. Adattare picchi diversi negli istogrammi alle funzioni gaussiane e determinare la percentuale di ogni popolazione l'area sotto la curva utilizzando il software Origin38.
NOTA: i picchi nel saggio di rilegatura corrispondono al complesso GEN1-HJ legato, mentre nell'HJ libero, i picchi rappresentano gli isomeri che si intercambiano. - Utilizzare il codice plotTimetraceALEX MATLAB per generare una traccia temporale per ogni molecola che mostra l'emissione del donatore per eccitazione diretta e le emissioni dell'accettatore dovute a FRET e all'eccitazione diretta.
NOTA: LE tracce temporali ALEX tengono tempo in modo indipendente per mostrare le emissioni sia del donatore che dell'accettatore, ma con una risoluzione temporale inferiore rispetto al FRET monocolore. Simile a FRET monocolore, le tracce temporali ALEX possono essere ulteriormente analizzate da vbFRET per identificare i diversi stati FRET e i rispettivi tempi di pergolato. - Determinare la costante di dissociazione adattando le percentuali della popolazione legata rispetto alla concentrazione di GEN1 a una funzione iperbolica.
- Time lapse monocolore FRET
- Impostare il tempo di esposizione del laser verde a 60 ms e il tempo di ciclo a 624 ms o inferiore, a seconda della velocità della dinamica osservata.
- Impostare la portata su 110 l/min in un canale della cella di flusso a sei canali. Avviare brevemente la registrazione prima dell'ingresso di GEN1 all'interno della cella di flusso.
NOTA: il flusso continuo porta al rapido fotosbiancamento dei fluorofori, sincronizzando quindi l'inizio dell'imaging e l'ingresso delle proteine massimizza il numero di eventi catturati. La lettura ottimale della pompa della siringa dipende dal volume morto e dall'esatto tubo utilizzato per assemblare la cella di flusso; nel nostro caso è di 25 dollari l. - Acquisire un filmato di 125 fotogrammi per un tempo totale di acquisizione di 78 s. Alla fine della registrazione, esporre il campione al laser rosso per 50 fotogrammi ciascuno con tempo di esposizione di 25 ms per sondare l'accettatore.
NOTA: Questo metodo prolunga la finestra di osservazione nell'esperimento di scissione riducendo il numero di cicli di eccitazione. I parametri cinetici come kon e koff of dimerization sono derivati adattando la distribuzione a un modello bi-esponenziale30,38.
7. Saggi di spostamento di mobilità elettroforetico (EMSA)
- In 50 l volume totale, incubare la concentrazione desiderata di GEN1 con 50 pM Cy5 etichettato HJ a RT per 30 min nel buffer di rilegatura EMSA (tabella 1).
- Caricare i campioni su 8 cm x 8 cm di gel Tris-borate-EDTA del 6%. Eseguire il gel utilizzando 100 V per 1 h e 20 min in 1x tampone TBE a RT.
- Determinare la percentuale di substrato legato a una concentrazione di GEN1 dal suo contributo relativo all'intensità totale di fluorescenza della rispettiva corsia.
NOTA: GEN1 monomer-HJ (banda I) è identificato dall'accordo delle sue dimensioni con l'associazione picomolar del monomero GEN1 al Nicked HJ21,30. GEN1-dimer-HJ è assegnato alla banda II a causa dell'associazione stepwise del monomer GEN1 all'HJ21,23. - Calcolare le costanti di associazione apparenti Kd-monomer-app-EMSA e Kd-dimer-app-EMSA utilizzando l'equazione:

Dove: Max è la concentrazione alla quale le rispettive specie hanno raggiunto il suo massimo legame (momero o dimero); n è il coefficiente Hill; K (in vi, d-app-EMSA è l'apparente costante di legame delle rispettive specie, che indica la concentrazione di GEN1 alla quale è presente metà del massimo di monomero o dimero.
Risultati
Conforme pregiudizio e isomerizzazione del HJ
L'isomerizzazione di HJ è stata ampiamente studiata da FRET attraverso l'etichettatura di due bracci adiacenti della giunzione17,18,39. Il donatore (Cy3) e l'accettatore (Alexa Fluor 647) sono posizionati rispettivamente ai due bracci vicini, R (filo 2) e X (filo 3),(Figura 2A). Gli isomeri stacked-X sono stati assegnati dai loro due fili continui [cioè, Iso(1,3) o Iso(2,4)]. L'istogramma ALEX FRET dell'etichetta adiacente X0 mostra due picchi che corrispondono allo scambio dell'iso più abbondante(1,3) (E -0,75) e meno abbondante Iso(2,4) (E .0.40) (Figura 2B).
FRET monocolore viene utilizzato per acquisire tracce temporali per registrare i rapidi cambiamenti conformazionali nell'HJ libero con alta risoluzione temporale di 10 ms riducendo l'area utilizzata della fotocamera EMCCD2. Una traccia temporale FRET a colore singolo rappresentativo di x0 junction mostra le transizioni tra isomeri FRET alto e basso (Figura 2B). I tassi di isomerizzazione kIso(1,3)-Iso(2,4) e kIso(2,4)-Iso(1,3) ottenuti dagli istogrammi del tempo di dimora di Iso(1,3) e Iso(2,4) (Figura 2C) sono coerenti con quelli precedentemente riportati17.
SMFRET dimostra la distorsione attiva dell'HJ da parte di GEN1
HJ subisce un riarrangiamento strutturale al momento del legame con GEN122. Pertanto, la spaziatura tra il donatore e l'accettatore è simile sia in Iso(1,3) che in Iso(2,4) (Figura 3A). I saggi vincolanti smFRET sono stati effettuati in presenza di Ca2 per evitare la scissione dell'HJ. Gli istogrammi FRET della giunzione X0 dell'etichetta adiacente a diverse concentrazioni GEN1 sono stati acquisiti da ALEX(Figura 3B). L'istogramma è adatto a due funzioni gaussiane: una corrispondente all'alto libero FRET Iso(1,3), e l'altra corrispondente alla popolazione GEN1-HJ legata dopo aver sottratto il contributo del Iso(2,4) dal picco basso FRET.
A saturazione della concentrazione di GEN1, l'istogramma FRET di X0 ha un solo picco basso di FRET corrispondente a GEN1 legato a uno dei opiù dell'HJ come previsto dal modello22. L'apparente costante di dissociazione del monomero (Kd-monomer-app) è determinata dall'adattamento iperbolico delle percentuali della popolazione legata a GEN1 in funzione della concentrazione di GEN1 (Figura 3C). L'etichetta adiacente nk-X0 rappresenta una versione sfusa HJ che imita il prodotto dopo la prima reazione di incisione. A causa del rilievo della deformazione di impilamento da parte del nick simulato, nk-X0 è una struttura non isometrice40 come evidente dal picco singolo substrato a E 0,40, a differenza di X0 (Figura 3D vs Figura 3B). La struttura del complesso GEN1-nk-X0 è simile a quella del complesso GEN1-X0, come indicato dalla somiglianza nelle efficienze FRET (E 0,25 per nk-X0 e 0,32 per X0) (Figura 3D vs. Figura 3B). L'associazione avanzata del monomer GEN1 a nk-X0 è dimostrata dal valore Kd-monomer-app inferiore di 40 volte rispetto a quello di X0 (Figura 3E e Figura 3C). Questo attaccamento stretto può fungere da meccanismo di salvaguardia contro la risoluzione incompleta dell'HJ nell'improbabile caso della dissociazione del dimero GEN1 o di uno dei suoi monomeri.
Rilegatura graduale del monomero GEN1 all'HJ
L'associazione del monomero GEN1 all'HJ seguito dalla formazione di dimer è una caratteristica unica per l'eucacrotico HJ resolvase GEN1 rispetto ai risolvasi procariotici, che esistono in forma dimeric nella soluzione21,23,41. EMSA di GEN1 a 50 pM X0 mostra l'associazione stepwise di GEN1 in complessi di ordine superiore, come indicato dai numeri romani nel pannello superiore (Figura 4A). La costante di dissociazione del monomere GEN1 determinata da EMSA (Kd-monomer-EMSA) coincide con la costante di dissociazione dal saggio di legame smFRET Kd-monomer-app (Figura 4A e Figura 3C , rispettivamente). La quantificazione della banda II viene utilizzata per calcolare la costante di dissociazione dell'equilibrio di GEN1 dimer (Kd-dimer-EMSA). EMSA di GEN1 a 50 pM nk-X0 dimostra l'attacco momnotille prominente come indicato dal molto basso Kd-monomer-app-EMSA che è 30 volte inferiore a quello di X0, mentre il suo Kd-dimer-EMSA è paragonabile a quella di X0 (Figura 4B).
Un'ulteriore prova del legame e distorce l'HJ di GEN1 è l'osservazione di un numero significativo di tracce di particelle con stato FRET basso stabile (Figura 4C) in presenza di Mg2 a basse concentrazioni di GEN1. Il numero di queste tracce è diminuito al momento dell'aumento della concentrazione di GEN1. La risoluzione dell'HJ è guidata dalla stretta legatura del monomero GEN1, che supporta la formazione di dimeri. L'associazione del monomero si osserva nelle tracce temporali dello zio nk-X0 in Mg2,che si estende fino a poche concentrazioni di nanomolare (Figura 4D). Il monomero GEN1 si lega strettamente per salvaguardare nk-X0, garantendo alla fine la piena risoluzione attraverso la formazione di dimeri.
Saggio di risoluzione SMFRET dell'HJ
Il termine "scissione" nei saggi smFRET viene utilizzato in modo intercambiabile con la "risoluzione" dell'HJ, poiché in questo esempio viene rilevato solo il rilascio del prodotto che segue il secondo evento di scissione. Gli eventi sono registrati dall'eccitazione a colore singolo time-lapse per ridurre al minimo il fotosbiancamento dell'accettatore fotosensibile durante il tempo di acquisizione di 1,3 min.
Lo schema nella figura 5A illustra le incisioni dei filamenti 1 e 3 di X0 Iso(1,3) dopo la rilegatura e la distorsione da parte di GEN1 di un X0 collegato al vetro funzionalizzato. Sia il donatore che l'accettatore entrano in soluzione con conseguente perdita dei loro segnali dopo la risoluzione HJ. La prima e la seconda incisione sono disaccoppiate in nk-X0, che esemplifica un prototipo per l'HJ parzialmente risolto. Al momento del binding di GEN1, nk-X0 adotta una struttura simile a X0. La risoluzione procede con una singola incisione nel filamento 1, come illustrato nella figura 5B.
La partenza simultanea del donatore e dell'accettatore dopo un basso stato FRET stabile nelle tracce di X0 risolto si è verificata senza l'emergere di un FRET intermedio (E - 0,40 USD) indica che la risoluzione completa avviene entro il ciclo di vita del GEN1-HJ complesso (Figura 5C). Pertanto, questi risultati suggeriscono che la risoluzione HJ si verifica all'interno della durata complessa GEN1-HJ. La risoluzione di nk-X0 procede anche dopo il riarrangiamento strutturale e si conclude con la partenza del duplex che trasporta due fluorofori (Figura 5D) simili a X0.
Cinetica della dimerizzazione GEN1 sul monomere GEN1 legato HJ
Time-lapse smFRET misuraprima della scissione che include principalmente il tempo necessario per la formazione di dimer e la risoluzione dell'HJ dopo la distorsione da parte del monomero GEN1. Applicando questa tecnica, viene fornita una prova diretta a sostegno dell'affermazione che la formazione di dimeri è necessaria per la risoluzione sia di X0 che di nk-X0, dal momento che la distribuzione di ,prima della scissione, è dipendente dalla concentrazione GEN1.
L'apparente tasso della risoluzione HJ (kapp) è definito come l'inverso della media disè prima-scissione alla rispettiva concentrazione di GEN1. Il termine "apparente" viene utilizzato per descrivere il tasso della risoluzione HJ, poiché la possibilità che GEN1 rimanga legata al prodotto dopo la risoluzione HJ non può essere esclusa.
Le funzioni di densità di probabilità (PDF) delle distribuzioniprima della scissione di X0 (Figura 6A) riflettono il tempo per la formazione di dimeri, che è più lungo a basse concentrazioni di GEN1, quindi più breve a concentrazioni di GEN1 più elevate. I tassi di associazione e di dissociazione per il dimer, kon-dimer e koff-dimer, rispettivamente, sono determinati da un modello bi-esponenziale30. Inoltre, i PDF di nk-X0 (Figura 6B) mostrano una distribuzione simile a X0 che indica il requisito per la formazione di dimeri.
La trama della concentrazione di kapp contro GEN1 è stata adattata ad una funzione iperbolica. Le costanti apparenti del tasso di catalisi (kMax-app) di X0 e nk-X0 sono 0,10711 s-1 e 0,231 - 0,036 s-1, rispettivamente (Figura 6C). I grafici di kapp per le giunzioni X0 e nk-X0 si intersecano a concentrazione GEN1 5,6 nM a causa della più veloce k kMax-app e del kpiù lentosul dimero del nick la giunzione intatta.
In sintesi, il relativamenteveloce kon-dimer e lento koff-dimer portano alla progressione della reazione in avanti verso la risoluzione HJ una volta che il dimero è formato. La forte legame del monomero GEN1 alla giunzione nk-X0 costituisce un meccanismo di sicurezza contro qualsiasi improbabile scissione di seconda interruzione o aiuta a raccogliere eventuali HJ incompleti e inrisolti lasciati dalle vie di risoluzione primaria nella cella.
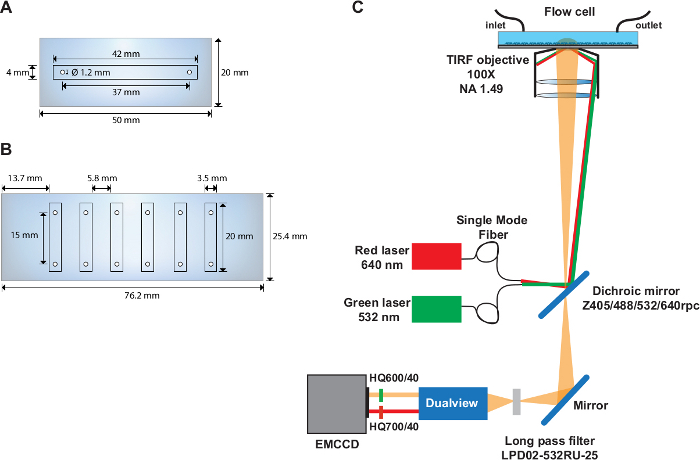
Figura 1: celle di flusso a canale singolo e multicanale e layout del set-up ottico.
(A) Schematico della cella di flusso a canale singolo. (B) Schema della cella di flusso a sei canali. (C) Layout dell'impostazione ottica raffigurante le fonti di eccitazione, l'obiettivo TIRF, lo specchio dicroico installato all'interno del cubo del filtro e i filtri di emissione utilizzati nel dispositivo di divisione delle immagini. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Pregiudizio conformatore e isomerizzazione dell'HJ osservato da FRET.
(A) Isomerizzazione dei conformatori HJ x-stacked etichetta adiacente che prendono il nome dai due fili continui. I fili sono numerati, mentre le braccia sono indicate da lettere. I siti di incisione sono indicati da frecce. Sono indicate le posizioni del donatore (verde) e dell'accettatore (rosso) e il cambiamento in FRET dopo l'isomerizzazione. (B) Pannello destro: traccia temporale FRET (nero) e traccia IDEALizzata FRET (rosso) di X0 a 50 mM Mg2. Pannello a sinistra: Istogramma FRET di X0 a 50 mM Mg2. Di seguito sono riportate le intensità di fluorescenza del donatore (verde) e dell'accettatore (rosso). (C) Gli istogrammi temporali di etichetta adiacente X0 Iso(1,3) e Iso(2,4) sono stati adattati a funzioni unexponential per determinare i tassi di isomerizzazione. Le incertezze indicano l'intervallo di confidenza del 95% dell'adeguato. Questa cifra è stata modificata dalla letteratura pubblicata in precedenza30.
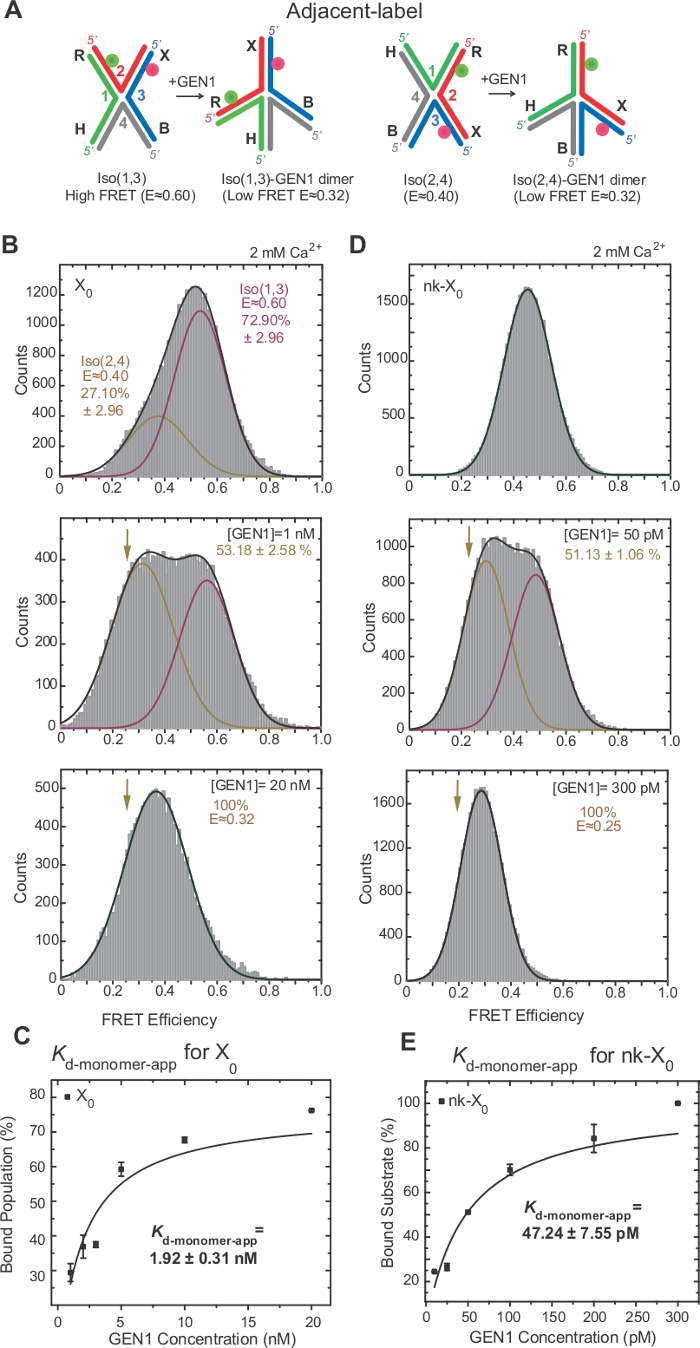
Figura 3: Distorsione attiva dell'HJ di GEN1.
(A) Modifica strutturale dell'etichetta adiacente HJ in base al modello proposto22. (B) L'istogramma ALEX FRET dell'etichetta adiacente X0 ha un picco alto importante di FRET (E - 0,6) corrispondente a Iso(1,3) e un picco FRET inferiore (E - 0,4 USD) per Iso(2,4). L'intero istogramma è adatto a due funzioni gaussiane: una corrispondente all'alto libero FRET Iso(1,3), e l'altra corrispondente alla popolazione vincolata meno il contributo iniziale di Iso(2,4) alla popolazione totale. (C) L'apparente costante di dissociazione del monomero (Kd-monomer-app) è determinata da un effetto iperbolico delle percentuali delle popolazioni legate a GEN1 in funzione della concentrazione di GEN1. (D) ISTOgrammi FRET dell'etichetta adiacente nk-X0 a diverse concentrazioni di GEN1. L'area sotto il basso FRET (E - 0,25) gaussiana corrisponde alla percentuale della popolazione vincolata. (E) L'app Kd-monome di nk-X0 è determinata dall'adattamento iperbolico della popolazione legata a GEN1. Le barre di errore rappresentano le deviazioni standard da due o più esperimenti. Questa cifra è stata modificata dalla letteratura pubblicata in precedenza30. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Rilegatura stepwise di GEN1 all'HJ.
(A) Saggio elettroforetico per il cambio di mobilità (EMSA) di GEN1 a 50 pM X0. Pannello superiore: i numeri romani indicano il numero di monomeri GEN1 nel complesso. Pannello inferiore: rilegatura del monomero GEN1 a X0. Le costanti di dissociazione apparente sono state ottenute da un adattamento sigmoidale delle rispettive specie e rappresentano la media di due esperimenti. (B) EMSA di GEN1 a 50 pM nk-X0 dimostra l'attacco monomero prominente come indicato dal molto basso Kd-monomer-app-EMSA. (C) FRET traccia temporale di legato ma ziviato etichetta adiacente X0 in Mg2 . È stata eseguita l'eccitazione del donatore per 1,3 min, seguita dall'eccitazione diretta dell'accettatore (regione rosa sfumata). (D) FRET traccia temporale di legato ma ziviato adiacente-etichetta nk-X0 in Mg2 . Questa cifra è stata modificata dalla letteratura pubblicata in precedenza30. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: saggio di risoluzione SMFRET dell'HJ.
(A) Schematico dell'etichetta adiacente X0 Iso(1,3) dopo la distorsione di GEN1. Il substrato è attaccato alla superficie funzionalizzata tramite collegamento biotina/avidin. La dissociazione di GEN1 dopo le due incisioni comporta la perdita sia del donatore che dell'accettatore che entrano in soluzione. (B) Schematico della risoluzione dell'etichetta adiacente nk-X0 mediante la filamento 1. (C) Traccia temporale (nera) a 2 mM Mg2o della scissione di Iso(1,3). L'insorgenza dell'associazione GEN1 forma uno stato FRET stabile basso fino a quando il segnale FRET non viene bruscamente perso a causa della scissione. Di conseguenza, l'aumento del donatore e la diminuzione delle intensità di fluorescenza dell'acceleratore al legame GEN1 sono seguiti dalla scomparsa simultanea della fluorescenza da entrambi i coloranti al momento della scissione. (D) Allo stesso modo, la traccia temporale di nk-X0 mostra uno stato FRET stabile e basso dopo il legame GEN1 che si conclude con la brusca perdita del segnale FRET. Questa cifra è stata modificata dalla letteratura pubblicata in precedenza30. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Cinetica di dimerizzazione GEN1 su MOmero GEN1 legato HJ.
(A) La funzione di densità di probabilità (PDF) delladistribuzione di X 0 di X0 illustra la sua dipendenza dalla concentrazione di GEN1. I tempi di permanenza dello stato basso del FRET(prima della scissione) presso la rispettiva concentrazione di GEN1 sono stati ottenuti da due o più esperimenti e utilizzati per ottenere tassi medi (kapp). Le tariffe di kapp elencate sono determinate dall'inverso della media -prima-cleavage alla rispettiva concentrazione di GEN1. I tassi di associazione (kon-dimer) e di dissociazione (koff-dimer) per la formazione dei dimeri sono calcolati da un modello bi-esponenziale38. Gli errori rappresentano SEM di kapp. (B) La trama PDF delle distribuzioniprima della scissione delle tariffe di nk-X0 e kapp. (C) Stampa dellaconcentrazione di kapp contro GEN1 adattata a una funzione iperbolica per determinare l'apparente velocità catalitica (kMax-app). La trama di kapp per X0 e nk-X0 illustral'app kiniziale più veloce di X0 che viene poi superata da nk-X0 sopra [GEN1] -5,6 nM. Questa cifra è stata modificata dalla letteratura pubblicata in precedenza30. Fare clic qui per visualizzare una versione più grande di questa figura.
| buffer m inv | Compostione | ||
| Buffer di associazione | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM CaCl2,1 mM DTT, 0,1% BSA e 5% (v/v) glicerol | ||
| Buffer A | 20 mM Tris-HCl pH 8.0, 1 mM DTT e 300 mM NaCl | ||
| Buffer B | 20 mM Tris-HCl pH 8.0, 1 mM DTT e 100 mM NaCl | ||
| Buffer C | 20 mM Tris-HCl pH 8.0 e 1 mM DTT | ||
| Buffer cleavage | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM MgCl2,1 mM DTT, 0,1% BSA e 5% (v/v) glicerol | ||
| Buffer di binding EMSA | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 1 mM DTT, 2 mM CaCl2, 0,1 mg/ml BSA, 5% (v/v) glicerol e 5 ng/l Poly-dI-dC | ||
| Buffer di imaging (associazione) | 40 L-L-6-Idrossi-2,5,7,8-tetrametilchismo-2-carboxylic acid (4 M), 60 PCA (6 nM), 60 PCD (60 nM) e 840 L di buffer di rilegatura | ||
| Buffer di imaging (scissione) | 40 L-L-6-Idrossi-2,5,7,8-tetrametilchismo-2-carboxylic acid (4 M), 60 PCA (6 nM), 60 PCD (60 nM) e 840 l di tampone Cleavage | ||
| Buffer di lisi | 20 mM Tris-HCl pH 8.0, 10 mM-mercaptoetanolo, 300 mM NaCl e 2 mM PMSF | ||
| Buffer di archiviazione PCD | 100 mM Tris-HCl pH 7,5, 1 mM EDTA, 50 mM KCl e 50% glicerolo | ||
| buffer di archiviazione | 20 mM Tris-HCl pH 8.0, 1 mM DTT, 0,1 mM EDTA, 100 mM NaCl e 10% glicerolo | ||
| Buffer TBE | 89 mM Tris-HCl, 89 mM di acido borico e 2 mM EDTA | ||
| Buffer di TE100 | 10 mM Tris.HCl pH 8.0 e 100 mM NaCl | ||
| Buffer Tris-EDTA | 50 mM Tris-HCl pH 8.0 e 1 mM EDTA pH 8.0 | ||
Tabella 1: L'elenco dei buffer e le relative composizioni utilizzate in questo studio.
| Oligo | sequenza | ||
| X0-st1 | ACGCTGCCGAATTCTACCAGTGCCTGCTAGTAGACATCTGCCCACCTGCAGTCACCCTCTCACCCACCCACCCACCCACCCACCCACCCACCCACCCACCCACCCACCC | ||
| X0-st2 | GGGTGAACCTGCAGGTGGG/iCy3/AAAGATGTCTCTCTTGTACGTCACTCTCTCTCTCTCTCT | ||
| X0-st3 | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATGAGAGMAGAGMAGGAGGAGGGGGGGCTAGAGGATCCGACTATCG | ||
| X0-st4 | 5'BiotinCGATAGTCGGATCCTAGACGCTCCATGTAGCAAGGCACTGGTAGAATTCGGCGGCGCGT | ||
| X0-Adj | X0-st1, X0-st2, X0-st3 & X0-st4 | ||
| X0In_st2 | GGGTGAACCTGCAGGTGGGCAAAGATGTCTCTGTTGTCGTCAAGCTCTTCCCT | ||
| X0In_st4 | 5'BiotinCGATAGTCGGATCCTAGACAGCTCCATGTAGCAAGGCA/iCy3/TGGTAGAATTCGGCAGCGT | ||
| Nk-X0 | X0-st1, X0-st2, X0-nk3a, X0-nk3b & X0-st4 | ||
| X0-nk3a | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATC | ||
| X0-nk3b | ATGGAGCTGTCTAGAGGATCCGACTC | ||
Tabella 2: Substrati SMFRET ed EMSA HJ. L'elenco degli oligonucleotidi utilizzati per la preparazione degli HJ fluorescenti etichettati per smFRET ed EMSA. Gli olighi sono stati ottenuti commercialmente. Gli oligo fluorescenti erano purificati dall'HPLC e, quando possibile, le oligo di 60 bp erano purificate da PAGE.
Discussione
In questo studio sono state implementate diverse tecniche smFRET per determinare la cinetica della risoluzione HJ da GEN130. Approcci smFRET simili sono stati utilizzati per seguire il fabbisogno conformealmente del DNA a doppio lembo e la scissione mediante la replicazione del DNA e la riparazione del lembo endonuclease 142,43,44. In questo caso vengono illustrati i passaggi critici di questo protocollo. La reazione di insilanizzazione dovrebbe essere libera da qualsiasi traccia di umidità. La soluzione di pegilazione deve essere applicata rapidamente al vetro silanizzato una volta che il PEG viene sciolto per evitare l'idrolisi. Nella cella di flusso multicanale, qualsiasi aria intrappolata nel foglio adesivo deve essere rimossa per evitare perdite tra i canali vicini. La soluzione PCA deve essere appena preparata in quanto si ossida nel tempo. L'aggiunta di 10 N NaOH dovrebbe essere dropwise, con vortice in mezzo. Lo sfondo a fluorescenza nel coperchio deve essere minimo prima di fluorescenza etichettato HJ. L'imaging nella cella di flusso deve essere eseguito in una direzione per evitare l'imaging di aree sbiancate. Negli esperimenti ALEX, la potenza del laser rosso dovrebbe essere ridotta per evitare uno sbiancamento rapido dell'accettatore. Negli esperimenti time-lapse, il tempo di ciclo deve essere più breve dell'evento più veloce.
smFRET è una tecnica sensibile che può fornire preziose intuizioni in tempo reale nelle reazioni biomolecolari. Tuttavia, questo metodo ha diverse sfide tecniche, tra le quali sta ottenendo un cambiamento misurabile in FRET durante la reazione biochimica. Ciò è necessario per ottenere caratteristiche ben separate negli istogrammi e negli stati distinguibili nelle tracce temporali. In molti casi, smFRET richiede un'attenta progettazione dei substrati, selezione delle coppie di fluorofori e delle loro posizioni, e amplificazione dei cambiamenti FRET nel substrato di DNA a causa dei piccoli cambiamenti strutturali nel substrato45. Un altro approccio per l'esecuzione di FRET è quello di utilizzare proteine etichettate46. La finestra di osservazione in FRET è limitata dalla stabilità dell'accettatore come Cy5 o Alexa Fluor 647 che tende a sbiancarsi più rapidamente del donatore (Cy3 in questo caso). Pertanto, FRET richiede una continua ricerca di fluorofori stabili per estendere la durata dell'esperimento e gli sforzi per sviluppare sistemi di scavenging di ossigeno per prolungare il segnale di fluorescenza e massimizzare il rapporto segnale-rumore47,48 .
Tra i suggerimenti per la risoluzione dei problemi in smFRET c'è il bilanciamento dei diversi parametri coinvolti nell'imaging come la potenza laser, il tempo di esposizione, il tempo di ciclo e il numero di cicli per massimizzare l'emissione di fluorescenza, prolungare la durata dell'esperimento e raggiungere adeguati intervalli di campionamento per la dinamica enzimatica. Tempi di osservazione più lunghi e effetti minimi derivanti dal fotosbiancamento sono essenziali per ottenere distribuzioni di tempo di perazione ad alta fedeltà che rappresentano la dinamica degli enzimi. ALEX genera istogrammi migliori poiché questo metodo è sottoposto a minori contributi da particelle fotosbiancate rispetto al FRET monocolore. Tuttavia, la risoluzione temporale in ALEX è inferiore a quella in FRET monocolore.
Infine, l'enfasi di smFRET sulla rilevazione di cambiamenti conformi/strutturali nelle singole molecole in tempo reale colma il divario tra le tecniche strutturali ad alta risoluzione (cioè la cristallografia a raggi X, la risonanza magnetica nucleare, la microscopia elettronica), che fornisce dettagli strutturali a risoluzione atomica in condizioni statiche e metodi di massa che producono la media dell'insieme di una proprietà misurabile. In molti aspetti, smFRET si è dimostrata una potente tecnica per studiare i sistemi biologici in tempo reale.
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da King Abdullah University of Science and Technology attraverso il finanziamento di base e il Premio di ricerca competitiva (CRG3) a S. M. H.
Materiali
| Name | Company | Catalog Number | Comments |
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox) | Sigma-Aldrich | 238813 | |
| 0.1 M sodium bicarbonate buffer | Fisher | 144-55-8 | |
| 10 % Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6275BOX | |
| 100 X TIRF objective | Olympus | NAPO 1.49 | |
| 3,4-dihroxybenzoic acid (PCA) | Sigma-Aldrich | P5630 | |
| 3-aminopropyltriethoxysilane (APTES) | Sigma-Aldrich | 741442 | |
| 6% Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6265BOX | |
| Adhesive sheet | Grace bio-labs | SA-S-1L | |
| Benchtop refrigerated centrifuge | Eppendorf | Z605212 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA 5000 | |
| Bovine Serum Albumin (BSA) | New England Biolabs | B9001S | |
| Calcium Chloride Dihydrate | Sigma-Aldrich | 31307 | |
| cation exchange column | GE healthcare | MonoS (4.6/100) | |
| Cell distruptor | Constant Cell Disruption System | TS5/40/CE/GA | |
| Coomassie Brilliant Blue | MP Biomedicals | 808274 | |
| Cy3 emission filter | Chroma | HQ600/40M-25 | |
| Cy5/Alexa Fluor 647 emission filter | Chroma | HQ700/40M-25 | |
| Dichroic for DV2 filter cube | Photometrics | 630dcxr-18x26 | |
| Dithiothreitol (DTT) | Thermo Scientific | R0861 | |
| Drill | Dremel | 200-1/21 | |
| Electronic cutter | Copam | CP-2500 | |
| EMCCD camera | Hamamatsu | C9100-13 | |
| Epoxy glue | Devcon | 14250 | |
| FPLC Aktapurifier UPC 10 | GE Healthcare | 28406268 | |
| GelQuant.NET software | biochemlabsolutions.com | Version 1.8.2 | |
| GEN1 entry vector | Harvard plasmid repository | HSCD00399935 | |
| Glycerol | Sigma Life Science | G5516 | |
| green laser (emission 532 nm) | Coherent | Compass 315M-100 | |
| Heparin column | GE healthcare | HiTrap Heparin column | |
| HEPES | BDH | BDH4162 | |
| Image splitter | Photometrics | Dualview (DV2) | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Inverted microscope | Olympus | IX81 | |
| Isopropyl-ß-D-thiogalactoside (IPTG) | Goldbio. | 12481C100 | |
| Laser scanner | GE healthcare | Typhoon Trio | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Long pass 532nm filter | Semrock | LPD02-532RU-25 | |
| Magnesium Chloride | Sigma Life Science | M8266 | |
| mPEG | Laysan Bio | mPEG-SVA 5000 | |
| Neutravidin | Pierce | 31000 | |
| Ni-NTA column | GE healthcare | HisTrap FF | |
| NuPAGE 10% Bis-Tris gels | Novex Life technologies | NP0301BOX | |
| NuPAGE 10% Bis-Tris Protein Gels | Thermo Fisher Scientific | NP0302PK2 | |
| Origin software | OriginLab Corporation | Version 8.5 | |
| Phenylmethylsulfonyl fluoride (PMSF) | Alexis Biochemicals | 270-184-G025 | |
| Phosphate-buffered saline | GIBCO | 14190 | |
| Polyethylene Tubing (I.D. 0.76 mm O.D. 1.22mm) | Fisher (Becton Dickinson) | 427416 | |
| Protocatechuate 3,4-dioxygenase (3,4-PCD) | Sigma-Aldrich | P8279-25UN | |
| Quad-band dichroic | Chroma Inc | Z405/488/532/640rpc | |
| red laser (emission 640 nm) | Coherent | Cube 640 100C | |
| Sodium Chloride | Fisher Chemical | S271 | |
| Sorvall RC-6 plus centrifuge | Thermo Fisher Scientific | 46910 | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000 | |
| Syringe pump | Harvard Apparatus | 70-3007 | |
| Teflon tweezers | Rubis | K35A | |
| Tris Base | Promega | H5135 | |
| Ultracentrifuge | Beckman Coulter | Optima L-90K | |
| Ultrafiltration membrane | Millipore | UFC90300 |
Riferimenti
- Moerner, W. E., Fromm, D. P. Methods of single-molecule fluorescence spectroscopy and microscopy. Review of Scientific Instruments. 74 (8), 3597-3619 (2003).
- Ha, T. Single-molecule fluorescence resonance energy transfer. Methods. 25 (1), 78-86 (2001).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nature Methods. 5 (6), 507-516 (2008).
- Walter, N. G., Huang, C. Y., Manzo, A. J., Sobhy, M. A. Do-it-yourself guide: how to use the modern single-molecule toolkit. Nature Methods. 5 (6), 475-489 (2008).
- Conchello, J. A., Lichtman, J. W. Optical sectioning microscopy. Nature Methods. 2 (12), 920-931 (2005).
- Axelrod, D. Total internal reflection fluorescence microscopy in cell biology. Methods in Enzymology. 361, 1-33 (2003).
- Kim, H. D., et al. Mg2+-dependent conformational change of RNA studied by fluorescence correlation and FRET on immobilized single molecules. Proceedings of the National Academy of Sciences of the United States of America. 99 (7), 4284-4289 (2002).
- Lee, T. H., et al. Measuring the folding transition time of single RNA molecules. Biophysical Journal. 92 (9), 3275-3283 (2007).
- Holliday, R. Mechanism for Gene Conversion in Fungi. Genetical Research. 5 (2), 282-304 (1964).
- West, S. C., et al. The Formation and Resolution of Holliday Junctions during the Recombinational Repair of DNA Damages. Journal of Cellular Biochemistry. , 269-269 (1995).
- Cox, M. M., et al. The importance of repairing stalled replication forks. Nature. 404 (6773), 37-41 (2000).
- West, S. C. Molecular views of recombination proteins and their control. Nature Reviews: Molecular Cell Biology. 4 (6), 435-445 (2003).
- Duckett, D. R., et al. The structure of the Holliday junction, and its resolution. Cell. 55 (1), 79-89 (1988).
- Clegg, R. M., et al. Fluorescence resonance energy transfer analysis of the structure of the four-way DNA junction. Biochemistry. 31 (20), 4846-4856 (1992).
- McKinney, S. A., Declais, A. C., Lilley, D. M., Ha, T. Structural dynamics of individual Holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2003).
- Joo, C., McKinney, S. A., Lilley, D. M., Ha, T. Exploring rare conformational species and ionic effects in DNA Holliday junctions using single-molecule spectroscopy. Journal of Molecular Biology. 341 (3), 739-751 (2004).
- Hyeon, C., Lee, J., Yoon, J., Hohng, S., Thirumalai, D. Hidden complexity in the isomerization dynamics of Holliday junctions. Nature Chemistry. 4 (11), 907-914 (2012).
- Ip, S. C., et al. Identification of Holliday junction resolvases from humans and yeast. Nature. 456 (7220), 357-361 (2008).
- Rass, U., et al. Mechanism of Holliday junction resolution by the human GEN1 protein. Genes & Development. 24 (14), 1559-1569 (2010).
- Liu, Y., et al. Crystal Structure of a Eukaryotic GEN1 Resolving Enzyme Bound to DNA. Cell Reports. 13 (11), 2565-2575 (2015).
- Chan, Y. W., West, S. GEN1 promotes Holliday junction resolution by a coordinated nick and counter-nick mechanism. Nucleic Acids Research. 43 (22), 10882-10892 (2015).
- van Gool, A. J., Hajibagheri, N. M., Stasiak, A., West, S. C. Assembly of the Escherichia coli RuvABC resolvasome directs the orientation of holliday junction resolution. Genes & Development. 13 (14), 1861-1870 (1999).
- Lee, S. H., et al. Human Holliday junction resolvase GEN1 uses a chromodomain for efficient DNA recognition and cleavage. eLife. 4, (2015).
- Chan, Y. W., West, S. C. Spatial control of the GEN1 Holliday junction resolvase ensures genome stability. Nature Communications. 5, 4844(2014).
- Liu, Y., Freeman, A. D., Declais, A. C., Lilley, D. M. J. A monovalent ion in the DNA binding interface of the eukaryotic junction-resolving enzyme GEN1. Nucleic Acids Research. 46 (20), 11089-11098 (2018).
- Zhou, R., et al. Junction resolving enzymes use multivalency to keep the Holliday junction dynamic. Nature Chemical Biology. 15 (3), 269-275 (2019).
- Bellendir, S. P., et al. Substrate preference of Gen endonucleases highlights the importance of branched structures as DNA damage repair intermediates. Nucleic Acids Research. 45 (9), 5333-5348 (2017).
- Sobhy, M. A., et al. Resolution of the Holliday junction recombination intermediate by human GEN1 at the single-molecule level. Nucleic Acids Research. 47 (4), 1935-1949 (2019).
- Sobhy, M. A., et al. Versatile single-molecule multi-color excitation and detection fluorescence set-up for studying biomolecular dynamics. Review of Scientific Instruments. 82 (11), 113702(2011).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Rashid, F., et al. Initial state of DNA-Dye complex sets the stage for protein induced fluorescence modulation. Nature Communications. 10 (1), 2104(2019).
- Sambrook, J., Russell, D. W. Standard ethanol precipitation of DNA in microcentrifuge tubes. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Holden, S. J., et al. Defining the limits of single-molecule FRET resolution in TIRF microscopy. Biophysical Journal. 99 (9), 3102-3111 (2010).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L. Jr, Wiggins, C. H. Learning rates and states from biophysical time series: a Bayesian approach to model selection and single-molecule FRET data. Biophysical Journal. 97 (12), 3196-3205 (2009).
- Kou, S. C., Cherayil, B. J., Min, W., English, B. P., Xie, X. S. Single-molecule Michaelis-Menten equations. Journal of Physical Chemistry B. 109 (41), 19068-19081 (2005).
- Clegg, R. M., Murchie, A. I., Lilley, D. M. The solution structure of the four-way DNA junction at low-salt conditions: a fluorescence resonance energy transfer analysis. Biophysical Journal. 66 (1), 99-109 (1994).
- Pohler, J. R., Duckett, D. R., Lilley, D. M. Structure of four-way DNA junctions containing a nick in one strand. Journal of Molecular Biology. 238 (1), 62-74 (1994).
- Fogg, J. M., Lilley, D. M. Ensuring productive resolution by the junction-resolving enzyme RuvC: large enhancement of the second-strand cleavage rate. Biochemistry. 39 (51), 16125-16134 (2000).
- Sobhy, M. A., Joudeh, L. I., Huang, X., Takahashi, M., Hamdan, S. M. Sequential and multistep substrate interrogation provides the scaffold for specificity in human flap endonuclease 1. Cell Reports. 3 (6), 1785-1794 (2013).
- Rashid, F., et al. Single-molecule FRET unveils induced-fit mechanism for substrate selectivity in flap endonuclease 1. eLife. 6, e21884(2017).
- Zaher, M. S., et al. Missed cleavage opportunities by FEN1 lead to Okazaki fragment maturation via the long-flap pathway. Nucleic Acids Research. 46 (6), 2956-2974 (2018).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31 (5), 1106-1116 (2001).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon