Method Article
Ein-Molekül Förster Resonanz Energietransfermethoden für Die Echtzeit-Untersuchung der Holliday Junction Resolution von GEN1
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll zur Durchführung der einmolekularen Förster-Resonanzenergieübertragung zur Untersuchung der HJ-Auflösung vorgestellt. Zur Bestimmung der Dissoziationskonstanten wird eine zweifarbige Wechselanregung verwendet. Einfarbigezeitraffe smFRET wird dann in Echtzeit-Spaltungstests angewendet, um die Verweilzeitverteilung vor hj-Auflösung zu erhalten.
Zusammenfassung
Massenmethoden messen das Ensembleverhalten von Molekülen, bei denen die individuellen Reaktionsraten der zugrunde liegenden Schritte in der gesamten Bevölkerung gemittelt werden. Die einmolekulare Förster-Resonanzenergieübertragung (smFRET) ermöglicht die Aufzeichnung der konformen Veränderungen, die von einzelnen Molekülen in Echtzeit stattfinden. Daher ist smFRET bei der Messung struktureller Veränderungen im Enzym oder Substrat während der Bindung und Katalyse leistungsfähig. Diese Arbeit stellt ein Protokoll zur Einzelmolekül-Bildgebung der Wechselwirkung einer Vier-Wege-Holliday-Kreuzung (HJ) und Spaltendonuklease I (GEN1), eines zytosolischen homologen Rekombinationsenzyms, vor. Ebenfalls vorgestellt werden einfarbige und zweifarbige abwechselnde Anregung (ALEX) smFRET Experimentelle Protokolle, um die Auflösung des HJ durch GEN1 in Echtzeit zu folgen. Die Kinetik der GEN1-Dimerisierung wird am HJ bestimmt, das eine Schlüsselrolle bei der Auflösung des HJ spielen soll und bis heute schwer fassbar ist. Die hier beschriebenen Techniken können weit verbreitet sein, um wertvolle mechanistische Erkenntnisse vieler Enzym-DNA-Systeme zu erhalten.
Einleitung
Einzelmolekül-Methoden auf Basis der Fluoreszenzdetektion bieten hohe Signal-Rausch-Verhältnisse1. FRET ist eine spektroskopische Technik, die Entfernungen im Bereich von 1–10 nm messen kann, wodurch diese Technik als molekulares Lineal zur Messung von Entfernungen im Nanometerbereich2,3dargestellt wird. Das Absorptionsspektrum des Akzeptors hat eine partielle spektrale Überlappung mit dem Emissionsspektrum des Spenders am kürzeren Wellenlängenende. FRET wird durch den strahlungsfreien Energietransfer zwischen Spender und Akzeptorpaar vermittelt, während die Effizienz der Energieübertragung von der Entfernung und Ausrichtung des Akzeptorsabhängt 4.
Mehrere Ansätze wurden implementiert, um den Hintergrund zu minimieren und die Detektionseffizienz des Fluoreszenzsignals5,6zu verbessern. Ein Ansatz ist die konfokale Mikroskopie, bei der ein Loch den Anregungspunkt auf eine Größe unterhalb der Beugungsgrenze7beschränkt. Ein weiterer Ansatz ist die totale interne Reflexionsfluoreszenz (TIRF), eine Weitfeldbeleuchtungstechnik, bei der das Licht außerhalb der Achse über einen kritischen Winkel8gerichtet wird. Das Licht wird dann an der Schnittstelle zwischen Glas und wässriger Lösung vollständig nach innen reflektiert, wodurch eine evaneszente Welle entsteht, die nur die an der Glasoberfläche befestigten Fluorophore beleuchtet und den Hintergrund der Fluorophore im Rest der die Lösung.
In der konfokalen Mikroskopie können die Moleküle entweder frei diffundiert oder oberflächenimmobilisiert werden. Die erreichte zeitliche Auflösung kann innerhalb von Mikrosekunden bis zu mehreren Millisekunden9liegen. Der konfokale Nachweis für ein einzelnes Molekül erfolgt durch Einphotonenlawinendiode (SPAD) und Punkt-für-Punkt-Scannen des Interessenbereichs10. In TIRF wird eine Zeitreihe von einigen hundert Molekülen, die auf der Oberfläche immobilisiert sind, von einem positionsempfindlichen zweidimensionalen Ladungs-gekoppelten Detektor (CCD) aufgezeichnet. Das CCD verstärkt das Fluoreszenzsignal entweder durch verstärkte Phosphorsieb und Mikrokanalplatte oder on-chip-Multiplikation von Photoelektronen (EMCCD). Die zeitliche Auflösung ist abhängig von der Auslesegeschwindigkeit und Quanteneffizienz des CCD und in der Regel in der Größenordnung von wenigen Dutzend Millisekunden6.
HJ ist ein zentrales Zwischenprodukt in der DNA-Reparatur und -Rekombination11,12,13,14. HJ hat zwei durchgehende und zwei kreuzende Stränge, die sich zwischen den durchgehenden Strängen verbinden, ohne sich gegenseitig zu überschneiden. HJ existiert in Lösung als X-gestapelte Konformere, die durch die kontinuierlichen Stränge, die sich kreuzen und die Kreuzungsstränge in den anderen Konformerenkontinuierlich werden, kontinuierlichwerden 15 . Die Isomerpräferenz des HJ ist abhängig von der Kernsequenz und der ionischen Umgebung und wurde von FRET16,17,18,19ausgiebig untersucht.
GEN120 ist ein monomeres Protein in Lösung21 und erfordert eine Dimerisierung, um die HJ zu spalten, wodurch eine ordnungsgemäße Trennung der rekombinierten Stränge22,23ermöglicht wird. Die Stapelkonformerpräferenz des HJ beeinflusst das Ergebnis der genetischen Rekombination, indem die Ausrichtung der Auflösung durch die HJ-Resolvasen24gesetzt wird. Verstehen, wie GEN1 die HJ bindet, koordiniert die beiden Einschnitte und stellt sicher, dass seine volle Auflösung alle unter intensiver Studie21,22,23,25,26 wurden ,27,28,29,30.
In dieser Studie wird eine objektive TIRF-Einrichtung verwendet, wie zuvorbeschrieben 31. Zweifarbige Wechselanregung (ALEX) wird angewendet, um die konformen Veränderungen bei der Wechselwirkung von GEN1 mit Fluorophor mit der Bezeichnung HJ zu bestimmen. ALEX produziert 2D-Histogramme auf der Grundlage von zwei ratiometrischen Parametern FRET-Effizienz E, die spender-akzeptierweise distanzabhängig ist, und dem Stoichiometrieparameter S, der den Spender-Akzeptor-Stoichiometrie32misst. ALEX ermöglicht die Sortierung fluoreszierender Arten auf der Grundlage der Stoichiometrien der Fluorophore, einschließlich nur für Spender, nur für Akzeptoren und gemischte Subpopulationen. ALEX kann den Einsatz von FRET auf den gesamten Bereich ausdehnen und Unterschiede in der Fluorophorhelligkeit und Oligomerisierung erkennen sowie Makromolekül-Liganden-Wechselwirkungen überwachen33.
Es wird festgestellt, dass es GEN1 konsequent gelingt, die HJ innerhalb der Lebensdauer des GEN1-HJ-Komplexes zu lösen. Die zeitabhängigen Konformationsänderungen werden aus den Zeitspuren einzelner Moleküle abgeleitet, während die Histogramme die Verteilung der zugrunde liegenden Populationen darstellen. Mit dem zeitraffigen einfarbigen FRET werden schnelle Einschaltraten und langsame Off-Raten für den GEN1-Dimer demonstriert, die die Affinität des montierten GEN1-Dimers beim ersten Schnittprodukt erhöhen.
Protokoll
1. Herstellung von oberflächenfunktionalisierten Abdeckungen
-
Reinigung
- Fünf Deckel (24 mm x 60 mm) in Ethanol in ein Coplin-Glas geben. In Ethanol dann in 1 M Kaliumhydroxid für 30 min für 3x beschallen. In Aceton 3x waschen und dekantieren.
-
Silanisierung
- Bereiten Sie eine Lösung von 2,8% 3-Aminopropyltriethoxysilan (APTES) in Aceton vor. Die APTES-Flasche mit einer Paraffinfolie versiegeln und bei 4 °C lagern.
HINWEIS: Verwenden Sie eine Schutzbrille und arbeiten Sie unter einer Dunstabzugshaube. Der Behälter der Silanlösung sollte vollständig trocken sein und unmittelbar vor und nach dem Gießen der Silanlösung in das Glas mit Aceton abspülen. - Gießen Sie 70 ml der 2,8% APTES-Lösung in das Coplin-Glas mit den Abdeckungen. Schütteln Sie das Glas für 4 min in einem Orbital-Shaker.
- Lassen Sie das Glas für 5 min auf der Bank stehen, beschallen Sie 1 min und halten Sie das Glas schließlich noch 10 min auf der Bank, damit die Silane mit den Hydroxylgruppen auf der Glasoberfläche reagiert.
- Die Reaktion durch Zugabe von 1 L entionisiertem Wasser durch Gießen von Wasser direkt in das Glas für einen schnellen Lösungsmittelaustausch ablöschen. Spülen Sie die Rutschen 3x in Wasser durch seitliches Schütteln des Glases auf einer ebenen Oberfläche.
- Nehmen Sie die Abdeckungen aus dem Glas und legen Sie sie auf ein Aluminiumfolienfach. Die Deckelinen im Ofen bei 110 °C 30 min backen, um die Deckelzulchen zu trocknen und die Silane auszuhärten. Lassen Sie das Fach auf der Bank, damit die Abdeckungen auf Raumtemperatur abkühlen.
- Bereiten Sie eine Lösung von 2,8% 3-Aminopropyltriethoxysilan (APTES) in Aceton vor. Die APTES-Flasche mit einer Paraffinfolie versiegeln und bei 4 °C lagern.
-
PEGylation
- Erwärmen Sie die biotinylierten PEG und PEG, die bei -20 °C auf Raumtemperatur (RT) gelagert werden, um die Kondensation von Feuchtigkeit beim Öffnen des Behälters zu verhindern.
- Legen Sie fünf Abdeckungen mit der silanisierten Oberfläche nach oben auf eine Box. Legen Sie zwei Abdeckungsglasschlupf (22 mm x 22 mm) als Abstandshalter an den Rändern der silanisierten Abdeckungen.
- Nach dem Aufwärmen biotinylierte PEG- und PEG-Lösungen im Verhältnis von 1:100 USD in 1 ml frischer, 0,1 M Natriumbicarbonatlösung herstellen, indem Sie 1,5 mg biotinylierte PEG und 150 mg PEG in ein 1,5 ml-Rohr geben.
- Wirbel nisch das Rohr PEG zu lösen und drehen nach unten, um Luftblasen zu entfernen.
ANMERKUNG: Gehen Sie von diesem Schritt, schnell sein, weil PEG hydrolysiert in Lösung innerhalb einer Zeitskala von min. - Tragen Sie schnell 100 l der PEG-Lösung auf jeden Deckelaufschlag auf. Nehmen Sie einen weiteren gebackenen Deckelschlupf und legen Sie die obere silanisierte Oberfläche mit der PEG-Lösung nach unten auf den Deckel, wodurch ein Glas-Lösungs-Glas-Sandwich entsteht, in dem die 22 mm x 22 mm nicht silanisierten Abdeckungen die beiden funktionalisierten Abdeckungen leicht getrennt werden.
- Inkubieren Sie die Abdeckungen über Nacht (16 h) im Dunkeln und bei RT. Sobald die Inkubation abgeschlossen ist, nehmen Sie die Abdeckungen auseinander, dann spülen Sie 10x mit entionisiertem Wasser durch Waschen von der Seite mit einer Spritzflasche.
- Trocknen Sie die Abdeckungen unter einem Fluss von trockenem Stickstoff. Bewahren Sie die trockenen Abdeckungen unter Vakuum auf.
HINWEIS: Die Folien können 1 Monat lang ohne Qualitätsverlust verwendet werden.
2. Vorbereitung der Durchflusszelle
-
Einkanalige Durchflusszelle
- Bohren Sie zwei Löcher mit 1,22 mm Durchmesser im mittleren Teil eines Quarzschlittens (50 mm x 20 mm), wobei die Mitten 37 mm voneinander entfernt und 6,5 mm vom Rand des Schlittens entfernt sind (Abbildung 1A).
- Schneiden Sie einen 41 mm x 2,25 mm Kanal mit einem elektronischen Fräser in ein 50 mm x 20 mm Stück einer Doppelklebefolie aus.
- Ziehen Sie die Kunststoffseite der Schutzhülle ab und richten Sie die Ränder des Stückes an den Rändern des Quarzschlittens aus. Entfernen Sie eingeschlossene Luftblasen, indem Sie vorsichtig mit einer Pinzette aus Polytetrafluorethylen drücken.
- Die Papierseite des Klebestücks abziehen. Montieren Sie das Stück auf der funktionalisierten Oberfläche des Deckels.
- Polyethylenschläuche (I.D. 1,22 mm) in eine Länge von 11 cm für den Einlass und 25 cm für den Auslass schneiden. Setzen Sie das Rohr als Ein- und Auslass für die Durchflusszelle in die zuvor gebohrten Löcher ein.
- Verwenden Sie 5 min Epoxidkleber, um die Ränder der Quarz-Abdeckungs-Schnittstelle und um die Rohre für den Ein- und Auslass zu versiegeln.
- Verwenden Sie die Durchflusszelle sofort, sobald sie trocknet, oder lagern Sie sie unter trockenem Vakuum für die spätere Verwendung.
- Avidin in PBS auf eine Konzentration von 0,03 mg/ml auflösen. Filtern Sie durch 0,2 m Spritzenfilter.
- Mit einer 1 ml Spritze wird Avidin in die Durchflusszelle fließen. Verwenden Sie eine andere Spritze mit Puffer gefüllt, um überschüssiges Avidin auszuwaschen. Achten Sie darauf, beim Austausch der Spritzen keine Luftblasen einzuführen.
-
Mehrkanal-Durchflusszelle
- Bohren Sie sechs Löcher mit einem Durchmesser von 1,22 mm an jeder der langen Seiten eines Quarzschlittens (76 mm x 25 mm)(Abbildung 1B). Machen Sie die Löcher 4,5 mm vom Rand des Schlittens und 9,3 mm voneinander entfernt. Stellen Sie sicher, dass der Abstand zwischen den Mitten jedes Lochpaares 15 mm beträgt.
- Schneiden Sie mit dem elektronischen Fräser sechs Kanäle (20 mm x 2,25 mm) in ein 76 mm x 25 mm Doppelklebeband aus.
- Ziehen Sie die Kunststoffseite der Schutzhülle ab und richten Sie die Ränder des Klebestücks an den Rändern des Quarzschlittens aus. Entfernen Sie alle eingeschlossenen Luftblasen durch sanftes Drücken mit einer Pinzette aus Polytetrafluorethylen.
- Die Papierseite des Klebestücks abziehen und auf die funktionalisierte Oberfläche des Deckels montieren.
HINWEIS: Manchmal funktioniert das Abschälen der Papierseite und das Anbringen an der Quarzrutsche gut in der Mehrkanal-Durchflusszelle. - Die Einlassrohre (11 cm) und die Auslassrohre (25 cm) für die sechs Kanäle schneiden. Bereiten Sie die Durchflusszelle wie in den Schritten 2.1.6–2.1.9 beschrieben vor.
- Schließen Sie den Ausgang des ersten Kanals an die Pumpe an. Legen Sie den Einlass mit OSS in 0,5 ml Rohr.
ANMERKUNG: Die Länge des Einlassrohrs wird gewählt, um die Anzahl der Ereignisse in den Spaltungsexperimenten zu maximieren, die unter kontinuierlichem Fluss durchgeführt werden, indem die Zeit des Enzymeintritts in die Durchflusszelle synchronisiert wird und die Bildgebung beginnt, wodurch die vorzeitige Photobleiche der Fluorophoren. - Wechseln Sie zu einem neuen Kanal, indem Sie den Ausgang des verwendeten Kanals trennen. Schließen Sie den Auslass mit einem Stecker aus einer Spritzennadel, die mit Kleber im Kunststoffteil versiegelt ist. Schließen Sie den Einlass des verwendeten Kanals.
3. Vorbereitung des Sauerstoffaufräumsystems (OSS)
- 0,2 g ()-6-Hydroxy-2,5,7,8-Tetramethylchroman-2-Carbonsäure (Triplet-Zustandsquencher, die das Blinken der Fluorophore minimiert) in 800 l Methanol auflösen.
- Fügen Sie 6 ml deionisiertes H2O hinzu und fügen Sie 1 N NaOH tropfenweise hinzu, bis es sich auflöst. Durch einen Spritzenfilter filtern, in 1 ml Aliquots einziehen und bei -80 °C lagern. Die Lagerkonzentration liegt bei 100 M.
- Bereiten Sie eine frische Lösung von 3,4-dihroxybenzoesäure (PCA) durch Auflösen von 61 mg PCA-Pulver in 4 ml ddH2O vor. Die Lagerkonzentration beträgt 100 nM.
- Fügen Sie 58 L von 10 N NaOH tropfenweise hinzu, stellen Sie sicher, dass sie nach jedem Tropfen wirbeln, bis PCA vollständig aufgelöst ist (pH = 9).
- 5,3 mg Protocatechuat 3,4-Dioxygenase (3,4-PCD) in 7 ml PCD-Speicherpuffer auflösen (Tabelle 1). 3,4-PCD entfernt Sauerstoff aus den Bindungs-/Spaltungspuffern, indem die Oxidation von Protokatchusäure34katalysiert wird.
- Teilen Sie die PCD-Lösung in 1 ml Aliquots auf. Die Lagerkonzentration beträgt 1 m. Die Aliquots in flüssigem Stickstoff einfrieren und bei -80 °C zur Langzeitlagerung oder bei -20 °C für die kurzfristige Lagerung lagern.
- Vorbereiten eines neuen Bindungspuffers (Tabelle 1). Ersetzen Sie 2 mM CaCl2 mit 2 mM MgCl2 für smFRET Spaltungsexperimente.
- Vorbereiten von 1 ml des Bildgebungspuffers (Tabelle 1). Halten Sie den Bildgebungspuffer auf Eis, bis er in die Durchflusszelle eingeführt wird, um die Aktivität des Sauerstoffaufräumsystems aufrechtzuerhalten.
4. Vorbereitung von fluoreszierend gekennzeichneten HJs
- Rekonstituieren Sie die lyophilisierten Oligos (Tabelle 2) im Tris-EDTA-Puffer (Tabelle 1) auf eine Konzentration von 100 m.
- Bereiten Sie die synthetische Verbindung vor, indem Sie äquimolare Portionen von jedem der in Tabelle 1aufgeführten X 0-Oligos mit 3 l mischen.
- Anneal durch Erhitzen bei 95 °C für 5 min, gefolgt von langsamer Kühlung zu RT mit einer Rate von 1 °C/min. Verwenden Sie entweder einen Wärmeblock oder PCR Thermocycler, um die gewünschte Abkühlrate zu erreichen.
- Die Mischung auf 8 cm x 8 cm 10% Tris-Borate-EDTA Polyacrylamidgel geben. Tragen Sie 100 V auf und führen Sie das Gel für 2 h aus. Die Bänder sind deutlich vom Auge gesehen, und ihre Farbe ist lila.
- Verbrauchen Sie das Band des geglühten Substrats mit einer sauberen Klinge. Übertragen Sie das Gelstück in ein autoklaviertes 1,5 ml-Rohr.
- Crush das Gelstück in der Röhre mit einem sauberen Kolben dann fügen Sie 100 L TE100 Puffer (Tabelle 1).
- Extrahieren Sie das HJ, indem Sie das Rohr bei 20 °C bei 1.500 U/min in einem Thermomixer für 2 h schütteln oder über Nacht bei 4 °C inkubieren.
- Führen Sie die Ethanolfällung auf der Lösung durch, die das Substrat35enthält.
- Setzen Sie das Substrat in 20 L DES TE100-Puffers aus (Tabelle 1). Die Endkonzentration liegt bei 1–3 m. Aliquot 2 l in jedem Rohr und lagert bei -20 °C.
5. Proteinexpression und Reinigung von GEN1
- Konstruieren Sie das Plasmid für die Expression von abgeschnittenen menschlichen GEN11,2,3,4,5 mit Hexa Histidin-Tag am C-terminus20 durch PCR des Eintragsvektors.
HINWEIS: N-Terminal-Tagging würde zur Inaktivierung von GEN1 führen. Der unstrukturierte C-Schwanz erschwert die Reinigung des Gen1 in voller Länge deutlich. Außerdem wurde berichtet, dass gen1 in voller Länge weniger Aktivität aufweist als die abgeschnittene Version23. - Transformieren Sie den Expressionsvektor in E. coli BL21-CodonPlus (DE3)-RIPL-Stamm.
- Impfen Sie die umgewandelten Zellen in zwei 6 L-Flaschen, die jeweils 2 L Luria-Brühemedien bei 37 °C enthalten, mit Schütteln bei 180 U/min, bis ein OD600 von 0,8 erreicht ist.
- Kühlen Sie die Kultur auf 16 °C ab und induzieren Sie den GEN1-Ausdruck mit 0,1 mM Isopropyl-D-Thiogalactopyranoside (IPTG) für 48 h.
- Ernten Sie die Zellen, indem Sie sie bei 4 °C bei 1000 x g in einer Zentrifuge nach unten drehen. Jeder Liter Kultur ergibt 5–6 g des Pellets.
- Entsorgen Sie den Überstand und setzen Sie die pelletierten Zellen im Lysepuffer (Tabelle 1) mit 4 ml/g Zellen wieder aus.
- Führen Sie die Zelllyse mit einem Zellstörer bei 30 kPsi durch und drehen Sie dann bei 10.000 x g für 1 h bei 4 °C nach unten. Sammeln Sie den Überstand und filtern Sie ihn auf Eis mit 0,45 m Filtern.
- Führen Sie die Proteinreinigung mit FPLC durch, indem Sie das Filtrat durch eine 5 ml Ni-NTA-Säule mit einer Durchflussrate von 2,5 ml/min mit Puffer A (Tabelle 1) passieren.
- Waschen mit 15 Säulenvolumen (CV). Elute mit einem linearen Gradienten von Puffer A und 500 mM Imidazol über 20 CV in 5 ml Fraktionen. GEN1 elutiert aus der Säule bei ca. 100 mM Imidazol.
- Pipette 10 L Aliquots aus den gesammelten Fraktionen, fügen gleiche Volumen von 2x SDS-Ladefarbstoff zu jedem Aliquot. Denaturieren Sie die Proben durch Erhitzen bei 90 °C für 5 min, kühlen und drehen Sie die Proben.
- Die Proben auf 10% Bis-Tris Gel auftragen. Führen Sie das Gel für 30–45 min bei 200 V. Stain mit Coomassie Brilliant Blue, dann destain. Sammeln Sie die Fraktionen, die gereinigtes GEN1 enthalten.
- Reduzieren Sie die Salzkonzentration der kombinierten Fraktionen durch Verdünnung mit Puffer C (Tabelle 1).
- Das salzarme Protein mit Puffer B (Tabelle 1) durch eine 5 ml Heparinsäule mit einer Durchflussrate von 3 ml/min passieren
- Mit 10 CV waschen. Elute mit einem Gradienten von 20 CV mit Puffer B und 1 M NaCl. Sammeln Sie 5 ml Brüche, in denen GEN1 ca. 360 mM NaCl eluiert.
- Überprüfen Sie die eluierten Fraktionen auf gereinigte GEN1-Fraktionen, wie in Schritt 5.8 beschrieben. Kombinieren Sie diese Fraktionen und verdünnen Sie auf 100 mM NaCl mit Puffer C.
- Das untere Salzprotein mit Puffer B auf eine Kationenaustauschsäule mit 1 ml/min Durchfluss laden.
- Elute mit einem Gradienten von 40 CV mit Puffer B und 1 M NaCl. Sammeln Sie 1,7 ml Fraktionen, in denen GEN1 ca. 300 mM NaCl eluiert.
- Prüfen Sie die Reinheit von GEN1 in den eluierten Fraktionen, wie in Schritt 5.8 beschrieben.
- Kombinieren Sie die reinsten Fraktionen und Dialyse bei 4 °C gegen Speicherpuffer (Tabelle 1). Führen Sie während der Dialyse mindestens einen Austausch des Puffers durch.
- Messen Sie die Proteinkonzentration von 0,5–1 mg/ml. Aliquot das dialysierte Protein in 10–15 L Volumen in kleinen Röhren, Blitz-Gefrierinze in flüssigem Stickstoff, und lagern bei -80 °C.
6. Einmolekulare FRET-Experimente
HINWEIS: Die smFRET-Experimente werden auf einer kundenspezifischen objektiven TIRF-Einrichtung durchgeführt (Abbildung 1C) beschrieben zuvor31.
- Einfarbige FRET-Experimente
- Tragen Sie einen Tropfen Tauchöl auf das 100x TIRF-Objektiv auf. Stellen Sie die EMCCD auf eine geeignete Verstärkung ein, um das Signal auf den Hintergrund zu optimieren und Sättigung zu verhindern.
HINWEIS: Schauen Sie nicht direkt in den Laserstrahl und tragen Sie schutzbeschützende Brillen, wenn Sie den Laser ausrichten. - Legen Sie die Durchflusszelle vorsichtig auf den Probenhalter. Erhöhen Sie das Ziel nach und nach mit grober Einstellung, bis das Öl den Deckelschlupf berührt.
- Schalten Sie den grünen Laser (532 nm) ein. Wechseln Sie in den Feineinstellungsmodus des Objektivs. Richten Sie die Emission an den Kameraanschluss, um das Bild auf dem Monitor zu beobachten.
- Passen Sie die Höhe des Objektivs an, bis die funktionalisierte Oberfläche des Abdeckslips in den Fokus gerückt wird und auf dem Monitor beobachtet werden kann.
HINWEIS: Die Bildaufnahme durch EMCCD löst die Lasererregung über den akusto-optischen Abstimmfilter (AOTF) aus, um Probenphotobleichungen zu verhindern, wenn keine Bilder aufgenommen werden. - Stellen Sie fest, dass der Hintergrund der funktionalisierten Oberfläche der Abdeckungen einige Stellen nicht überschreitet, bevor Sie in die fluoreszierend markierte HJ fließen.
- Das Stammsubstrat etwa 1000 Mal in TE100-Puffer (Tabelle 1) auf eine Endkonzentration von 1–5 nM verdünnen. Pipette 0,2–0,5 l des verdünnten Substrats in 120 l des Bildgebungspuffers mit OSS in ein 0,5 ml-Rohr.
- Schließen Sie den Auslass der Durchflusszelle an die Spritzenpumpe an. Setzen Sie das Einlassrohr der Durchflusszelle in ein 1,5 ml-Rohr ein und betreiben Sie die Spritzenpumpe mit einer Durchflussrate von 30–50 l/min, um die Lösung aus dem Rohr zu ziehen.
- Überprüfen Sie häufig die Oberfläche auf eine gute Abdeckung (100–300 homogen verteiltes, gut verteiltes Substrat) durch kurze Abbildung mit dem grünen Laser.
- Wenn die Oberflächenabdeckung immer noch nicht ausreicht, warten Sie entweder einige Minuten, bis sich die fluoreszierend markierte HJ von der Lösung auf die Oberfläche legt, oder wiederholen Sie den fließenden Schritt.
- Fließen Sie weitere 120 l Bildgebungspuffer (Tabelle 1) bei 30–50 l/min, um ungebunden fluoreszierend HJ zu waschen. Dann lassen Sie die Durchflusszelle für 5 min sitzen, damit das OSS gelösten Sauerstoff abbauen kann. Die Photobleichung der Fluorophore sollte zu Beginn der Bildgebung minimal sein.
- Legen Sie die Belichtungszeit (ca. 60 ms) fest, die Zykluszeit wird automatisch von der Software basierend auf der Geschwindigkeit der Datenübertragung (ca. 104 ms) eingestellt und die gewünschte Anzahl von Zyklen oder Frames (400) angegeben. Die Emission von Spender (Cy3) und Akzeptor (Alexa Fluor 647) wird durch ein Bildteilergerät in zwei Farbkanäle aufgeteilt.
- Finden Sie einen geeigneten Bereich auf der Oberfläche, fokussieren Sie das Bild, indem Sie die Höhe des Objektivs anpassen und den Film im 16-Bit-TIFF-Format speichern.
- Wechseln Sie zu einem neuen Bereich.
HINWEIS: Bewegen Sie sich immer nur in eine Richtung (d. h. vom Ausgang zum Einlass), um zu vermeiden, dass der gleiche Bereich zweimal gesiedet wird. - Bereiten Sie 1, 2, 5, 10, 25, 50, 75 und 100 nM GEN1 in 120 L Bildgebungspuffer nacheinander vor. Durchfluss der Lösung mit einer Durchflussrate von 30-50 l/min.
HINWEIS: Wenn die erforderliche Messung im stationären Zustand wie in der Bindung von HJ durch GEN1 oder der Isomerisierung des freien HJ durchgeführt wird, warten Sie 3–5 min, nachdem der Durchfluss angehalten hat, um den Film aufzunehmen. Erwerben Sie drei bis vier Filme aus neuen Bereichen für jede GEN1-Konzentration. - Wenn die Messung unter kontinuierlichem Durchfluss wie bei der Spaltung von HJ durch GEN1 durchgeführt wird, beginnen Sie die Aufzeichnung 5-10 s vor dem Eingang von GEN1 in die Durchflusszelle. Wiederholen Sie die Messung, indem Sie zu einem neuen Kanal in der Sechskanal-Durchflusszelle wechseln.
- Verwenden Sie am Ende eine feste fluoreszierende Perlenfolie, um die Spender- und Akzeptorpartikel im Bildtrenngerät einander zuzuordnen.
- Fügen Sie die Fluoreszenzperlen mit einem Durchmesser von 1 m in 500 l von 1 M Tris (pH = 8,0) ein, damit die Perlen an der Oberfläche kleben können.
- Schneiden Sie ein Quadrat (18 mm x 18 mm) in ein 22 mm x 22 mm Stück einer doppelseitigen Klebedichtung. Abziehen und auf die Mitte eines 76 mm x 25 mm Quarzschlittens kleben.
- Legen Sie 50 l der verdünnten Perlenlösung und lassen Sie es 5–10 min ansetzen. Befestigen Sie einen 22 mm x 22 mm Abdeckzettel auf dem quadratischen Stück. Überschüssige Perlenlösung mit Gewebe trocknen, dann die Kammer durch Epoxidkleber versiegeln.
- Erfassen Sie 100 Rahmen der Perlenrutsche bei einer Belichtungszeit von 60 ms.
VORSICHT: Senken Sie die Laserleistung und den EMVD-Gewinn auf ein Minimum, um eine Sättigung des Detektors zu vermeiden. - Installieren Sie das Softwarepaket (z. B. TwoTones) und öffnen Sie die darin enthaltenen Filme, wie in der Bedienungsanleitung36angegeben. Wählen Sie die Positionen der einzelnen Perlen in den Spender- und Akzeptorkanälen aus. Generieren Sie eine Transformationsmatrix, wie im Handbuch beschrieben.
HINWEIS: Diese Software verwendet Transformationsmatrix, um die Positionen der Partikel in den Spender- und Akzeptorkanälen abzugleichen und jede leichte Fehlausrichtung im Bildtrenngerät zu korrigieren. - Gehen Sie zu Datei, drücken Sie Film laden, wählen Sie dann die Filmdatei aus und drücken Sie Öffnen. Drücken Sie im Dateimenü TFORM laden und wählen Sie die Transformationsmatrix aus, die aus dem Perlenschieber generiert wurde. Passen Sie den Schwellenwert für die Spender- und Akzeptorkanäle an, bis keine falschen Positivmeldungen enthalten sind.
- Wählen Sie im Menü Kanalfilter die Option D&&A aus, um Partikel auszuwählen, die sowohl mit Spender als auch mit Akzeptor gekennzeichnet sind. Überprüfen Sie das Feld nächster Nachbar, um Moleküle auszuschließen, die sehr nahe beieinander liegen. Überprüfen Sie max Ellipsenheit, um sehr exzentrische Moleküle auszuschließen, und überprüfen Sie die Breite der Grenzen, um sehr breite oder sehr enge Moleküle auszuschließen.
- Geben Sie plotHistCW ein, wie im Twotones-Handbuch beschrieben, um Histogramme zu konstruieren.
HINWEIS: Die "scheinbare" FRET-Effizienz wird durch das Programm berechnet, indem die Emission des Akzeptors durch die Gesamtemissionen des Gebers und des Akzeptors dividiert wird. Twotones verwendet 100 Intervalle, um die Verteilung der Zustände der Moleküle gegen die FRET-Effizienz zu zentrieren. - Geben Sie plotTimetraceCW ein, wie im Twotones-Handbuch beschrieben, um die Zeitspuren für jedes Molekül zu generieren.
HINWEIS: Zeitablaufverfolgungen können von vbFRET37 weiter analysiert werden, um verschiedene FRET-Zustände, ihre jeweiligen Verweilzeiten und Übergangsraten zwischen verschiedenen Zuständen zu identifizieren.
- Tragen Sie einen Tropfen Tauchöl auf das 100x TIRF-Objektiv auf. Stellen Sie die EMCCD auf eine geeignete Verstärkung ein, um das Signal auf den Hintergrund zu optimieren und Sättigung zu verhindern.
- Zweifarbige wechselförmige Erregung FRET (ALEX) Experimente
- Zeichnen Sie einen Film auf, der aus aufeinanderfolgenden Frames von Spender- und Akzeptoremissionen durch direkte Anregungen mit den grünen bzw. roten Lasern mit jeweils 80 ms Dauer besteht.
- Öffnen Sie die erworbenen ALEX-Filme in Twotones. Legen Sie die geeignete Nachweisschwelle auf 300 US-Us-Liegenfür die drei Kanäle fest: Spenderemission aufgrund von Spendererregung (DexDem); Akzeptor-Emission aufgrund von Spenderanregung (DexAem); und Akzeptoremission durch direkte Anregung (AexAem).
- Wenden Sie den Kanalfilter DexDem&&DexAem&&AexAem an, um für die Partikel auszuwählen, die sowohl Spender als auch Akzeptor haben. Verknüpfen Sie die Partikel in den drei Kanälen mit 200–300 .
- Verwenden Sie den plotHistALEX MATLAB-Code, um ALEX-Histogramme zu generieren. Passen Sie verschiedene Spitzen in den Histogrammen an gauschsche Funktionen an und bestimmen Sie den Prozentsatz jeder Grundgesamtheit, die die Fläche unter der Kurve mit der Origin-Software38beträgt.
ANMERKUNG: Die Spitzen im Bindungstest entsprechen dem gebundenen GEN1-HJ-Komplex, während im freien HJ die Spitzen die sich verändernden Isomere darstellen. - Verwenden Sie den plotTimetraceALEX MATLAB-Code, um eine Zeitspur für jedes Molekül zu erzeugen, das die Spenderemission durch direkte Anregung und Akzeptanzemissionen durch FRET und direkte Anregung zeigt.
HINWEIS: ALEX-Zeitablaufe unabhängig, um die Emissionen des Spenders und des Akzeptors anzuzeigen, jedoch mit einer niedrigeren zeitlichen Auflösung als einfarbiges FRET. Ähnlich wie bei einfarbigem FRET können ALEX-Zeitspuren von vbFRET weiter analysiert werden, um verschiedene FRET-Zustände und deren jeweilige Verweilzeiten zu identifizieren. - Bestimmen Sie die Dissoziationskonstante, indem Sie die Prozentsätze der gebundenen Population im Vergleich zur GEN1-Konzentration einer hyperbolischen Funktion anpassen.
- Zeitraffer einfarbig FRET
- Stellen Sie die Belichtungszeit des grünen Lasers auf 60 ms und die Zykluszeit auf 624 ms oder niedriger, abhängig von der Geschwindigkeit der beobachteten Dynamik.
- Stellen Sie die Durchflussrate in einem Kanal der Sechskanal-Durchflusszelle auf 110 l/min ein. Starten Sie die Aufnahme kurz vor dem Eingang von GEN1 in die Durchflusszelle.
HINWEIS: Kontinuierlicher Fluss führt zu einer schnellen Photobleichung der Fluorophore, wodurch die Synchronisierung des Beginns der Bildgebung und des Proteineintrags die Anzahl der erfassten Ereignisse maximiert. Die optimale Spritzenpumpenablesung hängt vom Totvolumen und den genauen Schläuchen ab, die zur Zusammenbau der Durchflusszelle verwendet werden; in unserem Fall sind es 25 €. - Erwerben Sie einen Film mit 125 Bildern für eine Gesamterfassungszeit von 78 s. Setzen Sie die Probe am Ende der Aufnahme dem roten Laser für jeweils 50 Bilder mit einer Belichtungszeit von 25 ms aus, um den Akzeptor zu untersuchen.
HINWEIS: Diese Methode verlängert das Beobachtungsfenster im Spaltungsexperiment durch Verringern der Anzahl der Anregungszyklen. Kinetische Parameter wie kein und kaus der Dimerisierung werden abgeleitet, indem die Verteilung an ein zweiexponentiales Modell30,38anpasst.
7. Elektrophoretische Mobilitäts-Shift-Assays (EMSA)
- In 50 l Gesamtvolumen die gewünschte Konzentration von GEN1 mit 50 pM Cy5-beschrifteter HJ bei RT für 30 min im EMSA-Bindungspuffer inkubieren (Tabelle 1).
- Die Proben auf 8 cm x 8 cm 6% Tris-borate-EDTA Gel laden. Führen Sie das Gel mit 100 V für 1 h + 20 min in 1x TBE Puffer bei RT.
- Bestimmen Sie den Prozentsatz des gebundenen Substrats bei einer GEN1-Konzentration aus seinem relativen Beitrag zur Gesamtfluoreszenzintensität der jeweiligen Spur.
HINWEIS: GEN1 monomer-HJ (Band I) wird durch die Vereinbarung seiner Größe mit der picomolaren Bindung von GEN1 Monomer an die genickte HJ21,30identifiziert. GEN1-dimer-HJ wird Band II aufgrund der schrittweisen Bindung von GEN1 Monomer an die HJ21,23zugeordnet. - Berechnen Sie die scheinbaren Bindungskonstanten Kd-monomer-app-EMSA und Kd-dimer-app-EMSA mit der Gleichung:

Wo: Max ist die Konzentration, bei der die jeweilige Art ihre maximale Bindung erreicht hat (Monomer oder Dimer); n ist der Hill-Koeffizient; K d-app-EMSA ist die scheinbare Bindungskonstante der jeweiligen Art, die die Konzentration von GEN1 bezeichnet, bei der das halbmaximale Monomer oder Dimer vorhanden ist.
Ergebnisse
Conformer Bias und Isomerisierung des HJ
Die Isomerisierung von HJ wurde von FRET durch die Kennzeichnung von zwei angrenzenden Armen der Kreuzung17,18,39ausgiebig untersucht. Der Spender (Cy3) und der Akzeptor (Alexa Fluor 647) befinden sich an den beiden benachbarten Armen R (Strang 2) bzw. X (Strang 3)(Abbildung 2A). Die gestapelten X-Isomer wurden durch ihre beiden durchgehenden Stränge [d.h. Iso(1,3) oder Iso(2,4)] zugeordnet. Das ALEX FRET Histogramm des benachbarten Etiketts X0 zeigt zwei Spitzen, die dem Wechsel des reichlicheren Iso(1,3) (E 0,75) und des weniger reichlich vorhandenen Iso(2,4) (E 0,40) entsprechen (Abbildung 2B).
Einfarbiges FRET wird verwendet, um Zeitspuren für die Aufzeichnung der schnellen Konformationsänderungen im freien HJ mit hoher zeitlicher Auflösung von 10 ms zu erfassen, indem der verwendete Bereich der EMCCD2-Kamera reduziert wird. Eine repräsentative einfarbige FRET-Zeitablaufverfolgung der X0-Kreuzung zeigt die Übergänge zwischen hohen und niedrigen FRET-Isomern (Abbildung 2B). Die Isomerisierungsraten kIso(1,3)-Iso(2,4) und kIso(2,4)-Iso(1,3) aus den Verweilzeithistogrammen von Iso(1,3) und Iso(2,4) (Abbildung 2C) stimmen mit den zuvor gemeldeten17überein.
SMFRET zeigt aktive Verzerrung des HJ durch GEN1
HJ wird bei Bindung an GEN122einer strukturellen Umlagerung unterzogen. Somit ist der Abstand zwischen Spender und Akzeptor sowohl in Iso(1,3) als auch in Iso(2,4) ähnlich (Abbildung 3A). Die smFRET-Bindungstests wurden in Anwesenheit von Ca2+ durchgeführt, um eine Spaltung des HJ zu verhindern. FRET Histogramme der benachbarten X0-Kreuzung bei verschiedenen GEN1-Konzentrationen wurden von ALEX erworben (Abbildung 3B). Das Histogramm ist auf zwei Gaußsche Funktionen fit: eine, die dem freien hohen FRET Iso(1,3) entspricht, und die andere entspricht der gebundenen GEN1-HJ-Population, nachdem der Beitrag der Iso(2,4) von der niedrigen FRET-Spitze subtrahiert wurde.
Bei der Sättigung der GEN1-Konzentration hat das FRET-Histogramm von X0 nur einen einzigen niedrigen FRET-Peak, der GEN1 entspricht, der entweder an isomer des HJ gebunden ist, wie vom Modell22vorhergesagt. Die scheinbare Monomerdissoziationskonstante (Kd-monomer-app) wird aus der hyperbolischen Anpassung der Prozentsätze der GEN1-gebundenen Population in Abhängigkeit von der GEN1-Konzentration bestimmt (Abbildung 3C). Das benachbarte Etikett nk-X0 stellt eine singly nicked Version HJ dar, die das Produkt nach der ersten Einschnittreaktion imitiert. Aufgrund der Entlastung der Stapeldehnung durch den simulierten Nick ist nk-X0 eine nicht-isomerierende Struktur40, wie aus der Single-Substratspitze bei E 0,40 im Gegensatz zu X0 (Abbildung 3D vs. Abbildung 3B)ersichtlich. Die Struktur des GEN1-nk-X0-Komplexes ähnelt der des GEN1-X0-Komplexes, wie die Ähnlichkeit der FRET-Effizienzzeigten zeigt (E 0,25 für nk-X0 und 0,32 für X0) ( Abbildung3D vs. Abbildung 3B). Die starke Bindung von GEN1-Monomer an nk-X0 zeigt der 40-fach niedrigere Kd-monomer-app-Wert als der von X0 (Abbildung 3E vs. Abbildung 3C). Diese enge Bindung kann als Schutzmechanismus gegen die unvollständige Auflösung des HJ im unwahrscheinlichen Fall der Dissoziation von GEN1 Dimer oder einem seiner Monomere dienen.
Schrittweise Bindung von GEN1-Monomer an den HJ
Die Bindung von GEN1-Monomer an das HJ gefolgt von Dimer-Formation ist ein einzigartiges Merkmal für die eukaryotische HJ-Resolvase GEN1 im Vergleich zu prokaryotischen Resolvasen, die in dimerischer Form in Lösung21,23,41existieren. EMSA von GEN1 bei 50 pM X0 zeigt die schrittweise Assoziation von GEN1 in komplexere Komplexe höherer Ordnung, wie die römischen Ziffern im oberen Panel zeigen (Abbildung 4A). Die von EMSA (Kd-monomer-EMSA) ermittelte Dissoziationskonstante des GEN1-Monomers fällt mit der Dissoziationskonstante vom smFRET-Bindungstest Kd-monomer-app zusammen (Abbildung 4A und Abbildung 3C bzw. Die Quantifizierung von Band II wird verwendet, um die Gleichgewichtsdissoziationskonstante von GEN1 dimer (Kd-dimer-EMSA) zu berechnen. EMSA von GEN1 bei 50 pM nk-X0 zeigt die markante Monomerbindung, wie sie durch die sehr niedrige Kd-monomer-app-EMSA angezeigt wird, die 30-mal niedriger ist als die von X0, während ihre Kd-dimer-EMSA vergleichbar mit X0 (Abbildung 4B).
Ein weiterer Beweis dafür, dass GEN1-Monomer das HJ bindet und verzerrt, ist die Beobachtung einer signifikanten Anzahl von Spuren von uncleaved Partikeln mit stabilem niedrigem FRET-Zustand (Abbildung 4C) in Gegenwart von Mg2+ bei niedrigen GEN1-Konzentrationen. Die Anzahl dieser Spuren ging mit zunehmender GEN1-Konzentration zurück. Die Auflösung des HJ wird durch die enge Bindung von GEN1-Monomer angetrieben, das die Dimerbildung unterstützt. Die Monomerbindung wird in den Zeitspuren des onkeltierten nk-X0 in Mg2+beobachtet, das sich bis zu einer wenigen nanomolaren Konzentration erstreckt (Abbildung 4D). Das GEN1-Monomer bindet eng, um nk-X 0 zuschützen, und sorgt schließlich für die volle Auflösung durch Dimer-Bildung.
SMFRET-Auflösungstest des HJ
Der Begriff "Spaltung" in smFRET-Assays wird synonym mit "Auflösung" des HJ verwendet, da in diesem Test nur die Produktfreigabe, die auf das zweite Spaltungsereignis folgt, erkannt wird. Die Ereignisse werden durch einfarbige Sergung mit Zeitraffer aufgezeichnet, um die Photobleichung des fotoempfindlichen Akzeptors über die Erfassungszeit von 1,3 min zu minimieren.
Der Schaltplan in Abbildung 5A veranschaulicht die Einschnitte der Stränge 1 und 3 von X0 Iso(1,3) nach der Bindung und Verzerrung eines X0, das am funktionalisierten Glas befestigt ist, durch GEN1. Sowohl Spender als auch Akzeptor gehen in eine Lösung, die zum Verlust ihrer Signale nach der HJ-Auflösung führt. Der erste und der zweite Schnitt werden in nk-X0 entkoppelt, was einen Prototyp für das teilweise aufgelöste HJ veranschaulicht. Nach Bindung von GEN1 nimmt nk-X0 eine ähnliche Struktur wie X0an. Die Entschließung erfolgt durch einen einzigen Einschnitt in Strang 1, wie in Abbildung 5Bdargestellt.
Der gleichzeitige Abgang des Spenders und des Akzeptors nach einem stabilen niedrigen FRET-Zustand in Spuren von gelöstem X0 trat ohne die Entstehung eines Zwischen-FRET (E = 0,40) auf, was darauf hindeutet, dass die vollständige Auflösung innerhalb der Lebensdauer des GEN1-HJ auftritt. Komplex (Abbildung 5C). Daher deuten diese Ergebnisse darauf hin, dass die HJ-Auflösung innerhalb der GEN1-HJ-Komplexlebensdauer auftritt. Die Auflösung von nk-X0 erfolgt auch nach der strukturellen Umlagerung und schließt mit dem Weggang des Duplexmit zwei Fluorophore (Abbildung 5D) ähnlich X0.
Kinetik der GEN1-Dimerisierung auf GEN1-Monomer gebunden HJ
Zeitraffer-smFRET-Maßnahmenvor der Spaltung, die hauptsächlich die Zeit umfassen, die für die Dimerbildung und -auflösung des HJ nach der Verzerrung durch GEN1-Monomer benötigt wird. Bei Anwendung dieser Technik wird der direkte Nachweis erbracht, dass die Dimerbildung für die Auflösung von X0 und nk-X0erforderlich ist, da die Verteilung von-vor der Spaltung GEN1-konzentrationsabhängig ist.
Die scheinbare Rate der HJ-Auflösung (kapp) ist definiert als die Umkehrung des Mittelwerts von vor der Spaltung bei der jeweiligenGEN1-Konzentration. Der Begriff "offensichtlich" wird verwendet, um die Rate der HJ-Auflösung zu beschreiben, da die Möglichkeit, dass GEN1 nach der HJ-Auflösung an das Produkt gebunden bleibt, nicht ausgeschlossen werden kann.
Die Wahrscheinlichkeitsdichtefunktionen (PDF) der Vorspaltungsverteilungen von X0 (Abbildung 6A) spiegeln die Zeit für die Dimerbildung wider, die bei niedrigen GEN1-Konzentrationen länger und bei höheren GEN1-Konzentrationen kürzer ist. Die Assoziations- und Dissoziationsraten für dimer, kon-dimer bzw. koff-dimerwerden nach einem zweiexponentiellen Modell30bestimmt. Auch die PDFs von nk-X0 (Abbildung 6B) zeigen eine ähnliche Verteilung wie X0, was auf die Anforderung der Dimerbildung hinweist.
Das Diagramm der k-App-versus-GEN1-Konzentration wurde an eine hyperbolische Funktion angepasst. Die scheinbaren Katalyseratenkonstanten (kMax-app) von X0 und nk-X0 sind 0,107 x 0,011 s-1 bzw. 0,231 x 0,036 s-1, bzw. (Abbildung 6C). Die Plots derk-App für X0- und nk-X0-Knoten schneiden sich bei der GEN1-Konzentration um 5,6 nM aufgrund der schnelleren kMax-App und des langsameren k-On-Dimers der die intakte Kreuzung.
Zusammenfassend lässt sich sagen, dass die relativ schnellen kon-dimer und langsamkoff-dimer zur Progression der Vorwärtsreaktion in Richtung HJ-Auflösung führen, sobald der Dimer gebildet ist. Die starke Bindung des GEN1-Monomers an die nk-X0-Kreuzung stellt einen ausfallsicheren Mechanismus gegen jede unwahrscheinliche abgebrochene zweite Spaltung dar oder hilft, alle unvollständig ungelösten HJs aufzugreifen, die durch primäre Auflösungspfade in der Zelle zurückgelassen werden.
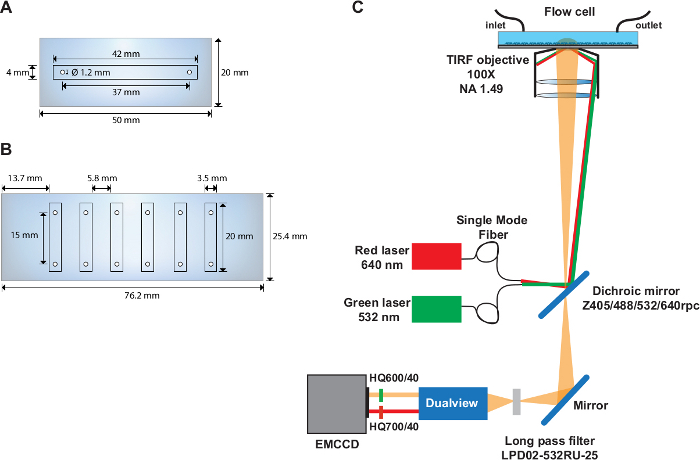
Abbildung 1: Ein- und Mehrkanal-Durchflusszellen und Layout der optischen Einrichtung.
(A) Schematic der einkanaligen Durchflusszelle. (B) Schemader der Sechskanal-Durchflusszelle. (C) Layout des optischen Aufbaus, der die Anregungsquellen, das IM Filterwürfel installierte TIRF-Objektiv, den dichroitischen Spiegel und die im Bildteiler verwendeten Emissionsfilter darstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Conformer Bias und Isomerisierung des HJ, beobachtet von FRET.
(A) Isomerisierung der benachbarten X-gestapelten HJ-Konformer, die nach den beiden durchgehenden Strängen benannt sind. Die Stränge sind nummeriert, während die Arme durch Buchstaben bezeichnet werden. Die Einschnittstellen werden durch Pfeile angezeigt. Die Positionen des Spenders (grün) und des Akzeptors (rot) und die Änderung des FRET nach der Isomerisierung sind angegeben. (B) Rechtes Panel: FRET-Zeitablauf (schwarz) und idealisierte FRET-Spur (rot) von X0 bei 50 mM Mg2+. Linke sprotte: FRET Histogramm von X0 bei 50 mM Mg2+. Die Fluoreszenzintensitäten des Spenders (grün) und des Akzeptors (rot) sind unten dargestellt. (C) Die Verweilzeit-Histogramme der benachbarten Bezeichnungen X0 Iso(1,3) und Iso(2,4) wurden an Einzelexponentialfunktionen angepasst, um die Isomerisierungsraten zu bestimmen. Die Unsicherheiten deuten auf das 95%-Konfidenzintervall der Passform hin. Diese Zahl wurde aus der zuvor veröffentlichten Literatur30geändert.
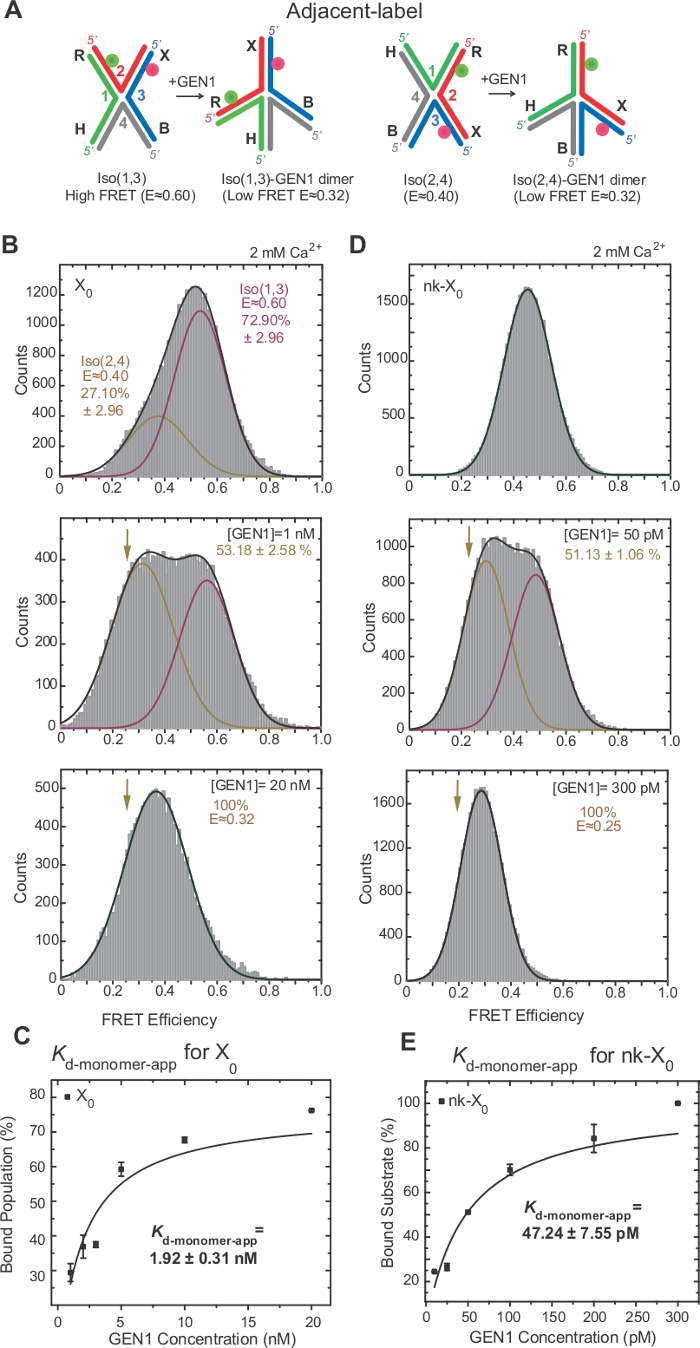
Abbildung 3: Aktive Verzerrung des HJ durch GEN1.
(A) Strukturelle Änderung des angrenzenden Etiketts HJ auf der Grundlage des vorgeschlagenen Modells22. (B) DAS ALEX FRET Histogramm des benachbarten Etiketts X0 hat einen großen hohen FRET-Peak (E = 0,6), der Iso(1,3) und ein niedrigerer FRET-Peak (E = 0,4) für Iso(2,4) entspricht. Das gesamte Histogramm ist für zwei Gaußsche Funktionen geeignet: eine, die der freien hohen FRET Iso(1,3) entspricht, und die andere, die der gebundenen Population abzüglich des anfänglichen Beitrags von Iso(2,4) zur Gesamtpopulation entspricht. (C) Die scheinbare Monomerdissoziationskonstante (Kd-monomer-app) wird aus einer hyperbolischen Anpassung der Prozentsätze der GEN1-gebundenen Populationen in Abhängigkeit von der GEN1-Konzentration bestimmt. (D) FRET Histogramme des angrenzenden Etiketts nk-X0 bei unterschiedlichen GEN1-Konzentrationen. Die Fläche unter dem niedrigen FRET (E = 0,25) Gaußian entspricht dem Prozentsatz der gebundenen Bevölkerung. (E) Die Kd-monomer-app von nk-X0 wird aus der hyperbolischen Passung der GEN1-gebundenen Population ermittelt. Die Fehlerbalken stellen die Standardabweichungen von zwei oder mehr Experimenten dar. Diese Zahl wurde aus der zuvor veröffentlichten Literatur30geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Schrittweise Bindung von GEN1 an das HJ.
(A) Elektrophoretische Mobilitätsschicht-Assay (EMSA) von GEN1 bei 50 pM X0. Oberplatte: Die römischen Ziffern geben die Anzahl der GEN1-Monomere im Komplex an. Untere Platte: Bindung des GEN1-Monomers an X0. Die scheinbaren Dissoziationskonstanten wurden aus einer sigmoidalen Passung der jeweiligen Art gewonnen und stellen den Durchschnitt von zwei Experimenten dar. (B) EMSA von GEN1 bei 50 pM nk-X0 zeigt die prominente Monomerbindung, wie sie durch die sehr niedrige Kd-monomer-app-EMSAangezeigt wird. (C) FRET-Zeitablauf des gebundenen, aber uncleavednten Nebenetiketts X0 in Mg2+. Es wurde eine Spendererregung von 1,3 min durchgeführt, gefolgt von direkter Akzeptorererererregung (schattierter rosa Bereich). (D) FRET-Zeitablauf des gebundenen, aber uncleavednbenachbarten Etiketts nk-X0 in Mg2+. Diese Zahl wurde aus der zuvor veröffentlichten Literatur30geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: SMFRET-Auflösungstest des HJ.
(A) Schemader des angrenzenden Etiketts X0 Iso(1,3) nach Verzerrung durch GEN1. Das Substrat wird über Biotin/Avidin-Verbindung an der funktionalisierten Oberfläche befestigt. Die Dissoziation von GEN1 nach den beiden Einschnitten führt zum Verlust von Spender und Akzeptor, die in Lösung gehen. (B) Schemat der Auflösung von benachbartem Etikett nk-X0 durch Klausen von Strang 1. (C) Zeitablauf (schwarz) bei 2 mM Mg2+ der Spaltung von Iso(1,3). Der Beginn der GEN1-Bindung bildet einen stabilen niedrigen FRET-Zustand, bis das FRET-Signal durch Spaltung abrupt verloren geht. Entsprechend folgt auf die Zunahme des Spenders und die Abnahme der Akzeptorfluoreszenzintensitäten bei GEN1-Bindung das gleichzeitige Verschwinden der Fluoreszenz aus beiden Farbstoffen bei Spaltung. (D) Ebenso zeigt die Zeitspur von nk-X0 einen stabilen niedrigen FRET-Zustand bei GEN1-Bindung, der durch den abrupten Verlust des FRET-Signals abgeschlossen wird. Diese Zahl wurde aus der zuvor veröffentlichten Literatur30geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Kinetik der GEN1-Dimerisierung auf GEN1-Monomer gebundenHJ.
(A) Das Wahrscheinlichkeitsdichtefunktionsdiagramm (PDF) der Verteilung x0 vor Spaltung veranschaulicht seine Abhängigkeit von derGEN1-Konzentration. Verweilzeiten des niedrigenFRET-Zustandes (vor der Spaltung) bei der jeweiligen GEN1-Konzentration wurden aus zwei oder mehr Experimenten gewonnen und verwendet, um Durchschnittsraten zu erhalten (kapp). Die aufgeführtenk-App-Raten werden aus der Umkehrung des Mittelwerts vor Derspaltung bei der jeweiligen GEN1-Konzentration ermittelt. Die Assoziations- (kon-dimer) und die Dissoziation (koff-dimer) Raten für die Dimerbildung werden aus einem zweiexponentiellen Modell38berechnet. Die Fehler stellen SEM der k-App dar. (B) Das PDF-Diagramm derVorspaltungsverteilungen von nk-X0 und der jeweiligenk-App-Raten. (C) Diagramm derk-App im Vergleich zur GEN1-Konzentration, die an eine hyperbolische Funktion angepasst ist, um die scheinbare katalytische Rate zu bestimmen (k Max-app). Das Diagramm derk-App für X0 und nk-X0 veranschaulicht die schnellere anfänglichek-App von X0, die dann von nk-X0 über [GEN1] 5,6 nM übertroffen wird. Diese Zahl wurde aus der zuvor veröffentlichten Literatur30geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| stoßdämpfer | Kompostion | ||
| Bindungspuffer | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM CaCl2, 1 mM DTT, 0,1% BSA und 5% (v/v) Glycerin | ||
| Puffer A | 20 mM Tris-HCl pH 8.0, 1 mM DTT und 300 mM NaCl | ||
| Puffer B | 20 mM Tris-HCl pH 8.0, 1 mM DTT und 100 mM NaCl | ||
| Puffer C | 20 mM Tris-HCl pH 8.0 und 1 mM DTT | ||
| Cleavage-Puffer | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 2 mM MgCl2, 1 mM DTT, 0,1% BSA und 5% (v/v) Glycerin | ||
| EMSA-Bindungspuffer | 40 mM Tris-HCl pH 7,5, 40 mM NaCl, 1 mM DTT, 2 mM CaCl2, 0,1 mg/ml BSA, 5% (v/v) Glycerin und 5 ng/l Poly-dI-dC | ||
| Imaging-Puffer (Bindung) | 40 l (-6-Hydroxy-2,5,7,8-Tetramethylchroman-2-Carbonsäure (4 m), 60 l PCA (6 nM), 60 l PCD (60 nM) und 840 l Bindungspuffer | ||
| Bildgebungspuffer (Spaltung) | 40 l (-6-Hydroxy-2,5,7,8-Tetramethylchroman-2-Carbonsäure (4 m), 60 l PCA (6 nM), 60 l PCD (60 nM) und 840 l Cleavage-Puffer | ||
| Lysis-Puffer | 20 mM Tris-HCl pH 8,0, 10 mM -Mercaptoethanol, 300 mM NaCl und 2 mM PMSF | ||
| PCD-Speicherpuffer | 100 mM Tris-HCl pH 7,5, 1 mM EDTA, 50 mM KCl und 50% Glycerin | ||
| Speicherpuffer | 20 mM Tris-HCl pH 8,0, 1 mM DTT, 0,1 mM EDTA, 100 mM NaCl und 10% Glycerin | ||
| TBE-Puffer | 89 mM Tris-HCl, 89 mM Borsäure und 2 mM EDTA | ||
| TE100-Puffer | 10 mM Tris.HCl pH 8,0 und 100 mM NaCl | ||
| Tris-EDTA-Puffer | 50 mM Tris-HCl pH 8,0 und 1 mM EDTA pH 8,0 | ||
Tabelle 1: Die Liste der In dieser Studie verwendeten Puffer und deren Zusammensetzungen.
| Oligo | folge | ||
| X0-st1 | ACGCTGCCGAATTCTACCAGTGCCTTGCTAGGACATCTTTGCCCACCTGTGCAGGTTCACCC | ||
| X0-st2 | GGGTGATGTGCAGGGGGG/iCy3/AAAGATGTCTGTTGTAATCGTCAAGCTTTATGCCGT | ||
| X0-st3 | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATCATGGAGCTCTCTAGAGGATCCGACTATCG | ||
| X0-st4 | 5'BiotinCGATAGTCGGATCCTCTAGACAGCTCCATGTAGCAAGGCACTTAGTAGAATTCGGCAGCGT | ||
| X0-Adj | X0-st1, X0-st2, X0-st3 & X0-st4 | ||
| X0In_st2 | GGGTGATGTGGGGGGGCAAAGATGTCCATCTGTTGTAATCGTCAAGCTTTATGCCGT | ||
| X0In_st4 | 5'BiotinCGATAGTCGGATCCTCTAGACAGCTCCATGTAGCAAGGCA/iCy3/TGGTAGAATTCGGCAGCGT | ||
| Nk-X0 | X0-st1, X0-st2, X0-nk3a, X0-nk3b & X0-st4 | ||
| X0-nk3a | ACGGCATAAAGCTTGACGA/iAF647-dT/TACAACAGATC | ||
| X0-nk3b | ATGGAGCTGTCTAGAGGATCCGACTATCG | ||
Tabelle 2: SMFRET- und EMSA HJ-Substrate. Die Liste der Oligonukleotide, die zur Herstellung der fluoreszierend gekennzeichneten HJs für smFRET und EMSA verwendet werden. Die Oligos wurden kommerziell erworben. Die fluoreszierend markierten Oligos wurden HPLC-gereinigt und, wenn möglich, Oligos von 60 bp wurden PAGE-gereinigt.
Diskussion
In dieser Studie wurden verschiedene smFRET-Techniken implementiert, um die Kinetik der HJ-Auflösung durch GEN130zu bestimmen. Ähnliche smFRET-Ansätze wurden verwendet, um die Doppelklappen-DNA-Konformationsanforderung und Spaltung durch die DNA-Replikation und Reparaturklappe Endonuklease 142,43,44zu folgen. Hier werden wichtige Schritte in diesem Protokoll erläutert. Die Silanisierungsreaktion sollte frei von Feuchtigkeitseinspuren sein. Die Pegylierungslösung sollte schnell auf das silanisierte Glas aufgetragen werden, sobald PEG gelöst ist, um eine Hydrolyse zu vermeiden. In der Mehrkanal-Durchflusszelle sollte jede eingeschlossene Luft in der Klebefolie entfernt werden, um Leckagen zwischen benachbarten Kanälen zu vermeiden. Die PCA-Lösung sollte frisch zubereitet werden, da sie im Laufe der Zeit oxidiert. Die Zugabe von 10 N NaOH sollte tropfenweise sein, mit Wirbel zwischendurch. Der Fluoreszenzhintergrund im Deckblatt sollte minimal sein, bevor der fluoreszierend beschriftete HJ geflossen ist. Die Bildgebung in der Durchflusszelle sollte in eine Richtung durchgeführt werden, um bildgebende gebleichte Bereiche zu vermeiden. In ALEX-Experimenten sollte die Leistung des roten Lasers reduziert werden, um ein schnelles Bleichen des Akzeptors zu vermeiden. In den Zeitrafferexperimenten muss die Zykluszeit kürzer sein als das schnellste Ereignis.
smFRET ist eine empfindliche Technik, die wertvolle Echtzeit-Einblicke in biomolekulare Reaktionen liefern kann. Diese Methode hat jedoch mehrere technische Herausforderungen, darunter die Erzielung einer messbaren Veränderung des FRET während der biochemischen Reaktion. Dies ist notwendig, um gut getrennte Merkmale in den Histogrammen und unterscheidbare Zustände in den Zeitspuren zu erhalten. In vielen Fällen erfordert smFRET eine sorgfältige Auslegung der Substrate, die Auswahl der Fluorophorpaare und ihrer Positionen sowie die Verstärkung von FRET-Änderungen im DNA-Substrat aufgrund der geringen strukturellen Veränderungen im Substrat45. Ein weiterer Ansatz für die Durchführung von FRET ist die Verwendung von markierten Proteinen46. Das Beobachtungsfenster in FRET wird durch die Stabilität des Akzeptors wie Cy5 oder Alexa Fluor 647 begrenzt, die tendenziell schneller bleicht als der Spender (in diesem Fall Cy3). Daher erfordert FRET eine kontinuierliche Suche nach stabilen Fluorophoren, um die Versuchsdauer zu verlängern und Anstrengungen zur Entwicklung von Sauerstoff-Aufräumsystemen zu unternehmen, um das Fluoreszenzsignal zu verlängern und das Signal-Rausch-Verhältnis zu maximieren47,48 .
Zu den Tipps für die Fehlerbehebung in smFRET gehört das Ausbalancieren der verschiedenen Parameter, die an der Bildgebung beteiligt sind, wie z. B. Laserleistung, Belichtungszeit, Zykluszeit und Anzahl der Zyklen, um die Fluoreszenzemission zu maximieren, die Versuchsdauer zu verlängern und geeignete Abtastintervalle für die Enzymdynamik. Längere Beobachtungszeiten und minimale Effekte durch Photobleichungen sind unerlässlich, um eine hohe Treue zu erhalten, um Zeitverteilungen zu erhalten, die die Enzymdynamik darstellen. ALEX erzeugt bessere Histogramme, da diese Methode niedrigeren Beiträgen von photobleached Partikeln im Vergleich zu einfarbigen FRET ausgesetzt ist. Die zeitliche Auflösung in ALEX ist jedoch niedriger als die im einfarbigen FRET.
Schließlich überbrückt smFRETs Schwerpunkt auf dem Nachweis von konformen/strukturellen Veränderungen einzelner Moleküle in Echtzeit die Lücke zwischen hochauflösenden Strukturtechniken (z. B. Röntgenkristallographie, Kernspinresonanz, Elektronenmikroskopie), die atomare Auflösung strukturelle Details unter statischen Bedingungen und Bulk-Methoden, die das Ensemble Durchschnitt einer messbaren Eigenschaft ergeben. In vielerlei Hinsicht hat sich smFRET als eine leistungsfähige Technik zur Erforschung biologischer Systeme in Echtzeit erwiesen.
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Diese Arbeit wurde von der King Abdullah University of Science and Technology durch Kernfinanzierung und Competitive Research Award (CRG3) an S. M. H. unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox) | Sigma-Aldrich | 238813 | |
| 0.1 M sodium bicarbonate buffer | Fisher | 144-55-8 | |
| 10 % Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6275BOX | |
| 100 X TIRF objective | Olympus | NAPO 1.49 | |
| 3,4-dihroxybenzoic acid (PCA) | Sigma-Aldrich | P5630 | |
| 3-aminopropyltriethoxysilane (APTES) | Sigma-Aldrich | 741442 | |
| 6% Novex Tris-Borate-EDTA gel | Thermo Fisher Scientific | EC6265BOX | |
| Adhesive sheet | Grace bio-labs | SA-S-1L | |
| Benchtop refrigerated centrifuge | Eppendorf | Z605212 | |
| Biotin-PEG | Laysan Bio | Biotin-PEG-SVA 5000 | |
| Bovine Serum Albumin (BSA) | New England Biolabs | B9001S | |
| Calcium Chloride Dihydrate | Sigma-Aldrich | 31307 | |
| cation exchange column | GE healthcare | MonoS (4.6/100) | |
| Cell distruptor | Constant Cell Disruption System | TS5/40/CE/GA | |
| Coomassie Brilliant Blue | MP Biomedicals | 808274 | |
| Cy3 emission filter | Chroma | HQ600/40M-25 | |
| Cy5/Alexa Fluor 647 emission filter | Chroma | HQ700/40M-25 | |
| Dichroic for DV2 filter cube | Photometrics | 630dcxr-18x26 | |
| Dithiothreitol (DTT) | Thermo Scientific | R0861 | |
| Drill | Dremel | 200-1/21 | |
| Electronic cutter | Copam | CP-2500 | |
| EMCCD camera | Hamamatsu | C9100-13 | |
| Epoxy glue | Devcon | 14250 | |
| FPLC Aktapurifier UPC 10 | GE Healthcare | 28406268 | |
| GelQuant.NET software | biochemlabsolutions.com | Version 1.8.2 | |
| GEN1 entry vector | Harvard plasmid repository | HSCD00399935 | |
| Glycerol | Sigma Life Science | G5516 | |
| green laser (emission 532 nm) | Coherent | Compass 315M-100 | |
| Heparin column | GE healthcare | HiTrap Heparin column | |
| HEPES | BDH | BDH4162 | |
| Image splitter | Photometrics | Dualview (DV2) | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Inverted microscope | Olympus | IX81 | |
| Isopropyl-ß-D-thiogalactoside (IPTG) | Goldbio. | 12481C100 | |
| Laser scanner | GE healthcare | Typhoon Trio | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Long pass 532nm filter | Semrock | LPD02-532RU-25 | |
| Magnesium Chloride | Sigma Life Science | M8266 | |
| mPEG | Laysan Bio | mPEG-SVA 5000 | |
| Neutravidin | Pierce | 31000 | |
| Ni-NTA column | GE healthcare | HisTrap FF | |
| NuPAGE 10% Bis-Tris gels | Novex Life technologies | NP0301BOX | |
| NuPAGE 10% Bis-Tris Protein Gels | Thermo Fisher Scientific | NP0302PK2 | |
| Origin software | OriginLab Corporation | Version 8.5 | |
| Phenylmethylsulfonyl fluoride (PMSF) | Alexis Biochemicals | 270-184-G025 | |
| Phosphate-buffered saline | GIBCO | 14190 | |
| Polyethylene Tubing (I.D. 0.76 mm O.D. 1.22mm) | Fisher (Becton Dickinson) | 427416 | |
| Protocatechuate 3,4-dioxygenase (3,4-PCD) | Sigma-Aldrich | P8279-25UN | |
| Quad-band dichroic | Chroma Inc | Z405/488/532/640rpc | |
| red laser (emission 640 nm) | Coherent | Cube 640 100C | |
| Sodium Chloride | Fisher Chemical | S271 | |
| Sorvall RC-6 plus centrifuge | Thermo Fisher Scientific | 46910 | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000 | |
| Syringe pump | Harvard Apparatus | 70-3007 | |
| Teflon tweezers | Rubis | K35A | |
| Tris Base | Promega | H5135 | |
| Ultracentrifuge | Beckman Coulter | Optima L-90K | |
| Ultrafiltration membrane | Millipore | UFC90300 |
Referenzen
- Moerner, W. E., Fromm, D. P. Methods of single-molecule fluorescence spectroscopy and microscopy. Review of Scientific Instruments. 74 (8), 3597-3619 (2003).
- Ha, T. Single-molecule fluorescence resonance energy transfer. Methods. 25 (1), 78-86 (2001).
- Weiss, S. Fluorescence spectroscopy of single biomolecules. Science. 283 (5408), 1676-1683 (1999).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nature Methods. 5 (6), 507-516 (2008).
- Walter, N. G., Huang, C. Y., Manzo, A. J., Sobhy, M. A. Do-it-yourself guide: how to use the modern single-molecule toolkit. Nature Methods. 5 (6), 475-489 (2008).
- Conchello, J. A., Lichtman, J. W. Optical sectioning microscopy. Nature Methods. 2 (12), 920-931 (2005).
- Axelrod, D. Total internal reflection fluorescence microscopy in cell biology. Methods in Enzymology. 361, 1-33 (2003).
- Kim, H. D., et al. Mg2+-dependent conformational change of RNA studied by fluorescence correlation and FRET on immobilized single molecules. Proceedings of the National Academy of Sciences of the United States of America. 99 (7), 4284-4289 (2002).
- Lee, T. H., et al. Measuring the folding transition time of single RNA molecules. Biophysical Journal. 92 (9), 3275-3283 (2007).
- Holliday, R. Mechanism for Gene Conversion in Fungi. Genetical Research. 5 (2), 282-304 (1964).
- West, S. C., et al. The Formation and Resolution of Holliday Junctions during the Recombinational Repair of DNA Damages. Journal of Cellular Biochemistry. , 269-269 (1995).
- Cox, M. M., et al. The importance of repairing stalled replication forks. Nature. 404 (6773), 37-41 (2000).
- West, S. C. Molecular views of recombination proteins and their control. Nature Reviews: Molecular Cell Biology. 4 (6), 435-445 (2003).
- Duckett, D. R., et al. The structure of the Holliday junction, and its resolution. Cell. 55 (1), 79-89 (1988).
- Clegg, R. M., et al. Fluorescence resonance energy transfer analysis of the structure of the four-way DNA junction. Biochemistry. 31 (20), 4846-4856 (1992).
- McKinney, S. A., Declais, A. C., Lilley, D. M., Ha, T. Structural dynamics of individual Holliday junctions. Nature Structural Biology. 10 (2), 93-97 (2003).
- Joo, C., McKinney, S. A., Lilley, D. M., Ha, T. Exploring rare conformational species and ionic effects in DNA Holliday junctions using single-molecule spectroscopy. Journal of Molecular Biology. 341 (3), 739-751 (2004).
- Hyeon, C., Lee, J., Yoon, J., Hohng, S., Thirumalai, D. Hidden complexity in the isomerization dynamics of Holliday junctions. Nature Chemistry. 4 (11), 907-914 (2012).
- Ip, S. C., et al. Identification of Holliday junction resolvases from humans and yeast. Nature. 456 (7220), 357-361 (2008).
- Rass, U., et al. Mechanism of Holliday junction resolution by the human GEN1 protein. Genes & Development. 24 (14), 1559-1569 (2010).
- Liu, Y., et al. Crystal Structure of a Eukaryotic GEN1 Resolving Enzyme Bound to DNA. Cell Reports. 13 (11), 2565-2575 (2015).
- Chan, Y. W., West, S. GEN1 promotes Holliday junction resolution by a coordinated nick and counter-nick mechanism. Nucleic Acids Research. 43 (22), 10882-10892 (2015).
- van Gool, A. J., Hajibagheri, N. M., Stasiak, A., West, S. C. Assembly of the Escherichia coli RuvABC resolvasome directs the orientation of holliday junction resolution. Genes & Development. 13 (14), 1861-1870 (1999).
- Lee, S. H., et al. Human Holliday junction resolvase GEN1 uses a chromodomain for efficient DNA recognition and cleavage. eLife. 4, (2015).
- Chan, Y. W., West, S. C. Spatial control of the GEN1 Holliday junction resolvase ensures genome stability. Nature Communications. 5, 4844(2014).
- Liu, Y., Freeman, A. D., Declais, A. C., Lilley, D. M. J. A monovalent ion in the DNA binding interface of the eukaryotic junction-resolving enzyme GEN1. Nucleic Acids Research. 46 (20), 11089-11098 (2018).
- Zhou, R., et al. Junction resolving enzymes use multivalency to keep the Holliday junction dynamic. Nature Chemical Biology. 15 (3), 269-275 (2019).
- Bellendir, S. P., et al. Substrate preference of Gen endonucleases highlights the importance of branched structures as DNA damage repair intermediates. Nucleic Acids Research. 45 (9), 5333-5348 (2017).
- Sobhy, M. A., et al. Resolution of the Holliday junction recombination intermediate by human GEN1 at the single-molecule level. Nucleic Acids Research. 47 (4), 1935-1949 (2019).
- Sobhy, M. A., et al. Versatile single-molecule multi-color excitation and detection fluorescence set-up for studying biomolecular dynamics. Review of Scientific Instruments. 82 (11), 113702(2011).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences of the United States of America. 101 (24), 8936-8941 (2004).
- Lee, N. K., et al. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophysical Journal. 88 (4), 2939-2953 (2005).
- Rashid, F., et al. Initial state of DNA-Dye complex sets the stage for protein induced fluorescence modulation. Nature Communications. 10 (1), 2104(2019).
- Sambrook, J., Russell, D. W. Standard ethanol precipitation of DNA in microcentrifuge tubes. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Holden, S. J., et al. Defining the limits of single-molecule FRET resolution in TIRF microscopy. Biophysical Journal. 99 (9), 3102-3111 (2010).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L. Jr, Wiggins, C. H. Learning rates and states from biophysical time series: a Bayesian approach to model selection and single-molecule FRET data. Biophysical Journal. 97 (12), 3196-3205 (2009).
- Kou, S. C., Cherayil, B. J., Min, W., English, B. P., Xie, X. S. Single-molecule Michaelis-Menten equations. Journal of Physical Chemistry B. 109 (41), 19068-19081 (2005).
- Clegg, R. M., Murchie, A. I., Lilley, D. M. The solution structure of the four-way DNA junction at low-salt conditions: a fluorescence resonance energy transfer analysis. Biophysical Journal. 66 (1), 99-109 (1994).
- Pohler, J. R., Duckett, D. R., Lilley, D. M. Structure of four-way DNA junctions containing a nick in one strand. Journal of Molecular Biology. 238 (1), 62-74 (1994).
- Fogg, J. M., Lilley, D. M. Ensuring productive resolution by the junction-resolving enzyme RuvC: large enhancement of the second-strand cleavage rate. Biochemistry. 39 (51), 16125-16134 (2000).
- Sobhy, M. A., Joudeh, L. I., Huang, X., Takahashi, M., Hamdan, S. M. Sequential and multistep substrate interrogation provides the scaffold for specificity in human flap endonuclease 1. Cell Reports. 3 (6), 1785-1794 (2013).
- Rashid, F., et al. Single-molecule FRET unveils induced-fit mechanism for substrate selectivity in flap endonuclease 1. eLife. 6, e21884(2017).
- Zaher, M. S., et al. Missed cleavage opportunities by FEN1 lead to Okazaki fragment maturation via the long-flap pathway. Nucleic Acids Research. 46 (6), 2956-2974 (2018).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31 (5), 1106-1116 (2001).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten