Method Article
サイトファストと上流クラスタリング法FlowSOMとサイトスプロアを用いた高次元サイトメトリーデータの可視化と定量
要約
Cytofastは、クラスタリングからの出力を分析するために使用される視覚化ツールです。Cytofastを使用して、FlowSOMとCytosploreの 2 つのクラスタリング方法を比較できます。Cytofastは、質量サイトメトリーデータの定量的および定性的な概要を迅速に生成し、異なるクラスタリングアルゴリズム間の主な違いを強調することができます。
要約
大量のサイトメトリーによって生成されるデータの複雑さは、分析結果を迅速に可視化するための新しいツールを必要としています。細胞球団やFlowSOMなどのクラスタリング方法は、細胞クラスターの可視化と同定に使用されます。下流分析のために、新しく開発されたRパッケージCytofastは、クラスタリング法から結果を迅速に可視化することができる。Cytofastは、細胞クラスターの表色特性を考慮に入れ、細胞クラスターの存在量を計算し、次いでグループを定量的に比較する。このプロトコルは、腫瘍の課題に続いて腫瘍の課題に対する免疫系の変調(すなわち、ナチュラルキラー[NK]細胞応答)に基づく質量サイトメトリーデータの使用に対する細胞化剤の適用を説明する(PD-L1遮断)。FlowSOMおよびサイトスプロアによるサイトファストの有用性の実証を示す。細胞化は、グループ関連の免疫細胞クラスターの視覚的表現と免疫系組成との相関を迅速に生成します。クラスタリング分析では違いが見られますが、グループ間の分離は両方のクラスタリング方法で表示されます。Cytofastは、活性化NK細胞サブセットのより高い存在量を含むPD-L1処理によって誘導されるパターンを視覚的に示し、活性化マーカーのより高い強度(すなわち、CD54またはCD11c)を発現する。
概要
質量サイトメトリー(飛行時間によるサイトメトリー、またはCyTOF)は、何百万もの単一細胞における幅広い細胞内または細胞外バイオマーカーの検出を可能にします。質量サイトメトリーデータの高次元性は、SPADE1、FlowMaps2、FlowSOM 3、フェノグラフ4、VorteX 5、および足場マップ6のような細胞クラスタリング技術などの特定の解析ツールを必要とする。また、様々な次元低減ベースの技術が開発されている(すなわち、主成分解析[PCA]7、t分散確率的近傍埋め込み[t-SNE]8、階層確率論的隣人埋め込み[HSNE]9、均一多様体近似図10、及び拡散マップ11)を開発し、高速化、解釈及び可視化を向上させる。
高次元流量と質量細胞測定データの下流分析では、クラスター周波数と臨床結果とのリンクに関する統計的検査を実行する自動プロセスが欠けていることがよくあります。以前は、CytosploreまたはFlowSOMによるクラスタリング技術の視覚的および定量的な下流分析を可能にする、Cytofast 12と呼ばれるRベースのワークフローを開発しました。
ここで説明するプロトコルは、RでのCytofastの使用を明確にし、定量的および定性的なヒートマップとグラフを生成する方法を示しています。さらに、観察された免疫表現型と臨床結果との間の接続の決定を促進する。このレポートでは、FlowSOMとCytosploreという 2 つの異なるクラスタリング手順を使用した特定の質量サイトメトリー データセットの分析についても説明します。両方のクラスタリング方法でCytofastを用いることで、NK細胞の活性化表現型がPD-L1免疫チェックポイント遮断の影響を受けることを示す。
プロトコル
すべての動物実験はLUMC動物実験委員会によって承認され、オランダおよび欧州委員会のガイドラインに従ってLUMCの動物実験ガイドラインに従って実行された。
注:実験的セットアップのために、C57BL/6マウスを、リン酸緩衝生理食塩水(PBS)の濃度0.3 x106細胞/200μLのマウス大腸腫瘍MC38で右脇腹に皮下接種した。10日後、腫瘍が触知可能な場合、マウスをPD-L1遮断抗体(クローンMIH-5、200μg/マウス、腹腔内注射)で治療するか、または模擬処理した。腫瘍をPD-L1注射の3日後に切除し、ex vivoを処理し、38マーカー13を用いてCyTOF質量サイトメトリーにより分析した。
1. データ分析のための機器とソフトウェア
メモ: 2.4 GHz 以上のコンピュータ (Windows 7 以降) とプロセッサ I5、インストールされているメモリ RAM 6 GB、および 10 GB のハード ドライブの空き容量を使用します。RパッケージCytofastは、主にflowCore、pheatmap、およびggplotの既存の機能を使用しています。 R で実行されるコマンド ラインは、プロトコルに含まれます。R 命令のリソースはhttps://education.rstudio.com/にあります。
- Cytofastパッケージをインストールするには、R (バージョン "3.6") を起動し、次のコードを入力してバイオコンダクタバージョン 3.9 をインストールします。
(!必須名前空間("バイオックマネージャー"、静かに = TRUE))
install.packages("バイオックマネージャー")
バイオクマネージャー::インストール(「サイトファスト」) - 次の手順を実行して、パッケージが目的の環境に読み込まれていることを確認します。
図書館(サイトファスト)
2. クラスターの作成
メモ:サイトファストを用いてサイトスプロアとFlowSOMの2つのクラスタリング方法を紹介するために、PD-L1治療の3日後に腫瘍マイクロ環境におけるNK細胞(CD161+)を分析する。
- によって実行されるクラスタリングCytosplore
- Cytosploreをダウンロードしてインストールした後、サイトスプロア.org>、[ ファイル ] メニューの [.fcs ファイル ] (補足ファイル 1.1 ~ 1.8 [Cytosplore 入力ファイル]) をアップロードします。サイトスプロアでFCSファイルを開きます。プロンプトが表示されたら[チャンネルとして一意のサンプルタグを追加]をクリックして、チャンネルとして一意のサンプルタグを追加し、双曲線アークシン変換の補因子を選択します(デフォルトは5)。
- [H-SNEの実行] を選択し、HSNE レベル 3 を実行して、マップが生成されるまで待ちます。
メモ:この手順は、分析されたセルの数と選択した HSNE のレベルに応じて、時間がかかる場合があります。 - 最初の HSNE レベルで、CD161 の正のセルを確認します。CD161+セルを選択し、[選択範囲にズームイン ] を右クリックします。2 番目のレベルでは、CD161+イベントのみを使用して 3 番目のレベルに到達する手順を繰り返します。
- 最後の tSNE マップが生成されたら、tSNE マップを右クリックしてCytosploreによって定義されたクラスターを保存し、[クラスターの保存] を選択します。Cytosploreによって求められた出力ファイルのディレクトリを選択し、このディレクトリを使用して .fcs ファイルを R に読み込むので、この場所をメモします。
メモ:サブセットの数は、sigma 値を変更することで手動で変更できます。sigma 値はデフォルトで 30 に設定されています。ただし、実際のサブセット数は入力によって異なります。ここでは、Cytosploreは 10 個の異なるサブセットを検出し、各ファイルは 1 つのサブセットを表します。 - 出力ファイルの名前を変更する場合は、簡易名 (文字のみ) を使用すると、識別が容易になり、処理が容易になります。[ 保存 ] を選択して出力ファイルを保存します。
メモ: 保存後、フォルダが .fcs ファイルを含むCytosploreによって作成され、各ファイルはCytosplore内の識別されたクラスタに対応しています。次のステップは、Cytofastの助けを借りてRにファイルをロードすることです。ここでは、生成された出力ファイルは補足ファイル 2.1 – 2.10 (Cytosplore 出力ファイル) で提供されます。 - 指定された関数を使用して、Cytosploreによって生成された出力ファイルを R にロードします。
dirFCS <- "C:\\ユーザー\\ユーザー名\\デスクトップ\\トースタディ"
cfデータ <- 読み取りサイトスプロールFCS(dir = dirFCS、colNames = "説明") - 「時間」や「背景」などのパラメータを削除して、データを消去します。不要なパラメータに関連する列の位置を確認し、マトリックスから削除します。
コル名(cfData@expr)
cfData@expr<- cfData@expr[-c(3,4,6,8:10,46:49,51:54)]
メモ: 不要な列は、生成されたマトリックスの列名を読み取ることによって表示されます。colnames(cfData@expr) をもう一度実行して、目的のパラメーターのみが取得されていることを確認します。 - 最初にリネージュ マーカーが表示され、次に機能マーカーが表示されるように、マーカーの順序を変更します。
cfData@expr<- cfData@expr[,c(1,2,3,35,36,31,9,10,18,8,37,20,
29,40,5,30,33,11,34,14,19,
32,28,6,7,4,12,13,17,16,15,
21,22,24,25,26,27,38,39)]
メモ: ステップ 2.1.8 はオプションです。 - 臨床情報を含むスプレッドシートメタファイルをアップロードして、メタデータファイルをCytosploreから生成されたデータにリンクします (補足ファイル 3)。
ライブラリ(読み取り)
メタ <- read_excel("C:\\ユーザー\\ユーザー名\\デスクトップ\\sample_id.xlsx")
cfData@samples
メモ: Cytosploreによって実行されているクラスタリングが完了しました。Cytosploreへの代替クラスタリング オプションはFlowSOMで、セクション 2.2 で説明されています。2 つのクラスタリング手順のいずれかを実行したら、視覚化手順 (セクション 3) に進みます。
- によって実行されるクラスタリングFlowSOM
- 最初に次のコマンドを実行して、R にFlowSOMをインストールします。
(!必須名前空間("バイオックマネージャー"、静かに = TRUE))
install.packages("バイオックマネージャー")
バイオクマネージャー::インストール(「フローソム」)
ライブラリ(フローソム) - 同様の方法を使用して flowCore パッケージをインストールし、次の実行で環境に読み込みます。
(!必須名前空間("バイオックマネージャー"、静かに = TRUE))
install.packages("バイオックマネージャー")
バイオクマネージャー::インストール(「フローコア」)
ライブラリ(フローソム) - 既に CD161+ イベントでゲートされていた補足ファイル 4.1 ~ 4.8 (FCS FlowSOM 入力) で提供される生データを Read.flowSet 関数を使用して R にロードします。
fcs_raw<- 読み取り.flowSet(パス="C:\\ユーザー\\ユーザー名\\\デスクトップ\\\トクラスタ"、パターン = ".fcs"、変換 = FALSE、truncate_max_range = FALSE、シード = 123) - 適切な列を選択して関連する生物学的マーカー(「背景」または「時間」を削除)を選択し、以下のコードに示すようにarcsinh5の方法でデータを変換します(ここでは、列1、2、4、5、6、17、21、24、25、34、35、37、38、51、任意の生物学的マーカーに対応していません)。Cytosploreで以前に適用した 5 の補因子を適用するには、以下の関数でcofactor=5を選択します。
fcs_raw<- fsApply(fcs_raw、 関数(x, 補因子=5){
コル名(x) <- fcs_raw[1]]@parameters@data$desc
expr <- exprs(x)
expr <- アシン(expr[,,c,5,6,17,21,24,25,34,35,37,38,51)]/ 補因子)
exprs(x) <- expr
戻る(x)}) - FlowSOM関数を使用してデータをクラスター化します。FlowSOMとCytosploreを比較するには、以前にCytosploreによって生成された出力として、10 個のサブセットにデータをクラスタ化することを選択します。
fsom <- フローソム(fcs_raw, 変換関数 = FALSE, スケール = FALSE,
スケーリングされた中心 = FALSE、スケーリングされたスケール = FALSE、サイレント = FALSE、colsToUse = c(1:37)
nClus = 10、maxMeta = 10、重要度 = NULL、シード = 123)
メモ: これはユーザーが手動で変更できます。 - 各セルを、識別されたサブセットとサンプル ID に割り当てます。
subset_id <- as.factor(fsom$FlowSOM$マップマッピング[,1])
レベル(subset_id) <- fsom$メタクラスタリング
ヘッド(subset_id) - グループ割り当てを含む R (補足ファイル 5で使用可能) にメタデータ ファイルを読み込み、.fcs ファイルにリンクします。
サンプル ID <- read_excel("C:\\ユーザー\\ユーザー名\\デスクトップ\\sample_id.xlsx")
サンプル ID <- na.省略 (サンプル)
サンプル ID サンプル ID <- as.factor(サンプル ID サンプル ID)
サンプル$グループ <- as.factor(サンプル$グループ)
サンプルid$CSPLR_ST .lt;- as.factor(サンプル$CSPLR_ST)
サンプル ID <- as.data.frame(サンプル ID)
名前(サンプルID)[3] <- "サンプルID"
サンプル ID <- lapply(fsom$FlowSOM$メタデータ, 関数 (x){{rep(x[1], 各 = 長さ(x[1]:x[2])})
attr(サンプル ID,'名前') <- NULL
サンプル ID <- as.factor(リスト解除(サンプル ID))
サンプル ID <- データ フレーム(サンプル ID)
レベル(サンプル ID) <- 貼り付け("ID", 1:dim(サンプルID)[1], sep="_")
df <- データ.フレーム(subset_id、サンプル ID、fsom$FlowSOM$データ[, c(1:37)])
名前を変更する <- データ.フレーム(コルパー=fcs_raw[1]]@parameters@data$desc)
コル名(df) <- c("クラスタ ID","サンプル ID", 名前の変更 $colpar[c(1:37)])
df$ クラスタ ID <- as.factor(df$ クラスタ ID)
df$サンプル ID <- as.factor(df$サンプル ID) - 次のスクリプトを実行して、FlowSOMから取得したデータフレームに基づいて cfList を作成します。
cfデータ <- cfList(サンプル = サンプル、
expr = df) - マーカーを順序を変更して、サイトスプロー解析の出力と同様に表示されます。
cfData@expr<- cfData@expr[,c(1,2,34,36,37,10,23,24,31,22,
38,15,8,3,27,9,11,28,35,26,
14,33,17,20,21,18,25,29,13,
30,12,16,32,4,5,6,7,19,39)]
メモ: FlowSOMによるクラスタリングが完了しました。次に、クラスタリング出力の視覚化を行う。
- 最初に次のコマンドを実行して、R にFlowSOMをインストールします。
3. 可視化:後処理クラスタリング分析
メモ: この手順は、両方のクラスタリング方法に共通する方法です。したがって、FlowSOMまたはサイトスプロアを使用してクラスタリングした後に実行できます。
- ヒートマップを作成する前に、次のコードに示すように、関数 cellCounts を使用してサンプルごとのカウント テーブルを生成します。一部のクラスターには他のクラスターよりも少ないセルが含まれているため、関数 cellCounts 内で "scale = TRUE" を指定してクラスターごとのデータをスケーリングし、サンプル間の分散を簡単に確認できるようにします。
cfデータ <- セル数(cfData,周波数 = TRUE,スケール = TRUE)
メモ: データを視覚化できるようになりました。 - ヒートマップで視覚化します。
メモ:このパッケージの主な機能の1つは、作成されたクラスターの表現型とサンプルに関する異質性を視覚化するために使用されるcytoHeatmapsです。
サイトヒートマップ (cfData,グループ="グループ",凡例=真) - 箱ひげ図を使用した視覚化
メモ: 関数 cytoBoxplots を呼び出すことで、データを定量的に表現できます。この関数の出力は、各クラスターの各サンプルの比率を表します。- 手順 3.1 で行ったようにセル数を生成しますが、各クラスターの頻度を取得するようにデータをスケーリングしないでください。
cfデータ <- セル数(cfData,周波数 = TRUE,スケール = FALSE)
サイトボックスプロット(cfData,グループ=「グループ」)
- 手順 3.1 で行ったようにセル数を生成しますが、各クラスターの頻度を取得するようにデータをスケーリングしないでください。
- 中央値強度信号ヒストグラムを使用した可視化
メモ:データは、中央値強度信号ヒストグラムを表すことによって視覚化することもできます。- CD45、CD11c、CD54の3つのマーカーの発現強度を視覚化します。
- 次の行を呼び出して、目的のマーカーの名前を確認します。マーカー名を書き留め、msiPlot 関数に含めます。
名前(cfData@expr)
メモ: ここでは、CD45、CD11c、および CD54 に焦点を当てます。マーカーの正確なスペルを確認し、必要に応じて調整します。
msiPlot(cfData, マーカー = c("89Y_CD45","167Er_CD11c","164Dy_CD54"), byグループ='グループ')
結果
ワークフロー Cytofast(図1)は、分析ソフトウェア(FlowSOMまたはCytosplore)によって最初にクラスタ化されたデータの定量的かつ定性的な概要を提供することを意図しています。Cytofastは、解析で識別されたすべてのクラスターのヒートマップを含む、いくつかの可能な出力を実行し、マーカー式に基づいています (図 2および図 3)。上部の樹形図は、識別されたクラスター間の階層的な類似性を表します。上部パネルには、各サンプル内の対応するサブセットの相対量を示す別のヒートマップが表示されます。右側の樹形図は、サンプル間の類似性を示し、サンプル間のユークリッド距離で実行される階層クラスタリングに基づいています。組み合わせたヒートマップはFlowSOMに続き、図2ではCytofast、続いて図3にCytosploreが続きます。 図 4および図 5に示すように、Cytofastを使用してデータを定量的に表示し、結果を箱ひげ図 (cytoBoxplots 関数を使用) で表示することもできます。
2つの異なる方法の間で同様のクラスターが見出され(例えば、Cytosploreからのクラスター8はFlowSOMからのクラスター10に対応)、PD-1およびLAG-3のようないくつかの阻害マーカーの共発現は両方の方法でまだ見られた。どちらのクラスタリング方法でも、PD-L1との間での判別が可能でした。PBSはマウスを治療した。対照的に、両方の方法の違いがいくつか強調表示されます。FlowSOMは 2 つのクラスタ (MHC-II+) を識別し、Cytosploreは 1 つのクラスタ (MHC-II+dim)のみを示します。これは、NK細胞がCD161+細胞上で手動でゲートされ、その後FlowSOMによってさらに処理された最初のゲーティング戦略によるものです。しかし、Cytosploreは最初のHSNEレベルでCD45+母集団から自動的に細胞をゲートし、その後、より高い階層レベルでクラスタリングされました。したがって、Cytosploreは、手動ゲーティングがCD161に焦点を当てた方法よりも正確にNK細胞サブセットを定義した。それにもかかわらず、サンプルの階層クラスタリングは、右側のデンドログラムに示すように保存され、2つのグループ間の分離(PD-L1とPBS)が選択されたクラスタリング方法に依存していないことを示す。
クラスターの数は、両方の方法を使用して手動で定義できます。Cytofastを使用すると、ユーザーはデータの異質性を評価し、データを分割するクラスターの数を選択する方法に関する洞察を提供できます。msiPlot 関数 (ステップ 3.4.2) などの他の機能は、グループごとの各マーカーの中央値信号強度 (MSI) プロットを示します (図 6および図7)。この機能は、PD-L1処理群のNK細胞におけるCD54またはCD11cの発現の増加などの世界的な変化を検出することを可能にする。オプション機能は、棒グラフやその他のデータ表現方法でのデータの表示など、Cytofastパッケージに組み込むことができます。後者は、Rによって生成することができるggplotツールを追加する必要があります。
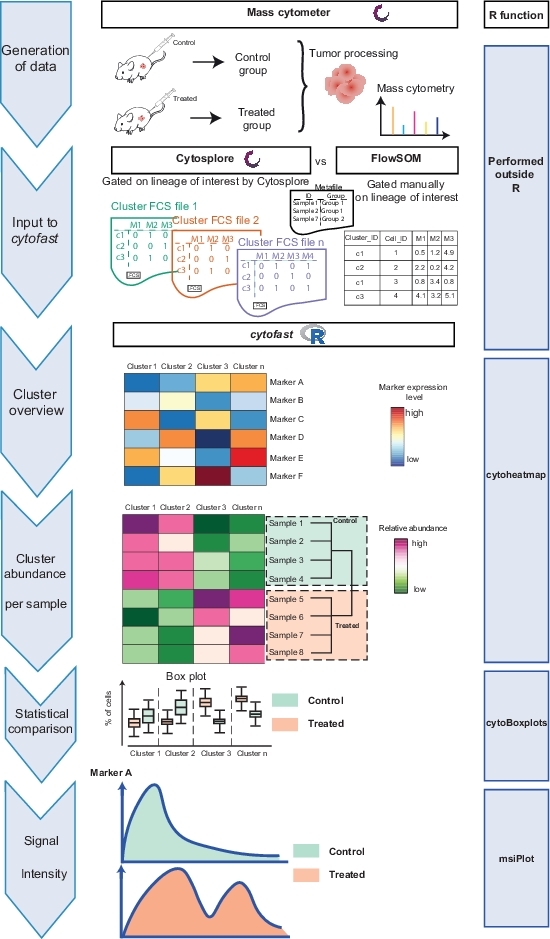
図1:サイトファストパッケージのワークフローこのデータは、免疫療法による治療の3日後に腫瘍から集団サイトメトリーによって生成されたか、または未治療のままにした。2 つの異なるクラスタリング技術を比較しました:サイトスプロールとFlowSOM.サイトファストは、2つの技術の違いを可視化するために使用された。この図の大きなバージョンを表示するには、ここをクリックしてください。
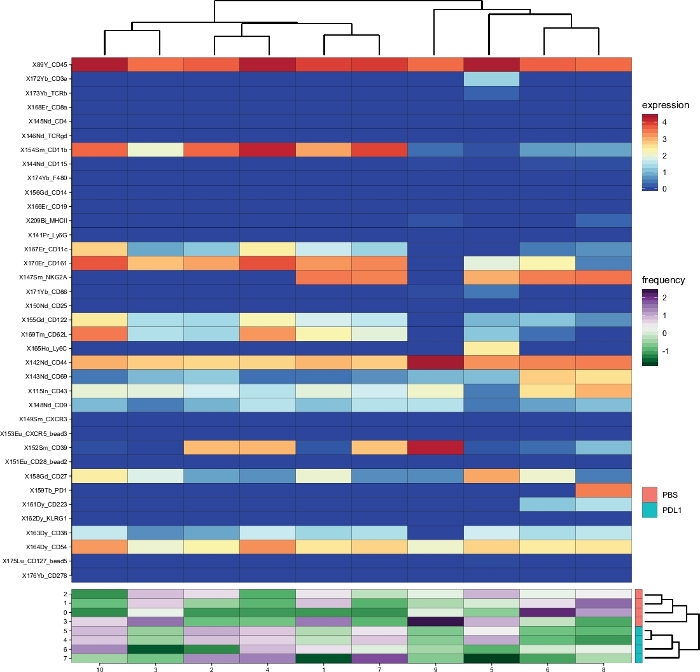
図 2: Cytosploreに続くCytofastによって分析されたグループごとのクラスターの概要とクラスターの存在量 。全NK細胞クラスターのヒートマップ(CD161+細胞は細胞スプロールによって自動的に定義される)、免疫療法の3日後に同定した(PD-L1)。示されたデータは、Cytosploreクラスタリングに基づいており、未処理およびPD-L1処理されたグループからプールされる。ArcSinh5 変換式マーカーのレベルは、虹のスケールで表示されます。下部パネルでは、各サンプルの相対的な存在量は緑色から紫色のスケールで表されます。右側の樹形図は、サブセット周波数に基づくサンプル間の類似性を表します。周波数スケールは平均の分散を表します。低周波または高周波は、それぞれ緑色または紫色で表されます。この図の大きなバージョンを表示するには、ここをクリックしてください。
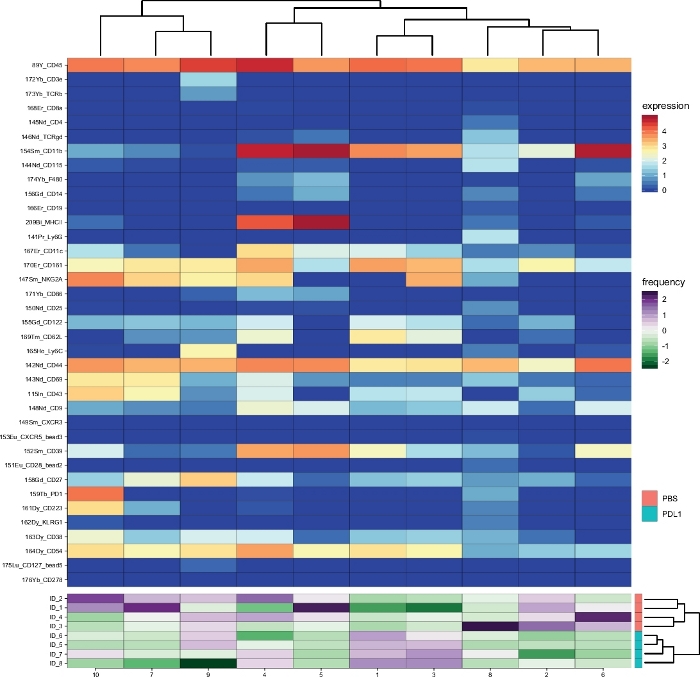
図 3: Cytofastフォロー FlowSOMによって分析されたグループごとのクラスターの概要とクラスターの存在量免疫療法(PD-L1)の3日後に同定された全NK細胞クラスター(CD161+イベントにプリゲート)のヒートマップ。表示されるデータはFlowSOMクラスタリングに基づいており、未処理のグループと PD-L1 処理されたグループからプールされます。ArcSinh5 変換式マーカーのレベルは、虹のスケールで表示されます。下部パネルでは、各サンプルの相対的な存在量は緑色から紫色のスケールで表されます。右側の樹形図は、サブセット周波数に基づくサンプル間の類似性を表します。周波数スケールは平均の分散を表します。低周波または高周波は、それぞれ緑色または紫色で表されます。この図の大きなバージョンを表示するには、ここをクリックしてください。
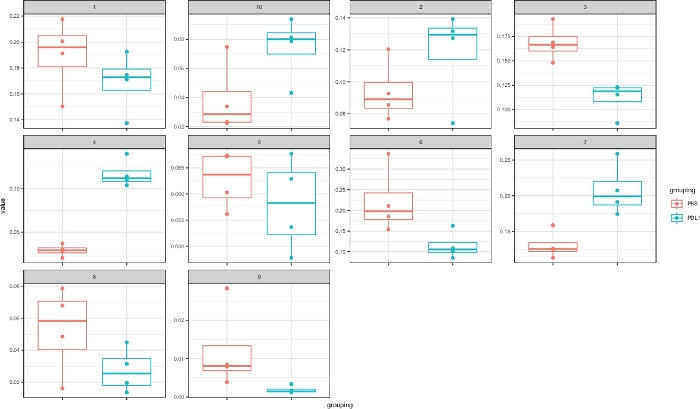
図 4: Cytosploreによって定義されたクラスターの箱ひげ図を使用したサイトファスト表現 。各クラスターの頻度は箱ひげ図で表され、2つのグループ(PBSとPD-L1)に分けられます。1 つの個々のドットは、1 つのマウスに対応します。この図の大きなバージョンを表示するには、ここをクリックしてください。
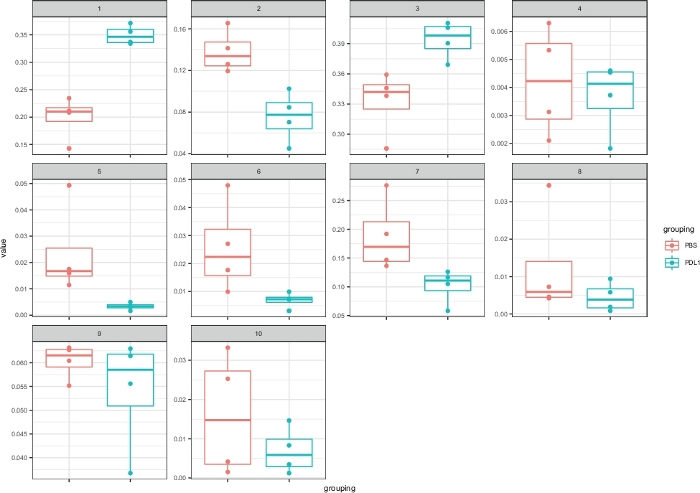
図 5: FlowSOMで定義されたクラスターの箱ひげ図を使用したサイトファスト表現 。各クラスターの頻度は箱ひげ図で表され、2つのグループ(PBSとPD-L1)に分けられます。1 つの個々のドットは、1 つのマウスに対応します。この図の大きなバージョンを表示するには、ここをクリックしてください。
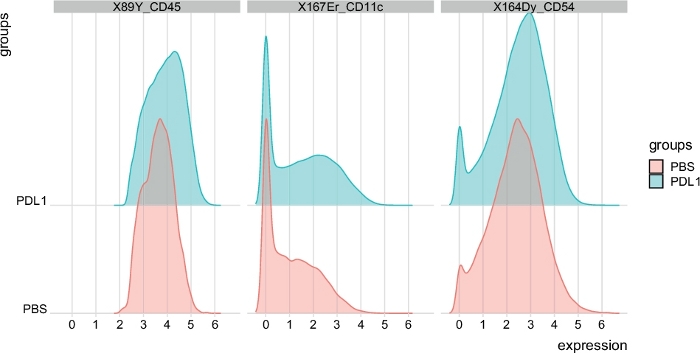
図6:Cytosploreによって自動的にゲートされたNK細胞からの信号強度プロットの分布。信号強度の分布は、CD45、CD11c、および CD54 の 3 つの特定のマーカーのヒストグラムに示されています。この図の大きなバージョンを表示するには、ここをクリックしてください。
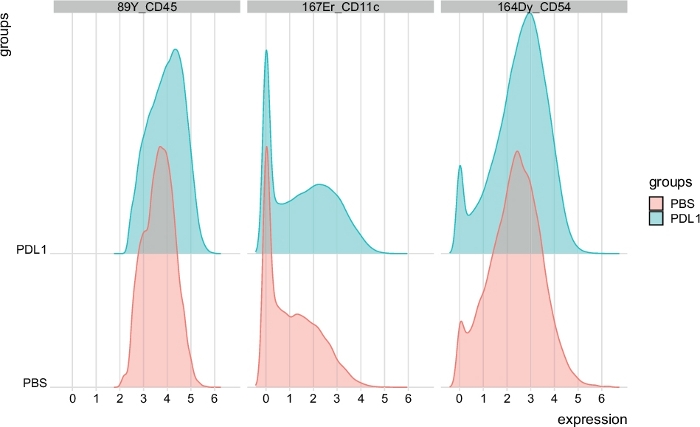
図7:FlowSOMによって自動的にゲートされたNKセルからの信号強度プロットの分布。信号強度の分布は、CD45、CD11c、およびCD54の3つの特定のマーカーのヒストグラムに示され、グループPBSおよびPD-L1によって分離される。この図の大きなバージョンを表示するには、ここをクリックしてください。
補足ファイル 1.1–1.8.このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
補足ファイル 2.1– 2.10.このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
補足ファイル 3.このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
補足ファイル 4.1–4.8.このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
ディスカッション
Cytofastは、治療固有の細胞サブセットを強調表示および定量することにより、細胞測定データの迅速かつグローバルな探索を提供する迅速な計算ツールです。説明したプロトコルは、CytosploreまたはFlowSOMを使用してクラスタリング分析をさらに処理することを目的としています。他のクラスタリング解析ツールはCytofastに適していますが、各セルをサブセットに割り当てるにはCytofastを使用する必要があります。ただし、Cytofastはクラスタリング方法ではないため、使用する前にクラスタリング手順が必要です。
ここで行った分析は、腫瘍微小環境における特定のCD161+NK細胞サブセットがPD-L1遮断に敏感であることを示した。これは、クラスタリング法としてCytosploreとFlowSOMの両方を使用して観察された表現型と豊富さの変化によって示されました。どちらの方法も、わずかに異なる周波数(Cytosploreの場合は15%-20%、FlowSOMの場合は30%~40%)でメインNK細胞クラスター(CD11b + NKG2A+)を区別しました。図2と図3の右側のパネルに表示される両方のデンドログラムが同様の結果を示したため、豊富さとこの近似の違いはグローバルパターンに影響を与えませんでした。Cytofastを使用することにより、NK細胞クラスター表現型および豊富さの分析に基づいてPD-L1処理および未処理マウスを分離することが可能である(選択されたクラスタリング方法とは無関係)。
記録されたパラメータに応じて、プロトコルを変更する必要があります。具体的には、クラスタリング分析の実行中に、時間や背景などの特定のパラメーターを削除する必要があります。さらに、各セルをサブセットに割り当てることが重要です。cfData 関数は、サンプルごとのクラスターごとの生のセル数を cfList に追加するだけです。このステップから、セクション 3 で説明したようにサイトヒートマップを構築できます。
Cytofastは、異なるクラスタリング方法13を比較するための可視化および定量ツールとして正常に使用されている。このRパッケージは、臨床変数を使用してクラスタのグループ間の関連をテストできるグローバルテスト14などの高度な機能とも互換性があります。将来的には、グローバルテストツールやその他のアルゴリズムをCytofastと統合して、より詳細な視覚化と定量化を実現できます。
開示事項
著者たちは何も開示する必要はない。
謝辞
我々は、提案番号675743(ISPIC)の下でH2020 MSCA賞の欧州委員会からの資金提供を認めます。私たちは、プロトコルをテストするためのテチェ・ファン・デル・スルイスとアイリス・パルディックに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Computer | Dell | NA | NA |
参考文献
- Anchang, B., et al. Visualization and cellular hierarchy inference of single-cell data using SPADE. Nature Protocols. 11 (7), 1264-1279 (2016).
- Zunder, E. R., Lujan, E., Goltsev, Y., Wernig, M., Nolan, G. P. A continuous molecular roadmap to iPSC reprogramming through progression analysis of single-cell mass cytometry. Cell Stem Cell. 16 (3), 323-337 (2015).
- Van Gassen, S., et al. FlowSOM: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry A. 87 (7), 636-645 (2015).
- Levine, J. H., et al. Data-Driven Phenotypic Dissection of AML Reveals Progenitor-like Cells that Correlate with Prognosis. Cell. 162 (1), 184-197 (2015).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nature Methods. 13 (6), 493-496 (2016).
- Spitzer, M. H., et al. An interactive reference framework for modeling a dynamic immune system. Science. 349 (6244), 1259425 (2015).
- Hotelling, H. Analysis of a complex of statistical variables into principal components. Journal of Educational Psychology. 24 (6), 417-441 (1933).
- van der Maaten, L., Hinton, G. Visualizing Data using t-SNE. Journal of Machine Learning Research. , (2008).
- Pezzotti, N., Hollt, T., Lelieveldt, B., Eisemann, E., Vilanova, A. Hierarchical Stochastic Neighbor Embedding. Computer Graphics Forum. 35 (3), 21-30 (2016).
- Becht, E., et al. Dimensionality reduction for visualizing single-cell data using UMAP. Nature Biotechnology. 37, 38 (2018).
- Haghverdi, L., Buettner, F., Theis, F. J. Diffusion maps for high-dimensional single-cell analysis of differentiation data. Bioinformatics. 31 (18), 2989-2998 (2015).
- Beyrend, G., Stam, K., Höllt, T., Ossendorp, F., Arens, R. Cytofast: A workflow for visual and quantitative analysis of flow and mass cytometry data to discover immune signatures and correlations. Computational and Structural Biotechnology Journal. 16, 435-442 (2018).
- Beyrend, G., et al. PD-L1 blockade engages tumor-infiltrating lymphocytes to co-express targetable activating and inhibitory receptors. Journal for ImmunoTherapy of Cancer. 7 (1), 217 (2019).
- Goeman, J. J., van de Geer, S. A., de Kort, F., van Houwelingen, H. C. A global test for groups of genes: testing association with a clinical outcome. Bioinformatics. 20 (1), 93-99 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved