Method Article
Cytofast 및 업스트림 클러스터링 방법 FlowSOM 및 Cytosplore를 사용하여 고차원 세포 측정 데이터의 시각화 및 정량화
요약
Cytofast는 클러스터링에서 출력을 분석하는 데 사용되는 시각화 도구입니다. Cytofast는 두 가지 클러스터링 방법을 비교하는 데 사용할 수 있습니다: FlowSOM 및 Cytosplore. Cytofast는 질량 세포 분석 데이터의 정량적 및 질적 개요를 신속하게 생성하고 서로 다른 클러스터링 알고리즘 간의 주요 차이점을 강조 할 수 있습니다.
초록
질량 세포 측정에 의해 생성된 데이터의 복잡성으로 인해 분석 결과를 신속하게 시각화할 수 있는 새로운 도구가 필요했습니다. Cytosplore 또는 FlowSOM과 같은 클러스터링 방법은 세포 클러스터의 시각화 및 식별에 사용됩니다. 다운스트림 분석을 위해 새로 개발된 R 패키지인 Cytofast는클러스터링 방법으로 결과를 신속하게 시각화할 수 있습니다. Cytofast는 세포 클러스터의 자형특성 특성화를 고려하여 세포 클러스터 풍부도를 계산한 다음 그룹을 정량적으로 비교합니다. 이 프로토콜은 종양 미세 환경(즉, 자연 살인자 [NK] 세포 반응)에서 면역 요법(PD-L1 봉쇄)에 이어 종양 챌린지에 기초하여 대량 세포 측정 데이터의 사용에 Cytofast의 적용을 설명한다. FlowSOM 및 Cytosplore와 사이토 패스트의 유용성의 데모가 표시됩니다. Cytofast는 급속하게 면역 계통 조성과 그룹 관련 면역 세포 클러스터 및 상관관계의 시각적 표현을 생성합니다. 클러스터링 분석에서는 차이점이 관찰되지만 두 군집 화 방법 모두에서 그룹 간 구분이 표시됩니다. Cytofast는 활성화된 NK 세포 서브세트의 더 높은 풍부함을 포함하는 PD-L1 처리에 의해 유도된 패턴을 시각적으로 나타내며, 활성화 마커(즉, CD54 또는 CD11c)의 더 높은 강도를 발현한다.
서문
질량 세포 분석 (비행 시간 또는 CyTOF에 의한 세포 분석)은 수백만 개의 단일 세포에서 광범위한 세포 내 또는 세포 외 바이오 마커를 검출 할 수 있습니다. 질량 세포 분석 데이터의 고차원 특성은 SPADE1, FlowMaps2, FlowSOM3, 현상지명4, VorteX5및 스캐폴드 맵6과같은 셀 클러스터링 기법과 같은 특정 분석 도구가 필요합니다. 또한, 다양한 차원 감소 기반 기술이 개발되었습니다 (즉, 주요 구성 요소 분석 [PCA]7,t-분산 확률 이웃 포함 [t-SNE]8,계층 적 확률 이웃 포함 [HSNE]9,균일 한 매니 폴드 근사치 및 프로젝션 [UMAP] 10, 및 확산 맵11)높은 차원 의 해석을 향상시키기 위해, 및 확산지도11)
고차원 유동 및 질량 세포 측정 데이터의 다운스트림 분석은 종종 클러스터 빈도에 대한 통계 적 테스트를 수행하고 임상 결과와 연결하는 자동 프로세스가 부족합니다. 이전에는 Cytosplore 또는 FlowSOM에의한 클러스터링 기술의 시각적 및 정량적 다운스트림 분석을 허용하는 Cytofast12로알려진 R 기반 워크플로를 개발했습니다.
여기에 설명된 프로토콜은 R에서 Cytofast의 사용을 명확히 하고 정량적 및 정성적 히트맵 및 그래프를 생성하는 방법을 보여줍니다. 또한, 관찰 된 면역 표현형과 임상 결과 사이의 연결의 결정을 용이하게합니다. 이 보고서는 또한 FlowSOM 및 Cytosplore: 두 개의 서로 다른 클러스터링 절차를 사용하여 특정 질량 세포 분석 데이터 세트의 분석을 설명합니다. 두 군집 방법 모두와 Cytofast를 사용하여, NK 세포의 활성화 표현형이 PD-L1 면역 체크포인트 봉쇄에 의해 영향을 받는 것으로 나타났다.
프로토콜
모든 동물 실험은 LUMC의 동물 실험 위원회의 승인을 받았으며 네덜란드 및 유럽 위원회의 지침에 따라 LUMC의 동물 실험 지침에 따라 실행되었습니다.
참고: 실험 적 설정의 경우, C57BL / 6 마우스는 0.3 x 106 세포 / 200 μL의 농도로 뮤린 결장 종양 MC38로 오른쪽 측면에 피하 접종되었습니다 .PBS (PBS). 10일 후, 종양이 만져졌을 때, 마우스를 PD-L1 차단 항체(클론 MIH-5, 200 μg/마우스, 복막 내 주사)로 처리하거나 모의 처리하였다. 종양은 PD-L1 주사 후 3일 후 절제되었고, 생체내 를 처리하고, 38개의 마커13을사용하여 CyTOF 질량 세포측정에 의해 분석하였다.
1. 데이터 분석을 위한 장비 및 소프트웨어
참고: 2.4 GHz 또는 이와 동등한 에서 컴퓨터 (Windows 7 이상) 및 프로세서 I5를 사용하여 설치된 메모리 RAM 6 GB 및 10 GB의 사용 사용 하드 드라이브 공간. R 패키지 Cytofast는 기존 기능을 사용합니다: 주로 flowCore, pheatmap및 ggplot. R에서 실행할 명령줄이 프로토콜에 포함됩니다. R 지침에 대한 리소스는 https://education.rstudio.com/.
- Cytofast 패키지를 설치하려면 R(버전 "3.6")을 시작하고 다음 코드를 입력하여 생체 전도체 버전 3.9를 설치합니다.
(!필요네임 스페이스("BiocManager", 조용히 = TRUE))
설치.패키지("BiocManager")
BiocManager::설치("세포 파기") - 다음을 실행하여 패키지가 원하는 환경에서 로드되었는지 확인합니다.
라이브러리(사이토패스트)
2. 클러스터 만들기
참고: 사이토파스트와함께 Cytosplore 및 FlowSOM을 두 개의 군집 방법을 선보이기 위해, 종양 미세 환경에서 NK 세포 (CD161+ +)를PD-L1 처리 후 3 일 후에 분석한다.
- 클러스터링에 의해 수행됩니다.Cytosplore
- 다운로드 및 Cytosplore를설치 한 후 , 에서 호스팅 Cytosplore.org>,파일을 클릭하여 .fcs 파일(보충 파일 1.1-1.8 [Cytosplore 입력 파일])를 업로드 | CYtosplore에서FCS 파일을 엽니다. 메시지가 표시될 때 고유한 샘플 태그 추가를 클릭하고 쌍곡선 아크신 변환을 위한 보조 팩터를 선택하여 고유한 샘플 태그를 채널로 추가합니다(기본값은 5).
- H-SNE 실행을선택하고 HSNE 수준 3을 실행하고 맵이 생성될 때까지 기다립니다.
참고: 이 단계는 분석 된 셀의 수와 선택한 HSNE 수준에 따라 약간의 시간이 필요할 수 있습니다. - 첫 번째 HSNE 수준에서 CD161에 대해 양수인 셀을 확인합니다. CD161+ 셀을 선택하고 선택 영역으로 확대/축소를마우스 오른쪽 단추로 클릭합니다. 두 번째 수준에서는 CD161+ 이벤트만 사용하여 세 번째 수준에 도달하는 절차를 반복합니다.
- 마지막 tSNE 맵이 생성되면 TSNE 맵을 마우스 오른쪽 단추로 클릭하여 Cytosplore에 의해 정의된 클러스터를 저장하고 클러스터 저장을 선택합니다. Cytosplore에 의해 프롬프트로 출력 파일의 디렉토리를 선택하고이 디렉토리는 R에 .fcs 파일을로드하는 데 사용되기 때문에이 위치를 기록하십시오.
참고: 하위 집합의 수는 시그마 값을 변경하여 수동으로 변경할 수 있습니다. 시그마 값은 기본적으로 30으로 설정됩니다. 그러나 실제 하위 집합 수는 입력에 따라 다릅니다. 여기서 Cytosplore는 각 파일이 하나의 하위 집합을 나타내는 10개의 서로 다른 하위 집합을 감지했습니다. - 출력 파일의 이름을 바꿀 때 간단한 이름(문자만 해당)을 사용하면 식별및 추가 처리가 더 쉬워집니다. 저장 을선택하여 출력 파일을 저장합니다.
참고: 저장 후, 폴더는 Cytosplore에서 식별 된 클러스터에 해당하는 각 파일과 .fcs 파일Cytosplore에의해 생성되고있다 . 다음 단계는 Cytofast의도움으로 파일을 R로 로드하는 것입니다. 여기서 생성된 출력 파일은 보조 파일 2.1-2.10(Cytosplore 출력 파일)에 제공됩니다. - Cytosplore에 의해 생성된 출력 파일을 지정된 기능인 readCytosploreFCS를 사용하여 R로 로드합니다.
dirFCS <- "C:\\\사용자\\\\바탕 화면\\\연구"
cfData <- 읽기CytosploreFCS (dir = dirFCS, 이름 = "설명") - "시간" 및 "백그라운드"와 같은 일부 매개 변수를 제거하여 데이터를 정리합니다. 불필요한 매개 변수와 관련된 열의 위치를 확인하고 행렬에서 제거합니다.
cfData@expr)
cfData@expr<- cfData@expr[-c(3,4,6,8:10,46:49,51:54)]
참고: 생성 된 행렬의 열 이름을 읽고 불필요한 열을 볼 수 있습니다. cfData@expr)를 다시 실행하여 원하는 매개 변수만 가져오도록 합니다. - 계보 마커가 먼저 표시되고 기능 마커가 표시되도록 마커를 다시 정렬합니다.
cfData@expr<- cfData@expr[, c(1,2,3,35,36,31,9,10,18,8,37,20,
29,40,5,30,33,11,34,14,19,
32,28,6,7,4,12,13,17,16,15,
21,22,24,25,26,27,38,39)]
참고: 2.1.8 단계는 선택 사항입니다. - 임상 정보가 포함된 스프레드시트 메타파일을 업로드하여 Cytosplore에서 생성된 데이터에 메타 데이터 파일을 연결합니다(보충파일 3).
라이브러리(읽기 슬)
메타 <- read_excel ("C:\\\사용자\\\\\바탕 화면\\\sample_id.xlsx")
cfData@samples&- 데이터.프레임(메타)
참고: Cytosplore에 의해 수행되는 클러스터링이 이제 완료되었습니다. Cytosplore에 대한 대체 클러스터링 옵션은 FlowSOM이며 섹션 2.2에 설명되어 있습니다. 두 클러스터링 단계 중 하나가 수행되면 시각화 단계(섹션 3)를 진행합니다.
- 클러스터링에 의해 수행됩니다.FlowSOM
- 다음 명령을 실행하여 R에 FlowSOM을 먼저 설치합니다.
(!필요네임 스페이스("BiocManager", 조용히 = TRUE))
설치.패키지("BiocManager")
BiocManager::설치("FlowSOM")
라이브러리(흐름솜) - 유사한 방법을 사용하여 flowCore 패키지를 설치하고 다음을 실행하여 환경에 로드합니다.
(!필요네임 스페이스("BiocManager", 조용히 = TRUE))
설치.패키지("BiocManager")
BiocManager::설치("흐름 코어")
라이브러리(흐름솜) - read.flowSet 함수를 사용하여 CD161+ 이벤트에서 이전에 게이트된 보조 파일 4.1-4.8(FCS FlowSOM 입력)에 제공된 원시 데이터를 R로 로드합니다.
fcs_raw <- read.- read.flowSet(path="C:\\\\사용자 이름\\\\\tocluster", 패턴 = ".fcs", 변환 = FALSE, truncate_max_range = FALSE, 시드=123) - 적절한 컬럼을 선택하여 관련 생물학적 마커("배경" 또는 "시간"제거)를 선택하고 아래 코드에 표시된 대로 arcsinh5 방식으로 데이터를 변환합니다(여기서, 열 1, 2, 4, 5, 6, 17, 21, 24, 25, 34, 35, 37, 38, 51, 어떤 생물학적 마커에 해당하지 않음). 아래 함수에서 보조 팩터=5를 선택하여 Cytosplore에이전에 적용한 대로 5의 보조 인자를 적용합니다.
fcs_raw <- fsApply(fcs_raw, 함수(x, 보조 팩터=5){
(x) <- fcs_raw[1]]@parameters@data$desc
expr;lt;- exprs(x)
expr<- 아신(expr[,c(1,2,4,5,6,17,21,24,25,34,35,37,38,51]//공동인자)
exprs(x) & exprs
반환(x)}) - FlowSOM 함수를 사용하여 데이터를 클러스터합니다. FlowSOM 및 Cytosplore를비교하려면 Cytosplore에서이전에 산출한 출력으로 10개의 하위 집합에서 데이터를 클러스터로 클러스터하도록 선택합니다.
fsom <- FlowSOM(fcs_raw, transformFunction = FALSE, 배율 = FALSE,
scaled.center = FALSE, scaled.scale = FALSE, 무음 = FALSE, colsToUse = c(1:37),
nClus = 10, maxMeta = 10, 중요도 = NULL, 시드 = 123)
참고: 사용자가 수동으로 변경할 수 있습니다. - 식별된 하위 집합 및 샘플 ID에 각 셀을 할당합니다.
subset_id<- as.factor(fsom$FlowSOM$맵$매핑[,1])
레벨(subset_id) & fsom$메타클러스터링
헤드(subset_id) - 그룹 할당이 포함된 R(보충 파일 5에서사용 가능)에서 메타데이터 파일을 로드하고 .fcs 파일에 연결합니다.
sampleid <- read_excel("C:\\\사용자\\\\\바탕 화면\\\\sample_id.xlsx")
샘플ID & lt;- na.omit(샘플id)
샘플리드$샘플ID &- as.factor(샘플리드$샘플ID)
샘플리드$그룹 & as.factor(샘플id$그룹)
sampleid$CSPLR_ST <- as.factor(샘플id$CSPLR_ST)
샘플ID 및 lt;- as.data.frame(샘플id)
이름(샘플ID)[3] <- "샘플ID"
sampleID <- lapply(fsom$FlowSOM$메타데이터, 함수(x){rep(x[1], 각 = 길이(x[1]:x:2]))))))))))))))
attr (샘플ID, '이름') <- NULL
샘플ID 및 lt;- as.factor(목록 해제(샘플ID) )
샘플ID 및 lt;- 데이터.프레임(샘플id)
레벨(sampleID) <- 페이스트("ID", 1:dim(샘플ID)[1], 9월="_")
df<- data.frame(subset_id, 샘플ID, fsom$FlowSOM$데이터[, c(1:37)])
이름 바꾸기 <- data.frame(콜파=fcs_raw[[1]]@parameters@data$desc)
colnames(df) <- c("clusterID", "sampleID", 이름 바꾸기$colpar[c(1:37)])
df$ 클러스터ID 및 lt;- as.factor(df$ 클러스터ID)
df$샘플ID 및 lt;- as.factor(df$샘플ID) - 다음 스크립트를 실행하여 FlowSOM에서 얻은 데이터 프레임을 기반으로 cfList를 만듭니다.
cfData <- cfList(샘플 = 샘플id,
expr = df) - 사이토스플로레 분석의 출력과 유사하게 표시되도록 마커를 다시 정렬합니다.
cfData@expr<- cfData@expr,c(1,2,34,36,37,10,23,24,31,22,
38,15,8,3,27,9,11,28,35,26,
14,33,17,20,21,18,25,29,13,
30,12,16,32,4,5,6,7,19,39)]
참고: FlowSOM에 의한 클러스터링이 완료되었습니다. 다음으로 클러스터링 출력의 시각화를 수행합니다.
- 다음 명령을 실행하여 R에 FlowSOM을 먼저 설치합니다.
3. 시각화: 후처리 클러스터링 분석
참고: 이 단계는 두 클러스터링 방법에 공통적인 메서드입니다. 따라서 FlowSOM 또는 Cytosplore로클러스터링한 후 수행할 수 있습니다.
- 히트맵을 만들기 전에 아래 코드와 같이 함수 cellCount를 사용하여 샘플당 카운트 테이블을 생성합니다. 일부 클러스터는 다른 클러스터보다 적은 셀을 포함하므로 함수 셀카운트 내부에 "scale = TRUE"를 지정하여 클러스터당 데이터를 확장하여 샘플 간의 분산을 쉽게 볼 수 있도록 합니다.
cfData <- 셀카운트(cfData, 빈도 = TRUE, 규모 = TRUE)
참고: 이제 데이터를 시각화 할 수 있습니다. - 히트맵으로 시각화합니다.
참고:이 패키지의 주요 기능 중 하나는 cytoHeatmaps, 생성된 클러스터의 표현형뿐만 아니라 샘플에 대한 이질성을 시각화하는 데 사용됩니다.
cytoHeatmaps (cfData, 그룹="그룹", 범례=TRUE) - 박스플롯이 있는 시각화
참고: 데이터는 함수 cytoBoxplots를 호출하여 정량적 인 방법으로 표현 될 수있다. 이 함수의 출력은 모든 클러스터에 대한 각 샘플의 비율을 나타냅니다.- 3.1단계에서 수행된 셀 수를 생성하지만 각 클러스터의 빈도를 얻기 위해 데이터를 확장하지는 않습니다.
cfData <- 셀카운트(cfData, 빈도 = TRUE, 배율 = FALSE)
사이토박스플롯(cfData, 그룹 = "그룹")
- 3.1단계에서 수행된 셀 수를 생성하지만 각 클러스터의 빈도를 얻기 위해 데이터를 확장하지는 않습니다.
- 중앙강도 신호 히스토그램으로 시각화
참고: 데이터는 중앙값 강도 신호 히스토그램을 표시하여 시각화 할 수 있습니다.- CD45, CD11c 및 CD54의 세 마커의 발현 강도를 시각화합니다.
- 다음 줄을 호출하여 원하는 마커의 이름을 확인합니다. 마커 이름을 기록하고 msiPlot 함수에 포함합니다.
이름(cfData@expr)
참고: 여기, CD45, CD11c 및 CD54에 초점을 맞춥니다. 마커의 정확한 철자를 확인하고 필요한 경우 조정합니다.
msiPlot (cfData, 마커 = c ("89Y_CD45", "167Er_CD11c", "164Dy_CD54"), byGroup='그룹')
결과
Cytofast 워크플로스트(그림1)는분석 소프트웨어(즉, FlowSOM 또는 Cytosplore)에의해 원래 클러스터된 데이터의 정량적 및 정성적 개요를 제공하기 위한 것입니다. Cytofast는 분석에서 확인된 모든 클러스터의 히트맵을 포함하여 마커 식에 따라 여러 가지 가능한출력을 실행합니다(그림 2 및 그림 3). 맨 위에 있는 덴드로그램은 식별된 클러스터 간의 계층적 유사성을 나타냅니다. 위쪽 패널에는 각 샘플에서 해당 하위 집합의 상대수량을 표시하는 다른 히트맵이 표시됩니다. 오른쪽의 덴드로그램은 샘플 간의 유사성을 나타내며 샘플 간의 유클리드 거리에서 수행된 계층적 군집성을 기반으로 합니다. 결합된 히트맵은 FlowSOM에 대해 표시되고 그림 2의 Cytofast 다음에는 Cytosplore가, 그림 3에서는 Cytofast가 표시됩니다. Cytofast는 그림 4 및 그림 5와 같이 데이터를 정량적으로 표시하고 결과를 박스플롯(cytoBoxplots 함수 사용)에 표시하는 데 사용할 수도 있습니다.
유사한 클러스터는 두 가지 다른 방법(예를 들어, Cytosplore로부터의 클러스터 8이 FlowSOM으로부터의클러스터 10에 해당함) 사이에 발견되었으며, PD-1 및 LAG-3와 같은 일부 억제 마커의 공동 발현은 두 방법 모두에서 여전히 볼 수 있었다. 두 클러스터링 방법 모두 PD-L1 과. 간의 차별을 허용했습니다. PBS 처리 마우스. 반대로 두 방법 간의 몇 가지 차이점을 강조 표시할 수 있습니다. FlowSOM은 2개의 클러스터(MHC-II+)를 식별하는 반면 Cytosplore는 하나의 클러스터(MHC-II+dim)만표시합니다. 이것은 NK 세포가 CD161+ 세포에 수동으로 게이트된 다음 FlowSOM에의해 추가로 처리된 초기 게이팅 전략 때문입니다. 그러나 Cytosplore는 첫 번째 HSNE 수준에서 CD45+ 모집단에서 자동으로 게이트된 셀을 다음 더 높은 계층 수준에서 클러스터화했습니다. 따라서 Cytosplore는 수동 게이팅이 CD161에 초점을 맞춘 방법보다 NK 세포 서브세트를 더 정확하게 정의했습니다. 그럼에도 불구하고, 샘플의 계층적 클러스터링은 오른쪽의 덴드로그램에 도시된 바와 같이 보존되었고, 이는 두 그룹(PD-L1 및 PBS) 간의 분리가 선택한 클러스터링 방법에 의존하지 않음을 나타낸다.
두 방법을 모두 사용하여 클러스터 수를 수동으로 정의할 수 있습니다. Cytofast를 사용하면 데이터의 이질성을 평가하고 데이터를 분할해야 하는 클러스터 수를 선택하는 방법에 대한 통찰력을 제공할 수 있습니다. msiPlot 함수(단계 3.4.2)와 같은 Cytofast 패키지에 포함된 다른 특징은 그룹당 모든 마커의 중앙값 신호 강도(MSI) 플롯을 도시합니다(도6 및 도 7). 이 함수는 PD-L1 처리군의 NK 세포에서 CD54 또는 CD11c의 발현증가와 같은 글로벌 변화를 검출할 수 있게 한다. 선택적 기능은 막대 그래프 및 기타 데이터 표현 방법에 데이터를 표시하는 것과 같이 Cytofast 패키지에 통합될 수 있습니다. 후자는 R에 의해 생성 될 수있는 ggplot 도구를 추가해야합니다.
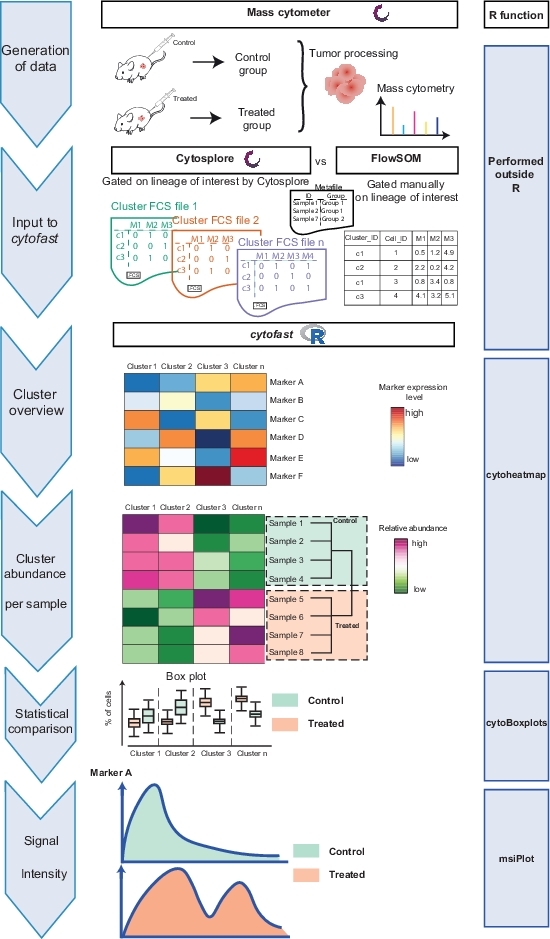
그림 1: 사이토패스트 패키지의 워크플로우. 데이터는 면역 요법으로 치료 한 후 3 일 종양으로부터 질량 세포 측정에 의해 생성되거나 치료되지 않은 상태로 방치되었다. 두 개의 서로 다른 군집 기술을 비교했다: 사이토스플로레와 FlowSOM. Cytofast는 두 기술 사이의 차이를 시각화하는 데 사용되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
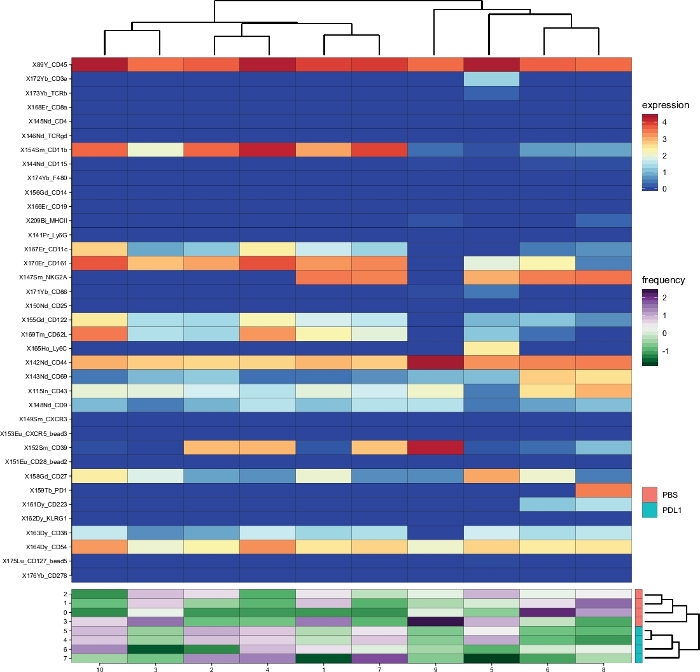
그림 2: Cytosplore다음 Cytofast에 의해 분석된 그룹당 클러스터 개요 및 클러스터 풍부도. 모든 NK 세포 클러스터의 히트맵(CD161+ Cytosplore에의해 자동으로 정의된 세포)은 면역요법 후 3일 후에 확인되었다(PD-L1). 도시된 데이터는 Cytosplore 클러스터링을 기반으로 하며 처리되지 않은 및 PD-L1 처리군으로부터 풀링됩니다. ArcSinh5 변환식 마커의 수준이 무지개 눈금으로 표시됩니다. 하부 패널에서 각 샘플의 상대적 풍부도는 녹색에서 보라색으로 표시됩니다. 오른쪽의 덴드로그램은 서브셋 주파수를 기반으로 한 샘플 간의 유사성을 나타냅니다. 주파수 눈금은 평균의 분산을 나타낸다. 낮거나 높은 주파수는 각각 녹색 또는 보라색으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
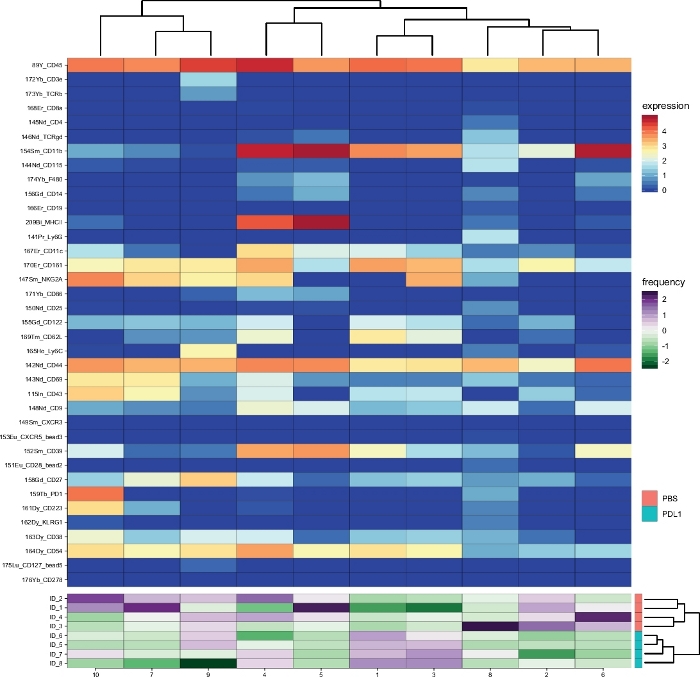
그림 3: FlowSOM다음에 Cytofast에 의해 분석된 그룹당 클러스터 개요 및 클러스터 풍부도. 면역 요법 (PD-L1) 후 3 일 확인 된 모든 NK 세포 클러스터 (CD161+ 이벤트에 사전 게이트)의 히트맵. 표시된 데이터는 FlowSOM 클러스터링을 기반으로 하며 처리되지 않은 및 PD-L1 처리 그룹에서 풀링됩니다. ArcSinh5 변환식 마커의 수준이 무지개 눈금으로 표시됩니다. 하부 패널에서 각 샘플의 상대적 풍부도는 녹색에서 보라색으로 표시됩니다. 오른쪽의 덴드로그램은 서브셋 주파수를 기반으로 한 샘플 간의 유사성을 나타냅니다. 주파수 눈금은 평균의 분산을 나타낸다. 낮거나 높은 주파수는 각각 녹색 또는 보라색으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
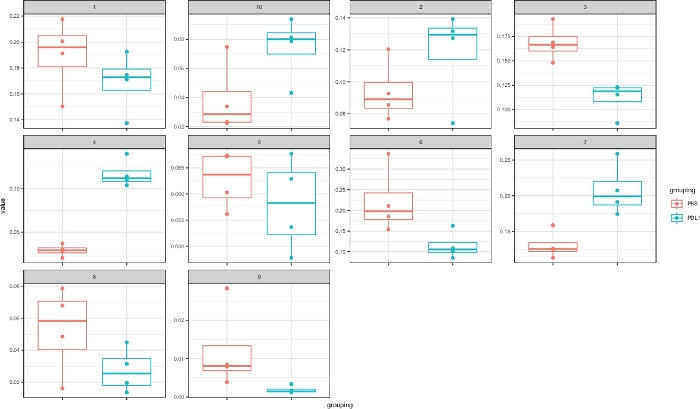
그림 4: Cytosplore에의해 정의된 클러스터의 박스플롯을 사용하여 Cytofast 표현. 각 클러스터의 빈도는 두 그룹(PBS 및 PD-L1)으로 구분된 박스플롯으로 표시됩니다. 하나의 개별 점은 하나의 마우스에 해당합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
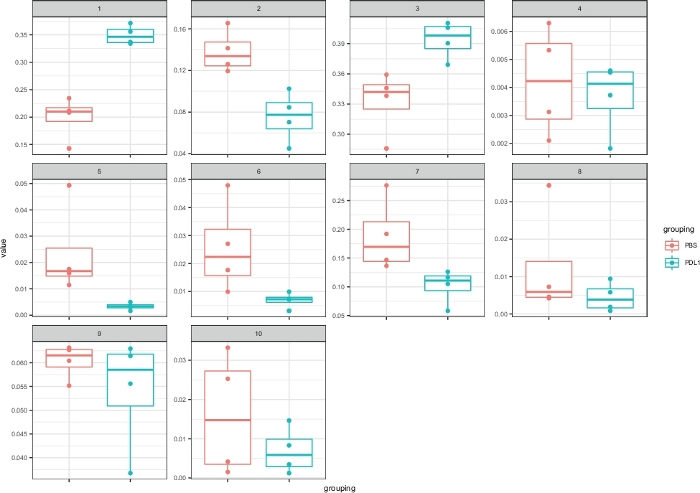
그림 5: FlowSOM에의해 정의된 클러스터의 박스플롯을 사용하여 Cytofast 표현. 각 클러스터의 빈도는 두 그룹(PBS 및 PD-L1)으로 구분된 박스플롯으로 표시됩니다. 하나의 개별 점은 하나의 마우스에 해당합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
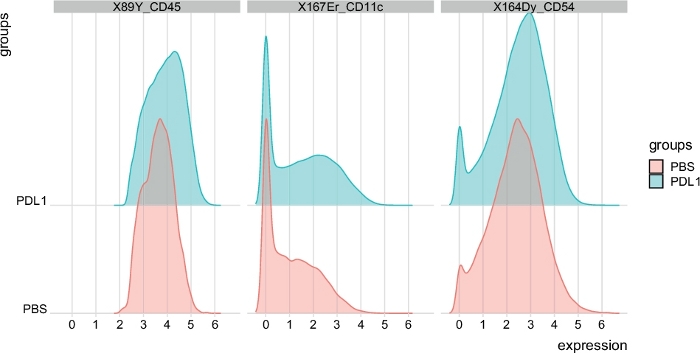
그림 6: Cytosplore에의해 자동으로 게이트된 NK 셀의 신호 강도 플롯 분포. 신호 강도의 분포는 CD45, CD11c 및 CD54의 세 가지 특정 마커에 대한 히스토그램에 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
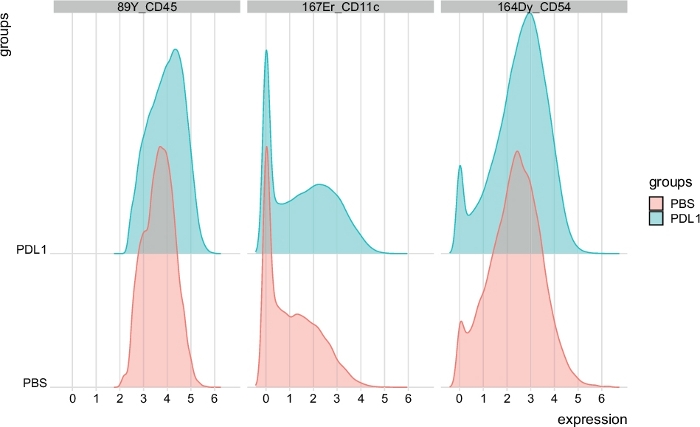
그림 7: FlowSOM에의해 자동으로 게이트된 NK 셀의 신호 강도 플롯 분포. 신호 강도의 분포는 그룹 PBS 및 PD-L1에 의해 분리된 CD45, CD11c 및 CD54의 세 가지 특정 마커에 대한 히스토그램에 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보조 파일 1.1-1.8. 이 파일을 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).
보조 파일 2.1-2.10. 이 파일을 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).
보충 파일 3. 이 파일을 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).
보조 파일 4.1-4.8. 이 파일을 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).
토론
Cytofast는 치료 별 세포 하위 집합을 강조하고 정량화하여 세포 측정 데이터의 신속하고 글로벌 탐색을 제공하는 빠른 계산 도구입니다. 설명된 프로토콜은 Cytosplore 또는 FlowSOM을사용하여 클러스터링 분석을 추가로 처리하는 것을 목표로 합니다. 다른 군집 분석 도구는 Cytofast에적합하지만 각 셀을 하위 집합에 할당하려면 Cytofast를 사용해야 합니다. 그러나 Cytofast는클러스터링 방법이 아니므로 사용하기 전에 클러스터링 프로시저가 필요합니다.
여기에서 수행된 분석은 종양 미세 환경에서 특정 CD161+ NK 세포 서브세트가 PD-L1 봉쇄에 민감하다는 것을 보여주었다. 이것은 그들의 표현형및 풍부에 있는 변경에 의해 입증되었습니다, 이는 사이토스플로레와 FlowSOM을 클러스터링 방법으로 둘 다 사용하여 관찰되었습니다. 두 방법 모두 주 NK 세포 클러스터(CD11b+ NKG2A+)를약간 다른 주파수(사이토스플로레의경우 15%-20%, FlowSOM의경우 30%-40%)로 구별했습니다. 도 2 와 도 3의 오른쪽 패널에 표시된 덴드로그램이 모두 유사한 결과를 보였기 때문에 풍부함과 이러한 근사치의 차이는 전역 패턴에 영향을 미치지 않았다. Cytofast를사용하여, NK 세포 클러스터 표현형 및 풍부의 분석에 기초하여 PD-L1 처리 및 처리되지 않은 마우스를 분리하는 것이 가능하다(선택된 군집 방법으로부터 독립적).
기록된 매개 변수에 따라 프로토콜을 수정해야 합니다. 특히 클러스터링 분석을 수행하는 동안 시간 및 배경과 같은 특정 매개 변수를 제거해야 합니다. 또한 각 셀이 하위 집합에 할당되는 것이 중요합니다. cfData 함수는 샘플당 클러스터당 원시 셀 수를 cfList에 추가하기만 하면 됩니다. 이 단계에서, cytoheatmap 섹션 3에 설명 된 대로 구축 될 수 있다.
Cytofast는 성공적으로 다른 클러스터링 방법13을비교하는 시각화 및 정량화 도구로 사용되었습니다. 이 R 패키지는 임상 변수를 사용하여 클러스터 그룹 간의 연관성을 테스트할 수 있는 globaltest14와같은 고급 기능과도 호환됩니다. 앞으로 글로벌 테스트 도구 및 기타 알고리즘을 Cytofast와 통합하여 보다 심층적인 시각화 및 정량화를 할 수 있습니다.
공개
저자는 공개 할 것이 없다.
감사의 말
우리는 제안 번호 675743 (ISPIC)에 따라 H2020 MSCA 수상의 유럽 위원회에서 자금을 인정합니다. 우리는 프로토콜을 테스트 테체 반 데르 슬루이스와 아이리스 Pardieck 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| Computer | Dell | NA | NA |
참고문헌
- Anchang, B., et al. Visualization and cellular hierarchy inference of single-cell data using SPADE. Nature Protocols. 11 (7), 1264-1279 (2016).
- Zunder, E. R., Lujan, E., Goltsev, Y., Wernig, M., Nolan, G. P. A continuous molecular roadmap to iPSC reprogramming through progression analysis of single-cell mass cytometry. Cell Stem Cell. 16 (3), 323-337 (2015).
- Van Gassen, S., et al. FlowSOM: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry A. 87 (7), 636-645 (2015).
- Levine, J. H., et al. Data-Driven Phenotypic Dissection of AML Reveals Progenitor-like Cells that Correlate with Prognosis. Cell. 162 (1), 184-197 (2015).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nature Methods. 13 (6), 493-496 (2016).
- Spitzer, M. H., et al. An interactive reference framework for modeling a dynamic immune system. Science. 349 (6244), 1259425 (2015).
- Hotelling, H. Analysis of a complex of statistical variables into principal components. Journal of Educational Psychology. 24 (6), 417-441 (1933).
- van der Maaten, L., Hinton, G. Visualizing Data using t-SNE. Journal of Machine Learning Research. , (2008).
- Pezzotti, N., Hollt, T., Lelieveldt, B., Eisemann, E., Vilanova, A. Hierarchical Stochastic Neighbor Embedding. Computer Graphics Forum. 35 (3), 21-30 (2016).
- Becht, E., et al. Dimensionality reduction for visualizing single-cell data using UMAP. Nature Biotechnology. 37, 38 (2018).
- Haghverdi, L., Buettner, F., Theis, F. J. Diffusion maps for high-dimensional single-cell analysis of differentiation data. Bioinformatics. 31 (18), 2989-2998 (2015).
- Beyrend, G., Stam, K., Höllt, T., Ossendorp, F., Arens, R. Cytofast: A workflow for visual and quantitative analysis of flow and mass cytometry data to discover immune signatures and correlations. Computational and Structural Biotechnology Journal. 16, 435-442 (2018).
- Beyrend, G., et al. PD-L1 blockade engages tumor-infiltrating lymphocytes to co-express targetable activating and inhibitory receptors. Journal for ImmunoTherapy of Cancer. 7 (1), 217 (2019).
- Goeman, J. J., van de Geer, S. A., de Kort, F., van Houwelingen, H. C. A global test for groups of genes: testing association with a clinical outcome. Bioinformatics. 20 (1), 93-99 (2004).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유