Method Article
Visualização e quantificação de dados de citometria de alta dimensão usando Cytofast e os métodos de agrupamento upstream FlowSOM e Cytosplore
Neste Artigo
Resumo
Cytofast é uma ferramenta de visualização usada para analisar a saída do agrupamento. Cytofast pode ser usado para comparar dois métodos de agrupamento: FlowSOM e Cytosplore. Cytofast pode rapidamente gerar uma visão quantitativa e qualitativa dos dados de citometria de massa e destacar as principais diferenças entre diferentes algoritmos de agrupamento.
Resumo
A complexidade dos dados gerados pela citometria de massa exigiu novas ferramentas para visualizar rapidamente os resultados analíticos. Métodos de agrupamento como Cytosplore ou FlowSOM são usados para visualização e identificação de aglomerados celulares. Para análise a jusante, um pacote R recém-desenvolvido, Cytofast,pode gerar uma visualização rápida dos resultados de métodos de agrupamento. Cytofast leva em conta a caracterização phenotypic de aglomerados celulares, calcula a abundância de aglomerados celulares, em seguida, compara quantitativamente grupos. Este protocolo explica as aplicações de Cytofast ao uso de dados de citometria de massa com base na modulação do sistema imunológico no microambiente tumoral (ou seja, a resposta natural da célula [NK]) após o desafio do tumor seguido de imunoterapia (bloqueio PD-L1). Demonstração da utilidade de Cytofast com FlowSOM e Cytosplore é mostrada. Cytofast rapidamente gera representações visuais de agrupamentos de células imunes relacionadas ao grupo e correlações com a composição do sistema imunológico. As diferenças são observadas na análise de agrupamento, mas a separação entre grupos é visível com ambos os métodos de agrupamento. Cytofast mostra visualmente os padrões induzidos pelo tratamento PD-L1 que incluem uma maior abundância de subconjuntos de células NK ativados, expressando uma maior intensidade de marcadores de ativação (ou seja, CD54 ou CD11c).
Introdução
Citometria em massa (citometria por tempo de voo, ou CyTOF) permite a detecção de uma ampla gama de biomarcadores intracelulares ou extracelulares em milhões de células individuais. A natureza multidimensional dos dados de citometria de massa requer certas ferramentas de análise, como técnicas de agrupamento celular como SPADE1, FlowMaps2, FlowSOM3, Phenograph4, VorteX5,e mapas de andaimes6. Além disso, várias técnicas baseadas em redução da dimensionalidade foram desenvolvidas (ou seja, análise de componentes principais [PCA]7, vizinho estocástico t-distribuído incorporando [t-SNE]8, vizinho estocástico hierárquico incorporando [HSNE]9, aproximação e projeção manifold uniformes [UMAP]10, e mapas de difusão11) para melhorar a velocidade, interpretação e visualização de conjuntos de dados de alta dimensão.
A análise a jusante de dados citommétricos de fluxo e massa de alta dimensão muitas vezes carece de processos automáticos para realizar testes estatísticos na frequência de cluster e links com resultados clínicos. Anteriormente, desenvolvemos um fluxo de trabalho baseado em R conhecido como Cytofast12, que permite análises visuais e quantitativas a jusante de técnicas de agrupamento por Cytosplore ou FlowSOM.
O protocolo aqui descrito esclarece o uso de Cytofast em R e mostra como gerar mapas e gráficos de calor quantitativos e qualitativos. Além disso, facilita a determinação das conexões entre fenótipos imunes observados e desfechos clínicos. Este relatório também descreve a análise de um conjunto de dados específicos de citometria de massa usando dois procedimentos de agrupamento diferentes: FlowSOM e Cytosplore. Usando Cytofast com ambos os métodos de agrupamento, é mostrado correspondentemente que o fenótipo de ativação das células NK é influenciado pelo bloqueio do ponto de verificação imunológico PD-L1.
Protocolo
Todas as experiências com animais foram aprovadas pelo Comitê de Experimentos Animais da LUMC e foram executadas de acordo com as diretrizes de experimentação animal do LUMC em conformidade com as diretrizes dos comitês holandeses e europeus.
NOTA:Para configuração experimental, camundongos C57BL/6 foram subcutâneamente inoculados no flanco direito com o tumor de cólon murine MC38 a uma concentração de 0,3 x 106 células/200 μL de soro lógico tampão de fosfato (PBS). Após 10 dias, quando os tumores eram palpáveis, os camundongos foram tratados com anticorpos de bloqueio pd-L1 (clone MIH-5, 200 μg/mouse, injeção intra-peritoneal) ou foram tratados com maquetes. Os tumores foram ressecados 3 dias depois, após a injeção de PD-L1, processado ex vivo, e analisados pela citometria de massa CyTOF usando 38 marcadores13.
1. Equipamentos e Software para Análise de Dados
NOTA:Use um computador (Windows 7 ou mais novo) e processador I5 em 2,4 GHz ou equivalente, memória instalada RAM 6 GB, e 10 GB de espaço de disco rígido gratuito. O pacote R Cytofast usa funções existentes: principalmente flowCore, pheatmape ggplot. As linhas de comando a serem executadas em R estão incluídas no protocolo. O recurso para instruções R pode ser encontrado em https://education.rstudio.com/.
- Para instalar o pacote Cytofast, inicie R (versão "3.6") e instale a versão Bioconductor 3.9, inserindo o seguinte código:
se (!requireNamespace ("BiocManager", silenciosamente = TRUE))
install.packages ("BiocManager")
BiocManager::instalar ("cytofast") - Certifique-se de que o pacote seja carregado no ambiente desejado, executando o seguinte:
biblioteca (cytofast)
2. Criando clusters
NOTA:Para mostrar os dois métodos de agrupamento Cytosplore e FlowSOM com Cytofast,as células NK (CD161+) no microambiente tumoral 3 dias após o tratamento PD-L1 são analisados.
- Agrupamento realizado porCytosplore
- Depois de baixar e instalar cytosplore, hospedado em Cytosplore.org>, carregar os arquivos .fcs(Arquivos Suplementares 1.1-1.8 [Arquivos de entrada Cytosplore]) clicando Arquivo | Arquivo FCS Aberto (s) em Cytosplore. Adicione uma marca de exemplo única como canal clicando adicionar a marca de exemplo única como canal quando solicitado e selecione um cofator para transformação de arcosinh hiperbólico (padrão é 5).
- Selecione Run H-SNE,executar um nível de HSNE de 3, e esperar que o mapa seja gerado.
NOTA:Esta etapa pode exigir algum tempo, dependendo do número de células analisadas e do nível de HSNE escolhido. - No primeiro nível hsne, verifique as células que são positivas para CD161. Selecione o CD161+ células e zoom clique direito em seleção. No segundo nível, repita o procedimento para atingir o terceiro nível com apenas CD161+ eventos.
- Uma vez que o último mapa tSNE é gerado, salvar os clusters definidos por Cytosplore por direito clicando no mapa tSNE e escolher Save Clusters. Escolha o diretório dos arquivos de saída, conforme solicitado por Cytosplore e observe este local, porque este diretório será então usado para carregar os arquivos .fcs em R.
NOTA:O número de subconjuntos pode ser alterado manualmente alterando o valor sigma. O valor sigma é definido por padrão em 30; no entanto, o número real de subconjuntos depende da entrada. Aqui, Cytosplore detectou 10 subconjuntos diferentes, com cada arquivo representando um subconjunto. - Use um nome simples (apenas caracteres) ao renomear os arquivos de saída, o que facilitará a identificação e o manuseio adicional. Salve os arquivos de saída selecionando Save.
NOTA:Depois de salvar, uma pasta está sendo criada pela Cytosplore com os arquivos .fcs, com cada arquivo correspondente aos clusters identificados em Cytosplore. O próximo passo será carregar os arquivos em R com a ajuda de Cytofast. Aqui, os arquivos de saída gerados são fornecidos em Arquivos Suplementares 2.1-2.10 (arquivos de saída Cytosplore). - Carregue os arquivos de saída gerados pela Cytosplore em R com a função designada: readCytosploreFCS.
dirFCS <- "C:\\Users\\username\\Desktop\\tostudy"
cfData <- readCytosploreFCS (dir = dirFCS, colNames = "descrição") - Limpe os dados removendo alguns parâmetros, como "Tempo" e "Fundo". Verifique a posição da coluna relacionada aos seus parâmetros desnecessários e retire-a da matriz.
colnomes (cfData@expr)
cfData@expr <- cfData@expr[,-c(3,4,6,8:10,46:49,51:54)]
NOTA:As colunas desnecessárias podem ser vistas lendo os nomes das colunas da matriz gerada. Ao executar colnomes (cfData@expr) mais uma vez, garantir que apenas os parâmetros desejados são obtidos. - Re-encomendar os marcadores para que os marcadores de linhagem são exibidos em primeiro lugar, seguido por marcadores funcionais.
cfData@expr e cfData@expr,c(1,2,3,35,36,31,9,10,18,8,37,20,
29,40,5,30,33,11,34,14,19,
32,28,6,7,4,12,13,17,16,15,
21,22,24,25,26,27,38,39)]
NOTA:Passo 2.1.8 é opcional. - Vincule o arquivo de dados meta aos dados gerados da Cytosplore, carregando o metaarquivo de planilha contendo informações clínicas(Arquivo Suplementar 3).
biblioteca (readxl)
meta <- read_excel ("C:\\Users\\username\\desktop\\sample_id.xlsx")
cfData@samples <- data.frame (meta)
NOTA:O agrupamento que está sendo realizado por Cytosplore está agora terminado. Uma opção alternativa de agrupamento para Cytosplore é FlowSOM e é descrita na seção 2.2. Uma vez realizada uma das duas etapas de agrupamento, prossiga com a etapa de visualização (seção 3).
- Agrupamento realizado porFlowSOM
- Instale pela primeira vez o FlowSOM em R, executando o seguinte comando:
se (!requireNamespace ("BiocManager", silenciosamente = TRUE))
install.packages ("BiocManager")
BiocManager::instalar ("FlowSOM")
biblioteca (FlowSOM) - Instale o pacote flowCore usando um método semelhante e carregue-o no ambiente executando o seguinte:
se (!requireNamespace ("BiocManager", silenciosamente = TRUE))
install.packages ("BiocManager")
BiocManager::instalar ("flowCore")
biblioteca (FlowSOM) - Carregue os dados brutos fornecidos em Arquivos Suplementares 4.1-4.8 (entrada FCS FlowSOM), que foram previamente fechados em eventos CD161+, em R com a função read.flowSet.
fcs_raw <- read.flowSet (path="C:\\Users\\username\\Desktop\\tocluster", padrão = ".fcs", transformação = FALSE, truncate_max_range = FALSE, semente=123) - Selecione os marcadores biológicos relevantes (remover "Background" ou "Time") selecionando as colunas adequadas e transforme os dados de forma arcsinh5, como mostrado no código abaixo (aqui, remova as colunas 1, 2, 4, 5, 6, 17, 21, 24, 25, 34, 35, 37, 38, 51, que não correspondem a nenhum marcador biológico). Aplique um cofator de 5 como aplicado anteriormente com Cytosplore,escolhendo cofator=5 na função abaixo.
fcs_raw <- fsApply (fcs_raw, função (x, cofactor=5){
colnames (x) <- fcs_raw[[1]]] @parameters@data$desc
expr <- exprs (x)
expr <- asinh (expr[,-c(1,2,4,5,6,17,21,24,25,35,37,38,51)/cofactor)
exprs (x) <- expr
retorno (x)}) - Cluster os dados usando a função FlowSOM. Para comparar FlowSOM e Cytosplore,opte por agrupar os dados em dez subconjuntos como a saída anteriormente produzida pela Cytosplore.
fsom <- FlowSOM (fcs_raw, transformFunction = FALSE, scale = FALSE,
scaled.center = FALSE, scaled.scale = FALSE, silent = FALSE, colsToUse = c(1:37),
nClus = 10, maxMeta = 10, importância = NUL, semente = 123)
NOTA:Isso pode ser alterado manualmente pelo usuário. - Atribuir cada célula ao seu subconjunto identificado e identificação da amostra.
subset_id <- as.factor (fsom$FlowSOM$map$mapping[,1])
níveis (subset_id) <- fsom$metaclustering
cabeça (subset_id) - Carregue o arquivo de metadados em R (disponível no Arquivo Suplementar 5)contendo a atribuição do grupo e vincule-o aos arquivos .fcs.
sampleid <- read_excel ("C:\\Users\\username\\Desktop\\sample_id.xlsx")
sampleid <- na.omit (sampleid)
sampleid$sampleID <- as.factor (sampleid$sampleID)
sampleid$group <- as.factor (sampleid$group)
sampleid$CSPLR_ST <- as.factor (sampleid$CSPLR_ST)
sampleid <- as.data.frame (sampleid)
nomes (sampleid)[3] <- "sampleID"
sampleID <- lapply (fsom$FlowSOM$metaData, função (x){rep (x[1], cada = comprimento (x[1]:x[2]))})
attr (sampleID, 'names') <- NULL
sampleID <- as.factor (unlist (sampleID))
sampleid <- data.frame (sampleid)
níveis (sampleID) <- colar ("ID", 1:dim (sampleid)[1], sep="_")
df <- data.frame (subset_id, sampleID, fsom$FlowSOM$data[, c (1:37)])
renome <- data.frame (colpar=fcs_raw[[1]]] @parameters@data$desc)
colnames (df) <- c ("clusterID", "sampleID", rebatizar$colpar[c(1:37)])
df$ clusterID <- as.factor (df$ clusterID)
df$sampleID <- as.factor (df$sampleID) - Crie uma cfList com base no quadro de dados obtido da FlowSOM executando o seguinte script:
cfData <- cfList (samples = sampleid, cfData <- cfList (samples = sampleid, cfData <- cfList (samples = sampleid, cfData
expr = df) - Reordenar os marcadores para aparecer de forma semelhante à saída da análise Cytosplore.
cfData@expr e cfData@expr,c(1,2,34,36,37,10,23,24,32, 22, 22,
38,15,8,3,27,9,11,28,35,26,
14,33,17,20,21,18,25,29,13,
30,12,16,32,4,5,6,7,19,39)]
NOTA:O agrupamento por FlowSOM está agora terminado. Em seguida, realize a visualização da saída de agrupamento.
- Instale pela primeira vez o FlowSOM em R, executando o seguinte comando:
3. Visualização: Análise de Agrupamento pós-processamento
NOTA:Esta etapa é um método que seja comum a ambos os métodos de agrupamento. Portanto, ele pode ser realizado após clustering com FlowSOM ou Cytosplore.
- Antes de criar os mapas de calor, gere a tabela de contagem por amostra usando a função cellCounts como mostrado no código abaixo. Uma vez que alguns clusters contêm menos células do que outras, dimensionar os dados por cluster, especificando "escala = TRUE" dentro da função cellCounts, de modo que a dispersão entre as amostras pode ser facilmente visto.
cfData <- cellCounts (cfData, frequency = TRUE, scale = TRUE) cfData <- cellCounts (cfData, frequency = TRUE, scale = TRUE) cfData <- cellCounts (cfData, frequency = TRUE, scale = TRUE) cfData
NOTA:Os dados agora podem ser visualizados. - Visualize com mapa de calor.
NOTA:Uma das principais funções deste pacote é cytoHeatmaps, que é usado para visualizar o fenótipo dos clusters criados, bem como a sua heterogeneidade em relação às amostras.
cytoHeatmaps (cfData, group="group", legend=TRUE) - Visualização com enredos de caixa
NOTA:Os dados podem ser representados de forma quantitativa, chamando a função cytoBoxplots. A saída desta função representa a proporção de cada amostra para cada cluster.- Gere a contagem celular como feito na etapa 3.1, mas não dimensione os dados para obter a frequência de cada cluster.
cfData <- cellCounts (cfData, frequency = TRUE, scale = FALSE) cfData <- cellCounts (cfData, frequency = TRUE, scale = FALSE) cfData <- cellCounts (cfData, frequency = TRUE, scale = FALSE) cfData
cytoBoxplots (cfData, grupo = "grupo")
- Gere a contagem celular como feito na etapa 3.1, mas não dimensione os dados para obter a frequência de cada cluster.
- Visualização com intensidade mediana sinaliza histograma
NOTA:Os dados também podem ser visualizados representando o histograma de sinal de intensidade mediana.- Visualize a intensidade de expressão de três marcadores: CD45, CD11c e CD54.
- Verifique os nomes dos marcadores desejados, chamando a seguinte linha. Observe os nomes dos marcadores e inclua-o na função msiPlot.
nomes (cfData@expr)
NOTA:Aqui, concentre-se em CD45, CD11c e CD54. Verifique a ortografia exata dos marcadores e ajuste, se necessário:
msiPlot (cfData, marcadores = c("89Y_CD45", "167Er_CD11c", "164Dy_CD54"), pelo Group='group')
Resultados
O fluxo de trabalho Cytofast (Figura 1) destina-se a fornecer uma visão quantitativa e qualitativa dos dados originalmente agrupados por software de análise (ou seja, FlowSOM ou Cytosplore). Cytofast executa várias saídas possíveis, incluindo o mapa de calor de todos os clusters identificados na análise e com base na expressão marcador(Figura 2 e Figura 3). O dendrogram na parte superior representa a similaridade hierárquica entre os conjuntos identificados. O painel superior exibe outro mapa de calor mostrando a quantidade relativa de subconjuntos correspondentes em cada amostra. O dendrogram à direita mostra a semelhança entre as amostras e é baseado no agrupamento hierárquico realizado nas distâncias euclides entre as amostras. Os mapas de calor combinados são mostrados para FlowSOM seguido por Cytofast na Figura 2 e para Cytosplore seguido por Cytofast na Figura 3. Cytofast também pode ser usado para apresentar os dados quantitativamente e exibir os resultados em boxplots (usando a função cytoBoxplots), como mostrado na Figura 4 e Figura 5.
Aglomerados semelhantes foram encontrados entre os dois métodos diferentes (por exemplo, o cluster 8 de Cytosplore corresponde ao cluster 10 do FlowSOM), e a co-expressão de alguns marcadores inibitórios como PD-1 e LAG-3 ainda eram visíveis em ambos os métodos). Ambos os métodos de agrupamento permitiram a discriminação entre PD-L1 vs. Pbs tratados ratos. Em contraste, algumas diferenças entre ambos os métodos podem ser destacadas. FlowSOM identifica 2 clusters (MHC-II+), enquanto Cytosplore mostra apenas um cluster (MHC-II+dim). Isto é devido à estratégia inicial gating em que as células NK foram manualmente fechado em CD161+ células, em seguida, processado s a flowsom. No entanto, Cytosplore automaticamente fechado células do CD45+ população no primeiro nível HSNE, que foram então agrupados em um nível hierárquico superior. Assim, Cytosplore definiu os subconjuntos de células NK com mais precisão do que como o trabalho manual se concentrou em CD161. No entanto, o agrupamento hierárquico das amostras foi preservado, como mostrado no dendrogram à direita, indicando que a segregação entre os dois grupos (PD-L1 e PBS) não dependia do método de agrupamento escolhido.
O número de clusters pode ser definido manualmente usando ambos os métodos. Cytofast permite ao usuário avaliar a heterogeneidade de seus dados e pode fornecer informações sobre como escolher o número de clusters em que os dados devem ser divididos. Outras características estão incluídas no pacote Cytofast, como a função msiPlot (passo 3.4.2), mostrando o gráfico mediano de intensidade de sinal (MSI) de cada marcador por grupo (Figura 6 e Figura 7). Esta função permite a detecção de mudanças globais, como aumentos na expressão de CD54 ou CD11c em células NK do grupo tratado pelo PD-L1. Os recursos opcionais podem ser incorporados no pacote Cytofast, como a exibição de dados em gráficos de barras e outros métodos de representação de dados. Este último requer a adição de ferramentas ggplot, que podem ser geradas por R.
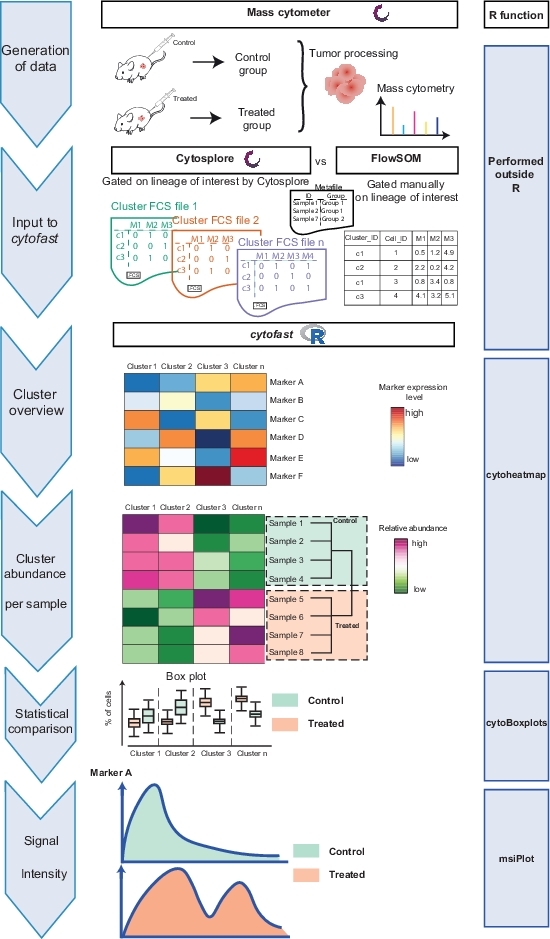
Figura 1: Fluxo de trabalho do pacote Cytofast. Os dados foram gerados pela citometria em massa de um tumor 3 dias após o tratamento com imunoterapia ou não tratada. Duas técnicas de agrupamento diferentes foram comparadas: Cytosplore e FlowSOM. Cytofast foi usado para visualizar diferenças entre as duas técnicas. Clique aqui para ver uma versão maior deste número.
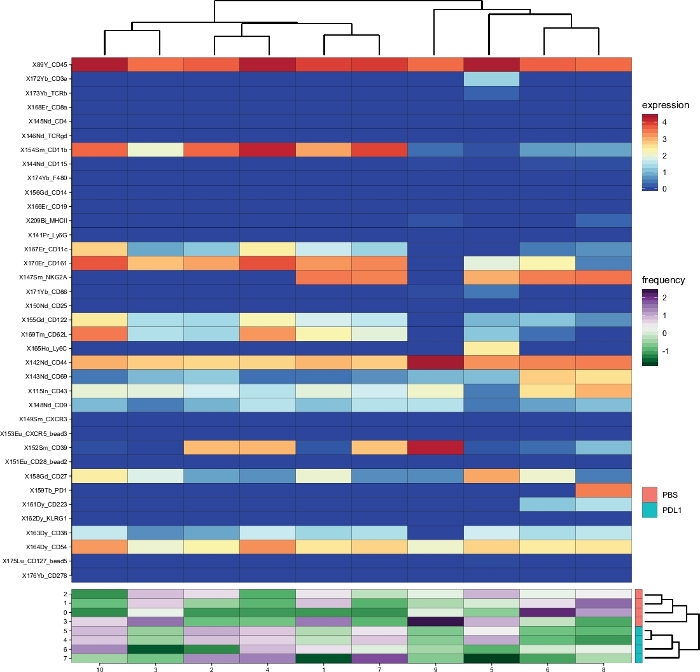
Figura 2: Visão geral de cluster e abundância de cluster por grupo, conforme analisado pela Cytofast após Cytosplore. Mapa térmico de todos os aglomerados de células NK (CD161+ células definidas automaticamente pela Cytosplore),que foram identificadas 3 dias após a imunoterapia (PD-L1). Os dados mostrados são baseados no agrupamento cytosplore e agrupados dos grupos tratados não tratados e PD-L1. Os níveis de marcador de expressão transformado em ArcSinh5 são exibidos em escala de arco-íris. No painel inferior, a abundância relativa de cada amostra é representada pela escala verde-roxa. O dendrogram à direita representa a semelhança entre amostras com base em frequências subdefinidas. A escala de frequência representa a dispersão da média. Uma baixa ou alta frequência é representada por uma cor verde ou roxa, respectivamente. Clique aqui para ver uma versão maior deste número.
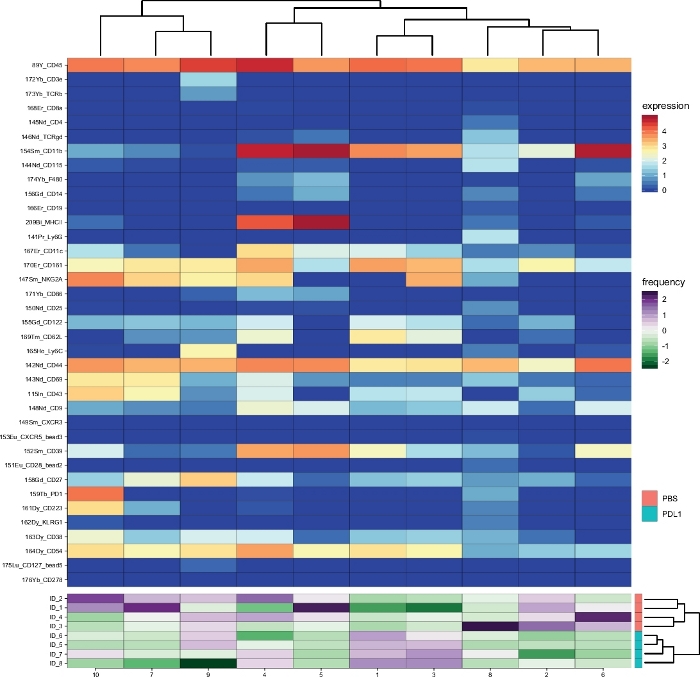
Figura 3: Visão geral de cluster e abundância de cluster por grupo, conforme analisado pela Cytofast após FlowSOM. Mapa de calor de todos os aglomerados de células NK (pré-fechados em CD161+ eventos), que foram identificados 3 dias após a imunoterapia (PD-L1). Os dados mostrados são baseados no agrupamento FlowSOM e agrupados a partir dos grupos tratados não tratados e PD-L1. Os níveis de marcador de expressão transformado em ArcSinh5 são exibidos em escala de arco-íris. No painel inferior, a abundância relativa de cada amostra é representada pela escala verde-roxa. O dendrogram à direita representa a semelhança entre amostras com base em frequências subdefinidas. A escala de frequência representa a dispersão da média. Uma baixa ou alta frequência é representada por uma cor verde ou roxa, respectivamente. Clique aqui para ver uma versão maior deste número.
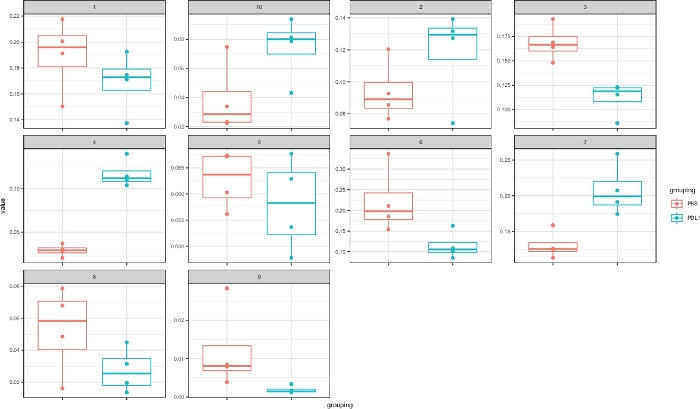
Figura 4: Representação cytofast com boxplots dos clusters definidos por Cytosplore. A frequência de cada cluster é representada em uma caixa, separada em dois grupos (PBS e PD-L1). Um pontão individual corresponde a um rato. Clique aqui para ver uma versão maior deste número.
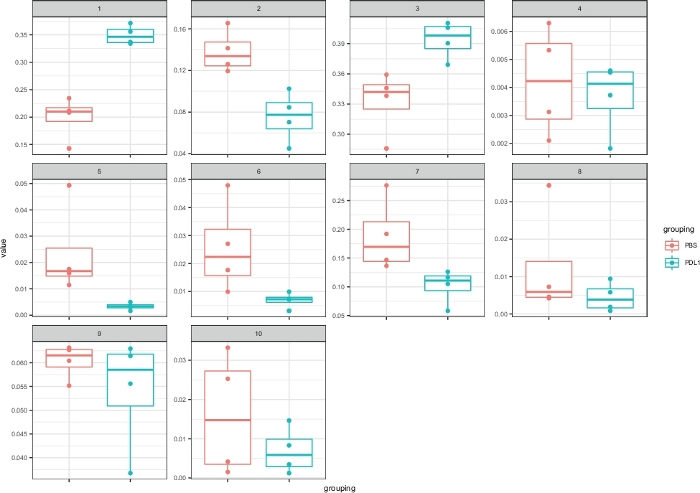
Figura 5: Representação citorápida com boxplots dos clusters definidos pela FlowSOM. A frequência de cada cluster é representada em uma caixa, separada em dois grupos (PBS e PD-L1). Um pontão individual corresponde a um rato. Clique aqui para ver uma versão maior deste número.
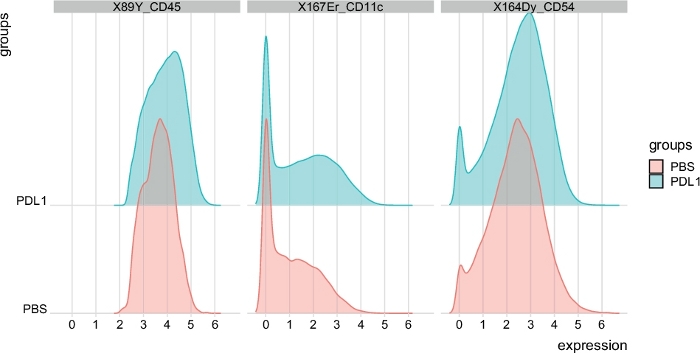
Figura 6: Distribuição de parcelas de intensidade de sinal das células NK automaticamente fechadas pela Cytosplore. A distribuição de intensidades de sinal é mostrada em um histograma para três marcadores específicos: CD45, CD11c e CD54. Clique aqui para ver uma versão maior deste número.
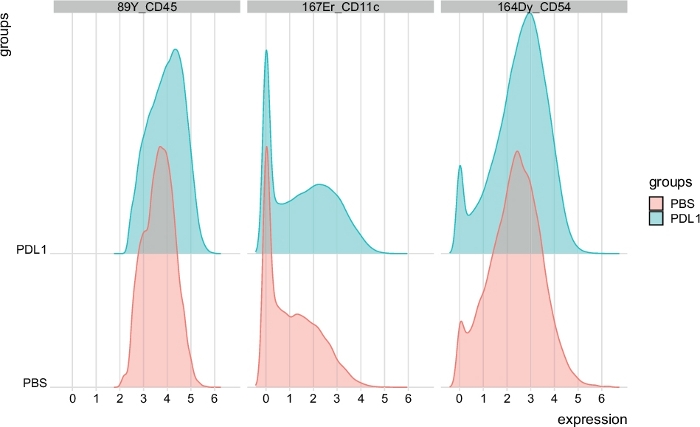
Figura 7: Distribuição de parcelas de intensidade de sinal das células NK automaticamente fechadas pela FlowSOM. A distribuição de intensidades de sinal é mostrada em um histograma para três marcadores específicos: CD45, CD11c e CD54, segregados pelos grupos PBS e PD-L1. Clique aqui para ver uma versão maior deste número.
Arquivos suplementares 1.1-1.8. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivos suplementares 2.1-2.10. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo Suplementar 3. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivos suplementares 4.1-4.8. Clique aqui para ver este arquivo (Clique certo para baixar).
Arquivo Suplementar 5. Clique aqui para ver este arquivo (Clique certo para baixar).
Discussão
Cytofast é uma ferramenta computacional rápida que fornece uma exploração rápida e global de dados citommétricos, destacando e quantificando subconjuntos celulares específicos do tratamento. O protocolo descrito visa novas análises de agrupamento de processos com Cytosplore ou FlowSOM. Outras ferramentas de análise de agrupamento são adequadas para Cytofast,mas isso requer o uso de Cytofast para atribuir cada célula a um subconjunto. Cytofast,no entanto, não é um método de agrupamento e, portanto, requer procedimentos de agrupamento antes do uso.
A análise realizada aqui mostrou que certos subconjuntos celulares CD161+ NK no microambiente tumoral eram sensíveis a um bloqueio PD-L1. Isso foi evidenciado por mudanças em seu fenótipo e abundância, que foram observadas usando Cytosplore e FlowSOM como métodos de agrupamento. Ambos os métodos distinguiram o principal cluster de células NK (CD11b+ NKG2A+) com frequências ligeiramente diferentes (15%-20% para Cytosplore, 30%-40% para FlowSOM). As diferenças de abundância e essa aproximação não afetaram o padrão global, pois ambos os dendrogramas exibidos nos painéis direitos da Figura 2 e da Figura 3 mostraram resultados semelhantes. Usando Cytofast,é assim possível (independente do método de agrupamento escolhido) segregar ratos pd-L1-tratados e não tratados baseados em análises do fenótipo e da abundância do conjunto da pilha de NK.
Dependendo dos parâmetros gravados, são necessárias modificações no protocolo. Especificamente, certos parâmetros, como tempo e fundo, devem ser removidos durante a realização da análise de agrupamento. Além disso, é importante que cada célula seja atribuída a um subconjunto. A função cfData simplesmente adicionará as contagens de células brutas por cluster por amostra no cfList. A partir desta etapa, o citoheatmap pode ser construído como explicado na seção 3.
Cytofast tem sido usado com sucesso como uma ferramenta de visualização e quantificação para comparar diferentes métodos de agrupamento13. Este pacote R também é compatível com características avançadas, como o globaltest14,que pode testar associações entre grupos de clusters usando variáveis clínicas. No futuro, a ferramenta globaltest e outros algoritmos podem ser integrados com Cytofast para visualização e quantificação mais aprofundadas.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Reconhecemos o financiamento da Comissão Europeia de um prémio H2020 MSCA ao abrigo da proposta número 675743 (ISPIC). Agradecemos a Tetje van der Sluis e Iris Pardieck por testarem o protocolo.
Materiais
| Name | Company | Catalog Number | Comments |
| Computer | Dell | NA | NA |
Referências
- Anchang, B., et al. Visualization and cellular hierarchy inference of single-cell data using SPADE. Nature Protocols. 11 (7), 1264-1279 (2016).
- Zunder, E. R., Lujan, E., Goltsev, Y., Wernig, M., Nolan, G. P. A continuous molecular roadmap to iPSC reprogramming through progression analysis of single-cell mass cytometry. Cell Stem Cell. 16 (3), 323-337 (2015).
- Van Gassen, S., et al. FlowSOM: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry A. 87 (7), 636-645 (2015).
- Levine, J. H., et al. Data-Driven Phenotypic Dissection of AML Reveals Progenitor-like Cells that Correlate with Prognosis. Cell. 162 (1), 184-197 (2015).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nature Methods. 13 (6), 493-496 (2016).
- Spitzer, M. H., et al. An interactive reference framework for modeling a dynamic immune system. Science. 349 (6244), 1259425 (2015).
- Hotelling, H. Analysis of a complex of statistical variables into principal components. Journal of Educational Psychology. 24 (6), 417-441 (1933).
- van der Maaten, L., Hinton, G. Visualizing Data using t-SNE. Journal of Machine Learning Research. , (2008).
- Pezzotti, N., Hollt, T., Lelieveldt, B., Eisemann, E., Vilanova, A. Hierarchical Stochastic Neighbor Embedding. Computer Graphics Forum. 35 (3), 21-30 (2016).
- Becht, E., et al. Dimensionality reduction for visualizing single-cell data using UMAP. Nature Biotechnology. 37, 38 (2018).
- Haghverdi, L., Buettner, F., Theis, F. J. Diffusion maps for high-dimensional single-cell analysis of differentiation data. Bioinformatics. 31 (18), 2989-2998 (2015).
- Beyrend, G., Stam, K., Höllt, T., Ossendorp, F., Arens, R. Cytofast: A workflow for visual and quantitative analysis of flow and mass cytometry data to discover immune signatures and correlations. Computational and Structural Biotechnology Journal. 16, 435-442 (2018).
- Beyrend, G., et al. PD-L1 blockade engages tumor-infiltrating lymphocytes to co-express targetable activating and inhibitory receptors. Journal for ImmunoTherapy of Cancer. 7 (1), 217 (2019).
- Goeman, J. J., van de Geer, S. A., de Kort, F., van Houwelingen, H. C. A global test for groups of genes: testing association with a clinical outcome. Bioinformatics. 20 (1), 93-99 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados