Method Article
液滴ベースのRNAシーケンシングによるハイスループット酵母株フェノタイピング
要約
微生物工学の「設計構築テスト」サイクルのボトルネックは、株の機能画面を実行できる速度です。液滴ベースのRNAシーケンシングを利用した実験ごとに数百~数千個の酵母細胞に適用される菌株スクリーニングのハイスループット法について述べています。
要約
酵母ゲノムを編集できる強力なツールにより、この微生物はエンジニアリングにとって貴重なプラットフォームとなっています。遺伝的に異なる株の何百万ものライブラリを構築することが可能になりましたが、所望の表現型のスクリーニングは依然として重要な障害です。既存のスクリーニング手法では、情報出力とスループットの間にトレードオフがあり、通常、ハイスループットスクリーニングは対象となる1つの製品で実行されます。そこで、遺伝子組み換え酵母株のアイソジェニックピコリットルコロニーに単一細胞RNAシーケンシングを適応させることにより、菌株スクリーニングを促進するアプローチを提示する。酵母細胞にRNAシーケンシングを行う独自の課題に対処するために、RNAシーケンシングを行う前に、ヒドロゲルおよびスフェロプラスト内でイソジェニック酵母コロニーを培養します。RNAシーケンシングデータを使用して、酵母の遺伝子型を推測し、操作された経路を選別することができます。当社の方法のスケーラビリティは、微生物工学における重大な閉塞に対処します。
概要
微生物工学の第一の目標は、微生物を改変して、貴重な化合物を1,22に産生するように誘導することです。S. cerevisiaeは,、培養の容易さと、そのゲノム3、4、54をエンジニアリングするために利用可能なツールの広さのために、3微生物工学の主要な生物となっています。5しかし、改変酵母で機能スクリーンを実行する上でハードルが残っている:スクリーニングスループットはゲノム工学に桁違いの遅れがある。スクリーニングは、典型的には、マイクロウェルプレート内の株を分離し、特定の化合物66,77の生産を測定することによってそれらを定型化することを含む。このプロセスのスループットは、100マイクロリットル反応で個々の株をアッセイするために必要な大量の試薬によって制限されます。液滴マイクロ流体は、通常ウェルプレート8で行われるダウンスケーリング反応によって、酵母スクリーニングのスループットを桁違いに向上させる魅力的な溶液を提供する。しかし、プレートスクリーンの井戸と同様に、液滴スクリーンは通常、単一の製品化合物を検出し、これは、設計された経路99、10、1110,11のグローバル機能に限られた情報を提供する。
RNAシーケンシング(RNA-seq)は、関連するすべての遺伝子の発現レベルを同時に12,13に評価できるようにすることで、13経路操作のより包括的な特徴付けが可能であり得る。さらに、液滴法は、実験ごとに何千もの細胞をプロファイリングすることを可能にし、工学的なバリアント14、15,15のライブラリをスクリーニングするために必要なスループットを提供する。しかし、RNA-seq法は哺乳動物細胞に最適化されています。酵母は、比較して、細胞当たりのmRNAが少なく、細胞壁は16を除去することが困難であり、既存の方法によるシーケンシングを排除する。酵母RNA-seqを可能にするために高スループットの液滴法を考案することができれば、酵母工学のためのスケーラブルで費用対効果の高い、情報豊富なフェノタイピングプラットフォームを提供します。
我々は、高スループットの液滴マイクロ流体17を用いて酵母細胞をシーケンシングするための我々の最近開発された方法の詳細なプロトコルを提示する。限られたRNAの課題を克服するために、ピコリットル水ゲル球に単一酵母細胞を封入し、培養します。培養は細胞を複製し、同じ設計された経路を共有する何百ものコピーを生成する。これは、単一細胞遺伝子発現による変動を減少させ、シーケンシングに利用できるRNAの量を大幅に増加させる。培養ベースの増幅後、細胞を球状にし、バルク酵素消化を介して細胞壁を除去する。細胞膜はそのまま残るので、各等元性コロニーおよび関連するmRNAは、そのヒドロゲル球に封入されたままである。これにより、個々のコロニーをmRNA捕捉試薬およびリシスバッファーと組み合わせることができ、また、MRNAを捕捉、バーコード化、および配列決定するDrop-Seqワークフロー14に従って行うことができます。我々の方法は、実験ごとに何千もの等原性酵母コロニーのトランスクリプトーム全体のスクリーニングを可能にする。
プロトコル
1. マイクロ流体デバイスの製造
- SU-8マスターファブリケーション
- コンピュータ支援設計ソフトウェアを使用してデバイスAおよびB(補足ファイル1および2)のマイクロ流体チャネル用の負のマスクを設計し、少なくとも10μmの解像度で回路基板フィルムに印刷します。
- スピンコーターに清潔な75mmシリコンウエハを置き、約1mLのSU-8を中央に注ぎます。真空をオンにして、ウエハをチャックに固定します。
- デバイスAの場合、スピンコートSU-8 2150は30sの500rpmで、次いで2,750 rpmで30 sが続きます。デバイスBの場合、スピンコートSU-8 2100は30sの500rpmで、次いで2,500rpmで30sが続く。これにより、それぞれ200 μmと120 μmの厚さのSU-8層が得られます。
- スピンコーターからウエハーを取り出し、ホットプレートの上に95°Cで60分間柔らかいベークします。
- ホットプレートからウエハーを取り出し、室温まで冷まします。マスクをウエハーの上に置き、コリメータ190mW、365 nmのUV LEDを2分間照射します。
- ウエハーは、露出後のベーキングのために5分間、95 °Cに設定されたホットプレートに置きます。
- ウエハーを取り出し、室温まで冷まします。ウエハーをプロピレングリコールモノメチルエーテルアセテート(PGMEA)の浴槽に20分間置きます。
- ウエハをPGMEAでリンスし、続いてイソプロパノールを使用します。このプロセス中に不透明な残留物が見える場合は、PGMEAおよびイソプロパノールでリンスを繰り返します。空気はウエハーを乾燥させる。
- ウエハーをホットプレートの上に95°Cで3分間置きます。
- ウェハーを取り出し、直径90mmのペトリ皿に入れます。
- SU-8マスターにポリジメチルシロキサン(PDMS)鋳造
- シリコーン基剤と硬化剤の10:1質量比を混ぜ合わせます。約30分間混合した後、PDMSを脱ガスする。
- 少なくとも5mmの厚さの層がウエハーの上に形成されるまで、SU-8マスターの上に脱気PDMSを注ぎます。
- 約30分間、ウエハーの上にPDMSをドガ。
- ウエハーを65°Cのオーブンに少なくとも80分間置き、PDMSを硬化させる。
- 硬化したPDMSスラブをウェハから切り取ります。
- マイクロ流体機能を上向きにしたPDMSスラブを配置し、0.75 mmのバイオプシーパンチで入口と出口穴をパンチします。
- イソプロパノールで50 mm x 75 mmガラススライドをクリーニングし、テープでPDMSスラブのマイクロ流体機能側からすべてのほこりを取り除きます。
- 洗浄されたガラススライドとPDMSスラブをマイクロ流体機能で100 Pa(1 mbar O2)プラズマに1分間露出させます。
- フィーチャーを使用する PDMS スラブをガラススライドの上に下に置き、ボンディングを可能にします。スライドを65°Cのオーブンに入れ、少なくとも30分間ボンディングを完了します。
- フッ素表面処理液で洗い流すことで、マイクロ流体チャネルをすべて処理します。65°Cのオーブンで少なくとも10分間焼き、液体を蒸発させます。
2. デバイスAを用いたヒドロゲル中の酵母カプセル化
- 懸濁液培養で成長している酵母を取り、ヘモサイトメーターを頼りにしてください。
- リン酸緩衝生理食塩分(PBS)の細胞を約750k/mLの濃度に再懸濁する。これにより、ヒドロゲルの30%が1つの酵母細胞を含みます。酵母細胞の約半分だけがコロニーに成長し、酵母コロニーを含むヒドロゲルの約15%につながる。
- PBSで2%w/vで超低融点アガロースを混合し、溶融するまで90°Cで加熱します。所要時間は約10分です。
- アガロースミックスを0.22 μmフィルターを取り付けたシリンジに、80°Cに設定されたスペースヒーターの前にあるシリンジポンプにセットします。
- 酵母懸濁液を充填したシリンジと、2%w/vイオン性フルオロサーファクタクタ18のフッ素化油を含むシリンジをシリンジポンプにロードします。
- セクション 1 で作成したコフロー ドロップ スプリッター デバイスを取り出し、スポイトからデバイスにチューブを接続します。滴収集のための氷のバケツの15 mL円錐形の管に出口から管を導く。
- 次の流量でデバイスに 3 つのソリューションをフローします。
- 3 mL/hの流量で酵母懸濁液を流します。
- アガロース混合物を流量3 mL/hで流す。
- フッ素化油を流量15mL/hで流します。
- 約1mLのエマルジョンを集める。アガロースが完全に設定できるように、さらに5分待ちます。
3. 培養用のゲルの割れと洗浄
- フッ素化油中の20%パーフルオロオクタンノール(PFO)をエマルションに等量添加する。円錐管を数回反転させて混合を可能にします。
- 2,000 x gで壊れたエマルジョンを 2 分間回転させます。ゲルは、油相およびPFO相の上にペレットになります。
- 油相を取り除き、2 mLのTE-TWバッファー(10 mM Tris pH = 8.0,1 mM EDTA,0.01%Tween-20)を加え、ゲルを再懸濁させます。サスペンションを新しい15 mL円錐形チューブに移します。
- ステップ3.2のようにゲルをペレットダウンし、TE-TWでさらに1回洗浄し、合計2回洗浄します。
- 上清を取り除き、2 mLの培地でゲルを再懸濁します。5 mL培養管に移す。
- 振盪下で一晩30°Cでインキュベートする。
注:一晩のインキュベーションの後、酵母ヒドロゲルは、数日間4°Cで保つことができます。
4. 酵母コロニーの起下
- 2,000 x gで 15 mL 円錐形チューブとペレットヒドロゲルにゲルを 2 分間移動します。
- ヒドロゲルをPBS 2xで洗浄します。
- 1xスフェロプチャーリング緩衝液1xで洗浄します。
- 球状緩衝液中の球状酵素の2~50倍の希釈を行い、ヒドロゲルに1 mLを加えます。
- 37°Cで1時間インキュベートする。処理された酵母はより透明に見えます(図3A)。
- ハイドロゲル懸濁液の底部0.8 mLを取り、1 mLの上限のないシリンジに移します。
- 3Dプリントされたシリンジホルダー(補足ファイル3)にシリンジを入れ、2分間2,000 x gで回転します。これにより、ヒドロゲルがシリンジヘッドのパックを閉じます。
5. デバイスBを用いた酵母コロニーからのmRNA捕捉
- 240,000個のDrop-Seqビーズを取り、15 mLの円錐形チューブに移します。
- ペレットドロップセクビーズは、1,000 x gで1分間回転します。
- 上清を取り除き、ビーズを120,000ビーズ/mLの濃度にする500 mMの塩化ナトリウムを含む0.9x酵母のリシスバッファーの2 mLでビーズを再懸濁します。
- ビーズの懸濁液を、攪拌棒を挿入して3 mLのシリンジに移します。
- フッ素油中の2%w/vパーフルオロポリエーテルポリエチレングリコール(PFPE-PEG)界面活性剤を数ミリリットル含むシリンジを調製します。
- 密に詰めたヒドロゲルを含む注射器のすべての水性ヘッドを避難させ、注射器をキャップします。
- ヒドロゲル、ビーズ懸濁液、オイルシリンジをシリンジポンプに挿入し、チューブを介してセクション1で作られたカプセル化装置に接続します。
- 出口チューブから氷上の50 mL円錐形チューブに接続します。
- 次の流量でデバイスに 3 つのソリューションをフローします。
- ヒドロゲルを0.4 mL/hで流します。
- ビードサスペンションを0.4 mL/hで流します。
- フッ素化した油を1.6 mL/hで流します。
- エマルジョンの〜1 mLを収集するか、残ったヒドロゲルがなくなるまでデバイスを実行します。
6. cDNA生成、シーケンスライブラリの準備、およびシーケンス
- 収集されたエマルジョンに、6x SSCバッファの30 mLと1 mLのPFOを追加します。
- 引き続き Drop-Seq プロトコルに従って、ビーズでキャプチャされた mRNA から cDNA を生成し、ライブラリの準備をシーケンシングし、シーケンシング データ分析を行います。
結果
先に発表した Drop-Seq ワークフロー14をイソジェニックコロニーシーケンシング(ICO-seq)に適応させて、イソジェニック酵母コロニーの遺伝子発現プロファイリングを行った。我々は単一酵母細胞を単離し、アガロースミクロゲルにカプセル化した(図1A)。ミクロゲルの一晩のインキュベーションの後、これらのカプセル化酵母細胞は等元性コロニーに成長した。mRNA捕捉用の第2マイクロ流体装置にゲルをロードする前に、酵母細胞壁を消化してmRNAをより使いやすくした(図1B、左)。これらのマイクロゲルを密に詰め込み、mRNA捕獲ビーズとリシスバッファーを結合しました。いくつかの液滴は、ちょうど1つのビーズを、酵母コロニーと組み合わせたものを含んでいた。エマルジョン中のすべてのビーズを収集し、cDNAを合成し、Drop-Seqプロトコルに従って配列した。
8滴スプリッタが付いたコカプセル化マイクロ流体装置を用いてアガロースミクロゲル内に単一酵母細胞カプセル化を介して等元性酵母コロニーを生成した(図2A)。マイクロゲルの30%が酵母を1個持つため、入力酵母懸濁液を約75万/mLの濃度に希釈しました。超低融解温度アガロースを装置に挿入する前に、高温で溶解し、この温度でシリンジを維持し、早期のゲル化を防ぎます。ドロップ生成接合部(図2B)では、酵母細胞を最初に160μmの液滴にカプセル化した。落下生成接合部に続いて、8つの折り畳みスプリッターがこれらの液滴を8個の80μmの液滴に分割した(図2C)。溶けたアガロースにシリンジフィルターを取り付けて、チャネル内に詰まりが生じないようにしました。我々は、すぐにアガロースのゲル化プロセスを開始した氷の上にエマルジョンを収集しました。典型的なエマルジョンの多分散度は~6%(補足図1)と計算したが、10%までの多分散値は許容できる。アガロースゲルがセットされると、エマルジョンを壊し、油相を取り除いた。ゲルを成長培地に浸漬する前に水性緩衝液で洗浄した。マイクロゲルの一晩のインキュベーションは、いくつかのマイクロゲル内で成長する等元性コロニーをもたらした(図2D)。少なくとも20個の細胞のコロニーを含むヒドロゲルの割合は、培養時間および培地組成を含む培養条件に依存した。C.アルビカンスを用いたデモンストレーションでは、ヒドロゲルの約15%が20時間の懸濁培養後にコロニーを含むと判断しました。
第2のカプセル化装置は、等原性コロニーからmRNAを抽出した(図3A)。酵母ミクロゲルをマイクロ流体デバイスにロードする前に、ゲルを溶液中に洗浄して浸漬して酵母細胞壁を消化しました。酵母細胞の適切な消化は顕微鏡法により検証され、より反射的な形態を有する処理酵母を有する(図3B)。マイクロゲルをシリンジに密閉し、ゲル入力流量を調整し、各ドロップに1つのゲルが入った。ドロップメイキング接合の前に、密に詰まったゲルストリームと混合されたリシスバッファー内のmRNA捕捉ビーズの流れ(図3C)。160μmの液滴のエマルジョンを採取し、コロニーが細胞内容物をライスして放出し始めた。複数のビーズを含む滴の数を最小限に抑えるために制限希釈液にビーズをロードしましたが、ドロップメイキング中のゲルの密閉充填により、溶解したコロニーを含む1つのビーズを含む回収された滴の約10%が得られた(図3D)。
ヒト腸内微生物叢に存在する酵母の一種であるC.アルビカンスの遺伝子発現を、ICO-seqワークフローを用いて分析した。C. アルビカンスは、白と不透明の2 つのセル状態を切り替える能力で注目されています。我々は、設計されたCアルビカン株、株RZY122を使用し、WH11遺伝子の1コピーを置き換え、白い細胞内でのみ活性をYFP20に置き換える。ワークフローを用いて遺伝子発現プロファイルを取得し、少なくとも300個のユニークな遺伝子を発現するコロニーの解析に使用しました。参照データセットとして、以前に発表された研究17から得られたC.Albicans発現データを使用し、600未満のユニークな遺伝子を発現するコロニーを除外しました。主成分(PC)解析とt-ストカシスニア埋め込み(tSNE)次元低減21を行った後、サンプルデータセットと参照との間に一般的な一般的な一角を見つけました(図4A)。PC分析の結果、YFPとWH11が最初の2台のPCに大きく貢献している。さらに、tSNE解析では3つのクラスタが明らかになった(図4B)。クラスター 2 はサンプル データセットのセルで主に構成されていましたが、クラスター 0 と 1 は両方のサンプルのセルで構成されていました。TSNE(図4C、上部パネル)にWH11発現を重ね合わせ、クラスタ1に白色コロニーが含まれている可能性が高いと判断した。また、クラスタ1(図4C、下部パネル)においてSTF2発現が増加し、以前に得られたデータ17と一致することがわかった。クラスタ 0 および 2 では、クラスタ 1 と比較して、WH11 と STF2 の規制が大幅に低下しました (図 4D)。ADH1などの発酵に関与する遺伝子は、クラスター0においてアップレギュレートされ、不透明細胞22の以前の研究と一致した。クラスター2のコロニーは、クラスター0および1のコロニーと比較してリボソームRNAが減少していることがわかった。サンプルと参照データセットは同じ細胞ストックを使用して取得されましたが、この結果は、実験的な取り扱いの微妙な違いでさえ遺伝子発現に影響を与える可能性があることを示唆しています。
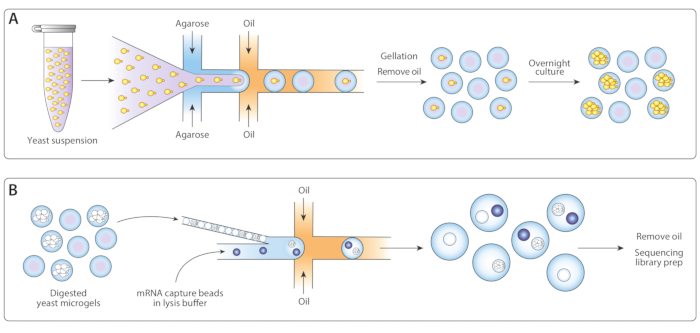
図 1: ICO-seq ワークフローの概要(A)懸濁液培養で増殖する酵母を緩衝液中で希釈し、溶融アガロースを流れ集中液滴発生装置に共封して、単一酵母細胞を有するアガロースミクロゲルのポアソンローディングを可能にした。アガロースが冷却し、油/水懸濁液が壊れ、油を取り除き、ゲルビーズを水中に懸濁させたときに設定されたゲル。一晩培養した後、酵母細胞はミクロゲル内の等元性コロニーに成長した。(B)コロニーは細胞壁分解緩衝液を受け、その後、近接パックされ、第2のマイクロ流体装置中にmRNA捕捉ビーズと共封された。マイクロゲルの密閉充填は、各滴が1つのゲルを持っていることを保証し、ビーズのポアソンローディングは1滴以内に複数のビーズの可能性を減らした。収集された滴は、cDNA合成およびシーケンシングライブラリの生成のために処理された。この図の大きなバージョンを表示するには、ここをクリックしてください。
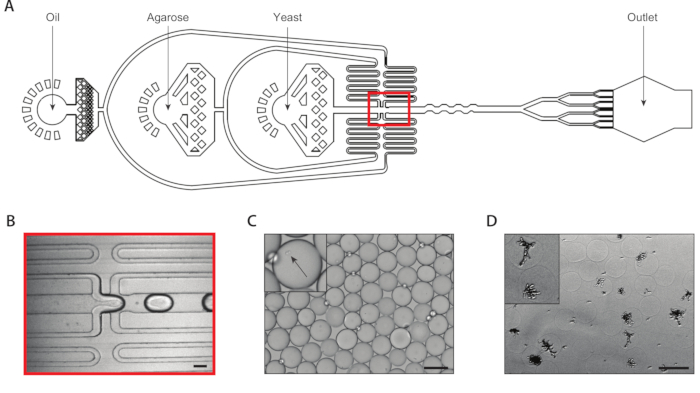
図2:デバイスAを用いたアガロースミクロゲル内の等元性酵母コロニーの生成。(A)マイクロ流体デバイスの概略、3つの入力および出力ポートの位置を示す。ドロップメイキング ジャンクションは赤で強調表示されます。(B) 通常のデバイス動作時のドロップメイキングジャンクションのクローズアップ。(C)回収された液滴の顕微鏡写真、カプセル化された細胞(インセット)を含む液滴のクローズアップを有する。(D) 24時間インキュベーション後のアガロースミクロゲル中のアイソジェニック酵母コロニーの顕微鏡写真を、2つのコロニー(インセット)のクローズアップで行う。すべてのスケールバー= 100 μm.この図の大きなバージョンを表示するには、ここをクリックしてください。
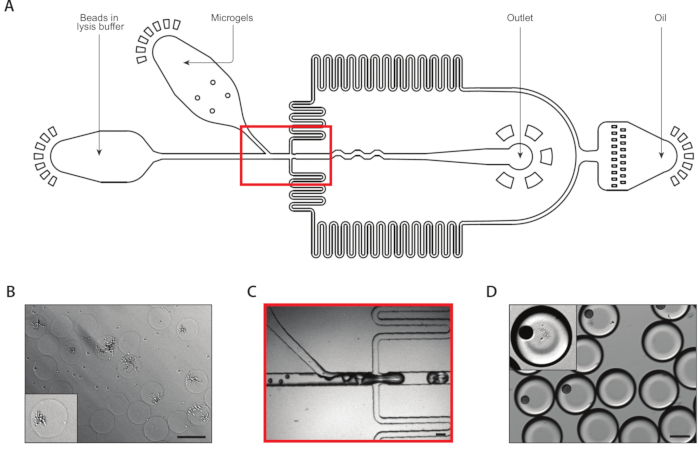
図3:デバイスBを用いたアイソジェニックコロニーからのリシスおよびmRNAの捕獲。(A)マイクロ流体デバイスの概略、3つの入力および出力ポートの位置を示す。ドロップメイキング ジャンクションは赤で強調表示されます。(B) 細胞壁消化後の酵母コロニーの顕微鏡写真を、1コロニー(インセット)のクローズアップで行う。(C)通常のデバイス動作時のドロップメイキングジャンクションのクローズアップ。(D) マイクロゲルとビーズのペアリング後に収集されたエマルジョンの顕微鏡写真、ビーズとリズングコロニー(インセット)でのドロップを示すクローズアップ付き。すべてのスケールバー= 100 μm.この図の大きなバージョンを表示するには、ここをクリックしてください。
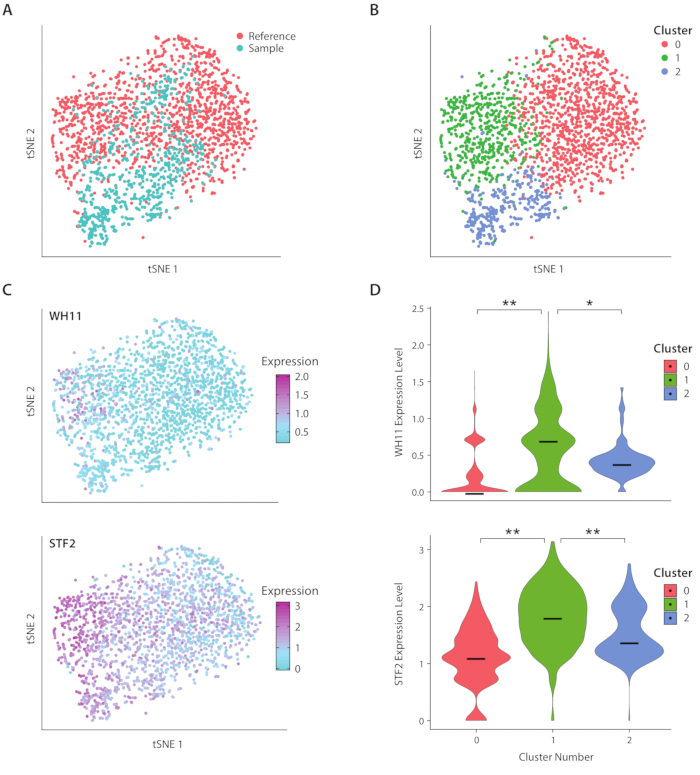
図4: C. アルビカンスにおける白色不透明切り替え応答の解析 .(A) サンプル データセットの tSNE プロットと、Liu17の参照データセットを組み合わせたもの 。(B) 転写体のクラスタリングは、tSNEプロット上で可視化された3つのクラスターを明らかにする。(C)白色不透明切り替え応答に関与する主要な遺伝子は、主成分分析を通じて決定された変動に寄与した。(D) tSNE プロットでマークされたクラスタによる YFP および WH11 の正規化された発現レベルのヴァイオリンプロット。**は p <<< 0.05 を示し、* は p << 0.05 を示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
補足図1と2.これらの汚いをダウンロードするには、ここをクリックしてください。
補助ファイル 1-3.これらのファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
イソジェニック酵母コロニーRNAシーケンシング(ICO-seq)の方法は、設計された酵母株のハイスループットスクリーニングのために、公表された単一細胞RNAシーケンシングプラットフォームDrop-Seqを適応させます。酵母細胞は、典型的な哺乳動物細胞のmRNAのコピーの10%未満を含み、mRNA捕捉前に分解する必要がある細胞壁を有する16。これらの2つの要因は、Drop-Seqまたは他の液滴ベースのscRNA-seqプラットフォームへの酵母の直接の適用を妨げる。これらの問題に対処するために、ヒドロゲル内に単一細胞をカプセル化し、RNAシーケンシングのための十分な入力材料を提供するためにコロニーに成長させ、酵母細胞壁を消化して、リシスおよびmRNA捕捉前にスフェロプラストを生成します。これらの変更は、元の Drop-Seq ワークフローと比較すると、ICO-seq ワークフローの複雑さを増し、ユーザーがスムーズに進める必要がある重要な手順です。
デバイスAの適切な動作は、アガロースヒドロゲル内の単一の酵母細胞をカプセル化するために必要です。入力酵母懸濁液の適切なカウントは、mRNA捕捉中の合理的な細胞捕捉効率を確保するために十分なヒドロゲルが単一の細胞を含み、一つ以上の酵母細胞を有するヒドロゲルの数を最小限に抑えるために従わなければならない。マイクロ流体デバイスの操作中に、アガロースゲル混合物はよく溶解し、デバイスの詰まりの可能性を最小限に抑えるために注射器フィルターを通過する必要があります。アガロースゲル混合物は粘性であり、単一のチャネルが8つに分割される領域は、特に詰まりを起こしやすい。高速カメラを中心にしてデバイスのその領域でデバイスの動作を可視化することで、ユーザーは8つのチャンネルのそれぞれから出てくる液滴の均一性を監視し、チャネルの詰まりによって均一性が変化した場合に迅速に反応することができます。顕微鏡下で少量の集められたエマルジョンを検査することで、高品質のエマルジョンを確認するための二次的な方法を提供します。
ヒドロゲル内の酵母コロニーの成長後、単一コロニーレベルでの高品質のmRNA抽出を確実にするためにいくつかの予防措置が必要です。酵母がヒドロゲル培養中の時間を最適化することが重要であり、酵母が培養中に長く放置されると、多くの人がヒドロゲルの閉じ込めを逃れ、RNAシーケンシング中に高いバックグラウンドシグナルを引き起こし、細胞型間で識別する際の感度が低下するためです。Zymolyaseを使用した球状体系の適切な生成は、mRNAがリシスバッファーへの細胞暴露後に放出されることを保証する。Zymolyaseに続く酵母コロニーの目視検査は、より光沢のある酵母細胞を得る必要があります。不適切な細胞壁の消化は、RNAの捕捉効率を低下させます。最後に、ハイドロゲルはデバイスBに注入される際に密接にパックする必要があります。高速カメラでヒドロゲル入力を監視すると、ハイドロゲルがデバイスへの入力時に閉じ込めされなくなったらエマルジョンコレクションを終了することができ、そうでなければ捕獲効率に影響を与えます。
我々の方法に関する潜在的な懸念は、酵母のミクロゲル培養が遺伝子発現を著しく変化させる可能性があるということです。マイクロゲルおよび寒天における酵母遺伝子発現を調査する以前の研究は、遺伝子発現平均の違いを示しているが、全体的に正の相関17を示しているが、様々な酵母株に関するこの主張のさらなる調査は慎重である。この方法はまた、ポアソン統計14に続くmRNA捕捉ビーズの確率的負荷による細胞捕捉効率も限定的である。現在、約10%の滴にはビーズとコロニーが含まれ、二重カプセル化率は1%以下になると予想されています。二重カプセル化はRNA-seqデータ分析中に交結合要素につながり、それらのフィルタリングは挑戦的な23;25%の捕獲率は、対応する二重カプセル化の5%への増加につながるだろう(補足図2)。Drop-Seq プラットフォームを使用した ICO-seq のデモンストレーションを行いますが、市販の 10x ゲノミクス クロム プラットフォーム15,,24など、統計的にではなく、決定的に mRNA キャプチャビーズを導入する他の液滴 RNA-seq プラットフォームがあります。これらのプラットフォームとICO-seqの統合は、ポアソン統計が許す以上のキャプチャ効率を高める可能性があります。最後に、液滴RNA-seqの根本的な制限は、シーケンシング後に目的の細胞を回収できないことです。この方法を使用して分析する酵母ライブラリの種類を考慮する際には、この制限を考慮する必要があります。
細胞間ヘテロジニアリティは、大腸菌25およびSなどの微生物のクローンレベルで実証されている。cerevisiae26は、バルクレベル分析がそうでなければマスクされることを新しい細胞状態を明らかにする。C.アルビカンスで行われたバルクRNA-seq分析は、集団全体の転写体変化を見るか、または白および不透明な細胞を2つの別々の集団27,28,28として見る傾向がある。ICO-seqの適用は、追加のサブステイトの発見につながり、他の酵母種内の新しい細胞状態を発見するための分析フレームワークを提供する可能性があります。しかし、ヒドロゲル内の細胞の増殖は、酵母に限定されない:哺乳動物、細菌、および他の真菌細胞のような他の細胞タイプも、ヒドロゲル29、30,30内で培養されてもよい。単一細胞に対する等原性コロニーのシーケンシングは、細胞間の変動による生物学的ノイズの平均化をもたらし、細胞タイプ間の差別を改善する。これは、遺伝的多様性が特定の合成経路に焦点を当て細胞を分析する際に役立つ可能性があります。.ICO-seqへの細胞型入力の可能性の拡大と、市販の液滴RNA-seqプラットフォームとの潜在的な統合は、ICO-seqを遺伝的レベルで細胞異質性を解剖するための有望なプラットフォームとして位置付けています。
開示事項
著者らは利益相反を宣言しない。
謝辞
このプロジェクトは、国立科学財団キャリアアワードDBI-1253293、国立衛生研究所新イノベーター賞DP2AR068129、助成金R01HG008978、国立科学財団技術センター補助金DBI-1548297、UCSF細胞構築センターによって支援されました。ARAとZJGはチャン・ザッカーバーグバイオハブの調査官です。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 um syringe filter | Milipore Sigma | SLGP033RS | |
| 0.5M EDTA, pH 8.0 | Thermo-Fisher | 15575020 | Used to make TE-TW buffer |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| 1 mL syringes | BD | 309628 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| 1M Tris-HCI, pH 8.0 | Thermo-Fisher | 15568025 | Used to make TE-TW buffer |
| 27 gauge needles | BD | 305109 | |
| 3 mL syringes | BD | 309657 | |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | See Supplemental Files for 3D print file |
| Agarose, low gelling temperature | Sigma-Aldrich | a9414 | |
| Aquapel (hydrophobic glass treatment) | Pittsburgh Glass Works | 47100 | |
| Drop-Seq Beads | ChemGenes | MACOSKO-2011-10 | |
| Glass microscope slides (75 mm x 50 mm) | Corning | 294775X50 | |
| Ionic Krytox Surfactant | Synthesis instructions in ref 14. Can substitute with PEG-PFPE surfactant. | ||
| Isopropanol | Sigma-Aldrich | 109827 | |
| NaCl | Sigma-Aldrich | S9888 | |
| Novec 7500 | 3M | 98-0212-2928-5 | Commonly knowns as HFE 7500 |
| PBS | Fisher Scientific | BP243820 | |
| PE-2 polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| PEG-PFPE surfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Photomasks | CadArt Servcies | (custom) | See Supplemental Files for mask designs |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| SSC Buffer | Sigma-Aldrich | S6639 | |
| SU-8 2100 | MicroChem | Y111075 | |
| SU-8 2150 | MicroChem | Y111077 | |
| Sylgard 184 silicone elastomer kit | Krayden | 4019862 | |
| Tween-20 | Sigma-Aldrich | P1379 | Used to make TE-TW buffer |
| YR Digestion buffer | Zymo Research | R1001-1 | Spheroplasting buffer |
| YR Lysis Buffer | Zymo Research | R1001-2 | |
| Zymolyase | Zymo Research | E1005 | Spheroplasting enzyme mixture |
参考文献
- Peralta-Yahya, P. P., Zhang, F., Del Cardayre, S. B., Keasling, J. D. Microbial engineering for the production of advanced biofuels. Nature. 488 (7411), 320-328 (2012).
- Curran, K. A., Alper, H. S. Expanding the chemical palate of cells by combining systems biology and metabolic engineering. Metabolic Engineering. 14 (4), 289-297 (2012).
- Botstein, D., Chervitz, S. A., Cherry, J. M. Yeast as a model organism. Science. 277 (5330), 1259-1260 (1997).
- Mager, W. H., Winderickx, J. Yeast as a model for medical and medicinal research. Trends in Pharmacological Sciences. 26 (5), 265-273 (2005).
- Krivoruchko, A., Siewers, V., Nielsen, J. Opportunities for yeast metabolic engineering: Lessons from synthetic biology. Biotechnology Journal. 6 (3), 262-276 (2011).
- Vanella, R., et al. Yeast-based assays for screening 11β-HSD1 inhibitors. Microbial Cell Factories. 15 (1), (2016).
- Zhuang, X., Chappell, J. Building terpene production platforms in yeast. Biotechnology and Bioengineering. 112 (9), 1854-1864 (2015).
- Agresti, J. J., et al. Ultrahigh-throughput screening in drop-based microfluidics for directed evolution. Proceedings of the National Academy of Sciences. 107 (9), 4004-4009 (2010).
- Wang, G., et al. RNAi expression tuning, microfluidic screening, and genome recombineering for improved protein production in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 116 (19), 9324-9332 (2019).
- Beneyton, T., et al. Droplet-based microfluidic high-throughput screening of heterologous enzymes secreted by the yeast Yarrowia lipolytica. Microbial Cell Factories. 16 (1), 18(2017).
- Sjostrom, S. L., et al. High-throughput screening for industrial enzyme production hosts by droplet microfluidics. Lab on a Chip. 14 (4), 806-813 (2014).
- Nadal-Ribelles, M., et al. Sensitive high-throughput single-cell RNA-seq reveals within-clonal transcript correlations in yeast populations. Nature Microbiology. 4 (4), 683-692 (2019).
- Gasch, A. P., et al. Single-cell RNA sequencing reveals intrinsic and extrinsic regulatory heterogeneity in yeast responding to stress. PLoS Biology. 15 (12), 2004050(2017).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- von der Haar, T. A quantitative estimation of the global translational activity in logarithmically growing yeast cells. BMC Systems Biology. 2, 87(2008).
- Liu, L., Dalal, C. K., Heineike, B. M., Abate, A. R. High throughput gene expression profiling of yeast colonies with microgel-culture Drop-seq. Lab on a Chip. 19 (10), 1838-1849 (2019).
- Fallah-Araghi, A., Baret, J. C., Ryckelynck, M., Griffiths, A. D. A completely in vitro ultrahigh-throughput droplet-based microfluidic screening system for protein engineering and directed evolution. Lab on a Chip. 12 (5), 882-891 (2012).
- Berman, J., Sudbery, P. E. Candida albicans: A molecular revolution built on lessons from budding yeast. Nature Reviews Genetics. 3 (12), 918-930 (2002).
- Srikantha, T., Soll, D. R. A white-specific gene in the white-opaque switching system of Candida albicans. Gene. 131 (1), 53-60 (1993).
- van der Maaten, L., Hinton, G. Visualizing data using t-SNE. Journal of Machine Learning Research. 9, 2579-2605 (2008).
- Sun, Y., et al. Deletion of a yci1 domain protein of Candida albicans allows homothallic mating in MTL heterozygous cells. mBio. 7 (2), 00465-00516 (2016).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet Detection in Single-Cell RNA Sequencing Data Using Artificial Nearest Neighbors. Cell Systems. 8 (4), 329-337 (2019).
- Baran-Gale, J., Chandra, T., Kirschner, K. Experimental design for single-cell RNA sequencing. Briefings in Functional Genomics. 17 (4), 233-239 (2018).
- Silander, O. K., et al. A Genome-Wide Analysis of Promoter-Mediated Phenotypic Noise in Escherichia coli. PLoS Genetics. 8 (1), 1002443(2012).
- Newman, J. R. S., et al. Single-cell proteomic analysis of S. cerevisiae reveals the architecture of biological noise. Nature. 441 (7095), 840-846 (2006).
- Tuch, B. B., et al. The Transcriptomes of Two Heritable Cell Types Illuminate the Circuit Governing Their Differentiation. PLoS Genetics. 6 (8), 1001070(2010).
- Romo, J. A., et al. Global Transcriptomic Analysis of the Candida albicans Response to Treatment with a Novel Inhibitor of Filamentation. mSphere. 4 (5), 00620(2019).
- Huang, H., et al. Generation and manipulation of hydrogel microcapsules by droplet-based microfluidics for mammalian cell culture. Lab on a Chip. 17 (11), 1913-1932 (2017).
- Lin, X., Nishio, K., Konno, T., Ishihara, K. The effect of the encapsulation of bacteria in redox phospholipid polymer hydrogels on electron transfer efficiency in living cell-based devices. Biomaterials. 33 (33), 8221-8227 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved