Method Article
微細藻類のモデル改良のための高スループット代謝プロファイリング
要約
このプロトコルは、表現型マイクロアレイ(PM)技術プラットフォームを使用して、緑の微細藻類 であるクラミドモナス・レインハルトイ の代謝要件を定義し、既存の代謝ネットワークモデルを改良することを実証しています。
要約
代謝モデルは、生物の利用可能なゲノム注釈に基づいて再構築され、システムレベルで代謝プロセスを研究するための予測ツールを提供します。ゲノムスケールの代謝モデルには、実験的に未確認の反応と同様にギャップが含まれる可能性があります。新たに単離された微細藻類種の再構築モデルは、通常、そのような分離株の代謝に利用可能なまばらな生化学的証拠があるので、これらのギャップによる弱点をもたらす。表現型マイクロアレイ(PM)技術は、幅広いエントリ代謝産物に応答して細胞代謝活動を機能的に決定する効果的で高スループットな方法です。高スループットの表現型アッセイと代謝モデリングを組み合わせることで、ゲノム証拠をサポートおよび拡張するための生化学的証拠を提供することで、既存の代謝ネットワークモデルを迅速に再構築または最適化することができます。本研究では、緑色の微細藻類モデル種クラミドマス・reinhardtiiを例に用いた微細藻類研究のためのPMアッセイの使用を示す。PMが得た254以上の反応の実験的証拠は、ゲノムスケールC.reinhardtii代謝ネットワークモデルi RC1080を約25%拡大し、精製するために使用された。ここで作成されたプロトコルは、既知の微細藻類変異体および新しい分離株を含む他の微細藻類の代謝を機能的にプロファイリングするための基礎として使用することができる。
概要
標的代謝産物の増強と安定した生産のために藻類代謝を最適化するには、代謝ネットワークのシステムレベルの解析を通じた複雑な代謝工学戦略の開発が必要です。代謝ネットワークモデルは、最適化戦略1、2、3、4の迅速な開発のための合理的な設計を導くことができます。約160種の微細種が5種配列化されているが、我々の知る限りでは、4,6,7の藻類代謝モデルは44種類しか存在しません。ゲノム情報の実験検証のための高スループット代謝型フェノミカルデータの取得が困難なため、高品質ネットワークモデルの再構築は藻類ゲノムシーケンシングの急速な発展に遅れをとっています。
C.reinhardtiiは藻類ベースの研究のための魅力的なモデルシステムです。この種は、光自己または不均一に成長することができ、基礎および応用研究のモデル生物として広く使用されています。そのゲノム配列は2007年8月に発表され、ゲノムスケールの代謝モデルは9、10、11種のために再構築された。C.reinhardtii(i RC1080)のゲノムスケールモデルをChangらによって再構築した。 10ゲノムおよび文献の証拠に基づく(〜250の情報源を伴う)。それは2,190反応10と1,706代謝物を有する;しかし、モデルの完全性は、当時公開された公開された実験証拠を超えて検証できませんでした。
表現型マイクロアレイ(PM)技術は、組織培養細胞だけでなく、ヘテロ栄養微生物の代謝プロファイリング情報を提供できるハイスループットプラットフォームです。特に、クロミドモナス・レインハルト12に対して最初に報告されたように、クロロイドニウム13およびクロレラ14の種に対して最初に報告されたように、微細藻類における表現型間の知識ギャップに対処するために使用することができる。何千もの代謝産物、シグナル伝達分子、浸透分子、およびエフェクター分子に対する細胞応答を研究することにより、PMアッセイは機能的代謝プロファイリングを提供し、機能、代謝、および環境感受性15、16、17に関する洞察を提供することができる。具体的には、PMアッセイは、各ウェルに含まれる異なる栄養素、代謝物、または浸透量が異なる96ウェルマイクロプレートにおける細胞代謝産物の利用を検出する。また、抗生物質やホルモンなどの生理活性分子をアッセイすることもできる。テトラゾリウム系酸化還元色素のNADH還元による色の強度によって決定されるように、基質の代謝利用率は細胞呼吸15、16、17の観点から評価される。96ウェルマイクロプレートでの実験は、表現型マイクロアレイ装置(PMI)プラットフォームを使用して、時間の経過とともに自動的に監視および決定することができます。20個の96ウェルマイクロプレートは、炭素、窒素、硫黄、リン源を利用する細胞表現型を研究するための共通のセット代謝産物を表すように設計されており、異なる浸透/イオンおよびpH効果を有する。PM技術は、微生物15、16、17、18の既存のゲノムスケール代謝モデルの数を更新し、アップグレードするために正常に使用されています。
ここに示すプロトコルとデータは、Chaiboonchoeらによって以前に公開された作品に基づいています 。12本発表の研究では、微小藻類の代謝表現型を特徴付け 、C.reinhardtii の既存の藻類代謝モデルを拡大し、新しい代謝モデルの再構築を導くために、PMアッセイ法の使用について詳しく説明した。
プロトコル
1. 表現型マイクロアレイ実験
- ミネソタ大学クラミドモラスリソースセンター(https://www.chlamycollection.org)から C.reinhardtii 株CC-503を取得します。
- 400 μg/mL インフィング、50 μg/mL アンピシリン、および 100 μg/mL カナマイシン (細菌増殖を阻害する) の新鮮なトリス-酢酸リン酸 (TAP) 培地19で細胞を成長させます(細菌増殖を抑制する) 400 マイクロモル光素子/ms の下で 25 °C, 25 °C, 中期段階ログから 2 日間.
- 22°Cで10分間2,000 x gで培養液をスピンダウンし、ペレットを邪魔することなく上清を捨てます。
- 0.1%テトラゾリウムバイオレット染料「D」を含む新鮮なTAP培地を準備します。
注:このステップでTAP培地を変更して、各プレートでテストされた代謝産物カテゴリに応じて栄養素を除外します(例えば、窒素源プレートの塩化アンモニウムを除外します)。 - ペレットを調製した新鮮なTAP培地(ステップ1.2から)で1 x 106 細胞/mLの最終濃度まで再懸濁します。
- 化学化合物アレイアッセイプレート(炭素源、窒素源、リンおよび硫黄源プレート、ペプチド窒素源)を使用します。
- 細胞含有培地の100μLアリコートをアッセイプレートの各ウェルに接種します。
注:必ずアッセイを複製してください。 - 酵母エキス/ペプトンプレート上のストリーク細胞は、スミスらのように、グラム染色を行 う。20の前および後の細菌汚染を監視するアッセイ。
- 化学化合物アレイアッセイプレートをマイクロプレートリーダーシステムに挿入します。
- 30°Cですべてのプレートを最大7日間インキュベートし、15分ごとに色素の色の変化を読み取るようにマイクロプレートリーダーシステムをプログラムします。
注:ほとんどのマイクロプレートリーダーは、インキュベーション中に連続光の源を提供しなくて、藻類は、ヘテロ栄養呼吸を行うことができるはずです。
2. データ分析
- マイクロプレートリーダーから生のキネティックデータをCSVファイルとしてエクスポートし、その後、R.のオムニログ表現型マイクロアレイ(OPM)パッケージへの入力として使用されます。
- PMキネティックデータコンバータソフトウェアを使用する。D5E データ ファイルをロードし、PM キネティック解析ソフトウェアの次のコマンドラインを使用して、OKA ファイルに変換します。
|を読み込むインポート (OKA ファイルのフォルダを検索する) |フィルタの|の設定インポート |すべてのプレートを追加|閉める。
輸出| [データの読み取り (キネティック)] を選択し、 フォーマット (CSV) (タブヘッダー)を選択して、プレート (各プレート (個々のファイル) ) を選択|データ |のエクスポートセーブ。 - 表現型マイクロアレイ(PM)データ解析を実行するには、Rソフトウェア環境内で動作するOPMソフトウェアパッケージ21,22を使用します。パッケージ、チュートリアル、およびリファレンス ドキュメントは、次の http://www.goeker.org/opm/ にあります。RStudio では、R のグラフィカル ユーザー インターフェイスで、次のコマンドを使用して opm パッケージとその依存関係をインストールします。
ソース (http://www.goeker.org/opm/install_opm.R)
図書館(opm) - キネティックデータの CSV ファイルを含むディレクトリに移動し、opm 関数 read_を使用してデータをインポートします。
x <- read_opm ("." " - 曲線パラメータ推定を使用して、運動データを集約し、分離します。
(i in 1 :length(x)) {
x[i]<私は_aggre(x[[i]、ブート = 0 L、コア= 1 L、メソッド="スプライン"、オプション=set_spline_options("smooth.spline"))
x[[i]<- do_disc(x[[i],カットオフ = FALSE)
}
メタデータの#Collection
メタデータ <- collect_template(")
メタデータ$ひず<-c("ブランク"""、CC- 503")
(I in 1 :length(x)) {x[i]<-include_metadata(x[i], md = メタデータ,置き換え = TRUE)} - xy_plot関数を使用して、呼吸(または成長)測定(y軸)をアッセイされた96ウェルプレートの時間(x軸)の関数としてマッピングします。
印刷 (xy_plot[x[]1]、="ひずみ"、セオ.max = FALSEを含む) - 関数 level_plot を使用してデータをヒート マップとして視覚化し、キネティック データの簡単な比較概要を確認します。
level_plot(x, main = list)、色 =opm_opt("color.borders")、panel.headers = メタデータ$ひずみ、cex = NULL、ストリップ.fmt = リスト)、ストリップテキスト.fmt = リスト)、レジェンド.sep ="、スペース="ラボ"、バイアス = 0.7、num.colors = 200 L) - 重要な生物学的情報を抽出し、曲線パラメータは、生の運動曲線から、ラグ相(λ)、増殖速度(μ λ)、最大細胞呼吸(A)、および曲線下面積(AUC)21を含む。正の代謝物を識別するには、各マイクロウェルプレートのブランクに加えて、培地との色素の非生物反応性を表す負のコントロールのA値を背景の減算値として使用する。抽出関数は、A パラメーターを取得するために使用されます。
opm_opt(「カーブ.パラム」)
パラム<- 抽出 (x, as.labels = リスト("ひずみ")) )
- PMキネティックデータコンバータソフトウェアを使用する。D5E データ ファイルをロードし、PM キネティック解析ソフトウェアの次のコマンドラインを使用して、OKA ファイルに変換します。
3. 新しい代謝産物に関連する反応と遺伝子の同定
- KEGG(京都遺伝子・ゲノム百科事典)(http://www.genome.jp/kegg/)とMetaCyc(http://metacyc.org/)を検索し、化学化合物アレイから見つかった代謝物を用いた反応について酵素委員会番号(AC)を同定する23,24,23,24.
- 同定されたEC番号を、関節ゲノム研究所(JGI)、フィトゾーム(http://www.phytozome.net)、査読出版23、25、26、27などの複数の利用可能な藻類注釈リソースの検索基準として使用する。
- クエリが特定のEC番号の遺伝的証拠を返さない場合は 、C.reinhardtiiに最も近い種から始めて、他の生物の関連する関連タンパク質を特定し、デフォルト設定でNCBI PSI-BLASTサーバーを使用してプロファイルベースの検索を行い 、C.reinhardtii(taxid:3055) で非冗長タンパク質(nr)を使用して反応12に関連する候補遺伝子を同定します。
- EMBL-EBI Pfam (http://pfam.xfam.org/search)またはInterPro(http://www.ebi.ac.uk/interpro/)タンパク質ドメイン予測サーバーを介してBLASTヒットを照会することにより、検索されたEC番号との関連性について、PsI-BLASTヒットを0.05のE <値で手動でキュレーションします。後者の2つのスキャンは、タンパク質の正しい酵素活性の同定を確実にするための重要なステップであることに注意してください。
4. モデルの改良と評価
- 最新のCOBRAツールボックスv.3.0を使用する28マットラボで29,30モデルの絞り込みのための次のステップを実行するプラットフォーム。COBRA ツールボックスは、「https://opencobra.github.io/cobratoolbox/stable/installation.html」の手順に従ってインストールできます。また、COBRA ツールボックスは Python (COBRApy) などの他のオープン ソース プログラミング言語にも実装されています。31)、https://opencobra.github.io/cobrapy/ で入手できます。
- COBRAツールボックスv.3.0をインストールした後、MATLABを開き、次のコマンドを実行してツールボックスを初期化します。
イニトコブラツールボックス; - COBRA ツールボックス関数addReactionとchangeGeneAssociationを使用して、関連する遺伝子と共に同定された反応を代謝モデル(iRC1080 など) に追加します。http://bigg.ucsd.edu/models/iRC1080 からダウンロードしたiRC1080 モデルを含むディレクトリに移動し、次のコマンドを実行してモデルをロードし、名前を変更し、新しいリアクションとそれに関連するジーンを追加します。
ロード('iRC 1080 .mat')
モデル新しい = iRC 1080;
モデル新しい = 反応を追加する(モデル、'D-ALA 2'、..
{'d-ala[c]'、'atp[c]'、..
'D-アラデータ[c]、'adp[c]'、'pi[c]'、..
'h[c]' },,2 - 1 1 1 1 1 [,false);
モデルNEW = 変更遺伝子協会(モデル新、..
'D-ALA 2','au.g 14655 _t 1' ); - 代謝産物が細胞内で産生されないが培地から取り込まれる場合には、新しい代謝産物の輸送反応をモデルに追加する。これらの輸送反応は、細胞外媒体から細胞質体への代謝産物の受動的拡散を表す。さらに 、addExchangeRxn 関数を使用して対応する人工交換反応を追加して、代謝産物を細胞外媒体に入力または出力します。
モデル新しい = 反応を追加する(モデル、'CYCPt'、..
{'cycp[e]','cycp[c]' },,1 1 1 [,真)'
モデル新しい = addExchangeRxn(モデル、'cycp[e]' ,- 1000,1000); - 新しい結果モデルの挙動をテストし、例 えば、BD1106は、光と闇 条件下で 、バイオマスを最大化するための光と暗い条件下でフラックスバランス解析(FBA)を実施し、目的関数として機能を発揮します。光の成長のために、PRISM太陽リチョの光反応の下限と上限を646.07(最大レート)に設定します。暗い成長のために、すべてのPRISM光反応の限界をゼロに設定します。暗い条件と明るい条件下での成長のために、以前に定義されたバイオマス関数を使用してください。FBA溶液は、反応流束(solution.v)とコスト削減(solution.w)に対応する2つのベクトルと、代謝産物の影の価格(solution.y)に対応する1つのベクトルを出力します。
%光条件下での成長をシミュレートする:
モデル新しい = 変更Rxn境界(モデル新しい、{..
%'PRISM_solar_litho'、..
'PRISM_solar_exo' , ..
'PRISM_incandescent_ 60 W'、..
'PRISM_fluorescent_cool_ 215 W' ,..
'PRISM_metal_halide'、..
'PRISM_high_pressure_sodium'、..
'PRISM_growth_room'、..
'PRISM_white_LED'、..
'PRISM_red_LED_array_ 653 nm' ,..
'PRISM_red_LED_ 674 nm' ,..
'PRISM_fluorescent_warm_18 W'、..
'PRISM_design_growth'、..
}, 0, 'b' );
モデル新しい = 変更目的(モデル、新しい、'BIOMASS_Chlamy_mixo')。
FBAsolution 新しい = 最適化CbModel(モデル新、'最大'); - ステップ 4.1.4 を iRC1080 で繰り返して 、iBD1106 用に得られた FBA ソリューションと iRC1080 用に得られたものを比較します。
- モデルの比較に使用できるさまざまなCOBRA法があります(例えば、フラックス変動解析、遺伝子欠失解析、ロバストネス分析、フラックス分割予測、FBA、サンプリングなど)。詳細なチュートリアルは、https://opencobra.github.io/cobratoolbox/stable/tutorials/index.html で見つけることができます。ここでは、iRC1080モデルを精製版 iBD106と比較する例を提供し、各モデルで説明する代謝産物の影価格(バイオマス目的関数のシステム変数の変化に対する感度)を得ることによって提供される。
代謝物の影の価格を取得します。
シャドウ価格 = テーブル(モデルNew.mets、..
モデル新しい.メットネーム、FBAソリューション新しい.y);
- COBRAツールボックスv.3.0をインストールした後、MATLABを開き、次のコマンドを実行してツールボックスを初期化します。
結果
モデル藻体クラミドモナス・レイアルドティの表現型マイクロアレイスクリーニング
PMアッセイは、最小限の媒体で炭素、窒素、硫黄、リンの様々な供給源を利用する藻類の能力をテストします。本手法では、PMアッセイを用いて炭素と窒素の代謝を同定する方法を示した。炭素および窒素利用速度論をマイクロプレートリーダーで測定した。データはPMIソフトウェアを用いて分析した。選択したPMアッセイプレート(PM01およびPM03)の概略運動量を 図1に示す。「xyプロット」は、96ウェルプレートのアッセイのためにプロットされた時間の経過に関する呼吸測定を表示し、y軸とx軸はそれぞれ生の測定値と時間の値を表します。データをヒートマップパターンに変換し、運動データの組立を比較的解析した。
PMデータを用いたゲノム規模の代謝ネットワークの精製パイプライン(図2)は、高スループットPMアッセイと、ゲノム検索によって提供される実験証拠の統合が代謝ネットワークモデルを拡大できることを示している。
PM01-04およびPM10プレートから得られたPMデータの再現性を判定するために、線形回帰を分析し、互いに対して2つの独立した反復実験からのデータをプロットした(図3)。図3は、データの大部分が45°ラインに落ちるにつれてほぼ類似しており、外れ値はごくわずかであり、測定係数R2は0.9であったことを示しています。藻類の実験の一貫性と再現性は、このプロットによって検証されます。
新しい代謝物の同定
PMアッセイは7つのプレートで662の代謝産物を同定した。PM01-PM04およびPM06-PM08は、ガスクロマトグラフィー時間-飛行時間(GC-TOF)が77の代謝産物32を同定した(図4)。これら2つの集合を iRC1080で説明した1068の代謝産物と比較すると、3つのセットの間で重なった代謝物は6つだけで 、149個はiRC1080とPMの間で重なった。この結果は、代謝プロファイリングプラットフォームが新しい代謝情報の重要な供給源となり得ことを示しています。
酢酸は、背景信号を減算した後に支持炭素としてプレートPM01で検出された唯一の炭素源であった。この知見は、文献33と一致し、PMアッセイの特異性を示す。PMアッセイは 、C.reinhardtii が成長のために利用できる新しい硫黄、リン、および窒素源を明らかにした。硫黄代謝物は硫酸塩、チオ硫酸、テトラチオン酸、DL-リポアミドであった。リン源はチオリン酸、ジチオリン酸、D-3-ホスホグリセリン酸、システアミン-S-リン酸であった。窒素源代謝物は、Lアミノ酸とDアミノ酸であり、あまり一般的でないアミノ酸を含む。L-ホモセリン、L-ピログルタミン、メチルアミン、エチルアミン、エタノールアミン、およびD、L-α-アミノ酪酸、および108ジペプチドおよび5個のトリペプチド(表 1)新たに同定された128個の代謝産物はすべて、KEGGおよびMetaCycで、関連する反応、EC番号、および経路について検索された。
新しい128の代謝物は49の一意のEC番号に関連付けられていた。このうち、15個のICが、5つの情報源を用いてゲノム証拠にリンクされていた。フィトゾームバージョン10.0.234 JGIバージョン435、AUGUSTUS5.0、および5.210のマニチャイクルらからの注釈36とKEGG13.ゲノムエビデンスのない代謝産物は、ユニバーサルタンパク質資源のウェブサイト(UniProt、http://www.uniprot.org/)37,38に入力され、そこで他の生物に関連する配列が見つかった。C.reinhardtiiの相同配列は、有意なアライメントを生み出した配列のみを考慮してNCBIウェブサイトから位置特異的反復BLAST(PSI-BLAST、https://blast.ncbi.nlm.nih.gov/Blast.cgi)を実行することによって同定された(E値<0.005)。
モデルの絞り込み
新しい128代謝産物に関連する反応は、そのコードされた遺伝子と共に 、iRC1080モデルに追加され、ネットワークを拡大した。得られたモデル iBD1106は、2,444反応、1,959個の代謝産物、および1,106遺伝子を占める(表 2)。新たに254個の反応を加えたのは、20個のアミノ酸酸化反応、108個のジペプチド加水分解反応、5つのトリペプチド加水分解反応、および120個の輸送反応で、4つの遺伝子(Cre02.g096350.t1.3、au.g14655_t1、e_gwW.1.243.1、Cre12.g48635.t.3.t
合計113の追加の新しい反応は、ジペプチドと三ペプチドの加水分解を占めています。ジペプチドと三ペプチドの加水分解は、2つの遺伝子、1つは、ジペプチド(Cre02.g078650.t1.3)と3ペプチド用(Cre16.g675350.t1.3)に関連付けられている。
リンの供給源に関しては、システアミンS-リン酸のシステアミンおよびリン酸への加水分解に対する反応を、その遺伝子JLM_162926に関連付けて添加した。
WoLF PSORTツール39(http://www.genscript.com/psort/wolf_psort.html)とガムサリらによって報告された結果。35は、新しい反応が行われる細胞コンパートメントの仕様を得るために適用された。新しい反応に関連するタンパク質配列を分析することにより、WoLF PSORTは細胞コンパートメントとして細胞質を反応させると予測した。
生まれた代謝モデルは、生化学的情報が不完全な場合にギャップを含む可能性がある。このような場合は、gapFind、COBRA コマンドが使用されます。ルート ギャップを一覧表示し、新しいモデルで新しいギャップの導入を識別できるようにします。代謝モデルでは生成できない代謝産物を、根隙40,41と呼びます。ルートギャップを分析すると、iRC1080とiBD1106モデルの両方に同じ91ギャップが含まれていることが示されました。これは、新しい代謝産物とそれに関連する反応を追加しても、追加の根間ギャップが導入されないことを示しています。原発性の根間間代謝物には、表現型アッセイでは取り上げられなかった輸送機構や生産機構がないため、このプロトコルで使用される表現型法は根隙を埋めずに行う必要がある。フラックスバランス解析は、光と暗い条件下でiBD1106の代謝挙動をテストするために行った;(酢酸塩なし)と(酢酸塩を含む)をそれぞれ、それぞれ。このアルゴリズムは、目的関数(バイオマス成長)のためのバイオマス前駆体反応を最大化します。各代謝産物が設定された目的関数に関与することを評価するために、全ての代謝物に対する「影の価格」を計算した。代謝物の流束変化に関する目的関数の変化は、代謝産物30,42の影の価格を定義する。代謝産物が「過剰」であるか、または「制限」している目的関数の指標は、例えばバイオマス生産の影の価格分析によって決定することができる。負または正の影の価格値は、追加されると、目的関数を減少または増加させる代謝物を明らかにします。影の価格のゼロ値は、目的関数に影響を与えない代謝物を明らかにします。図5のiBD1106とiRC1080の影の価格の比較は、ほとんどの代謝産物にとって、大きな変化は観察されないことを示している。しかし、それぞれ明暗の成長条件下で105と70のケースに違いが見られます。表4にそのような代謝物の例が含まれる。
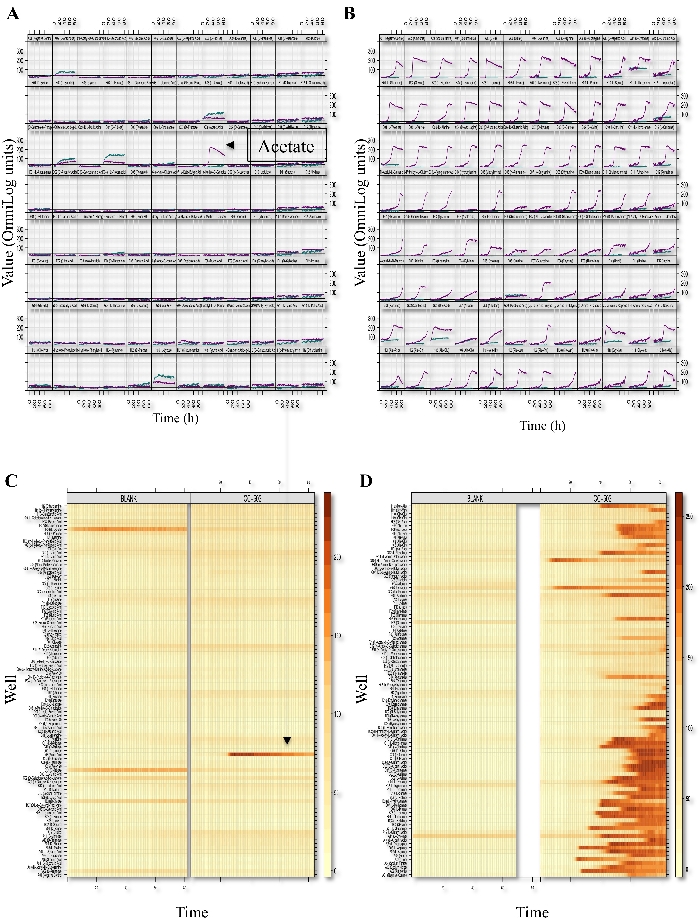
図1: C.reinhardtiiのフェドミピックマイクロアレイプロファイリングPM01の呼吸XYプロットとレベルプロット(炭素源;A,C)およびPM03(窒素源;B,D)アッセイプレートが示されている。図は8x12配列で、各セルはウェルプレートを表し、したがって、所定の代謝物または成長環境を表します。各セルまたはウェル表現内では、曲線は時間(x軸)の関数として、減少(y軸)による色素変換を表します。CC-503およびブランクウェルからのPM呼吸曲線は各セルに示され、色で示されます(色は白いウェルを表し、紫色はCC-503を表します)。レベルプロットは、各呼吸曲線を、時間の経過とともに色を変化させる(または変化しない)細い水平線として表します。ヒートマップの色の変化は、淡黄色(ほとんどまたは全く呼吸が行われなかった)から濃いオレンジまたは茶色(大幅な呼吸が行われている)に変化します。C.reinhardtii(CC-503)およびブランクプレートで利用される代謝物が示されている。この図は、チャイブーンチョーらによる以前に出版された作品からである。12この図の大きなバージョンを表示するには、ここをクリックしてください。
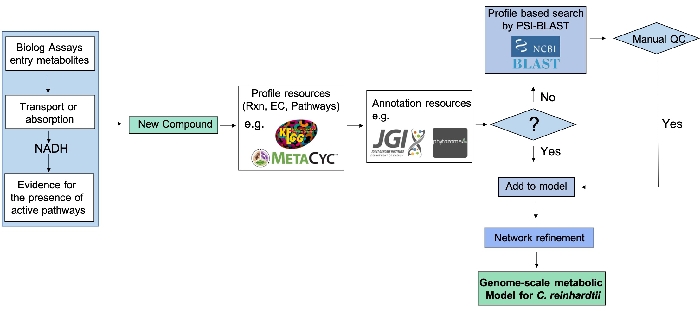
図2PMデータを用いたゲノムスケール代謝ネットワーク改良パイプライン PMアッセイで陽性反応を示す新しい化合物の後、その酵素委員会数(EC)、反応、経路は、KEGGおよびMetaCycなどの利用可能なデータベースから同定されます。ゲノム証拠は、その後、利用可能な場合にゲノムおよび注釈リソースから抽出され、遺伝子型と表現型の間のリンクを構成する。直接ゲノム証拠が利用できない場合、タンパク質配列はEC番号から同定され、遺伝子証拠はPSI-Blastを介して同定される。再構築された代謝ネットワークは、新たに同定された化合物に基づいて洗練されますが、関連するデータベースを使用してタンパク質ドメインを照会する品質管理ステップの後にのみ行われます。この図は、Chaiboonchoeらによって以前に出版された作品から変更 されています。12 この図の大きなバージョンを表示するには、ここをクリックしてください。
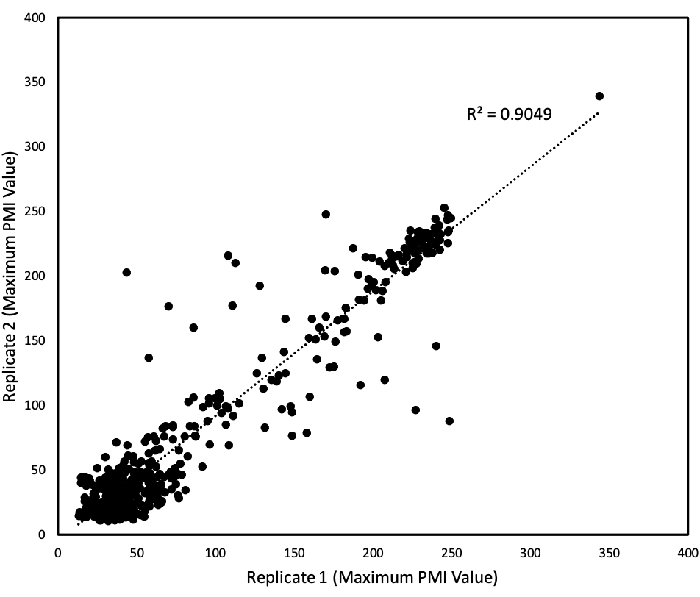
図3:PM試験の再現性 PMI値は168時間にわたって収集され、最大PMI値は2つの反復研究のためにプロットされた。各軸は、各スタディの最大 PMI 値を表します (X 軸は 1 つの反復スタディ、y 軸は別です)。再現された値は各軸から等距離です。各ポイントは、1 つの最大値を表します。線形回帰は、スプレッドシートソフトウェアによって行われ、結果の決定係数(R2)が示されている。この図は、Chaiboonchoeらによって以前に出版された作品から変更 されています。12 この図の大きなバージョンを表示するには、ここをクリックしてください。

図4: 代謝物のベン図 ベン図は、PMプレート 、iRC1080代謝モデル、およびガスクロマトグラフィー飛行時間(GC-TOF)実験によって同定された代謝産物を列挙する。各円は、それぞれの研究方法に存在する代謝産物の総数を示します。同時に、重複する領域は、それらのメソッド間で共有される代謝産物の数を表します。 iRC1080代謝モデルには、合計1,068個のユニークな代謝産物が含まれています。GC-TOFは合計77個の代謝産物32を同定し、PMプレートを使用して同定された代謝産物は合計662個ある。この図は、チャイブーンチョーらによる以前に出版された作品から である。 12この図の大きなバージョンを表示するには、ここをクリックしてください。
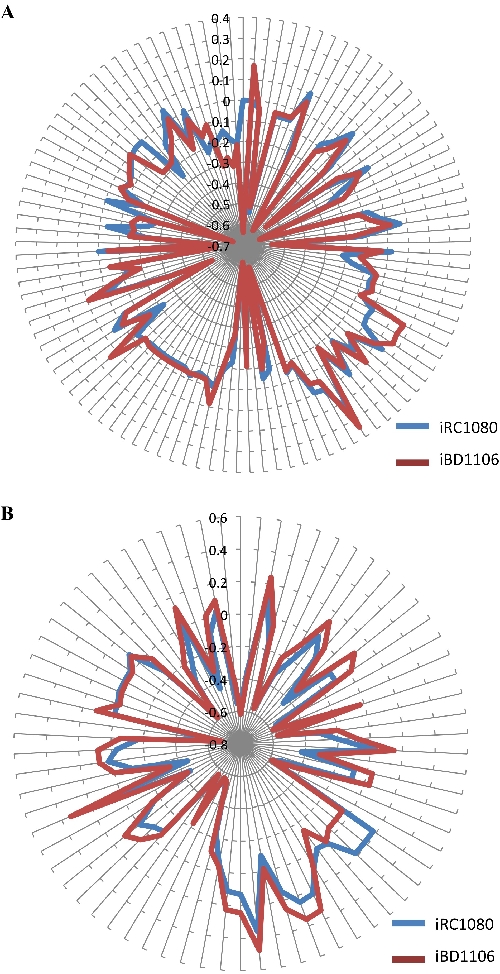
図5:バイオマスの最大化のための異なる条件下でのiRC1080およびiBD1106における代謝産物の影の価格。「レーダープロット」の各円はシャドウ価格の値に対応し、プロットの中心から伸びる各線は代謝産物を示します。(A)光の成長条件下でのiRC1080およびiBD1106の影の価格と代謝挙動;(B)、暗い成長状態下でのiRC1080およびiBD1106の異なる代謝挙動。この図は、チャイブーンチョーらによる以前に出版された作品からである。12この図の大きなバージョンを表示するには、ここをクリックしてください。
| 生物学ケミカル | EC* | 遺伝子注釈 | PSIブラスト |
| システアミン-S-リン酸 | 3.1.3.1 | JLM_1629261,2,3,4 | |
| テトラチオネート | 1.8.2.2 | 重要でない E 値 | |
| 1.8.5.2 | 重要でない E 値 | ||
| D-アラニン | 1.4.1.1 | XP_001700222.1 | |
| 1.5.1.22 | 失敗した手動 QC | ||
| 2.1.2.7 | 重要でない E 値 | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 2.3.2.10 | 重要でない E 値 | ||
| 2.3.2.14 | 重要でない E 値 | ||
| 2.3.2.16 | 重要でない E 値 | ||
| 2.3.2.17 | 重要でない E 値 | ||
| 2.3.2.18 | 重要でない E 値 | ||
| 2.6.1.21 | 失敗した手動 QC | ||
| 3.4.13.22 | XP_001698572.1, XP_001693532.1, XP_001701890.1, XP_001700930.1 | ||
| 3.4.16.4 | Chlre2_kg.scaffold_ 140000391,2,3 | ||
| 3.4.17.8 | 失敗した手動 QC | ||
| 3.4.17.13 | 重要でない E 値 | ||
| 3.4.17.14 | 重要でない E 値 | ||
| 4.5.1.2 | 重要でない E 値 | ||
| 6.1.1.13 | 失敗した手動 QC | ||
| 6.1.2.1 | 失敗した手動 QC | ||
| 6.3.2.4 | au.g14655_t11、2、3 | ||
| 6.3.2.10 | 失敗した手動 QC | ||
| 6.3.2.16 | 重要でない E 値 | ||
| 6.3.2.35 | 重要でない E 値 | ||
| D-アスパラギン | 1.4.5.1 | 重要でない E 値 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 3.1.1.96 | 重要でない E 値 | ||
| 2.3.1.36 | 重要でない E 値 | ||
| 1.4.99.1 | XP_001692123.1 | ||
| 3.5.1.77 | e_gwW.1.243.11,2 | ||
| 3.5.1.81 | 重要でない E 値 | ||
| 5.1.1.10 | 失敗した手動 QC | ||
| D-アスパラギン酸 | 6.3.1.12 | 重要でない E 値 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-グルタミン酸 | 1.4.3.7 | 重要でない E 値 | |
| 1.4.3.3 | 重要でない E 値 | ||
| D-リジン | 5.4.3.4 | 重要でない E 値 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 6.3.2.37 | 失敗した手動 QC | ||
| D-セリン | 2.7.11.8 | 重要でない E 値 | |
| 2.7.11.17 | Cre12.g486350.t1.31,2,3,4 | ||
| 3.4.21.78 | 失敗した手動 QC | ||
| 3.4.21.104 | 失敗した手動 QC | ||
| 4.3.1.18 | g6244.t14 | 失敗した手動 QC | |
| 6.3.2.35 | 重要でない E 値 | ||
| 6.3.3.5 | 重要でない E 値 | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-ヴァリン | 1.21.3.1 | 失敗した手動 QC | |
| 6.3.2.26 | 失敗した手動 QC | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| L-パイログルタミン酸 | |||
| チオリン酸 | |||
| ジチオリン酸 | |||
| エチルアミン | 6.3.1.6 | ||
| D,L-a-アミノブチル酸 | 2.1.1.49 | 重要でない E 値 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| ジペプチド | 3.4.13.18 | Cre02.g078650.t1.31 | |
| トリペプチド | 3.4.11.4 | Cre16.g675350.t1.31 |
表1:同定された陽性基質利用代謝産物(C、P、S、N)のリストは、i RC1080代謝モデルに存在しない。 ※遺伝子が同定されなかった場合は反応は含まれません。 1植物ゾームバージョン10.0.2 (http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii)。 2JGI バージョン 4 35. 3アウグストゥスバージョン510. 4ケッグ(http://www.genome.jp/kegg/kegg1.html)。 5JGI バージョン 3.136. この表は、チャイブーンチョーらによって以前に出版された作品からです。12
| モデル | 反応 | 代謝 産物 | 遺伝子 |
| iRC1080 | 2,191 | 1,706 | 1,086 |
| iBD1106 | 2,445 | 1,959 | 1,106 |
表2:iRC1080とiBD1106の内容. この表は、チャイブーンチョーらによって以前に出版された作品からです。12
| 反応のカテゴリまたはクラス | 反応数 |
| アミノ酸 | 20 |
| ジペプチド | 108 |
| トリペプチド | 5 |
| 輸送反応 | 120 |
表3: iBD1106における新たな反応の概要この表は、チャイブーンチョーらによって以前に出版された作品からです。12
| 成長条件 | 代謝 産物 | 名前 | iRC1080 | iBD1106 | |
| 4r5au | 4-(1-D-リビチルアミノ)-5-アミノウラシル | 0 | 0.168 | ||
| 5aprbu | 5-アミノ-6-(5'-リン酸化アミノ)ウラシル | -0.009 | 0.158 | ||
| 光 | pa1819Z18111Z | 1-(9Z)-オクタデセノイル,2-(11Z)-オクタデセノイル-sn-グリセロール3-リン酸 | -0.009 | -0.65 | |
| 暗い | 4abut | 4-アミノブタノエート | 0.18 | -0.05 | |
表4: iRC1080およびiBD1106の重要な影の価格の例。この表は、チャイブーンチョーらによって以前に出版された作品からです。12
ディスカッション
緑色の微細藻類 C.reinhardtiiの代謝表現型は、高スループットPMアッセイプレートおよび未改変PMIを用いてここで説明した。このアッセイは、合計190個の炭素源(PM01およびPM02)、95個の窒素源(PM03)、59リン源、35個の硫黄源(PM04)、ペプチド窒素源(PM06-08)に対して利用された。陽性呼吸は148の栄養素(C源利用のための1つの陽性アッセイ、各S源およびP源利用のための4つの肯定的なアッセイ、およびN源利用のための139陽性アッセイ)に対して観察された。これらの各ソースをテストする関連するPMマイクロプレートに適用する場合、メディアの基質または栄養素(炭素、窒素、リン、または硫黄)成分を定義された培地に添加しないでください。
ここに示す方法は、既存の代謝ネットワークモデルを拡張したり、新しいモデルの再構築を指示するために使用することができる代謝微細藻類の表型を特徴付けるために有効である。また、ほとんどの微細藻類の栄養要件が知られていないので、このプラットフォームはこれらを迅速に定義するために使用することができる。ネルソン ら43は、これらの方法を適用して、微細藻類 クロロイドウム sp.UTEX 3007の成長を支える新しい化合物を同定し、得られた情報を使用して、クラミドマとは異なり、40種類の炭素源を含む種の侵入代謝物を定義した。
微細藻類のプロファイリングのためのPMの大きな制限の1つは、PMIがインキュベーションチャンバーに照明を持たないことであり、微細藻類は不均栄養代謝を行うことができる必要がある。光の欠如は、代謝フラックスを計算するために光を組み込むモデルの解釈に影響を与える可能性があります。協調機能を持つ遺伝子ペアは、代謝ネットワークハブを構成するために共進化しており、光合成ネットワークハブと非光合成ネットワークハブの区別を44にすることができる。一般に、光合成ネットワークハブ(モデル内の高度に接続されたノード)は、ヘテロトロピケートモデルから除外されます。ミキソトロピケート種におけるモデリングの異種のモデリングは、光によって駆動される反応を省略し、条件間のエネルギーバランスの違いを考慮する必要があります。従って、クラミドモラス代謝モデリング6,45における光依存性および光非依存代謝のモデリングが標準的な実践である。
トレブキシオフィテスのようないくつかの緑色の微細藻類は、成長のために様々な炭素分子を同化することが知られており、これは地衣類46のメンバーとしての長い進化の歴史から生じたと考えられている。クラミドメナスのようなクロロファイトは、成長に酢酸を使用することができるが、非常に長鎖多価不飽和脂肪酸(VLC-PUFAs)を商業的に生産する可能性で知られる褐海の微細藻類チソクリシスルテアは、酢酸を使用することはできないが、成長にグリセロールを使用することができる47。100 g l-1以上の乾電池重量のバイオマス濃度は、供給バッチモード48で有機炭素源を最適化してクロレラで達成されています。また、クロレリャヴルガリスへの糖の添加は、CO2の隔離を高めることができ、したがって、光合成成長49中に付加的な利益を提供する。ほとんどの異栄養性微細藻類もミキソトロフィカルに成長することができるが、クロロファイトクロモクロリスゾフィンギエンシスは、糖50を添加すると光合成を遮断することが示されている。
分裂バチラリオフィタに属する珪藻類は、植物プランクトンの主要なグループである。ほとんどの珪藻は光自己対光的にしか成長できないが、それらのいくつかは、ミキソトロフィックまたは異様51を培養することができる。例えば、グリセロールは、モデル種であるフェオダクチラムトリコルヌトゥタム52を含む一部の珪藻類においてCO2が存在しない場合の光の成長を支えることを発見した。また、ニッツキアリニアのようないくつかの底生珪藻は、暗い53の炭水化物に成長することができます。PMアッセイは、細胞が不均一に成長できるように適切な有機炭素源を補うことによって、糖尿病および他の藻類群にPMアッセイを拡張する可能性があり、かつ、必要最小限の光供給を提供する義務的な自己栄養性微細藻類にもミキソトロフィー戦略を使用する可能性がある。
データの再現性を評価するために、全てのプレートに対して重複アッセイを行うことを強く推奨します。アッセイは、負のコントロールとそれぞれのブランクウェルからの減算後に、吸光度(PMI値)が正の場合にのみ正であると考えられる。この説明は、試験化合物の存在下で、色素の培地との不生物反応の反映である。
開示事項
著者らは開示するものは何もない。
謝辞
この研究に対する主要な支援は、ニューヨーク大学アブダビ研究所助成金(73 71210 CGSB9)とNYUアブダビ教員研究基金(AD060)の下でタムキーンが資金を提供したNYUADゲノミクス・システム生物学センター(CGSB)によって提供されました。W.F.は浙江大学の百人タレントプログラムによってさらにサポートされました。私たちは、ビデオを記録する上で助けを求めてAshish Jaiswalに感謝します。代謝表現型データを生成してくれたホンカイに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Ampicillin | VWR | 97062-796 | |
| Biolog assay plates [ PM01-08] | Biolog, Hayward, CA, USA | ||
| Biolog Omnilog Instrument | Biolog, Hayward, CA, USA | ||
| Chlamydomonas reinhardtii strain CC-503 | Chlamydomonas Resource Center at the University of Minnesota, USA. | Regents of the University of Minnesota | |
| Kanamycin | VWR | 0408-EU-10G | |
| Tetrazolium Violet Dye “D” | Biolog, Hayward, CA, USA | ||
| Timentin | GlaxoSmithKline Australia Pty Ltd | 42010012-2 |
参考文献
- Oberhardt, M. A., Palsson, B. Ø, Papin, J. A. J. M. Applications of genome-scale metabolic reconstructions. Molecular Systems Biology. 5 (1), 320(2009).
- Schmidt, B. J., Lin-Schmidt, X., Chamberlin, A., Salehi-Ashtiani, K., Papin, J. A. Metabolic systems analysis to advance algal biotechnology. Biotechnology Journal. 5 (7), 660-670 (2010).
- Koskimaki, J. E., Blazier, A. S., Clarens, A. F., Papin, J. A. J. I. B. Computational models of algae metabolism for industrial applications. Industrial Biotechnology. 9 (4), 185-195 (2013).
- Koussa, J., Chaiboonchoe, A., Salehi-Ashtiani, K. J. B. Computational approaches for microalgal biofuel optimization: a review. BioMed Research. 2014, 649453(2014).
- Nelson, D. R., et al. Large-scale genome sequencing reveals the driving forces of viruses in microalgal evolution. Cell Host & Microbe. 29 (2), 250-266 (2021).
- Shene, C., Asenjo, J. A., Chisti, Y. Metabolic modelling and simulation of the light and dark metabolism of Chlamydomonas reinhardtii. The Plant Journal. 96 (5), 1076-1088 (2018).
- Tibocha-Bonilla, J. D., Zuñiga, C., Godoy-Silva, R. D., Zengler, K. Advances in metabolic modeling of oleaginous microalgae. Biotechnology for Biofuels. 11 (1), 241(2018).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318 (5848), 245-250 (2007).
- May, P., Christian, J. -O., Kempa, S., Walther, D. J. B. G. ChlamyCyc: an integrative systems biology database and web-portal for Chlamydomonas reinhardtii. BMC Genomics. 10 (1), 209(2009).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), (2011).
- de Oliveira Dal'Molin, C. G., Quek, L. -E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM - a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. BMC Genomics. 12 (5), (2011).
- Chaiboonchoe, A., et al. Microalgal metabolic network model refinement through high-throughput functional metabolic profiling. Frontiers in Bioengineering and Biotechnology. 2, 68(2014).
- Kanehisa, M., et al. Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Research. 42 (1), 199-205 (2014).
- Zuñiga, C., et al. Genome-scale metabolic model for the green alga Chlorella vulgaris UTEX 395 accurately predicts phenotypes under autotrophic, heterotrophic, and mixotrophic growth conditions. Plant Physiology. 172 (1), 589-602 (2016).
- Bochner, B. R. New technologies to assess genotype-phenotype relationships. Nature Reviews Genetics. 4 (4), 309-314 (2003).
- Bochner, B. R. Global phenotypic characterization of bacteria. FEMS Microbiology Reviews. 33 (1), 191-205 (2009).
- Bochner, B. R., Gadzinski, P., Panomitros, E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 11 (7), 1246-1255 (2001).
- Bartell, J. A., Yen, P., Varga, J. J., Goldberg, J. B., Papin, J. A. Comparative metabolic systems analysis of pathogenic Burkholderia. Journal of Bacteriology. 196 (2), 210-226 (2014).
- Gorman, D. S., Levine, R. J. P. otN. A. oS. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. PNAS. 54 (6), 1665-1669 (1965).
- Smith, A. C., Hussey, M. A. Gram stain protocols. American Society for Microbiology. , 1-9 (2005).
- Vaas, L. A. I., et al. opm: an R package for analysing OmniLog phenotype microarray data. Bioinformatics. 29 (14), 1823-1824 (2013).
- Vaas, L. A. I., Sikorski, J., Michael, V., Göker, M., Klenk, H. -P. Visualization and Curve-Parameter Estimation Strategies for Efficient Exploration of Phenotype Microarray Kinetics. PLoS ONE. 7 (4), 34846(2012).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Research. 48 (1), 445-453 (2020).
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Research. , (2020).
- Lopez, D., Casero, D., Cokus, S. J., Merchant, S. S., Pellegrini, M. Algal Functional Annotation Tool: a web-based analysis suite to functionally interpret large gene lists using integrated annotation and expression data. BMC Bioinformatics. 12 (1), 282(2011).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Research. 46, 633-639 (2018).
- Sahoo, S., et al. dEMBF v2. 0: An Updated Database of Enzymes for Microalgal Biofuel Feedstock. Plant and Cell Physiology. 61 (5), 1019-1024 (2020).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 14 (3), 639-702 (2019).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 1, (2019).
- Orth, J. D., Thiele, I., Palsson, B. Ø What is flux balance analysis. Nature Biotechnology. 28 (3), 245(2010).
- Ebrahim, A., Lerman, J. A., Palsson, B. O., Hyduke, D. R. COBRApy: constraints-based reconstruction and analysis for python. BMC Systems Biology. 7 (1), 74(2013).
- Bölling, C., Fiehn, O. Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiology. 139 (4), 1995-2005 (2005).
- Harris, E. H. The Chlamydomonas sourcebook: introduction to Chlamydomonas and its laboratory use. 1, Academic Press. (2009).
- Goodstein, D. M., et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research. 40, 1178-1186 (2012).
- Ghamsari, L., et al. Genome-wide functional annotation and structural verification of metabolic ORFeome of Chlamydomonas reinhardtii. BMC Genomics. 12 (1), 4(2011).
- Manichaikul, A., et al. Metabolic network analysis integrated with transcript verification for sequenced genomes. Nature Methods. 6 (8), 589-592 (2009).
- Apweiler, R., et al. UniProt: the Universal Protein knowledgebase. Nucleic Acids Research. 32, 115-119 (2004).
- Consortium, T. U. Activities at the universal protein resource (UniProt). Nucleic Acids Research. 42 (11), 7486-7486 (2014).
- Horton, P., et al. PSORT: protein localization predictor. Nucleic Acids Research. 35, suppl_2 585-587 (2007).
- Becker, S. A., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox. Nature Protocols. 2 (3), 727-738 (2007).
- Schellenberger, J., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6 (9), 1290(2011).
- Varma, A., Boesch, B. W., Palsson, B. O. Stoichiometric interpretation of Escherichia coli glucose catabolism under various oxygenation rates. Applied and Environmental Microbiology. 59 (8), 2465-2473 (1993).
- Nelson, D. R., et al. The genome and phenome of the green alga Chloroidium sp. UTEX 3007 reveal adaptive traits for desert acclimatization. eLife. , 25783(2017).
- Chaiboonchoe, A., et al. Systems level analysis of the Chlamydomonas reinhardtii metabolic network reveals variability in evolutionary co-conservation. Molecular BioSystems. 12 (8), 2394-2407 (2016).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), 518(2011).
- Rajendran, A., Hu, B. Mycoalgae biofilm: development of a novel platform technology using algae and fungal cultures. Biotechnology for Biofuels. 9 (1), 112(2016).
- Hu, H., et al. Effect of cultivation mode on the production of docosahexaenoic acid by Tisochrysis lutea. AMB Express. 8 (1), 50(2018).
- Bumbak, F., Cook, S., Zachleder, V., Hauser, S., Kovar, K. Best practices in heterotrophic high-cell-density microalgal processes: achievements, potential and possible limitations. Applied Microbiology and Biotechnology. 91 (1), 31(2011).
- Fu, W., et al. Sugar-stimulated CO2 sequestration by the green microalga Chlorella vulgaris. Science of the Total Environment. 654, 275-283 (2019).
- Roth, M. S., et al. Regulation of oxygenic photosynthesis during trophic transitions in the green alga Chromochloris zofingiensis. The Plant Cell. , (2019).
- Villanova, V., et al. Investigating mixotrophic metabolism in the model diatom Phaeodactylum tricornutum. Philosophical Transactions of the Royal Society B: Biological Sciences. 372 (1728), 20160404(2017).
- Cerón-García, M., et al. Mixotrophic growth of Phaeodactylum tricornutum on fructose and glycerol in fed-batch and semi-continuous modes. Bioresource Technology. 147, 569-576 (2013).
- Tuchman, N. C., Schollett, M. A., Rier, S. T., Geddes, P. Advances in Algal Biology: A Commemoration of the Work of Rex Lowe. , Springer. 167-177 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved