Method Article
用于微藻模型改进的高通量代谢分析
摘要
该协议展示了使用表型微阵列(PM)技术平台来定义 衣原体reinhardtii( 一种绿色微藻)的代谢需求,并完善现有的代谢网络模型。
摘要
代谢模型根据生物体可用的基因组注释进行重建,并提供预测工具来研究系统级的代谢过程。基因组规模的代谢模型可能包括间隙以及未经实验验证的反应。由于这些间隙,新分离的微藻物种的重建模型将导致弱点,因为通常有稀疏的生化证据可用于这些分离物的代谢。表型微阵列(PM)技术是一种有效的高通量方法,可响应于各种进入代谢物的功能性确定细胞代谢活动。将高通量表型测定与代谢建模相结合,可以通过提供生化证据来支持和扩展基因组证据,从而快速重建或优化现有的代谢网络模型。这项工作将通过使用绿色微藻模型物种 衣原体reinhardtii 作为示例,展示PM测定在微藻研究中的使用。本研究使用PM获得的超过254种反应的实验证据来扩展和完善基因组规模 的C. reinhardtii 代谢网络模型 iRC1080,大约25%。这里创建的方案可以用作功能分析其他微藻代谢的基础,包括已知的微藻突变体和新分离物。
引言
优化藻类代谢以增强和稳定地生产靶向代谢物需要通过代谢网络的系统级分析来开发复杂的代谢工程策略。代谢网络模型可以指导合理设计,用于优化策略1、2、3、4的快速发展。虽然大约160种微藻物种已经测序5,但据我们所知,只有44种藻类代谢模型可用4,6,7。由于难以获得用于基因组信息实验验证的高通量代谢表型数据,高质量网络模型的重建滞后于藻类基因组测序的快速发展。
C. reinhardtii是一个有吸引力的基于藻类研究的模型系统。该物种可以光自养或异养生长,并已被广泛用作基础和应用研究中的模式生物。它的基因组序列发表于2007年8,随后重建了9、10、11物种的基因组规模代谢模型。C. reinhardtii(i RC1080)的基因组尺度模型由Chang等人重建。 10个基于基因组和文献证据(需要约250个来源)。它有1,706个代谢物,2,190个反应10;然而,除了当时已发表的实验证据之外,无法验证该模型的完整性。
表型微阵列(PMs)技术是一种高通量平台,可以为异养微生物以及组织培养细胞提供代谢分析信息。特别是,它可用于解决微藻中的表型间知识差距,如首次报道的衣原体reinhardtii12以及随后的氯铱13和小球藻14物种。通过研究细胞对数千种代谢物,信号分子,渗透压分解物和效应分子的反应,PM测定可以提供功能代谢分析,并提供对功能,代谢和环境敏感性的见解15,16,17。具体而言,PM测定检测96孔微孔板中的细胞代谢物利用,每个孔中含有不同的营养物质,代谢物或渗透压解质。此外,还可以测定生物活性分子,例如抗生素和激素。由基于四唑的氧化还原染料的NADH还原产生的颜色强度确定,底物的代谢利用根据细胞呼吸15,16,17进行评估。96孔微孔板中的实验可以通过表型微阵列仪(PMI)平台随时间自动监测和确定。设计了20个96孔微孔板,以代表常见的代谢物,以研究细胞表型以利用碳,氮,硫和磷源,以及不同的渗透/离子和pH效应。PM技术已成功用于更新和升级一些现有的微生物基因组尺度代谢模型15、16、17、18。
这里显示的协议和数据基于Chaiboonchoe等人先前发表的工作 。12所提出的工作详细介绍了使用PM测定方法来表征微藻的代谢表型,并扩展 了C. reinhardtii 的现有藻类代谢模型以及指导新代谢模型的重建。
研究方案
1. 表型微阵列实验
- 从美国明尼苏达大学衣原体资源中心获得 C. reinhardtii 菌株CC-503(https://www.chlamycollection.org)。
- 在新鲜的Tris-Acetate-Phosphate(TAP)培养基19中培养细胞,终浓度为400μg/ mL timentin,50μg/ mL氨苄西林和100μg/ mL卡那霉素(以抑制细菌生长)在400μmol光子/m2s下,在25°C下,持续两天至对数中期。
- 在22°C下以2,000×g离心培养物10分钟,并在不干扰沉淀的情况下丢弃上清液。
- 制备含有0.1%四唑紫染料"D"的新鲜TAP培养基。
注意:在此步骤中修改TAP培养基,以根据每个板中测试的代谢物类别排除某些营养素(例如,排除氮源板的氯化铵)。 - 将沉淀重悬于制备的新鲜TAP培养基中(从步骤1.2)至终浓度为1×106 个细胞/ mL。
- 使用化合物阵列测定板(碳源、氮源、磷和硫源板以及肽氮源)。
- 将100μL等分试样的含细胞培养基接种到测定板的每个孔中。
注意:确保重复测定。 - 在酵母提取物/蛋白胨板上条纹细胞并进行革兰氏染色,如Smith 等人。20.测定前后监测细菌污染。
- 将化合物阵列测定板插入酶标仪系统。
- 将所有板在30°C下孵育长达7天,并对酶标仪系统进行编程,以每15分钟读取染料颜色变化。
注意:由于大多数酶标仪在孵育期间不提供连续光源,藻类应该能够进行异养呼吸。
2. 数据分析
- 将微孔板读数器中的原始动力学数据导出为CSV文件,随后将用作R中的Omnilog表型微阵列(OPM)包的输入,将生物信息添加为元数据(例如,菌株名称,生长培养基,温度等)。
- 使用PM动力学数据转换器软件;加载D5E数据文件,并使用PM动力学分析软件中的以下命令行将其转换为OKA文件:
负载|导入(找到OKA文件的文件夹)|填充筛选器|导入|添加所有板|关闭。
出口| 选择读取数据 (动力学), 选择格式 (CSV)(表格标题),然后选择板 (每个板(单个文件))|导出数据|救。 - 为了进行表型微阵列(PM)数据分析,请使用OPM软件包21,22在R软件环境中运行。该软件包、教程和参考文档位于以下位置:http://www.goeker.org/opm/。在 RStudio(R 的图形用户界面)中,使用以下命令安装 opm 包及其依赖项:
源 (http://www.goeker.org/opm/install_opm.R)
库 (opm) - 导航到包含动力学数据的 CSV 文件的目录,并使用read_opm 函数导入数据:
x <- read_opm(".", convert="grp", include=list ("csv:")) - 使用曲线参数估计对动力学数据进行聚合和离散化。
For (i in 1 :length(x)) {
x[[i]]
x[[i]] <- do_disc(x[[i]], cutoff = FALSE)
}
元数据的#Collection
元数据<- collect_template(".")
metadata$Strain <- c("BLANK","CC- 503")
for (I in 1 :length(x)) {x[[i]] <- include_metadata(x[[i]], md = metadata, replace = TRUE)} - 使用 xy_plot 函数将呼吸(或生长)测量值(y轴)映射为已测定的96孔板的时间函数(x轴)。
print (xy_plot(x[[ 1 ]], include ="Strain", theor.max = FALSE)) - 使用 level_plot 函数将数据可视化为热图,以便对动力学数据进行快速比较概述。
level_plot(x, main = list(), colors = opm_opt("color.borders"), panel.headers = metadata$Strain, cex = NULL, strip.fmt = list(), striptext.fmt = list(), legend.sep =" ", space ="Lab", bias = 0.7 , num.colors = 200 L) - 从原始动力学曲线中提取重要的生物学信息,曲线参数,包括滞后相位(λ)、生长速率(μ)、最大细胞呼吸量(A)和曲线下面积(AUC)21。为了识别阳性代谢物,使用阴性对照的A值,它表示染料与培养基的非生物反应性,以及每个微孔板的空白作为背景减法值。提取函数用于获取 A 参数。
opm_opt("curve.param")
param <- extract (x, as.labels = list("Strain")))
- 使用PM动力学数据转换器软件;加载D5E数据文件,并使用PM动力学分析软件中的以下命令行将其转换为OKA文件:
3. 与新代谢物相关的反应和基因的鉴定
- 搜索KEGG(京都基因和基因组百科全书)(http://www.genome.jp/kegg/)和MetaCyc(http://metacyc.org/)以识别酶委员会编号(EC),以使用从化合物阵列中发现的代谢物进行反应23,2423,24。
- 使用已识别的EC编号作为多个可用藻类注释资源的搜索基础,例如联合基因组研究所(JGI),植物组(http://www.phytozome.net)和同行评审的出版物23,25,26,27。
- 如果查询未返回给定EC编号的遗传证据,则从最接近C. reinhardtii的物种开始,确定其他生物体中的相关蛋白质,然后使用NCBI PSI-BLAST服务器(默认设置)进行基于配置文件的搜索,并使用C. reinhardtii中的非冗余蛋白质(nr)(taxid:3055)来识别与反应相关的候选基因12。
- 通过 EMBL-EBI Pfam (http://pfam.xfam.org/search) 或 InterPro (http://www.ebi.ac.uk/interpro/) 蛋白质域预测服务器查询这些 PSI-BLAST 命中,手动整理 E 值为 < 0.05 的 PSI-BLAST 命中,以获得与搜索的 EC 编号的相关性。请注意,后两种扫描是确保鉴定蛋白质的正确酶活性的关键步骤。
4. 模型优化与评估
- 使用最新的 COBRA 工具箱 v.3.028在 MATLAB 中29,30平台,用于执行以下步骤进行模型细化。可以按照中的步骤安装 COBRA 工具箱:https://opencobra.github.io/cobratoolbox/stable/installation.html。或者,请注意,COBRA Toolbox也是在其他开源编程语言中实现的,例如Python(COBRApy)。31),并可在以下位置找到:https://opencobra.github.io/cobrapy/ 。
- 安装 COBRA 工具箱 v.3.0 后,打开 MATLAB 并执行以下命令以初始化工具箱:
initCobraToolbox; - 将已识别的反应及其相关基因添加到代谢模型中,例如iRC1080,使用COBRA工具箱功能addReaction和changeGeneAssociation。导航到包含 i RC1080 模型的目录,从 http://bigg.ucsd.edu/models/iRC1080 并执行以下命令来加载模型、重命名模型并添加新的反应及其相关基因。
Load('iRC 1080 .mat')
模型新 = iRC 1080;
modelNew = addReaction(modelNew, 'D-ALA 2' , ...
{'d-ala[c]' , 'atp[c]' , ...
'D-aladata[c]' , 'adp[c]' , 'pi[c]' , ...
'h[c]' },[- 2 - 1 1 1 1 1 ],false);
modelNEW = changeGeneAssociation(modelNew, ...
'D-ALA 2','au.g 14655 _t 1' ); - 在某些情况下,当代谢物不是细胞内产生的,而是从培养基中吸收时,将新代谢物的转运反应添加到模型中。这些转运反应代表代谢物从细胞外培养基被动扩散到细胞质基质。此外,使用 addExchangeRxn功能添加 相应的人工交换反应,将代谢物输入或输出到细胞外培养基中。
modelNew = addReaction(modelNew, 'CYCPt' , ...
{'cycp[e]','cycp[c]' },[- 1 1 ],true)'
modelNew = addExchangeRxn(modelNew, 'cycp[e]' ,- 1000 ,1000 ); - 测试新结果模型(例如 ,iBD1106)的行为,方法是在光暗条件下使用函数 optimizeCbModel 进行通量平衡分析(FBA),以实现生物量作为目标函数的最大化。对于光生长,将PRISM太阳能光反应的下限和上限设置为646.07(最大速率)。对于暗增长,将所有 PRISM 光反应的界限设置为零。使用先前定义的生物质函数10在黑暗和明亮条件下生长。FBA解决方案将输出两个对应于反应通量(solution.v)和降低成本(solution.w)的向量,以及一个对应于代谢物影子价格(solution.y)的向量。
%模拟光照条件下的生长:
modelNew = changeRxnBounds(modelNew,{ ...
% 'PRISM_solar_litho' , ...
"PRISM_solar_exo" , ...
"PRISM_incandescent_ 60 W' , ...
"PRISM_fluorescent_cool_ 215 W' , ...
"PRISM_metal_halide" , ...
"PRISM_high_pressure_sodium" , ...
"PRISM_growth_room" , ...
"PRISM_white_LED" , ...
"PRISM_red_LED_array_ 653 nm' , ...
"PRISM_red_LED_ 674 nm' , ...
"PRISM_fluorescent_warm_ 18 W' , ...
"PRISM_design_growth" , ...
}, 0, 'b' );
modelNew = changeObjective(modelNew, 'BIOMASS_Chlamy_mixo');
FBAsolutionNew = optimizeCbModel(modelNew, 'max'); - 对 iRC1080 重复步骤 4.1.4,以比较从 iBD1106 获得的 FBA 解决方案与为 iRC1080 获得的解决方案。
- 有一系列COBRA方法可用于比较模型(例如,通量变异性分析,基因缺失研究,稳健性分析,通量分裂预测,FBA,采样等)。详细的教程可以在 https://opencobra.github.io/cobratoolbox/stable/tutorials/index.html 上找到。这里提供了一个例子,其中i RC1080模型与其改进版本iBD1106进行比较,方法是获得每个模型中所考虑的代谢物的影子价格(生物量目标函数对系统变量变化的敏感性)。
获取代谢物的影子价格:
shadowPrices = table(modelNew.mets, ...
modelNew.metNames, FBAsolutionNew.y);
- 安装 COBRA 工具箱 v.3.0 后,打开 MATLAB 并执行以下命令以初始化工具箱:
结果
模型藻类衣原体的表型微阵列筛选
PM测定测试藻类在最小介质中利用各种碳,氮,硫和磷源的能力。在该方法描述中,我们展示了如何使用PM测定来鉴定碳和氮代谢。用酶标仪测量碳和氮利用动力学。使用PMI软件分析数据。所选PM测定板(PM01和PM03)的摘要动力学如图 1所示。"xy 图"显示为 96 孔板测定绘制的随时间变化的呼吸测量值,其中 y 轴和 x 轴分别表示原始测量值和时间值。将数据转换为热图模式,以比较分析动力学数据的组装。
使用PM数据提炼基因组规模代谢网络的管道(图2)说明了高通量PM测定与基因组搜索提供的实验证据的整合可以扩展代谢网络模型。
为了确定从PM01 - 04和PM10板获得的PM数据的再现性,分析了线性回归以绘制来自两个独立重复实验的数据(图3)。图3显示,大多数数据几乎相似,因为它们落在45°线上,只有少数异常值存在,它们的决定系数R2为0.9。该图验证了藻类实验的一致性和可重复性。
鉴定新代谢物
PM测定在七个板中鉴定出662个代谢物;PM01-PM04和PM06-PM08,而气相色谱飞行时间(GC-TOF)已经鉴定出77种代谢物32(图4)。当将这两组与iRC1080中占的1068个代谢物进行比较时,三组之间只有六个代谢物重叠,iRC1080和PM之间有149个代谢物重叠。这一结果表明,代谢分析平台可以成为新代谢信息的重要来源。
醋酸是减去背景信号后在板PM01中检测到的唯一碳源作为支撑碳。这一发现与文献33一致,并显示了PM测定的特异性。PM测定揭示了新的硫,磷和氮源 ,C. reinhardtii 可以利用这些来源进行生长。硫代谢物为硫酸盐、硫代硫酸盐、四亚硫酸盐和DL-脂酰胺。磷源为硫代磷酸盐、二硫代磷酸盐、D-3-磷酸甘油酯和半胱胺-S-磷酸盐。氮源代谢物为L-氨基和D-氨基酸,包括不太常见的氨基酸;L-高丝氨酸,L-焦谷氨酸,甲胺,乙胺,乙醇胺和D,L-α氨基丁酸,以及108个二肽和5个三肽(表 1)。在KEGG和MetaCyc中搜索了所有128种新鉴定的代谢物,以查找其相关反应,EC编号和途径。
新的128种代谢物与49个唯一的EC编号相关联。其中,15个EC使用五个来源与其基因组证据相关联,包括;Phytozome Version 10.0.234 JGI Version 435,AUGUSTUS 5.0, and 5.210 Manichaikul et al.36和 KEGG13。没有基因组证据的代谢物被输入到Universal Protein Resource网站(UniProt,http://www.uniprot.org/)37,38中,在其他生物体中发现了它们的相关序列。通过运行NCBI网站上的位置特异性迭代BLAST(PSI-BLAST,https://blast.ncbi.nlm.nih.gov/Blast.cgi)来鉴定C. reinhardtii中的同源序列,仅考虑产生显着比对的序列(E值<0.005)。
模型细化
与新的128种代谢物及其编码基因相关的反应被添加到 iRC1080模型中,从而扩大了网络。所得模型 iBD1106占2,444个反应,1,959个代谢物和1,106个基因(表 2)。新增的254个反应分别为20个氨基酸氧化反应、108个二肽水解反应、5个三肽水解反应和120个转运反应,由4个基因编码(Cre02.g096350.t1.3,au.g14655_t1,e_gwW.1.243.1,Cre12.g486350.t1.3)。
总共有113种新添加的反应,用于二肽和三肽的水解。二肽和三肽的水解与两个基因有关,一个用于二肽(Cre02.g078650.t1.3),一个用于三肽(Cre16.g675350.t1.3)。
关于磷的来源,添加了与基因JLM_162926相关的将半胱胺-S-磷酸水解成胱胺和磷酸盐的反应。
WoLF PSORT工具39(http://www.genscript.com/psort/wolf_psort.html)和Ghamsari等人报告的结果。应用35个以获得发生新反应的细胞区室的规格。通过分析与新反应相关的蛋白质序列,WoLF PSORT预测细胞质基质是反应的细胞区室。
当生化信息不完整时,生成的代谢模型可能包含间隙。在这种情况下,将使用gapFind,COBRA 命令。它列出了根差距,并允许识别新模型中引入的新差距。代谢模型中不能产生的代谢物被称为根间隙40,41。分析根间隙表明,iRC1080和iBD1106型号都包含相同的91个间隙。这表明添加新的代谢物及其相关反应不会引入任何额外的根间隙。应该注意的是,该协议中使用的表型方法不会关闭根间隙,因为原始的根间隙代谢物缺乏运输或产生机制,这在表型测定中没有得到解决。采用通量平衡分析法测试iBD1106在明暗条件下的代谢行为;(不含醋酸盐)和(含醋酸盐)。该算法最大化目标函数(生物量生长)的生物质前体反应。为了评估每种代谢物对设定目标功能的参与,计算了所有代谢物的"影子价格"。关于代谢物通量变化的客观函数的变化定义了代谢物的影子价格30,42。代谢物是否处于"过量"或"限制"目标函数的指示可以通过影子价格分析来确定,例如生物质生产。负或正影子价格值显示代谢物,这些代谢物在添加时将降低或增加目标函数。影子价格的零值揭示了不会影响目标函数的代谢物。图5中iBD1106和iRC1080之间的影子价格比较表明,对于大多数代谢物,没有观察到显着变化;然而,在光明和黑暗生长条件下,分别在105例和70例中发现了差异。表4包括此类代谢物的示例。
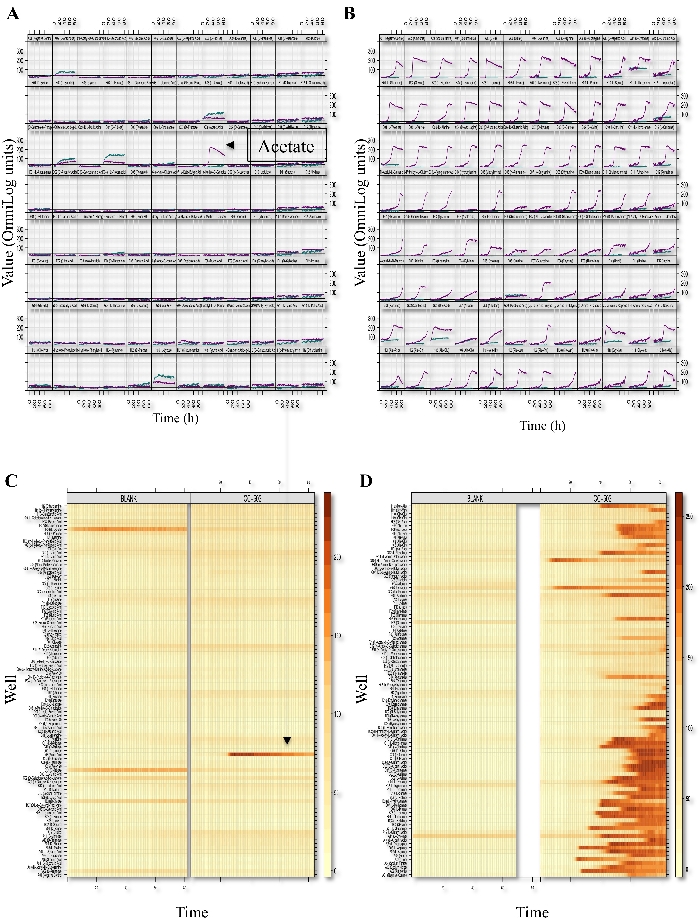
图1:C. reinhardtii的表型微阵列分析。PM01(碳源;A,C)和PM03(氮源;B,D)显示测定板。该图是一个8x12阵列,其中每个细胞代表一个孔板,因此,给定的代谢物或生长环境。在每个细胞或孔表示中,曲线表示通过还原(y轴)作为时间(x轴)的函数的染料转化。来自CC-503和空白孔的PM呼吸曲线显示在每个单元格中,并用颜色表示(蓝绿色代表空白孔,紫色代表CC-503)。水平图将每个呼吸曲线表示为一条细水平线,随时间变化(或保持不变)。热图颜色变化从浅黄色(很少或没有呼吸)到深橙色或棕色(发生了显着的呼吸)。图中显示了C. reinhardtii(CC-503)和空白板使用的代谢物。 这个数字来自Chaiboonchoe等人以前发表的一篇作品。12请点击此处查看此图的放大版本。
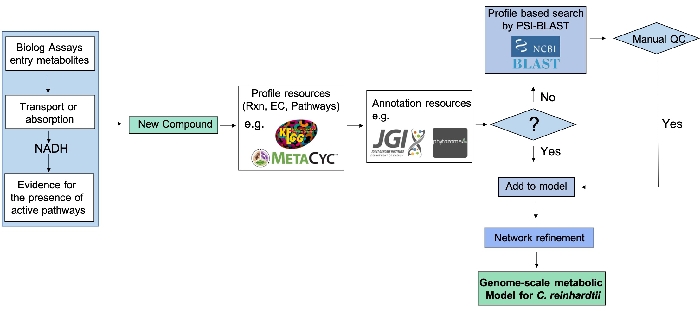
图2:使用PM数据的基因组规模代谢网络细化管道。 在PM测定中新化合物检测呈阳性后,其酶委员会编号(EC),反应和途径从可用数据库(例如KEGG和MetaCyc)中鉴定出来。然后从基因组和注释资源中提取基因组证据(如果可用),并构成基因型和表型之间的联系。当没有直接的基因组证据时,从EC编号中鉴定蛋白质序列,并通过PSI-Blast鉴定遗传证据。然后根据新鉴定的化合物对重建的代谢网络进行精炼,但只有在质量控制步骤之后,需要使用相关数据库查询蛋白质结构域。这个数字是从Chaiboonchoe等人先前发表的工作中修改 而来的。12 请点击此处查看此图的放大版本。
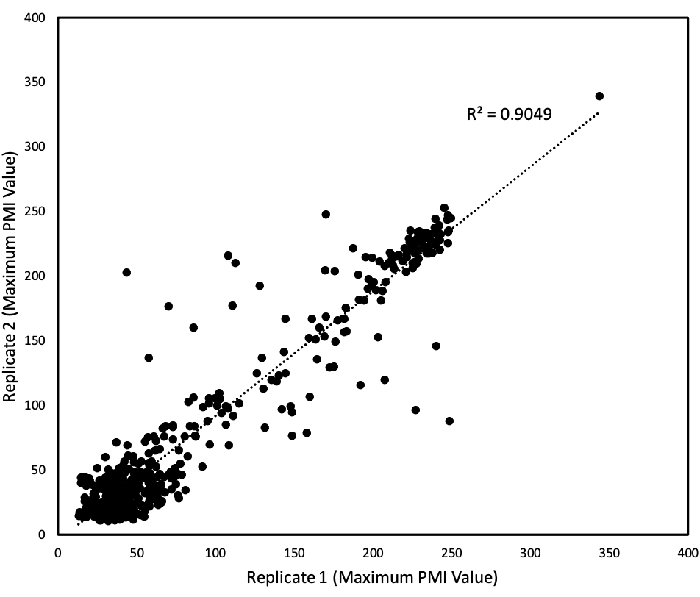
图3:PM测试的再现性。 PMI值是在168小时内收集的,并且为两项重复研究绘制了最大PMI值。每个轴表示每个算例的最大 PMI 值(x 轴表示一个重复算例,y 轴表示另一个重现算例)。再现的值与每个轴的距离相等。每个点表示一个最大值。线性回归由电子表格软件执行,并显示结果决定系数(R2)。这个数字是从Chaiboonchoe等人先前发表的工作中修改 而来的。12 请点击此处查看此图的放大版本。

图4:代谢物维恩图。 维恩图列举了通过PM板 ,iRC1080代谢模型和气相色谱飞行时间(GC-TOF)实验鉴定的代谢物。每个圆圈表示每种研究方法中存在的代谢物总数。同时,重叠区域表示这些方法之间共享的代谢物数量。 iRC1080代谢模型总共包含1,068种独特的代谢物。GC-TOF共鉴定出77个代谢物32个,而使用PM板共鉴定出662个代谢物。这个数字来自Chaiboonchoe等人以前发表的工作 。 12请点击此处查看此图的放大版本。
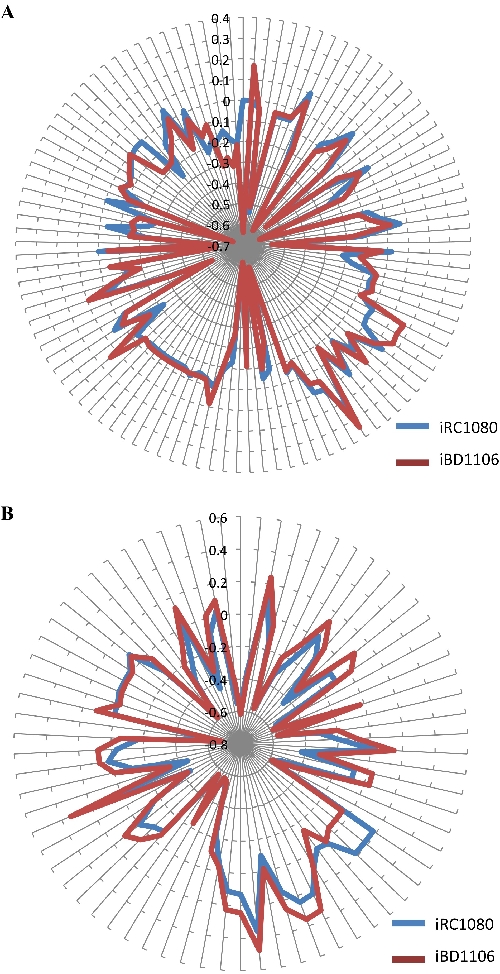
图5:不同条件下iRC1080和iBD1106中代谢物的影子价格,以实现生物质最大化。"雷达图"上的每个圆圈都对应于一个影子价格值,而从图中心延伸的每条线都表示代谢物。(A)光生长条件下iRC1080和iBD1106的影子价格和代谢行为;(B),i RC1080和iBD1106在暗生长条件下的不同代谢行为。 这个数字来自Chaiboonchoe等人以前发表的工作。12请点击此处查看此图的放大版本。
| 生物化学 | 欧共体* | 基因注释 | PSI-爆炸 |
| 半胱胺-S-磷酸盐 | 3.1.3.1 | JLM_162926 1,2,3,4 | |
| 四硫代亚磺酸盐 | 1.8.2.2 | 微不足道的电子价值 | |
| 1.8.5.2 | 微不足道的电子价值 | ||
| D-丙氨酸 | 1.4.1.1 | XP_001700222.1 | |
| 1.5.1.22 | 失败的手动质量控制 | ||
| 2.1.2.7 | 微不足道的电子价值 | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 2.3.2.10 | 微不足道的电子价值 | ||
| 2.3.2.14 | 微不足道的电子价值 | ||
| 2.3.2.16 | 微不足道的电子价值 | ||
| 2.3.2.17 | 微不足道的电子价值 | ||
| 2.3.2.18 | 微不足道的电子价值 | ||
| 2.6.1.21 | 失败的手动质量控制 | ||
| 3.4.13.22 | XP_001698572.1, XP_001693532.1, XP_001701890.1, XP_001700930.1 | ||
| 3.4.16.4 | Chlre2_kg.脚手架_ 14000039 1,2,3 | ||
| 3.4.17.8 | 失败的手动质量控制 | ||
| 3.4.17.13 | 微不足道的电子价值 | ||
| 3.4.17.14 | 微不足道的电子价值 | ||
| 4.5.1.2 | 微不足道的电子价值 | ||
| 6.1.1.13 | 失败的手动质量控制 | ||
| 6.1.2.1 | 失败的手动质量控制 | ||
| 6.3.2.4 | au.g14655_t1 1,2,3 | ||
| 6.3.2.10 | 失败的手动质量控制 | ||
| 6.3.2.16 | 微不足道的电子价值 | ||
| 6.3.2.35 | 微不足道的电子价值 | ||
| D-天冬酰胺 | 1.4.5.1 | 微不足道的电子价值 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 3.1.1.96 | 微不足道的电子价值 | ||
| 2.3.1.36 | 微不足道的电子价值 | ||
| 1.4.99.1 | XP_001692123.1 | ||
| 3.5.1.77 | e_gwW.1.243.11,2 | ||
| 3.5.1.81 | 微不足道的电子价值 | ||
| 5.1.1.10 | 失败的手动质量控制 | ||
| D-天冬氨酸 | 6.3.1.12 | 微不足道的电子价值 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-谷氨酸 | 1.4.3.7 | 微不足道的电子价值 | |
| 1.4.3.3 | 微不足道的电子价值 | ||
| D-赖氨酸 | 5.4.3.4 | 微不足道的电子价值 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 6.3.2.37 | 失败的手动质量控制 | ||
| D-丝氨酸 | 2.7.11.8 | 微不足道的电子价值 | |
| 2.7.11.17 | Cre12.g486350.t1.31,2,3,4 | ||
| 3.4.21.78 | 失败的手动质量控制 | ||
| 3.4.21.104 | 失败的手动质量控制 | ||
| 4.3.1.18 | g6244.t14 | 失败的手动质量控制 | |
| 6.3.2.35 | 微不足道的电子价值 | ||
| 6.3.3.5 | 微不足道的电子价值 | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-缬氨酸 | 1.21.3.1 | 失败的手动质量控制 | |
| 6.3.2.26 | 失败的手动质量控制 | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| L-焦谷氨酸 | |||
| 硫代磷酸盐 | |||
| 二硫代磷酸盐 | |||
| 乙胺 | 6.3.1.6 | ||
| D,L-a-氨基丁酸 | 2.1.1.49 | 微不足道的电子价值 | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 二肽 | 3.4.13.18 | Cre02.g078650.t1.31 | |
| 三肽 | 3.4.11.4 | Cre16.g675350.t1.31 |
表1:i RC1080代谢模型中不存在的已鉴定的阳性底物利用代谢物(C,P,S,N)列表。 *如果未鉴定出基因,则不包括反应。 1 个植物基因组版本 10.0.2 (http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii)。 阿拉伯数字JGI 版本 4 35. 3 个奥古斯都版本 510. 4 个凯格(http://www.genome.jp/kegg/kegg1.html)。 5 个JGI 版本 3.136. 此表来自Chaiboonchoe等人先前发表的工作。11
| 型 | 反应 | 代谢 产物 | 基因 |
| iRC1080 | 2,191 | 1,706 | 1,086 |
| iBD1106 | 2,445 | 1,959 | 1,106 |
表 2: i RC1080 和 iBD1106 的内容。 此表来自Chaiboonchoe等人先前发表的工作。11
| 反应类别或类别 | 反应次数 |
| 氨基酸 | 20 |
| 二肽 | 108 |
| 三肽 | 5 |
| 运输反应 | 120 |
表3:iBD1106中新反应的摘要。此表来自Chaiboonchoe等人先前发表的工作。11
| 生长条件 | 代谢 产物 | 名字 | iRC1080 | iBD1106 | |
| 4r5au | 4-(1-D-核苷氨基)-5-氨基尿嘧啶 | 0 | 0.168 | ||
| 5aprbu | 5-氨基-6-(5'-磷酸化氨基)尿嘧啶 | -0.009 | 0.158 | ||
| 光 | pa1819Z18111Z | 1-(9Z)-十八烯酰基,2-(11Z)-十八烯酰基-sn-甘油3-磷酸酯 | -0.009 | -0.65 | |
| 黑暗 | 4阿布特 | 4-氨基丁酸酯 | 0.18 | -0.05 | |
表 4 :iRC1080 和i BD1106 的重要影子价格示例。此表来自Chaiboonchoe等人先前发表的工作。11
讨论
绿色微藻 C. reinhardtii的代谢表型在这里使用高通量PM测定板和未修饰的PMI进行描述。这些测定共用于190个碳源(PM01和PM02),95个氮源(PM03),59个磷源和35个硫源(PM04),以及肽氮源(PM06-08)。对148种营养素观察到正呼吸(C源利用一次阳性测定,S源和P源利用各4次阳性测定,以及139次N源利用阳性测定)。当应用于测试每种来源的相关PM微孔板时,不应将介质的底物或营养物质(碳,氮,磷或硫)组分添加到定义的介质中。
这里显示的方法对于表征可用于扩展现有代谢网络模型或指导新模型重建的代谢微藻表型是有效的。此外,由于大多数微藻的营养需求尚不清楚,该平台可用于快速定义这些微藻。纳尔逊 等人。43成功地应用了这些方法来鉴定支持微藻 Chlorodiium sp. UTEX 3007生长的新化合物,并使用获得的信息来定义物种进入代谢物,与衣原体不同,它包括40种不同的碳源。
PM用于分析微藻的一个主要限制是PMI在孵育室中没有照明,并且微藻需要能够进行异养代谢。光的缺乏可能会影响对包含光来计算代谢通量的模型的解释。具有协调功能的基因对已经共同进化为构成代谢网络枢纽,并且可以区分光合和非光合网络枢纽44。通常,光合网络集线器(即模型中高度连接的节点)将被排除在异养模型之外。出于实际目的,模拟混合营养物种中的异养性应省略已知由光驱动的反应,并考虑条件之间的能量平衡差异。因此,模拟光依赖性和光无关代谢是衣原体代谢建模的标准做法6,45。
一些绿色微藻,如Trebouxiophytes,已知会同化各种碳分子以进行生长,这被认为是从它们作为地衣成员的漫长进化历史46中产生的。虽然像衣原体这样的叶绿植物可以使用乙酸盐进行生长,但棕色海洋微藻Tisochrysis lutea以其商业生产超长链多不饱和脂肪酸(VLC-PUFAs)的潜力而闻名,不能使用乙酸盐,但可以使用甘油进行生长47。使用小球藻在进料批次模式下优化添加有机碳源,实现了超过100 g l−1干细胞重量的生物质浓度。此外,向小球藻中添加糖可以提高其对CO2的封存,从而在光合生长期间提供添加剂益处49。大多数异养微藻也可以混合营养生长,但叶绿体铬藻已被证明在添加糖50时会关闭光合作用。
硅藻,属于Bacillariophyta部门,是浮游植物的主要群体。虽然大多数硅藻只能光自养生长,但其中一些可以混合养或异养培养51。例如,发现甘油在一些硅藻中没有CO2的情况下支持光照下的生长,包括模型物种 Phaeodactylum tricornutum52。此外,一些底栖硅藻,如 Nitzschia linearis 可以在黑暗中的碳水化合物上生长53。通过补充合适的有机碳源,可以将PM测定扩展到硅藻和其他藻类,以使细胞异养生长,并且混合营养策略也可以潜在地用于专性自养微藻,提供最低限度的光供应。
为了评估数据的再现性,强烈建议对所有板进行重复测定。只有当从阴性对照和相应的空白孔中减去吸光度(PMI值)为正时,才能将测定视为阳性。在测试化合物存在的情况下,这种描述反映了染料与介质的非生物反应。
披露声明
作者没有什么可透露的。
致谢
这项工作的主要支持由NYUAD基因组学和系统生物学中心(CGSB)提供,该中心由Tamkeen在纽约大学阿布扎比研究所资助(73 71210 CGSB9)和纽约大学阿布扎比学院研究基金(AD060)资助下提供。W.F.还得到了浙江大学百人计划的支持。我们感谢Ashish Jaiswal帮助录制视频。我们感谢蔡宏生成代谢表型数据。
材料
| Name | Company | Catalog Number | Comments |
| Ampicillin | VWR | 97062-796 | |
| Biolog assay plates [ PM01-08] | Biolog, Hayward, CA, USA | ||
| Biolog Omnilog Instrument | Biolog, Hayward, CA, USA | ||
| Chlamydomonas reinhardtii strain CC-503 | Chlamydomonas Resource Center at the University of Minnesota, USA. | Regents of the University of Minnesota | |
| Kanamycin | VWR | 0408-EU-10G | |
| Tetrazolium Violet Dye “D” | Biolog, Hayward, CA, USA | ||
| Timentin | GlaxoSmithKline Australia Pty Ltd | 42010012-2 |
参考文献
- Oberhardt, M. A., Palsson, B. Ø, Papin, J. A. J. M. Applications of genome-scale metabolic reconstructions. Molecular Systems Biology. 5 (1), 320(2009).
- Schmidt, B. J., Lin-Schmidt, X., Chamberlin, A., Salehi-Ashtiani, K., Papin, J. A. Metabolic systems analysis to advance algal biotechnology. Biotechnology Journal. 5 (7), 660-670 (2010).
- Koskimaki, J. E., Blazier, A. S., Clarens, A. F., Papin, J. A. J. I. B. Computational models of algae metabolism for industrial applications. Industrial Biotechnology. 9 (4), 185-195 (2013).
- Koussa, J., Chaiboonchoe, A., Salehi-Ashtiani, K. J. B. Computational approaches for microalgal biofuel optimization: a review. BioMed Research. 2014, 649453(2014).
- Nelson, D. R., et al. Large-scale genome sequencing reveals the driving forces of viruses in microalgal evolution. Cell Host & Microbe. 29 (2), 250-266 (2021).
- Shene, C., Asenjo, J. A., Chisti, Y. Metabolic modelling and simulation of the light and dark metabolism of Chlamydomonas reinhardtii. The Plant Journal. 96 (5), 1076-1088 (2018).
- Tibocha-Bonilla, J. D., Zuñiga, C., Godoy-Silva, R. D., Zengler, K. Advances in metabolic modeling of oleaginous microalgae. Biotechnology for Biofuels. 11 (1), 241(2018).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318 (5848), 245-250 (2007).
- May, P., Christian, J. -O., Kempa, S., Walther, D. J. B. G. ChlamyCyc: an integrative systems biology database and web-portal for Chlamydomonas reinhardtii. BMC Genomics. 10 (1), 209(2009).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), (2011).
- de Oliveira Dal'Molin, C. G., Quek, L. -E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM - a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. BMC Genomics. 12 (5), (2011).
- Chaiboonchoe, A., et al. Microalgal metabolic network model refinement through high-throughput functional metabolic profiling. Frontiers in Bioengineering and Biotechnology. 2, 68(2014).
- Kanehisa, M., et al. Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Research. 42 (1), 199-205 (2014).
- Zuñiga, C., et al. Genome-scale metabolic model for the green alga Chlorella vulgaris UTEX 395 accurately predicts phenotypes under autotrophic, heterotrophic, and mixotrophic growth conditions. Plant Physiology. 172 (1), 589-602 (2016).
- Bochner, B. R. New technologies to assess genotype-phenotype relationships. Nature Reviews Genetics. 4 (4), 309-314 (2003).
- Bochner, B. R. Global phenotypic characterization of bacteria. FEMS Microbiology Reviews. 33 (1), 191-205 (2009).
- Bochner, B. R., Gadzinski, P., Panomitros, E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 11 (7), 1246-1255 (2001).
- Bartell, J. A., Yen, P., Varga, J. J., Goldberg, J. B., Papin, J. A. Comparative metabolic systems analysis of pathogenic Burkholderia. Journal of Bacteriology. 196 (2), 210-226 (2014).
- Gorman, D. S., Levine, R. J. P. otN. A. oS. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. PNAS. 54 (6), 1665-1669 (1965).
- Smith, A. C., Hussey, M. A. Gram stain protocols. American Society for Microbiology. , 1-9 (2005).
- Vaas, L. A. I., et al. opm: an R package for analysing OmniLog phenotype microarray data. Bioinformatics. 29 (14), 1823-1824 (2013).
- Vaas, L. A. I., Sikorski, J., Michael, V., Göker, M., Klenk, H. -P. Visualization and Curve-Parameter Estimation Strategies for Efficient Exploration of Phenotype Microarray Kinetics. PLoS ONE. 7 (4), 34846(2012).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Research. 48 (1), 445-453 (2020).
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Research. , (2020).
- Lopez, D., Casero, D., Cokus, S. J., Merchant, S. S., Pellegrini, M. Algal Functional Annotation Tool: a web-based analysis suite to functionally interpret large gene lists using integrated annotation and expression data. BMC Bioinformatics. 12 (1), 282(2011).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Research. 46, 633-639 (2018).
- Sahoo, S., et al. dEMBF v2. 0: An Updated Database of Enzymes for Microalgal Biofuel Feedstock. Plant and Cell Physiology. 61 (5), 1019-1024 (2020).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 14 (3), 639-702 (2019).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 1, (2019).
- Orth, J. D., Thiele, I., Palsson, B. Ø What is flux balance analysis. Nature Biotechnology. 28 (3), 245(2010).
- Ebrahim, A., Lerman, J. A., Palsson, B. O., Hyduke, D. R. COBRApy: constraints-based reconstruction and analysis for python. BMC Systems Biology. 7 (1), 74(2013).
- Bölling, C., Fiehn, O. Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiology. 139 (4), 1995-2005 (2005).
- Harris, E. H. The Chlamydomonas sourcebook: introduction to Chlamydomonas and its laboratory use. 1, Academic Press. (2009).
- Goodstein, D. M., et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research. 40, 1178-1186 (2012).
- Ghamsari, L., et al. Genome-wide functional annotation and structural verification of metabolic ORFeome of Chlamydomonas reinhardtii. BMC Genomics. 12 (1), 4(2011).
- Manichaikul, A., et al. Metabolic network analysis integrated with transcript verification for sequenced genomes. Nature Methods. 6 (8), 589-592 (2009).
- Apweiler, R., et al. UniProt: the Universal Protein knowledgebase. Nucleic Acids Research. 32, 115-119 (2004).
- Consortium, T. U. Activities at the universal protein resource (UniProt). Nucleic Acids Research. 42 (11), 7486-7486 (2014).
- Horton, P., et al. PSORT: protein localization predictor. Nucleic Acids Research. 35, suppl_2 585-587 (2007).
- Becker, S. A., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox. Nature Protocols. 2 (3), 727-738 (2007).
- Schellenberger, J., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6 (9), 1290(2011).
- Varma, A., Boesch, B. W., Palsson, B. O. Stoichiometric interpretation of Escherichia coli glucose catabolism under various oxygenation rates. Applied and Environmental Microbiology. 59 (8), 2465-2473 (1993).
- Nelson, D. R., et al. The genome and phenome of the green alga Chloroidium sp. UTEX 3007 reveal adaptive traits for desert acclimatization. eLife. , 25783(2017).
- Chaiboonchoe, A., et al. Systems level analysis of the Chlamydomonas reinhardtii metabolic network reveals variability in evolutionary co-conservation. Molecular BioSystems. 12 (8), 2394-2407 (2016).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), 518(2011).
- Rajendran, A., Hu, B. Mycoalgae biofilm: development of a novel platform technology using algae and fungal cultures. Biotechnology for Biofuels. 9 (1), 112(2016).
- Hu, H., et al. Effect of cultivation mode on the production of docosahexaenoic acid by Tisochrysis lutea. AMB Express. 8 (1), 50(2018).
- Bumbak, F., Cook, S., Zachleder, V., Hauser, S., Kovar, K. Best practices in heterotrophic high-cell-density microalgal processes: achievements, potential and possible limitations. Applied Microbiology and Biotechnology. 91 (1), 31(2011).
- Fu, W., et al. Sugar-stimulated CO2 sequestration by the green microalga Chlorella vulgaris. Science of the Total Environment. 654, 275-283 (2019).
- Roth, M. S., et al. Regulation of oxygenic photosynthesis during trophic transitions in the green alga Chromochloris zofingiensis. The Plant Cell. , (2019).
- Villanova, V., et al. Investigating mixotrophic metabolism in the model diatom Phaeodactylum tricornutum. Philosophical Transactions of the Royal Society B: Biological Sciences. 372 (1728), 20160404(2017).
- Cerón-García, M., et al. Mixotrophic growth of Phaeodactylum tricornutum on fructose and glycerol in fed-batch and semi-continuous modes. Bioresource Technology. 147, 569-576 (2013).
- Tuchman, N. C., Schollett, M. A., Rier, S. T., Geddes, P. Advances in Algal Biology: A Commemoration of the Work of Rex Lowe. , Springer. 167-177 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。