Method Article
Perfil metabólico de alta produtividade para refinamentos de modelo de microalgas
Neste Artigo
Resumo
Este protocolo demonstra o uso de uma plataforma tecnológica de microarray fenotipo (PM) para definir os requisitos metabólicos de Chlamydomonas reinhardtii, uma microalga verde, e refinar um modelo de rede metabólica existente.
Resumo
Modelos metabólicos são reconstruídos com base na anotação do genoma disponível de um organismo e fornecem ferramentas preditivas para estudar processos metabólicos em nível de sistemas. Modelos metabólicos em escala de genoma podem incluir lacunas, bem como reações que não são verificadas experimentalmente. Modelos reconstruídos de espécies microalgal recém-isoladas resultarão em fraquezas devido a essas lacunas, pois geralmente há evidências bioquímicas esparsas disponíveis para o metabolismo de tais isolados. A tecnologia de microarraia de fenótipo (PM) é um método eficaz e de alto rendimento que determina funcionalmente atividades metabólicas celulares em resposta a uma ampla gama de metabólitos de entrada. A combinação dos ensaios fenotípicos de alto rendimento com a modelagem metabólica pode permitir que modelos de rede metabólica existentes sejam rapidamente reconstruídos ou otimizados, fornecendo evidências bioquímicas para apoiar e expandir evidências genômicas. Este trabalho mostrará como exemplo o uso de ensaios pm para o estudo de microalgas usando a espécie modelo microalgal verde Chlamydomonas reinhardtii. Evidências experimentais para mais de 254 reações obtidas pela PM foram utilizadas neste estudo para expandir e refinar um modelo de rede metabólica C. reinhardtii em escala genoma, iRC1080, em aproximadamente 25%. O protocolo criado aqui pode ser usado como base para traçar o perfil funcional do metabolismo de outras microalgas, incluindo mutantes conhecidos de microalgas e novos isolados.
Introdução
A otimização do metabolismo algal para uma produção aprimorada e estável de metabólitos requer o desenvolvimento de estratégias complexas de engenharia metabólica através de análises em nível de sistemas de redes metabólicas. Modelos de rede metabólica podem orientar os projetos racionais para o rápido desenvolvimento das estratégias de otimização1,2,3,4. Embora aproximadamente 160 espécies microánguas tenham sido sequenciadas5, existem, pelo que sabemos, apenas 44 modelos metabólicos de algas disponíveis4,6,7. Devido à dificuldade em obter dados fenotípicos metabólicos de alto rendimento para validação experimental de informações genômicas, a reconstrução de modelos de rede de alta qualidade está por trás do rápido desenvolvimento do sequenciamento do genoma de algas.
C. reinhardtii é um sistema de modelo atraente para estudos baseados em algas. Esta espécie pode crescer fotoautotrófica ou heterotrofoficamente e tem sido amplamente utilizada como um organismo modelo em pesquisas básicas e aplicadas. Sua sequência de genomas foi publicada em 20078, com modelos metabólicos em escala de genoma posteriormente reconstruídos para a espécie9,10,11. O modelo em escala de genoma para C. reinhardtii (iRC1080) foi reconstruído por Chang et al. 10 com base em evidências genômicas e bibliográficos (implicando ~250 fontes). Tem 1.706 metabólitos com 2.190 reações10; no entanto, a com completeidade do modelo não pôde ser verificada além das evidências experimentais publicadas disponíveis na época.
A tecnologia de microarrays fenotipos (PMs) é uma plataforma de alto rendimento que pode fornecer informações metabólicas de perfil para microrganismos heterotróficos, bem como células de cultura tecidual. Em particular, pode ser usado para abordar a lacuna de conhecimento fenótipo-genótipo em microalgas, como relatado pela primeira vez para chlamydomonas reinhardtii12 e posteriormente para uma espécie de Cloroidium13 e Chlorella14. Ao estudar as respostas celulares a milhares de metabólitos, moléculas de sinalização, osmolitos e moléculas de efeitos, os ensaios da PM podem fornecer perfis metabólicos funcionais e oferecer insights sobre a função, metabolismo e sensibilidade ambiental15,16,17. Especificamente, os ensaios pm detectam a utilização de células metabólicas em microplatas de 96 poços com diferentes nutrientes, metabólitos ou osmólises contidos em cada poço. Além disso, também é possível avaliar moléculas bioativas, como antibióticos e hormônios. Conforme determinado pela intensidade da produção de cores pela redução nadh de um corante de redox à base de tetrazolium, a utilização metabólica dos substratos é avaliada em termos de respiração celular15,16,17. Os experimentos em microplacos de 96 poços podem ser monitorados e determinados automaticamente ao longo do tempo com a plataforma de instrumentos de microarraia de fenótipo (PMI). Vinte microplacas de 96 poços são projetadas para representar os metabólitos comuns para estudar fenótipos celulares para utilizar fontes de carbono, nitrogênio, enxofre e fósforo, juntamente com diferentes efeitos osmóticos/íons e pH. A tecnologia PM tem sido usada com sucesso para atualizar e atualizar uma série de modelos metabólicos em escala de genoma existentes para microrganismos15,16,17,18.
O protocolo e os dados aqui mostrados são baseados em trabalhos publicados anteriormente por Chaiboonchoe et al. 12 O trabalho apresentado detalha o uso do método de ensaio pm para caracterizar os fenótipos metabólicos das microalgas e ampliar um modelo metabólico de algas existente de C. reinhardtii, bem como orientar a reconstrução de novos modelos metabólicos.
Protocolo
1. Experimentos de Microarray do Fenótipo
- Obtenha C. reinhardtii cep CC-503 do Centro de Recursos Chlamydomonas na Universidade de Minnesota, EUA (https://www.chlamycollection.org).
- Cultivar as células em mídia fresca Tris-Acetato-Fosfato (TAP)19 com concentrações finais de 400 μg/mL timentin, 50 μg/mL ampicillin, e 100 μg/mL kanamycin (para inibir o crescimento bacteriano) sob 400 fótons de micromol/m2s, a 25 °C, durante dois dias até a fase de registro médio.
- Gire a cultura a 2.000 x g por 10 min a 22 °C e descarte o supernatante sem perturbar a pelota.
- Prepare as mídias tap frescas contendo 0,1% de corante violeta tetrazolium "D".
NOTA: Modifique a mídia TAP nesta etapa para excluir alguns nutrientes dependendo da categoria metabólito testada em cada placa (por exemplo, exclua o cloreto de amônio para placas de origem de nitrogênio). - Resuspende a pelota em mídia TAP fresca preparada (a partir da etapa 1.2) para uma concentração final de 1 x 106 células/mL.
- Use placas de ensaio de matriz de compostos químicos (fontes de carbono, fontes de nitrogênio, placas de fontes de fósforo e enxofre, e as fontes de nitrogênio peptídeo).
- Inocular uma alíquota de 100 μL de mídia contendo células em cada poço das placas de ensaio.
NOTA: Certifique-se de duplicar os ensaios. - Células de listras em placas de extrato de levedura/peptone e realizar coloração grama, como em Smith et al. 20 antes e depois do ensaio para monitorar a contaminação bacteriana.
- Insira as placas de ensaio do conjunto composto químico no sistema de leitor de microplacas.
- Incubar todas as placas a 30 °C por até 7 dias e programar o sistema de leitor de microplacas para ler a mudança de corante a cada 15 minutos.
NOTA: Como a maioria dos leitores de microplaca não fornece uma fonte de luz contínua durante a incubação, as algas devem ser capazes de realizar respiração heterotrófica.
2. Análise de dados
- Exporte os dados cinéticos brutos do leitor de microplapes como arquivos CSV, que posteriormente serão usados como entrada para o pacote Omnilog Phenotype Microarray (OPM) em R. Adicione as informações biológicas como metadados (por exemplo, designação de cepa, mídia de crescimento, temperatura, etc.).
- Utilizando o software de conversor de dados PM Kinetic; carregar os arquivos de dados D5E e convertê-los em arquivos OKA usando as seguintes linhas de comando no software de análise cinética PM:
| de carga Importação (localizar a pasta dos arquivos OKA) | Filtros de preenchimento | | de importação Adicione todas as placas | Fechar.
| de exportação escolha os dados de leitura (Cinético), escolha o formato (CSV) (Cabeçalho Tabula)e escolha placas (cada placa (arquivos individuais)) | Dados de exportação | Salvar. - Para realizar a análise de dados Phenotype Microarray (PM), utilize o pacote de software OPM21,22 que funciona dentro do ambiente de software R. O pacote, tutorial e documentação de referência estão disponíveis em: http://www.goeker.org/opm/. No RStudio, uma interface de usuário gráfica para R, instale o pacote opm e suas dependências usando os seguintes comandos:
fonte (http://www.goeker.org/opm/install_opm.R)
biblioteca (opm) - Navegue até o diretório que contém os arquivos CSV dos dados cinéticos e importe os dados usando a função read_opm:
x <- read_opm(".", convert="grp", include=list ("csv:")) - Agregar e discretizar os dados cinéticos usando a estimativa de parâmetro de curva.
Para (i em 1:comprimento(x)) {
x[[i]]
x[[i]] <- do_disc(x[[i]], corte = FALSO)
}
#Collection dos metadados
metadados <- collect_template(".")
metadados$Strain <-c("BLANK","CC- 503")
para (I em 1:comprimento(x)) {x[[[i]] <- include_metadata(x[[[i]], md = metadados, substitua = TRUE)} - Use a função xy_plot para mapear as medidas de respiração (ou crescimento) (eixo y) em função do tempo (eixo x) para as placas de 96 poços.
imprimir (xy_plot(x[[1]] incluem ="Tensão", teor.max = FALSO)) - Visualize os dados como um mapa de calor usando a função level_plot para permitir uma visão geral comparativa rápida dos dados cinéticos.
level_plot(x, principal = lista(), cores = opm_opt ("cor.fronteiras"), painel.cabeçalhos = metadados $Strain, cex = NULL, strip.fmt = list(), striptext.fmt = list(), legend.sep =" ", space ="Lab", bias = 0.7 , num.colors = 200 L) - Extrair informações biológicas importantes, os parâmetros da curva, a partir das curvas cinéticas brutas e incluem a fase de lag (λ), a taxa de crescimento (μ), a respiração celular máxima (A) e a área sob a curva (AUC)21. Para identificar metabólitos positivos, utilize os valores A do controle negativo, que representa a reatividade abiótica do corante com o meio, além do vazio de cada placa de microwell como valores de subtração de fundo. A função extrato é usada para obter o parâmetro A.
opm_opt("curve.param")
param <- extrato (x, as.labels = lista("Tensão"))
- Utilizando o software de conversor de dados PM Kinetic; carregar os arquivos de dados D5E e convertê-los em arquivos OKA usando as seguintes linhas de comando no software de análise cinética PM:
3. Identificação de Reações e Genes Associados a Novos Metabólitos
- Procure KEGG (Enciclopédia de Quioto de Genes e Genomas) (http://www.genome.jp/kegg/) e MetaCyc (http://metacyc.org/) para identificar números da Comissão de Enzimas (CES) para reações usando metabólitos encontrados a partir de matrizes de compostos químicos23,2423,24.
- Use os números de CE identificados como base de pesquisa em múltiplos recursos de anotação de algas disponíveis, como Joint Genome Institute (JGI), Phytozome (http://www.phytozome.net) e publicações revisadas por pares23,25,26,27.
- Se uma consulta não retornar nenhuma evidência genética para um determinado número ce, identifique as proteínas associadas relevantes em outros organismos, começando com espécies mais próximas do C. reinhardtii, em seguida, realize uma pesquisa baseada em perfil usando o servidor NCBI PSI-BLAST com configurações padrão e use proteínas não redundantes (nr) em C. reinhardtii (taxid:3055) para identificar genes candidatos associados à reação12.
- Faça a curadoria manual dos hits PSI-BLAST com valores E de < 0,05 para relevância para o número de CE pesquisado através da consulta desses hits BLAST através de servidores de domínio de proteínas EMBL-EBI Pfam (http://pfam.xfam.org/search) ou InterPro (http://www.ebi.ac.uk/interpro/). Observe que os dois últimos exames são passos críticos para garantir a identificação da atividade enzimática correta para a proteína.
4. Refinamento e Avaliação do Modelo
- Use a última caixa de ferramentas COBRA v.3.028no MATLAB29,30plataforma para realizar as seguintes etapas para refinamento de modelo. A caixa de ferramentas COBRA pode ser instalada seguindo as etapas em: https://opencobra.github.io/cobratoolbox/stable/installation.html . Alternativamente, observe que a caixa de ferramentas COBRA também é implementada em outras linguagens de programação de código aberto, como python (COBRApy31), e está disponível em: https://opencobra.github.io/cobrapy/ .
- Após a instalação da caixa de ferramentas COBRA v.3.0, abra o MATLAB e execute o seguinte comando para inicializar a caixa de ferramentas:
initCobraToolbox; - Adicione as reações identificadas com seus genes associados ao modelo metabólico, como o iRC1080, usando as funções da caixa de ferramentas COBRA adicionando Reação e changeGeneAssociation. Navegue até o diretório que contém o modelo iRC1080, baixado de http://bigg.ucsd.edu/models/iRC1080 e execute os seguintes comandos para carregar o modelo, renomeá-lo e adicionar uma nova reação e seu gene associado.
Carga ('iRC 1080 .mat')
modeloNova = iRC 1080;
modelNew = addReaction (modelNew, 'D-ALA 2' , ...
{'d-ala[c]' , 'atp[c]' , ...
'D-aladata[c]' , 'adp[c]' , 'pi[c]' , ...
'h[c]' },[-2 - 11 11 11 ],falso);
modelNEW = changeGeneAssociation (modelNew, ...
'D-ALA 2','au.g 14655 _t 1' ); - Em alguns casos, quando o metabólito não é produzido intracelularmente, mas é retirado do meio, adicione reações de transporte para os novos metabólitos ao modelo. Estas reações de transporte representam difusão passiva de um metabólito do meio extracelular ao citosol. Além disso, adicione uma reação de troca artificial correspondente usando a função addExchangeRxn para inserir ou saída do metabólito no meio extracelular.
modelNew = addReaction (modelNew, 'CYCPt' , ...
{'cycp[e]','cycp[c]' },[- 11 ],true)'
modelNew = addExchangeRxn (modelNew, 'cycp[e]' - 1000 ,1000 ); - Teste o comportamento do novo modelo resultante, por exemplo, iBD1106, realizando análise de equilíbrio de fluxo (FBA) utilizando a função otimizar O Modelo de pressão sob condições claras e escuras para a maximização da biomassa como função objetiva. Para o crescimento da luz, defina as retrações de luz do lito solar PRISM para 646,07 (taxa máxima). Para o crescimento escuro, coloque os limites de todas as reações de luz PRISM a zero. Use a função biomassa definida anteriormente10 para crescimento em condições escuras e claras. A solução FBA produzirá dois vetores correspondentes a fluxos de reação (solution.v) e redução de custo (solution.w), bem como um vetor correspondente aos preços de sombra dos metabólitos (solution.y).
%Simular crescimento sob condição leve:
modelNew = changeRxnBounds (modelNew,{ ...
% 'PRISM_solar_litho', ...
'PRISM_solar_exo', ...
'PRISM_incandescent_ 60 W', ...
'PRISM_fluorescent_cool_ 215 W', ...
'PRISM_metal_halide', ...
'PRISM_high_pressure_sodium', ...
'PRISM_growth_room', ...
'PRISM_white_LED', ...
'PRISM_red_LED_array_ 653 nm', ...
'PRISM_red_LED_ 674 nm', ...
'PRISM_fluorescent_warm_ 18 W', ...
'PRISM_design_growth', ...
}, 0, 'b' );
modelNew = changeObjective (modelNew, 'BIOMASS_Chlamy_mixo');
FBAsolutionNew = optimizeCbModel (modelNew, 'max'); - Repita o passo 4.1.4 para iRC1080 para comparar as soluções FBA obtidas para iBD1106 com as obtidas para iRC1080.
- Há uma gama de métodos COBRA disponíveis que podem ser usados para comparar modelos (por exemplo, análise de variabilidade de fluxo, estudos de exclusão genética, análises de robustez, previsões de divisão de fluxo, FBA, amostragem, etc.). Tutoriais detalhados podem ser encontrados em https://opencobra.github.io/cobratoolbox/stable/tutorials/index.html. Aqui, é fornecido um exemplo onde o modelo iRC1080 é comparado com sua versão refinada, iBD1106, obtendo os preços de sombra (sensibilidade da função objetiva de biomassa às mudanças na variável sistema) dos metabólitos contabilizados em cada modelo.
Obtenha os preços das sombras para os metabólitos:
shadowPrices = tabela(modelNew.mets, ...
modelNew.metNames, FBAsolutionNew.y);
- Após a instalação da caixa de ferramentas COBRA v.3.0, abra o MATLAB e execute o seguinte comando para inicializar a caixa de ferramentas:
Resultados
Triagem de fenótipo microarray do modelo alga Chlamydomonas reinhardtii
Os ensaios da PM testam a capacidade das algas de utilizar várias fontes de carbono, nitrogênio, enxofre e fósforo em um meio mínimo. Nesta descrição dos métodos, demonstramos como os ensaios pm foram usados para identificar o metabolismo de carbono e nitrogênio. Cinéticas de utilização de carbono e nitrogênio foram medidas com um leitor de microplacão. Os dados foram analisados por meio de software PMI. As cinéticas sumárias das placas de ensaio PM selecionadas (PM01 e PM03) são mostradas na Figura 1. As "parcelas de xy" exibem as medidas de respiração ao longo do tempo traçadas para os ensaios das placas de 96 poços, onde o eixo y e o eixo x representam os valores das medições brutas e do tempo, respectivamente. Os dados foram convertidos em um padrão de mapa de calor para analisar comparativamente a montagem dos dados cinéticos.
O pipeline de refinação da rede metabólica em escala genoma usando dados pm (Figura 2) ilustra a integração dos ensaios pm de alto rendimento com evidências experimentais fornecidas por pesquisas genômicas pode expandir um modelo de rede metabólica.
Para determinar a reprodutibilidade dos dados pm obtidos das placas PM01 - 04 e PM10, foi analisada uma regressão linear para traçar os dados de dois experimentos independentes de réplica um contra o outro (Figura 3). A Figura 3 mostra que a maioria dos dados foi quase semelhante à queda na linha 45°, com apenas alguns outliers presentes, e seu coeficiente de determinação R2 foi de 0,9. A consistência e a reprodutibilidade dos experimentos para as algas são verificadas por esta trama.
Identificação de novos metabólitos
O relatório da PM identificou 662 metabólitos em sete placas; PM01-PM04 e PM06-PM08, enquanto o Tempo de Voo (GC-TOF) identificou 77 metabólitos32 (Figura 4). Ao comparar esses dois conjuntos com os metabólitos 1068 contabilizados no iRC1080, apenas seis metabólitos se sobrepuseram entre os três conjuntos, e 149 se sobrepuseram entre o iRC1080 e o PM. Este resultado demonstra que a plataforma de perfil metabólico pode ser uma fonte significativa de novas informações metabólicas.
O ácido acético foi a única fonte de carbono detectada na placa PM01 como um carbono de suporte depois de subtrair o sinal de fundo. Esse achado é consistente com a literatura33 e mostra a especificidade dos ensaios da PM. Os ensaios da PM revelaram novas fontes de enxofre, fósforo e nitrogênio que C. reinhardtii pode utilizar para o crescimento. Os metabólitos de enxofre foram sulfato, tiossulfato, tetrathionato e DL-Lipoamida. As fontes de fósforo foram tiofosfato, dithiofosfato, ácido D-3-fosfo-glicerico, e cisteamina-S-fosfato. Os metabólitos de fonte de nitrogênio foram l-amino e D-aminoácidos, incluindo aminoácidos menos comuns; L-homoserina, L-piroglutámica, metilamina, etilamina, etanolamina e D,L-α-amino-butírico, e 108 Di-peptídeos e cinco Tri-peptídeos(Tabela 1). Todos os 128 metabólitos recém-identificados foram pesquisados em KEGG e MetaCyc por suas reações associadas, números ce e caminhos.
Os novos 128 metabólitos foram associados a 49 números únicos de CE. Destes, 15 CEs foram vinculados às suas evidências genômicas usando cinco fontes, incluindo; Fitozoma Versão 10.0.234 JGI Versão 435, AUGUSTUS 5.0, e 5.210 anotações de Manichaikul et al. 36 e KEGG13. Metabólitos sem evidência genômica foram inseridos no site do Universal Protein Resource (UniProt, http://www.uniprot.org/)37,38 onde suas sequências relacionadas foram encontradas em outros organismos. Sequências homólogos em C. reinhardtii foram identificadas executando A BLAST Iterada Específica da Posição (PSI-BLAST, https://blast.ncbi.nlm.nih.gov/Blast.cgi) do site da NCBI considerando apenas sequências que produziram alinhamentos significativos (valor E <0,005).
Refinamento de modelo
Reações associadas aos novos 128 metabólitos, juntamente com seus genes codificados, foram adicionadas ao modelo iRC1080, expandindo a rede. O modelo resultante iBD1106, responde por 2.444 reações, 1.959 metabólitos e 1.106 genes(Tabela 2). As novas 254 reações adicionadas foram 20 reações de oxidação de aminoácidos, 108 reações de hidrólise di-peptídeo, cinco reações de hidrólise de tripeptídeos e 120 reações de transporte, codificadas por quatro genes (Cre02.g096350.t1.3, au.g14655_t1, e_gwW.1.243.1, Cre12.g486350.t1.3).
Um total de 113 novas reações adicionadas explicam a hidrólise de di-peptídeos e tri-peptídeos. A hidrólise de di-peptídeos e tri-peptídeos estão associadas a dois genes, um para di-peptídeos (Cre02.g078650.t1.3) e um para tri-peptídeos (Cre16.g675350.t1.3).
Quanto às fontes de fósforo, foi adicionada uma reação para hidrólise da cisteamina-S-fosfato em cisteamina e fosfato associado ao gene JLM_162926.
A ferramenta WoLF PSORT39 (http://www.genscript.com/psort/wolf_psort.html) e os resultados relatados por Ghamsari et al. 35 foram aplicados para obter a especificação dos compartimentos celulares onde ocorrem as novas reações. Analisando sequências proteicas associadas às novas reações, o WoLF PSORT previu o citosol como compartimento celular para as reações.
Um modelo metabólico gerado pode conter lacunas quando as informações bioquímicas estão incompletas. Nesses casos, gapFind, um comando COBRA, é usado. Ele lista lacunas radicais e permite a identificação de novas lacunas introdução no novo modelo. Os metabólitos que não podem ser produzidos em um modelo metabólico são referidos como lacunas radiculares40,41. Analisando a lacuna raiz indicou que ambos os modelos iRC1080 e iBD1106 contêm as mesmas 91 lacunas. Isso mostra que a adição dos novos metabólitos e suas reações associadas não introduziu lacunas adicionais. Deve-se notar que o método de fenotipagem utilizado neste protocolo não fecha lacunas radiculares, pois os metabólitos originais da lacuna raiz carecem de mecanismos de transporte ou produção, que não foram abordados nos ensaios de fenotipagem. A análise do balanço de fluxo foi realizada para testar o comportamento metabólico do iBD1106 em condições claras e escuras; (sem acetato) e (com acetato), respectivamente. O algoritmo maximiza as reações precursoras da biomassa para uma função objetiva (crescimento da biomassa). Para avaliar o envolvimento de cada metabólito à função objetiva definida, foram calculados os "preços das sombras" para todos os metabólitos. A mudança na função objetiva relativa às alterações de fluxo do metabólito define o preço sombra de um metabólito30,42. A indicação de se um metabólito está em "excesso" ou está "limitando" a função objetiva pode ser determinada pela análise de preço de sombra, por exemplo, produção de biomassa. Os valores negativos ou positivos dos preços das sombras revelam metabólitos que, ao adição, diminuirão ou aumentarão a função objetiva. Valores zero de preços de sombra revelam metabólitos que não afetarão a função objetiva. A comparação dos preços das sombras entre iBD1106 e iRC1080 na Figura 5 mostra que, para a maioria dos metabólitos, não se observa uma mudança significativa; no entanto, as diferenças são encontradas em 105 e 70 casos em condições claras e de crescimento escuro, respectivamente. A Tabela 4 inclui exemplos desses metabólitos.
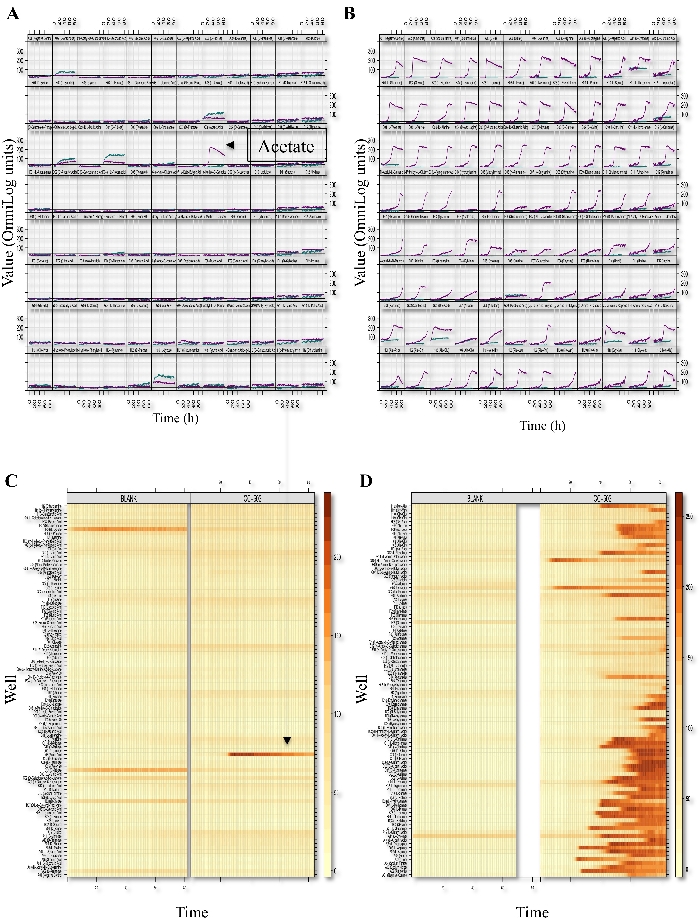
Figura 1: Perfil de microarray fenotípico de C. reinhardtii. Respiração XY-plots e parcelas de nível do PM01 (fontes de carbono; A, C) e PM03 (fontes de nitrogênio; Placas de ensaio B, D) são mostradas. A figura é uma matriz 8x12 onde cada célula representa uma placa de bem e, portanto, um determinado metabólito ou ambiente de crescimento. Dentro de cada célula ou representação de poço, as curvas representam a conversão de corante por redução (eixo y) em função do tempo (eixo x). As curvas de respiração PM do CC-503 e os poços em branco são mostrados em cada célula e são indicadas por cor (a cor teal representa poços em branco e a cor roxa representa CC-503). O gráfico de nível representa cada curva de respiração como uma fina linha horizontal mudando de cor (ou permanecendo inalterada) ao longo do tempo. As mudanças de cor do mapa de calor são de amarelo claro (pouca ou nenhuma respiração ocorreu) para laranja escuro ou marrom (respiração significativa ocorreu). Metabólitos utilizados por C. reinhardtii (CC-503) e as placas em branco são mostrados. Este valor é de um trabalho publicado anteriormente por Chaiboonchoe et al. 12Clique aqui para ver uma versão maior desta figura.
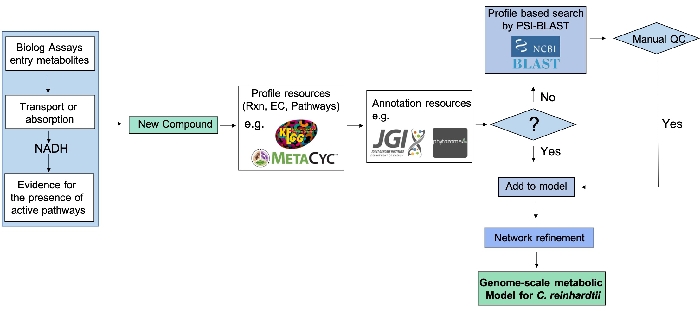
Figura 2: Tubulação de refinamento de rede metabólica em escala de genoma usando dados pm. Após um novo composto testar positivo em um ensaio pm, seu número da Comissão de Enzimas (CE), reação e via são identificados a partir de bancos de dados disponíveis, por exemplo, KEGG e MetaCyc. A evidência genômica é então extraída de recursos genômicos e de anotação quando disponíveis e constitui uma ligação entre genótipo e fenótipo. Quando evidências genômicas diretas não estão disponíveis, a sequência de proteínas é identificada a partir dos números da CE, e evidências genéticas são identificadas via PSI-Blast. A rede metabólica reconstruída é então refinada com base em compostos recém-identificados, mas somente após uma etapa de controle de qualidade que implica consultar os domínios de proteínas usando bancos de dados relevantes. Este valor foi modificado a partir de trabalhos publicados anteriormente por Chaiboonchoe et al. 12Clique aqui para ver uma versão maior desta figura.
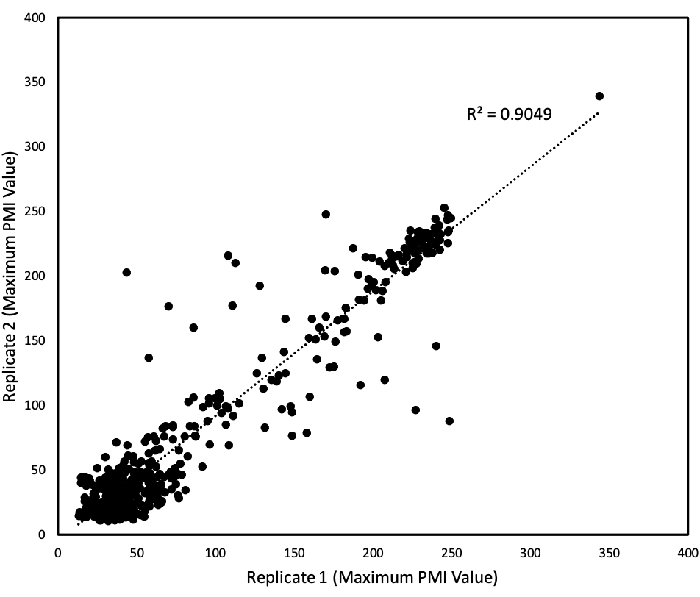
Figura 3: Reprodutibilidade dos testes pm. Os valores do PMI foram coletados em um período de 168 horas, e os valores máximos do PMI foram traçados para dois estudos de replicação. Cada eixo representa os valores máximos do PMI para cada estudo (sendo o eixo x um estudo de réplica e o eixo y outro). Os valores reproduzidos são equidistantes de cada eixo. Cada ponto representa um único valor máximo. A regressão linear foi realizada por um software de planilha, e o coeficiente resultante de determinação (R2) é mostrado. Este valor foi modificado a partir de trabalhos publicados anteriormente por Chaiboonchoe et al. 12Clique aqui para ver uma versão maior desta figura.

Figura 4: Diagrama de venn de metabólitos. O diagrama de Venn enumera metabólitos identificados por placas pm, o modelo metabólico iRC1080 e experimentos de Tempo de Voo de Cromatografia Gas (GC-TOF). Cada círculo indica o número total de metabólitos que existem em cada método de estudo. Ao mesmo tempo, as regiões sobrepostas representam o número de metabólitos compartilhados entre esses métodos. O modelo metabólico iRC1080 contém um total de 1.068 metabólitos únicos. O GC-TOF identificou um total de 77 metabólitos32,enquanto há um total de 662 metabólitos identificados utilizando as placas da PM. Este valor é de trabalho publicado anteriormente por Chaiboonchoe et al. 12Clique aqui para ver uma versão maior desta figura.
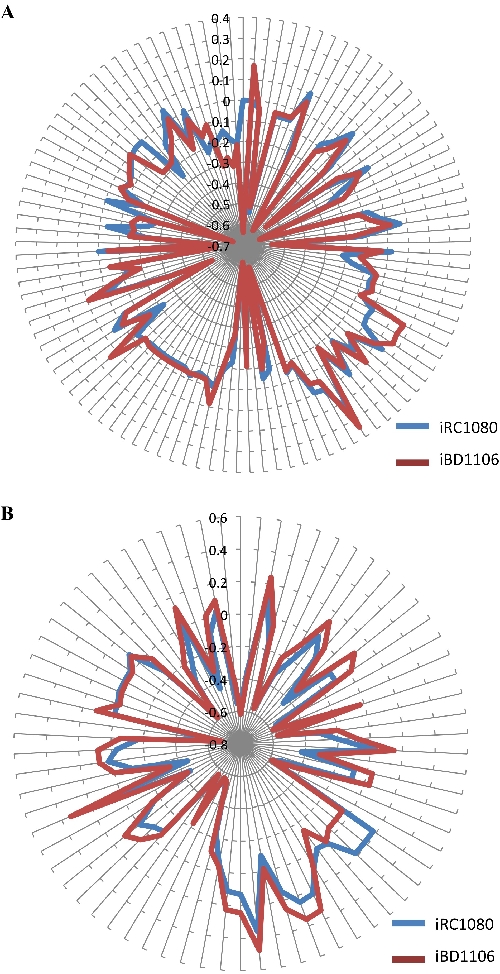
Figura 5: Preços de sombra de metabólitos em iRC1080 e iBD1106 em diferentes condições para maximização da biomassa. Cada círculo nas "parcelas de radar" corresponde a um valor de preço de sombra, enquanto cada linha que se estende do centro de uma parcela indica um metabólito. (A) Preços das sombras e comportamentos metabólicos de iRC1080 e iBD1106 sob uma condição de crescimento leve; (B), diferentes comportamentos metabólicos de iRC1080 e iBD1106 sob uma condição de crescimento escuro. Este valor é de trabalho publicado anteriormente por Chaiboonchoe et al. 12Clique aqui para ver uma versão maior desta figura.
| Química biológica | CE* | Anotação de Genes | PSI-BLAST |
| Cisteamina-Fosfato | 3.1.3.1 | JLM_1629261,2,3,4 | |
| Tetrathionato | 1.8.2.2 | insignificante valor E | |
| 1.8.5.2 | insignificante valor E | ||
| D-Alanina | 1.4.1.1 | XP_001700222.1 | |
| 1.5.1.22 | QC manual falhou | ||
| 2.1.2.7 | insignificante valor E | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 2.3.2.10 | insignificante valor E | ||
| 2.3.2.14 | insignificante valor E | ||
| 2.3.2.16 | insignificante valor E | ||
| 2.3.2.17 | insignificante valor E | ||
| 2.3.2.18 | insignificante valor E | ||
| 2.6.1.21 | QC manual falhou | ||
| 3.4.13.22 | XP_001698572,1, XP_001693532,1, XP_001701890,1, XP_001700930,1 | ||
| 3.4.16.4 | Chlre2_kg.scaffold_ 140000391,2,3 | ||
| 3.4.17.8 | QC manual falhou | ||
| 3.4.17.13 | insignificante valor E | ||
| 3.4.17.14 | insignificante valor E | ||
| 4.5.1.2 | insignificante valor E | ||
| 6.1.1.13 | QC manual falhou | ||
| 6.1.2.1 | QC manual falhou | ||
| 6.3.2.4 | au.g14655_t11,2,3 | ||
| 6.3.2.10 | QC manual falhou | ||
| 6.3.2.16 | insignificante valor E | ||
| 6.3.2.35 | insignificante valor E | ||
| D-Asparagine | 1.4.5.1 | insignificante valor E | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 3.1.1.96 | insignificante valor E | ||
| 2.3.1.36 | insignificante valor E | ||
| 1.4.99.1 | XP_001692123.1 | ||
| 3.5.1.77 | e_gwW.1.243.11,2 | ||
| 3.5.1.81 | insignificante valor E | ||
| 5.1.1.10 | QC manual falhou | ||
| Ácido D-Aspartic | 6.3.1.12 | insignificante valor E | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Ácido D-Glutamico | 1.4.3.7 | insignificante valor E | |
| 1.4.3.3 | insignificante valor E | ||
| D-Lysine | 5.4.3.4 | insignificante valor E | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 6.3.2.37 | QC manual falhou | ||
| D-Serine | 2.7.11.8 | insignificante valor E | |
| 2.7.11.17 | Cre12.g486350.t1.31,2,3,4 | ||
| 3.4.21.78 | QC manual falhou | ||
| 3.4.21.104 | QC manual falhou | ||
| 4.3.1.18 | g6244.t14 | QC manual falhou | |
| 6.3.2.35 | insignificante valor E | ||
| 6.3.3.5 | insignificante valor E | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-Valine | 1.21.3.1 | QC manual falhou | |
| 6.3.2.26 | QC manual falhou | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Ácido L-Pyroglutamic | |||
| Tiofosfato | |||
| Dithiofosfato | |||
| Etilamina | 6.3.1.6 | ||
| D,L-a-Amino-Butyric Acid | 2.1.1.49 | insignificante valor E | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Di-peptídeo | 3.4.13.18 | Cre02.g078650.t1.31 | |
| Tri-peptídeo | 3.4.11.4 | Cre16.g675350.t1.31 |
Tabela 1: Lista de metabólitos de utilização de substrato positivo identificados (C, P, S, N) não presentes no modelo metabólico iRC1080. *A reação não foi incluída se nenhum gene foi identificado. 1 Fitozoma versão 10.0.2 (http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii). algarismo JGI versão 4 35. 3 Augustus versão 510. 4 KEGG (http://www.genome.jp/kegg/kegg1.html). 5 Versão JGI 3.136. Esta tabela é de trabalhos publicados anteriormente por Chaiboonchoe et al. 12
| Modelo | Reações | Metabolitos | Genes |
| iRC1080 | 2,191 | 1,706 | 1,086 |
| iBD1106 | 2,445 | 1,959 | 1,106 |
Tabela 2: Conteúdo de iRC1080 e iBD1106. Esta tabela é de trabalhos publicados anteriormente por Chaiboonchoe et al. 12
| Categoria ou Classe de reações | Número de reações |
| Aminoácidos | 20 |
| Dipeptides | 108 |
| Tripeptídeos | 5 |
| Reação de transporte | 120 |
Tabela 3: Resumo das novas reações no iBD1106. Esta tabela é de trabalhos publicados anteriormente por Chaiboonchoe et al. 12
| Condição de crescimento | Metabólito | Nome | iRC1080 | iBD1106 | |
| 4r5au | 4-(1-D-Ribitylamino)-5-aminouracil | 0 | 0.168 | ||
| 5aprbu | 5-Amino-6-(5'-phosphoribitylamino)uracil | -0.009 | 0.158 | ||
| Luz | pa1819Z18111Z | 1-(9Z)-octadecenoyl,2-(11Z)-octadecenoyl-sn-gliceol3-fosfato | -0.009 | -0.65 | |
| Escuro | 4abut | 4-aminobutanoato | 0.18 | -0.05 | |
Tabela 4: Exemplo de preços de sombra significativos para iRC1080 e iBD1106. Esta tabela é de trabalhos publicados anteriormente por Chaiboonchoe et al. 12
Discussão
Fenotipagem metabólica da microalga verde, C. reinhardtii,foi descrito aqui usando placas de ensaio pm de alta taxa de rendimento e um PMI não modificado. Os ensaios foram utilizados para um total de 190 fontes de carbono (PM01 e PM02), 95 fontes de nitrogênio (PM03), 59 fontes de fósforo e 35 fontes de enxofre (PM04), além de fontes de nitrogênio peptídeo (PM06-08). A respiração positiva foi observada para 148 nutrientes (um ensaio positivo para utilização da fonte C, quatro ensaios positivos para cada utilização da fonte S e fonte P e 139 ensaios positivos para utilização da fonte N). Os substratos ou nutrientes (carbono, nitrogênio, fósforo ou enxofre) da mídia não devem ser adicionados ao meio definido quando aplicados às microplatas pm relevantes que testam para cada uma dessas fontes.
O método aqui mostrado é eficaz para caracterizar fenótipos de microalgas metabólicas que podem ser usados para estender modelos de rede metabólica existentes ou direcionar a reconstrução de novos modelos. Além disso, como não são conhecidos os requisitos nutricionais da maioria das microalgas, essa plataforma pode ser usada para defini-las rapidamente. Nelson et al. 43 aplicou com sucesso esses métodos para identificar novos compostos que suportam o crescimento da microalgae Cloroidium sp. UTEX 3007 e usou as informações obtidas para definir os metabólitos de entrada da espécie, que, ao contrário das Clamídias, incluem 40 diferentes fontes de carbono.
Uma das principais limitações da PM para o perfil das microalgas é que o PMI não tem iluminação na câmara de incubação, e as microalgas precisam ser capazes de realizar o metabolismo heterotrófico. A ausência de luz poderia afetar a interpretação de modelos que incorporam luz para calcular fluxos metabólicos. Os pares genéticos com funções de coordenação têm co-evoluído para constituir hubs de rede metabólica, e a distinção entre hubs de rede fotossintéticos e não fotossintéticos pode ser feita44. Em geral, os hubs de rede fotossintéticos (ou seja, nódulos altamente conectados no modelo) ficariam de fora dos modelos heterotróficos. Para fins práticos, a modelagem do heterotrofismo em espécies mixotróficas deve omitir reações conhecidas por serem impulsionadas pela luz e explicar as diferenças de equilíbrio energético entre as condições. Assim, modelar o metabolismo dependente da luz e da luz é a prática padrão na modelagem metabólica clamídia6,45.
Algumas microalgas verdes, como trebouxiophytes, são conhecidas por assimilar uma variedade de moléculas de carbono para o crescimento, e acredita-se que isso tenha surgido de sua longa história evolutiva como membros dos líquens46. Enquanto clorofilas como as Clamídias podem usar acetato para crescimento, a microalga marinha marrom Tisochrysis lutea, conhecida por seu potencial de produzir comercialmente ácidos graxos poli-insaturados de cadeia muito longa (VLC-PUFAs), não pode usar acetato, mas pode usar glicerol para crescimento47. A concentração de biomassa de mais de 100 g l-1 de peso celular seco foi alcançada com chlorella com adição otimizada de fontes orgânicas de carbono em um modo de lotealimentado 48. Além disso, a adição de açúcar aos Clorovulgaris pode elevar seu sequestro de CO2, proporcionando assim um benefício aditivo durante o crescimento fotossintético49. A maioria das microalgas heterotróficas também pode crescer mixotropo, mas a clorofila chromoclolo zofingiensis tem sido mostrada para desligar a fotossíntese após a adição de açúcar50.
Diatomas, pertencentes à divisão Bacillariophyta, são um grande grupo de fitoplâncton. Embora a maioria dos diatomas só possa crescer fotoautotropoticamente, alguns deles podem ser cultivados misotroficamente ou heterotroficamente51. Por exemplo, o glicerol foi encontrado para apoiar o crescimento da luz na ausência de CO2 em algumas diatomas, incluindo a espécie modelo Phaeodactylum tricornutum52. Além disso, alguns diatomás benéticos como nitzschia linearis podem crescer em carboidratos no escuro53. É provável que estenda os ensaios pm a diatomas e outros grupos de algas, suplementando fontes de carbono orgânicas adequadas para permitir que as células cresçam heterotroficamente, e uma estratégia de mixotrofia também pode ser potencialmente usada para a microalga autotrófica obrigatória, fornecendo um fornecimento de luz minimamente necessário.
Para avaliar a reprodutibilidade dos dados, é altamente recomendável realizar ensaios duplicados para todas as placas. Um ensaio só pode ser considerado positivo se, após a subtração do controle negativo e dos respectivos poços em branco, a absorção (valor pmi) for positiva. Esta descrição, na presença do composto testado, é um reflexo da reação abiótica do corante com o meio.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O grande apoio para este trabalho foi fornecido pelo NYUAD Center for Genomics and Systems Biology (CGSB), financiado pela Tamkeen sob a bolsa do Instituto de Pesquisa de Abu Dhabi da Universidade de Nova York (73 71210 CGSB9) e nyu Abu Dhabi Faculty Research Funds (AD060). W.F. foi adicionalmente apoiado pelo Programa de Cem Talentos da Universidade de Zhejiang. Agradecemos a Ashish Jaiswal pela ajuda na gravação do vídeo. Agradecemos a Hong Cai por gerar os dados do fenótipo metabólico.
Materiais
| Name | Company | Catalog Number | Comments |
| Ampicillin | VWR | 97062-796 | |
| Biolog assay plates [ PM01-08] | Biolog, Hayward, CA, USA | ||
| Biolog Omnilog Instrument | Biolog, Hayward, CA, USA | ||
| Chlamydomonas reinhardtii strain CC-503 | Chlamydomonas Resource Center at the University of Minnesota, USA. | Regents of the University of Minnesota | |
| Kanamycin | VWR | 0408-EU-10G | |
| Tetrazolium Violet Dye “D” | Biolog, Hayward, CA, USA | ||
| Timentin | GlaxoSmithKline Australia Pty Ltd | 42010012-2 |
Referências
- Oberhardt, M. A., Palsson, B. &. #. 2. 1. 6. ;., Papin, J. A. J. M. Applications of genome-scale metabolic reconstructions. Molecular Systems Biology. 5 (1), 320 (2009).
- Schmidt, B. J., Lin-Schmidt, X., Chamberlin, A., Salehi-Ashtiani, K., Papin, J. A. Metabolic systems analysis to advance algal biotechnology. Biotechnology Journal. 5 (7), 660-670 (2010).
- Koskimaki, J. E., Blazier, A. S., Clarens, A. F., Papin, J. A. J. I. B. Computational models of algae metabolism for industrial applications. Industrial Biotechnology. 9 (4), 185-195 (2013).
- Koussa, J., Chaiboonchoe, A., Salehi-Ashtiani, K. J. B. Computational approaches for microalgal biofuel optimization: a review. BioMed Research. 2014, 649453 (2014).
- Nelson, D. R., et al. Large-scale genome sequencing reveals the driving forces of viruses in microalgal evolution. Cell Host & Microbe. 29 (2), 250-266 (2021).
- Shene, C., Asenjo, J. A., Chisti, Y. Metabolic modelling and simulation of the light and dark metabolism of Chlamydomonas reinhardtii. The Plant Journal. 96 (5), 1076-1088 (2018).
- Tibocha-Bonilla, J. D., Zuñiga, C., Godoy-Silva, R. D., Zengler, K. Advances in metabolic modeling of oleaginous microalgae. Biotechnology for Biofuels. 11 (1), 241 (2018).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318 (5848), 245-250 (2007).
- May, P., Christian, J. -. O., Kempa, S., Walther, D. J. B. G. ChlamyCyc: an integrative systems biology database and web-portal for Chlamydomonas reinhardtii. BMC Genomics. 10 (1), 209 (2009).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), (2011).
- de Oliveira Dal'Molin, C. G., Quek, L. -. E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM - a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. BMC Genomics. 12 (5), (2011).
- Chaiboonchoe, A., et al. Microalgal metabolic network model refinement through high-throughput functional metabolic profiling. Frontiers in Bioengineering and Biotechnology. 2, 68 (2014).
- Kanehisa, M., et al. Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Research. 42 (1), 199-205 (2014).
- Zuñiga, C., et al. Genome-scale metabolic model for the green alga Chlorella vulgaris UTEX 395 accurately predicts phenotypes under autotrophic, heterotrophic, and mixotrophic growth conditions. Plant Physiology. 172 (1), 589-602 (2016).
- Bochner, B. R. New technologies to assess genotype-phenotype relationships. Nature Reviews Genetics. 4 (4), 309-314 (2003).
- Bochner, B. R. Global phenotypic characterization of bacteria. FEMS Microbiology Reviews. 33 (1), 191-205 (2009).
- Bochner, B. R., Gadzinski, P., Panomitros, E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 11 (7), 1246-1255 (2001).
- Bartell, J. A., Yen, P., Varga, J. J., Goldberg, J. B., Papin, J. A. Comparative metabolic systems analysis of pathogenic Burkholderia. Journal of Bacteriology. 196 (2), 210-226 (2014).
- Gorman, D. S., Levine, R. J. P. o. t. N. A. o. S. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. PNAS. 54 (6), 1665-1669 (1965).
- Smith, A. C., Hussey, M. A. Gram stain protocols. American Society for Microbiology. , 1-9 (2005).
- Vaas, L. A. I., et al. opm: an R package for analysing OmniLog phenotype microarray data. Bioinformatics. 29 (14), 1823-1824 (2013).
- Vaas, L. A. I., Sikorski, J., Michael, V., Göker, M., Klenk, H. -. P. Visualization and Curve-Parameter Estimation Strategies for Efficient Exploration of Phenotype Microarray Kinetics. PLoS ONE. 7 (4), 34846 (2012).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Research. 48 (1), 445-453 (2020).
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Research. , (2020).
- Lopez, D., Casero, D., Cokus, S. J., Merchant, S. S., Pellegrini, M. Algal Functional Annotation Tool: a web-based analysis suite to functionally interpret large gene lists using integrated annotation and expression data. BMC Bioinformatics. 12 (1), 282 (2011).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Research. 46, 633-639 (2018).
- Sahoo, S., et al. dEMBF v2. 0: An Updated Database of Enzymes for Microalgal Biofuel Feedstock. Plant and Cell Physiology. 61 (5), 1019-1024 (2020).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 14 (3), 639-702 (2019).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 1, (2019).
- Orth, J. D., Thiele, I., Palsson, B. &. #. 2. 1. 6. ;. What is flux balance analysis. Nature Biotechnology. 28 (3), 245 (2010).
- Ebrahim, A., Lerman, J. A., Palsson, B. O., Hyduke, D. R. COBRApy: constraints-based reconstruction and analysis for python. BMC Systems Biology. 7 (1), 74 (2013).
- Bölling, C., Fiehn, O. Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiology. 139 (4), 1995-2005 (2005).
- Harris, E. H. . The Chlamydomonas sourcebook: introduction to Chlamydomonas and its laboratory use. 1, (2009).
- Goodstein, D. M., et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research. 40, 1178-1186 (2012).
- Ghamsari, L., et al. Genome-wide functional annotation and structural verification of metabolic ORFeome of Chlamydomonas reinhardtii. BMC Genomics. 12 (1), 4 (2011).
- Manichaikul, A., et al. Metabolic network analysis integrated with transcript verification for sequenced genomes. Nature Methods. 6 (8), 589-592 (2009).
- Apweiler, R., et al. UniProt: the Universal Protein knowledgebase. Nucleic Acids Research. 32, 115-119 (2004).
- Consortium, T. U. Activities at the universal protein resource (UniProt). Nucleic Acids Research. 42 (11), 7486-7486 (2014).
- Horton, P., et al. PSORT: protein localization predictor. Nucleic Acids Research. 35, 585-587 (2007).
- Becker, S. A., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox. Nature Protocols. 2 (3), 727-738 (2007).
- Schellenberger, J., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6 (9), 1290 (2011).
- Varma, A., Boesch, B. W., Palsson, B. O. Stoichiometric interpretation of Escherichia coli glucose catabolism under various oxygenation rates. Applied and Environmental Microbiology. 59 (8), 2465-2473 (1993).
- Nelson, D. R., et al. The genome and phenome of the green alga Chloroidium sp. UTEX 3007 reveal adaptive traits for desert acclimatization. eLife. , 25783 (2017).
- Chaiboonchoe, A., et al. Systems level analysis of the Chlamydomonas reinhardtii metabolic network reveals variability in evolutionary co-conservation. Molecular BioSystems. 12 (8), 2394-2407 (2016).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), 518 (2011).
- Rajendran, A., Hu, B. Mycoalgae biofilm: development of a novel platform technology using algae and fungal cultures. Biotechnology for Biofuels. 9 (1), 112 (2016).
- Hu, H., et al. Effect of cultivation mode on the production of docosahexaenoic acid by Tisochrysis lutea. AMB Express. 8 (1), 50 (2018).
- Bumbak, F., Cook, S., Zachleder, V., Hauser, S., Kovar, K. Best practices in heterotrophic high-cell-density microalgal processes: achievements, potential and possible limitations. Applied Microbiology and Biotechnology. 91 (1), 31 (2011).
- Fu, W., et al. Sugar-stimulated CO2 sequestration by the green microalga Chlorella vulgaris. Science of the Total Environment. 654, 275-283 (2019).
- Roth, M. S., et al. Regulation of oxygenic photosynthesis during trophic transitions in the green alga Chromochloris zofingiensis. The Plant Cell. , (2019).
- Villanova, V., et al. Investigating mixotrophic metabolism in the model diatom Phaeodactylum tricornutum. Philosophical Transactions of the Royal Society B: Biological Sciences. 372 (1728), 20160404 (2017).
- Cerón-García, M., et al. Mixotrophic growth of Phaeodactylum tricornutum on fructose and glycerol in fed-batch and semi-continuous modes. Bioresource Technology. 147, 569-576 (2013).
- Tuchman, N. C., Schollett, M. A., Rier, S. T., Geddes, P. . Advances in Algal Biology: A Commemoration of the Work of Rex Lowe. , 167-177 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados