Method Article
都市環境のスワッピング - 環境貯留層からのSARS-CoV-2のサンプリングと検出のためのパイプライン
要約
市民科学プロジェクトは、SARS-CoV-2の環境サンプルを収集するためにサンディエゴの住民を募集するように設計されました。多言語 Web ベースのプラットフォームは、使いやすいモバイル デバイス インターフェイスを使用してデータ送信用に作成されました。ラボ情報管理システムは、リアルタイムの結果追跡により、数千もの地理的に多様なサンプルの収集を促進しました。
要約
2020年の世界的なパンデミックの間に重度の急性呼吸器症候群コロナウイルス2(SARS-CoV-2)のコミュニティ伝送を制御するために、ほとんどの国は、直接ヒトのテスト、顔の覆い、および表面消毒に基づく戦略を実施しました。主な透過経路にはエアロゾルと呼吸液滴が含まれるという仮定の下で、フラミテ中のSARS-CoV-2を検出する取り組みは、有病率が高いと疑われる場所(例えば、病院の病棟、クルーズ船、大量輸送システム)に焦点を当てています。めったに清掃されず、めったに消毒されない都市環境の表面にSARS-CoV-2が存在することを調査するために、350人の市民がより大きなサンディエゴ郡から入隊しました。合計で、これらの市民科学者は4,080サンプルを収集しました。サンプリングキットの配達とピックアップを監視し、サンプルデータを収集するオンラインプラットフォームが開発されました。サンプリングキットは、主にパンデミックストレスの店舗で入手可能な物資から構築されました。サンプルは、再発供給不足にもかかわらずアクセスしやすい試薬を用いて処理した。使用される方法は、環境試料中に一般的に存在する阻害剤に対して高感度かつ耐性であった。提案された実験計画と処理方法は、多様な表面積からサンプルを効果的に収集した多数の市民科学者を巻き込むことに成功しました。ここで説明するワークフローと方法は、公衆衛生上の懸念事項であり、将来のパンデミックに脅威を与える他のウイルスの都市環境を調査することに関連しています。
概要
SARS-CoV-2は、汚染されたエアロゾルや液滴を感染者1、2、3、4と直接接触して吸入することにより、主に伝染すると考えられている。しかし、世界的なCOVID-19パンデミックの初期段階では、SARS-CoV-2の透過制御に向けた取り組みは、表面の消毒、手洗い、および消毒に強く焦点を当てました。2020年末までに、世界保健機関(WHO)5および米国疾病予防管理センター(CDC)6からの送電ガイドラインは、主に感染者と密接に接触する場合(<2m)またはエアロゾル生成医療処置の存在下で、空中送電が危険であると考えました。汚染された表面と接触した後の自己接種またはエアロゾル化されたフミットの吸入は、SARS-CoV-2の伝達経路としてまだ排除されていない。
COVID-19症例は、空中感染が7,8と思われる症例が報告されている。SARS-CoV-2ビリオンは、銅に対して最大8時間、段ボールとステンレス鋼で最大24時間、プラスチック上で最大48時間9回感染する。クルーズ船のキャビンでは、乗客が7を出発した17日後にSARS-CoV-2 RNAが検出されました。病院や大量輸送システムからの空気および表面サンプルは、SARS-CoV-2および他のコロナウイルス8、10、11、12、13、14の陽性をテストした。無症候性および中等度/軽度の症候性COVID-19患者によって処理されるハロウィーンキャンディの外側の包装に関して行われた研究は、ハンドソープによるキャンディーのハンドラーと洗浄による手洗いの組み合わせがSARS-CoV-2 RNAを閾値レベル15以下に減少したと結論付けた。
SARS-CoV-2診断のためのいくつかの方法は、リアルタイム逆転写ポリメラーゼ連鎖反応(RT-qPCR)16、17、逆転写ループ媒介等熱増幅(RT-LAMP)11、18、19、20、21、およびCRISPR-Cas技術18、19、22、23に基づいて公開されている。大部分は、世界的に重要な需要の期間中に不足することが多いRNA抽出キットを必要とし、ウイルス24の環境スクリーニングに使用されたものはほとんどありません。RT-LAMPを用いたSARS-CoV-2 RNAの検出は、RT-qPCRを用いた83%以上の一致であることが実証されています。さらに、RT-LAMPはRT-qPCR15と比較して決定的な結果で25%の減少をもたらした。
RT-LAMPは、RNAテンプレート25からcDNAを合成するために逆転写酵素を使用する単純な技術であり、続いて一定温度でDNAを合成する強い鎖変位活性を有するDNAポリメラーゼ(すなわち、等温増幅)26。ウイルスゲノム検出のより高い特異性は、標的DNAの6または8の領域を認識する4つまたは6つのプライマーを使用することによって達成される。増幅は内部プライマーから開始され、半二重鎖DNA構造をもたらす。その後、リーディングストランドは外プライマーによって増幅されます。これらの増幅は、リバースプライマーに対して繰り返される。いずれかの端の内部および外側のプライマーは、増幅産物26,27においてループを形成する内部逆自己相補部位を有する。等温鎖変位では、非同期DNA合成は、連続重合が反応11、20、28あたりわずか10コピーの信号を増幅する大量の増幅産物を生成する。着色RT-LAMPミックスは弱くバッファリングされ、pHインジケータとしてフェノールレッドを使用しています。ポリメラーゼはヌクレオチドを組み込むので、プロトンを放出し、そして十分な陽子は溶液のpHならびにピンクから黄色11、20、28、29に色を変える。
RT-LAMPは、完全に装備された実験室25 を欠いている末梢医療施設における蚊媒介性疾患の検出と、ヒト免疫不全ウイルス30のような他のRNAウイルスの迅速な検出のために開発された。WHOの定義に従って、流行の流行における最も脆弱な人口は、多くの場合、十分な経済資源と検出を実行するための適切な設備を欠いています(国連グローバル公衆衛生アジェンダ)。現在のSARS-CoV-2パンデミックでは、特に非製造業の国では、医療グレードの綿棒やRNA抽出キット用の試薬などの供給が世界的な需要を満たしていません。提案されたプロトコルは、グアニジニウム・チオシアネート(GITC)ベースの粗RNA抽出を用いて、RNAをコールドチェーンに依存しない方法で効果的に保存し、サンプルからの阻害剤の持続性を有意に低下させた。さらに、GITC-クロロホルム抽出プロトコルは、DNAとタンパク質からのRNAの分離に基づいており、その後にそれぞれの沈殿物が続き、ほとんどの遺伝物質の回収が可能です。これらの利点は、適切にリスクを知らせる措置が講じられた場合、化学物質を取り扱う市民科学者の潜在的な危険を上回る。
提案されたワークフローは、一般的に使用される材料および試薬を使用する。それは基本的な、しばしば田舎、実験室の設定で利用できる装置を要求する。これらの方法は、環境サンプルや抽出キットで処理できないサンプルにしばしば見られる阻害剤に対して安価で高い耐性を有し、高精度サーモサイクラーの必要性を排除します。本研究は、家庭や都市環境の一般的に触れ、めったに感染しない表面上の環境貯水池からのSARS-CoV-2のサンプリングと検出のためのパイプラインを提示する。
プロトコル
カタログ番号、製造メーカー、および対応するコストを含む試薬と供給の詳細なリストについては、 材料表 を参照してください。
1. 都市環境のサンプリング
- 市民科学者のアウトリーチ
- 地元やソーシャルメディアを通じて公開された直接的かつ明確な行動を呼び出して市民科学者を募集します。ソーシャルメディアのコンテンツ間でトピックを接続するために、ソーシャルメディアハンドル(例えば 、#swab4corona)を作成します。
- 関心のある地域の主要言語(例えば、サンディエゴ郡のスペイン語と英語)に堪能な人によって管理されている、研究室のチームと各市民科学者との間の直接のコミュニケーションのための電子メールアカウントを作成します。
- 安全な Web ベースのサンプル管理プラットフォーム (SMP) を構築し、データベース、ラボ情報管理システム (LIMS) として機能し、市民科学者と通信します。
注: SMP は、ユーザーがキットを要求し、サンプル収集プロトコルにアクセスし、サンプル メタデータを送信し、完成したサンプル キットの集荷を要求する一元的な場所を提供します。 - オンラインフォームで指定されたバイオセーフティ関連の質問に答えることによって、個人が環境サンプリングの取り組みに参加するために申請するためのSMP(例えば、https://demo.covidsample.org/)(図1)へのリンクを作成します。
- クラウド コンピューター サービス プロバイダーによって容易に提供される認証アプリケーション プログラミング インターフェイスを使用して、SMP へのセキュアなアクセス。承認されたユーザーに SMP へのアクセス権を付与します。
注: 認証と承認については、OAuth 2.0 プロトコル31 の説明については、資料を参照してください。これは、市民科学者のボランティアのための摩擦のないサインインプロセスを提供します。また、ユーザーは既存のアカウントでサインインできるため、カスタム サインイン ソリューションを作成してユーザー資格情報を管理する必要がなくなり、時間を大幅に節約し、参加を促進できます。最近のレポートによると、選択したクラウド コンピューター サービス プロバイダーで利用可能な無料の電子メール サービスには、約 15 億人の月間アクティブな電子メール ユーザーがいます。参加のためにこのサービスプロバイダーから電子メールアカウントを要求することは、落胆する要因とは見なされません。 - 研究の目的とバイオセーフティの考慮事項を、最初のキットを要求する前にSMPの市民科学者に説明します。クラウド・コンピューター・サービス・プロバイダーが提供する多言語の機械翻訳サービスから利用できる言語でナビゲーションを可能にする多言語プラグインを提供します。
- 英語とスペイン語のサンプリング セクショングラフィックとオーディオビジュアル プロトコルに含めます。
- 各キットに一意の識別子を割り当て、サンプル ID にリンクされたボタンを使用してデータ入力プロセスを効率化するようにユーザー インターフェイスを設計します (図 1A)。
- 配達/ピックアップ ルートを最適化し、到着の正確な推定時間を市民科学者に通知するためにドライバーが使用するモバイル デバイス アプリケーションで配信ルート計画ソフトウェアを使用します。
- LIMS プラットフォームを PHP Web サービススタック上に構築し、商用ホスティングプラットフォームでホストします (推奨されるオペレーティングシステム、Web サーバーソフトウェア、およびデータベースソフトウェアは 、資料一覧に記載されています)。
- 安全な Web ベースのアプリケーション インターフェイスを提供し、LIMS でデータを迅速かつ簡単に管理できるようにラボ担当者を実現します。クラウド コンピューター サービス プロバイダーによって容易に行われたデータ グラフ アプリケーション プログラミング インターフェイスを使用してデータの視覚化を提供します。
- クラウド コンピューター サービス プロバイダーが容易に行う地理空間アプリケーション プログラミング インターフェイスを使用して、地理空間データを視覚化します。SMP を介して LIMS に送信されたデータを保存して、プロジェクト データの集中保存を容易にします。(2) サンプル/データ処理ワークフローの追跡(3)市民科学者へのサンプルキット配布の物流管理。
- 送信されたメタデータをベスト プラクティス (たとえば 、https://demo.covidsample.org/)を使用してセキュリティで保護します。
- サンプルキットID、サンプルID、日付、時刻、全地球測位システム(GPS)座標(サイトの画像から自動的に収集)などの事前読み込み情報は、データタイプのコンプライアンスを可能にし、ユーザーによる誤ったデータまたは欠落データの送信を最小限に抑えます(図1B)。収集の日付と時刻、場所の簡単な説明、サンプリング サイトの画像など、市民の科学者によって手動で迅速に (<1 分) 入力するフィールドを含めます。
- アップロードされたすべてのデータをサニタイズし、データタイプを検証します。たとえば、.jpgファイルを選択するためにユーザーがアップロードした画像データを検証し、サンプルと迅速に関連付けるための Sample ID に名前を変更し、アップロードした画像をユーザーがアクセスできない別の安全な場所に保存します。
- すべてのサンプル (16) が完了したら、キットの配達と集荷を要求するオプションを有効にします。さらに、前のキットのピックアップ時に新しいキットを配信するよう要求するオプションを有効にします(図1A)。
注:非ウェブベースのプラットフォームを好むボランティアや、GPSの位置を開示することを心配している人(例えば、移行状況を懸念するコミュニティのメンバー)のために、キットは合意された会議の場所で配達することができ、ボランティアはデータ収集の書面によるバージョンを記録するように求められます。研究室と各市民科学者との間の通信のために、電話やテキストのために利用可能なプロジェクトのバイリンガルメンバーを持っています。
- コロナのための綿棒
- サンプリング作業に対して疫学的に関連する時間枠を特定します。
- 必要な個人用保護具 (マスク、手袋など)、サンプリング プロトコル、およびバイオセーフティ関連情報を含む、すべてのサンプリング用品を含むキットを構築します (図 2)。各チューブに一意の識別子(サンプル ID)を事前にラベル付けします。
- スワブは、家庭や都市環境でエアロゾル化されたフミットにさらされる表面を滅菌することはめったにありません。
- クロスコンタミネーションを避けるために、公共の場で提供されたマスクと各サンプルの収集のための手袋の新しいペアを着用してください。サンプリングを終了した後、付属の手指消毒剤を使用してください。
- 1cm2ポリエステル吸収性綿棒(例えば、モップパッド)を洗浄剤(例えば、0.5%ドデシル硫酸ナトリウム(SDS))で湿潤し、そのエンベロープを破壊してウイルスを不活性化させ、RNases32の展開を誘導して裸のRNAを安定化させる。
- 10cm2の表面をスワブ。爪楊枝に助けられ、200 μLのグアニジニウム・チオシアネート溶液(GITC)を含む対応する事前標識チューブに各サンプル綿棒を完全に沈めます。彼らは実験室に輸送されるまで4 °Cでチューブを保管してください。サンプルが実験室に到着したら、-80 °Cで保管してください。
注意: GITCは有毒刺激性です。皮膚との接触を避ける。GITCソリューションは、レシピのために、一般的な実験室の化学物質から準備するのは簡単です33,34.ウイルスを不活性化し、RNases34、35、36を変性してRNAを安定化させ、室温でサンプルを安定化させます。しかし、キットには、家庭用冷蔵庫を保管するために使用することなく、サンプルを冷たく保つためのアイスパックが含まれています。
2. SARS-CoV-2 検出
- トータルRNA分離
- 2 mM硫酸銅と3%過酸化水素の溶液で表面、機器、およびピペットを消毒;その後、10%漂白剤、90 mM重炭酸ナトリウム、5%SDS、および2.5%NaOHの溶液が続いた。蒸留水で十分に拭き、その後75%エタノールを加えます。
注: これらのソリューションは、市販のソリューションに代わるソリューションです。 - 氷の上のサンプルを解凍します。中速で2分間の渦サンプル。
- スクリーニングの速度を上げるには、サンプルをプールで処理します。プールが陽性の場合は、各サンプルのRNAを個別に抽出して、陽性サンプル/sを見つけます。各サンプリングキット(合計16個)のサンプルを、8つのサンプルの2つのプールに組み合わせます。
注: プールあたり 8 個のサンプルを用意すると、キットごとに 2 つのプールのみを処理する必要があります。プールが正の場合、個々のサンプルは個々のRT-LAMP分析のために再処理されます。これにより、時間、コスト、試薬を削減できます。 - マイクロ遠心チューブ(総容積400 μL)に8サンプルのそれぞれ50 μLをプールします。残りのサンプルを-80 °Cで保存します。 0.2ボリューム(80μL)のクロロホルム、渦を15秒加え、4°Cで20分間インキュベートします。 遠心分離機13,000×gで20分4°Cで。
- 水性(透明な液体)層を新しいマイクロ遠心チューブに移します。残りのインターフェースとピンクの液体を-80 °Cの冷凍庫に保管してください。これらの画分は、DNAおよびタンパク質33,36を含む。
- 等量のイソプロパノール(〜200 μL)とグリコーゲンコ沈降剤2.6 μL(15 mg mL-1)37を加える。よく混ぜ、-20°Cで少なくとも1時間インキュベートし、続いて4°Cで10分間RNAを沈殿させた。
注: このプロトコルは、1 時間ではなく-20 °C でサンプルを一晩インキュベートすることで、ここで一時停止できます。 - 遠心分離機13,000×gで20分4°Cで。 ペレットを邪魔することなく上清を除去します。ペレットをジエチルピロカーボネート(DEPC)処理水50μLに再懸濁し、RNaseフリー5 M酢酸アンモニウムと2.5容量(250μl)の等容量(50μL)を加え、100%エタノール7,38を加える。
注: アンモニウムイオンは、下流工程38で使用する場合にポリヌクレオチドキナーゼを阻害する。この混合は、溶液38中にデオキシヌクレオシド三リン酸およびオリゴ糖を残しながらRNAを沈殿させる。 - よく混ぜ、-20°Cで少なくとも1時間インキュベートし、続いて4°Cで10分間RNAを沈殿させた。
注: このプロトコルは、1 時間ではなく-20 °C でサンプルを一晩インキュベートすることで、ここで一時停止できます。 - 遠心分離機13,000×gで20分4°Cで。 ペレットを1mLの冷(-20°C)で洗浄し、75%エタノールを作りたてから。遠心分離機 8,000 × gで 5 分 4 °C. ペレットを乱さないようにP10ピペットで上清を取り除く。
- 残りのエタノールがなくなるまで、ペレットを10〜15分間乾燥させます。ペレットをDEPC処理水50μLに再懸濁し、10x DNaseバッファー+1μLのDNase(2単位μL-1)を5μL加え、37°Cで30分間インキュベートします。
- DNase不活性化試薬の0.1ボリューム(5.6 μL)を加え、室温で5分間インキュベートし、毎分穏やかに混ぜます。2分間13,000×gで遠心分離機を、上清を新しいチューブ(〜50μL)に移します。RT-qPCRまたはRT-LAMP反応を調製しながらすぐにチューブを氷の上に置くか、-20°Cの冷凍庫に保管してください。
注:持ち越し汚染を避けるために、アンプリコンフリーの部屋でRNAの隔離を行ってください。
- 2 mM硫酸銅と3%過酸化水素の溶液で表面、機器、およびピペットを消毒;その後、10%漂白剤、90 mM重炭酸ナトリウム、5%SDS、および2.5%NaOHの溶液が続いた。蒸留水で十分に拭き、その後75%エタノールを加えます。
- マルチプレックス逆転写ループ媒介等温増幅(RT-LAMP)
- プライマーのセットごとに20Xプライマーミックス溶液 (表1)を 用意 する(表2)。10%の過剰な体積で室温でRT-LAMP反応ミックス (表3)を 準備し、ピペットロスを考慮します。
注:南極熱ラビシルウラシルDNAグリコシラーゼ(UDG)との比色LAMP 2Xマスターミックスは、以前の反応20、28からのDNA汚染の増幅を防ぎます。 - 渦と混合物をスピンダウン。各反応管にミックスの20 μLを分配する: サンプル、 スパイク、 正の対照、および 陰性対照。チューブ内の反応を室温で10分間インキュベートし、UDGが潜在的な持ち越し汚染に作用できるようにします。
- サンプル反応に5μLのRNAを、スパイク反応に合成SARS-CoV-2 RNAの5μL+2.5μL(450コピー)、陽性対照反応に合成SARS-CoV-2 RNAの2.5 μL(450コピー)、および5μLのH2Oを陰性制御反応に加えます。よく混ぜて、反応をスピンダウンします。氷の上のすべてのRNAを解凍します。
- サーモサイクラー、または水浴を65°Cに加熱しながら、すべての反応が室温で維持されるようにします。UDGは>50°Cで不活性化されます。 サーモサイクラーに反応を入れ(熱蓋を使用)、65°Cで40分間インキュベートし、室温(5分間〜22°C)に達させたり、氷上で1分間冷却したりします。色分けフィーチャー(単純な観測)を使用するか、またはアガロースゲルで製品を実行して結果を分析します。
注: 検出制限(LOD)は反応あたり10コピーですが、コピー数が反応ごとに500コピーに近づくにつれて検出の頻度が増加します(図3A)。測色観測では、負の結果はピンク(pH = 8.8)で示され、正の結果は黄色(pH = 5)で示されることに注意してください(図3B)。着色オプションは、増幅後のRT-LAMPチューブの開口を回避し、作業環境におけるRT-LAMP製品の体積を低減し、汚染を持ち込みます。ゲル電気泳動の場合、0.5%トリ/ボレート/EDTA(TBE)バッファーに1X DNAゲル染色を含む1.5%アガロースゲルを調製します。各ウェルに反応+ 5 μL の 6X 負荷染料をロードします。ゲルを100 Vで60分間動かします。正のサンプルはラダーパターンを示すので分子マーカーは必要ありません(図3C)。
- プライマーのセットごとに20Xプライマーミックス溶液 (表1)を 用意 する(表2)。10%の過剰な体積で室温でRT-LAMP反応ミックス (表3)を 準備し、ピペットロスを考慮します。
結果
SARS-CoV-2の検出のために市民科学者によって収集されたサンプル。8ヶ月間(2020年11月3日から3週目)に、482人の市民がこのプロジェクトへの参加を承認し、そのうち350人(73%)が参加しました。キットを要求しました。合計362キットが納入され(例えば、一部の参加者は複数のキットを要求した)、246キット(70%)が返された(図 4A、B)。これらのキットに含まれる4,080サンプルすべてが処理されました。コレクションサイトは、郡の北沿岸、北中部、中央、南部の各地区と、東部地区にいくつか分布していました(図 4A)。これらの地区は、サンディエゴ郡の人口密度が最も高く、最も文書化されたCOVID-19症例を有する。
市民は、ほとんどのサンプリングされたキットのピックアップを要求しました(すなわち、平均成功率:70.4%)。毎日1~16キットが要求され、0~14キットが研究室に返却されました(図4B)。市民科学者の調査によると、完全なキット(16サンプル)のコレクションは、平均8日間にわたって分布する1〜3時間かかった(図4Aおよび表4)。キットの大部分は完成しており(91.1%)、16個のサンプルチューブの中に綿棒が含まれ、対応するサンプリングデータがLIMSにアップロードされた(図4Aおよび表4)。
GITC-クロロホルム抽出および多重逆転写酵素ループ媒介等温増幅(RT-LAMP)を用いたSARS-CoV-2の検出。この着色RT-LAMPアッセイでは、2組のプライマー11,20,28を用いて、ヌクレオキャプシド(N2)およびエンベロープ(E1)遺伝子を標的とした(表2)。これらのプライマーによって認識される配列は、RT-qPCRによるCOVID-19のヒト診断のためにCDC40および欧州連合(EU)41によって承認されたプライマーおよびプローブと同じ領域にある。これらの結果は、Zhangら28が説明したものを裏付け、反応に60 mMグアニジン塩酸塩を添加すると、多重で実行するとLODが増加する。100%の周波数でのLODは、25 μL反応あたり500コピーであった(図3A、B)。カラーメトリック RT-LAMP では、pH シフトが ~8 から 5.5 (図 3B)のため、正のサンプルがピンク色から黄色に変化しました。反応が低コピー数でオレンジ色に変わったとき、サンプルを1.5%アガロースゲルで実行して陽性であることが確認され、ラダー状のパターンが生じた(図3C)。RT-LAMPはプールされたRNAサンプルのSARS-CoV-2を検出するために使用された。
反応阻害剤による偽陰性を制御するために、各サンプルを、合成SARS-CoV-2の500コピーでスパイクした追加の反応で試験した。ポジティブプールサンプルは、プール内の個々のサンプルのRNAを分離して脱複製し、RT-LAMP反応で実行して陽性サンプルの同一性を決定した。検出結果は LIMS にアップロードされ、一意のサンプル ID と、日付、時刻、GPS 座標、サイト、およびサンプルの画像に関する情報が組み合わせられました。
リアルタイムおよび従来のRT-PCR法:サンプル汚染物質による阻害。 提案された検出パイプラインに適した最良の方法を選択するために、他のRNA増幅方法を、市民科学者のパイロットコホートによって収集された環境サンプルで試験した。これらの各方法の結果の例は、低ウイルスコピー数濃度における環境阻害剤およびバックグラウンド信号ノイズに対するそれらの感受性を描写するために 図5 に示されている。
CDCおよびWHOによって承認された6つのRT-qPCR製剤(材料表)は、環境試料上のウイルスの検出について試験された。プロトコルは、臨床現場でのSARS-CoV-2の検出のためのCDCガイドラインと同様に、製造業者の指示に従って従った。合成SARS-CoV-2 RNA制御の異なる濃度を含む反応は、粗RNA単離後にスワッピング表面サンプルにスパイクされた。すべてのマスターミックスは、陽性対照のLOD濃度で阻害剤に敏感であった(図5A)。
これらのリアルタイム技術の阻害剤をバイパスするために、従来のRT-PCRシステムがテストされました。1ステップRT-PCRシステム(材料表)を用いて、プライマーセットN1、N2、および、CDC(USA)およびECDC(EU)によって承認されたプライマーセットE1を用いてエンベロープ遺伝子をそれぞれ使用して、ヌクレオキャプシド遺伝子を増幅した(表2)。 プロトコルは、臨床現場でのSARS-CoV-2の検出のためのCDCガイドラインと同様に、製造業者の指示に従って従った。このプライマーは、CDCによって設計されたN1とN2を設定し、〜70 bpの製品を生成します。しかし、低コピー数陽性は、結果に誤検出を導入した陰性制御(図5B)の増幅バックグラウンドノイズと区別されませんでした。E1プライマーの積は低コピー数で弱いシグナルを持っていました(図5B),結果に偽陰性を導入.さらに、試験したRT-PCR法は、環境試料中に存在する阻害剤に対しても依然として感受性であった(データは示していない)。
他の方法は、ターゲットシーケンスの非常に少量を検出するために開発されています。これらの方法の1つは、ローリングサークル増幅(RCA)であり、その後、標的配列、RNAまたはDNAの認識、特異的な線形プローブによって、リガーゼがテンプレートを円形化する。プローブとハイブリダイズするように設計されたプライマーを用いて、ストランド変位活性を有するDNAポリメラーゼは、等温反応42においてプローブを増幅する。ターゲットを特定したプローブであり、増幅されるものであり、ターゲットシーケンスではなく、この方法は非常に敏感な43である。Wangら44は、SARS-CoV-1 RNAの直接検出のためのRCAプロトコルを発表した。この方法は、SARS-CoV-2に固有のプライマーを使用するように変更した。残念ながら、非テンプレートコントロール(NTC)では、プローブは、SNP感受性リガーゼを含む多種多様なリガサーを使用する場合でも、RNAテンプレートがない場合に製品を円形化し、生成します。リガーゼがない場合、NTCは線形化されたプローブ(図5C)からの増幅を示さなかった。

図1: モバイルデバイス用のサンプル収集データインターフェイスを備えたWebベースのサンプリングプラットフォーム (A) 多言語プラグインを含むウェブサイトは、研究室と市民科学者の間の相互作用を仲介するために作成されました。このプラットフォームは、サンプルキットの配達/集荷要求およびサンプルデータ提出に使用されました。プラットフォームは、詳細な英語/スペインのグラフィックと視聴覚サンプリングプロトコルが含まれていました。(B) サンプル データのアップロードに使用されるモバイル デバイス ビュー:日付、時刻、GPS 座標、サンプル サイトの説明、およびコレクション サイトの画像。略語: SARS-CoV-2 = 重度の急性呼吸器症候群コロナウイルス 2;GPS = 全地球測位システム この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:サンプルコレクションキット 市民科学者は、2つのアイスパック、GITCソリューションの取り扱いの危険性についてボランティアに知らせるための安全データシート、詳細なサンプリングとマスク着用プロトコルを含むクーラーを受け取りました。 KN95マスク、廃棄物袋、ハンドサニタイザー付きスプレーボトル、0.5%SDS付きスプレーボトル、16組の手袋、16個の爪楊枝と16個のポリエステル綿棒を備えた小さなバッグ、200μLのGITC溶液を含む16個のラベル付きマイクロ遠心チューブ、サンプリングチューブを含む箱、および二次容器の二次容器として使用されるバッグ。略語: GITC = グアニジニウム・チオシアネート;SDS =ドデシル硫酸ナトリウム。 この図の大きなバージョンを表示するには、ここをクリックしてください。
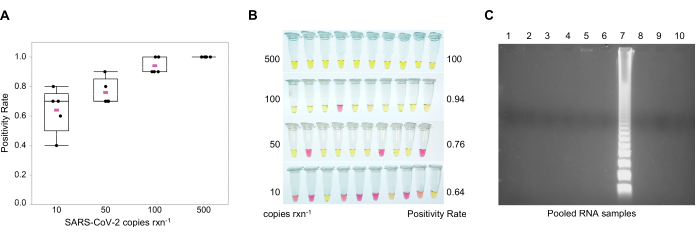
図3:多重化逆転写ループ媒介等熱増幅(RT-LAMP)アッセイ SARS-CoV-2ヌクレオキャプシド(N2)およびエンベロープ(E1)遺伝子のプライマーを用いた多重化反応は、反応中のウイルスの10コピーを検出する。合成SARS-CoV-2 RNAを陽性対照として用いた。(A)SARS-CoV-2の多重色分けRT-LAMPにおける検出の頻度を、反応ごとに異なるゲノムコピー数で行う。5つの平均値はピンクで複製します。(B) 多重色度 RT-LAMP における SARS-CoV-2 の検出限界 (LOD)黄色 = 正 (pH ~5);ピンク = 負 (pH ~8)(C)1.5%アガロースゲル電気泳動における正のSARS-CoV-2 RT-LAMP反応のラダーパターン。略語: SARS-CoV-2 = 重度の急性呼吸器症候群コロナウイルス 2;rxn = 反応。 この図の大きなバージョンを表示するには、ここをクリックしてください。
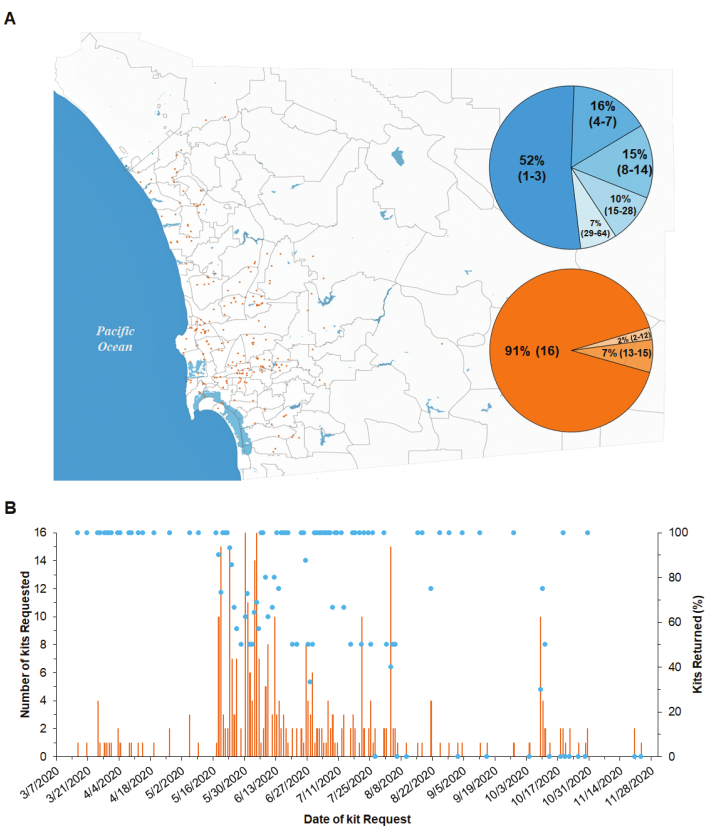
図4:サンディエゴ郡の市民科学者サンプリングキットの位置と要求キットの成功率. (A) オレンジ色のドットは、16 サンプルを含む 1 サンプリング キットの位置を表します。青い円グラフは、市民科学者に納入された時から研究室に戻ったときまで、さまざまな日を要したキットの割合を示しています。括弧内の日数。オレンジ色の円グラフは、合計 16 サンプルの完成サンプル数が異なるキットの割合を示します。サンプルチューブ内の綿棒を含む完成サンプル数と、LIMSにアップロードされた対応するサンプリングデータを括弧で囲んで示したデータ。(B) キットが要求された日付に対する、実験室に返却されたキットの割合 (ドット) と、要求されたキット (棒) の総数。 この図の大きなバージョンを表示するには、ここをクリックしてください。
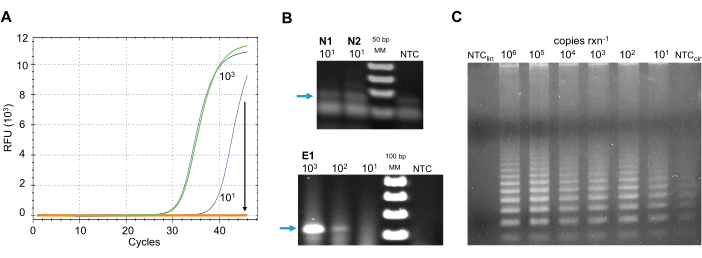
図5:代替SARS-CoV-2 RNA検出法(A) プライマーセットN2を用いたSARS-CoV-2ヌクレオキャプシド(N)遺伝子のRT-qPCR検出。プールされた環境サンプルは、SARS-CoV-2の900(緑)、または9(オレンジ)コピーでスパイクしました。同じコピー番号の正のコントロールを青色で表示します。矢印は、環境試料が存在する場合の低コピー数陽性対照の蛍光検出の減少を示す。(B) SARS-CoV-2の従来のRT-PCR検出。上: プライマーセットN1およびN2を使用したヌクレオキャプシド遺伝子の RT-PCR 産物 。テンプレートなしのコントロールでは、かすかなバックグラウンド信号が観察されます。下:プライマーセットE1を用いたエンベロープ遺伝子のRT-PCR産物。LOD濃度では極めて低い信号が観測される。青い矢印は、期待される陽性生成物を示す:(上)〜70bp及び(下)2%アガロースゲル電気泳動で113bp。(C) SARS-CoV-2 RNA の RCA.円形プローブは、リガーゼの存在下で増幅し、RNAテンプレート(NTCcir)の不在。リニアプローブ(NTClin)を増幅しないリニアプローブとリニアリニアの存在しない場合。略語: SARS-CoV-2 = 重度の急性呼吸器症候群コロナウイルス 2;RFU = 相対蛍光単位;bp = 基本ペア。rxn = 反応;MM = 分子マーカー;RT-PCR = 逆転写ポリメラーゼ連鎖反応;RT-qPCR = リアルタイム定量 RT-PCR;RCA = ローリングサークル増幅;NTC= テンプレートなしのコントロール。LOD = 検出の限界;rxn = 反応。この図の大きなバージョンを表示するには、ここをクリックしてください。
| 入門 | 20X濃度(μM) | 1X濃度(μM) |
| FIP | 32 | 1.6 |
| BIP | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 | 4 | 0.2 |
| ループフ | 8 | 0.4 |
| ループB | 8 | 0.4 |
表1:20X RT-LAMPプライマーミックスの処方。 RT-LAMP反応では、6つのプライマーが標的DNAの8つの領域を認識する。略語: 逆転写ループ媒介等温増幅;FIP = 前方内側プライマー。BIP = 後方内側プライマー;F3 = 前方変位プライマー;B3 = 後方変位プライマー;LoopF = フォワードループプライマー;LoopB = 逆方向ループプライマー。
| 入門 | 順序 | ターゲット | 製品サイズ | |||
| RTランプ9,18,19 | ||||||
| E1-F3 | タグタックガアクタクトクトクトキャット | E | はしごのようなパターン | |||
| E1-B3 | TTCAガットッタカッチャガガグ | |||||
| E1-FIP | アカクガグカーガーアガガグツクトクトクトCGガーガガガサック | |||||
| E1-BIP | TTGCTAGCACタグコッカタックトタットゥッタッタアガクトクタット | |||||
| E1ループB | GCGCTCGATGTGTGTGT | |||||
| E1-ループフ | CGCTタッタタッタッタアCG | |||||
| N2-F3 | アカガアクアトカガウグ | N | ||||
| N2-B3 | ガクトットガクトクトトクトトッグガッガクト | |||||
| N2-FIP | TTCCGAACGCTGAAGCGGAACTGATAAACATTGGCC | |||||
| N2-BIP | CGCATTGGGGGGAGTカカタットガッグカッテクトGTタ | |||||
| N2ループフ | GGGGGCAATTGTGCAATTTG | |||||
| N2ループB | CTTCGGGAACGTTGACC | |||||
| RT-qPCR38 | ||||||
| 2019-nCoV_N1-F | ガマクカーアアトカGCガート | N | 72 bp | |||
| 2019-nCoV_N1-R | TCTGGTACTGCCAGTTGAATCTG | |||||
| 2019-nCoV_N1-P | 5'-FAM-ACC CCG CAT GTT TGG TGG ACC-BHQ1-3' | |||||
| 2019-nCoV_N2-F | タカアキャットコッチャア | N | 67 bp | |||
| 2019-nCoV_N2-R | GCGCGCGCGCTGCGCGCGAAガーガー | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CAG CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | アカッタックッタタッタタグクト | E | 113 bp | |||
| E1_Sarbeco_R | アタツコガガタカカ | |||||
表 2: RT ランプ、RT-qPCR、および RT-PCR に使用するプライマー。プライマー配列、標的遺伝子、期待される産物サイズ、および対応する基準がリストされている。略語: RT-LAMP = 逆転写ループ媒介等温増幅;RT-PCR = 逆転写ポリメラーゼ連鎖反応;bp = 基本ペア。RT-qPCR = リアルタイム定量 RT-PCR; E1 = エンベロープ遺伝子; N2 = ヌクレオキャプシド遺伝子;F = フォワードプライマー;R = リバースプライマー;P = プローブ;FIP = 前方内側プライマー。BIP = 後方内側プライマー;F3 = 前方変位プライマー;B3 = 後方変位プライマー;LoopF = フォワードループプライマー;LoopB = 逆方向ループプライマー。
| 試薬 | ボリューム(μL) |
| UDGとウォームスタートのカラーメトリックランプ2Xマスターミックス | 12.5 |
| N2 プライマーミックス (20x) | 1.25 |
| E1 プライマーミックス (20x) | 1.25 |
| グアニジン塩酸塩(600 mM)* | 2.5 |
| 標的RNA | 5 |
| ヌクレアーゼフリー H2O | 2.5 |
| 総ボリューム | 25 |
表3:多重色分けRT-LAMPの反応マスターミックス。 (*)グアニジン塩酸塩は、特徴のない機構28によって反応の感度および速度を増加させることが示されている。略語: LAMP = ループ媒介等温増幅;UDG = ウラシル-DNAグリコシラーゼ; N2 = ヌクレオキャプシド遺伝子; E1 = エンベロープ遺伝子;DEPC = ジエチルピロカーボネート.
| 承認済みの市民 | キットを要求した市民 | 納入キット | 返却されたサンプルキット | サンプリング専用の日数 | 完成率キット | 不完全なキットの割合 | 加工サンプル | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | 意味する | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| 中央値 | 3 | |||||||
表 4:SARS-CoV-2 のスワビングを数値で指定する。 アウトリーチとサンプリング成功率。略語: SARS-CoV-2 = 重症急性呼吸器症候群コロナウイルス 2.
ディスカッション
市民科学者の関与。市民科学者は、都市環境におけるSARS-CoV-2の存在をサンプリングし、検出するために、サンディエゴ郡全体の表面を綿棒にするために募集されました。提供されたサンプリングキットの大半(70%)実験室に戻され、それらの、ほとんどすべてのサンプルが完了した(91%)(図3A、B、表4)ボランティアは、Webベースのプラットフォームを介してキットの配達/ピックアップを簡単に要求することができ、配達ルート計画ソフトウェアは、観測された成功のための両方の可能性の高い重要な要因である到着の推定時間を市民科学者に通知しました。キットが市民科学者に納入されてから実験室に戻された時点までの平均時間は8日で、中央値は3日、範囲は1~64日であった(図3Aおよび表4)。ボランティアへのより頻繁なリマインダーは、この遅れ時間を減らす可能性が高いです。
データ収集プラットフォームは、大多数のユーザーによって正常に使用されました (73%)(表 4)市民科学者の努力は測定されなかったが、フィールドテストは、データ収集プラットフォームがサンプル収集を適切に完了するために必要な労力と時間を大幅に削減したことを示した。したがって、簿記の量を減らすことは、市民科学者の関与を奨励しました。Webベースのプラットフォームは、多言語ニューラル機械翻訳サービスを提供し、英語とスペイン語でグラフィックと視聴覚プロトコルを提供することにより、人口統計学的な制限を克服することを意図していました。これは、郡のヒスパニック/ラテン系の人口のほとんどが45に住んでいるサウスベイとノース郡の両方から収集されたサンプルが少なかったので、部分的にしか成功しませんでした。これらの地域はまた、サンディエゴ郡のCOVID-19症例全体の63%(100,000例あたり1,700例)を抱えており、疾患の罹患率が最も高く、入院率は47,48でした。サンプルのほとんどはセントラル郡から来ましたが、代表的な数は最もCOVID-19影響を受けた地区から収集され、サンプルのごく一部だけが陽性であり、都市環境におけるSARS-CoV-2の表面貯水池は比較的まれであることを示唆しています。
サンプル処理。サンプリング綿棒はSDSで湿潤し、エンベロープを破壊してウイルスを不活性化し、RNases32を展開して裸のRNAを安定させた。回収中に便利に、綿棒の洗剤がサンプリングされた表面をきれいにした。環境サンプルには、多くの場合、非常に少量のRNAが含まれています。回収を最大化するために、RNAの単離は、GITCベースのカラムフリーの粗抽出法を用いて行った。強力なカオトロピック剤であるGITCは、タンパク質の折りたたみを維持する水素結合(すなわち疎水性効果)を破壊する。この作用は、ウイルス粒子の不活性化をもたらし、RNAはRNAses34、35、36の阻害のために安定したままである。GITCソリューションは、厳格なコールドチェーンの検討なしにRNAサンプルの安定性を維持し、提供されたアイスパックの冷凍庫が利用できない場合、市民は室温でサンプルを維持することができました。この試薬が直接皮膚または粘膜接触が発生したときに生じる潜在的な危険を減らすために、市民はキットに提供される物質的な安全データシートを含めることによってこれらのリスクを認識させ、チューブを含む箱に警告シールを置いた。
粗いGITC-クロロホルム抽出法は、綿棒からのRNAの痕跡の回収を助け、スパイクされたサンプルの増幅によって示されるように、抽出後にサンプル中に阻害剤がほとんど持続しない。サンプルはSARS-CoV-2に対して陰性であり、RT-LAMP阻害を示さなかったが、真の陰性を表すか、または100%周波数でLODよりも低いコピー数を示した。逆に、表面上のウイルスRNAの検出は、陽性サンプルからのウイルスの感染性をテストする必要があるため、接触を介して伝達されるリスクを直接意味するものではありません。環境の迅速なスクリーニングは、洗練された供給物や高度な資格を持つ人員の入手可能性によって制限されず、表面がウイルス貯蔵所を構成しているかどうかを評価し、より良い直接的な予防および封じ込めの取り組みのために重要です。
RT-LAMPは、提案された検出パイプラインに適した最良の方法として選択された。これは、残りの阻害剤のほとんどに対して非常に耐性があり、他のRT-qPCR法と同様に敏感かつ特異的な迅速かつ安価な方法であることが判明した。SARS-CoV-2のパンデミックの間の臨床の設定の使用のために、RT-qPCRキットの利用可能性は世界的な需要の影響を受けた。さらに、RT-qPCR技術は、阻害剤に抵抗するために処方されたものであっても、市民科学者のパイロットコホートによって収集された環境サンプルに含まれる物質に対して感受性であった。これらの知見は、COVID-19患者が取り扱うキャンディーから綿棒サンプル上のSARS-CoV-2を検出する両方の方法を比較し、83%以上の結果を併合させ、RT-LAMP15によって分析されたサンプル中の阻害が25%低いという最近の研究によって裏付けられている。さらに、GITC-クロロホルム粗抽出は、RT-LAMPと相まって、RNAキット抽出およびRT-qPCR( 材料表)と比較して、試薬および供給のコストを42%削減した。
この方法により、数千の表面綿棒サンプルの高スループット分析が可能になります。640個のサンプルを表す最大80個のプールを、RT-LAMPによるSARS-CoV-2検出まで、RNA抽出から2日間で処理した。提案されたプロトコルは、半定量的であり、ウイルスRNAの検出に限定され、感染性ウイルス粒子の存在を示すものではない。スワッピングされた表面に存在する感染したフミットからのSARS-CoV-2の伝染のリスクを評価するためにさらなる分析が必要である。
この研究は、伝染性疾患を伴う健康上の緊急事態に直面したときの効果的なワークフローを含むテスト戦略を迅速に設定するためのプロトコルを提示する。提案されたサンプリングプロトコルは単純で、家庭で一般的に見られる供給を使用し、ウイルス検出方法はサーモサイクラーの代わりに水浴のような基本的な実験室の設定で利用できる装置で行われる。RT-LAMP試薬のコストは、RT-qPCRに必要なものよりも大幅に低く、高いグローバル需要シナリオの影響を受けにくい。この研究は、将来の流行の流行と世界的なパンデミックにおける環境ウイルス貯留層の評価のための枠組みとして機能する。
開示事項
すべての著者は、競合する利益は存在しないと宣言しています。
謝辞
ウイルス情報研究所(VII)の調査官、アンカ・M・セガール博士、ウィロー・セガール、パトリシア・L・ローワー、ゲイリー・ローワー、キャリー・L・ローワー、マグダ・シルビア・ピネッタ、エリザベス・クルーズ・カノ、グレゴリー・ピーターズ博士、スチュアート・A・サンディン博士、ジェニファー・スミス博士に感謝します。また、カリフォルニア州サンディエゴ校(UCSD)の小児科のロブ・ナイト博士、ジャック・ギルバート博士、ペドロ・バルダ=フェレ博士、サラ・アラード博士に感謝します。ステイシー・カロタ(SDSU科学大学)とジーナ・スピデル(SDSU)のロジスティクスサポート、フアン・ロドリゲスのサンプリング・プロトコルのアートとグラフィックデザインに感謝します。非常に困難な時期に、このプロジェクトに対する献身と献身に対して、すべての参加者に感謝します。この研究は、ジョー・アン・レーン博士(SDSU科学大学)と国立科学財団RAPID:SARS-CoV-2助成金の環境貯水池(賞番号:2030479)からの寛大な寄付によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
参考文献
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved