Method Article
Swabbing the Urban Environment - Eine Pipeline zur Probenahme und zum Nachweis von SARS-CoV-2 aus Umweltreservoirs
In diesem Artikel
Zusammenfassung
Ein Citizen-Science-Projekt wurde entwickelt, um Einwohner von San Diego zu rekrutieren, um Umweltproben für SARS-CoV-2 zu sammeln. Für die Datenübermittlung über eine benutzerfreundliche Oberfläche für mobile Geräte wurde eine mehrsprachige webbasierte Plattform geschaffen. Ein Laborinformationsmanagementsystem erleichterte die Entnahme von Tausenden von geografisch unterschiedlichen Proben mit Echtzeit-Ergebnisverfolgung.
Zusammenfassung
Um die Übertragung des schweren akuten respiratorischen Syndroms Coronavirus 2 (SARS-CoV-2) während der globalen Pandemie 2020 in der Gemeinschaft zu kontrollieren, implementierten die meisten Länder Strategien, die auf direkten menschlichen Tests, Gesichtsbedeckung und Oberflächendesinfektion basieren. Unter der Annahme, dass der Hauptübertragungsweg Aerosole und Atemtröpfchen umfasst, haben sich die Bemühungen zum Nachweis von SARS-CoV-2 in Fomiten auf Orte konzentriert, an denen eine hohe Prävalenz vermutet wird (z. B. Krankenhausstationen, Kreuzfahrtschiffe und Massentransportsysteme). Um das Vorhandensein von SARS-CoV-2 auf Oberflächen in der städtischen Umgebung zu untersuchen, die selten gereinigt und selten desinfiziert werden, wurden 350 Bürger aus dem Großraum San Diego County angeworben. Insgesamt sammelten diese Citizen Scientists 4.080 Proben. Es wurde eine Online-Plattform entwickelt, um die Lieferung und Abholung von Probenahmesätzen zu überwachen und Probendaten zu sammeln. Die Probenahme-Kits wurden größtenteils aus Vorräten gebaut, die in pandemiegestressten Geschäften erhältlich waren. Die Proben wurden mit Reagenzien verarbeitet, die trotz der wiederkehrenden Versorgungsengpässe leicht zugänglich waren. Die verwendeten Methoden waren hochempfindlich und resistent gegen Inhibitoren, die üblicherweise in Umweltproben vorhanden sind. Die vorgeschlagenen experimentellen Design- und Verarbeitungsmethoden waren erfolgreich bei der Einbeziehung zahlreicher Bürgerwissenschaftler, die effektiv Proben aus verschiedenen Oberflächen sammelten. Der hier beschriebene Workflow und die hier beschriebenen Methoden sind relevant, um die städtische Umwelt auf andere Viren zu untersuchen, die für die öffentliche Gesundheit von Belang sind und eine Bedrohung für zukünftige Pandemien darstellen.
Einleitung
Es wird angenommen, dass SARS-CoV-2 hauptsächlich durch das Einatmen kontaminierter Aerosole und Tröpfchen aus direktem Kontakt mit infizierten Personen übertragen wird1,2,3,4. In den Anfangsphasen der globalen COVID-19-Pandemie konzentrierten sich die Bemühungen zur Kontrolle der Übertragung von SARS-CoV-2 jedoch stark auf die Desinfektion von Oberflächen, das Händewaschen und die Desinfektion. Bis Ende 2020 stusten die Übertragungsrichtlinien der Weltgesundheitsorganisation (WHO)5 und der US-amerikanischen Centers for Disease Control and Prevention (CDC)6 die Übertragung in der Luft hauptsächlich bei engem Kontakt (<2 m) mit einer infizierten Person oder in Gegenwart von aerosolerzeugenden medizinischen Verfahren als Gefahr ein. Eine Selbstimpfung nach Kontakt mit kontaminierten Oberflächen oder Inhalation von aerosolisierten Fomiten als Übertragungsweg von SARS-CoV-2 ist noch nicht ausgeschlossen.
ES WURDEN COVID-19-Fälle gemeldet, bei denen eine Übertragung in der Luft unwahrscheinlich erscheint7,8. SARS-CoV-2-Virionen bleiben bis zu 8 h auf Kupfer, auf Karton und Edelstahl bis zu 24 h und auf Kunststoff bis zu 48 h9infektiös. In Kreuzfahrtschiffkabinen wurde SARS-CoV-2 RNA 17 Tage nach der Abfahrt der Passagiere7nachgewiesen. Luft- und Oberflächenproben aus Krankenhäusern und Nahverkehrssystemen wurden positiv auf SARS-CoV-2 und andere Coronavirengetestet 8,10,11,12,13,14. Eine Studie, die auf der äußeren Verpackung von Halloween-Süßigkeiten durchgeführt wurde, die von asymptomatischen und mittelschweren/leicht symptomatischen COVID-19-Patienten gehandhabt wurde, kam zu dem Schluss, dass die Kombination aus Händewaschen durch den Handler und Waschen der Süßigkeiten mit Handseife die SARS-CoV-2-RNA auf unter die Schwellenwerte15reduzierte.
Mehrere Methoden zur SARS-CoV-2-Diagnostik wurden auf der Grundlage der Echtzeit-Reverse-Transkriptionspolymerase-Kettenreaktion (RT-qPCR)16,17, Reverse-Transkriptionsschleifen-vermittelten isothermen Amplifikation (RT-LAMP)11,18,19,20,21und CRISPR-Cas-Technologien18,19,22,23veröffentlicht. Die meisten benötigen RNA-Extraktionskits, die in Zeiten erheblicher globaler Nachfrage oft knapp sind, und nur sehr wenige wurden für das Umweltscreening des Virusverwendet 24. Der Nachweis von SARS-CoV-2-RNA mit RT-LAMP stimmt nachweislich zu über 83% mit der Verwendung von RT-qPCR überein. Darüber hinaus führte RT-LAMP zu einer Reduzierung der nicht schlüssigen Ergebnisse um 25% im Vergleich zu RT-qPCR15.
RT-LAMP ist eine einfache Technik, die eine reverse Transkriptase verwendet, um cDNA aus einer RNA-Vorlage25 zusynthetisieren, gefolgt von einer DNA-Polymerase mit starker Strangverschiebungsaktivität, die DNA bei konstanter Temperatur synthetisiert (dh isotherme Amplifikation)26. Eine höhere Spezifität des viralen Genomnachweises wird durch die Verwendung von vier oder sechs Primern erreicht, die sechs oder acht Regionen der Ziel-DNA erkennen. Die Amplifikation wird von einem internen Primer initiiert und ergibt eine semi-doppelsträngige DNA-Struktur. Der führende Strang wird dann durch eine äußere Grundierung verstärkt. Diese Verstärkungen werden für die umgekehrten Primer wiederholt. Interne und äußere Grundierungen an beiden Enden haben eine interne umgekehrte selbsterlehnte Stelle, die eine Schleife im Verstärkungsprodukt26,27bildet. Bei der isothermen Strangverschiebung erzeugt die asynchrone DNA-Synthese hohe Mengen an amplifiziertem Produkt, wobei die kontinuierliche Polymerisation das Signal von nur 10 Kopien pro Reaktion verstärkt11,20,28. Der kolorimetrische RT-LAMP-Mix ist schwach gepuffert und verwendet Phenolrot als pH-Indikator. Da die Polymerase ein Nukleotid enthält, setzt sie ein Proton frei, und genügend Protonen ändern den pH-Wert der Lösung sowie ihre Farbe von rosa nach gelb11,20,28,29.
RT-LAMP wurde für den Nachweis von durch Mücken übertragenen Krankheiten in peripheren Gesundheitseinrichtungen entwickelt, in denen es an voll ausgestatteten Laborsmangelt 25 und für den schnellen Nachweis anderer RNA-Viren wie dem humanen Immundefizienzvirus30. Den am stärksten gefährdeten Bevölkerungsgruppen bei Epidemienausbrüchen – gemäß der Definition der WHO – mangelt es oft an ausreichenden wirtschaftlichen Ressourcen und der geeigneten Ausrüstung, um die Erkennung durchzuführen (Global Public Health Agenda der Vereinten Nationen). In der aktuellen SARS-CoV-2-Pandemie konnten Lieferungen wie medizinische Abstriche und Reagenzien für RNA-Extraktionskits die globale Nachfrage nicht decken, insbesondere in Nicht-Produktionsländern. Das vorgeschlagene Protokoll verwendete eine Guanidiniumthiocyanat (GITC)-basierte Roh-RNA-Extraktion, die die RNA effektiv kühlkettenunabhängig konservierte und die Persistenz der Inhibitoren aus der Probe signifikant reduzierte. Darüber hinaus basiert das GITC-Chloroform-Extraktionsprotokoll auf der Trennung von RNA von DNA und Proteinen, gefolgt von der jeweiligen Ausfällung, was die Rückgewinnung des größten Teils des genetischen Materials ermöglicht. Diese Vorteile überwiegen die potenziellen Gefahren, die Bürgerwissenschaftler im Umgang mit der Chemikalie haben, wenn Maßnahmen ergriffen werden, um sie angemessen über die Risiken zu informieren.
Der vorgeschlagene Workflow verwendet Materialien und Reagenzien, die von allgemeinem Nutzen sind. Es erfordert Geräte, die in einfachen, oft ländlichen Laborumgebungen verfügbar sind. Diese Methoden sind kostengünstig, hochbeständig gegen Inhibitoren, die häufig in Umweltproben oder Proben vorkommen, die nicht mit Extraktionskits verarbeitet werden können, und machen einen hochpräzisen Thermocycler überflüssig. Diese Studie stellt eine Pipeline für die Probenahme und den Nachweis von SARS-CoV-2 aus Umweltreservoirs auf häufig berührten und selten desinfizierten Oberflächen von Haushalten und der städtischen Umwelt vor.
Protokoll
Eine detaillierte Liste der Reagenzien und Verbrauchsmaterialien, einschließlich Katalognummern, Hersteller und entsprechender Kosten, finden Sie in der Materialtabelle.
1. Beprobung der städtischen Umwelt
- Citizen Scientist Outreach
- Rekrutieren Sie Citizen Scientists mit einem direkten und klaren Call-to-Action, der über lokale und soziale Medien veröffentlicht wird. Erstellen Sie ein Social-Media-Handle (z. B. #swab4corona),um das Thema über Social-Media-Inhalte hinweg zu verbinden.
- Erstellen Sie ein E-Mail-Konto für die direkte Kommunikation zwischen dem Laborteam und jedem Citizen Scientist, das von einer Person verwaltet wird, die die wichtigsten Sprachen der Region von Interesse fließend spricht (z. B. Spanisch und Englisch für San Diego County).
- Aufbau einer sicheren webbasierten Probenmanagementplattform (SMP), die als Datenbank, Laborinformationsmanagementsystem (LIMS) und zur Kommunikation mit Citizen Scientists dient.
HINWEIS: Das SMP bietet einen zentralen Speicherort, an dem Benutzer ein Kit anfordern, auf Probensammlungsprotokolle zugreifen, Beispielmetadaten übermitteln und eine Abholung für abgeschlossene Probenkits anfordern. - Erstellen Sie einen Link zum SMP (z. B. https://demo.covidsample.org/) (Abbildung 1), damit Einzelpersonen die Teilnahme an den Umweltprobenahmebemühungen beantragen können, indem sie Fragen zur biologischen Sicherheit beantworten, die in einem Online-Formular angegeben sind.
- Sicherer Zugriff auf das SMP über eine Authentifizierungs-Anwendungsprogrammierschnittstelle, die von einem Cloud-Computerdienstanbieter unterstützt wird. Gewähren Sie genehmigten Benutzern Zugriff auf das SMP.
HINWEIS: Eine Beschreibung des OAuth 2.0-Protokolls31 für Authentifizierung und Autorisierung finden Sie in der Literatur. Es bietet einen reibungslosen Anmeldeprozess für Freiwillige von Citizen Scientist. Außerdem können sich Benutzer mit einem vorhandenen Konto anmelden, sodass keine benutzerdefinierte Anmeldelösung erstellt und Benutzeranmeldeinformationen verwaltet werden müssen, was viel Zeit spart und die Teilnahme fördert. Jüngsten Berichten zufolge hat der kostenlose E-Mail-Dienst, der für den ausgewählten Cloud-Computer-Dienstanbieter verfügbar ist, etwa 1,5 Milliarden monatlich aktive E-Mail-Benutzer; die Anforderung eines E-Mail-Kontos von diesem Dienstleister für die Teilnahme wird nicht als entmutigender Faktor angesehen. - Erläutern Sie den Citizen Scientists am SMP das Ziel der Studie und Überlegungen zur biologischen Sicherheit, bevor sie ihr erstes Kit anfordern. Stellen Sie ein mehrsprachiges Plugin bereit, um die Navigation in einer der Sprachen zu ermöglichen, die von einem mehrsprachigen neuronalen maschinellen Übersetzungsdienst verfügbar sind, der von einem Cloud-Computerdienstanbieter unterstützt wird.
- Fügen Sie im Sampling-Abschnitt grafische und audiovisuelle Protokolle in Englisch und Spanisch ein.
- Weisen Sie jedem Kit eine eindeutige Kennung zu, und entwerfen Sie die Benutzeroberfläche so, dass Schaltflächen verwendet werden, die mit der Beispiel-ID verknüpft sind, um den Dateneingabeprozess zu optimieren (Abbildung 1A).
- Verwenden Sie eine Lieferroutenplanungssoftware mit einer Anwendung für mobile Geräte, die von Fahrern verwendet werden kann, um Liefer- / Abholrouten zu optimieren und Bürgerwissenschaftler über genaue geschätzte Ankunftszeiten zu informieren.
- Erstellen Sie die LIMS-Plattform auf einem PHP-Webdienst-Stack und hosten Sie sie auf einer kommerziellen Hosting-Plattform (vorgeschlagenes Betriebssystem, Webserver-Software und Datenbanksoftware sind in der Materialtabelleangegeben).
- Stellen Sie eine sichere webbasierte Anwendungsschnittstelle bereit, damit das Laborpersonal Daten schnell und einfach im LIMS verwalten kann. Stellen Sie Datenvisualisierung mithilfe einer Datendiagramm-Anwendungsprogrammierschnittstelle bereit, die von einem Cloud-Computerdienstanbieter unterstützt wird.
- Visualisieren Sie Geodaten mithilfe einer Geodaten-Anwendungsprogrammierschnittstelle, die von einem Cloud-Computerdienstanbieter unterstützt wird. Speichern Sie die daten, die über das SMP an das LIMS übermittelt werden, um (1) die zentrale Speicherung von Projektdaten zu erleichtern; (2) Verfolgung von Proben-/Datenverarbeitungsworkflows; und (3) Management der Logistik der Probenkit-Verteilung an Citizen Scientists.
- Sichern Sie übermittelte Metadaten mithilfe von Best Practices (z. B. https://demo.covidsample.org/).
- Laden Sie Informationen wie Sample Kit ID, Sample ID, Datum, Uhrzeit und GPS-Koordinaten (Global Positioning System) vor (automatisch aus einem Bild des Standorts gesammelt), um die Datentypkonformität zu ermöglichen und die Übermittlung fehlerhafter oder fehlender Daten durch den Benutzer zu minimieren (Abbildung 1B). Fügen Sie die folgenden Felder ein, die vom Citizen Scientist manuell und schnell (<1 min) ausgefüllt werden sollen: Datum und Uhrzeit der Sammlung, eine kurze Beschreibung des Standorts und ein Bild der Probenahmestelle.
- Berefizieren Sie alle hochgeladenen Daten und überprüfen Sie sie auf Datentyp. Überprüfen Sie beispielsweise von Benutzern hochgeladene Bilddaten, um .jpg Dateien auszuwählen, benennen Sie sie mit Sample ID um, um sie schnell mit dem Beispiel zu assoziieren, und speichern Sie hochgeladene Bilder an einem separaten sicheren Ort, auf den Benutzer nicht zugreifen können.
- Aktivieren Sie die Option, die Lieferung und Abholung des Kits anzufordern, wenn alle Proben (16) abgeschlossen sind. Aktivieren Sie außerdem die Option, ein neues Kit anzufordern, das bei abholung des vorherigen kits geliefert werden soll (Abbildung 1A).
HINWEIS: Für Freiwillige, die eine nicht webbasierte Plattform bevorzugen, und für diejenigen, die sich Sorgen um die Offenlegung ihres GPS-Standorts machen (z. B. Mitglieder der Gemeinschaft, die über ihren Migrationsstatus besorgt sind), können Kits an einem vereinbarten Treffpunkt geliefert und Freiwillige gebeten werden, eine schriftliche Version der Datenerfassung aufzuzeichnen. Für die Kommunikation zwischen dem Labor und jedem Citizen Scientist steht ein zweisprachiges Projektmitglied für Telefonate und SMS zur Verfügung.
- Tupfer für Corona
- Identifizieren Sie ein epidemiologisch relevantes Zeitfenster für den Probenahmeaufwand.
- Erstellen Sie ein Kit, das alle Probenahmematerialien enthält, einschließlich der erforderlichen persönlichen Schutzausrüstung (z. B. Maske, Handschuhe), eines Probenahmeprotokolls und für die biologische Sicherheit relevanter Informationen (Abbildung 2). Beschriften Sie jedes Röhrchen mit der zugewiesenen eindeutigen Kennung (Proben-ID).
- Tupfer desinfiziert selten Oberflächen, die aerosolisierten Fomiten in Haushalten und der städtischen Umgebung ausgesetzt sind.
- Tragen Sie die mitgelieferte Maske in der Öffentlichkeit und ein neues Paar Handschuhe für die Entnahme jeder Probe, um Kreuzkontaminationen zu vermeiden. Nachdem Sie die Probenahme abgeschlossen haben, verwenden Sie das mitgelieferte Händedesinfektionsmittel.
- Befeuchten Sie einen 1 cm2 Polyester absorbierenden Tupfer (z. B. Wischpads) mit einem Reinigungsmittel (z. B. 0,5% Natriumdodecylsulfat (SDS)), um das Virus durch Störung seiner Hülle zu inaktivieren und die nackte RNA durch Induktion der Entfaltung von RNasen zu stabilisieren32.
- Tupfen Sie eine Fläche von 10 cm2ab. Tauchen Sie mit Hilfe eines Zahnstochers jeden Probentupfer vollständig in das entsprechende vormarkierte Röhrchen mit 200 μL Guanidiniumthiocyanatlösung (GITC) ein. Lagern Sie die Röhrchen bei 4 °C, bis sie ins Labor transportiert werden. Sobald die Proben im Labor angekommen sind, lagern Sie sie bei -80 °C.
HINWEIS: GITC ist ein giftiger Reizstoff; Kontakt mit der Haut vermeiden. GITC-Lösung ist einfach aus gängigen Laborchemikalien herzustellen, für das Rezept siehe33,34. Es inaktiviert das Virus, stabilisiert RNA durch Denaturierung der RNasen34,35,36und stabilisiert Proben bei Raumtemperatur. Das Kit enthält jedoch Eisbeutel, um die Proben kalt zu halten, ohne dass Haushaltskühlschränke für die Lagerung verwendet werden müssen.
2. SARS-CoV-2 Nachweis
- Totale RNA-Isolierung
- Desinfizieren Sie Oberflächen, Geräte und Pipettierer mit einer Lösung von 2 mM Kupfersulfat und 3% Wasserstoffperoxid; gefolgt von einer Lösung aus 10% Bleichmittel, 90 mM Natriumbicarbonat, 5% SDS und 2,5% NaOH. Gründlich mit destilliertem Wasser abwischen, gefolgt von 75% Ethanol.
HINWEIS: Diese Lösungen sind eine Alternative zu den handelsüblichen Lösungen. - Proben auf Eis auftauen. Wirbelproben für 2 min bei mittlerer Geschwindigkeit.
- Um die Geschwindigkeit des Screenings zu erhöhen, verarbeiten Sie die Proben in Pools. Wenn ein Pool positiv ist, extrahieren Sie die RNA jeder Probe unabhängig voneinander, um die positive Probe (n) zu finden. Kombinieren Sie die Proben aus jedem Probenahmeset (insgesamt 16) in 2 Pools mit 8 Proben.
HINWEIS: 8 Proben pro Pool bedeuten, dass nur 2 Pools pro Kit verarbeitet werden müssen. Ist ein Pool positiv, werden die einzelnen Proben für die individuelle RT-LAMP-Analyse wieder aufbereitet. Dies reduziert Zeit, Kosten und Reagenzien. - 50 μL von jeweils 8 Proben in einem Mikrozentrifugenröhrchen (Gesamtvolumen 400 μL) bündeln; Speichern Sie die restliche Probe bei -80 °C. 0,2 Volumina (80 μL) Chloroform, Wirbel für 15 s hinzufügen und dann 20 min bei 4 °C inkubieren. Zentrifuge bei 13.000 × g für 20 min bei 4 °C.
- Die wässrige (klare Flüssigkeit) Schicht in ein neues Mikrozentrifugenröhrchen geben. Lagern Sie die restliche Schnittstelle und die rosa Flüssigkeit im -80 °C-Gefrierschrank; diese Fraktionen enthalten DNA und Proteine33,36.
- Fügen Sie ein gleiches Volumen Isopropanol (~ 200 μL) und 2,6 μL Glykogen-Coprecipitant (15 mg ml-1)hinzu 37. Gut mischen und bei -20 °C für mindestens 1 h inkubieren, gefolgt von 4 °C für 10 min, um RNA auszufälschen.
HINWEIS: Das Protokoll kann hier pausiert werden, indem Proben bei -20 °C über Nacht statt 1 h inkubiert werden. - Zentrifuge bei 13.000 × g für 20 min bei 4 °C. Entfernen Sie den Überstand, ohne das Pellet zu stören. Das Pellet wird in 50 μL Diethylpyrocarbonat (DEPC)-behandeltem Wasser resuspendiert und ein gleiches Volumen (50 μL) RNase-freies 5 M Ammoniumacetat und 2,5 Volumina (250 μl) 100% Ethanol7,38hinzugefügt.
HINWEIS: Ammoniumionen hemmen die Polynukleotidkinase, wenn sie in einem nachgeschalteten Prozess verwendet werden38. Die Mischung fällt RNA aus, während Desoxynukleosidtriphosphate und Oligosaccharide in Lösung38zurückblassen. - Gut mischen und bei -20 °C für mindestens 1 h inkubieren, gefolgt von 4 °C für 10 min, um RNA auszufälschen.
HINWEIS: Das Protokoll kann hier pausiert werden, indem Proben bei -20 °C über Nacht statt 1 h inkubiert werden. - Zentrifuge bei 13.000 × g für 20 min bei 4 °C. Waschen Sie das Pellet mit 1 ml kalt (-20 °C), frisch hergestellt 75% Ethanol. Zentrifuge bei 8.000 × g für 5 min bei 4 °C. Entfernen Sie den Überstand mit einer P10-Pipette, um eine Störung des Pellets zu vermeiden.
- Trocknen Sie das Pellet 10-15 minuten an der Luft, bis kein Ethanol mehr vorhanden ist. Das Pellet in 50 μL DEPC-behandeltem Wasser wieder aufsuchen, 5 μL 10x DNase-Puffer + 1μL DNase (2 EinheitenμL-1)hinzufügen und bei 37 °C für 30 min inkubieren.
- Fügen Sie 0,1 Volumen (5,6 μL) DNase-Inaktivierungsreagenz hinzu, inkubieren Sie bei Raumtemperatur für 5 minuten und mischen Sie jede Minute vorsichtig. Zentrifuge bei 13.000 × g für 2 min und überlagern Sie den Überstand in ein neues Röhrchen (~50 μL). Legen Sie das Röhrchen sofort während der Vorbereitung der RT-qPCR- oder RT-LAMP-Reaktionen auf Eis oder lagern Sie es in einem -20 °C-Gefrierschrank.
HINWEIS: Führen Sie die RNA-Isolierung in einem ampliconfreien Raum durch, um eine Verschleierung zu vermeiden.
- Desinfizieren Sie Oberflächen, Geräte und Pipettierer mit einer Lösung von 2 mM Kupfersulfat und 3% Wasserstoffperoxid; gefolgt von einer Lösung aus 10% Bleichmittel, 90 mM Natriumbicarbonat, 5% SDS und 2,5% NaOH. Gründlich mit destilliertem Wasser abwischen, gefolgt von 75% Ethanol.
- Multiplex-Reverse-Transkriptionsschleifen-vermittelte isotherme Verstärkung (RT-LAMP)
- Bereiten Sie für jeden Satz Primer (Tabelle 2) eine 20-fache Primer-Mixlösung (Tabelle 1) vor. Bereiten Sie die RT-LAMP-Reaktionsmischung (Tabelle 3) bei Raumtemperatur mit 10% Übervolumen vor, um den Pipettierverlust zu berücksichtigen.
HINWEIS: Der kolorimetrische LAMP 2X Master-Mix mit antarktischer thermolabiler Uracil-DNA-Glykosylase (UDG) verhindert die Amplifikation der DNA-Kontamination aus früheren Reaktionen20,28. - Wirbel und spinnen Sie die Mischung herunter. Dosieren Sie 20 μL der Mischung in jedes Reaktionsröhrchen: Probe, Spiked, Positivkontrolleund Negativkontrolle. Inkubieren Sie die Reaktionen in den Röhrchen bei Raumtemperatur für 10 Minuten, damit das UDG auf mögliche Verschränkungskontaminationen einwirken kann.
- 5 μL RNA zur Probenreaktion, 5 μL RNA + 2,5 μL (450 Kopien) synthetische SARS-CoV-2 RNA zur Spiked-Reaktion, 2,5 μL (450 Kopien) synthetische SARS-CoV-2 RNA zur positiven Kontrollreaktion und 5 μLH2O zur negativen Kontrollreaktion. Mischen Sie gut und drehen Sie die Reaktionen herunter; alle RNA auf Eis auftauen.
- Während der Thermocycler oder das Wasserbad auf 65 °C erhitzt wird, lassen Sie alle Reaktionen auf Raumtemperatur bleiben. Das UDG wird bei >50 °C inaktiviert. Legen Sie die Reaktionen in den Thermocycler (Wärmedeckel verwenden), inkubieren Sie bei 65 °C für 40 min und lassen Sie die Reaktionen Raumtemperatur erreichen (~22 °C für 5 min), oder kühlen Sie sie auf Eis für 1 min ab. Analysieren Sie die Ergebnisse mit der kolorimetrischen Funktion (einfache Beobachtung) oder indem Sie die Produkte in einem Agarosegel laufen lassen.
HINWEIS: Obwohl die Nachweisgrenze (LOD) 10 Kopien pro Reaktion beträgt, nimmt die Häufigkeit der Erkennung zu, wenn sich die Kopienzahl 500 Kopien pro Reaktion nähert (Abbildung 3A). Für die kolorimetrische Beobachtung ist zu beachten, dass ein negatives Ergebnis durch Rosa (pH = 8,8) angezeigt wird, während ein positives Ergebnis durch Gelb (pH = 5) angezeigt wird(Abbildung 3B). Die kolorimetrische Option vermeidet das Öffnen der RT-LAMP-Röhren nach der Verstärkung, wodurch das Volumen der RT-LAMP-Produkte in der Arbeitsumgebung und die Verschleierung der Kontamination reduziert wird. Für die Gelelektrophorese bereiten Sie ein 1,5% iges Agarosegel mit 1X DNA-Gelfleck in 0,5% Tris / Borat / EDTA (TBE) Puffer vor. Laden Sie 25 μL der Reaktion + 5 μL 6X Ladefarbstoff in jede Vertiefung. Führen Sie das Gel bei 100 V für 60 Minuten aus. Ein molekularer Marker ist nicht erforderlich, da positive Proben ein Leitermuster aufweisen (Abbildung 3C).
- Bereiten Sie für jeden Satz Primer (Tabelle 2) eine 20-fache Primer-Mixlösung (Tabelle 1) vor. Bereiten Sie die RT-LAMP-Reaktionsmischung (Tabelle 3) bei Raumtemperatur mit 10% Übervolumen vor, um den Pipettierverlust zu berücksichtigen.
Ergebnisse
Proben, die von Citizen Scientists zum Nachweis von SARS-CoV-2 gesammelt wurden. Während eines Zeitraums von 8 Monaten (Mitte März bis zur dritten Novemberwoche 2020) wurden 482 Bürgerinnen und Bürger zur Teilnahme an diesem Projekt zugelassen, von denen 350 (73%) ein Kit angefordert. Insgesamt wurden 362 Kits geliefert (d.h. einige Teilnehmer forderten mehrere Kits an), und 246 (70%) zurückgegeben wurden (Abbildung 4A,B). Alle 4.080 Proben, die in diesen Kits enthalten waren, wurden verarbeitet. Die Sammelstellen waren über die Distrikte North Coastal, North Central, Central und Southern sowie einige wenige in den östlichen Distrikten verteilt(Abbildung 4A). Diese Bezirke haben die höchste Bevölkerungsdichte von San Diego County und die am meisten dokumentierten COVID-19-Fälle, wie von der San Diego Human Health Service Agency39berichtet.
Die Bürger forderten die Abholung der meisten beprobten Kits (d. H. Durchschnittliche Erfolgsquote: 70,4%). Jeden Tag wurden 1-16 Kits angefordert und 0-14 Kits an das Labor zurückgegeben (Abbildung 4B). Eine Befragung der Citizen Scientists ergab, dass die Entnahme eines kompletten Kits (16 Proben) 1-3 h verteilt auf durchschnittlich 8 Tage dauerte (Abbildung 4A und Tabelle 4). Die große Mehrheit der Kits war vollständig (91,1%), d.h. sie enthielten einen Tupfer in allen 16 Probenröhrchen, und die entsprechenden Probenahmedaten wurden in das LIMS hochgeladen (Abbildung 4A und Tabelle 4).
Nachweis von SARS-CoV-2 mittels GITC-Chloroform-Extraktion und Multiplex-Reverse-Transkriptase-Loop-vermittelter isothermer Amplifikation (RT-LAMP). Für den kolorimetrischen RT-LAMP-Assay wurden zwei Sätze von Primern11,20,28 verwendet, um die Nukleokapsid- (N2) und die Hüllkurve (E1) -Gene anzusprechen (Tabelle 2). Die von diesen Primern erkannten Sequenzen befinden sich in der gleichen Region wie die Primer und Sonden, die von der CDC40 und der Europäischen Union (EU)41 für die menschliche Diagnose von COVID-19 durch RT-qPCR zugelassen sind. Diese Ergebnisse bestätigen, was Zhang et al.28 beschrieben haben, wobei die Zugabe von 60 mM Guanidinhydrochlorid zur Reaktion die LOD erhöht, wenn sie im Multiplex ausgeführt wird. Die LOD bei einer Frequenz von 100% betrug 500 Kopien pro 25 μL Reaktion (Abbildung 3A,B). Im kolorimetrischen RT-LAMP änderten positive Proben aufgrund einer pH-Verschiebung von ~8 auf 5,5 die Farbe von rosa zu gelb (Abbildung 3B). Als die Reaktion bei niedrigen Kopienzahlen orange wurde, wurden Proben in einem 1,5% igen Agarosegel durchgeführt, um zu bestätigen, dass diese positiv waren und zu einem leiterartigen Muster führten (Abbildung 3C). RT-LAMP wurde verwendet, um SARS-CoV-2 in gepoolten RNA-Proben nachzuweisen.
Um falsch negative Ergebnisse aufgrund von Reaktionshemmern zu kontrollieren, wurde jede Probe in einer zusätzlichen Reaktion mit 500 Kopien von synthetischem SARS-CoV-2 getestet. Positive gepoolte Proben wurden durch Isolierung der RNA jeder einzelnen Probe im Pool entepliziert und in einer RT-LAMP-Reaktion ausgeführt, um die Identität der positiven Probe zu bestimmen. Die Erkennungsergebnisse wurden dann in das LIMS hochgeladen, wo die eindeutige Proben-ID mit den Informationen zu Datum, Uhrzeit, GPS-Koordinaten, Standort und Bild der Probe gepaart wurde.
Echtzeit- und traditionelle RT-PCR-Methoden: Hemmung durch Probenverunreinigungen. Um die beste Methode für die vorgeschlagene Nachweispipeline auszuwählen, wurden andere RNA-Amplifikationsmethoden mit Umweltproben getestet, die von einer Pilotkohorte von Citizen Scientists gesammelt wurden. Beispiele für die Ergebnisse jeder dieser Methoden sind in Abbildung 5 dargestellt, um ihre Empfindlichkeit gegenüber Umweltinhibitoren und Hintergrundsignalrauschen bei niedrigen viralen Kopienzahlkonzentrationen darzustellen.
Sechs rt-qPCR-Formulierungen (Table of Materials), die von der CDC und der WHO zugelassen wurden, wurden auf den Nachweis des Virus an Umweltproben getestet. Protokolle wurden gemäß den Anweisungen des Herstellers sowie CDC-Richtlinien zum Nachweis von SARS-CoV-2 in klinischen Umgebungenbefolgt 40. Reaktionen, die unterschiedliche Konzentrationen synthetischer SARS-CoV-2-RNA-Kontrollen enthielten, wurden nach roher RNA-Isolierung in Abgetupferoberflächenproben gespiked. Alle Mastermischungen waren empfindlich gegenüber Inhibitoren bei LOD-Konzentrationen der Positivkontrolle (Abbildung 5A).
Um Inhibitoren dieser Echtzeittechnologien zu umgehen, wurde ein herkömmliches RT-PCR-System getestet. Ein einstufiges RT-PCR-System (Table of Materials) wurde verwendet, um das Nukleokapsid-Gen mit den Primer-Sets N1, N2und dem Hüll-Gen mit dem von der CDC (USA) bzw. dem ECDC (EU) zugelassenen Primer-Set E1 zu amplifizieren (Tabelle 2). Protokolle wurden gemäß den Anweisungen des Herstellers sowie CDC-Richtlinien zum Nachweis von SARS-CoV-2 in klinischen Umgebungenbefolgt 40. Die von der CDC entwickelten Primer-Sets N1 und N2 ergeben ein Produkt von ~ 70 bp; Positive mit niedriger Kopienzahl wurden jedoch nicht von den Amplifikationshintergrundgeräuschen der Negativkontrolle (Abbildung 5B)unterschieden, wodurch Fehlalarme in die Ergebnisse eingebracht wurden. Das Produkt der E1-Primer hatte ein schwaches Signal bei niedriger Kopienzahl ( Abbildung5B),wodurch falsche Negative in die Ergebnisse einführten. Darüber hinaus war die getestete RT-PCR-Methode immer noch empfindlich gegenüber Inhibitoren, die in den Umweltproben vorhanden waren (Daten nicht gezeigt).
Andere Methoden wurden entwickelt, um sehr kleine Mengen der Zielsequenz zu detektieren. Eine dieser Methoden ist die Rolling Circle Amplification (RCA), bei der die Erkennung der Zielsequenz, RNA oder DNA, durch eine spezifische lineare Sonde, eine Ligase die Vorlage zirkularisiert. Unter Verwendung von Primern, die für die Hybridisierung mit der Sonde entwickelt wurden, verstärkt eine DNA-Polymerase mit Strangverschiebungsaktivität die Sonde in einer isothermen Reaktion42. Es ist die Sonde, die das Ziel identifiziert, das verstärkt wird, und nicht die Zielsequenz, die diese Methode hochempfindlich macht43. Wang et al.44 veröffentlichten ein RCA-Protokoll zum direkten Nachweis von SARS-CoV-1-RNA. Das Verfahren wurde modifiziert, um Primer speziell für SARS-CoV-2 zu verwenden. Leider zirkularisiert und liefert die Sonde bei der Non-Template-Kontrolle (NTC) das Produkt in Abwesenheit einer RNA-Vorlage, selbst wenn eine Vielzahl von Ligasen verwendet wird, einschließlich einer SNP-sensitiven Ligase. In Abwesenheit einer Ligase zeigte der NTC keine Verstärkung von der linearisierten Sonde (Abbildung 5C).

Abbildung 1: Webbasierte Probenahmeplattform mit Probensammeldatenschnittstelle für mobile Endgeräte. (A) Eine Website mit einem mehrsprachigen Plugin wurde erstellt, um die Interaktion zwischen dem Labor und den Citizen Scientists zu vermitteln. Die Plattform wurde für die Lieferung von Musterkits / Abholanforderungen und die Einreichung von Musterdaten verwendet. Die Plattform enthielt detaillierte englisch/spanische Grafik- und audiovisuelle Sampling-Protokolle. (B) Ansicht des mobilen Geräts, die zum Hochladen von Beispieldaten verwendet wird: Datum, Uhrzeit, GPS-Koordinaten, Beispielstandortbeschreibung und ein Bild des Sammelstandorts. Abkürzungen: SARS-CoV-2 = schweres akutes respiratorisches Syndrom Coronavirus 2; GPS = Global Positioning System. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2:Probensammelkit. Citizen Scientists erhielten einen Kühler mit zwei Eisbeuteln, einem Sicherheitsdatenblatt, um die Freiwilligen über die Gefahren beim Umgang mit der GITC-Lösung zu informieren, ein detailliertes Probenahme- und Maskentrageprotokoll, eine KN95-Maske, einen Abfallbeutel, eine Sprühflasche mit Händedesinfektionsmittel, eine Sprühflasche mit 0,5% SDS, 16 Paar Handschuhe, einen kleinen Beutel mit 16 Zahnstochern und 16 Polyestertupfern, 16 voretikettierte Mikrozentrifugenröhrchen mit 200 μL GITC-Lösung, eine Schachtel mit den Probenahmeröhrchen und einen Beutel, der als Sekundärbehälter für die Tubenbox im Falle eines Verschüttens verwendet wird. Abkürzungen: GITC = Guanidiniumthiocyanat; SDS = Natriumdodecylsulfat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
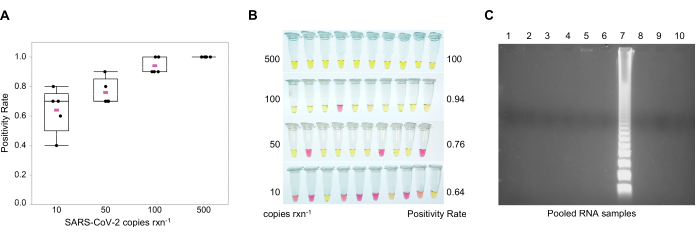
Abbildung 3: Multiplexed Reverse-Transcription Loop-Mediated Isothermal Amplification (RT-LAMP) Assay. Multiplexreaktionen mit Primern für SARS-CoV-2-Nukleokapsid (N2) und Hüllkurven (E1) Gene, um nur 10 Kopien des Virus in der Reaktion nachzuweisen. Als Positivkontrolle wurde synthetische SARS-CoV-2-RNA eingesetzt. (A) Häufigkeit des Nachweises von SARS-CoV-2 in multiplex kolorimetrischem RT-LAMP bei unterschiedlichen Genomkopienzahlen pro Reaktion. Mittelwert von fünf Replikaten in Rosa. (B) Nachweisgrenze (LOD) von SARS-CoV-2 in multiplex kolorimetrischen RT-LAMP; gelb = positiv (pH ~5); rosa = negativ (pH ~8). (C) Leitermuster positiver SARS-CoV-2 RT-LAMP-Reaktionen bei 1,5%iger Agarosegelelektrophorese. Abkürzungen: SARS-CoV-2 = schweres akutes respiratorisches Syndrom Coronavirus 2; rxn = Reaktion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
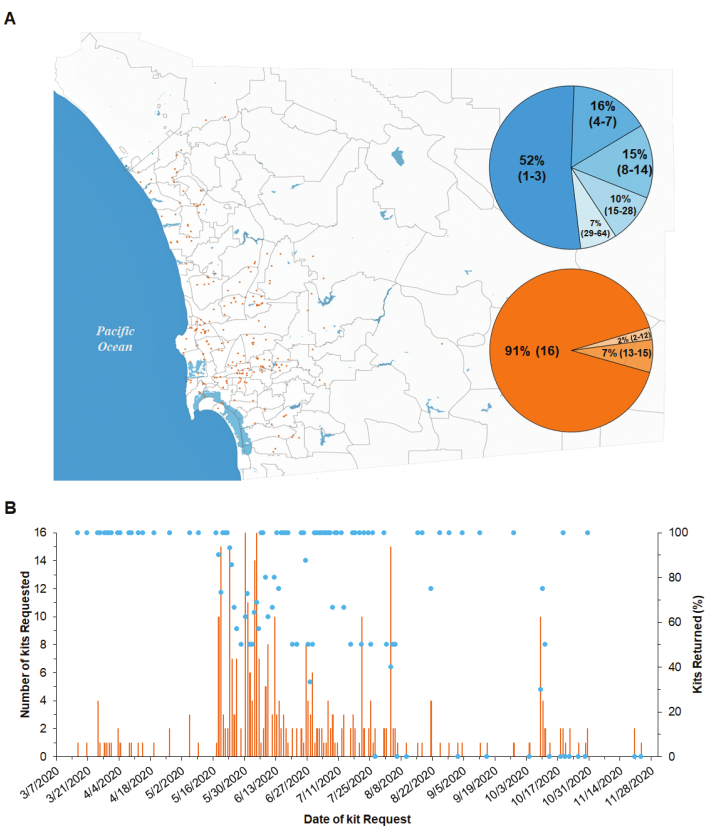
Abbildung 4: Lage der Citizen Scientist-Probenahmekits in San Diego County und Erfolgsrate der angeforderten Kits. (A) Orange Punkte stellen den Standort von 1 Probenahmekit dar, das 16 Proben enthält. Das blaue Kreisdiagramm zeigt den Prozentsatz der Kits, die verschiedene Tage von der Auslieferung an die Citizen Scientists bis zur Rückgabe an das Labor dauerten. Anzahl der Tage in Klammern. Das orangefarbene Kreisdiagramm zeigt den Prozentsatz der Kits mit unterschiedlicher Anzahl abgeschlossener Proben aus insgesamt 16 Proben. Anzahl der ausgefüllten Proben, die einen Tupfer im Probenröhrchen und die entsprechenden Probenahmedaten enthalten, die in Klammern in das LIMS hochgeladen wurden. (B) Prozentsatz der Kits, die an das Labor zurückgegeben wurden (Punkte), und Gesamtzahl der angeforderten Kits (Riegel) im Verhältnis zum Datum, an dem das Kit angefordert wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
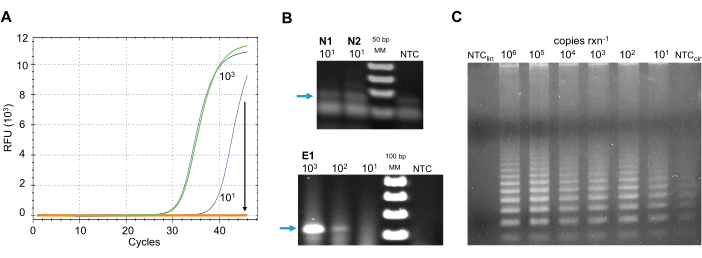
Abbildung 5: Alternative SARS-CoV-2 RNAdetection-Methoden. (A) RT-qPCR-Nachweis des SARS-CoV-2-Nukleokapsidsids (N) Gens unter Verwendung des Primersatzes N2. Gepoolte Umweltproben mit 900 (grün) oder 9 (orange) Kopien von SARS-CoV-2. Positivkontrollen der gleichen Kopiennummern in blau. Arrow zeigt die Abnahme der Fluoreszenzdetektion der Positivkontrolle mit niedriger Kopienzahl an, wenn eine Umgebungsprobe vorhanden ist. (B) Traditioneller RT-PCR-Nachweis von SARS-CoV-2. Oben: RT-PCR-Produkte des Nukleokapsid-Gens unter Verwendung der Primer-Sets N1 und N2. Im Steuerelement ohne Vorlage wird ein schwaches Hintergrundsignal beobachtet. Unten: RT-PCR-Produkte des Hüllgens mit Primer-Set E1. Bei der LOD-Konzentration wird ein sehr niedriges Signal beobachtet. Blaue Pfeile zeigen das erwartete positive Produkt: (oben) ~ 70 bp und (unten) 113 bp in 2% Agarosegelelektrophorese. (C) RCA von SARS-CoV-2 RNA. Kreisförmige Sonde amplifiziert in Gegenwart von Ligase und Abwesenheit von RNA-Vorlage (NTCcir); in Abwesenheit von Ligase und RNA-Vorlage wird die lineare Sonde nicht amplifizieren (NTClin). Abkürzungen: SARS-CoV-2 = schweres akutes respiratorisches Syndrom Coronavirus 2; RFU = relative Fluoreszenzeinheiten; bp = Basenpaare; rxn = Reaktion; MM = molekularer Marker; RT-PCR = Reverse-Transkriptions-Polymerase-Kettenreaktion; RT-qPCR = quantitative RT-PCR in Echtzeit; RCA = Rollkreisverstärkung; NTC= Steuerelement ohne Vorlage; LOD = Nachweisgrenze; rxn = Reaktion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Fibel | 20X Konzentration (μM) | 1x Konzentration (μM) |
| Fip | 32 | 1.6 |
| BIP | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 | 4 | 0.2 |
| LoopF | 8 | 0.4 |
| LoopB | 8 | 0.4 |
Tabelle 1: Formulierung für 20X RT-LAMP Primer Mix. In der RT-LAMP-Reaktion erkennen 6 Primer 8 Regionen der Ziel-DNA. Abkürzungen: Reverse-Transkriptionsschleifen-vermittelte isotherme Amplifikation; FIP = vordere innere Grundierung; BIP = rückwärts innere Grundierung; F3 = Vorwärtsverschiebungsprimer; B3 = Rückwärtsverschiebungsprimer; LoopF = Vorwärtsschleifenprimer; LoopB = Rückwärtsschleifenprimer.
| Fibel | Reihenfolge | Ziel | Produktgröße | |||
| RT-LAMPE 9,18,19 | ||||||
| E1-F3 | TGAGTACGAACTTATGTACTCAT | E | leiterartiges Muster | |||
| E1-B3 | TTCAGATTTTTAACACGAGAGT | |||||
| E1-FIP | ACCACGAAAGCAAGAAAAAGAAGTTCGTTTCGGAAGAGACAG | |||||
| E1-BIP | TTGCTAGTTACACTAGCCATCCTTAGGTTTTACAAGACTCACGT | |||||
| E1-LoopB | GCGCTTCGATTGTGTGCGT | |||||
| E1-LoopF | CGCTATTAACTATTAACG | |||||
| N2-F3 | ACCAGGAACTAATCAGACAAG | n | ||||
| N2-B3 | GACTTGATCTTTGAAATTTGGATCT | |||||
| N2-FIP | TTCCGAAGAACGCTGAAGCGGAACTGATTACAAACATTGGCC | |||||
| N2-BIP | CGCATTGGCATGGAAGTCACAATTTGATGGCACCTGTGTA | |||||
| N2-LoopF | GGGGGCAAATTGTGCAATTTG | |||||
| N2-LoopB | CTTCGGGAACGTGGTTGACC | |||||
| RT-qPCR38 | ||||||
| 2019-nCoV_N1-F | GACCCCAAAATCAGCGAAAT | n | 72 Bp | |||
| 2019-nCoV_N1-R | TCTGGTTACTGCCAGTTGAATCTG | |||||
| 2019-nCoV_N1-P | 5'-FAM-ACC CCG CAT TAC GTT TGG TGG ACC-BHQ1-3' | |||||
| 2019-nCoV_N2-F | TTACAAACATTGGCCGCAAA | n | 67 Bp | |||
| 2019-nCoV_N2-R | GCGCGACATTCCGAAGAA | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CAG CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | ACAGGTACGTTAATAGTTAATAGCGT | E | 113 Bp | |||
| E1_Sarbeco_R | ATATTGCAGCAGTACGCACACA | |||||
Tabelle 2: Primer für RT-LAMP, RT-qPCR und RT-PCR. Primersequenzen, Zielgen, erwartete Produktgröße und entsprechende Referenz werden aufgelistet. Abkürzungen: RT-LAMP = Reverse-Transkriptionsschleifen-vermittelte isotherme Verstärkung; RT-PCR = Reverse-Transkriptions-Polymerase-Kettenreaktion; bp = Basenpaare; RT-qPCR = quantitative RT-PCR in Echtzeit; E1 = Hüllgen; N2 = Nukleokapsid-Gen; F = Vorwärtsgrundierung; R = umgekehrte Grundierung; P = Sonde ; FIP = vordere innere Grundierung; BIP = rückwärts innere Grundierung; F3 = Vorwärtsverschiebungsprimer; B3 = Rückwärtsverschiebungsprimer; LoopF = Vorwärtsschleifenprimer; LoopB = Rückwärtsschleifenprimer.
| Reagenz | Volumen (μL) |
| WarmStart Colorimetric LAMP 2X Master Mix mit UDG | 12.5 |
| N2 Primer Mix (20x) | 1.25 |
| E1 Primer Mix (20x) | 1.25 |
| Guanidinhydrochlorid (600 mM)* | 2.5 |
| Ziel-RNA | 5 |
| NukleasefreiesH2O | 2.5 |
| Gesamtvolumen | 25 |
Tabelle 3: Reaktionsmaster-Mix für multiplex kolorimetrische RT-LAMP. (*) Es wurde gezeigt, dass Guanidinhydrochlorid die Empfindlichkeit und Geschwindigkeit der Reaktion durch einen uncharakteristischen Mechanismus erhöht28. Abkürzungen: LAMP = schleifenvermittelte isotherme Verstärkung; UDG = Uracil-DNA-Glykosylase; N2 = Nukleokapsid-Gen; E1 = Hüllgen; DEPC = Diethylpyrocarbonat.
| Zugelassene Bürger | Bürger, die ein Kit angefordert haben | Gelieferte Kits | Zurückgegebene sampled Kits | Tage für die Probenahme | % Komplette Kits | % Unvollständige Kits | Verarbeitete Proben | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | bedeuten | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| Median | 3 | |||||||
Tabelle 4:Abstrich für SARS-CoV-2 nach Den Zahlen. Reichweite und Stichprobenerfolgsraten. Abkürzung: SARS-CoV-2 = schweres akutes respiratorisches Syndrom Coronavirus 2.
Diskussion
Citizen Scientist Engagement. Citizen Scientists wurden rekrutiert, um Oberflächen im gesamten San Diego County abzutupfen, um das Vorhandensein von SARS-CoV-2 in der städtischen Umgebung zu untersuchen und nachzuweisen. Die Mehrzahl der gelieferten Probenahmekits (70 %) wurden ins Labor zurückgebracht, und von diesen waren fast alle Proben vollständig (91%) (Abbildung 3A,B und Tabelle 4). Freiwillige konnten die Lieferung / Abholung des Kits einfach über die webbasierte Plattform anfordern, und die Lieferroutenplanungssoftware informierte die Bürgerwissenschaftler über die geschätzten Ankunftszeiten, beides wahrscheinlich wichtige Faktoren für den beobachteten Erfolg. Die durchschnittliche Zeit von der Auslieferung des Kits an den Citizen Scientist bis zur Rückgabe an das Labor betrug 8 Tage, mit einem Median von 3 Tagen und einem Bereich von 1-64 Tagen (Abbildung 3A und Tabelle 4). Häufigere Erinnerungen an Freiwillige würden diese Verzögerung wahrscheinlich reduzieren.
Die Datenerfassungsplattform wurde von einer großen Mehrheit der Nutzer (73%) (Tabelle 4). Während die Bemühungen der Citizen Scientists nicht gemessen wurden, zeigten Feldtests, dass die Datenerfassungsplattform den Aufwand und die Zeit, die für den ordnungsgemäßen Abschluss der Probenentnahme erforderlich sind, erheblich reduzierte. So förderte die Reduzierung des Buchhaltungsaufwands das Engagement von Citizen Scientist. Die webbasierte Plattform sollte demografische Einschränkungen überwinden, indem sie einen mehrsprachigen neuronalen maschinellen Übersetzungsdienst bereitstellt und grafische und audiovisuelle Protokolle in Englisch und Spanisch bereitstellt. Dies war nur teilweise erfolgreich, da weniger Proben sowohl aus der South Bay als auch aus dem North County gesammelt wurden, wo der größte Teil der hispanischen / lateinamerikanischen Bevölkerung des Countys45lebt. Diese Gebiete beherbergten auch 63% (1.700 Fälle pro 100.000) der gesamten COVID-19-Fälle in San Diego County mit der höchsten Prävalenz der Krankheit46 und der Rate der Krankenhausaufenthalte (62%)47,48. Obwohl die meisten Proben aus Central County stammten, wurde eine repräsentative Anzahl aus den am stärksten von COVID-19 betroffenen Bezirken gesammelt und nur ein kleiner Bruchteil der Proben war positiv, was darauf hindeutet, dass Oberflächenreservoirs von SARS-CoV-2 in der städtischen Umgebung relativ selten sind.
Probenverarbeitung. Probenahmeabstriche wurden mit SDS benetzt, das das Virus inaktivierte, indem es seine Hülle störte und die nackte RNA stabilisierte, indem es RNasen32entfaltete. Praktischerweise reinigte das Reinigungsmittel im Tupfer während der Entnahme die beprobte Oberfläche. Umweltproben enthalten oft sehr geringe Mengen an RNA. Um die Rückgewinnung zu maximieren, wurde die RNA-Isolierung mit einer GITC-basierten, säulenfreien, rohen Extraktionsmethode durchgeführt. GITC, ein starkes chaotropes Mittel, stört die Wasserstoffbrückenbindungen, die die Proteinfaltung aufrechterhalten (dh hydrophobe Wirkung). Diese Wirkung führt zur Inaktivierung viraler Partikel, und die RNA bleibt aufgrund der Hemmung der RNAsen34,35,36stabil . Die GITC-Lösung hielt die Stabilität der RNA-Proben ohne strenge Kühlkettenüberlegungen aufrecht, was es den Bürgern ermöglichte, die Proben bei Raumtemperatur zu halten, wenn kein Gefrierschrank für die bereitgestellten Eisbeutel verfügbar war. Um die potenzielle Gefahr zu verringern, die dieses Reagenz bei direktem Haut- oder Schleimhautkontakt darstellt, wurden die Bürger durch die Aufnahme eines Sicherheitsdatenblatts im Kit auf diese Risiken aufmerksam gemacht und ein Warnsiegel in die Box mit den Röhrchen gelegt.
Die rohe GITC-Chloroform-Extraktionsmethode unterstützte die Rückgewinnung von Spuren von RNA aus den Tupfern, und wie die Amplifikation von Spike-Proben zeigte, blieben Inhibitoren nach der Extraktion selten in den Proben bestehen. Proben, die negativ für SARS-CoV-2 waren und keine RT-LAMP-Hemmung zeigten, stellten echte Negative dar oder hatten eine niedrigere Kopienzahl als die LOD bei 100% Häufigkeit. Umgekehrt bedeutet der Nachweis viraler RNA auf einer Oberfläche nicht direkt das Risiko einer Übertragung durch Kontakt, da die Infektiosität des Virus aus positiven Proben getestet werden muss. Ein unverzügliches Screening der Umwelt, das nicht durch die Verfügbarkeit von hochentwickelten Vorräten oder hochqualifiziertem Personal eingeschränkt ist, ist von entscheidender Bedeutung, um zu beurteilen, ob Oberflächen ein Viralreservoir darstellen, und um bessere direkte Präventions- und Eindämmungsbemühungen zu unternehmen.
RT-LAMP wurde als die beste Methode ausgewählt, die für die vorgeschlagene Detektionspipeline geeignet ist. Es erwies sich als eine schnelle und kostengünstige Methode, die gegen die meisten der verbleibenden Inhibitoren sehr resistent war und so empfindlich und spezifisch war wie andere RT-qPCR-Methoden. Aufgrund ihres Einsatzes in klinischen Umgebungen während der SARS-CoV-2-Pandemie wurde die Verfügbarkeit von RT-qPCR-Kits durch die globale Nachfrage beeinflusst. Darüber hinaus waren RT-qPCR-Techniken - selbst solche, die formuliert wurden, um Inhibitoren zu widerstehen - empfindlich gegenüber Substanzen, die in den Umweltproben enthalten waren, die von einer Pilotkohorte von Bürgerwissenschaftlern gesammelt wurden, selbst nach der Verwendung anderer gängiger Strategien zur Verringerung der Inhibitorkonkurrenz um die Enzymbindung49. Diese Ergebnisse werden durch eine aktuelle Studie bestätigt, die beide Methoden zum Nachweis von SARS-CoV-2 auf Tupferproben von Süßigkeiten, die von COVID-19-Patienten behandelt wurden, verglich und eine Übereinstimmung der Ergebnisse von über 83% mit einer um 25% geringeren Hemmung in Proben fand, die von RT-LAMP15analysiert wurden. Darüber hinaus reduzierte die GITC-Chloroform-Rohextraktion in Verbindung mit RT-LAMP die Kosten für Reagenzien und -vorräte um 42% im Vergleich zur RNA-Kit-Extraktion und RT-qPCR (Table of Materials).
Diese Methode ermöglichte eine Hochdurchsatzanalyse von Tausenden von Oberflächentupferproben. Bis zu 80 Pools, die 640 Proben repräsentieren, wurden in 2 Tagen von der RNA-Extraktion bis zum SARS-CoV-2-Nachweis durch RT-LAMP verarbeitet. Das vorgeschlagene Protokoll ist semiquantitativ, beschränkt sich auf den Nachweis viraler RNA und weist nicht auf das Vorhandensein infektiöser Viruspartikel hin. Weitere Analysen sind erforderlich, um das Risiko einer Übertragung von SARS-CoV-2 von infizierten Fomiten an den abtupfern Oberflächen zu bewerten.
Diese Studie stellt ein Protokoll zur schnellen Einrichtung einer Teststrategie vor, die einen effektiven Workflow bei einem Gesundheitsnotfall mit einer übertragbaren Krankheit umfasst. Das vorgeschlagene Probenahmeprotokoll ist einfach und verwendet Verbrauchsmaterialien, die üblicherweise in Haushalten zu finden sind, und die Viralnachweismethode wird auf Geräten durchgeführt, die in grundlegenden Laborumgebungen wie einem Wasserbad anstelle eines Thermocyclers verfügbar sind. Die Kosten für RT-LAMP-Reagenzien sind deutlich niedriger als die für RT-qPCR und weniger anfällig für Szenarien mit hoher globaler Nachfrage. Diese Studie dient als Rahmen für die Bewertung von umweltbedingten Virusreservoirs bei zukünftigen Epidemienausbrüchen und globalen Pandemien.
Offenlegungen
Alle Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Wir danken den Forschern des Viral Information Institute (VII), Dr. Anca M. Segall, Willow Segall, Patricia L. Rohwer, Gary Rohwer, Cary L. Rohwer, Magda Silvia Pinetta, Elizabeth Cruz Cano, Dr. Gregory Peters, Dr. Stuart A. Sandin und Dr. Jennifer Smith, dass sie sich die Zeit genommen haben, zahlreiche Proben zu sammeln. Wir danken auch Dr. Rob Knight, Dr. Jack Gilbert, Dr. Pedro Balda-Ferre und Dr. Sarah Allard von der Abteilung für Pädiatrie an der School of Medicine University of San Diego California (UCSD) für die Erleichterung positiver Kontrollen und nützliches Feedback. Wir danken Stacey Carota (SDSU College of Sciences) und Gina Spidel (SDSU) für die logistische Unterstützung und Juan Rodríguez für die Kunst und grafische Gestaltung des Sampling-Protokolls. Wir danken allen Teilnehmern für ihr Engagement und ihren Einsatz für dieses Projekt in sehr schwierigen Zeiten. Diese Arbeit wurde durch eine großzügige Spende von Dr. Jo Ann Lane (SDSU College of Sciences) und der National Science Foundation RAPID: Environmental Reservoirs of SARS-CoV-2 Grant (Award Number: 2030479) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
Referenzen
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten