Method Article
Tamponare l'ambiente urbano - Una pipeline per il campionamento e il rilevamento della SARS-CoV-2 dai bacini ambientali
In questo articolo
Riepilogo
Un progetto di citizen science è stato progettato per reclutare residenti a San Diego per raccogliere campioni ambientali per SARS-CoV-2. È stata creata una piattaforma multilingue basata sul Web per l'invio dei dati utilizzando un'interfaccia di dispositivo mobile intuitiva. Un sistema di gestione delle informazioni di laboratorio ha facilitato la raccolta di migliaia di campioni geograficamente diversi con il monitoraggio dei risultati in tempo reale.
Abstract
Per controllare la trasmissione comunitaria della sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) durante la pandemia globale del 2020, la maggior parte dei paesi ha implementato strategie basate su test umani diretti, copertura del viso e disinfezione superficiale. Nell'ipotesi che la principale via di trasmissione includa aerosol e goccioline respiratorie, gli sforzi per rilevare la SARS-CoV-2 nei fomiti si sono concentrati su luoghi sospettati di alta prevalenza (ad esempio reparti ospedalieri, navi da crociera e sistemi di trasporto di massa). Per indagare sulla presenza di SARS-CoV-2 sulle superfici dell'ambiente urbano che raramente vengono pulite e raramente disinfettate, 350 cittadini sono stati arruolati dalla grande contea di San Diego. In totale, questi scienziati cittadini hanno raccolto 4.080 campioni. È stata sviluppata una piattaforma online per monitorare la consegna e il ritiro dei kit di campionamento, nonché per raccogliere i dati di esempio. I kit di campionamento sono stati costruiti principalmente con forniture disponibili nei negozi stressati dalla pandemia. I campioni sono stati elaborati utilizzando reagenti di facile accesso nonostante la ricorrente carenza di approvvigionamento. I metodi utilizzati erano altamente sensibili e resistenti agli inibitori che sono comunemente presenti nei campioni ambientali. I metodi sperimentali di progettazione e lavorazione proposti sono riusciti a coinvolgere numerosi scienziati cittadini che hanno effettivamente raccolto campioni da diverse aree di superficie. Il flusso di lavoro e i metodi qui descritti sono rilevanti per l'indagine sull'ambiente urbano per altri virus, che sono di interesse per la salute pubblica e rappresentano una minaccia per le future pandemie.
Introduzione
Si ritiene che la SARS-CoV-2 sia trasmessa principalmente attraverso l'inalazione di aerosol e goccioline contaminati dal contattodiretto con individui infetti 1,2,3,4. Tuttavia, durante le fasi iniziali della pandemia globale di COVID-19, gli sforzi per controllare la trasmissione della SARS-CoV-2 si sono concentrati fortemente sulla disinfezione delle superfici, sul lavaggio delle mani e sulla sanificazione. Entro la fine del 2020, le linee guida di trasmissione dell'Organizzazione Mondiale della Sanità (OMS)5 e dei Centri statunitensi per il controllo e la prevenzione delle malattie (CDC)6 consideravano la trasmissione aerea un pericolo principalmente quando era a stretto contatto (<2 m) con una persona infetta o in presenza di procedure mediche che generano aerosol. L'autoimulazione dopo il contatto con superfici contaminate o l'inalazione di fomiti aerosolizzati non è ancora stata esclusa come via di trasmissione della SARS-CoV-2.
Sono stati segnalati casi di COVID-19 in cui la trasmissione aerea sembra improbabile7,8. I virioni SARS-CoV-2 rimangono infettivi sul rame fino a 8 ore, su cartone e acciaio inossidabile fino a 24 ore e sulla plastica fino a 48 h9. Nelle cabine delle navi da crociera, l'RNA SARS-CoV-2 è stato rilevato 17 giorni dopo che i passeggeri erano partiti7. Campioni di aria e superficie provenienti da ospedali e sistemi di transito di massa sono risultati positivi alla SARS-CoV-2 e ad altri coronavirus8,10,11,12,13,14. Uno studio condotto sulla confezione esterna di caramelle di Halloween gestite da pazienti covid-19 asintomatici e moderata / leggermente sintomatici, ha concluso che la combinazione di lavaggio a mano da parte del gestore e lavaggio delle caramelle con sapone per le mani ha ridotto l'RNA SARS-CoV-2 al di sotto dei livellidi soglia 15.
Diversi metodi per la diagnostica SARS-CoV-2 sono stati pubblicati sulla base della reazione a catena della polimerasi a trascrizione inversa in tempo reale (RT-qPCR)16,17, amplificazione isotermica mediata ad anello di trascrizione inversa (RT-LAMP)11,18,19,20,21e crispr-castecnologie 18,19,22,23. La maggior parte richiede kit di estrazione dell'RNA che sono spesso a corto di offerta durante i periodi di significativa domanda globale e pochissimi sono stati utilizzati per lo screening ambientale del virus24. Il rilevamento dell'RNA SARS-CoV-2 utilizzando RT-LAMP ha dimostrato di essere più dell'83% concorde con l'utilizzo di RT-qPCR. Inoltre, RT-LAMP ha comportato una riduzione del 25% dei risultati inconcludenti rispetto a RT-qPCR15.
RT-LAMP è una tecnica semplice che utilizza una transcriptasi inversa per sintetizzare cDNA da un modello di RNA25, seguita da una DNA polimerasi con forte attività di spostamento del filamento che sintetizza il DNA a temperatura costante (cioè amplificazione isotermica)26. Una maggiore specificità del rilevamento del genoma virale si ottiene utilizzando quattro o sei primer che riconoscono sei o otto regioni del DNA bersaglio. L'amplificazione è iniziata da un primer interno e produce una struttura di DNA semi-a doppio filamento. Il filamento principale viene quindi amplificato da un primer esterno. Queste amplificazioni vengono ripetute per i primer inversi. I primer interni ed esterni su entrambe le estremità hanno un sito interno auto-complementare inverso che forma un ciclo nel prodotto diamplificazione 26,27. Nello spostamento del filamento isotermico, la sintesi asincrona del DNA genera elevate quantità di prodotto amplificato dove la polimerizzazione continua amplifica il segnale di appena 10 copie per reazione11,20,28. Il mix colorimetrico RT-LAMP è debolmente tamponato e utilizza il rosso fenolo come indicatore di pH. Poiché la polimerasi incorpora un nucleotide, rilascia un protone, e abbastanza protoni cambieranno il pH della soluzione e il suo colore dal rosa al giallo11,20,28,29.
RT-LAMP è stato sviluppato per il rilevamento di malattie trasmesse dalle zanzare in strutture sanitarie periferiche che mancano di laboratoricompletamente attrezzati 25 e per il rapido rilevamento di altri virus dell'RNA come il virus dell'immunodeficienzaumana 30. Le popolazioni più vulnerabili in epidemie , secondo la definizione dell'OMS, spesso non dispongono di risorse economiche sufficienti e delle attrezzature adeguate per effettuare il rilevamento (Agenda globale delle Nazioni Unite per la salute pubblica). Nell'attuale pandemia di SARS-CoV-2, forniture come tamponi e reagenti di livello medico per kit di estrazione dell'RNA non sono stati in grado di soddisfare la domanda globale, specialmente nei paesi non manifatturieri. Il protocollo proposto utilizzava un'estrazione di RNA grezzo a base di tiocianato di guanidinio (GITC), che preservava efficacemente l'RNA in modo indipendente dalla catena del freddo e riduceva significativamente la persistenza degli inibitori dal campione. Inoltre, il protocollo di estrazione GITC-cloroformio si basa sulla separazione dell'RNA dal DNA e dalle proteine seguita dalle rispettive precipitazioni, consentendo il recupero della maggior parte del materiale genetico. Questi vantaggi superano i rischi potenziali degli scienziati cittadini che maneggiano la sostanza chimica se vengono adottate misure per informarli adeguatamente dei rischi.
Il flusso di lavoro proposto utilizza materiali e reagenti di uso generale. Richiede attrezzature disponibili in ambienti di laboratorio di base, spesso rurali. Questi metodi sono economici, altamente resistenti agli inibitori spesso presenti in campioni ambientali o campioni che non possono essere elaborati con kit di estrazione ed eliminano la necessità di un termociclometro ad alta precisione. Questo studio presenta una pipeline per il campionamento e il rilevamento della SARS-CoV-2 da serbatoi ambientali su superfici comunemente toccate e raramente disinfettate delle famiglie e dell'ambiente urbano.
Protocollo
Per un elenco dettagliato dei reagenti e delle forniture, inclusi i numeri di catalogo, il produttore e i costi corrispondenti, vedere la tabella dei materiali.
1. Campionamento dell'ambiente urbano
- Sensibilizzazione degli scienziati cittadini
- Recluta scienziati cittadini utilizzando un invito all'azione diretto e chiaro rilasciato tramite i media locali e sociali. Crea un handle di social media (ad esempio, #swab4corona ) perconnettere l'argomento attraverso i contenuti dei social media.
- Creare un account e-mail per la comunicazione diretta tra il team di laboratorio e ogni scienziato cittadino, gestito da una persona fluente nelle principali lingue della regione di interesse (ad esempio, spagnolo e inglese per la contea di San Diego).
- Creare una piattaforma di gestione dei campioni (SMP) sicura basata sul Web per fungere da database, un sistema di gestione delle informazioni di laboratorio (LIMS) e per comunicare con gli scienziati cittadini.
NOTA: L'SMP fornisce una posizione centralizzata in cui gli utenti richiedono un kit, accedono ai protocolli di raccolta di esempio, inviano metadati di esempio e richiedono un ritiro per i kit di esempio completati. - Creare un collegamento all'SMP (ad esempio, https://demo.covidsample.org/) (Figura 1) affinché le persone possano presentare domanda per partecipare allo sforzo di campionamento ambientale rispondendo alle domande relative alla biosicurezza specificate in un modulo online.
- Accesso sicuro all'SMP utilizzando un'interfaccia di programmazione delle applicazioni di autenticazione facilitata da un provider di servizi cloud computer. Concedere agli utenti approvati l'accesso all'SMP.
NOTA: fare riferimento alla letteratura per una descrizione del protocollo OAuth 2.031 per l'autenticazione e l'autorizzazione. Fornisce un processo di accesso senza attriti per i volontari scienziati cittadini. Consente inoltre agli utenti di accedere con un account esistente, eliminando la necessità di creare una soluzione di accesso personalizzata e gestire le credenziali utente, risparmiando una quantità significativa di tempo e incoraggiando la partecipazione. Secondo recenti rapporti, il servizio di posta elettronica gratuito disponibile per il fornitore di servizi cloud di computer scelto ha circa 1,5 miliardi di utenti di posta elettronica attivi mensilmente; richiedere un account di posta elettronica da questo fornitore di servizi per la partecipazione non è considerato un fattore scoraggiante. - Spiegare l'obiettivo dello studio e le considerazioni sulla biosicurezza agli scienziati cittadini dell'SMP prima di richiedere il loro primo kit. Fornire un plug-in multilingue per consentire la navigazione in una qualsiasi delle lingue disponibili da un servizio di traduzione automatica neurale multilingue facilitato da un provider di servizi di cloud computer.
- Includere nella sezione di campionamento protocolli grafici e audiovisivi in inglese e spagnolo.
- Assegnare un identificatore univoco a ogni kit e progettare l'interfaccia utente in modo che utilizzi i pulsanti collegati all'ID di esempio per semplificare il processo di immissione dei dati (Figura 1A).
- Utilizza un software di pianificazione del percorso di consegna con un'applicazione per dispositivi mobili che deve essere utilizzata dai conducenti per ottimizzare i percorsi di consegna / ritiro e informare gli scienziati cittadini degli orari di arrivo stimati accurati.
- Creare la piattaforma LIMS su uno stack di servizi Web PHP e ospitarla su una piattaforma di hosting commerciale (il sistema operativo suggerito, il software del server Web e il software di database sono specificati nella tabella dei materiali).
- Fornire un'interfaccia applicativa sicura basata sul Web per consentire al personale di laboratorio di gestire i dati in modo rapido e semplice nel LIMS. Fornire la visualizzazione dei dati utilizzando un'interfaccia di programmazione delle applicazioni di charting dei dati facilitata da un provider di servizi cloud computer.
- Visualizza i dati geospaziali utilizzando un'interfaccia di programmazione di applicazioni geospaziali facilitata da un provider di servizi cloud computer. Memorizzare i dati inviati al LIMS attraverso l'SMP per facilitare (1) l'archiviazione centralizzata dei dati di progetto; 2) monitoraggio dei flussi di lavoro di campionamento/elaborazione dei dati; e (3) gestione della logistica della distribuzione di kit campione agli scienziati cittadini.
- Proteggere i metadati inviati utilizzando le procedure consigliate (ad esempio, https://demo.covidsample.org/).
- Pre-caricamento di informazioni quali l'ID del kit di esempio, l'ID di esempio, la data, l'ora e le coordinate GPS (Global Positioning System) (raccolte automaticamente da un'immagine del sito) per consentire la conformità al tipo di dati e ridurre al minimo l'invio di dati erronei o mancanti da parte dell'utente (Figura 1B). Includi i seguenti campi da riempire manualmente e rapidamente (<1 min) da parte dello scienziato cittadino: data e ora di raccolta, una breve descrizione del luogo e un'immagine del sito di campionamento.
- Sanificare tutti i dati caricati e convalidare per il tipo di dati. Ad esempio, convalidare i dati delle immagini caricati dagli utenti per selezionare i file .jpg, rinominarli con ID di esempio per un'associazione rapida con l'esempio e archiviare le immagini caricate in un percorso sicuro separato non accessibile agli utenti.
- Attiva l'opzione per richiedere la consegna e il ritiro del kit quando tutti i campioni (16) sono stati completati. Inoltre, attivare l'opzione per richiedere la consegna di un nuovo kit al momento del ritiro del precedente (Figura 1A).
NOTA: Per i volontari che preferiscono una piattaforma non basata sul web e per coloro che sono preoccupati di divulgare la loro posizione GPS (ad esempio, i membri della comunità preoccupati per il loro stato migratorio), i kit possono essere consegnati in un luogo di incontro concordato e i volontari hanno chiesto di registrare una versione scritta della raccolta dei dati. Per la comunicazione tra il laboratorio e ogni scienziato cittadino, avere un membro bilingue del progetto disponibile per telefonate e messaggi.
- Tampone per Corona
- Identificare un lavanza epidemiologicamente rilevante per lo sforzo di campionamento.
- Creare un kit contenente tutte le forniture di campionamento, compresi i necessari dispositivi di protezione individuale (ad esempio maschera, guanti), un protocollo di campionamento e informazioni pertinenti sulla biosicurezza (figura 2). Pre-etichettare ogni tubo con l'identificatore univoco assegnato (ID campione).
- Tampone raramente disinfettato superfici che sono esposte a fomiti aerosolizzati nelle famiglie e nell'ambiente urbano.
- Indossare la maschera fornita in pubblico e un nuovo paio di guanti per la raccolta di ogni campione per evitare la contaminazione incrociata. Dopo aver terminato il campionamento, utilizzare il disinfettante per le mani fornito.
- Bagnare un tampone assorbente in poliestereda 1 cm 2 (ad esempio, tamponi per mop) con un detergente (ad esempio, solfato di dodecil di sodio allo 0,5%) per inattivare il virus interrompendone l'involucro e stabilizzare l'RNA nudo inducendo lo spiegamento delle RNasi32.
- Tamponare una superficie di 10 cm2. Aiutati da uno stuzzicadenti, immergere completamente ogni tampone campione nel corrispondente tubo preetichettiato contenente 200 μL di soluzione di tiocianato di guanidinio (GITC). Conservare i tubi a 4 °C fino a quando non vengono trasportati in laboratorio. Una volta che i campioni arrivano in laboratorio, conservarli a -80 °C.
NOTA: GITC è irritante tossico; evitare il contatto con la pelle. La soluzione GITC è semplice da preparare da comuni prodotti chimici di laboratorio, per la ricettavedi 33,34. Inattiva il virus, stabilizza l'RNA denaturando la RNasi34,35,36e stabilizza i campioni a temperatura ambiente. Il kit, tuttavia, include confezioni di ghiaccio per mantenere i campioni freddi senza la necessità di utilizzare frigoriferi domestici per la conservazione.
2. Rilevamento SARS-CoV-2
- Isolamento totale dell'RNA
- Disinfettare superfici, attrezzature e pipettor con una soluzione di solfato di rame da 2 mM e perossido di idrogeno al 3%; seguita da una soluzione di candeggina al 10%, bicarbonato di sodio da 90 mM, 5% SDS e 2,5% di NaOH. Pulire accuratamente con acqua distillata seguita dal 75% di etanolo.
NOTA: Queste soluzioni sono un'alternativa alle soluzioni disponibili in commercio. - Scongelare i campioni sul ghiaccio. Campioni di vortice per 2 min a media velocità.
- Per aumentare la velocità dello screening, elaborare i campioni in pool. Se una piscina è positiva, estrarre l'RNA di ciascun campione in modo indipendente per trovare il campione o i campioni positivi. Unire i campioni di ciascun kit di campionamento (16 in totale) in 2 pool di 8 campioni.
NOTA: Avere 8 campioni per piscina significa che solo 2 piscine devono essere elaborate per kit. Se un pool è positivo, i singoli campioni vengono ri-elaborati per l'analisi RT-LAMP individuale. Ciò riduce tempi, costi e reagenti. - Pool 50 μL di ciascuno degli 8 campioni in un tubo di microcentrifugo (volume totale 400 μL); salvare il campione rimanente a -80 °C. Aggiungere 0,2 volumi (80 μL) di cloroformio, vortice per 15 s, quindi incubare per 20 minuti a 4 °C. Centrifuga a 13.000 × g per 20 min a 4 °C.
- Trasferire lo strato acquoso (liquido trasparente) in un nuovo tubo di microcentrifugo. Conservare l'interfaccia rimanente e il liquido rosa nel congelatore a -80 °C; queste frazioni contengono DNA eproteine 33,36.
- Aggiungere un volume uguale di isopropanolo (~200 μL) e 2,6 μL di coprecipio di glicogeno (15 mg di mL-1)37. Mescolare bene e incubare a -20 °C per almeno 1 h, seguito da 4 °C per 10 minuti per precipitare l'RNA.
NOTA: Il protocollo può essere messo in pausa qui incubando campioni a -20 °C durante la notte invece di 1 h. - Centrifuga a 13.000 × g per 20 min a 4 °C. Rimuovere il supernatante senza disturbare il pellet. Rimescolare il pellet in 50 μL di acqua trattata con dietilpirrocarbonato (DEPC) e aggiungere un volume uguale (50 μL) di acetato di ammonio 5 M esometo privo di RNasi e 2,5 volumi (250 μl) di etanolo al 100%7,38.
NOTA: Gli ioni di ammonio inibiscono la polinucleotide chinasi se utilizzati in un processo avalle 38. La miscela precipita l'RNA lasciando trifosfati e oligosaccaridi deossinucleoside nellasoluzione 38. - Mescolare bene e incubare a -20 °C per almeno 1 h, seguito da 4 °C per 10 minuti per precipitare l'RNA.
NOTA: Il protocollo può essere messo in pausa qui incubando campioni a -20 °C durante la notte invece di 1 h. - Centrifuga a 13.000 × g per 20 min a 4 °C. Lavare il pellet con 1 mL di etanolo freddo (-20 °C), appena fatto al 75%. Centrifuga a 8.000 × g per 5 min a 4 °C. Rimuovere il supernatante con una pipetta P10 per evitare di disturbare il pellet.
- Asciugare all'aria il pellet per 10-15 minuti fino a quando non rimane etanolo. Rimescolare il pellet in 50 μL di acqua trattata con DEPC, aggiungere 5 μL di tampone di 10x DNasi + 1μL di DNasi (2 unità μL-1)e incubare a 37 °C per 30 min.
- Aggiungere 0,1 volumi (5,6 μL) di reagente di inattivazione della DNasi, incubare a temperatura ambiente per 5 minuti e mescolare delicatamente ogni minuto. Centrifuga a 13.000 × g per 2 min e trasferisci il supernatante su un nuovo tubo (~ 50 μL). Posizionare immediatamente il tubo sul ghiaccio durante la preparazione delle reazioni RT-qPCR o RT-LAMP o conservare in un congelatore a -20 °C.
NOTA: Eseguire l'isolamento dell'RNA in una stanza priva di ampliconi per evitare la contaminazione da riporto.
- Disinfettare superfici, attrezzature e pipettor con una soluzione di solfato di rame da 2 mM e perossido di idrogeno al 3%; seguita da una soluzione di candeggina al 10%, bicarbonato di sodio da 90 mM, 5% SDS e 2,5% di NaOH. Pulire accuratamente con acqua distillata seguita dal 75% di etanolo.
- Amplificazione isotermica mediata da loop di trascrizione inversa multiplex (RT-LAMP)
- Preparare una soluzione di miscela di primer 20X (tabella 1) per ogni insieme di primer (tabella 2). Preparare la miscela di reazione RT-LAMP (tabella 3) a temperatura ambiente con un volume in eccesso del 10% per tenere conto della perdita di pipettazione.
NOTA: Il master mix colorimetrico LAMP 2X con glicosilasi uracil-DNA termolabile antartica (UDG) previene l'amplificazione della contaminazione da DNA da reazioniprecedenti 20,28. - Vortice e spin giù la miscela. Distribuire 20 μL della miscela in ogni tubo di reazione: campione, chiodato, controllopositivo e controllo negativo. Incubare le reazioni nei tubi a temperatura ambiente per 10 minuti per consentire all'UDG di agire su una potenziale contaminazione da riporto.
- Aggiungere 5 μL di RNA alla reazione del campione, 5 μL di RNA + 2,5 μL (450 copie) di RNA sintetico SARS-CoV-2 alla reazione chiodata, 2,5 μL (450 copie) di SARS-CoV-2 sintetico alla reazione di controllo positivo e 5 μL di H2O alla reazione di controllo negativo. Mescolare bene, e spin down le reazioni; scongelare tutto l'RNA sul ghiaccio.
- Mentre il termociclo, o il bagno d'acqua, viene riscaldato a 65 °C, lasciare che tutte le reazioni rimangano a temperatura ambiente. L'ODG sarà disattivato a >50 °C. Posizionare le reazioni nel termociclo (utilizzare il coperchio termico), incubare a 65 °C per 40 minuti e lasciare che le reazioni raggiungano la temperatura ambiente (~ 22 °C per 5 minuti) o raffreddare sul ghiaccio per 1 minuto. Analizzare i risultati utilizzando la funzione colorimetrica (semplice osservazione) o eseguendo i prodotti in un gel di agarosio.
NOTA: Sebbene il limite di rilevamento (LOD) sia di 10 copie per reazione, la frequenza di rilevamento aumenta man mano che il numero di copie si avvicina a 500 copie per reazione (Figura 3A). Per l'osservazione colorimetrica, si noti che un risultato negativo è indicato dal rosa (pH = 8,8), mentre un risultato positivo è indicato dal giallo (pH = 5) (Figura 3B). L'opzione colorimetrica evita l'apertura dei tubi RT-LAMP dopo l'amplificazione, che ridurrà il volume dei prodotti RT-LAMP nell'ambiente di lavoro e la contaminazione da riporto. Per l'elettroforesi gel, preparare un gel di agarosio all'1,5% con una macchia di gel di DNA 1X nel tampone Tris/Borate/EDTA (TBE) allo 0,5%. Caricare 25 μL della reazione + 5 μL di colorante di carico 6X in ogni pozzo. Eseguire il gel a 100 V per 60 minuti. Non è necessario un marcatore molecolare in quanto campioni positivi mostrano un modello di scala (Figura 3C).
- Preparare una soluzione di miscela di primer 20X (tabella 1) per ogni insieme di primer (tabella 2). Preparare la miscela di reazione RT-LAMP (tabella 3) a temperatura ambiente con un volume in eccesso del 10% per tenere conto della perdita di pipettazione.
Risultati
Campioni raccolti da scienziati cittadini per l'individuazione della SARS-CoV-2. Durante un periodo di 8 mesi (da metà marzo alla terza settimana di novembre 2020), sono stati approvati 482 cittadini per partecipare a questo progetto, di cui 350 (73%) richiesto un kit. Sono stati consegnati in totale 362 kit (cioè alcuni partecipanti hanno richiesto più kit) e 246 (70%) sono stati restituiti (figura 4A,B). Tutti i 4.080 campioni contenuti in questi kit sono stati elaborati. I siti di raccolta sono stati distribuiti nei distretti costieri, centro-settentrionali, centrali e meridionali della contea, così come alcuni nei distretti orientali (Figura 4A). Questi distretti hanno la più alta densità di popolazione della contea di San Diego e i casi di COVID-19 più documentati, come riportato dalla San Diego Human Health Service Agency39.
I cittadini hanno richiesto il ritiro della maggior parte dei kit campionati (cioè un tasso medio di successo: 70,4%). Ogni giorno sono stati richiesti 1-16 kit e 0-14 kit sono stati restituiti al laboratorio(Figura 4B). Da un'indagine condotta presso gli scienziati cittadini è emerso che la raccolta di un kit completo (16 campioni) ha richiesto 1-3 ore distribuite in una media di 8 giorni(figura 4A e tabella 4). La maggior parte dei kit è stata completa (91,1%), il che significa che contenevano un tampone all'interno di tutti i 16 tubi campione e i corrispondenti dati di campionamento sono stati caricati sul LIMS(figura 4A e tabella 4).
Rilevamento di SARS-CoV-2 mediante estrazione GITC-cloroformio e amplificazione isotermica mediata da loop con trascrittasi multipla inversa (RT-LAMP). Per il saggio colorimetrico RT-LAMP, due serie di primer11,20,28 sono stati utilizzati per colpire il nucleocapsid (N2) e i geni dell'involucro (E1) (Tabella 2). Le sequenze riconosciute da questi primer si trovano nella stessa regione dei primer e delle sonde approvate dal CDC40 e dall'Unione Europea (UE)41 per la diagnosi umana di COVID-19 da parte di RT-qPCR. Questi risultati confermano ciò che Zhang etal. Il LOD ad una frequenza del 100% era di 500 copie per reazione di 25 μL (figura 3A,B). Nella colorimetrica RT-LAMP, i campioni positivi hanno cambiato colore dal rosa al giallo a causa di uno spostamento del pH da ~8 a 5,5 (Figura 3B). Quando la reazione è diventata arancione a numeri a bassa copia, i campioni sono stati eseguiti in un gel di agarosio dell'1,5% per confermare che erano positivi e hanno portato a un modello simile a una scala (Figura 3C). RT-LAMP è stato utilizzato per rilevare SARS-CoV-2 in campioni di RNA raggruppati.
Per controllare i falsi negativi dovuti agli inibitori di reazione, ogni campione è stato testato in una reazione aggiuntiva con picco con 500 copie di SARS-CoV-2 sintetico. Campioni positivi raggruppati sono stati dereplicati isolando l'RNA di ogni singolo campione nella piscina ed eseguiti in una reazione RT-LAMP per determinare l'identità del campione positivo. I risultati del rilevamento sono stati quindi caricati nel LIMS, dove l'ID campione univoco è stato associato alle informazioni su data, ora, coordinate GPS, sito e immagine dell'esempio.
Metodi RT-PCR tradizionali e in tempo reale: inibizione da parte dei contaminanti del campione. Per selezionare il metodo migliore adatto alla pipeline di rilevamento proposta, altri metodi di amplificazione dell'RNA sono stati testati con campioni ambientali raccolti da una coorte pilota di scienziati cittadini. Esempi dei risultati di ciascuno di questi metodi sono presentati nella figura 5 per illustrarne la sensibilità agli inibitori ambientali e al rumore del segnale di fondo a basse concentrazioni virali di numero di copie.
Sei formulazioni RT-qPCR (Table of Materials) approvate dal CDC e dall'OMS sono state testate per l'individuazione del virus su campioni ambientali. I protocolli sono stati seguiti secondo le istruzioni del produttore e le linee guida CDC per il rilevamento di SARS-CoV-2 in contesti clinici40. Le reazioni contenenti diverse concentrazioni di controlli sintetici dell'RNA SARS-CoV-2 sono state spiked in campioni di superficie tamponata dopo l'isolamento dell'RNA grezzo. Tutte le miscele master erano sensibili agli inibitori a concentrazioni LOD del controllo positivo (Figura 5A).
Per bypassare gli inibitori di queste tecnologie in tempo reale, è stato testato un tradizionale sistema RT-PCR. Un sistema RT-PCR in un solo passaggio (Table of Materials) è stato utilizzato per amplificare il gene nucleocapsid usando i set di primer N1, N2e il gene dell'involucro utilizzando il set di primer E1 approvato rispettivamente dal CDC (USA) e dall'ECDC (EU), (Tabella 2). I protocolli sono stati seguiti secondo le istruzioni del produttore e le linee guida CDC per il rilevamento di SARS-CoV-2 in contesti clinici40. I set di primer N1 e N2 progettati dal CDC producono un prodotto ~70 bp; Tuttavia, i positivi al numero di copie basse non sono stati distinti dal rumore di fondo di amplificazione del controllo negativo (Figura 5B),che ha introdotto falsi positivi nei risultati. Il prodotto dei primer E1 aveva un segnale debole a basso numero di copie ( Figura5B),introducendo falsi negativi nei risultati. Inoltre, il metodo RT-PCR testato era ancora sensibile agli inibitori presenti nei campioni ambientali (dati non mostrati).
Altri metodi sono stati sviluppati per rilevare quantità molto piccole di sequenza bersaglio. Uno di questi metodi è la Rolling Circle Amplification (RCA), dopodi clic il riconoscimento della sequenza bersaglio, RNA o DNA, da una specifica sonda lineare, una ligasi circolarizza il modello. Utilizzando primer progettati per ibridarsi con la sonda, una DNA polimerasi con attività di spostamento del filamento amplifica la sonda in una reazione isotermica42. È la sonda che ha identificato la destinazione, che viene amplificata, e non la sequenza di destinazione, il che rende questo metodo altamente sensibile43. Wang et al.44 published an RCA protocol for the direct detection of SARS-CoV-1 RNA. Il metodo è stato modificato per utilizzare primer specifici per SARS-CoV-2. Sfortunatamente, nel controllo non modello (NTC), la sonda circolarizza e produce prodotto in assenza di modello di RNA, anche quando si utilizza un'ampia varietà di ligasi, inclusa una ligasi sensibile all'SNP. In assenza di ligasi, l'NTC non ha mostrato amplificazione dalla sonda linearizzata (Figura 5C).

Figura 1: Piattaforma di campionamento basata sul Web con interfaccia dati di raccolta di campioni per dispositivi mobili. (A) È stato creato un sito Web contenente un plug-in multilingue per mediare l'interazione tra il laboratorio e gli scienziati cittadini. La piattaforma è stata utilizzata per la richiesta di consegna/ritiro del kit di esempio e l'invio di dati di esempio. La piattaforma conteneva protocolli dettagliati di campionamento grafico e audiovisivo inglese/spagnolo. (B) Visualizzazione dispositivo mobile utilizzata per caricare dati di esempio: data, ora, coordinate GPS, descrizione del sito di esempio e immagine del sito di raccolta. Abbreviazioni: SARS-CoV-2 = sindrome respiratoria acuta grave coronavirus 2; GPS = Sistema di posizionamento globale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Kit di raccolta dei campioni. Gli scienziati cittadini hanno ricevuto un dispositivo di raffreddamento contenente due confezioni di ghiaccio, una scheda di dati di sicurezza per informare i volontari sui rischi della manipolazione della soluzione GITC, un protocollo dettagliato di campionamento e di uso della maschera, una maschera KN95, un sacchetto di rifiuti, un flacone spray con disinfettante per le mani, un flacone spray con 0,5% SDS, 16 paia di guanti, una piccola borsa con 16 stuzzicadenti e 16 tamponi in poliestere, 16 tubi microcentrifuge preeticati contenenti 200 μL di soluzione GITC, una scatola contenente i tubi di campionamento e un sacchetto utilizzato come contenitore secondario per la scatola del tubo in caso di fuoriuscita. Abbreviazioni: GITC = guanidinium thiocyanate; SDS = solfato di dodecil solfato di sodio. Clicca qui per visualizzare una versione più grande di questa figura.
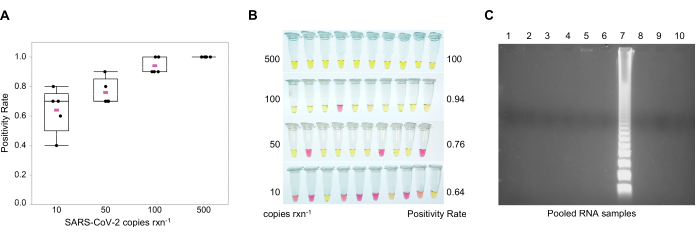
Figura 3: Saggio rt-LAMP (Multiplexed Reverse-Transcription Loop-Mediated Isothermal Amplification). Reazioni multiplexate che utilizzano primer per i geni nucleocapsidi SARS-CoV-2 (N2) e busta (E1) per rilevare fino a 10 copie del virus nella reazione. L'RNA sintetico SARS-CoV-2 è stato utilizzato come controllo positivo. (A) Frequenza di rivelazione in RT-LAMP colorimetrico multiplex di SARS-CoV-2 con diversi numeri di copia del genoma per reazione. Valore medio di cinque repliche in rosa. (B) Limite di rivelazione (LOD) del SARS-CoV-2 in RT-LAMP colorimetrico multiplex; giallo = positivo (pH ~5); rosa = negativo (pH ~8). (C) Modello di scala delle reazioni positive SARS-CoV-2 RT-LAMP nell'elettroforesi del gel di agarosio all'1,5%. Abbreviazioni: SARS-CoV-2 = sindrome respiratoria acuta grave coronavirus 2; rxn = reazione. Clicca qui per visualizzare una versione più grande di questa figura.
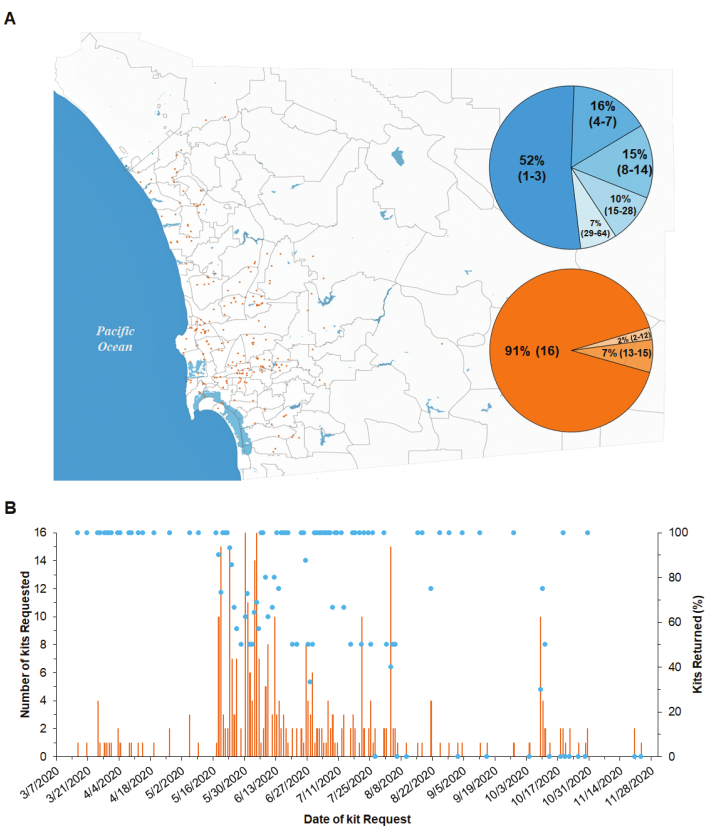
Figura 4: Ubicazione dei kit di campionamento degliscienziati cittadini nella contea di San Diego e tasso di successo dei kit richiesti. (A) I punti arancioni rappresentano la posizione di 1 kit di campionamento, che contiene 16 campioni. Il grafico a torta blu mostra la percentuale di kit che hanno richiesto vari giorni da quando sono stati consegnati agli scienziati cittadini a quando sono stati restituiti al laboratorio. Numero di giorni tra parentesi. Il grafico a torta arancione mostra la percentuale di kit con un numero diverso di campioni completati da un totale di 16 campioni. Numero di campioni completati contenenti un tampone all'interno del tubo campione e i corrispondenti dati di campionamento caricati nel LIMS tra parentesi. (B) Percentuale di kit restituiti al laboratorio (punti) e numero totale di kit richiesti (barre), rispetto alla data in cui è stato richiesto il kit. Clicca qui per visualizzare una versione più grande di questa figura.
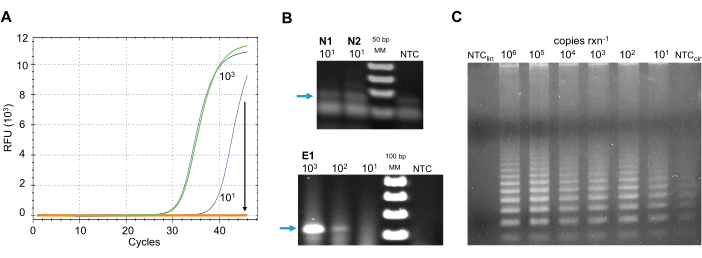
Figura 5: Metodi alternativi di aspirazione SARS-CoV-2 RN. (A) Rilevamento RT-qPCR del gene nucleocapside SARS-CoV-2(N)utilizzando il set di primer N2. Campioni ambientali raggruppati con 900 copie (verdi) o 9 (arancioni) di SARS-CoV-2. Controlli positivi degli stessi numeri di copia in blu. Arrow indica la diminuzione del rilevamento della fluorescenza del controllo positivo del numero di copie basse quando è presente un campione ambientale. (B) Rilevamento tradizionale RT-PCR della SARS-CoV-2. Top: prodotti RT-PCR del gene nucleocapsid utilizzando set primer N1 e N2. Un debole segnale di fondo si osserva nel controllo senza modello. Inbasso: prodotti RT-PCR del gene dell'involucro utilizzando il set di primer E1. Un segnale molto basso si osserva alla concentrazione di LOD. Le frecce blu mostrano il prodotto positivo atteso: (in alto) ~ 70 bp e (in basso) 113 bp nell'elettroforesi del gel di agarosio al 2%. (C) RCA di SARS-CoV-2 RNA. La sonda circolare amplifica in presenza di ligasi e assenza di modello di RNA(CIR NTC); in assenza di ligasi e RNA modello sonda lineare non amplifica (NTClin). Abbreviazioni: SARS-CoV-2 = sindrome respiratoria acuta grave coronavirus 2; RFU = unità di fluorescenza relativa; bp = coppie di basi; rxn = reazione; MM = marcatore molecolare; RT-PCR = reazione a catena della polimerasi a trascrizione inversa; RT-qPCR = RT-PCR quantitativo in tempo reale; RCA = amplificazione del cerchio di laminazione; NTC= controllo senza modello; LOD = limite di rilevamento; rxn = reazione. Clicca qui per visualizzare una versione più grande di questa figura.
| abbecedario | Concentrazione 20X (μM) | 1X Concentrazione (μM) |
| Fip | 32 | 1.6 |
| Bip | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 B3 | 4 | 0.2 |
| LoopF | 8 | 0.4 |
| LoopB | 8 | 0.4 |
Tabella 1: Formulazione per miscela di primer RT-LAMP 20X. Nella reazione RT-LAMP, 6 primer riconoscono 8 regioni del DNA mirato. Abbreviazioni: amplificazione isotermica mediata da loop di trascrizione inversa; FIP = primer interno in avanti; BIP = primer interno all'indietro; F3 = primer di spostamento in avanti; B3 = primer di spostamento all'indietro; LoopF = primer ad anello in avanti; LoopB = primer ad anello all'indietro.
| abbecedario | sequenza | bersaglio | Dimensioni del prodotto | |||
| RT-LAMP9,18,19 | ||||||
| E1-F3 | TGAGTACGAACTTATGTACTCAT | ecstasy | ladder-like modello | |||
| E1-B3 | TTCAGATTTTTAACACGAGAGT | |||||
| E1-FIP | ACCACGAAAGCAAGAAAAGAAGTTCGTTTCGGAAGAGACAG | |||||
| E1-BIP | TTGCTAGTTACACTAGCCATCCTTAGGTTTTACAAGACTCACGT | |||||
| E1-LoopB | GCGCTTCGATTGTGTGCGT | |||||
| E1-LoopF | CGCTATTAACTATTAACG | |||||
| N2-F3 | ACCAGGAACTAATCAGACAAG | N | ||||
| N2-B3 | GACTTGATCTTTGAAATTTGGATCT | |||||
| N2-FIP | TTCCGAAGAACGCTGAAGCGGAACTGATTACAAACATTGGCC | |||||
| N2-BIP | CGCATTGGCATGGAAGTCAATTTGATGGCACCTGTGTA | |||||
| N2-LoopF | GGGGGCAAATTGTGCAATTTG | |||||
| N2-LoopB | CTTCGGGAACGTGGTTGACC | |||||
| RT-qPCR38 | ||||||
| 2019-nCoV_N1-F | GACCCCAAAATCAGCGAAAT | N | 72 pb | |||
| 2019-nCoV_N1-R | TCTGGTTACTGCCAGTTGAATCTG | |||||
| -nCoV_N1-P 2019 | 5'-FAM-ACC CCG CAT TAC GTT TGG TGG ACC-BHQ1-3' | |||||
| 2019-nCoV_N2-F | TTACAAACATTGGCCGCAAA | N | 67 pb | |||
| 2019-nCoV_N2-R | GCGCGACATTCCGAAGAA | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CAG CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | ACAGGTACGTTAATAGTTAATAGCGT | ecstasy | 113 pb | |||
| E1_Sarbeco_R | ATATTGCAGCAGTACGCACACA | |||||
Tabella 2: Primer utilizzati per RT-LAMP, RT-qPCR e RT-PCR. Vengono elencate le sequenze primer, il gene target, la dimensione prevista del prodotto e il riferimento corrispondente. Abbreviazioni: RT-LAMP = amplificazione isotermica mediata da loop di trascrizione inversa; RT-PCR = reazione a catena della polimerasi a trascrizione inversa; bp = coppie di basi; RT-qPCR = RT-PCR quantitativo in tempo reale; E1 = gene busta; N2 = gene nucleocapsidico; F = primer in avanti; R = primer inverso; P = Sonda; FIP = primer interno in avanti; BIP = primer interno all'indietro; F3 = primer di spostamento in avanti; B3 = primer di spostamento all'indietro; LoopF = primer ad anello in avanti; LoopB = primer ad anello all'indietro.
| Reagente | Volume (μL) |
| WarmStart Colorimetric LAMP 2X Master Mix con UDG | 12.5 |
| N2 Primer Mix (20x) | 1.25 |
| E1 Primer Mix (20x) | 1.25 |
| Guanidina Cloridrato (600 mM)* | 2.5 |
| RNA bersaglio | 5 |
| Senza nucleasi H2O | 2.5 |
| Volume totale | 25 |
Tabella 3: Mix master di reazione per RT-LAMP colorimetrico multiplex. (*) Il cloridrato di guanidina ha dimostrato di aumentare la sensibilità e la velocità della reazione con un meccanismo non caratteristiche28. Abbreviazioni: LAMP = amplificazione isotermica mediata da loop; UDG = glicosilasi uracil-DNA; N2 = gene nucleocapsidico; E1 = gene busta; DEPC = dietilpirrocarbonato.
| Cittadini approvati | Cittadini che hanno richiesto un kit | Kit consegnati | Kit campionati restituiti | Giornate dedicate al campionamento | % kit completi | % kit incompleti | Campioni elaborati | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | Significare | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| mediano | 3 | |||||||
Tabella 4:Tampone per SARS-CoV-2 per i numeri. Tassi di successo di sensibilizzazione e campionamento. Abbreviazione: SARS-CoV-2 = sindrome respiratoria acuta grave coronavirus 2.
Discussione
Coinvolgimento degli scienziati cittadini. Gli scienziati cittadini sono stati reclutati su superfici tampone in tutta la contea di San Diego per campionare e rilevare la presenza di SARS-CoV-2 nell'ambiente urbano. La maggior parte dei kit di campionamento consegnati (70%) sono stati restituiti al laboratorio, e di questi, quasi tutti i campioni sono stati completi (91%) (Figura3A,B e tabella 4). I volontari potrebbero facilmente richiedere la consegna / ritiro del kit attraverso la piattaforma basata sul web e il software di pianificazione del percorso di consegna ha notificato agli scienziati cittadini i tempi stimati di arrivo, entrambi fattori probabilmente significativi per il successo osservato. Il tempo medio da quando il kit è stato consegnato allo scienziato cittadino a quando è stato restituito al laboratorio è stato di 8 giorni, con una mediana di 3 giorni e un intervallo di 1-64 giorni (figura 3A e tabella 4). I promemoria più frequenti ai volontari ridurrebbero probabilmente questo ritardo.
La piattaforma di raccolta dati è stata utilizzata con successo dalla stragrande maggioranza degli utenti (73%) (Tabella 4). Sebbene gli sforzi degli scienziati cittadini non siano stati misurati, i test sul campo hanno dimostrato che la piattaforma di raccolta dei dati ha ridotto significativamente lo sforzo e il tempo necessari per completare correttamente la raccolta dei campioni. Pertanto, la riduzione della quantità di contabilità ha incoraggiato il coinvolgimento degli scienziati cittadini. La piattaforma basata sul web intendeva superare i limiti demografici fornendo un servizio multilingue di traduzione automatica neurale e fornendo protocolli grafici e audiovisivi in inglese e spagnolo. Questo ebbe solo parzialmente successo poiché furono raccolti meno campioni sia dalla South Bay che dalla Contea del Nord, dove la maggior parte della popolazione ispanica/latina della contea risiede45. Queste aree ospitavano anche il 63% (1.700 casi ogni 100.000) dei casi totali di COVID-19 nella contea di San Diego con la più alta prevalenzadella malattia 46 e il tasso di ricoveri (62%)47,48. Sebbene la maggior parte dei campioni proveniva dalla Contea Centrale, è stato raccolto un numero rappresentativo dai distretti più colpiti dal COVID-19 e solo una piccola frazione dei campioni è stata positiva, il che suggerisce che i serbatoi di superficie della SARS-CoV-2 nell'ambiente urbano sono relativamente rari.
Elaborazione dei campioni. I tamponi di campionamento sono stati bagnati con SDS, che ha inattivato il virus interrompendo il suo involucro e stabilizzato l'RNA nudo dispiegando RNasi32. Comodamente durante la raccolta, il detergente nel tampone ha pulito la superficie campionare. I campioni ambientali spesso contengono quantità molto piccole di RNA. Per massimizzare il recupero, l'isolamento dell'RNA è stato eseguito utilizzando un metodo di estrazione grezzo basato su GITC, privo di colonne. Il GITC, un forte agente chaotropico, interrompe i legami idrogeno che mantengono il ripiegamento proteico (cioè l'effetto idrofobico). Questa azione si traduce nell'inattivazione delle particelle virali e l'RNA rimane stabile a causa dell'inibizione delleRNAsi 34,35,36. La soluzione GITC ha mantenuto la stabilità dei campioni di RNA senza rigorose considerazioni sulla catena del freddo, il che ha permesso ai cittadini di mantenere i campioni a temperatura ambiente se non era disponibile un congelatore per le confezioni di ghiaccio fornite. Per ridurre il rischio potenziale che questo reagente rappresenta quando si verifica un contatto diretto con la pelle o la mucosa, i cittadini sono stati informati di questi rischi con l'inclusione di una scheda di dati di sicurezza dei materiali fornita nel kit ed è stato inserito un sigillo di avvertimento nella scatola contenente i tubi.
Il metodo di estrazione gitc-cloroformio grezzo ha aiutato il recupero di tracce di RNA dai tamponi e, come dimostrato dall'amplificazione dei campioni chiodati, gli inibitori raramente persistettero nei campioni dopo l'estrazione. I campioni, che erano negativi per il SARS-CoV-2 e non mostravano inibizioni RT-LAMP, rappresentavano veri negativi o avevano un numero di copia inferiore rispetto al LOD con una frequenza del 100%. Al contrario, il rilevamento dell'RNA virale su una superficie non implica direttamente il rischio di trasmissione attraverso il contatto in quanto l'infettività del virus da campioni positivi deve essere testata. Un rapido esame dell'ambiente, non limitato dalla disponibilità di forniture sofisticate o di personale altamente qualificato, è fondamentale per valutare se le superfici costituiscono un serbatoio virale e per migliorare gli sforzi diretti di prevenzione e contenimento.
RT-LAMP è stato selezionato per essere il metodo migliore adatto alla tubazione di rilevamento proposta. Si dimostrò un metodo rapido ed economico che era altamente resistente alla maggior parte degli inibitori rimanenti e sensibile e specifico come altri metodi RT-qPCR. A causa del loro uso in contesti clinici durante la pandemia di SARS-CoV-2, la disponibilità di kit RT-qPCR è stata influenzata dalla domanda globale. Inoltre, le tecniche RT-qPCR , anche quelle formulate per resistere agli inibitori, erano sensibili alle sostanze contenute nei campioni ambientali raccolti da una coorte pilota di scienziati cittadini, anche dopo l'uso di altre strategie comuni per ridurre la concorrenza degli inibitori per il legameenzimatico 49. Questi risultati sono confermati da un recente studio che ha confrontato entrambi i metodi per rilevare SARS-CoV-2 su campioni di tampone da caramelle gestite da pazienti COVID-19 e ha trovato oltre l'83% di concordanza dei risultati, con un'inibizione inferiore del 25% nei campioni analizzati da RT-LAMP15. Inoltre, l'estrazione di greggio GITC-cloroformio, abbinata a RT-LAMP, ha ridotto il costo dei reagenti e delle forniture del 42% rispetto all'estrazione del kit di RNA e rt-qPCR (Tabella dei materiali).
Questo metodo ha permesso un'analisi ad alta produttività di migliaia di campioni di tamponi superficiali. Fino a 80 piscine, che rappresentano 640 campioni, sono state elaborate in 2 giorni dall'estrazione dell'RNA al rilevamento SARS-CoV-2 da RT-LAMP. Il protocollo proposto è semiquantitativo, limitato al rilevamento dell'RNA virale, e non indica la presenza di particelle virali infettive. Sono necessarie ulteriori analisi per valutare il rischio di trasmissione della SARS-CoV-2 da fomiti infetti presenti sulle superfici tamponate.
Questo studio presenta un protocollo per impostare rapidamente una strategia di test che include un flusso di lavoro efficace quando si affronta un'emergenza sanitaria con una malattia trasmissibile. Il protocollo di campionamento proposto è semplice e utilizza forniture comunemente presenti nelle famiglie, e il metodo di rilevazione virale viene eseguito su apparecchiature disponibili in ambienti di laboratorio di base come un bagno d'acqua al posto di un termociclo. I costi dei reagenti RT-LAMP sono significativamente inferiori a quelli necessari per RT-qPCR e meno suscettibili agli elevati scenari di domanda globale. Questo studio funge da quadro per la valutazione dei serbatoi virali ambientali nelle future epidemie e pandemie globali.
Divulgazioni
Tutti gli autori dichiarano che non esistono interessi concorrenti.
Riconoscimenti
Ringraziamo gli investigatori del Viral Information Institute (VII), la Dott.ssa Anca M. Segall, Willow Segall, Patricia L. Rohwer, Gary Rohwer, Cary L. Rohwer, Magda Silvia Pinetta, Elizabeth Cruz Cano, il Dr. Gregory Peters, il Dr. Stuart A. Sandin e la Dott.ssa Jennifer Smith per aver preso il tempo necessario per raccogliere numerosi campioni. Ringraziamo anche il Dr. Rob Knight, il Dr. Jack Gilbert, il Dr. Pedro Balda-Ferre e la Dott.ssa Sarah Allard del Dipartimento di Pediatria della School of Medicine University di San Diego California (UCSD) per aver facilitato controlli positivi e feedback utili. Ringraziamo Stacey Carota (SDSU College of Sciences) e Gina Spidel (SDSU) per il supporto logistico e Juan Rodríguez per l'arte e la progettazione grafica del protocollo di campionamento. Ringraziamo tutti i partecipanti per il loro impegno e dedizione a questo progetto in momenti molto difficili. Questo lavoro è stato supportato da una generosa donazione da parte della Dott.ssa Jo Ann Lane (SDSU College of Sciences) e della National Science Foundation RAPID: Environmental Reservoirs of SARS-CoV-2 grant (Premio: 2030479).
Materiali
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
Riferimenti
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon