Method Article
Écouvillonner l’environnement urbain - Un pipeline pour l’échantillonnage et la détection du SARS-CoV-2 dans les réservoirs environnementaux
Dans cet article
Résumé
Un projet de science citoyenne a été conçu pour recruter des résidents de San Diego afin de recueillir des échantillons environnementaux pour le SARS-CoV-2. Une plate-forme Web multilingue a été créée pour la soumission de données à l’aide d’une interface conviviale pour les appareils mobiles. Un système de gestion de l’information de laboratoire a facilité la collecte de milliers d’échantillons géographiquement diversifiés grâce au suivi des résultats en temps réel.
Résumé
Pour contrôler la transmission communautaire du coronavirus 2 du syndrome respiratoire aigu sévère (SARS-CoV-2) pendant la pandémie mondiale de 2020, la plupart des pays ont mis en œuvre des stratégies basées sur des tests humains directs, le couvre-visage et la désinfection des surfaces. En supposant que la principale voie de transmission comprend les aérosols et les gouttelettes respiratoires, les efforts pour détecter le SARS-CoV-2 chez les vecteurs ennemis se sont concentrés sur les endroits soupçonnés d’une prévalence élevée (p. ex. les services hospitaliers, les navires de croisière et les systèmes de transport en commun). Pour enquêter sur la présence du SARS-CoV-2 sur des surfaces de l’environnement urbain qui sont rarement nettoyées et rarement désinfectées, 350 citoyens ont été enrôlés du grand comté de San Diego. Au total, ces citoyens scientifiques ont recueilli 4 080 échantillons. Une plateforme en ligne a été mise au point pour surveiller la livraison et le ramassage des trousses d’échantillonnage, ainsi que pour recueillir des données d’échantillons. Les trousses d’échantillonnage ont été principalement construites à partir de fournitures disponibles dans les magasins stressés par la pandémie. Les échantillons ont été traités à l’aide de réactifs faciles d’accès malgré la pénurie récurrente d’approvisionnement. Les méthodes utilisées étaient très sensibles et résistantes aux inhibiteurs couramment présents dans les échantillons environnementaux. La conception expérimentale et les méthodes de traitement proposées ont réussi à mobiliser de nombreux citoyens scientifiques qui ont recueilli efficacement des échantillons dans diverses zones de surface. Le flux de travail et les méthodes décrits ici sont pertinents pour étudier l’environnement urbain à la recherche d’autres virus, qui sont préoccupants pour la santé publique et constituent une menace pour de futures pandémies.
Introduction
On pense que le SARS-CoV-2 se transmet principalement par inhalation d’aérosols et de gouttelettes contaminés par contact direct avec des personnes infectées1,2,3,4. Cependant, au cours des phases initiales de la pandémie mondiale de COVID-19, les efforts visant à contrôler la transmission du SARS-CoV-2 se sont fortement concentrés sur la désinfection des surfaces, le lavage des mains et la désinfection. À la fin de 2020, les directives de transmission de l’Organisation mondiale de la Santé (OMS)5 et des Centers for Disease Control and Prevention (CDC) des États-Unis6 ont jugé que la transmission par voie aérienne présentait un danger, principalement lorsqu’elle était en contact étroit (<2 m) avec une personne infectée ou en présence d’interventions médicales génératrices d’aérosols. L’auto-inoculation après un contact avec des surfaces contaminées ou l’inhalation de vecteurs fomites aérosolisés n’a pas encore été exclue comme voie de transmission du SARS-CoV-2.
Des cas de COVID-19 ont été signalés où la transmission par voie aérienne semble peu probable7,8. Les virions du SARS-CoV-2 restent infectieux sur le cuivre jusqu’à 8 h, sur le carton et l’acier inoxydable jusqu’à 24 h, et sur le plastique jusqu’à 48 h9. Dans les cabines des navires de croisière, l’ARN du SARS-CoV-2 a été détecté 17 jours après le départ des passagers7. Des échantillons d’air et de surface provenant d’hôpitaux et de systèmes de transport en commun ont été testés positifs pour le SARS-CoV-2 et d’autres coronavirus8,10 , 11,12,13,14. Une étude réalisée sur l’emballage extérieur des bonbons d’Halloween manipulés par des patients COVID-19 asymptomatiques et modérés / légèrement symptomatiques a conclu que la combinaison du lavage des mains par le manipulateur et du lavage des bonbons avec du savon pour les mains réduisait l’ARN du SARS-CoV-2 à des niveaux inférieurs au seuil15.
Plusieurs méthodes de diagnostic du SARS-CoV-2 ont été publiées sur la base de la réaction en chaîne de la polymérase à transcription inverse en temps réel (RT-qPCR)16,17,de l’amplification isotherme médiée par boucle de transcription inverse (RT-LAMP)11,18,19,20,21et des technologies CRISPR-Cas18,19,22,23. La plupart nécessitent des kits d’extraction d’ARN qui sont souvent en pénurie pendant les périodes de demande mondiale importante, et très peu ont été utilisés pour le dépistage environnemental du virus24. Il a été démontré que la détection de l’ARN du SARS-CoV-2 à l’aide de RT-LAMP est concordante à plus de 83 % avec l’utilisation de RT-qPCR. En outre, RT-LAMP a entraîné une réduction de 25% des résultats non concluants par rapport à RT-qPCR15.
RT-LAMP est une technique simple qui utilise une transcriptase inverse pour synthétiser l’ADNc à partir d’un modèle d’ARN25,suivie d’une ADN polymérase avec une forte activité de déplacement de brin qui synthétise l’ADN à température constante (c’est-à-dire l’amplification isotherme)26. Une plus grande spécificité de la détection du génome viral est obtenue en utilisant quatre ou six amorces qui reconnaissent six ou huit régions de l’ADN cible. L’amplification est initiée à partir d’un apprêt interne et donne une structure d’ADN semi-double brin. Le brin avant est ensuite amplifié par un apprêt extérieur. Ces amplifications sont répétées pour les amorces inverses. Les amorces internes et externes à chaque extrémité ont un site auto-complémentaire inverse interne qui forme une boucle dans le produit d’amplification26,27. Dans le déplacement de brins isothermes, la synthèse asynchrone de l’ADN génère de grandes quantités de produit amplifié où la polymérisation continue amplifie le signal d’aussi peu que 10 copies par réaction11,20,28. Le mélange colorimétrique RT-LAMP est faiblement tamponné et utilise du rouge de phénol comme indicateur de pH. Comme la polymérase incorpore un nucléotide, elle libère un proton, et suffisamment de protons vont changer le pH de la solution ainsi que sa couleur du rose au jaune11,20,28,29.
RT-LAMP a été développé pour la détection des maladies transmises par les moustiques dans les établissements de santé périphériques qui ne disposent pas de laboratoires entièrement équipés25 et pour la détection rapide d’autres virus à ARN comme le virus de l’immunodéficience humaine30. Les populations les plus vulnérables en cas d’épidémie – selon la définition de l’OMS – manquent souvent de ressources économiques suffisantes et de l’équipement approprié pour effectuer la détection (Programme de santé publique mondiale des Nations Unies). Dans la pandémie actuelle de SARS-CoV-2, les fournitures telles que les écouvillons de qualité médicale et les réactifs pour les kits d’extraction d’ARN n’ont pas été en mesure de répondre à la demande mondiale, en particulier dans les pays non manufacturiers. Le protocole proposé utilisait une extraction d’ARN brut à base de thiocyanate de guanidinium (GITC), qui a effectivement préservé l’ARN d’une manière indépendante de la chaîne du froid et a réduit de manière significative la persistance des inhibiteurs de l’échantillon. De plus, le protocole d’extraction gitc-chloroforme est basé sur la séparation de l’ARN de l’ADN et des protéines suivie de la précipitation respective, permettant la récupération de la majeure partie du matériel génétique. Ces avantages l’emportent sur les dangers potentiels des citoyens scientifiques manipulant le produit chimique si des mesures sont prises pour les informer de manière appropriée des risques.
Le flux de travail proposé utilise des matériaux et des réactifs d’utilisation générale. Il nécessite un équipement disponible dans des laboratoires de base, souvent ruraux. Ces méthodes sont peu coûteuses, très résistantes aux inhibiteurs souvent trouvés dans les échantillons environnementaux ou les échantillons qui ne peuvent pas être traités avec des kits d’extraction, et éliminent le besoin d’un thermocycleur de haute précision. Cette étude présente un pipeline pour l’échantillonnage et la détection du SARS-CoV-2 à partir de réservoirs environnementaux sur des surfaces communément touchées et rarement désinfectées des ménages et de l’environnement urbain.
Protocole
Consultez la table des matériaux pour obtenir une liste détaillée des réactifs et des fournitures, y compris les numéros de catalogue, le fabricant et les coûts correspondants.
1. Échantillonnage de l’environnement urbain
- Sensibilisation des citoyens scientifiques
- Recruter des citoyens scientifiques à l’aide d’un appel à l’action direct et clair publié via les médias locaux et sociaux. Créez un handle de médias sociaux (par exemple, #swab4corona)pour connecter le sujet à travers le contenu des médias sociaux.
- Créez un compte de messagerie pour une communication directe entre l’équipe du laboratoire et chaque citoyen scientifique, géré par une personne parlant couramment les principales langues de la région d’intérêt (par exemple, l’espagnol et l’anglais pour le comté de San Diego).
- Construire une plateforme web sécurisée de gestion des échantillons (SMP) qui servira de base de données, de système de gestion de l’information de laboratoire (LIMS) et de communication avec les citoyens scientifiques.
Remarque : le SMP fournit un emplacement centralisé où les utilisateurs demandent un kit, accèdent aux protocoles de collecte d’échantillons, soumettent des métadonnées d’échantillon et demandent un ramassage pour les kits d’échantillons terminés. - Créer un lien vers le PSM (p. ex. https://demo.covidsample.org/)(figure 1) pour que les personnes présentent une demande de participation à l’effort d’échantillonnage environnemental en répondant aux questions liées à la biosécurité précisées dans un formulaire en ligne.
- Accès sécurisé au SMP à l’aide d’une interface de programmation d’applications d’authentification facilitée par un fournisseur de services informatiques en nuage. Donnez aux utilisateurs approuvés l’accès au SMP.
Remarque : reportez-vous à la documentation pour une description du protocole OAuth 2.031 pour l’authentification et l’autorisation. Il fournit un processus de connexion sans friction pour les citoyens scientifiques bénévoles. Il permet également aux utilisateurs de se connecter avec un compte existant, éliminant ainsi la nécessité de créer une solution de connexion personnalisée et de gérer les informations d’identification de l’utilisateur, ce qui permet de gagner beaucoup de temps et d’encourager la participation. Selon des rapports récents, le service de messagerie gratuit disponible pour le fournisseur de services informatiques cloud choisi compte environ 1,5 milliard d’utilisateurs de messagerie actifs mensuellement; exiger un compte de courriel de ce fournisseur de services pour participer n’est pas considéré comme un facteur décourageant. - Expliquer l’objectif de l’étude et les considérations en matière de biosécurité aux citoyens scientifiques du SMP avant qu’ils ne demandent leur première trousse. Fournissez un plug-in multilingue pour permettre la navigation dans l’une des langues disponibles à partir d’un service de traduction automatique neuronal multilingue facilité par un fournisseur de services informatiques cloud.
- Inclure dans la section d’échantillonnage des protocoles graphiques et audiovisuels en anglais et en espagnol.
- Attribuez un identificateur unique à chaque kit et concevez l’interface utilisateur pour utiliser des boutons liés à Sample ID afin de rationaliser le processus de saisie des données(Figure 1A).
- Utilisez un logiciel de planification des itinéraires de livraison avec une application pour appareil mobile à utiliser par les chauffeurs afin d’optimiser les itinéraires de livraison et de ramassage et d’informer les citoyens scientifiques des heures d’arrivée estimées avec précision.
- Créez la plate-forme LIMS sur une pile de services Web PHP et hébergez-la sur une plate-forme d’hébergement commerciale (le système d’exploitation suggéré, le logiciel de serveur Web et le logiciel de base de données sont spécifiés dans la table des matériaux).
- Fournir une interface d’application Web sécurisée pour permettre au personnel de laboratoire de gérer les données rapidement et facilement dans le LIMS. Fournir la visualisation des données à l’aide d’une interface de programmation d’applications de cartographie de données facilitée par un fournisseur de services informatiques cloud.
- Visualisez les données géospatiales à l’aide d’une interface de programmation d’applications géospatiales facilitée par un fournisseur de services informatiques cloud. Stocker les données soumises au SGIL par l’intermédiaire du PSM afin de faciliter (1) le stockage centralisé des données du projet; (2) le suivi des flux de travail de traitement des échantillons et des données; et (3) la gestion de la logistique de la distribution des trousses d’échantillons aux citoyens scientifiques.
- Sécuriser les métadonnées soumises à l’aide des meilleures pratiques (p. ex., https://demo.covidsample.org/).
- Informations de préchargement telles que l’ID du kit d’échantillons, l’ID d’échantillon, la date, l’heure et les coordonnées gps (gps) (automatiquement collectées à partir d’une image du site) pour permettre la conformité du type de données et minimiser la soumission de données erronées ou manquantes par l’utilisateur(Figure 1B). Inclure les champs suivants à remplir manuellement et rapidement (<1 min) par le citoyen scientifique : la date et l’heure de la collecte, une brève description de l’emplacement et une photo du site d’échantillonnage.
- Nettoyez toutes les données téléchargées et validez le type de données. Par exemple, validez les données d’image téléchargées par les utilisateurs pour sélectionner des fichiers .jpg, renommez-les avec Sample ID pour une association rapide avec l’exemple et stockez les images téléchargées dans un emplacement sécurisé distinct non accessible aux utilisateurs.
- Activez l’option de demander la livraison et le ramassage du kit lorsque tous les échantillons (16) ont été complétés. De plus, activez l’option permettant de demander la livraison d’un nouveau kit lors du ramassage du précédent(Figure 1A).
REMARQUE : Pour les bénévoles qui préfèrent une plateforme non web et pour ceux qui s’inquiètent de divulguer leur position GPS (p. ex., les membres de la communauté préoccupés par leur statut migratoire), les trousses peuvent être livrées à un lieu de réunion convenu et les bénévoles peuvent être invités à enregistrer une version écrite de la collecte de données. Pour la communication entre le laboratoire et chaque citoyen scientifique, avoir un membre bilingue du projet disponible pour les appels téléphoniques et les textos.
- Écouvillon pour Corona
- Déterminer une fenêtre de temps pertinente sur le plan épidémiologique pour l’effort d’échantillonnage.
- Construire une trousse qui contient toutes les fournitures d’échantillonnage, y compris l’équipement de protection individuelle nécessaire (c.-à-d. masque, gants), un protocole d’échantillonnage et des renseignements pertinents sur la biosécurité (figure 2). Pré-étiquetez chaque tube avec l’identificateur unique assigné (id d’échantillon).
- Écouvillonner rarement les surfaces désinfectées qui sont exposées à des vecteurs fomites aérosolisés dans les ménages et l’environnement urbain.
- Portez le masque fourni en public et une nouvelle paire de gants pour le prélèvement de chaque échantillon afin d’éviter la contamination croisée. Après avoir terminé l’échantillonnage, utilisez le désinfectant pour les mains fourni.
- Mouiller un écouvillon absorbant le polyester de 1 cm2 (p. ex. tampons de vadrouille) avec un détergent (p. ex. sulfate de dodécyle de sodium (FDS) à 0,5 %) pour inactiver le virus en perturbant son enveloppe et stabiliser l’ARN nu en induisant le dépliage des RNases32.
- Écouvillonner une surface de 10 cm2. Aidé par un cure-dent, submergez complètement chaque écouvillon d’échantillon dans le tube pré-marqué correspondant contenant 200 μL de solution de thiocyanate de guanidinium (GITC). Entreposer les tubes à 4 °C jusqu’à ce qu’ils soient transportés au laboratoire. Une fois les échantillons arrivés au laboratoire, conservez-les à -80 °C.
REMARQUE: GITC est un irritant toxique; éviter tout contact avec la peau. La solution GITC est simple à préparer à partir de produits chimiques de laboratoire courants, pour la recette voir33,34. Il inactive le virus, stabilise l’ARN en dénaturant RNases34,35,36et stabilise les échantillons à température ambiante. La trousse, cependant, comprend des blocs réfrigérants pour garder les échantillons au froid sans avoir besoin d’utiliser des réfrigérateurs ménagers pour l’entreposage.
2. Détection du SARS-CoV-2
- Isolement total de l’ARN
- Désinfecter les surfaces, l’équipement et les pipettors avec une solution de sulfate de cuivre à 2 mM et de peroxyde d’hydrogène à 3 %; suivi d’une solution de 10% d’eau de Javel, 90 mM de bicarbonate de sodium, 5% SDS, et 2,5% NaOH. Essuyez soigneusement avec de l’eau distillée suivie d’éthanol à 75%.
REMARQUE: Ces solutions sont une alternative aux solutions disponibles dans le commerce. - Décongeler les échantillons sur la glace. Échantillons de vortex pendant 2 min à vitesse moyenne.
- Pour accélérer le dépistage, traitez les échantillons dans des piscines. Si un pool est positif, extraire l’ARN de chaque échantillon indépendamment pour trouver le ou les échantillons positifs. Combinez les échantillons de chaque trousse d’échantillonnage (16 au total) en 2 bassins de 8 échantillons.
REMARQUE: Avoir 8 échantillons par pool signifie que seulement 2 pools doivent être traités par kit. Si un pool est positif, les échantillons individuels sont traités à nouveau pour l’analyse RT-LAMP individuelle. Cela réduit le temps, les coûts et les réactifs. - Regrouper 50 μL de chacun des 8 échantillons dans un tube à microcentrifugation (volume total de 400 μL); conserver l’échantillon restant à -80 °C. Ajouter 0,2 volume (80 μL) de chloroforme, de vortex pendant 15 s, puis incuber pendant 20 min à 4 °C. Centrifuger à 13 000 × g pendant 20 min à 4 °C.
- Transférer la couche aqueuse (liquide clair) dans un nouveau tube à microcentrifugation. Conserver l’interface restante et le liquide rose dans le congélateur à -80 °C; ces fractions contiennent de l’ADN et des protéines33,36.
- Ajouter un volume égal d’isopropanol (~200 μL) et 2,6 μL de glycogène coprecipitant (15 mg mL-1)37. Bien mélanger et incuber à -20 °C pendant au moins 1 h, puis 4 °C pendant 10 min pour précipiter l’ARN.
REMARQUE: Le protocole peut être mis en pause ici en incubant des échantillons à -20 °C pendant la nuit au lieu de 1 h. - Centrifuger à 13 000 × g pendant 20 min à 4 °C. Retirez le surnageant sans déranger la pastille. Ressusciter la pastille dans 50 μL d’eau traitée au diéthylpyrocarbonate (DEPC) et ajouter un volume égal (50 μL) d’acétate d’ammonium 5 M sans RNase et 2,5 volumes (250 μl) d’éthanol à 100 %7,38.
REMARQUE : Les ions ammonium inhibent la polynucléotide kinase s’ils sont utilisés dans un processus en aval38. Le mélange précipite l’ARN tout en laissant des triphosphates désoxynucléosides et des oligosaccharides en solution38. - Bien mélanger et incuber à -20 °C pendant au moins 1 h, puis 4 °C pendant 10 min pour précipiter l’ARN.
REMARQUE: Le protocole peut être mis en pause ici en incubant des échantillons à -20 °C pendant la nuit au lieu de 1 h. - Centrifuger à 13 000 × g pendant 20 min à 4 °C. Laver le culot avec 1 mL d’éthanol froid (-20 °C), fraîchement fait à 75 %. Centrifuger à 8 000 × g pendant 5 min à 4 °C. Retirez le surnageant avec une pipette P10 pour éviter de perturber la pastille.
- Sécher à l’air le culot pendant 10-15 min jusqu’à ce qu’il ne reste plus d’éthanol. Ressusciter la pastille dans 50 μL d’eau traitée au DEPC, ajouter 5 μL de tampon DNase 10x + 1μL de DNase (2 unités μL-1)et incuber à 37 °C pendant 30 min.
- Ajouter 0,1 volume (5,6 μL) de réactif d’inactivation de la DNase, incuber à température ambiante pendant 5 min et mélanger doucement toutes les minutes. Centrifuger à 13 000 × g pendant 2 min et transférer le surnageant dans un nouveau tube (~50 μL). Placez immédiatement le tube sur de la glace pendant la préparation des réactions RT-qPCR ou RT-LAMP, ou conservez-le dans un congélateur à -20 °C.
REMARQUE: Effectuez l’isolement de l’ARN dans une pièce sans amplicon pour éviter la contamination par le report.
- Désinfecter les surfaces, l’équipement et les pipettors avec une solution de sulfate de cuivre à 2 mM et de peroxyde d’hydrogène à 3 %; suivi d’une solution de 10% d’eau de Javel, 90 mM de bicarbonate de sodium, 5% SDS, et 2,5% NaOH. Essuyez soigneusement avec de l’eau distillée suivie d’éthanol à 75%.
- Amplification isotherme multiplex à médiation par boucle de transcription inverse (RT-LAMP)
- Préparer une solution de mélange d’apprêts 20X (tableau 1) pour chaque ensemble d’apprêts (tableau 2). Préparer le mélange réactionnel RT-LAMP (tableau 3) à température ambiante avec un volume excédentaire de 10 % pour tenir compte de la perte de pipetage.
NOTE: Le mélange maître colorimétrique LAMP 2X avec l’uracile thermolabile antarctique-ADN glycosylase (UDG) empêche l’amplification de la contamination de l’ADN des réactions précédentes20,28. - Vortex et faire tourner le mélange. Distribuer 20 μL du mélange dans chaque tube réactionnel : échantillon, enrichi, témoin positifet témoin négatif. Incuber les réactions dans les tubes à température ambiante pendant 10 min pour permettre à l’UDG d’agir sur la contamination potentielle de report.
- Ajouter 5 μL d’ARN à la réaction de l’échantillon, 5 μL d’ARN + 2,5 μL (450 copies) d’ARN synthétique du SARS-CoV-2 à la réaction enrichie, 2,5 μL (450 copies) d’ARN synthétique du SARS-CoV-2 à la réaction témoin positive et 5 μL deH2O à la réaction témoin négative. Bien mélanger et faire tourner les réactions; décongeler tout l’ARN sur la glace.
- Pendant que le thermocycleur, ou le bain-marie, est chauffé à 65 °C, laissez toutes les réactions rester à température ambiante. L’UDG sera inactivé à >50 °C. Placez les réactions dans le thermocycleur (utilisez un couvercle de chaleur), incuber à 65 °C pendant 40 min et laissez les réactions atteindre la température ambiante (~ 22 °C pendant 5 min) ou refroidir sur de la glace pendant 1 min. Analysez les résultats à l’aide de la fonction colorimétrique (observation simple) ou en exécutant les produits dans un gel d’agarose.
REMARQUE : Bien que la limite de détection (LD) soit de 10 copies par réaction, la fréquence de détection augmente à mesure que le nombre de copies approche les 500 copies par réaction(figure 3A). Pour l’observation colorimétrique, notez qu’un résultat négatif est indiqué par le rose (pH = 8,8), tandis qu’un résultat positif est indiqué par le jaune (pH = 5)(Figure 3B). L’option colorimétrique évite l’ouverture des tubes RT-LAMP après amplification, ce qui réduira le volume des produits RT-LAMP dans l’environnement de travail et la contamination par report. Pour l’électrophorèse sur gel, préparez un gel d’agarose à 1,5% avec une coloration de gel d’ADN 1X dans un tampon Tris / Borate / EDTA (TBE) à 0,5%. Chargez 25 μL de la réaction + 5 μL de colorant de chargement 6X dans chaque puits. Faire fonctionner le gel à 100 V pendant 60 min. Un marqueur moléculaire n’est pas nécessaire car les échantillons positifs montrent un motif d’échelle(figure 3C).
- Préparer une solution de mélange d’apprêts 20X (tableau 1) pour chaque ensemble d’apprêts (tableau 2). Préparer le mélange réactionnel RT-LAMP (tableau 3) à température ambiante avec un volume excédentaire de 10 % pour tenir compte de la perte de pipetage.
Résultats
Échantillons prélevés par des citoyens scientifiques pour la détection du SARS-CoV-2. Au cours d’une période de 8 mois (de la mi-mars à la troisième semaine de novembre 2020), 482 citoyens ont été approuvés pour participer à ce projet, dont 350 (73 %) a demandé une trousse. Au total, 362 trousses ont été livrées (c.-à-d. que certains participants ont demandé plusieurs trousses), et 246 (70 %) ont été retournés (figure 4A,B). Les 4 080 échantillons contenus dans ces trousses ont été traités. Les sites de collecte ont été répartis dans les districts côtiers nord, centre-nord, centre-ville et sud du comté, ainsi que quelques-uns dans les districts de l’est (figure 4A). Ces districts ont la densité de population la plus élevée du comté de San Diego et les cas de COVID-19 les plus documentés, comme indiqué par l’Agence des services de santé humaine de San Diego39.
Les citoyens ont demandé le ramassage de la plupart des trousses échantillonnées (c.-à-d. taux de réussite moyen : 70,4 %). Chaque jour, de 1 à 16 trousses ont été demandées, et de 0 à 14 trousses ont été retournées au laboratoire(figure 4B). Une enquête auprès des citoyens scientifiques a montré que la collecte d’un kit complet (16 échantillons) a pris 1-3 h répartis sur une moyenne de 8 jours(Figure 4A et Tableau 4). La grande majorité des trousses étaient complètes (91,1 %), ce qui signifie qu’elles contenaient un écouvillon à l’intérieur des 16 tubes d’échantillonnage, et les données d’échantillonnage correspondantes ont été téléchargées dans le SGIL(figure 4A et tableau 4).
Détection du SARS-CoV-2 à l’aide de l’extraction gitc-chloroforme et de l’amplification isotherme multiplex à médiation par boucle de transcriptase inverse (RT-LAMP). Pour le dosage colorimétrique RT-LAMP, deux ensembles d’amorces11,20,28 ont été utilisés pour cibler les gènes nucléocapside(N2)et enveloppe(E1) (tableau 2). Les séquences reconnues par ces amorces se trouvent dans la même région que les amorces et sondes approuvées par le CDC40 et l’Union européenne (UE)41 pour le diagnostic humain de COVID-19 par RT-qPCR. Ces résultats corroborent ce que Zhang et al.28 ont décrit, dans lequel l’ajout de chlorhydrate de guanidine de 60 mM à la réaction augmente la LD lorsqu’il est exécuté en multiplex. La LD à une fréquence de 100 % était de 500 copies par réaction de 25 μL (figure 3A,B). Dans le RT-LAMP colorimétrique, les échantillons positifs ont changé de couleur du rose au jaune en raison d’un décalage du pH d’environ 8 à 5,5 (Figure 3B). Lorsque la réaction est devenue orange à faible nombre de copies, des échantillons ont été prélevés dans un gel d’agarose à 1,5 % pour confirmer qu’ils étaient positifs et ont donné lieu à un motif semblable à une échelle (figure 3C). Rt-LAMP a été utilisé pour détecter le SARS-CoV-2 dans des échantillons d’ARN groupés.
Pour contrôler les faux négatifs dus aux inhibiteurs de réaction, chaque échantillon a été testé dans une réaction supplémentaire enrichie de 500 copies de SARS-CoV-2 synthétique. Les échantillons groupés positifs ont été dérepliqués en isolant l’ARN de chaque échantillon individuel dans le pool et exécutés dans une réaction RT-LAMP pour déterminer l’identité de l’échantillon positif. Les résultats de la détection ont ensuite été téléchargés dans le SGIL où l’ID d’échantillon unique a été jumelé avec les informations sur la date, l’heure, les coordonnées GPS, le site et l’image de l’échantillon.
Méthodes RT-PCR en temps réel et traditionnelles: inhibition par les contaminants de l’échantillon. Afin de sélectionner la meilleure méthode adaptée au pipeline de détection proposé, d’autres méthodes d’amplification de l’ARN ont été testées à l’aide d’échantillons environnementaux prélevés par une cohorte pilote de citoyens scientifiques. Des exemples des résultats de chacune de ces méthodes sont présentés à la figure 5 pour illustrer leur sensibilité aux inhibiteurs environnementaux et au bruit de fond du signal à de faibles concentrations de copies virales.
Six formulations de RT-qPCR (table des matières) approuvées par les CDC et l’OMS ont été testées pour la détection du virus sur des échantillons environnementaux. Les protocoles ont été suivis conformément aux instructions du fabricant ainsi qu’aux lignes directrices des CDC pour la détection du SARS-CoV-2 en milieu clinique40. Des réactions contenant différentes concentrations de témoins synthétiques de l’ARN du SARS-CoV-2 ont été enrichies dans des échantillons de surface écouvillonnée après l’isolement de l’ARN brut. Tous les mélanges maîtres étaient sensibles aux inhibiteurs aux concentrations de LD du témoin positif (figure 5A).
Pour contourner les inhibiteurs de ces technologies en temps réel, un système RT-PCR traditionnel a été testé. Un système RT-PCR en une étape (Table of Materials) a été utilisé pour amplifier le gène nucléopside en utilisant les ensembles d’amorces N1, N2et le gène d’enveloppe en utilisant l’ensemble d’amorces E1 approuvé par le CDC (USA) et l’ECDC (UE), respectivement (Tableau 2). Les protocoles ont été suivis conformément aux instructions du fabricant ainsi qu’aux lignes directrices des CDC pour la détection du SARS-CoV-2 en milieu clinique40. Les ensembles d’amorces N1 et N2 conçus par le CDC donnent un produit d’environ 70 pb; cependant, le nombre de copies positives à faible copie n’a pas été distingué du bruit de fond d’amplification du témoin négatif (figure 5B),qui a introduit des faux positifs dans les résultats. Le produit des amorces E1 avait un signal faible au nombre de copie faible (Figure 5B),introduisant des faux négatifs dans lesrésultats. De plus, la méthode RT-PCR testée était encore sensible aux inhibiteurs présents dans les échantillons environnementaux (données non présentées).
D’autres méthodes ont été développées pour détecter de très petites quantités de séquences cibles. L’une de ces méthodes est l’amplification du cercle roulant (RCA), sur laquelle la reconnaissance de la séquence cible, de l’ARN ou de l’ADN, par une sonde linéaire spécifique, une ligase circularise le gabarit. À l’aide d’amorces conçues pour s’hybrider avec la sonde, une ADN polymérase avec une activité de déplacement de brin amplifie la sonde dans une réaction isotherme42. C’est la sonde qui a identifié la cible, qui est amplifiée, et non la séquence cible, ce qui rend cette méthode très sensible43. Wang et coll.44 ont publié un protocole RCA pour la détection directe de l’ARN du SARS-CoV-1. La méthode a été modifiée pour utiliser des amorces spécifiques au SARS-CoV-2. Malheureusement, dans le contrôle sans gabarit (NTC), la sonde circularise et produit en l’absence de modèle d’ARN, même en utilisant une grande variété de ligases, y compris une ligase sensible au SNP. En l’absence de ligase, le NTC n’a pas montré d’amplification à partir de la sonde linéarisée (figure 5C).

Figure 1: Plate-forme d’échantillonnage en ligne avec interface de données de collecte d’échantillons pour appareils mobiles. (A) Un site web, contenant un plugin multilingue, a été créé pour servir de médiateur dans l’interaction entre le laboratoire et les citoyens scientifiques. La plateforme a été utilisée pour la livraison et la collecte d’échantillons et la soumission de données d’échantillons. La plate-forme contenait des protocoles d’échantillonnage graphiques et audiovisuels détaillés anglais/espagnols. (B) Vue de l’appareil mobile utilisée pour télécharger des données d’échantillon : date, heure, coordonnées GPS, description de l’échantillon du site et image du site de collecte. Abréviations : SARS-CoV-2 = coronavirus du syndrome respiratoire aigu sévère 2; GPS = Système mondial de localisation. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 2: Trousse de prélèvement d’échantillons. Les citoyens scientifiques ont reçu une glacière contenant deux blocs réfrigérants, une fiche de données de sécurité pour informer les bénévoles des dangers de la manipulation de la solution GITC, un protocole détaillé d’échantillonnage et de port du masque, un masque KN95, un sac à déchets, un vaporisateur avec désinfectant pour les mains, un vaporisateur avec 0,5 % de FDS, 16 paires de gants, un petit sac avec 16 cure-dents et 16 écouvillons en polyester, 16 tubes de microcentrifugation pré-étiquetés contenant 200 μL de solution GITC, une boîte contenant les tubes d’échantillonnage et un sac utilisé comme contenant secondaire pour la boîte à tubes en cas de déversement. Abréviations : GITC = thiocyanate de guanidinium; FDS = dodécyl sulfate de sodium. Veuillez cliquer ici pour voir une version plus grande de cette figure.
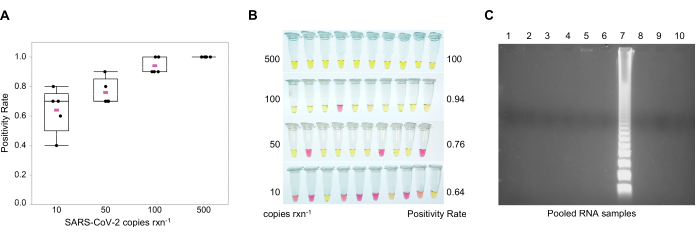
Figure 3: Dosage d’amplification isotherme multiplexée à médiation par boucle de transcription inverse (RT-LAMP). Réactions multiplexées utilisant des amorces pour les gènes nucléocosides(N2)et enveloppe(E1)du SARS-CoV-2 pour détecter aussi peu que 10 copies du virus dans la réaction. L’ARN synthétique du SARS-CoV-2 a été utilisé comme témoin positif. (A) Fréquence de détection en RT-LAMP colorimétrique multiplex du SARS-CoV-2 à différents nombres de copies du génome par réaction. Valeur moyenne de cinq répétitions en rose. B)Limite de détection (LD) du SARS-CoV-2 dans le RT-LAMP colorimétrique multiplex; jaune = positif (pH ~ 5); rose = négatif (pH ~8). (C) Modèle d’échelle des réactions positives de SARS-CoV-2 RT-LAMP dans l’électrophorèse de gel d’agarose de 1,5%. Abréviations : SARS-CoV-2 = coronavirus du syndrome respiratoire aigu sévère 2; rxn = réaction. Veuillez cliquer ici pour voir une version plus grande de cette figure.
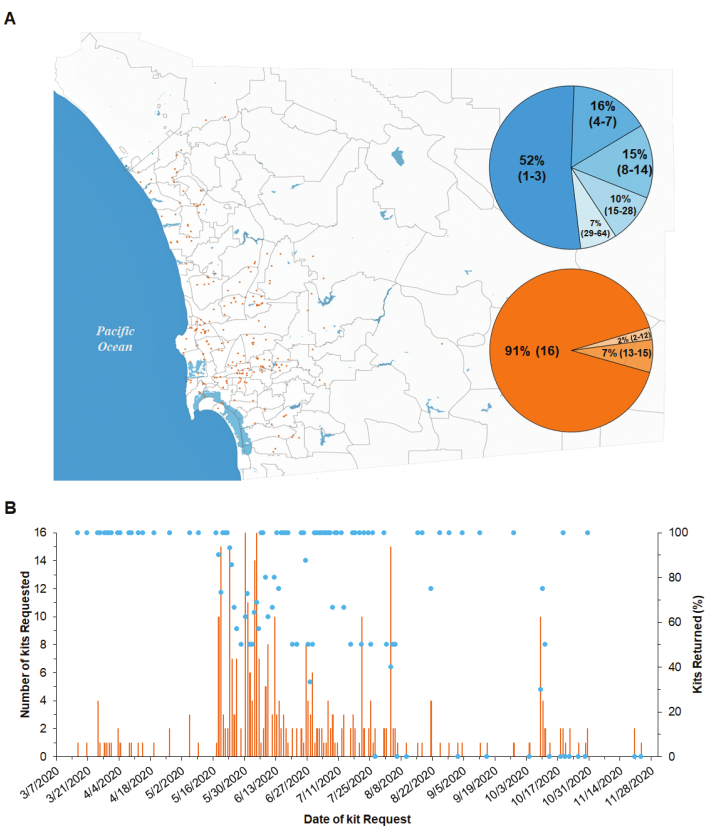
Figure 4: Emplacement des trousses d’échantillonnage des citoyens scientifiques dans le comté de San Diego et taux de réussite des trousses demandées. (A)Les points orange représentent l’emplacement de 1 trousse d’échantillonnage, qui contient 16 trousses. Le diagramme circulaire bleu montre le pourcentage de trousses qui ont pris plusieurs jours entre le moment où elles ont été livrées aux citoyens scientifiques et le moment où elles ont été retournées au laboratoire. Nombre de jours entre parenthèses. Le diagramme circulaire orange montre le pourcentage de trousses avec un nombre différent d’échantillons complétés sur un total de 16 échantillons. Nombre d’échantillons complétés contenant un écouvillon à l’intérieur du tube d’échantillonnage et les données d’échantillonnage correspondantes téléchargées dans le SGIL entre parenthèses. (B) Pourcentage de trousses qui ont été retournées au laboratoire (points) et nombre total de trousses demandées (barres), par rapport à la date à laquelle la trousse a été demandée. Veuillez cliquer ici pour voir une version plus grande de cette figure.
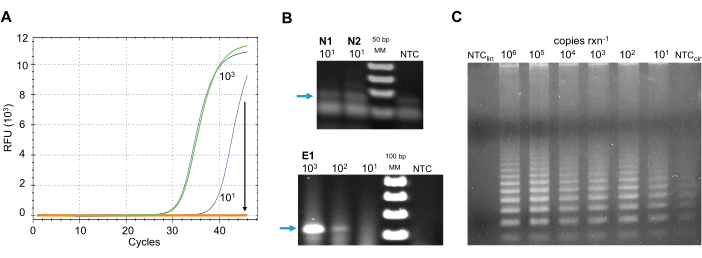
Figure 5: Méthodes alternatives d’adétection du SARS-CoV-2 RN. (A) Détection RT-qPCR du gène nucléocopside(N)du SARS-CoV-2 à l’aide de l’ensemble d’amorces N2. Des échantillons environnementaux groupés enrichis de 900 copies (vertes) ou 9 copies (orange) du SARS-CoV-2. Contrôles positifs des mêmes numéros de copie en bleu. Arrow indique la diminution de la détection de fluorescence du contrôle positif du nombre de copies faibles lorsque l’échantillon environnemental est présent. (B) Détection traditionnelle rt-PCR du SARS-CoV-2. Haut: Produits RT-PCR du gène nucléocopside utilisant les ensembles d’amorces N1 et N2. Un signal d’arrière-plan faible est observé dans le contrôle sans gabarit. En bas: Produits RT-PCR du gène d’enveloppe utilisant l’ensemble d’amorces E1. Un signal très faible est observé à la concentration de LOD. Les flèches bleues montrent le produit positif attendu: (en haut) ~ 70 pb et (en bas) 113 pb dans l’électrophorèse sur gel d’agarose à 2%. (C) RCA de l’ARN du SARS-CoV-2. Sonde circulaire amplifie en présence de ligase et absence de modèle d’ARN (NTCcir); en l’absence de ligase et d’ARN, la sonde linéaire ne s’amplifie pas (NTClin). Abréviations : SARS-CoV-2 = coronavirus du syndrome respiratoire aigu sévère 2; RFU = unités de fluorescence relative; bp = paires de bases; rxn = réaction; MM = marqueur moléculaire; RT-PCR = réaction en chaîne de la polymérase à transcription inverse; RT-qPCR = RT-PCR quantitative en temps réel; RCA = amplification du cercle roulant; NTC = contrôle sans modèle; LOD = limite de détection; rxn = réaction. Veuillez cliquer ici pour voir une version plus grande de cette figure.
| amorce | Concentration 20X (μM) | Concentration 1X (μM) |
| Fip | 32 | 1.6 |
| BIP | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 | 4 | 0.2 |
| BoucleF | 8 | 0.4 |
| BoucleB | 8 | 0.4 |
Tableau 1: Formulation pour le mélange d’apprêt RT-LAMP 20X. Dans la réaction RT-LAMP, 6 amorces reconnaissent 8 régions de l’ADN ciblé. Abréviations: amplification isotherme médiée par boucle de transcription inverse; FIP = amorce intérieure avant; BIP = amorce intérieure arrière; F3 = amorce de déplacement vers l’avant; B3 = amorce de déplacement vers l’arrière; LoopF = amorce de boucle avant; LoopB = amorce de boucle arrière.
| amorce | séquence | cible | Taille du produit | |||
| RT-LAMP9,18,19 | ||||||
| E1-F3 | TGAGTACGAACTTATGTACTCAT | E | motif en forme d’échelle | |||
| E1-B3 | TTCAGATTTTTAACACGAGAGT | |||||
| E1-FIP | ACCACGAAAGCAAGAAAAAGAAGAG | |||||
| E1-BIP | TTGCTAGTTACACTAGCCATCCTTAGGTTTTACAAGACTCACGT | |||||
| E1-BoucleB | GCGCTTCGATTGTGTGCGT | |||||
| E1-LoopF | CGCTATTAACTATTAACG | |||||
| N2-F3 | ACCAGGAACTAATCAGACAAG | N | ||||
| N2-B3 | GACTTGATCTTTGAAATTTGGATCT | |||||
| N2-FIP | TTCCGAAGAACGCTGAAGCGGAACTGATTACAAACATTGGCC | |||||
| N2-BIP | CGCATTGGCATGGAAGTCACAATTTGATGGCACCTGTGTA | |||||
| Boucle N2F | GGGGGCAAATTGTGCAATTTG | |||||
| Boucle N2B | CTTCGGGAACGTGGTTGACC | |||||
| RT-qPCR38 | ||||||
| 2019-nCoV_N1-F | GACCCCAAAATCAGCGAAAT | N | 72 points de base | |||
| 2019-nCoV_N1-R | TCTGGTTACTGCCAGTTGAATCTG | |||||
| 2019-nCoV_N1-P | 5'-FAM-ACC GCC CAT TAC GTT TGG TGG ACC-BHQ1-3' | |||||
| 2019-nCoV_N2-F | TTACAAACATTGGCCGCAAA | N | 67 points de base | |||
| 2019-nCoV_N2-R | GCGCGACATTCCGAAGAA | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CAG CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | ACAGGTACGTTAATAGTTAATAGCGT | E | 113 points de base | |||
| E1_Sarbeco_R | ATATTGCAGCAGTACGCACACA | |||||
Tableau 2 : Amorces utilisées pour rt-lamp, rt-qPCR, et RT-PCR. Les séquences d’amorce, le gène cible, la taille prévue du produit et la référence correspondante sont énumérés. Abréviations : RT-LAMP = amplification isotherme médiée par la boucle de transcription inverse; RT-PCR = réaction en chaîne de la polymérase à transcription inverse; bp = paires de bases; RT-qPCR = RT-PCR quantitative en temps réel; E1 = gène de l’enveloppe; N2 = gène nucléocopside; F = amorce avant; R = amorce inverse; P = Sonde ; FIP = amorce intérieure avant; BIP = amorce intérieure arrière; F3 = amorce de déplacement vers l’avant; B3 = amorce de déplacement vers l’arrière; LoopF = amorce de boucle avant; LoopB = amorce de boucle arrière.
| Réactif | Volume (μL) |
| WarmStart Colorimetric LAMP 2X Master Mix avec UDG | 12.5 |
| N2 Primer Mix (20x) | 1.25 |
| E1 Primer Mix (20x) | 1.25 |
| Chlorhydrate de guanidine (600 mM)* | 2.5 |
| ARN cible | 5 |
| H2Osans nucléase | 2.5 |
| Total Volume | 25 |
Tableau 3 : Mélange maître de réaction pour RT-LAMP colorimétrique multiplex. (*) Il a été démontré que le chlorhydrate de guanidine augmente la sensibilité et la vitesse de la réaction par un mécanisme non caractéristique28. Abréviations : LAMP = amplification isotherme à médiation en boucle; UDG = uracile-ADN glycosylase; N2 = gène nucléocopside; E1 = gène de l’enveloppe; DEPC = diéthylpyrocarbonate.
| Citoyens agréés | Citoyens qui ont demandé une trousse | Kits livrés | Kits échantillonnés retournés | Journées dédiées à l’échantillonnage | % kits complets | % Kits incomplets | Échantillons traités | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | méchant | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| médian | 3 | |||||||
Tableau 4 :Écouvillonnage du SARS-CoV-2 en chiffres. Taux de réussite de la sensibilisation et de l’échantillonnage. Abréviation : SARS-CoV-2 = coronavirus du syndrome respiratoire aigu sévère 2.
Discussion
Engagement des citoyens scientifiques. Des citoyens scientifiques ont été recrutés pour écouvillonner les surfaces dans tout le comté de San Diego afin d’échantillonner et de détecter la présence du SARS-CoV-2 dans l’environnement urbain. La majorité des trousses d’échantillonnage livrées (70 %) ont été retournés au laboratoire, et de ce nombre, presque tous les échantillons étaient complets (91 %) (Figure 3A,B et Tableau 4). Les bénévoles pouvaient facilement demander la livraison ou le ramassage des trousses par l’entremise de la plateforme Web, et le logiciel de planification des itinéraires de livraison demandait aux citoyens scientifiques d’estimer les heures d’arrivée, deux facteurs probablement importants pour le succès observé. Le délai moyen entre la livraison de la trousse au citoyen scientifique et le moment où elle a été retournée au laboratoire était de 8 jours, avec une médiane de 3 jours et une fourchette de 1 à 64 jours (figure 3A et tableau 4). Des rappels plus fréquents aux bénévoles réduiraient probablement ce délai.
La plateforme de collecte de données a été utilisée avec succès par une grande majorité d’utilisateurs (73 %) (Tableau 4). Bien que les efforts des citoyens scientifiques n’aient pas été mesurés, des tests sur le terrain ont montré que la plate-forme de collecte de données réduisait considérablement les efforts et le temps nécessaires pour mener à bien le prélèvement d’échantillons. Ainsi, la réduction de la quantité de comptabilité a encouragé l’engagement des citoyens scientifiques. La plate-forme Web visait à surmonter les limitations démographiques en fournissant un service de traduction automatique neuronal multilingue et en fournissant des protocoles graphiques et audiovisuels en anglais et en espagnol. Cela n’a été que partiellement réussi, car moins d’échantillons ont été prélevés à la fois dans la baie Sud et dans le comté de North, où réside la majeure partie de la population hispanique / latino ducomté, soit 45personnes. Ces zones abritaient également 63% (1 700 cas pour 100 000) du nombre total de cas de COVID-19 dans le comté de San Diego avec la prévalence la plus élevée de la maladie46 et le taux d’hospitalisations (62%)47,48. Bien que la plupart des échantillons proviennent du comté de Central, un nombre représentatif a été recueilli dans les districts les plus touchés par la COVID-19 et seule une petite fraction des échantillons était positive, ce qui suggère que les réservoirs de surface du SARS-CoV-2 dans l’environnement urbain sont relativement rares.
Traitement des échantillons. Les écouvillons d’échantillonnage ont été mouillés avec des FDS, qui ont désactivé le virus en perturbant son enveloppe et stabilisé l’ARN nu en dépliant RNases32. Idéalement, pendant le prélèvement, le détergent dans l’écouvillon a nettoyé la surface échantillonnée. Les échantillons environnementaux contiennent souvent de très petites quantités d’ARN. Pour maximiser la récupération, l’isolement de l’ARN a été effectué à l’aide d’une méthode d’extraction brute, sans colonne, basée sur GITC. Gitc, un agent chaotrope fort, perturbe les liaisons hydrogène qui maintiennent le repliement des protéines (c.-à-d. effet hydrophobe). Cette action se traduit par l’inactivation des particules virales, et l’ARN reste stable en raison de l’inhibition des RNAses34,35,36. La solution GITC a maintenu la stabilité des échantillons d’ARN sans tenir strictement compte de la chaîne du froid, ce qui a permis aux citoyens de maintenir les échantillons à température ambiante si un congélateur pour les blocs réfrigérants fournis n’était pas disponible. Afin de réduire le danger potentiel que pose ce réactif en cas de contact direct avec la peau ou les muqueuses, les citoyens ont été sensibilisés à ces risques par l’inclusion d’une fiche signalétique fournie dans la trousse et un sceau d’avertissement a été placé dans la boîte contenant les tubes.
La méthode brute d’extraction de GITC-chloroforme a aidé la récupération des traces d’ARN des écouvillons, et comme le montre l’amplification des échantillons enrichis, les inhibiteurs ont rarement persisté dans les échantillons après extraction. Les échantillons, qui étaient négatifs pour le SARS-CoV-2 et ne présentaient aucune inhibition de RT-LAMP, représentaient de vrais négatifs ou avaient un nombre de copies inférieur à celui de la LD à une fréquence de 100 %. Inversement, la détection de l’ARN viral sur une surface n’implique pas directement un risque de transmission par contact, car l’infectiosité du virus à partir d’échantillons positifs doit être testée. Un dépistage rapide de l’environnement, qui n’est pas limité par la disponibilité de fournitures sophistiquées ou de personnel hautement qualifié, est essentiel pour évaluer si les surfaces constituent un réservoir viral et pour mieux orienter les efforts de prévention et de confinement.
RT-LAMP a été choisi pour être la meilleure méthode adaptée au pipeline de détection proposé. Il s’est avéré être une méthode rapide et peu coûteuse qui était fortement résistante à la plupart des inhibiteurs restants et aussi sensible et spécifique que d’autres méthodes RT-qPCR. En raison de leur utilisation en milieu clinique pendant la pandémie de SARS-CoV-2, la disponibilité des kits RT-qPCR a été affectée par la demande mondiale. De plus, les techniques rt-qPCR , même celles formulées pour résister aux inhibiteurs, étaient sensibles aux substances contenues dans les échantillons environnementaux recueillis par une cohorte pilote de citoyens scientifiques, même après l’utilisation d’autres stratégies communes pour réduire la concurrence des inhibiteurs pour la liaison enzymatique49. Ces résultats sont corroborés par une étude récente qui a comparé les deux méthodes pour détecter le SARS-CoV-2 sur des échantillons d’écouvillons de bonbons manipulés par des patients COVID-19 et a trouvé une concordance de résultats de plus de 83%, avec une inhibition inférieure de 25% dans les échantillons analysés par RT-LAMP15. En outre, l’extraction du brut GITC-chloroforme, couplée à RT-LAMP, a réduit le coût des réactifs et des fournitures de 42% par rapport à l’extraction du kit d’ARN et au RT-qPCR (Table des matériaux).
Cette méthode a permis d’analyser à haut débit des milliers d’échantillons d’écouvillonnage de surface. Jusqu’à 80 piscines, représentant 640 échantillons, ont été traitées en 2 jours, de l’extraction de l’ARN à la détection du SARS-CoV-2 par RT-LAMP. Le protocole proposé est semi-quantitative, limité à la détection de l’ARN viral, et n’indique pas la présence de particules virales infectieuses. Une analyse plus approfondie est nécessaire pour évaluer le risque de transmission du SARS-CoV-2 par les vecteurs ennemis infectés présents aux surfaces écouvillonnés.
Cette étude présente un protocole pour mettre en place rapidement une stratégie de test qui comprend un flux de travail efficace face à une urgence sanitaire avec une maladie transmissible. Le protocole d’échantillonnage proposé est simple et utilise des fournitures que l’on trouve couramment dans les ménages, et la méthode de détection virale est effectuée sur de l’équipement disponible dans des laboratoires de base tels qu’un bain d’eau au lieu d’un thermocycleur. Les coûts des réactifs RT-LAMP sont nettement inférieurs à ceux nécessaires pour le RT-qPCR et moins sensibles aux scénarios de demande mondiale élevée. Cette étude sert de cadre à l’évaluation des réservoirs viraux environnementaux lors de futures épidémies et pandémies mondiales.
Déclarations de divulgation
Tous les auteurs déclarent qu’il n’existe pas d’intérêts concurrents.
Remerciements
Nous remercions les chercheurs du Viral Information Institute (VII), la Dre Anca M. Segall, Willow Segall, Patricia L. Rohwer, Gary Rohwer, Cary L. Rohwer, Magda Silvia Pinetta, Elizabeth Cruz Cano, le Dr Gregory Peters, le Dr Stuart A. Sandin et la Dre Jennifer Smith d’avoir pris le temps de prélever de nombreux échantillons. Nous remercions également le Dr Rob Knight, le Dr Jack Gilbert, le Dr Pedro Balda-Ferre et la Dre Sarah Allard du département de pédiatrie de l’École de médecine de l’Université de San Diego en Californie (UCSD) d’avoir facilité les contrôles positifs et les commentaires utiles. Nous remercions Stacey Carota (SDSU College of Sciences) et Gina Spidel (SDSU) pour leur soutien logistique et Juan Rodríguez pour l’art et la conception graphique du protocole d’échantillonnage. Nous remercions tous les participants pour leur engagement et leur dévouement à ce projet en ces temps très difficiles. Ce travail a été soutenu par un généreux don de la Dre Jo Ann Lane (SDSU College of Sciences) et de la subvention RAPID: Environmental Reservoirs of SARS-CoV-2 de la National Science Foundation (numéro de bourse : 2030479).
matériels
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
Références
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon