Method Article
도시 환경 면봉 - 환경 저수지에서 SARS-CoV-2의 샘플링 및 검출을 위한 파이프라인
요약
시민 과학 프로젝트는 SARS-CoV-2에 대한 환경 샘플을 수집하기 위해 샌디에고 주민을 모집하도록 설계되었습니다. 다국어 웹 기반 플랫폼은 사용자 친화적인 모바일 장치 인터페이스를 사용하여 데이터 제출을 위해 만들어졌습니다. 실험실 정보 관리 시스템은 실시간 결과 추적을 통해 지리적으로 다양한 수천 개의 샘플을 수집할 수 있도록 했습니다.
초록
2020년 글로벌 전염병 중 중 심각한 급성 호흡기 증후군 코로나바이러스 2(SARS-CoV-2)의 지역 사회 전염을 제어하기 위해 대부분의 국가는 직접적인 인간 검사, 얼굴 덮개 및 표면 소독에 기반한 전략을 구현했습니다. 전송의 주요 경로는 에어로졸과 호흡 물방울을 포함한다는 가정하에, fomites에서 SARS-CoV-2를 검출하는 노력은 높은 보급의 의심 되는 위치 (예를 들어, 병원 병동, 유람선, 및 대량 운송 시스템)에 초점을 맞추고있다. 거의 청소되지 않는 도시 환경의 표면에 SARS-CoV-2의 존재를 조사하기 위해, 350 시민은 더 큰 샌디에고 카운티에서 입대했다. 이 시민 과학자들은 총 4,080개의 샘플을 수집했습니다. 샘플링 키트 배달 및 픽업을 모니터링하고 샘플 데이터를 수집하기 위해 온라인 플랫폼이 개발되었습니다. 샘플링 키트는 대부분 전염병 스트레스 매장에서 사용할 수 있는 소모품으로 제작되었습니다. 시료는 재발공급 부족에도 불구하고 쉽게 접근할 수 있는 시약을 사용하여 처리되었습니다. 사용된 방법은 환경 견본에 일반적으로 존재하는 억제제에 높게 민감하고 저항했습니다. 제안된 실험 설계 및 처리 방법은 다양한 표면 영역에서 샘플을 효과적으로 수집한 수많은 시민 과학자들을 참여시키는 데 성공했습니다. 여기에 설명된 워크플로우와 방법은 공중 보건에 관한 것이고 미래의 전염병에 위협이 되는 다른 바이러스에 대한 도시 환경을 조사하는 데 관련이 있습니다.
서문
SARS-CoV-2는 감염된개인1,2,3,4와직접 접촉하여 오염된 에어로졸및 물방울의 흡입을 통해 주로 전염되는 것으로 생각된다. 그러나, 글로벌 COVID-19 전염병의 초기 단계에서 SARS-CoV-2의 전송을 통제하기 위한 노력은 표면 소독, 손씻기 및 살균에 강하게 집중했습니다. 2020년 말까지 세계보건기구(WHO)5와 미국 질병통제예방센터(CDC)의 전송 지침은 감염된 사람과 가까운 접촉(<2m)에 있거나 에어로졸 생성 의료 절차가 있을 때 주로 공중 전염위험이 있다고 판단했습니다. 오염된 표면과의 접촉 또는 에어로졸화 된 포미트의 흡입 과 접촉 한 후 자가 접종은 SARS-CoV-2의 전송 경로로 아직 배제되지 않았습니다.
COVID-19 케이스는 공중 전송이 가능성이 보인다보고되었습니다 7,8. SARS-CoV-2 virions는 최대 8시간 동안 구리, 골판지 및 스테인리스 스틸에서 최대 24시간 동안, 최대 48h9의플라스틱에 전염성을 유지합니다. 유람선 선빈에서 SARS-CoV-2 RNA는 승객이7번출발한 지 17일 후에 검출되었습니다. 병원 및 대중 교통 시스템에서 공기 및 표면 샘플SARS-CoV-2 및 기타 코로나바이러스8,10, 11,12,13,14에대해 양성 반응을 보이고 있다. 무증상 및 중등/경미한 증상 COVID-19 환자에 의해 처리된 할로윈 사탕의 외부 포장에 수행된 연구는 손잡이에 의한 손 세척과 손비누로 사탕을 세척하는 조합으로 SARS-CoV-2 RNA를 임계값 수준15이하로 감소시키는 것으로 결론을 내렸다.
SARS-CoV-2 진단에 대한 여러 가지 방법은 실시간 역전사 폴리머라제 연쇄 반응(RT-qPCR)16,17,역전사 루프 매개 이소성 증폭(RT-LAMP)11,18,19,20,21, 및 CRISPR-Cas 기술 18, 19, 19, 20, 21, 및 CRISPR-Cas 기술18, 19,19, 19, 20, 21 및 CRISPR-Cas 기술 18, 19, 19, 20, 21 및 CRISPR-Cas 기술 18,19, 19, 19,20, 21, 및 CRISPR-Cas 기술18, 19, 19,20,21,및 CRISPR-Cas 기술 18, 19,293을기반으로 발표되었습니다. 대부분은 상당한 글로벌 수요기간 동안 공급이 부족한 RNA 추출 키트를 필요로 하며,바이러스(24)의환경 검진에는 거의 사용되지 않았다. RT-LAMP를 사용하여 SARS-CoV-2 RNA의 검출은 RT-qPCR을 사용하는 데 83% 이상의 일치하는 것으로 입증되었습니다. 또한 RT-LAMP는 RT-qPCR 15에 비해 결정적인 결과를25%감소시켰습니다.
RT-LAMP는 RNA템플릿(25)으로부터cDNA를 합성하기 위해 역실을 사용하는 간단한 기술이며, 이어서 일정한 온도(즉, 이더스피증폭)에서DNA를 합성하는 강한 가닥 변위 활성을 가진 DNA 중합체를 이룬다. 바이러스 게놈 검출의 높은 특이성은 표적 DNA의 6 개 또는 8개의 지구를 인식하는 4개 또는 6개의 프라이머를 사용하여 달성됩니다. 증폭은 내부 프라이머에서 시작되고 반 이중 가닥 DNA 구조를 산출합니다. 선행 가닥은 바깥 쪽 프라이머에 의해 증폭됩니다. 이러한 증폭은 역 프라이머에 대해 반복됩니다. 양쪽 끝에 내부 및 외부 프라이머는 증폭제품(26)및27에서루프를 형성하는 내부 역방향 자체 보완 부위를 가지고 있다. 비등형 가닥 변위에서 비동기 DNA 합성은 연속 중합이 반응 당 10부로 적은 신호를 증폭하는 증폭된 제품의다량(11,20,28)을생성한다. 색수정 RT-LAMP 믹스는 약하게 버퍼링되고 페놀 레드를 pH 표시기로 사용합니다. 중합체는 뉴클레오티드를 통합함에 따라 양성자를 방출하고, 충분한 양성자는 용액의 pH뿐만 아니라 분홍색에서 노란색11,20,28,29로색상을 변경합니다.
RT-LAMP는 완전히 갖춰진실험실(25)이 부족한 주변 의료 시설에서 모기 매개 질병의 검출을 위해 개발되었으며, 인간 면역결핍바이러스(30)와같은 다른 RNA 바이러스의 신속한 검출을 위해 개발되었다. WHO 정의에 따라 전염병 발병에서 가장 취약한 인구는 종종 충분한 경제적 자원과 탐지를 수행하기위한 적절한 장비가 부족합니다 (유엔 글로벌 공중 보건 의제). 현재 SARS-CoV-2 전염병에서는 RNA 추출 키트에 대한 의료급 면봉 및 시약과 같은 공급이 특히 비제조 국가에서 는 글로벌 수요를 충족할 수 없었습니다. 제안된 프로토콜은 구안디늄 틸오카네이트(GITC)기반의 원유 RNA 추출을 사용하여, 이는 효과적으로 콜드 체인 독립적 인 방식으로 RNA를 보존하고 샘플에서 억제제의 지속성을 현저히 감소시키는. 더욱이, GITC-클로로폼 추출 프로토콜은 DNA와 단백질로부터의 RNA 분리를 기반으로 하며, 그 다음에는 각각의 강수량이 뒤따르며, 대부분의 유전 물질을 회수할 수 있다. 이러한 장점은 위험을 적절하게 알리기 위한 조치를 취하면 화학 물질을 취급하는 시민 과학자의 잠재적 위험보다 큽니다.
제안된 워크플로우는 일반적인 용도의 재료 및 시약을 사용합니다. 그것은 기본, 종종 시골, 실험실 설정에서 사용할 수있는 장비가 필요합니다. 이러한 방법은 추출 키트로 처리할 수 없는 환경 샘플이나 샘플에서 발견되는 억제제에 대한 고도로 저항력이 뛰어나며 고정밀 열순환기의 필요성을 제거합니다. 이 연구는 가정과 도시 환경의 일반적으로 만지고 드물게 감염된 표면에 환경 저수지에서 SARS-CoV-2의 샘플링 및 검출을위한 파이프 라인을 제시한다.
프로토콜
카탈로그 번호, 제조업체 및 해당 비용을 포함한 자세한 시약 및 소모품 목록은 재료 표를 참조하십시오.
1. 도시 환경 샘플링
- 시민 과학자 봉사 활동
- 지역 및 소셜 미디어를 통해 발표 된 직접적이고 명확한 행동을 사용하여 시민 과학자를 모집하십시오. 소셜 미디어 핸들(예: #swab4corona)을만들어 주제를 소셜 미디어 콘텐츠에 연결합니다.
- 관심 지역의 주요 언어(예: 샌디에이고 카운티의 스페인어 및 영어)에 능통하는 사람이 관리하는 실험실 팀과 각 시민 과학자 간의 직접 통신을 위한 이메일 계정을 만듭니다.
- 데이터베이스, 실험실 정보 관리 시스템(LIMS) 역할을 하고 시민 과학자들과 통신할 수 있는 안전한 웹 기반 샘플 관리 플랫폼(SMP)을 구축합니다.
참고: SMP는 사용자가 키트를 요청하고, 샘플 수집 프로토콜에 액세스하고, 샘플 메타데이터를 제출하고, 완성된 샘플 키트에 대한 픽업을 요청하는 중앙 집중식 위치를 제공합니다. - 온라인 형태로 지정된 생물안전 관련 질문에 답하여 개인이 환경 샘플링 활동에 참여할 수 있도록 SMP(예: https://demo.covidsample.org/)(그림1)에대한 링크를 만듭니다.
- 클라우드 컴퓨터 서비스 공급자가 촉진하는 인증 응용 프로그램 프로그래밍 인터페이스를 사용하여 SMP에 안전하게 액세스할 수 있습니다. 승인된 사용자에게 SMP에 대한 액세스 권한을 부여합니다.
참고: 인증 및 권한 부여를 위해 OAuth 2.0 프로토콜31에 대한 설명은 문헌을 참조하십시오. 그것은 시민 과학자 자원 봉사자를위한 마찰없는 로그인 프로세스를 제공합니다. 또한 사용자가 기존 계정으로 로그인할 수 있으므로 사용자 지정 로그인 솔루션을 만들고 사용자 자격 증명을 관리할 필요가 없으므로 상당한 시간을 절약하고 참여를 유도합니다. 최근 보고서에 따르면 선택한 클라우드 컴퓨터 서비스 공급자가 사용할 수 있는 무료 이메일 서비스에는 매월 약 15억 명의 활성 이메일 사용자가 있습니다. 참여를 위해 이 서비스 제공업체의 이메일 계정을 요구하는 것은 실망스러운 요인으로 간주되지 않습니다. - 첫 번째 키트를 요청하기 전에 SMP의 시민 과학자들에게 연구 및 생물 안전 고려 사항의 목적을 설명하십시오. 다국어 플러그인을 제공하여 클라우드 컴퓨터 서비스 공급자가 촉진하는 다국어 신경 기계 번역 서비스에서 사용할 수 있는 모든 언어로 탐색할 수 있도록 합니다.
- 영어와 스페인어로 샘플링 섹션 그래픽 및 시청각 프로토콜을 포함합니다.
- 각 키트에 고유 식별자를 할당하고 샘플 ID에 연결된 단추를 사용하여 데이터 입력프로세스(그림 1A)를간소화하도록 사용자 인터페이스를 설계합니다.
- 배달/픽업 경로를 최적화하고 시민 과학자들에게 정확한 예상 도착 시간을 알리기 위해 운전자가 사용하는 모바일 장치 응용 프로그램과 함께 배달 경로 계획 소프트웨어를 사용합니다.
- PHP 웹 서비스 스택에서 LIMS 플랫폼을 구축하고 상용 호스팅 플랫폼에서 호스팅합니다(제안된 운영 체제, 웹 서버 소프트웨어 및 데이터베이스 소프트웨어는 재료 표에지정됨).
- LIMS에서 랩 직원이 데이터를 빠르고 쉽게 관리할 수 있도록 안전한 웹 기반 응용 프로그램 인터페이스를 제공합니다. 클라우드 컴퓨터 서비스 공급자가 촉진하는 데이터 차트 응용 프로그램 프로그래밍 인터페이스를 사용하여 데이터 시각화를 제공합니다.
- 클라우드 컴퓨터 서비스 공급자가 촉진하는 지리 공간 응용 프로그램 프로그래밍 인터페이스를 사용하여 지리 공간 데이터를 시각화합니다. 프로젝트 데이터의 중앙 집중식 저장을 용이하게 하기 위해 SMP를 통해 LIMS에 제출된 데이터를 저장합니다. (2) 샘플/데이터 처리 워크플로우 추적; (3) 시민 과학자들에게 샘플 키트 배포물류의 관리.
- 모범 사례(예: https://demo.covidsample.org/)를사용하여 제출된 메타데이터를 보호합니다.
- 샘플 키트 ID, 샘플 ID, 날짜, 시간 및 글로벌 포지셔닝 시스템(GPS)과 같은 사전 로드 정보는 데이터 유형 준수를 가능하게 하고 사용자가 잘못하거나 누락된 데이터의 제출을 최소화하기 위해(사이트의 사진에서 자동으로 수집)를 조정합니다(그림1B). 시민 과학자가 입력한 다음 필드(<1분)를 수집 날짜와 시간, 위치 설명 및 샘플링 사이트의 그림과 같은 다음 필드를 포함합니다.
- 업로드된 모든 데이터를 소독하고 데이터 형식의 유효성을 검사합니다. 예를 들어 사용자가 업로드한 이미지 데이터의 유효성을 검사하여 파일을 .jpg 선택하고, 샘플과 빠르게 연결하기 위해 샘플 ID로 이름을 변경하고, 업로드된 이미지를 사용자가 액세스할 수 없는 별도의 보안 위치에 저장합니다.
- 모든 샘플(16)이 완료되었을 때 키트 배달 및 픽업을 요청하는 옵션을 활성화합니다. 또한 이전 키트를 픽업할 때 새 키트를 배달하도록 요청하는 옵션을 활성화합니다(그림1A).
참고: 비웹 기반 플랫폼을 선호하는 자원봉사자와 GPS 위치 공개에 대해 걱정하는 자원봉사자(예: 철새 상태에 대해 우려하는 커뮤니티 회원)의 경우 합의된 모임 위치에서 키트를 배달할 수 있으며 자원 봉사자는 데이터 수집의 서면 버전을 기록하도록 요청합니다. 실험실과 각 시민 과학자 사이의 통신을 위해, 전화 및 문자에 사용할 수있는 프로젝트의 이중 언어 구성원이있다.
- 코로나를 위한 면봉
- 샘플링 노력에 대한 역학적으로 관련된 시간 창을 식별합니다.
- 필요한 개인 보호 장비(즉, 마스크, 장갑), 샘플링 프로토콜 및 생물 안전 관련정보(그림 2)를포함하여 모든 샘플링 소모품을 포함하는 키트를 작성합니다. 할당된 고유 식별자(샘플 ID)로 각 튜브에 미리 레이블을 지정합니다.
- Swab은 가정과 도시 환경에서 에어로졸화 된 포미트에 노출되는 거의 소독되지 않습니다.
- 제공된 마스크와 각 시료의 수집을 위한 새로운 장갑을 착용하여 교차 오염을 방지합니다. 샘플링을 마친 후 제공된 손 소독제를 사용하십시오.
- 1cm2 폴리에스테르 흡수성 면봉(예를 들어, 걸프 패드)을 세제(예를 들어, 0.5% 나트륨 도데실 황산염(SDS)로 적어 봉투를 방해하여 바이러스를 비활성화하고 RNases32의전개를 유도하여 알몸 RNA를 안정화한다.
- 10cm2의표면을 면봉. 이쑤시개에 의해 지원, 완전히 구니디늄 티오야네이트 용액 (GITC)의 200 μL을 포함하는 해당 사전 라벨 튜브에 각 샘플 면봉을 침수. 튜브를 실험실로 운반할 때까지 4°C로 저장합니다. 샘플이 실험실에 도착하면 -80 °C에 보관하십시오.
참고: GITC는 독성 자극제입니다. 피부와의 접촉을 피하십시오. GITC 솔루션은 일반적인 실험실 화학 물질에서 준비하는 것이 간단합니다, 조리법은33,34를참조하십시오. 그것은 바이러스를 비활성화하고, RNases34,35,36을데스팅하여 RNA를 안정시키고, 실온에서 샘플을 안정화시. 그러나 이 키트에는 가정용 냉장고를 보관할 필요 없이 샘플을 차갑게 유지하기 위한 아이스 팩이 포함되어 있습니다.
2. SARS-CoV-2 감지
- 총 RNA 격리
- 2mM 구리 황산염 및 3% 과산화수소의 용액으로 표면, 장비 및 파이펫토를 소독하는 단계; 다음으로 표백제 10%, 중탄산나트륨 90m, SDS 5%, NaOH 2.5% 용액이 그 뒤를 이었다. 증류수로 철저히 닦아, 75% 에탄올이 뒤따릅니다.
참고: 이러한 솔루션은 시판되는 솔루션의 대안입니다. - 얼음에 샘플을 해동. 중간 속도로 2 분 동안 소용돌이 샘플을 채취한다.
- 스크리닝 속도를 높이려면 풀에서 샘플을 처리합니다. 풀이 양성인 경우, 양수 시료/s를 찾기 위해 각 샘플의 RNA를 독립적으로 추출한다. 각 샘플링 키트(총 16개)의 샘플을 8개의 샘플 2개의 풀에 결합합니다.
참고: 풀당 8개의 샘플을 보유하면 키트당 2개의 풀만 처리해야 합니다. 풀이 양수인 경우 개별 RT-LAMP 분석을 위해 개별 샘플을 다시 처리합니다. 이렇게 하면 시간, 비용 및 시약이 줄어듭니다. - 8개의 샘플 각각의 풀 50 μL을 미세 원심분리기 튜브로 넣습니다(총 부피 400 μL); 나머지 샘플을 -80°C에서 저장합니다. 클로로폼의 0.2부부(80μL)를 추가한 다음, 15초 동안 소용돌이를 한 다음 4°C에서 20분 동안 배양합니다. 원심분리기는 13,000 × g에서 4°C에서 20분 동안.
- 수성(투명 액체) 층을 새로운 미세원심분리기 튜브로 옮기다. -80°C 냉동고에 남은 인터페이스와 분홍색 액체를 저장; 이러한 분획은 DNA및단백질(33,36)을함유하고 있다.
- 동일한 부피량의 이소프로판올(~200 μL)과 글리코겐 계수(15mg mL-1)37의2.6 μL을 추가한다. 잘 섞고 -20°C에서 적어도 1시간 동안 배양하고, RNA를 침전시키기 위해 10분 동안 4°C가 뒤따릅니다.
참고: 프로토콜은 1h 대신 하룻밤 -20°C의 샘플을 배양하여 여기에서 일시 중지할 수 있습니다. - 원심분리기는 13,000 × g에서 4°C에서 20분 동안. 펠릿을 방해하지 않고 상체를 제거합니다. 50 μL의 디틸피로카보네이트(DEPC)처리된 물로 펠릿을 재연하고, RNase 가 없는 5M 암모늄 아세테이트의 동일한 부피(50 μL)와 100% 에탄올7,38의2.5부피량(250 μl)을 첨가한다.
참고: 암모늄 이온은 하류공정(38)에사용되는 경우 폴리뉴클레오티드 키나아제억제한다. 혼합은 탈옥뉴클레오사이드 삼중산염및 올리고당(38)을 방치하면서 RNA를침전시한다. - 잘 섞고 -20°C에서 적어도 1시간 동안 배양하고, RNA를 침전시키기 위해 10분 동안 4°C가 뒤따릅니다.
참고: 프로토콜은 1h 대신 하룻밤 -20°C의 샘플을 배양하여 여기에서 일시 중지할 수 있습니다. - 원심분리기는 13,000 × g에서 4°C에서 20분 동안. 펠릿을 1mL의 감기(-20°C)로 씻어내고, 갓 만든 75% 에탄올을 만들어 낸다. 원심분리기는 8,000 × g에서 4°C에서 5분 동안. 펠릿을 방해하지 않도록 P10 파이펫으로 상체를 제거합니다.
- 남은 에탄올이 없을 때까지 10-15분 동안 펠릿을 공기 건조시키십시오. DEPC 처리된 물의 50 μL에서 펠릿을 재연하고, 10배 DNase 버퍼 + 1μL의 5μL을 DNase(2단위μL-1)로추가하고 30분 동안 37°C에서 배양합니다.
- DNase 비활성화 시약의 0.1 부피 (5.6 μL)를 추가하고 실온에서 5 분 동안 배양하고 분마다 부드럽게 섞습니다. 원심분리기는 13,000 × g에서 2분 동안, 새로운 튜브(~50 μL)로 상류체를 전송합니다. RT-qPCR 또는 RT-LAMP 반응을 준비하는 동안 튜브를 얼음 에 즉시 놓거나 -20 °C 냉동고에 보관하십시오.
참고: 이월 오염을 방지하기 위해 앰플리턴이 없는 방에서 RNA 격리를 수행합니다.
- 2mM 구리 황산염 및 3% 과산화수소의 용액으로 표면, 장비 및 파이펫토를 소독하는 단계; 다음으로 표백제 10%, 중탄산나트륨 90m, SDS 5%, NaOH 2.5% 용액이 그 뒤를 이었다. 증류수로 철저히 닦아, 75% 에탄올이 뒤따릅니다.
- 멀티플렉스 역전사 루프-중재된 이더피 증폭(RT-LAMP)
- 프라이머의 각 세트에 대해 20X 프라이머 믹스 솔루션(표 1)을 준비한다(표 2) 피펫팅 손실을 고려하여 10%의 초과 부피로 실온에서 RT-LAMP 반응 믹스(표 3)를 준비합니다.
참고: 남극 열람실 우라실-DNA 글리코슬라제(UDG)와 컬러메트릭 램프 2X 마스터 믹스는 이전 반응20,28로부터DNA 오염의 증폭을 방지한다. - 소용돌이와 혼합물을 아래로 회전. 각 반응 튜브에 믹스의 20 μL을 분배 : 샘플, 스파이크, 긍정적 인 제어,및 음의 제어. UDG가 잠재적인 이월 오염에 작용할 수 있도록 실온에서 10분 동안 튜브의 반응을 배양합니다.
- 샘플 반응에 RNA 5μL, RNA 5μL + 합성 SARS-CoV-2 RNA의 2.5 μL(450부) 및 합성 SARS-CoV-2 RNA의 2.5 μL(450카피) 양성 대조군 반응에,H2O의 5μL을 첨가하여 음의 대조반응을 일으킨다. 잘 섞어서 반응을 줄입니다. 얼음에 모든 RNA를 해동.
- 열순환기 또는 수조는 65°C로 가열되는 동안 모든 반응이 실온에서 유지되도록 합니다. UDG는 >50°C에서 비활성화됩니다. 체온사이클러(열 뚜껑 사용) 반응을 65°C에서 40분 동안 배양하고, 반응이 실온(~22°C 5분)에 도달하도록 하거나, 얼음위에서 1분간 식힙니다. 착색 기능(간단한 관찰)을 사용하거나 아가로즈 젤로 제품을 실행하여 결과를 분석합니다.
참고: 검출 제한(LOD)은 반응당 10부이지만, 복사 수가 반응당 500부(그림3A)에가까워지면서 검출 빈도가 증가합니다. 색색 관찰의 경우 음수 결과가 분홍색(pH = 8.8)으로 표시되는 반면, 양수 결과는 노란색(pH = 5)(도3B)으로표시됩니다. 색상 옵션은 증폭 후 RT-LAMP 튜브의 개방을 방지하여 작업 환경에서 RT-LAMP 제품의 부피를 줄이고 이월 오염을 방지합니다. 젤 전기 전도의 경우, 0.5 % 트리스 / Borate / EDTA (TBE) 버퍼에 1X DNA 젤 얼룩으로 1.5 % 아가로즈 젤을 준비하십시오. 반응의 25 μL + 각 우물에 6X 로딩 염료의 5 μL을 로드합니다. 젤을 100 V에서 60분 동안 실행합니다. 양극 시료가 사다리패턴(도 3C)을나타내기 때문에 분자 마커가 필요하지 않다.
- 프라이머의 각 세트에 대해 20X 프라이머 믹스 솔루션(표 1)을 준비한다(표 2) 피펫팅 손실을 고려하여 10%의 초과 부피로 실온에서 RT-LAMP 반응 믹스(표 3)를 준비합니다.
결과
SARS-CoV-2의 검출을 위해 시민 과학자에 의해 집합된 견본. 8개월(3월 중순~2020년 11월 3주차) 동안 482명의 시민이 이 프로젝트에 참여하도록 승인되었으며, 그 중 350명(73%)이 이 사업에 참여할 수 있도록 승인되었습니다. 키트를 요청했습니다. 총 362개의 키트(즉, 일부 참가자는 여러 키트를 요청함) 및 246개(70%)가 전달되었습니다. 반환되었습니다 (그림 4A,B). 이 키트에 포함된 4,080개의 샘플은 모두 처리되었습니다. 수거 현장은 북부 해안, 노스 센트럴, 중부 및 남부 지방뿐만 아니라 동부 지역 (그림 4A)에배포되었습니다. 이 지구는 샌디에고 카운티의 가장 높은 인구 밀도와 가장 문서화 된 COVID-19 케이스를 가지고, 샌디에고 인간 보건 서비스 국에 의해보고 된 바와 같이39.
시민들은 대부분의 샘플링 키트(즉, 평균 성공률: 70.4%)를 픽업해 달라고 요청했습니다. 매일 1-16 키트가 요청되었고 0-14 키트가 실험실로 반환되었습니다(그림4B). 시민 과학자들을 대상으로 실시한 설문조사에 따르면 전체 키트(16개 샘플)의 수집은 평균 8일 동안 1~3시간 분포한 것으로나타났다(그림 4A 및 표 4). 키트의 대부분이 완료되었다 (91.1%), 그들은 모든 16 샘플 튜브 내부에 면봉을 포함 의미, 해당 샘플링 데이터는 LIMS에 업로드되었다(그림 4A 및 표 4).
GITC-클로로폼 추출 및 멀티플렉스 역전사 루프-중재된 이더피 증폭(RT-LAMP)을 사용하여 SARS-CoV-2검출. 색색RT-LAMP 분석법을 위해,프라이머(11,20,28)의 2세트는 뉴클레오캡시드(N2) 및 봉투(E1) 유전자(표 2)를표적으로 하는 데 사용되었다. 이러한 프라이머에 의해 인식 된 서열은 RT-qPCR에 의해 COVID-19의 인간 진단을 위해 CDC40 및 유럽 연합 (EU)41에 의해 승인 된 프라이머 및 프로브와 동일한 지역에 있습니다. 이러한 결과는 Zhang 외28이 설명한 것을 확증하며, 반응에 60mM guanidine 염산염을 첨가하면 멀티플렉스에서 실행될 때 LOD가 증가합니다. LOD는 100% 주파수에서 25 μL 반응당 500부였다(그림 3A,B). 색색 RT-LAMP에서, 양수 샘플은 ~8에서 5.5 (도 3B)에서pH 변화로 인해 분홍색에서 노란색으로 색상을 변경했습니다. 낮은 카피 숫자에서 반응이 주황색으로 바뀌었을 때, 샘플은 1.5% 아가로즈 젤로 실행되어 양성 반응을 보이고 사다리와 같은 패턴(도 3C)을초래하였다. RT-LAMP는 풀드린 RNA 샘플에서 SARS-CoV-2를 검출하는 데 사용되었습니다.
반응 억제제로 인한 거짓 네거티브를 제어하기 위해 각 샘플은 합성 SARS-CoV-2의 500사본으로 스파이크된 추가 반응에서 테스트되었습니다. 양성 풀로 된 샘플은 풀에서 각 개별 샘플의 RNA를 격리하고 RT-LAMP 반응으로 실행하여 양성 샘플의 정체성을 결정함으로써 복제하였다. 그런 다음 고유한 샘플 ID가 샘플의 날짜, 시간, GPS 좌표, 사이트 및 이미지와 페어링된 LIMS에 검색 결과를 업로드했습니다.
실시간 및 전통적인 RT-PCR 방법: 시료 오염물질에 의한 억제. 제안된 검출 파이프라인에 적합한 최상의 방법을 선택하기 위해, 시민 과학자의 파일럿 코호트에 의해 수집된 환경 샘플로 다른 RNA 증폭 방법을 테스트하였다. 이러한 각 방법의 결과의 예는 낮은 바이러스 복사 수 농도에서 환경 억제제 및 배경 신호 잡음에 대한 민감도를 나타내기 위해 도 5에 제시된다.
CDC와 WHO가 승인한 6개의 RT-qPCR 제형(재료표)은 환경 샘플에 대한 바이러스의 검출을 위해 테스트되었다. 프로토콜은 임상 설정40에서SARS-CoV-2의 검출을 위한 CDC 지침뿐만 아니라 제조업체의 지침에 따라 수행되었다. 합성 SARS-CoV-2 RNA 컨트롤의 상이한 농도를 포함하는 반응은 조루 RNA 절연 후에 면봉된 표면 샘플로 스파이크되었다. 모든 마스터 믹스는 양성 대조군(도5A)의LOD 농도에서 억제제에 민감하였다.
이러한 실시간 기술의 억제제를 우회하기 위해 기존의 RT-PCR 시스템을 테스트했습니다. 1단계 RT-PCR 시스템(재료의 표)은 CDC(미국)와 ECDC(EU)에 의해 승인된 프라이머 세트 E1을 이용하여 프라이머 세트 N1, N2,및 봉투 유전자를 이용하여 뉴클레오캡시드 유전자를 증폭시키는 데 사용되었다(표 2). 프로토콜은 임상 설정40에서SARS-CoV-2의 검출을 위한 CDC 지침뿐만 아니라 제조업체의 지침에 따라 수행되었다. 프라이머세트 N1 및 N2는 CDC가 설계한 제품으로 ~70bp 의 생성물을 산출한다; 그러나, 낮은 카피 수 긍정은 결과에 거짓 긍정을 도입한 음수 제어(도 5B)의증폭 배경 잡음과 구별되지 않았다. E1 프라이머의 제품은 낮은 복사 번호(도 5B)에서약한 신호를 보였으며, 결과에 거짓 네거티브를 도입하였다. 더욱이, 시험된 RT-PCR 방법은 환경 샘플에 존재하는 억제제에 여전히 민감하였다(데이터는 도시되지 않음).
다른 방법은 표적 서열의 매우 소량을 검출하기 위해 개발되었습니다. 이들 방법 중 하나는 압연 원 증폭(RCA)이며, 표적 서열, RNA 또는 DNA의 인식에 따라, 특정 선형 프로브에 의해, 리구선은 템플릿을 원형화한다. 프로브와 혼성화하도록 설계된 프라이머를 사용하여 가닥 변위 활성을 가진 DNA 폴리머라제는 이소성반응(42)에서프로브를 증폭시한다. 표적 시퀀스가 아니라 증폭되는 표적을 식별하는 프로브로, 이 방법은 매우 민감하게43이다. Wang et al.44는 SARS-CoV-1 RNA의 직접 검출을 위한 RCA 프로토콜을 발표했다. 이 방법은 SARS-CoV-2에 특정한 프라이머를 사용하도록 수정되었습니다. 불행히도, 비템플릿 제어(NTC)에서, 프로브는 SNP에 민감한 리구아제(ligase)를 포함한 다양한 리구슬을 사용하는 경우에도 RNA 템플릿이 없는 상태에서 제품을 순환하고 산출한다. 리그가 없는 경우, NTC는 선형 프로브(도 5C)로부터증폭을 나타내지 않았다.

그림 1: 모바일 기기용 샘플 수집 데이터 인터페이스가 있는 웹 기반 샘플링 플랫폼. (A)다국어 플러그인을 포함하는 웹 사이트가 실험실과 시민 과학자 간의 상호 작용을 중재하기 위해 만들어졌습니다. 이 플랫폼은 샘플 키트 전달/픽업 요청 및 샘플 데이터 제출에 사용되었습니다. 이 플랫폼에는 상세한 영어/스페인어 그래픽 및 시청각 샘플링 프로토콜이 포함되어 있습니다. (B)샘플 데이터를 업로드하는 데 사용되는 모바일 장치 보기: 날짜, 시간, GPS 좌표, 샘플 사이트 설명 및 수집 사이트의 이미지. 약어: SARS-CoV-2 = 심한 급성 호흡기 증후군 코로나바이러스 2; GPS = 글로벌 포지셔닝 시스템. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 샘플 수집 키트. 시민 과학자들은 GITC 솔루션, 상세한 샘플링 및 마스크 착용 프로토콜, KN95 마스크, GITC 솔루션 처리의 위험에 대해 자원 봉사자에게 알리기 위해 두 개의 얼음 팩, 안전 데이터 시트가 들어있는 쿨러를 받았습니다. 폐봉투, 손소독제가 있는 스프레이 병, 0.5%의 SDS가 있는 스프레이 병, 장갑 16켤레, 이쑤시개 16개, 폴리에스테르 면봉 16개, GITC 용액 200μL가 들어있는 마이크로센트심분리튜브 16개, 샘플링 튜브가 들어있는 상자, 이차 상자로 사용되는 가방. 약어: GITC = 구니디늄 티오야네이트; SDS = 나트륨 도데실 황산염. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
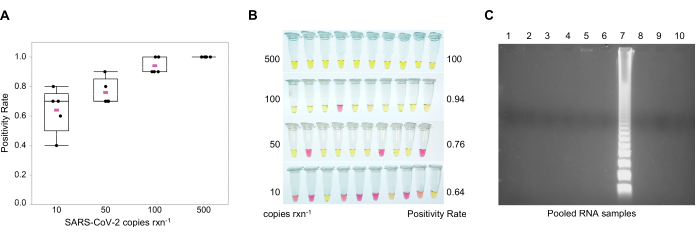
그림 3: 멀티플렉스 역전사 루프-중재 이소레말 증폭 (RT-LAMP) 분석. SARS-CoV-2 뉴클레오캡시드(N2) 및 봉투(E1) 유전자에 대한 프라이머를 이용한 멀티플렉스 반응은 반응에서 바이러스의 10개 카피만큼 적은 것을 검출한다. 합성 SARS-CoV-2 RNA는 양성 대조군으로 사용되었다. (A)반응당 상이한 게놈 카피 번호에서 SARS-CoV-2의 멀티플렉스 색법 RT-LAMP에서 검출 빈도. 5개의 평균 값이 분홍색으로 복제됩니다. (B)멀티플렉스 색색 RT-LAMP에서 SARS-CoV-2의 검출 제한(LOD); 노란색 = 양수(pH ~5); 분홍색 = 네거티브 (pH ~ 8). (C)1.5% 아가로즈 겔 전기포고에서 양성 SARS-CoV-2 RT-LAMP 반응의 사다리 패턴. 약어: SARS-CoV-2 = 심한 급성 호흡기 증후군 코로나바이러스 2; rxn = 반응. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
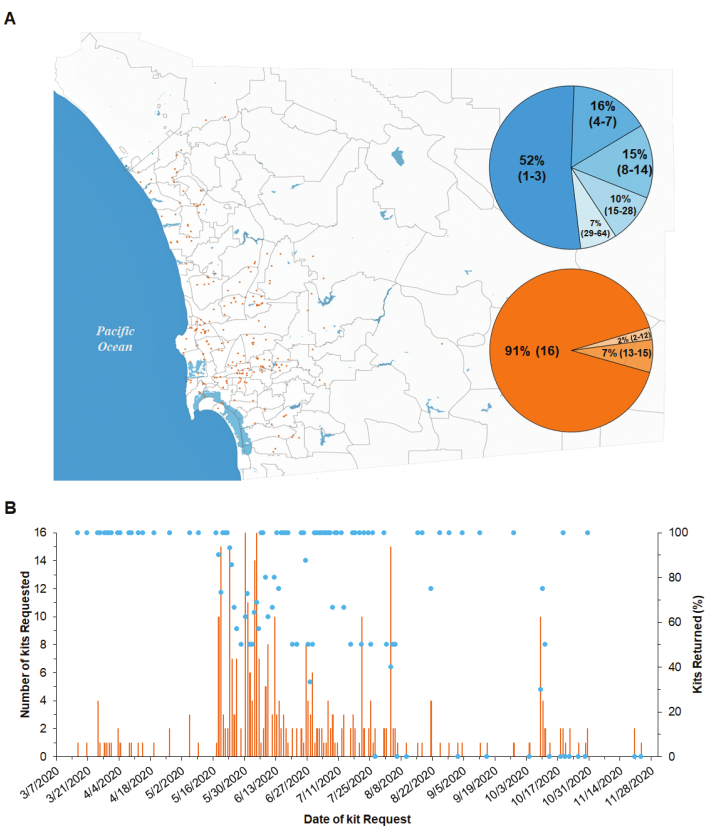
그림 4: 샌디에고 카운티의 시민 과학자 샘플링 키트의 위치와 요청 된 키트의 성공률. (A)오렌지 점은 16 개의 샘플을 포함하는 1 개의 샘플링 키트의 위치를 나타냅니다. 파란색 원형 차트는 시민 과학자들에게 전달된 때부터 실험실로 돌아왔을 때까지 다양한 며칠이 걸린 키트의 비율을 보여줍니다. 괄호 안에 있는 일 수입니다. 주황색 원형 차트는 총 16개의 샘플에서 완료된 샘플 수가 다른 키트의 백분율을 보여줍니다. 샘플 튜브 내부의 면봉을 포함하는 완성된 샘플 수와 괄호 안에 LIMS에 업로드된 해당 샘플링 데이터. (B)키트가 요청된 날짜를 기준으로 실험실(점)에 반환된 키트의 백분율 및 요청된 키트(막대)의 총 수입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
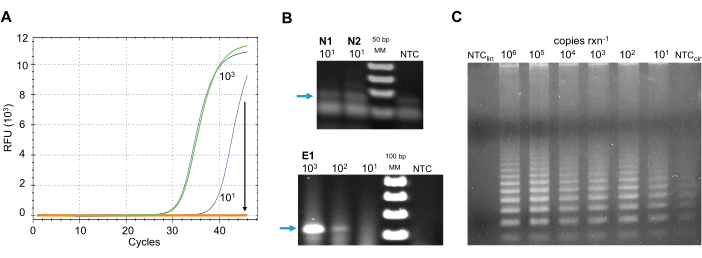
그림 5: 대체 SARS-CoV-2 RNAdetection 방법. (A)RT-qPCR 검출SARS-CoV-2 뉴클레오캡시드(N) 유전자를 사용하여 프라이머 세트 N2. 풀이 된 환경 샘플은 SARS-CoV-2의 900(녹색) 또는 9(주황색) 사본으로 급증했습니다. 파란색으로 동일한 복사 번호의 양수 컨트롤입니다. 화살표는 환경 샘플이 존재할 때 낮은 복사 수 양성 제어의 형광 검출의 감소를 나타냅니다. (B)SARS-CoV-2의 전통적인 RT-PCR 검출. 상단: 프라이머 세트를 사용하는 뉴클레오캡시드 유전자의 RT-PCR 제품은 N1 및 N2를설정한다. 템플릿 없음 컨트롤에서 희미한 배경 신호가 관찰됩니다. 아래쪽: 프라이머 세트 E1을이용한 봉투 유전자의 RT-PCR 제품. LOD 농도에서 매우 낮은 신호가 관찰됩니다. 파란색 화살표는 예상 된 긍정적 인 제품을 보여줍니다 : (상단) ~ 70 bp 및 (아래) 2 % 아가로즈 젤 전기 포에 113 bp. (C)SARS-CoV-2 RNA의 RCA. 원형 프로브는 RNA 템플릿(NTCcir)의리게아제 및 부재의 존재 속에서 증폭됩니다. 리구아제 및 RNA 템플릿 선형 프로브가 없는 경우 증폭되지 않는다(NTClin). 약어: SARS-CoV-2 = 심한 급성 호흡기 증후군 코로나바이러스 2; RFU = 상대형 광채 단위; bp = 기본 쌍; rxn = 반응; MM = 분자 마커; RT-PCR = 역전사 중합체 연쇄 반응; RT-qPCR = 실시간 정량적 RT-PCR; RCA = 롤링 원 증폭; NTC = 템플릿 없음 제어; LOD = 검출 제한; rxn = 반응. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 입문서 | 20배 농도(μM) | 1X 농도(μM) |
| FIP | 32 | 1.6 |
| BIP | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 | 4 | 0.2 |
| 루프F | 8 | 0.4 |
| 루프B | 8 | 0.4 |
표 1: 20X RT-LAMP 프라이머 믹스용 제형. RT-LAMP 반응에서, 6 프라이머는 표적 DNA의 8개의 영역을 인식합니다. 약어: 역전사 루프 중재 이더스몰 증폭; FIP = 전방 내부 프라이머; BIP = 후진 내부 프라이머; F3 = 전방 변위 프라이머; B3 = 후진 변위 프라이머; 루프F = 전방 루프 프라이머; 루프B = 뒤로 루프 프라이머.
| 입문서 | 순서 | 과녁 | 제품 크기 | |||
| RT 램프9,18,19 | ||||||
| E1-F3 | 트가타크가액트타트타트박트캣 | E | 사다리 와 같은 패턴 | |||
| E1-B3 | TTCAGATTTTAACACGAGAGT | |||||
| E1-FIP | ACCACGAAAGCAAAAAAA아가아가 | |||||
| E1-BIP | TTGC타택택타카틱스CCT타그타카아박틱 | |||||
| E1 루프B | GCGCTTCGATTGTGTGCGTGTGTGTGTGTGTGTGTGTGTGTGT | |||||
| E1 루프F | CGCTATTAACTATTAACG | |||||
| N2-F3 | 아가액타에카가카AGAGAG | N | ||||
| N2-B3 | GACTTGATCTTTGAAATTTGGATCTCT | |||||
| N2-FIP | TTCCGAAGAACGCTGAAGCGGAACTGAACTGAACTGAAAAACATGGCC | |||||
| N2-BIP | CGCATTGGCATGGAAGTCACAATTTGATGCACCTGTA | |||||
| N2 루프F | GGGGGCAAATTGTGCAATTTG | |||||
| N2-루프B | CTTCGGGAACGTGTTGACC | |||||
| RT-qPCR38 | ||||||
| 2019-nCoV_N1-F | 가카아아에카GCGAAAT | N | 72 bp | |||
| 2019-nCoV_N1-R | TCTGGTTACTGCCAGTTGAATCTG | |||||
| 2019-nCoV_N1-P | 5'-FAM-ACC CCG CAT TAC GTT TGG TGG ACC-BHQ1-3' | |||||
| 2019-nCoV_N2-F | 타카아카타카테그CCGCAAAAAAAAAAAAA | N | 67 bp | |||
| 2019-nCoV_N2-R | GCGCGACATTCCGA아가아 | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CAG CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | 아카그타그타타타그CGT | E | 113 bp | |||
| E1_Sarbeco_R | 아타트GCAGTACG카카 | |||||
표 2: RT-LAMP, RT-qPCR 및 RT-PCR에 사용되는 프라이머. 프라이머 서열, 표적 유전자, 예상 제품 크기 및 해당 참조가 나열됩니다. 약어: RT-LAMP = 역전사 루프-중재된 이더피 증폭; RT-PCR = 역전사 중합체 연쇄 반응; bp = 기본 쌍; RT-qPCR = 실시간 정량적 RT-PCR; E1 = 봉투 유전자; N2 = 뉴클레오캡시드 유전자; F = 포워드 프라이머; R = 역 프라이머; P = 프로브; FIP = 전방 내부 프라이머; BIP = 후진 내부 프라이머; F3 = 전방 변위 프라이머; B3 = 후진 변위 프라이머; 루프F = 전방 루프 프라이머; 루프B = 뒤로 루프 프라이머.
| 시약 | 부피(μL) |
| UDG와 웜 스타트 컬러측정 램프 2X 마스터 믹스 | 12.5 |
| N2 프라이머 믹스 (20x) | 1.25 |
| E1 프라이머 믹스 (20x) | 1.25 |
| 과니딘 염산염 (600 mM)* | 2.5 |
| 표적 RNA | 5 |
| 뉴클레아제 프리 H2O | 2.5 |
| 총 볼륨 | 25 |
표 3: 멀티플렉스 컬러메트릭 RT-LAMP용 반응 마스터 믹스. (*) 과니딘 염산염은 특징이 없는메커니즘(28)에의해 반응의 감도 및 속도를 증가시키는 것으로 나타났다. 약어: LAMP = 루프 매개 이더스몰 증폭; UDG = 우라실-DNA 글리코슬라세; N2 = 뉴클레오캡시드 유전자; E1 = 봉투 유전자; DEPC = 디틸피로카보네이트.
| 승인된 시민 | 키트를 요청한 시민 | 배달 된 키트 | 반환된 샘플링 키트 | 샘플링 전용 일 | % 완전 키트 | % 불완전한 키트 | 가공된 샘플 | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | 의미하다 | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| 중앙값 | 3 | |||||||
표 4:SARS-CoV-2용 스와빙 아웃리치 및 샘플링 성공률. 약어: SARS-CoV-2 = 중증 급성 호흡기 증후군 코로나바이러스 2.
토론
시민 과학자 참여. 시민 과학자들은 도시 환경에서 SARS-CoV-2의 존재를 샘플링하고 감지하기 위해 샌디에고 카운티 전역의 면봉 표면에 모집되었습니다. 배달된 샘플링 키트의 대부분(70%) 실험실로 반환되었고, 그 중 거의 모든 샘플이 완료되었습니다 (91%) (그림3A,B 및 표 4). 자원 봉사자는 웹 기반 플랫폼을 통해 키트 배달 / 픽업을 쉽게 요청할 수 있으며 배달 경로 계획 소프트웨어는 시민 과학자들에게 예상 도착 시간을 통보했으며, 관찰 된 성공에 중요한 요소가 될 수 있습니다. 키트가 시민 과학자에게 전달되었을 때실험실로 반환된 시점까지의 평균 시간은 8일, 중앙값은 3일, 1-64일(그림 3A 및 표 4)이었다. 자원봉사자들에게 더 자주 알림이 더 자주 주어질수록 이 지연 시간이 단축될 수 있습니다.
데이터 수집 플랫폼은 대다수의 사용자(73%)가 성공적으로 사용했습니다. (표4). 시민 과학자들의 노력은 측정되지 않았지만, 현장 테스트결과 데이터 수집 플랫폼은 샘플 수집을 제대로 완료하는 데 필요한 노력과 시간을 크게 줄였습니다. 따라서, 부기의 양을 줄이는 것은 시민 과학자참여를 장려했다. 웹 기반 플랫폼은 다국어 신경 기계 번역 서비스를 제공하고 영어와 스페인어로 그래픽 및 시청각 프로토콜을 제공함으로써 인구 통계학적 한계를 극복하기 위한 것입니다. 이것은 카운티의 히스패닉 / 라틴계 인구의 대부분이거주하는사우스 베이와 노스 카운티 모두에서 수집 된 샘플수가 적기 때문에 부분적으로만 성공했습니다. 이 지역은 또한 샌디에고 카운티에서 총 COVID-19 케이스의 63%(100,000명 당 1,700례)를 수용했으며, 질병46의 가장 높은 보급률과 입원률(62%)47,48. 대부분의 샘플은 센트럴 카운티에서 나왔지만, 가장 COVID-19에 영향을 받은 지역에서 대표적인 숫자를 수집했으며 샘플의 극히 일부만 양성으로 도시 환경에서 SARS-CoV-2의 표면 저수지가 상대적으로 드물다는 것을 시사합니다.
샘플 처리. 샘플링 면봉은 SDS로 젖어 봉투를 방해하여 바이러스를 활성화하고 RNases32를전개하여 알몸 RNA를 안정화시켰습니다. 수거 중 편리하게 면봉의 세제는 샘플링된 표면을 세척했습니다. 환경 샘플은 종종 매우 소량의 RNA를 함유하고 있습니다. 회복을 극대화하기 위해, RNA 절연은 GITC 기반, 컬럼 프리, 조잡한 추출 방법을 사용하여 수행되었다. GITC, 강한 chaotropic 에이전트, 단백질 접이식 유지 하는 수소 결합을 방해 (즉, 소수 성 효과). 이러한 작용은 바이러스 성 입자의 불활성화를 초래하고, RNA는RNA(34),35,36의억제로 인해 안정적으로 유지된다. GITC 용액은 엄격한 콜드 체인 고려 없이 RNA 샘플의 안정성을 유지하여 제공된 얼음 팩용 냉동고를 사용할 수 없는 경우 시민들이 실온에서 시료를 유지할 수 있도록 했습니다. 직접적인 피부 나 점막 접촉이 발생할 때 이 시약이 발생할 때 이러한 위험 위험을 줄이기 위해 시민들은 키트에 제공된 재료 안전 데이터 시트를 포함시킴으로써 이러한 위험을 인식하고 튜브를 포함하는 상자에 경고 씰을 배치했습니다.
조잡한 GITC-클로로폼 추출 방법은 면봉에서 RNA의 흔적의 회복을 도왔으며, 스파이크 샘플의 증폭에 의해 도시된 바와 같이, 억제제는 추출 후 샘플에서 거의 지속되지 않습니다. SARS-CoV-2에 대해 부정적이고 RT-LAMP 억제가 나타나지 않은 샘플은 진정한 네거티브를 나타내거나 100% 주파수에서 LOD보다 낮은 복사 번호를 가졌다. 반대로, 표면에서 바이러스 RNA의 검출은 양성 샘플에서 바이러스의 감염성을 테스트해야 하기 때문에 접촉을 통한 전염위험을 직접적으로 의미하지는 않는다. 정교한 공급품또는 고도의 자격을 갖춘 인력의 가용성에 의해 제한되지 않는 환경의 신속한 선별은 표면이 바이러스 성 저수지를 구성하는지 여부를 평가하고 보다 직접적인 예방 및 봉쇄 노력을 하는 데 매우 중요합니다.
RT-LAMP는 제안된 검출 파이프라인에 적합한 최상의 방법으로 선택되었다. 그것은 나머지 억제제의 대부분에 매우 저항 하고 다른 RT-qPCR 방법으로 민감 하 고 특정 신속 하 고 저렴 한 방법 입증. SARS-CoV-2 전염병 동안 임상 설정에서 사용했기 때문에 RT-qPCR 키트의 가용성은 글로벌 수요에 의해 영향을 받았습니다. 더욱이, RT-qPCR 기술은 억제제에 저항하기 위하여 공식화된 그조차 - 효소 결합에 대한 억제제 경쟁을 감소시키기 위하여 그밖 일반적인 전략의 사용 후에도 시민 과학자의 파일럿 코호트에 의해 집합된 환경 견본에 포함된 물질에 민감했습니다49. 이러한 사실 인정은 COVID-19 환자에 의해 취급된 사탕에서 면봉 견본에 SARS-CoV-2를 검출하는 두 가지 방법을 비교하고 RT-LAMP15에의해 분석된 견본에 있는 25% 더 낮은 억제와 더불어 83% 결과 일치를 찾아낸 최근 연구 결과에 의해 확증됩니다. 또한, GITC-클로로폼 원유 추출은 RT-LAMP와 결합되어 RNA 키트 추출 및 RT-qPCR(재료 표)에 비해 시약 및 공급 비용을 42% 절감하였다.
이 방법을 사용하면 수천 개의 표면 면봉 샘플에 대한 높은 처리량 분석을 허용했습니다. 640개의 샘플을 나타내는 최대 80개의 풀은 RNA 추출에서 RT-LAMP에 의한 SARS-CoV-2 검출까지 2일 동안 처리되었습니다. 제안된 프로토콜은 반정성, 바이러스 RNA의 검출에 국한되고, 감염성 바이러스 입자의 존재를 나타내지 않는다. 추가 분석은 swabbed 표면에 존재하는 감염된 포미트에서 SARS-CoV-2의 전송의 리스크를 평가하기 위하여 요구됩니다.
이 연구는 전염성 질환으로 건강 비상 사태에 직면 했을 때 효과적인 워크플로우를 포함 하는 테스트 전략을 신속 하 게 설정 하는 프로토콜을 제시 합니다. 제안된 샘플링 프로토콜은 간단하고 가정에서 흔히 발견되는 소모품을 사용하며, 바이러스 검출 방법은 열사이클기 대신 수조와 같은 기본 실험실 환경에서 사용할 수 있는 장비에서 수행됩니다. RT-LAMP 시약의 비용은 RT-qPCR에 필요한 비용보다 현저히 낮으며 높은 글로벌 수요 시나리오에 덜 취약합니다. 이 연구는 미래의 전염병 발병과 글로벌 전염병에 환경 바이러스 성 저수지의 평가를위한 프레임 워크 역할을합니다.
공개
모든 저자는 경쟁적인 이해관계가 존재하지 않는다고 선언합니다.
감사의 말
우리는 바이러스 정보 연구소 (VII) 조사관, 박사 앙카 M. 세갈, 윌로우 세갈, 패트리샤 L. 로워, 게리 로워, 캐리 L. 로워, 막다 실비아 피네타, 엘리자베스 크루즈 카노, 박사 그레고리 피터스, 박사 스튜어트 A. 샌딘, 박사 제니퍼 스미스에게 많은 샘플을 수집하기 위해 시간을 복용. 우리는 또한 긍정적인 통제 및 유용한 피드백을 촉진하기위한 샌디에고 캘리포니아 의과 대학 (UCSD)의 소아과부에서 박사 롭 나이트, 잭 길버트 박사, 페드로 발다 - 페레 박사, 박사 사라 알라드 감사합니다. 스테이시 카로타(SDSU 과학대학)와 지나 스피델(SDSU)에게 물류 지원에 감사드리며, 후안 로드리게스는 샘플링 프로토콜의 예술과 그래픽 디자인에 감사드립니다. 우리는 매우 어려운 시기에이 프로젝트에 대한 헌신과 헌신에 대한 모든 참가자들에게 감사드립니다. 이 작품은 조 앤 레인 박사 (SDSU 과학 대학)와 국립 과학 재단 RAPID : SARS-CoV-2 보조금의 환경 저수지 (상 번호 : 2030479)의 관대 한 기부에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
참고문헌
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유