Method Article
Limpando o Ambiente Urbano - Um Pipeline para Amostragem e Detecção de SARS-CoV-2 de Reservatórios Ambientais
Neste Artigo
Resumo
Um projeto de ciência cidadã foi projetado para recrutar residentes de San Diego para coletar amostras ambientais para sars-cov-2. Uma plataforma multilíngue baseada na Web foi criada para envio de dados usando uma interface de dispositivo móvel fácil de usar. Um sistema de gerenciamento de informações laboratorial facilitou a coleta de milhares de amostras geograficamente diversas com rastreamento de resultados em tempo real.
Resumo
Para controlar a transmissão comunitária da síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2) durante a pandemia global de 2020, a maioria dos países implementou estratégias baseadas em testes humanos diretos, cobertura facial e desinfecção da superfície. Sob o pressuposto de que a principal rota de transmissão inclui aerossóis e gotículas respiratórias, os esforços para detectar SARS-CoV-2 em fomites se concentraram em locais suspeitos de alta prevalência (por exemplo, enfermarias hospitalares, navios de cruzeiro e sistemas de transporte de massa). Para investigar a presença de SARS-CoV-2 em superfícies no ambiente urbano que raramente são limpas e raramente desinfetadas, 350 cidadãos foram alistados do grande condado de San Diego. No total, esses cientistas cidadãos coletaram 4.080 amostras. Uma plataforma online foi desenvolvida para monitorar a entrega e coleta do kit de amostragem, bem como para coletar dados amostrais. Os kits de amostragem foram construídos principalmente a partir de suprimentos disponíveis em lojas com estresse pandêmico. As amostras foram processadas com reagentes de fácil acesso, apesar da recorrente escassez de oferta. Os métodos utilizados foram altamente sensíveis e resistentes aos inibidores comumente presentes em amostras ambientais. Os métodos experimentais de design e processamento propostos foram bem sucedidos em envolver numerosos cientistas cidadãos que efetivamente coletaram amostras de diversas áreas de superfície. O fluxo de trabalho e os métodos aqui descritos são relevantes para o levantamento do ambiente urbano para outros vírus, que são de interesse da saúde pública e representam uma ameaça para futuras pandemias.
Introdução
Acredita-se que o SARS-CoV-2 seja transmitido principalmente através da inalação de aerossóis contaminados e gotículas de contato direto com indivíduos infectados1,2,3,4. No entanto, durante as fases iniciais da pandemia GLOBAL COVID-19, os esforços para controlar a transmissão do SARS-CoV-2 se concentraram fortemente na desinfecção de superfícies, lavagem das mãos e higienização. Até o final de 2020, as diretrizes de transmissão da Organização Mundial da Saúde (OMS)5 e dos Centros de Controle e Prevenção de Doenças dos EUA (CDC)6 consideraram a transmissão aérea um perigo principalmente quando em contato próximo (<2 m) com uma pessoa infectada ou na presença de procedimentos médicos geradores de aerossol. A auto-inoculação após o contato com superfícies contaminadas ou inalação de fomites aerossolizadas ainda não foram descartadas como uma rota de transmissão de SARS-CoV-2.
Os casos COVID-19 foram relatados onde a transmissão aérea parece improvável7,8. Os virions SARS-CoV-2 permanecem infecciosos em cobre por até 8h, em papelão e aço inoxidável por até 24 h, e em plástico por até 48 h9. Nas cabines de navios de cruzeiro, o SARS-CoV-2 RNA foi detectado 17 dias após os passageiros terem partido7. Amostras de ar e superfície de hospitais e sistemas de transporte coletivo deram positivo para SARS-CoV-2 e outros coronavírus8,10,11,12,13,14. Um estudo realizado na embalagem externa de doces de Halloween manuseados por pacientes assintomáticos e moderados/levemente sintomáticos COVID-19, concluiu que a combinação de lavagem das mãos pelo manipulador e lavagem dos doces com sabão manual reduziu sars-cov-2 RNA para níveis inferioresa 15.
Vários métodos para diagnóstico sars-cov-2 foram publicados com base na reação em cadeia de polimerase de transcrição reversa em tempo real (RT-qPCR)16,17, amplificação isotéria mediada por loop reverso (RT-LAMP)11,18,19,20,21, e tecnologias CRISPR-Cas18,19,22,23. A maioria exige kits de extração de RNA que muitas vezes estão em escassez durante períodos de demanda global significativa, e muito poucos têm sido usados para a triagem ambiental do vírus24. A detecção de RNA SARS-CoV-2 usando RT-LAMP demonstrou ser mais de 83% concordante com o uso de RT-qPCR. Além disso, o RT-LAMP resultou em redução de 25% nos resultados inconclusivos em comparação com o RT-qPCR15.
RT-LAMP é uma técnica simples que usa uma transcriptase reversa para sintetizar cDNA a partir de um modelo RNA25, seguido por uma polimerase de DNA com forte atividade de deslocamento de fios que sintetiza o DNA em temperatura constante (ou seja, amplificação isotemal)26. A maior especificidade da detecção do genoma viral é alcançada usando quatro ou seis primers que reconhecem seis ou oito regiões do DNA alvo. A amplificação é iniciada a partir de uma cartilha interna e produz uma estrutura de DNA semi-duplamente encalhada. A linha principal é então amplificada por uma cartilha externa. Essas amplificações são repetidas para os primers invertidos. Primers internos e externos em ambos os lados possuem um site interno reverso auto-complementar que forma um loop no produto de amplificação26,27. No deslocamento isoteérmico da vertente, a síntese de DNA assíncrono gera altas quantidades de produto amplificado onde a polimerização contínua amplifica o sinal de apenas 10 cópias por reação11,20,28. A mistura RT-LAMP colorimétrica é fracamente tamponada e usa o vermelho fenol como indicador de pH. Como a polimerase incorpora um nucleotídeo, ele libera um próton, e prótons suficientes mudarão o pH da solução, bem como sua cor de rosa para amarelo11,20,28,29.
O RT-LAMP foi desenvolvido para a detecção de doenças transmitidas por mosquitos em unidades de saúde periféricas que não possuem laboratórios totalmente equipados25 e para a detecção rápida de outros vírus de RNA, como o vírus da imunodeficiência humana30. As populações mais vulneráveis em surtos epidêmicos — de acordo com a definição da OMS — muitas vezes não têm recursos econômicos suficientes e equipamentos apropriados para realizar a detecção (Agenda Global de Saúde Pública das Nações Unidas). Na atual pandemia SARS-CoV-2, os suprimentos como cotonetes de grau médico e reagentes para kits de extração de RNA não foram capazes de atender à demanda global, especialmente em países não manufaturados. O protocolo proposto utilizou uma extração de RNA bruta baseada em guanidinium (GITC), que efetivamente preservou o RNA de forma independente da cadeia fria e reduziu significativamente a persistência dos inibidores da amostra. Além disso, o protocolo de extração gitc-clorofórmio baseia-se na separação do RNA do DNA e proteínas seguida pela respectiva precipitação, permitindo a recuperação da maior parte do material genético. Essas vantagens superam os perigos potenciais dos cientistas cidadãos que lidam com o produto químico se forem tomadas medidas para informá-los adequadamente dos riscos.
O fluxo de trabalho proposto utiliza materiais e reagentes de uso geral. Requer equipamentos disponíveis em ambientes básicos, muitas vezes rurais, laboratoriais. Esses métodos são baratos, altamente resistentes a inibidores frequentemente encontrados em amostras ambientais ou amostras que não podem ser processadas com kits de extração, e eliminam a necessidade de um termociclador de alta precisão. Este estudo apresenta um pipeline para amostragem e detecção de SARS-CoV-2 de reservatórios ambientais em superfícies comumente tocadas e raramente desinfetadas das residências e do ambiente urbano.
Protocolo
Consulte a Tabela de Materiais para obter uma lista detalhada de reagentes e suprimentos, incluindo números de catálogo, fabricante e custos correspondentes.
1. Amostragem do ambiente urbano
- Divulgação cientista cidadã
- Recrute cientistas cidadãos usando um chamado direto e claro lançado através de mídias locais e sociais. Crie uma alça de mídia social (por exemplo, #swab4corona) para conectar o tópico através do conteúdo das mídias sociais.
- Crie uma conta de e-mail para comunicação direta entre a equipe do laboratório e cada cientista cidadão, gerenciada por uma pessoa fluente nas principais línguas da região de interesse (por exemplo, espanhol e inglês para o condado de San Diego).
- Construa uma plataforma segura de gerenciamento de amostras baseada na Web (SMP) para servir como banco de dados, um sistema de gerenciamento de informações laboratorial (LIMS) e para se comunicar com cientistas cidadãos.
NOTA: O SMP fornece um local centralizado onde os usuários solicitam um kit, acessam protocolos de coleta de amostras, enviam metadados de amostra e solicitam uma coleta para kits de amostra concluídos. - Crie um link para o SMP (por exemplo, https://demo.covidsample.org/)(Figura 1) para que os indivíduos se inscrevam para participar do esforço de amostragem ambiental, respondendo perguntas relacionadas à biossegurança especificadas de forma online.
- Acesso seguro ao SMP usando uma interface de programação de aplicativos de autenticação facilitada por um provedor de serviços de computador em nuvem. Dê aos usuários aprovados acesso ao SMP.
NOTA: Consulte a literatura para obter uma descrição do protocolo OAuth 2.031 para autenticação e autorização. Ele fornece um processo de login sem atrito para voluntários cientistas cidadãos. Ele também permite que os usuários façaem login com uma conta existente, eliminando a necessidade de criar uma solução de login personalizada e gerenciar credenciais do usuário, o que economiza uma quantidade significativa de tempo e incentiva a participação. De acordo com relatórios recentes, o serviço de e-mail gratuito disponível para o provedor de serviços de computador em nuvem escolhido tem aproximadamente 1,5 bilhão de usuários de e-mail ativos mensais; exigir uma conta de e-mail deste provedor de serviços para participação não é considerado um fator desanimador. - Explique o objetivo do estudo e considerações de biossegurança aos cientistas cidadãos do SMP antes de solicitarem seu primeiro kit. Forneça um plugin multilíngue para permitir a navegação em qualquer um dos idiomas disponíveis a partir de um serviço de tradução de máquina neural multilíngue facilitado por um provedor de serviços de computador em nuvem.
- Inclua na seção de amostragem protocolos gráficos e audiovisuais em inglês e espanhol.
- Atribua um identificador exclusivo a cada kit e projete a interface do usuário para utilizar botões vinculados ao Sample ID para agilizar o processo de entrada de dados (Figura 1A).
- Use um software de planejamento de rota de entrega com um aplicativo de dispositivo móvel para ser usado pelos motoristas para otimizar rotas de entrega/coleta e notificar os cientistas cidadãos dos tempos estimados precisos de chegada.
- Construa a plataforma LIMS em uma pilha de serviços web PHP e hospede-a em uma plataforma de hospedagem comercial (sistema operacional sugerido, software de servidor web e software de banco de dados são especificados na Tabela de Materiais).
- Forneça uma interface de aplicativo baseada na Web segura para permitir que o pessoal do laboratório gerencie dados de forma rápida e fácil no LIMS. Forneça visualização de dados usando uma interface de programação de aplicativos de mapeamento de dados facilitada por um provedor de serviços de computador em nuvem.
- Visualize dados geoespaciais usando uma interface de programação de aplicativos geoespacial facilitada por um provedor de serviços de computador em nuvem. Armazenar os dados enviados ao LIMS através do SMP para facilitar (1) o armazenamento centralizado dos dados do projeto; (2) rastreamento dos fluxos de trabalho de processamento de amostras/dados; e (3) gestão da logística de distribuição de kits de amostras para cientistas cidadãos.
- Garantir metadados enviados usando as melhores práticas (por exemplo, https://demo.covidsample.org/).
- As coordenadas de Pré-carga, como ID do Kit de Amostra, ID de amostra, data, hora e GPS (coletados automaticamente a partir de uma imagem do site) para permitir a conformidade do tipo de dados e minimizar o envio de dados errôneos ou ausentes pelo usuário(Figura 1B). Inclua os seguintes campos a serem manualmente e rapidamente (<1 min) preenchidos pelo cientista cidadão: data e hora da coleta, uma breve descrição do local e uma foto do local de amostragem.
- Higienize todos os dados carregados e valide para o tipo de dados. Por exemplo, valide os dados de imagem carregados pelos usuários para selecionar .jpg arquivos, renomeá-los com Sample ID para rápida associação com a amostra e armazenar imagens carregadas em um local seguro separado não acessível aos usuários.
- Ative a opção de solicitar a entrega e a coleta do kit quando todas as amostras (16) tiverem sido concluídas. Além disso, ative a opção de solicitar um novo kit a ser entregue no momento da retirada do anterior (Figura 1A).
NOTA: Para voluntários que preferem uma plataforma não baseada na Web e para aqueles preocupados em divulgar sua localização gps (por exemplo, membros da comunidade preocupados com seu status migratório), os kits podem ser entregues em um local de reunião acordado e os voluntários solicitados a gravar uma versão por escrito da coleta de dados. Para comunicação entre o laboratório e cada cientista cidadão, tenha um membro bilíngue do projeto disponível para chamadas telefônicas e textos.
- Cotonete para Corona
- Identificar uma janela de tempo epidemiologicamente relevante para o esforço amostral.
- Construa um kit que contenha todos os suprimentos de amostragem, incluindo os equipamentos de proteção individual necessários (ou seja, máscara, luvas), um protocolo de amostragem e informações relevantes de biossegurança(Figura 2). Pré-rotule cada tubo com o identificador único atribuído (Sample ID).
- O swab raramente desinfetou superfícies expostas a fomites aerossolizadas em domicílios e no ambiente urbano.
- Use a máscara fornecida em público e um novo par de luvas para a coleta de cada amostra para evitar contaminação cruzada. Após o término da amostragem, use o desinfetante para as mãos fornecido.
- Molhe um cotonete absorvente de poliésterde 1 cm 2 (por exemplo, pastilhas de esfregão) com um detergente (por exemplo, sulfato de dodecyl de sódio de 0,5% (SDS)) para inativar o vírus interrompendo seu envelope e estabilizando o RNA nu induzindo o desdobramento de RNases32.
- Swab uma superfície de 10 cm2. Auxiliado por um palito, submerga completamente cada amostra no tubo pré-rotulado correspondente contendo 200 μL de solução de tiocianato de guanidinium (GITC). Armazene os tubos a 4 °C até serem transportados para o laboratório. Assim que as amostras chegarem ao laboratório, armazene-as a -80 °C.
NOTA: O GITC é um irritante tóxico; evite o contato com a pele. Solução GITC é simples de preparar a partir de produtos químicos de laboratório comuns, para a receita ver33,34. Inativa o vírus, estabiliza o RNA desnaturando RNases34,35,36e estabiliza amostras à temperatura ambiente. O kit, no entanto, inclui blocos de gelo para manter as amostras frias sem a necessidade de usar geladeiras domésticas para armazenamento.
2. Detecção de SARS-CoV-2
- Isolamento total do RNA
- Superfícies desinfetante, equipamentos e pipettors com solução de sulfato de cobre de 2 mM e peróxido de hidrogênio de 3%; seguido por uma solução de 10% alvejante, 90 mM de bicarbonato de sódio, 5% SDS e 2,5% NaOH. Limpe bem com água destilada seguida de 75% de etanol.
NOTA: Essas soluções são uma alternativa às soluções disponíveis comercialmente. - Descongelar amostras no gelo. Amostras de vórtice por 2 minutos em velocidade média.
- Para aumentar a velocidade da triagem, processe as amostras em piscinas. Se um pool for positivo, extraia o RNA de cada amostra independentemente para encontrar a amostra/s positivo. Combine as amostras de cada kit amostral (16 total) em 2 piscinas de 8 amostras.
NOTA: Ter 8 amostras por piscina significa que apenas 2 piscinas precisam ser processadas por kit. Se um pool for positivo, as amostras individuais ão reprocessadas para análise individual de RT-LAMP. Isso reduz o tempo, os custos e os reagentes. - Piscina 50 μL de cada uma das 8 amostras em um tubo de microcentrífuga (volume total 400 μL); salvar a amostra restante a -80 °C. Adicione 0,2 volumes (80 μL) de clorofórmio, vórtice para 15 s e, em seguida, incubar por 20 min a 4 °C. Centrifugar a 13.000 × g por 20 min a 4 °C.
- Transfira a camada aquosa (líquido claro) para um novo tubo de microcentrífuga. Armazenar a interface restante e o líquido rosa no congelador -80 °C; essas frações contêm DNA e proteínas33,36.
- Adicione um volume igual de isopropanol (~200 μL) e 2,6 μL de coprecipitante glicogênio (15 mg mL-1)37. Misture bem e incubar a -20 °C por pelo menos 1 h, seguido de 4 °C por 10 min para precipitar o RNA.
NOTA: O protocolo pode ser pausado aqui incubando amostras a -20 °C durante a noite em vez de 1h. - Centrifugar a 13.000 × g por 20 min a 4 °C. Remova o supernatante sem perturbar a pelota. Resuspenque a pelota em 50 μL de água tratada com dietilpiroto (DEPC) e adicione um volume igual (50 μL) de acetato de amônio sem RNase 5 M e 2,5 volumes (250 μl) de 100% de etanol7,38.
NOTA: Íons de amônio inibem a quinase de polinucleotídeo se usado em um processo a jusante38. A mistura precipita o RNA ao deixar triphosfatos e oligossacarídeos desoxicomíno na solução38. - Misture bem e incubar a -20 °C por pelo menos 1 h, seguido de 4 °C por 10 min para precipitar o RNA.
NOTA: O protocolo pode ser pausado aqui incubando amostras a -20 °C durante a noite em vez de 1h. - Centrifugar a 13.000 × g por 20 min a 4 °C. Lave a pelota com 1 mL de frio (-20 °C), recém-feito 75% de etanol. Centrifugar a 8.000 × g por 5 min a 4 °C. Remova o supernatante com uma pipeta P10 para evitar perturbar a pelota.
- Aeródromo secou a pelota por 10-15 min até que não haja etanol restante. Resuspenque a pelota em 50 μL de água tratada com DEPC, adicione 5 μL de 10x DNase tampão + 1μL de DNase (2 Unidades μL-1), e incubar a 37 °C por 30 min.
- Adicione 0,1 volumes (5,6 μL) de reagente de inativação DNase, incubar à temperatura ambiente por 5 minutos e misturar suavemente a cada minuto. Centrifugar a 13.000 × g por 2 min, e transferir supernante para um novo tubo (~50 μL). Coloque o tubo no gelo imediatamente durante o preparo das reações RT-qPCR ou RT-LAMP, ou armazene em um congelador de -20 °C.
NOTA: Realize o isolamento do RNA em uma sala livre de amplicon para evitar contaminação por atropelamento.
- Superfícies desinfetante, equipamentos e pipettors com solução de sulfato de cobre de 2 mM e peróxido de hidrogênio de 3%; seguido por uma solução de 10% alvejante, 90 mM de bicarbonato de sódio, 5% SDS e 2,5% NaOH. Limpe bem com água destilada seguida de 75% de etanol.
- Amplificação isotérmica mediada por loop de transcrição reversa multiplex (RT-LAMP)
- Prepare uma solução de mix de primer 20X (Tabela 1) para cada conjunto de primers (Tabela 2). Prepare a mistura de reação RT-LAMP (Tabela 3) à temperatura ambiente com volume de 10% de excesso para explicar a perda de tubulação.
NOTA: A mistura colorimétrica LAMP 2X master com uracil-DNA termolabile antártico-DNA glicosylase (UDG) impede a amplificação da contaminação do DNA de reações anteriores20,28. - Vórtice e gire a mistura. Dispense 20 μL da mistura em cada tubo de reação: amostra, pico, controle positivoe controle negativo. Incubar as reações nos tubos à temperatura ambiente por 10 minutos para permitir que o UDG aja sobre uma possível contaminação por atropelamento.
- Adicione 5 μL de RNA à reação amostral, 5 μL de RNA + 2,5 μL (450 cópias) de RNA SARS-CoV-2 sintético à reação cravada, 2,5 μL (450 cópias) de RNA SARS-CoV-2 sintético à reação de controle positivo, e 5 μL de H2O à reação de controle negativo. Misture bem, e gire as reações; descongelar todo o RNA no gelo.
- Enquanto o termociclador, ou o banho de água, é aquecido a 65 °C, permita que todas as reações permaneçam à temperatura ambiente. O UDG será inativado a >50 °C. Coloque as reações no termociclador (use tampa de calor), incubar a 65 °C por 40 min e deixe as reações atingirem a temperatura ambiente (~22 °C por 5 min) ou esfrie no gelo por 1 min. Analise os resultados utilizando o recurso colorimétrico (observação simples) ou executando os produtos em um gel de agarose.
NOTA: Embora o limite de detecção (LOD) seja de 10 cópias por reação, a frequência de detecção aumenta à medida que o número de cópia se aproxima de 500 cópias por reação(Figura 3A). Para observação colorimétrica, observe que um resultado negativo é indicado por rosa (pH = 8,8), enquanto um resultado positivo é indicado por amarelo (pH = 5) (Figura 3B). A opção colorimétrica evita a abertura dos tubos RT-LAMP após a amplificação, o que reduzirá o volume dos produtos RT-LAMP no ambiente de trabalho e a contaminação por transporte. Para eletroforese de gel, prepare um gel de 1,5% de agarose com mancha de gel de DNA 1X em 0,5% Tampão Tris/Borate/EDTA (TBE). Carregar 25 μL da reação + 5 μL de corante de carregamento 6X em cada poço. Execute o gel a 100 V por 60 min. Um marcador molecular não é necessário, pois amostras positivas mostram um padrão de escada(Figura 3C).
- Prepare uma solução de mix de primer 20X (Tabela 1) para cada conjunto de primers (Tabela 2). Prepare a mistura de reação RT-LAMP (Tabela 3) à temperatura ambiente com volume de 10% de excesso para explicar a perda de tubulação.
Resultados
Amostras coletadas por cientistas cidadãos para a detecção do SARS-CoV-2. Durante um período de 8 meses (meados de março até a terceira semana de novembro de 2020), 482 cidadãos foram aprovados para participar deste projeto, dos quais 350 (73%) solicitou um kit. Foram entregues 362 kits (ou seja, alguns participantes solicitaram vários kits) e 246 (70%) foram devolvidos (Figura 4A,B). Todas as 4.080 amostras contidas nesses kits foram processadas. Os locais de coleta foram distribuídos nos distritos da Costa Norte, Centro Norte, Central e Sul do município, bem como alguns nos distritos orientais (Figura 4A). Esses distritos têm a maior densidade populacional do Condado de San Diego e os casos mais documentados do COVID-19, conforme relatado pela Agência de Serviços de Saúde Humana de San Diego39.
Os cidadãos solicitaram a retirada da maioria dos kits amostrados (ou seja, taxa média de sucesso: 70,4%). Todos os dias, foram solicitados 1-16 kits, e 0-14 kits foram devolvidos ao laboratório (Figura 4B). Um levantamento dos cientistas cidadãos mostrou que a coleta de um kit completo (16 amostras) teve 1-3 h distribuídos ao longo de uma média de 8 dias (Figura 4A e Tabela 4). A grande maioria dos kits foram completos (91,1%), o que significa que continham um cotonete dentro de todos os 16 tubos de amostra, e os dados amostrais correspondentes foram enviados para o LIMS (Figura 4A e Tabela 4).
Detecção de SARS-CoV-2 usando extração de clorofórmio GITC e amplificação isotérmica mediada por loop (RT-LAMP). Para o ensaio colorimétrico RT-LAMP, dois conjuntos de primers11,20,28 foram usados para atingir os genes nucleocapsídeos(N2) e o envelope(E1) genes (Tabela 2). As sequências reconhecidas por esses primers estão na mesma região que os primers e sondas aprovados pelo CDC40 e pela União Europeia (UE)41 para diagnóstico humano do COVID-19 pela RT-qPCR. Esses resultados corroboram o que Zhang et al.28 descreveram, no qual adicionar cloridrato de guanidina de 60 mM à reação aumenta o LOD quando executado em multiplex. O LOD em uma frequência de 100% foi de 500 cópias por reação de 25 μL (Figura 3A, B). Na RT-LAMP colorimétrica, amostras positivas mudaram de cor de rosa para amarelo devido a uma mudança de pH de ~8 para 5,5 (Figura 3B). Quando a reação ficou laranja em números de cópia baixa, as amostras foram executadas em um gel de 1,5% para confirmar que eram positivas e resultaram em um padrão semelhante a uma escada (Figura 3C). O RT-LAMP foi usado para detectar SARS-CoV-2 em amostras de RNA agrupadas.
Para controlar falsos negativos devido aos inibidores de reação, cada amostra foi testada em uma reação adicional cravada com 500 cópias de SARS-CoV-2 sintéticos. Amostras agrupadas positivas foram desreplicadas isolando o RNA de cada amostra individual na piscina e executadas em uma reação RT-LAMP para determinar a identidade da amostra positiva. Os resultados de detecção foram então enviados para o LIMS, onde o ID único da amostra foi emparelhado com as informações em data, hora, coordenadas gps, local e imagem da amostra.
Métodos RT-PCR em tempo real e tradicionais: inibição por contaminantes amostrais. Para selecionar o melhor método adequado para o pipeline de detecção proposto, outros métodos de amplificação de RNA foram testados com amostras ambientais coletadas por uma coorte piloto de cientistas cidadãos. Exemplos dos resultados de cada um desses métodos são apresentados na Figura 5 para retratar sua sensibilidade aos inibidores ambientais e ruído de sinal de fundo em baixas concentrações de número de cópia viral.
Seis formulações de RT-qPCR (Tabela de Materiais) aprovadas pelo CDC e pela OMS foram testadas para detecção do vírus em amostras ambientais. Os protocolos foram seguidos de acordo com as instruções do fabricante, bem como as diretrizes do CDC para a detecção do SARS-CoV-2 em ambientes clínicos40. Reações contendo diferentes concentrações de controles RNA SARS-CoV-2 sintéticos foram cravadas em amostras de superfície limpa após o isolamento bruto do RNA. Todas as misturas mestras foram sensíveis aos inibidores nas concentrações de LOD do controle positivo (Figura 5A).
Para contornar os inibidores dessas tecnologias em tempo real, foi testado um sistema RT-PCR tradicional. Um sistema RT-PCR de uma etapa (Tabela de Materiais) foi usado para amplificar o gene nucleocapsídeo usando os conjuntos de primer N1, N2, e o gene envelope usando o conjunto de primer E1 aprovado pelo CDC (EUA) e o ECDC (UE), respectivamente (Tabela 2). Os protocolos foram seguidos de acordo com as instruções do fabricante, bem como as diretrizes do CDC para a detecção do SARS-CoV-2 em ambientes clínicos40. Os conjuntos de primer N1 e N2 projetados pelo CDC produzem um produto de ~70 bp; no entanto, os positivos de números de cópia baixa não foram distinguidos do ruído de fundo de amplificação do controle negativo (Figura 5B),que introduziu falsos positivos nos resultados. O produto dos primers E1 tinha um sinal fraco no número de cópia baixa (Figura 5B),introduzindo falsos negativos nos resultados. Além disso, o método RT-PCR testado ainda era sensível aos inibidores presentes nas amostras ambientais (dados não apresentados).
Outros métodos foram desenvolvidos para detectar quantidades muito pequenas de sequência de alvos. Um desses métodos é a Amplificação do Círculo De Rolamento (RCA), onde o reconhecimento da sequência de destino, RNA ou DNA, por uma sonda linear específica, uma ligase circulariza o modelo. Usando primers projetados para hibridizar com a sonda, uma polimerase de DNA com atividade de deslocamento de fios amplifica a sonda em uma reação isoteérmica42. É a sonda que identificou o alvo, que é amplificado, e não a sequência de destino, o que torna este método altamente sensível43. Wang et al.44 publicaram um protocolo RCA para a detecção direta do RNA SARS-CoV-1. O método foi modificado para o uso de primers específicos para SARS-CoV-2. Infelizmente, no controle não-modelo (NTC), a sonda circulariza e produz produto na ausência de modelo RNA, mesmo quando se usa uma grande variedade de ligases, incluindo um ligase sensível ao SNP. Na ausência de ligase, o NTC não mostrou amplificação da sonda linearizada (Figura 5C).

Figura 1: Plataforma de amostragem baseada na Web com interface de dados de coleta de amostras para dispositivos móveis. (A) Um site, contendo um plugin multilíngue, foi criado para mediar a interação entre o laboratório e os cientistas cidadãos. A plataforma foi utilizada para solicitação de entrega/coleta de kits de amostra e envio de dados de amostra. A plataforma continha protocolos detalhados de amostragem gráfica/espanhola e audiovisual. (B) Visualização de dispositivo móvel usado para carregar dados amostrais: data, hora, coordenadas GPS, descrição do local da amostra e uma imagem do local de coleta. Abreviaturas: SARS-CoV-2 = síndrome respiratória aguda grave coronavírus 2; GPS = Sistema de Posicionamento Global. Clique aqui para ver uma versão maior desta figura.

Figura 2: Kit de coleta de amostras. Cientistas cidadãos receberam um refrigerador contendo duas bolsas de gelo, uma ficha de dados de segurança para informar os voluntários sobre os perigos de manusear a solução GITC, um protocolo detalhado de amostragem e uso de máscaras, uma máscara KN95, um saco de lixo, uma garrafa de spray com desinfetante para as mãos, uma garrafa de spray com 0,5% SDS, 16 pares de luvas, um saco pequeno com 16 palitos de dente e 16 cotonetes de poliéster, 16 tubos de microcentrifuuge pré-rotulados contendo 200 μL de solução GITC, uma caixa contendo os tubos de amostragem, e um saco usado como recipiente secundário para a caixa de tubo em caso de derramamento. Abreviaturas: GITC = tiocianato de guanidinium; SDS = sulfato de dodecyl de sódio. Clique aqui para ver uma versão maior desta figura.
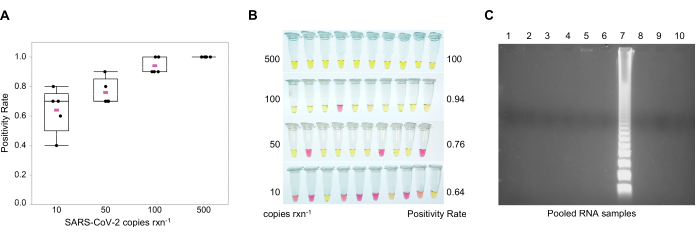
Figura 3: Ensaio de amplificação isotérmica mediada por loop de transcrição reversa multiplexada (RT-LAMP). Reações multiplexadas usando primers para genes nucleocapsídeos SARS-CoV-2(N2) e envelope(E1)para detectar apenas 10 cópias do vírus na reação. O RNA sintético SARS-CoV-2 foi utilizado como controle positivo. (A) Frequência de detecção em MULTIPLEX colorimétrico RT-LAMP de SARS-CoV-2 em diferentes números de cópia de genoma por reação. Valor médio de cinco réplicas em rosa. (B) Limite de detecção (LOD) de SARS-CoV-2 em RT-LAMP colorimétrico multiplex; amarelo = positivo (pH ~5); rosa = negativo (pH ~8). (C) Padrão de escada de reações positivas SARS-CoV-2 RT-LAMP em eletroforese de gel de 1,5% agarose. Abreviaturas: SARS-CoV-2 = síndrome respiratória aguda grave coronavírus 2; rxn = reação. Clique aqui para ver uma versão maior desta figura.
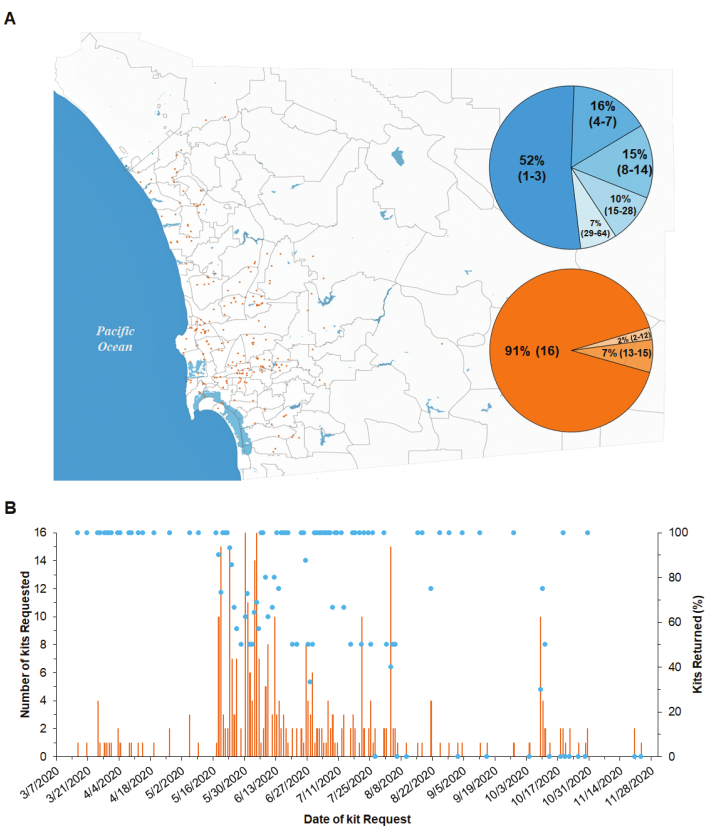
Figura 4: Localização de kits de amostragem de cientistas cidadãos no condado de San Diego e taxa de sucesso de kits solicitados. (A) Os pontos laranja representam a localização de 1 kit de amostragem, que contém 16 amostras. O gráfico de tortas azuis mostra a porcentagem de kits que levaram vários dias desde quando foram entregues aos cientistas cidadãos até quando foram devolvidos ao laboratório. Número de dias entre parênteses. O gráfico da torta de laranja mostra a porcentagem de kits com número diferente de amostras completas de um total de 16 amostras. Número de amostras completas contendo um cotonete dentro do tubo de amostra e os dados amostrais correspondentes enviados ao LIMS entre parênteses. (B) Percentual de kits que foram devolvidos ao laboratório (pontos), e número total de kits solicitados (barras), em relação à data em que o kit foi solicitado. Clique aqui para ver uma versão maior desta figura.
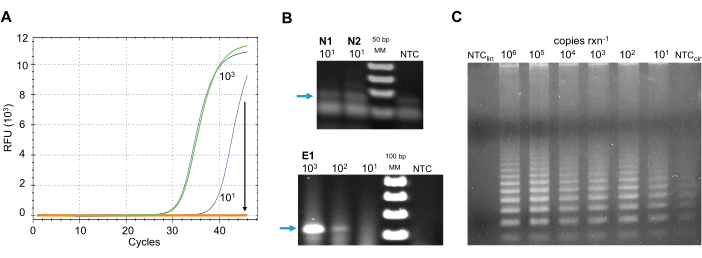
Figura 5: Métodos alternativos SARS-CoV-2 RNAdetection. (A) Detecção RT-qPCR do gene nucleocapsídeo SARS-CoV-2(N)utilizando o conjunto de primer N2. Amostras ambientais agrupadas cravaram com 900 cópias (verde), ou 9 (laranja) de SARS-CoV-2. Controles positivos dos mesmos números de cópia em azul. A seta indica a diminuição na detecção de fluorescência do controle positivo do número de cópia baixa quando a amostra ambiental está presente. (B) Detecção tradicional de RT-PCR de SARS-CoV-2. Topo: Produtos RT-PCR do gene nucleocapsídeo usando conjuntos de primer N1 e N2. Um sinal de fundo fraco é observado no controle sem modelo. Inferior: Produtos RT-PCR do gene envelope usando o conjunto de primer E1. Sinal muito baixo é observado na concentração de LOD. As setas azuis mostram o produto positivo esperado: (topo) ~70 bp e (inferior) 113 bp em 2% de eletroforese de gel agarose. (C) RCA de SARS-CoV-2 RNA. A sonda circular amplifica na presença de ligase e ausência de modelo RNA (NTCcir); na ausência de sonda linear de modelo ligase e RNA não amplifica (NTClin). Abreviaturas: SARS-CoV-2 = síndrome respiratória aguda grave coronavírus 2; RFU = unidades de fluorescência relativa; bp = pares de base; rxn = reação; MM = marcador molecular; RT-PCR = reação em cadeia de polimerase de transcrição reversa; RT-qPCR = RT-PCR quantitativo em tempo real; RCA = amplificação do círculo de rolamento; NTC= controle sem modelo; LOD = limite de detecção; rxn = reação. Clique aqui para ver uma versão maior desta figura.
| cartilha | Concentração de 20X (μM) | Concentração 1X (μM) |
| Fip | 32 | 1.6 |
| BIP | 32 | 1.6 |
| F3 | 4 | 0.2 |
| B3 | 4 | 0.2 |
| LoopF | 8 | 0.4 |
| LoopB | 8 | 0.4 |
Tabela 1: Formulação para mistura de primer RT-LAMP 20X. Na reação RT-LAMP, 6 primers reconhecem 8 regiões do DNA alvo. Abreviaturas: amplificação isotéria mediada por loop de transcrição reversa; FIP = primer interno para a frente; BIP = primer interno para trás; F3 = primer de deslocamento para a frente; B3 = primer de deslocamento para trás; LoopF = primer de loop dianteiro; LoopB = primer de loop para trás.
| cartilha | seqüenciar | alvo | Tamanho do produto | |||
| RT-LAMP9,18,19 | ||||||
| E1-F3 | TGAGTACGAACTTATGTACTCAT | ecstasy | padrão escada-like | |||
| E1-B3 | TTCAGATTTTTAACACGAGAGT | |||||
| E1-FIP | ACCACGAAAGCAAGAAAAAGAAGTTCTTTCGGAAGAGACAG | |||||
| E1-BIP | TTGCTAGTTACACTAGCCATCCCCTTAGGTTTTACAACTCACGT | |||||
| E1-LoopB | GCGCTTCGATTGTGTGTGTGTGT | |||||
| E1-LoopF | CGCTATTAACTATTAACG | |||||
| N2-F3 | ACCAGGAACTAATCAGACAAGAG | n | ||||
| N2-B3 | GACTTGATCTTTGAAATTTGGATCT | |||||
| N2-FIP | TTCCGAAGAACGCTGAAGCGGAACTGATTACAAACATTGGCC | |||||
| N2-BIP | CGCATTGGCATGGAAGACAATTTGATGGCACCTGTGTA | |||||
| N2-LoopF | GGGGGCAAAATTGTGCAATTTG | |||||
| N2-LoopB | CTTCGGGAACGTGGTTGACC | |||||
| RT-qPCR38 | ||||||
| nCoV_N1-F 2019 | GACCCCAAAAATCAGCGAAAT | n | 72 bp | |||
| 2019-nCoV_N1-R | TCTGGTTACTGCCAGTTGAATCTG | |||||
| 2019-nCoV_N1-P | 5'-FAM-ACC CCG CAT TAC GTT TGG TGG ACC-BHQ1-3' | |||||
| nCoV_N2-F 2019 | TTACAAACATTGGCCGCAAAA | n | 67 bp | |||
| 2019-nCoV_N2-R | GCGCGACATTCCGAAGAA | |||||
| 2019-nCoV_N2-P | 5'-FAM-ACA ATT TGC CCC CCC CGC TTC AG-BHQ1-3' | |||||
| RT-PCR39 | ||||||
| E1_Sarbeco_F | ACAGGTACGTTAATAGTTAATAGCGT | ecstasy | 113 bp | |||
| E1_Sarbeco_R | ATATTGCAGCAGTACACACACA | |||||
Tabela 2: Primers usados para RT-LAMP, RT-qPCR e RT-PCR. Sequências de primer, gene de destino, tamanho esperado do produto e referência correspondente estão listadas. Abreviaturas: RT-LAMP = amplificação isotemal mediada por loop de transcrição reversa; RT-PCR = reação em cadeia de polimerase de transcrição reversa; bp = pares de base; RT-qPCR = RT-PCR quantitativo em tempo real; E1 = gene envelope; N2 = gene nucleocapsídeo; F = primer para a frente; R = primer reverso; P = Sonda ; FIP = primer interno para a frente; BIP = primer interno para trás; F3 = primer de deslocamento para a frente; B3 = primer de deslocamento para trás; LoopF = primer de loop dianteiro; LoopB = primer de loop para trás.
| Reagente | Volume (μL) |
| WarmStart Colorimetric LAMP 2X Master Mix com UDG | 12.5 |
| N2 Primer Mix (20x) | 1.25 |
| E1 Primer Mix (20x) | 1.25 |
| Cloridrato de Guanidine (600 mM)* | 2.5 |
| RNA alvo | 5 |
| H2O sem nuclease | 2.5 |
| Total Volume | 25 |
Tabela 3: Mistura mestre de reação para multiplex colorimétrico RT-LAMP. (*) O cloridrato de Guanidina tem sido mostrado para aumentar a sensibilidade e a velocidade da reação por um mecanismo não característico28. Abreviaturas: LAMP = amplificação isotemal mediada por loop; UDG = uracil-DNA glicosylase; N2 = gene nucleocapsídeo; E1 = gene envelope; DEPC = ditilpirocarbonato.
| Cidadãos aprovados | Cidadãos que solicitaram um kit | Kits entregues | Kits amostrados devolvidos | Dias dedicados à amostragem | % Kits completos | % Kits incompletos | Amostras processadas | |
| 482 | 72.6% (350/482) | 362 | 70.4% (255/362) | significar | 8 | 91.1 (224/246) | 8.9 (22/246) | 4,080 |
| mediana | 3 | |||||||
Tabela 4:Swabbing para SARS-CoV-2 pelos números. Taxas de sucesso de divulgação e amostragem. Abreviação: SARS-CoV-2 = síndrome respiratória aguda grave coronavírus 2.
Discussão
Engajamento de cientistas cidadãos. Cientistas cidadãos foram recrutados para limpar superfícies em todo o condado de San Diego para amostrar e detectar a presença de SARS-CoV-2 no ambiente urbano. A maioria dos kits de amostragem entregues (70%) foram devolvidas ao laboratório, e dessas, quase todas as amostras foram completas (91%) (Figura 3A, B e Tabela 4). Os voluntários poderiam facilmente solicitar a entrega/coleta de kits através da plataforma baseada na Web, e o software de planejamento de rotas de entrega notificou os cientistas cidadãos dos tempos estimados de chegada, ambos provavelmente fatores significativos para o sucesso observado. O tempo médio de quando o kit foi entregue ao cientista cidadão para quando foi devolvido ao laboratório foi de 8 dias, com mediana de 3 dias e intervalo de 1 a 64 dias (Figura 3A e Tabela 4). Lembretes mais frequentes para os voluntários provavelmente reduziriam esse tempo de atraso.
A plataforma de coleta de dados foi usada com sucesso pela grande maioria dos usuários (73%) (Tabela 4). Embora os esforços dos cientistas cidadãos não tenham sido medidos, testes de campo mostraram que a plataforma de coleta de dados reduziu significativamente o esforço e o tempo necessários para completar adequadamente a coleta de amostras. Assim, reduzir a quantidade de escrituração incentivou o engajamento dos cientistas cidadãos. A plataforma baseada na Web pretendia superar as limitações demográficas, fornecendo um serviço multilíngue de tradução de máquinas neurais e fornecendo protocolos gráficos e audiovisuais em inglês e espanhol. Isso só foi parcialmente bem sucedido, pois menos amostras foram coletadas tanto da Baía do Sul quanto do Condado do Norte, onde a maioria da população hispânica/latina do condado reside45. Essas áreas também abrigavam 63% (1.700 casos por 100.000) do total de casos de COVID-19 no condado de San Diego com a maior prevalência da doença46 e taxa de internações (62%)47,48. Embora a maioria das amostras tenha vindo do Condado Central, um número representativo foi coletado dos distritos mais impactados pelo COVID-19 e apenas uma pequena fração das amostras foi positiva, o que sugere que os reservatórios superficiais de SARS-CoV-2 no ambiente urbano são relativamente raros.
Processamento de amostras. Amostras de amostras foram molhadas com SDS, que inativaram o vírus interrompendo seu envelope e estabilizando o RNA nu desdobrando RNases32. Convenientemente durante a coleta, o detergente no cotonete limpou a superfície amostrada. Amostras ambientais geralmente contêm quantidades muito pequenas de RNA. Para maximizar a recuperação, o isolamento do RNA foi realizado usando um método de extração bruta baseado em COLUNAs, sem colunas. O GITC, um agente chaotrópico forte, interrompe as ligações de hidrogênio que mantêm a dobra de proteínas (ou seja, efeito hidrofóbico). Esta ação resulta na inativação de partículas virais, e o RNA permanece estável devido à inibição de RNAses34,35,36. A solução GITC manteve a estabilidade das amostras de RNA sem considerações rigorosas da cadeia fria, o que permitiu aos cidadãos manter as amostras à temperatura ambiente se um freezer para as bolsas de gelo fornecidas não estivesse disponível. Para reduzir o risco potencial que este reagente representa quando ocorre contato direto da pele ou mucosa, os cidadãos foram informados desses riscos pela inclusão de uma folha de dados de segurança material fornecida no kit e um selo de advertência foi colocado na caixa contendo os tubos.
O método bruto de extração gitc-clorofórmio auxiliou na recuperação de traços de RNA dos cotonetes, e como mostrado pela amplificação de amostras cravadas, os inibidores raramente persistiam nas amostras após a extração. As amostras, que foram negativas para SARS-CoV-2 e não mostraram inibição de LÂMPADA-RT, representaram verdadeiros negativos ou apresentaram um número de cópia menor do que o LOD em 100% de frequência. Por outro lado, a detecção de RNA viral em uma superfície não implica diretamente o risco de transmissão através do contato, pois a infectividade do vírus a partir de amostras positivas precisa ser testada. A triagem rápida do ambiente, não limitada pela disponibilidade de suprimentos sofisticados ou pessoal altamente qualificado, é crucial para avaliar se as superfícies constituem um reservatório viral e para melhor direcionar os esforços de prevenção e contenção.
O RT-LAMP foi selecionado para ser o melhor método adequado para o pipeline de detecção proposto. Provou ser um método rápido e barato que era altamente resistente à maioria dos inibidores restantes e tão sensível e específico quanto outros métodos RT-qPCR. Devido ao seu uso em ambientes clínicos durante a pandemia SARS-CoV-2, a disponibilidade de kits RT-qPCR foi impactada pela demanda global. Além disso, as técnicas de RT-qPCR — mesmo aquelas formuladas para resistir aos inibidores — eram sensíveis às substâncias contidas nas amostras ambientais coletadas por uma coorte piloto de cientistas cidadãos, mesmo após o uso de outras estratégias comuns para reduzir a concorrência inibidora para a ligação enzimático49. Esses achados são corroborados por um estudo recente que comparou ambos os métodos para detectar SARS-CoV-2 em amostras de swab de doces manuseadas por pacientes COVID-19 e encontraram mais de 83% de concordância de resultados, com inibição 25% menor em amostras analisadas pela RT-LAMP15. Além disso, a extração de petróleo bruto GITC-clorofórmio, juntamente com a RT-LAMP, reduziu o custo dos reagentes e suprimentos em 42% em comparação com a extração do kit RNA e RT-qPCR (Tabela de Materiais).
Este método permitiu a análise de alto rendimento de milhares de amostras de swab de superfície. Até 80 piscinas, representando 640 amostras, foram processadas em 2 dias desde a extração de RNA até a detecção de SARS-CoV-2 pela RT-LAMP. O protocolo proposto é semiquantitativo, limitado à detecção de RNA viral, e não indica a presença de partículas virais infecciosas. É necessária uma análise mais aprofundada para avaliar o risco de transmissão de SARS-CoV-2 de fomitas infectadas presentes nas superfícies limpas.
Este estudo apresenta um protocolo para configurar rapidamente uma estratégia de teste que inclua um fluxo de trabalho eficaz ao enfrentar uma emergência de saúde com uma doença transmissível. O protocolo de amostragem proposto é simples e utiliza suprimentos comumente encontrados em domicílios, e o método de detecção de viral é realizado em equipamentos disponíveis em ambientes básicos de laboratório, como um banho de água em vez de um termciclista. Os custos dos reagentes RT-LAMP são significativamente menores do que os necessários para o RT-qPCR e menos suscetíveis a cenários de alta demanda global. Este estudo serve como um marco para a avaliação de reservatórios virais ambientais em futuros surtos epidêmicos e pandemias globais.
Divulgações
Todos os autores declaram que não existem interesses concorrentes.
Agradecimentos
Agradecemos aos investigadores do Instituto de Informações Virais (VII), Dr. Anca M. Segall, Willow Segall, Patricia L. Rohwer, Gary Rohwer, Cary L. Rohwer, Magda Silvia Pinetta, Elizabeth Cruz Cano, Dr. Gregory Peters, Dr. Stuart A. Sandin, e Dr. Jennifer Smith por tirarem o tempo para coletar inúmeras amostras. Agradecemos também ao Dr. Rob Knight, Dr. Jack Gilbert, Dr. Pedro Balda-Ferre e Dr. Sarah Allard do departamento de Pediatria da Universidade de Medicina de San Diego Califórnia (UCSD) por facilitar controles positivos e feedback útil. Agradecemos a Stacey Carota (Faculdade de Ciências da SDSU) e Gina Spidel (SDSU) pelo apoio logístico e a Juan Rodríguez pelo design artístico e gráfico do protocolo amostral. Agradecemos a todos os participantes pelo empenho e dedicação a este projeto em tempos muito difíceis. Este trabalho foi apoiado por uma generosa doação do Dr. Jo Ann Lane (SDSU College of Sciences) e da National Science Foundation RAPID: Environmental Reservoirs of SARS-CoV-2 grant (Número de Prêmio: 2030479).
Materiais
| Name | Company | Catalog Number | Comments |
| SMP, LIMS, and community outreach: | |||
| Authentication Application Programming Interface | Google Sign-In | ||
| Commercial hosting platform | GoDaddy | ||
| Data Charting Application Programming Interface | Google Charts | ||
| Database software | MySQL | ||
| Delivery route planning software | Circuit | Circuit for Teams | |
| Free email service | Google Email | ||
| Geospatial Application Programming Interface | Google Maps API | ||
| Multilingual neural machine translation service | Google Translate | ||
| Online form | Google Form | ||
| Operating system | Linux | ||
| Web and database development | Big Rose Web Design | ||
| Web server software | Apache | ||
| Sampling kit: | |||
| Coolers | Coleman (Amazon) | B00363X3F2 | Cost (US$) per 100 rxns: 70 |
| Gallon Ziploc bags | Solimo (Amazon) | B07BJ495GL | Cost (US$) per 100 rxns: 18 |
| Glycerol (hand sanitizer) | FischerScientific | G33-4 | Cost (US$) per 100 rxns: 9 |
| Ice packs | Ice-Brix (Amazon) | B075GLD3X1 | Cost (US$) per 100 rxns: 110 |
| Isopropanol (hand sanitizer) | FischerScientific | AA36644K7 | Cost (US$) per 100 rxns: 43 |
| KN95 masks | Echo-Sigma | Echo-Sigma | Cost (US$) per 100 rxns: 400 |
| Paper for Protocols and Trizol Safety Sheet | Office Depot | 348037 | Cost (US$) per 100 rxns: 36 |
| 30 mL spray bottles (SDS and hand sanitizer) | Anyumocz (Amazon) | B07T64FHXR | Cost (US$) per 100 rxns: 80 |
| RNase, DNase, DNA & PCR inhibitors free Microcentrifuge tubes | Genesee Scientific | 22-281 | Cost (US$) per 100 rxns: 83 |
| Sample ID solvent resistant labels | LABTAG | XST-10C1-1WH | Cost (US$) per 100 rxns: 68 |
| Swiffer WetJet pads (swabs) | Swiffer (Amazon) | B001F0RBT2 | Cost (US$) per 100 rxns: 8 |
| Toothpicks | Kitchen Essential (Amazon) | B00PBK4NG6 | Cost (US$) per 100 rxns: 8 |
| Trizol Reagent (guanidinium isothiocyanate solution - GITC), not LS | Invitrogen | 15596018 | Cost (US$) per 100 rxns: 40 |
| Tube boxes | Genesee Scientific | 21-119 | Cost (US$) per 100 rxns: 180 |
| Small Ziploc bags | Ziploc (Amazon) | B01LRKEI9K | Cost (US$) per 100 rxns: 8 |
| Zebra Thermal Transfer Desktop Printer | Zebra | GK420t | |
| Total Sampling kit Cost (US$) per 100 rxns: 1,160 | |||
| Trizol RNA extraction: | |||
| Ammonium Acetate RNase-free | Invitrogen | AM9070G | Cost (US$) per 100 rxns: 2 |
| Chloroform | FisherScientific | C298-500 | Cost (US$) per 100 rxns: 2 |
| GlycoBlue (glycogen 15 mg/mL) | Invitrogen | AM9515 | Cost (US$) per 100 rxns: 80 |
| Molecular-grade absolute (200 proof) Ethanol | FisherScientific | BP2818500 | Cost (US$) per 100 rxns: 30 |
| Molecular-grade Isopropanol | FisherScientific | BP2618500 | Cost (US$) per 100 rxns: 3 |
| TURBO DNA-free Kit | Invitrogen | AM1907 | Cost (US$) per 100 rxns: 110 |
| Multiplexed colorimetric RT-LAMP: | |||
| Guanidine Hydrochloride | Alfa Aesar | AAJ6548522 | Cost (US$) per 100 rxns: 1 |
| RT-LAMP E1-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| RT-LAMP N2-Primers | IDT | n/a | Cost (US$) per 100 rxns: 7 |
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| WarmStart Colorimetric LAMP 2X Master Mix with UDG | NEB | M1800S | Cost (US$) per 100 rxns: 210 |
| Eppendorf Mastercycler Pro Thermal Cycler | Eppendorf | 950030010 | |
| Total Trizol RNA extraction + LAMP Cost (US$) per 100 rxns: 470 | |||
| Kit for RNA extraction: | |||
| QIAamp DSP Viral RNA Mini Kit | Qiagen | 61904 | Cost (US$) per 100 rxns: 570 |
| RT-qPCR: | |||
| Synthetic SARS-CoV-2 RNA | ATCC | VR-3276SD | Cost (US$) per 100 rxns: 14 |
| TaqMan Fast Virus 1-Step Master Mix | Applied Biosystems | 4444432 | Cost (US$) per 100 rxns: 180 |
| SARS-CoV-2 (2019-nCoV) N1,N2 Primers and Probes | IDT | 10006713 | Cost (US$) per 100 rxns: 20 |
| qScript XLT 1-Step RT-qPCR ToughMix | Quantabio | 95132-100 | |
| QuantiNova Pathogen | Qiagen | 208652 | |
| QuantiNova Probe | Qiagen | 208352 | |
| UltraPlex 1-Step ToughMix | Quantabio | 95166-100 | |
| CFX96 Touch Real-Time PCR Detection System | BioRad | 1855196 | |
| Kit for RNA extraction + RT-qPCR Cost (US$) per 100 rxns: 790 | |||
| RT-PCR: | |||
| SuperScript IV One-Step RT-PCR | Invitrogen | 12594025 | |
| Lab cleanup: | |||
| DNAZap | Invitrogen | AM9890 | |
| RNAZap | Invitrogen | AM9780 | |
Referências
- Alsved, M., et al. Exhaled respiratory particles during singing and talking. Aerosol Science and Technology. 54 (11), 1245-1248 (2020).
- Morawska, L., Cao, J. Airborne transmission of SARS-CoV-2: The world should face the reality. Environment International. 139, 105730(2020).
- Stadnytskyi, V., Bax, C. E., Bax, A., Anfinrud, P. The airborne lifetime of small speech droplets and their potential importance in SARS-CoV-2 transmission. Proceedings of the National Academy of Sciences of the United States of America. 117 (22), 11875-11877 (2020).
- Yu, I. T. S., et al. Evidence of Airborne Transmission of the Severe Acute Respiratory Syndrome Virus. New England Journal of Medicine. 350 (17), 1731-1739 (2004).
- Coronavirus disease (COVID-19): How is it transmitted. World Health Organization. , Available from: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/question-and-answers-hub/q-a-detail/coronavirus-disease-covid-19-how-is-it-transmitted (2020).
- How COVID-19 Spreads. Centers for Disease Control and Prevention. , Available from: https://www.cdc.gov/coronavirus/2019-ncov/prevent-getting-sick/how-covid-spreads.html (2020).
- Moriarty, L. F., et al. Public Health Responses to COVID-19 Outbreaks on Cruise Ships - Worldwide, February-March 2020. MMWR. Morbidity and Mortality Weekly Report. 69 (12), 347-352 (2020).
- Cheng, V. C. -C., et al. Air and environmental sampling for SARS-CoV-2 around hospitalized patients with coronavirus disease 2019 (COVID-19). Infection Control and Hospital Epidemiology. , 1-8 (2020).
- Van Doremalen, N., et al. Aerosol and surface stability of SARS-CoV-2 as compared with SARS-CoV-1. New England Journal of Medicine. 382 (16), 1564-1567 (2020).
- Liu, Y., et al. Aerodynamic analysis of SARS-CoV-2 in two Wuhan hospitals. Nature. 582, 557-560 (2020).
- Butler, D. J., et al. Shotgun transcriptome and isothermal profiling of SARS-CoV-2 infection reveals unique host responses, viral diversification, and drug interactions. bioRxiv. , (2020).
- Döhla, M., et al. SARS-CoV-2 in environmental samples of quarantined households. medRxiv. , (2020).
- Ikonen, N., et al. Deposition of respiratory virus pathogens on frequently touched surfaces at airports. BMC Infectious Diseases. 18, 437(2018).
- Chia, P. Y., et al. Detection of air and surface contamination by SARS-CoV-2 in hospital rooms of infected patients. Nature Communications. 11 (1), 2800(2020).
- Salido, R. A., et al. Handwashing and detergent treatment greatly reduce SARS-CoV-2 viral load on Halloween candy handled by COVID-19 patients. mSystems. 5, 01074(2020).
- Chan, J. F. W., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. Journal of Clinical Microbiology. 58 (5), 00310-00320 (2020).
- Dao Thi, V. L., et al. A colorimetric RT-LAMP assay and LAMP-sequencing for detecting SARS-CoV-2 RNA in clinical samples. Science Translational Medicine. 12 (556), (2020).
- Rauch, J., et al. A scalable, easy-to-deploy, protocol for Cas13-based detection of SARS-CoV-2 genetic material. bioRxiv. , (2020).
- Zhang, F., Abudayyeh, O. O., Gootenberg, J. S. A protocol for detection of COVID-19 using CRISPR diagnostics. , Available from: https://www.broadinstitute.org/files/publications/special/COVID-119%20detection%20(updated).pdf (2020).
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-185 (2020).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nature Biotechnology. 38, 870-874 (2020).
- Lucia, C., Federico, P. -B., Alejandra, G. C. An ultrasensitive, rapid, and portable coronavirus SARS-CoV-2 sequence detection method based on CRISPR-Cas12. bioRxiv. , (2020).
- Danko, D., et al. Global genetic cartography of urban metagenomes and anti-microbial resistance. bioRxiv. , (2020).
- Parida, M., et al. Rapid detection and differentiation of dengue virus serotypes by a real-time reverse transcription-loop-mediated isothermal amplification assay. Journal of Clinical Microbiology. 43 (6), 2895-2903 (2005).
- Notomi, T., et al. Loop-mediated isothermal amplification of DNA. Nucleic acids research. 28 (12), 63(2000).
- Nagamine, K., Hase, T., Notomi, T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16 (3), 223-229 (2002).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. BioTechniques. 69 (3), 179-186 (2020).
- Tanner, N. A., Zhang, Y., Evans, T. C. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58 (2), 59-68 (2015).
- Curtis, K. A., Rudolph, D. L., Owen, S. M. Rapid detection of HIV-1 by reverse-transcription, loop-mediated isothermal amplification (RT-LAMP). Journal of Virological Methods. 151 (2), 264-270 (2008).
- OAuth 2.0. Internet Engineering Task Force (IETF. , Available from: https://oauth.net/2/ (2012).
- Naidu, K. T., Prabhu, N. P. Protein-surfactant interaction: Sodium dodecyl sulfate-induced unfolding of ribonuclease A. Journal of Physical Chemistry B. 115 (49), 14760-14767 (2011).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: Twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Hummon, A. B., Lim, S. R., Difilippantonio, M. J., Ried, T. Isolation and solubilization of proteins after TRIzol® extraction of RNA and DNA from patient material following prolonged storage. Biotechniques. 42 (4), 467-472 (2007).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols. , (2010).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Ethanol precipitation of RNA and the use of carriers. Cold Spring Harbor Protocols. , (2010).
- Wallace, D. M. Precipitation of nucleic acids. Methods in Enzymology. 152, 41-48 (1987).
- COVID-19 Dashboard. County of San Diego Health and Human Services Agency. , Available from: https://www.arcgis.com/apps/opsdashboard/index.html#/96feda77f12f46638b984fcb1d17bd24 (2020).
- CDC 2019-novel Coronavirus (2019-nCoV) real-time RT-PCR diagnostic panel. Centers for Disease Control and Prevention. , Available from: https://www.fda.gov/media/134922/download (2020).
- Corman, V. M., et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 25 (3), 2000045(2020).
- Lizardi, P. M., et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification. Nature Genetics. 19, 225-232 (1998).
- Johne, R., Müller, H., Rector, A., van Ranst, M., Stevens, H. Rolling-circle amplification of viral DNA genomes using phi29 polymerase. Trends in Microbiology. 17 (5), 205-211 (2009).
- Wang, B., et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification. Journal of Clinical Microbiology. 43 (5), 2339-2344 (2005).
- Population of Mexican origin in San Diego County. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/CHS/ENGLISH VERSION_Mexican Origin.pdf (2020).
- COVID-19 city of residence MAP. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/Epidemiology/COVID-19 City of Residence_MAP.pdf (2020).
- COVID-19 hospitalizations summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/phs/ Epidemiology/COVID-19 Hospitalizations Summary_ALL.pdf (2020).
- COVID-19 race and ethnicity Summary. County of San Diego Health and Human Services Agency. , Available from: https://www.sandiegocounty.gov/content/dam/sdc/hhsa/programs/ phs/Epidemiology/COVID-19 Race and Ethnicity Summary.pdf (2020).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados