Method Article
コラーゲンベースの足場を用いた神経芽細胞腫の3次元 in vitro 生体模倣モデル
* これらの著者は同等に貢献しました
Erratum Notice
要約
この論文では、神経芽腫細胞株を以前に説明した3次元コラーゲンベースの足場に播種し、所定の時間枠で細胞増殖を維持し、さまざまな実験目的を満たすために適応可能な、いくつかの細胞増殖および細胞挙動分析およびダウンストリームアプリケーション用の足場を回収するために必要な手順をリストしています。
要約
神経芽腫は小児に最も多くみられる頭蓋外固形腫瘍であり、小児がんによる死亡全体の15%を占める。天然腫瘍組織は、細胞外マトリックス(ECM)に囲まれたがん細胞と非がん細胞の層を含む複雑な3次元(3D)微小環境です。ECMは、物理的および生物学的なサポートを提供し、疾患の進行、患者の予後、および治療反応に貢献します。
この論文では、神経芽腫細胞株とコラーゲンベースの足場を使用して、神経芽腫微小環境を模倣する3D足場ベースのシステムを組み立てるためのプロトコルについて説明します。足場には、神経芽腫の最も一般的な転移部位である骨および骨髄に高濃度で自然に見られるナノヒドロキシアパタイト(nHA)またはグリコサミノグリカン(GAG)のいずれかが添加されています。これらの足場の3D多孔質構造は、神経芽細胞腫細胞の接着、増殖および遊走、および細胞クラスターの形成を可能にする。この3Dマトリックスでは、治療薬に対する細胞応答は、 in vivo の状況をより反映しています。
足場ベースの培養システムは、従来の2次元(2D)細胞培養よりも高い細胞密度を維持できます。したがって、初期播種細胞数の最適化プロトコルは、目的の実験時間枠に依存します。このモデルは、DNA定量による細胞増殖、代謝アッセイによる細胞生存率、組織学的染色による足場内の細胞分布を評価することによってモニターされます。
このモデルのアプリケーションには、遺伝子およびタンパク質発現プロファイルの評価、および従来の薬剤およびmiRNAを使用した細胞毒性試験が含まれます。3D培養システムにより、細胞およびECMコンポーネントの正確な操作が可能になり、生理学的に天然の腫瘍組織に近い環境を作り出します。したがって、この3D in vitroモデルは、疾患の病因の理解を深め、in vitro 、 in vivo の動物モデル、およびヒト被験者で得られた結果との相関を改善します。
概要
神経芽細胞腫は、神経堤細胞の形質転換により、胚発生期または出生後早期に発生する交感神経系の小児がんである1。小児に最も多くみられる固形頭蓋外腫瘍であり、15歳未満の患者で診断される悪性腫瘍の8%を占め、小児がんによる死亡全体の15%の原因となっている。この疾患は、特定の染色体、遺伝的およびエピジェネティックな変化、および組織病理学的特徴により、非常に不均一な臨床行動を示します。
これらの変化は、神経芽腫の侵攻性および小児患者の転帰不良に寄与する。したがって、現在の治療法は、臨床的に侵攻性疾患の患者のほぼ80%に対して長期的には効果がないことが証明されており2、このグループの患者に対する治療が依然として困難であるという事実を浮き彫りにしています。これは、神経芽腫の不均一性と転移のメカニズムがまだ完全に理解されていないことが原因である可能性があります。しかし、腫瘍微小環境(TME)は現在、多くのがんの進行に関与していると広く信じられています。しかし、神経芽細胞腫では研究が不十分なままです3,4。
ネイティブTMEは、ECMに囲まれたがん細胞と非がん細胞を含む複雑な3D微小環境です。ECMとは、組織の無細胞成分を指し、細胞の常在員に構造的および生化学的サポートを提供し、疾患の進行、患者の予後、および治療反応に寄与します5。この疾患進行の促進は、細胞とECMとの間の「動的互恵性」または継続的な双方向通信によるものです6,7,8。がんが進行するにつれて、間質コラーゲンはしばしば間質とがんの界面に垂直な線状に再編成され、がん細胞はこれを転移への移動経路として利用します9,10,11。
この天然の機能的生物学的足場の主成分には、I型およびII型コラーゲンの繊維状ネットワーク、およびエラスチン、ラミニンなどの糖タンパク質、ならびにさまざまなプロテオグリカンおよびその他の可溶性成分が含まれます12,13。天然ECMのこれらのタンパク質は、現在、3D in vitroモデルを開発するための魅力的な天然生体分子となっています3。in vitro細胞培養用の3Dスキャフォールドの用途は、従来の2D単層培養と比較してTMEの生理学的表現が優れているため、人気が高まっています。製造された3Dスキャフォールドは、細胞の接着、増殖、遊走、代謝、およびin vivoの生物学的システムに見られる刺激への応答を支援します。
これらの3D足場の主成分はコラーゲンであり、組織修復、血管新生、組織形態形成、細胞接着、遊走など、多くの正常な生物学的プロセスにおいて重要な役割を担っています11。コラーゲンベースの3Dマトリックスは、ECMをモデル化する堅牢な機能を示しており、in vitroの生体模倣微小環境として機能し、細胞とECMの相互作用、および細胞の遊走と浸潤を可能にします。また、これらの3Dマトリックスは、神経芽細胞腫17,18を含む多くのがんモデルにおいて、従来の2Dまたは「フラット」培養よりも、化学療法薬に対する細胞応答のより正確な分析を提供します14,15,16。3D細胞培養の遺伝子解析では、動物モデルと比較しても、ヒト組織プロファイルとの相関が高いことが報告されています19。全体として、これらの3Dスキャフォールドの基礎となるのは、細胞に適切なin vitro環境を提供することであり、これは天然の組織構造を再現し、双方向の分子クロストークを促進します8。
コラーゲンベースのモデルの複雑さを増すために、他の一般的なECMコンポーネントが組織工学プロセスに組み込まれているため、さまざまな組織のニッチなTMEを反映する、より生理学的に関連性のあるモデルが作成されます。例えば、すべての哺乳類組織に存在する負に帯電した多糖類であるGAGは20、細胞の接着、遊走、増殖、および分化を促進する。コンドロイチン硫酸は、骨や軟骨に見られる特定のタイプのGAGであり、以前は骨修復のための組織工学用途で使用されてきました21,22,23,24,25。ナノハイドロキシアパタイト(nHA)は、人間の骨組織のミネラル組成の主要な無機成分であり、骨の重量で最大65%を構成する26ため、骨の置換と再生に広く使用されています27。したがって、GAGとnHAは、原発性神経芽腫ECMを再建し、神経芽腫、骨髄(70.5%)、および骨(55.7%)の最も一般的な転移部位をモデル化するための魅力的な複合材料です28。
これらのECMコンポーネントを組み込んだ足場は、もともと骨組織工学アプリケーション向けに開発され、それらの生体適合性、毒性、および骨伝導性および骨誘導性の特徴を広範囲に分析しました29,30。これらは、凍結乾燥技術を使用して製造された多孔質のコラーゲンベースのマトリックスであり、物理的および生物学的特性を制御します。nHA(Coll-I-nHA)またはコンドロイチン-6-硫酸(Coll-I-GAG)のいずれかを添加したコラーゲン足場は、乳がん31の原発性TMEを模倣することに成功し、前立腺がん15および神経芽細胞腫17の骨への転移を模倣することに成功しました。これらの複合足場を製造するために使用される凍結乾燥技術は、足場内の細孔サイズと気孔率に再現可能な均質性をもたらします22,23,24。簡単に説明すると、線維性コラーゲンと0.05M酢酸をブレンドしてコラーゲンスラリー(0.5重量%)を作製します。Coll-I-GAGでは、サメの軟骨から単離された0.05wt%のクロノイチン-6-硫酸塩をコラーゲンスラリーに添加しながらブレンドします。複合Coll-I-nHAスキャフォールドでは、ナノサイズのハイドロキシアパタイト粒子が前述のように合成され27、ブレンドプロセス中にコラーゲンの重量に対して2:1の比率でコラーゲンスラリーに添加されます。すべての足場は物理的に架橋され、105°Cで24時間25分の脱水熱処理を使用して滅菌されます。円筒形の足場(直径6mm、高さ4mm)は、生検パンチを用いて得られ、3mMのN-(3-ジメチルアミノプロピル)-N'-エチルカルボジイミド塩酸塩および5.5mMのN-ヒドロキシコハク酸イミド(EDAC/NHS)で化学的に架橋して、構築物の機械的特性を改善することができる30。2つのコラーゲン足場のこの最適化された製造プロセスにより、細孔径、気孔率、剛性(kPa)などの再現性のある機械的特性を備えた足場が作成されます。Coll-I-GAGとColl-I-nHAの足場はどちらも物理的特性が異なるため、環境条件も異なります。各スキャフォールドの特性を表1に示す。
| Coll-I-GAGの | Coll-I-nHA(コリ・イ・インハ) | |
| 足場サイズ (直径[mm]×高さ[mm]) | 6×4 17 | 6×4 17 |
| コラーゲン濃度(wt. %) | 0.5 17 | 0.5 17 |
| 基質濃度(wt. %) [コラーゲンの重量に基づく] | 0.05 15.17 | 200 17 |
| 平均細孔径(mm) | 96歳22歳 | 96 - 120 29 |
| 気孔率(%) | 99.5 23 | 98.9 - 99.4 27 |
| 剛性(kPa) | 1.5°C 27°C | 5.5〜8.63 29 |
表1:神経芽腫生物学の研究に採用された2つの足場の機械的特性の概要。
この論文では、神経芽細胞腫細胞株と、nHA(Coll-I-nHA)またはコンドロイチン-6-硫酸塩(Coll-I-GAG)のいずれかを添加した以前に説明したコラーゲンベースの足場を使用して、神経芽細胞腫の微小環境をよりよく模倣するために3D足場ベースのシステムを組み立てるプロトコルについて説明します。プロトコルには、2D単層培養 図1から適応された以前に最適化された安価な方法を使用して、より生理学的に関連する環境で神経芽腫細胞の成長メカニズムを分析する下流の方法が含まれています。
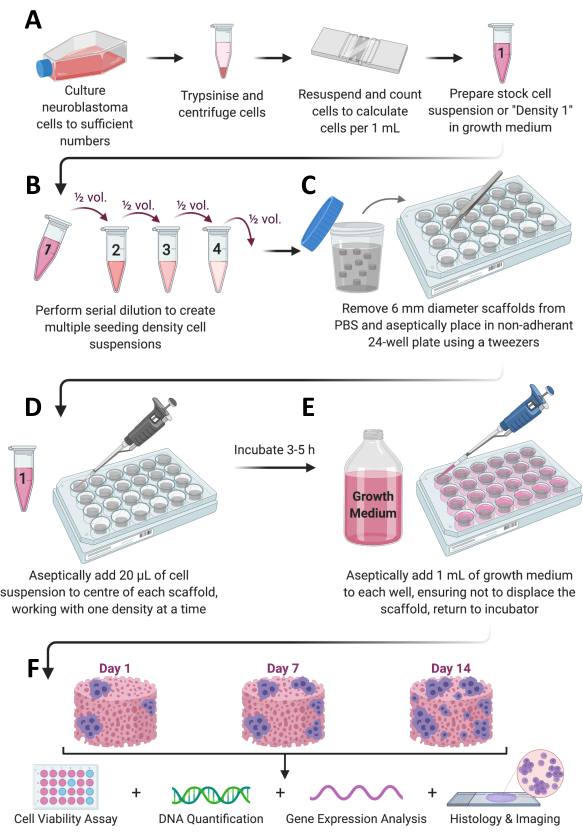
図1:プロトコルの全体的なワークフロー (A)細胞を十分な数まで増殖させ、分割し、計数し、適切な量の培地に再懸濁します。(B)次に、この細胞ストックを段階希釈して、密度の異なる合計4つの細胞懸濁液を調製します。(C)コラーゲンベースの足場を非接着性の24ウェルプレートに無菌播種し、(D)20μLの細胞懸濁液を各足場の中央に添加し、37°C、5%CO2、湿度95%で3〜5時間インキュベートします。 (E)次に、完全な増殖培地(1 mL)を各足場にゆっくりと添加し、プレートをインキュベーターに戻して、目的の時間枠で細胞増殖できるようにします。(f)所定の各時点で、細胞生存率および増殖評価、遺伝子発現解析、および組織学的染色のために、いくつかの足場を回収する。 この図の拡大版をご覧になるには、ここをクリックしてください。
プロトコル
1. 実験計画
注:各実験に必要な足場とセルの数は、実験の規模に依存し、実験計画に関するこのセクションのツールを使用して計算できます。
- 必要な足場の数
- 細胞生物学の変化(細胞増殖や代謝など)の全体的な実験タイムラインと評価間隔を決定します。
注:たとえば、実験期間は14日間で、細胞増殖の評価は7日ごとに行われます。したがって、実験時点は 1 日目、7 日目、14 日目であり、合計 3 つの時点が与えられます。 - 次に、各時点で適用する実験的アプリケーションの数を決定します。すべての時点で同じ分析を完了して、変化を監視してください。
注:スキャフォールド上での細胞増殖の典型的な分析には、細胞生存率アッセイ、DNA定量、組織学的染色とイメージング、および遺伝子発現解析が含まれます。これらについては、セクション 5 で詳しく説明します。 - 例えば、適切なアッセイを用いた細胞生存率評価の後、DNA/RNA単離(5.1.11で説明)にスキャフォールドを再利用するなど、複数のダウンストリームアプリケーションに同じスキャフォールドを使用するタイミングを決定します。
- アプリケーションごとに最低3つの生物学的複製物(例えば、細胞生存率とDNA定量用に3つの足場、組織学用に3つ、遺伝子発現解析用に3つ)を保管してください。これは時点ごとに 9 つのスキャフォールドに等しいため、3 つのタイム ポイントに 27 個のスキャフォールディングを使用することを計画します。
- 最後に、この数値に実験パラメータまたは条件の数を掛けます(例:複数の細胞株、初期播種密度、足場組成の評価)。
注:このプロトコルでは、4つの異なる初期播種密度が1つの細胞株に対して評価されており、108(27の足場 x 4つの条件)の足場が必要でした。人為的ミスを考慮し、追加のカバレッジを与えるには、この数に~10%を追加します(たとえば、108個の足場が必要な場合は、120個の足場を準備します)。
- 細胞生物学の変化(細胞増殖や代謝など)の全体的な実験タイムラインと評価間隔を決定します。
- 必要なセルの数
注:0日目に円筒形の足場(直径6 mm、高さ4 mm)に細胞を播種するには、20 μLの容量の細胞懸濁液が推奨されます。この20μLの細胞懸濁液あたりの細胞数は、セクション1.1で計算された実験計画に従って調整されます。一般的な初期播種密度は、20μLあたり2×105 細胞であり、以下のプロトコルの例として使用されます。- 実験に必要な細胞の総量を計算するには、20 μLあたり最初の2×105 細胞に、必要な足場の数を掛けます。
注:例えば、30個の足場に20 μLを掛けると、総容量は600 μLになります。各足場が2×105細胞を必要とする場合、600μLの懸濁液は合計6×10 6細胞(2×105×30)を含み、600μL中に6×106細胞の最終的な必要量を与える。実験パラメータの数によって、必要な細胞の総数が決まります。したがって、このプロトコルでは、従来の175 cm2フラスコ10個と同じ数の細胞を処理できる多層細胞培養フラスコを使用して細胞培養の概要を説明します。
- 実験に必要な細胞の総量を計算するには、20 μLあたり最初の2×105 細胞に、必要な足場の数を掛けます。
2. コラーゲンベースの足場の作製
注:Coll-I-nHAおよびColl-I-GAG円筒形足場(直径6 mm、高さ4 mm)は、確立された方法15、21、27を使用して準備されます。以前に発表された方法17に従って化学的に架橋されたら、足場は1週間以内に使用する必要があります。
- 目的の機械的特性を備えた足場を製造した後、足場が完全に水和し、リン酸緩衝生理食塩水(PBS)で完全に洗浄されていることを確認してください。
注:これは通常、足場の架橋後~12時間かかり、4°Cの100 mL組織培養廃液容器で実施でき、容器あたり最大50個の足場と足場あたり2 mLのPBSで実行できます。 - スキャフォールドをPBSで4°Cで保存し、使用する準備が整います。
3. 多層細胞培養フラスコで神経芽腫細胞を増殖させる
注意: 多層フラスコの最適な播種密度は異なります。この実験で使用したフラスコの場合、製造元の指示による最適密度は1 × 107セルです。多層フラスコを播種する前に、適切な組織培養フラスコ(例えば、T175cm2組織培養フラスコ)で1〜107細胞以上の×密度で細胞を増殖させる。細胞を多層フラスコ(セクション3.1)に播種するには、細胞を70〜80%のコンフルエントまで増殖させ、細胞カウントを実行するためのステップ3.2.16〜3.2.20を参照して、mLあたりの細胞数を収穫し、カウントします。細胞懸濁液を数えたら、すぐに多層フラスコの播種に進みます。細胞培養作業は、無菌性を維持するために層流フードで行う必要があります。
- 多層細胞培養フラスコの播種
- 550 mLの完全増殖培地(使用する細胞株によって異なります)と100 mLの滅菌PBSを37°Cのウォーターバスで20分間予温します。
- 回収した細胞懸濁液を用いて、式(1)を用いて、1×107細胞の最適な播種密度を達成するために必要な細胞懸濁液の必要量を計算します(WANTは多層フラスコの播種に必要な細胞数、HAVEは細胞懸濁液中の細胞数/mLを指します)。
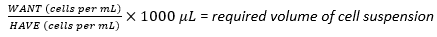 (1)
(1)
例えば。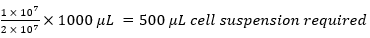
- 予熱した増殖培地100mLに、必要な量の細胞懸濁液を加えます。
- フードに新しい多層フラスコを取り、キャップを取り外して、フラスコを60°の角度で保持します。細胞懸濁液100 mLすべてをフラスコにゆっくりとピペットで移し、ネックの角度の付いた側を下ります。フラスコに蓋をして横に置き、細胞が層全体に均等に分散できるようにします。
- 60°の角度で、予熱した増殖培地400 mLを、ネックの角度のついた側にゆっくりと注ぐかピペッティングして、多層フラスコに加えます。首がいっぱいになった場合は、フラスコを直立位置に戻すか、フラスコに蓋をして横に置いてから注ぎに戻します。
注意: 過度の気泡形成を避けるために、ゆっくりと注いでください。フラスコを直立位置で軽くたたいて、すべての気泡が上部に浮き上がるようにし、10 mLピペットで取り除きます。フラスコが首の底の糸まで満たされていることを確認します。これを実現するには、必要に応じて培地を追加します。 - 多層フラスコに蓋をし、角度のついた首を下に向けて、37°C、5%CO2、湿度95%でインキュベートします。
- 2〜3日ごとに成長をチェックし、コンフルエントを確認します。多層フラスコの下部2層のコンフルエントを確認するには、倒立顕微鏡の4倍対物レンズで観察します。
注:10×7 個の神経芽腫細胞を播種した場合、10層フラスコがコンフルエントになるまでに通常1週間かかりますが、これは使用する細胞株によって異なる場合があります。
- 多層フラスコ内の細胞の定期メンテナンス
- 550 mLの完全増殖培地(使用する細胞株によって異なります)、50〜100 mLのトリプシン、および300 mLの滅菌PBSを37°Cのウォーターバスで20分間予温します。
- 多層フラスコの濃度が70〜80%であることを確認します。
- 多層フラスコを層流フードに入れ、使用済みの培地をフラスコから注いで廃棄します。最初に、フラスコを傾けて、媒体がエアダムを越えて廃棄物容器に注がれるようにします。注いでいる間、媒体がフラスコの角度の付いた首を流れ落ちるまで、フラスコを180°回転させます。フラスコをこの軸に沿って前後に回転させて、残っている液体を取り除きます。
- 予熱した滅菌PBS100 mLを斜めのネックにゆっくりと加えて細胞を洗浄します。フラスコに蓋をして横に置き、PBSが均一に分布するようにし、フラスコを前後に回転させて細胞を洗浄します。
- PBS洗浄は3.2.3と同じ要領で廃棄してください。洗浄手順を繰り返します。
- 予熱したトリプシン 50 mL を、予熱した滅菌 PBS 50 mL で希釈します。希釈したトリプシン溶液100 mLを多層フラスコに加え、キャップをしてフラスコを横にして置き、トリプシンが均一に分布するようにします。細胞株の接着性が高い場合は、100 mLの希釈されていないトリプシンを使用します。
- フラスコを37°C、5%CO 2、湿度95%で2〜5分間インキュベートし、顕微鏡下で細胞剥離をモニタリングします。必要に応じて、フラスコをしっかりと叩いて取り外しを助けるか、インキュベーターにさらに1分間戻します。
- 多層フラスコを層流フードに入れ、100 mLの成長培地でトリプシンを中和します。フラスコに蓋をして横に置き、前後に揺らして完全に中和します。
- 中和した細胞懸濁液を4 x 50 mLのコニカル遠心チューブに注ぎます。
注:完全な細胞回収が必要な場合は、多層フラスコを100 mLの滅菌PBSで再度洗浄し、2 x 50 mL遠心分離チューブに注ぎます。 - 細胞懸濁液を50mL遠心チューブで340×gで3〜4分間遠心分離し、細胞をペレット化します。
- 遠心分離管を層流フードに戻し、各ペレットからできるだけ多くの上清を慎重に廃棄します。
注意: ペレットは大きく、比較的緩いため、簡単に破壊される可能性があります。 - 各ペレットに1〜5 mLの成長培地を加え、ピペッティングで数回上下させて再懸濁します。
- 再懸濁した4つのペレットを1本の50 mL遠心チューブにプールし、ピペットで完全に混合し、総量を記録します。
- 血球計算盤の各外側の正方形に30〜100個の細胞が含まれるように、細胞カウント用の細胞懸濁液を適切に希釈します。
注:適切な開始希釈は1:100です。このために、10 μLの細胞懸濁液を新しい15 mLのコニカル遠心チューブにピペットで移し、990 μLの滅菌PBSで希釈します。混合物を上下に数回ピペットで移し、完全に混合します。 - 細胞ストックの入った50 mL遠心チューブをインキュベーターに入れながらカウントします。
- きれいな血球計算盤を取り出し、 図2に示すように、きれいなカバーガラスをグリッドの上に置きます。
- 希釈した細胞懸濁液10μLを血球計算盤チャンバーにピペットで移します。10 μLの分注がすべて完了する前にチャンバーが満杯になった場合は、ピペッティングを中止してください。
- 血球計算盤を光学顕微鏡の4倍対物レンズの下に置きます。粗い焦点と細かい焦点を調整して、セルを視覚化します。
- 図 2 で強調表示されているように、チャンバーの外側の 4 つの角の正方形のセルの数を数えます。4 つのカウントを加算し、4 で割って、正方形あたりの平均セルを計算します。
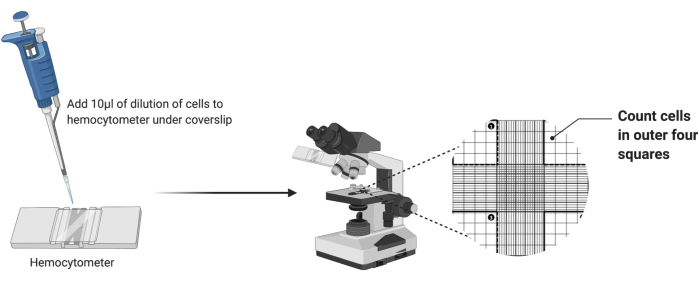
図2:血球計算盤を用いた細胞カウント。 10マイクロリットルの細胞懸濁液をカバーガラスの下の血球計算盤に加えます。次に、チャンバーを顕微鏡の4倍対物レンズの下に置き、グリッドの外側の4つの角にある細胞の数をカウントします。 この図の拡大版をご覧になるには、ここをクリックしてください。
- 平均カウントに希釈倍率(例:100)を掛け、この数に10,000を掛けて、式(2)を用いてmLあたりの細胞数を求めます。
 (2)
(2) - ストック溶液中の細胞の総数を掛けて計算します(例えば、プールした4つの再懸濁ペレットが20 mLの溶液になった場合は、細胞/mLに20を掛けます)。
- 多層フラスコを維持するには、ステップ3.1.2で概説した式(1)を使用して、1〜107 個の細胞をフラスコに戻すのに必要な細胞ストックの量を計算し×ステップ3.1.3-3.1.6を実行してフラスコを再播種します。スキャフォールドをシードする準備ができたら、次のセクションに進みます。
4. 足場に神経芽腫細胞を播種する
- ストックセル懸濁液の調製
注:このプロトコルは各密度間の2の増倍係数のneuroblastomaのセルの4つの播種密度を作成するためのステップを、概説する。したがって、段階希釈を使用して、ストック懸濁液からさらに3つの細胞懸濁液を作成します。細胞培養作業は、無菌性を維持するために層流フードで行う必要があります。- 式(3)を使用して、多層フラスコ内の細胞の総数(セクション3.2.19でカウント)から必要な細胞の体積を計算し、最初の播種密度または細胞ストック懸濁液を調製します。
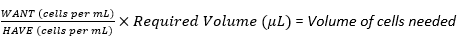 (3)
(3)
注:例えば、30個の足場に必要な足場あたり最大播種密度が6×10個の5細胞であり、各足場に20 μLの細胞懸濁液が供給される場合、ストック細胞懸濁液には、総容量600 μL(20 μL×30個の足場)で1.8 × 107個の細胞(6 × 105細胞×30個の足場)が必要です。
この調製物から段階希釈を行うため、これらの数を2倍に、すなわち、1200μLの総容量で3.6×107 細胞にする必要があります。これをmLあたりの細胞数でWANTに変換するには、3.6 × 107 を1200 μLで分割し、1000 μLを掛けて、1 mLあたり3 × 107 細胞を得る。

- 必要な量の細胞ストックを滅菌済み2 mLまたは15 mL遠心チューブに添加し、増殖培地で最終容量を1200 μLにします。このチューブに「Density 1」というラベルを付けます(図3)。
- 式(3)を使用して、多層フラスコ内の細胞の総数(セクション3.2.19でカウント)から必要な細胞の体積を計算し、最初の播種密度または細胞ストック懸濁液を調製します。
- 段階希釈を実行して、複数の播種密度細胞懸濁液を作成します。
- ステップ 4.1.1 で調製した密度 1 から、図 3 のように段階希釈によりさらに 3 つの密度の細胞懸濁液を調製します。
- 増殖培地で各密度を2倍に希釈します。まず、必要な最終容量(前の例では600 μL)の増殖培地を3本の滅菌済み2 mLまたは15 mL遠心分離チューブに加えます。
- 密度 1 の半分(600 μL)をチューブの 1 つに移し、細胞懸濁液を培地と完全に混合して希釈します。このチューブに 密度 2 というラベルを付けます。
- 密度2の半分(600μL)を次のチューブに移し、細胞懸濁液を培地と完全に混合して希釈します。このチューブに密度 3 というラベルを付けます。
- 密度3の半分(600 μL)を次のチューブに移し、細胞懸濁液を培地と完全に混合して希釈します。このチューブに密度 4 というラベルを付けます。
- 密度 4 から 600 μL を廃棄し、4 つの調製物すべての最終容量が 600 μL になるようにします。
- ネガティブコントロールとして、600 μLの増殖培地を滅菌済みの2 mL遠心チューブにのみ添加します。段階希釈プロセスの概略図については、 図3 を参照してください。
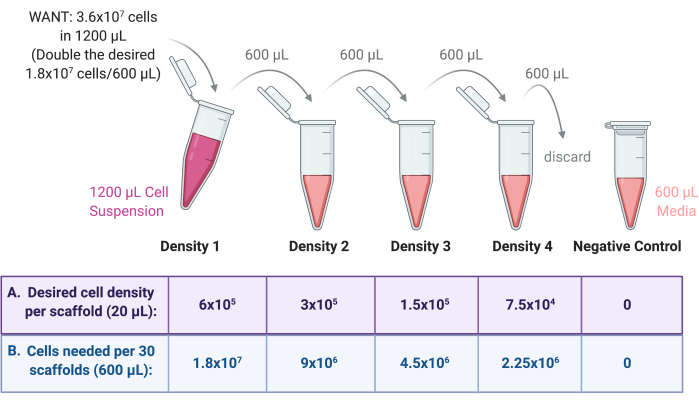
図3:細胞ストックを段階希釈して、4つの異なる足場播種密度の4つの懸濁液を調製 。(A)数は、足場当たりの所望の播種密度に合うように調整することができ、(B)密度当たりの足場の総数を掛けて、各足場に20μLの細胞懸濁液を投与する。この例では、密度 1 は、30 個の足場に対して 600 μL 中の 1.8 ×× 10 7 個の細胞に相当する、1 つの足場あたり 6 つの 10 個の5 個の細胞 を必要とします。この数を2倍にして段階希釈を開始し、600 μLを次のチューブの600 μLの増殖培地で移し、希釈します。このプロセスは、それぞれの間に係数が2の4つの細胞懸濁液が存在するまで続きます。ネガティブコントロールは、600 μLの培地をチューブにのみ添加することによって行われます。 この図の拡大版をご覧になるには、ここをクリックしてください。
- スキャフォールドへの細胞懸濁液の添加
注意: スキャフォールド(PBSで保管)を冷蔵庫から取り出し、室温(RT)に戻してからセルを追加します。- PBSの足場を層流フードに運びます。
- 滅菌ピンセットを使用して、ウェルごとに1つの足場を持つ非接着性の24ウェルプレートに足場を配置します(図1C)。足場の角をそっと持ち上げ、容器の側面に軽く押し付けて、余分なPBSを取り除きます。足場をウェルの中央にスキン側を下にして追加します(足場の光沢のある層側で、プラスチック製の24ウェルプレートに下向きにします)。
- 24ウェルプレートに、細胞株の詳細、関連するパラメーター(播種密度など)、および時点を標識します。一度に1つの細胞播種密度で作業し、残りの密度は使用準備が整うまで37°Cインキュベーターで保持します。
- 層流フードで、P20ピペットと滅菌チップを使用して、関連する細胞懸濁液20 μLを各足場の中央に静かに添加します(図1D)。細胞を足場に加えながらよく混合することにより、細胞を懸濁液中に完全に保持します。懸濁液が足場の上に留まり、ウェルの底部に滑り落ちないようにしてください。
- 細胞を添加したら、プレートを3〜5時間(37°C、5%CO2、湿度95%)インキュベートして、ほとんどの細胞が付着できるようにします。
- インキュベーション後、予熱した増殖培地1 mLを各ウェルにゆっくりと静かに添加します(図1E)。P1000ピペットを使用して培地を追加すると、よりゆっくりと制御された動きが可能になり、足場のずれが防止されます。非常に多くの足場を扱う場合は、ピペットガンを「ドロップ」と「ロー」に設定した10 mLピペットを使用します。
- 24ウェルプレートを一晩(37°C、5%CO2、湿度95%)インキュベートします。
- 足場上のセルのメンテナンス
- 細胞接着の最初の24時間後(1日目)、播種した足場を新しい非接着性の24ウェルプレートに移し、1〜2 mLの新鮮な増殖培地を加えます。
注:このステップでは、プラスチック製の24ウェルプレートの底に落ちた細胞を、足場上で成長させるのではなく、除去します。1日目として指定された足場複製は、セクション5で説明したように、24時間後に削除されます。したがって、メンテナンスはこれらの足場には適用されません。 - 最初に2〜3日ごとに足場を監視して、増殖培地の色が変化しないようにします。時間が経ち、足場内で細胞が増殖するにつれて、細胞に餌を与えることがより頻繁になります。
- 10 mLのピペットガンを使用し、スローモードで使用済みの培地の1 mLを取り除き、廃棄します。馴化培地を必要とする実験を行う場合は、使用済みの生物学的複製培地を15 mLの遠心チューブに回収し、340 × g で2分間遠心分離して細胞破片をペレット化し、上清を新しいチューブに移し、-80°Cで保存します。
- 予熱した増殖培地2 mLを、ピペットのドリップ機能を使用してスキャフォールドに静かに添加し、24ウェルプレートをインキュベーター(37°C、5%CO2、湿度95%)に戻します。培地が所望の成長期間にわたって費やされるたびに繰り返します。
- 細胞接着の最初の24時間後(1日目)、播種した足場を新しい非接着性の24ウェルプレートに移し、1〜2 mLの新鮮な増殖培地を加えます。
5. 足場の回収と応用
注:各時点で、いくつかのアプリケーションを使用して、足場上の細胞増殖をモニターしたり、遺伝子およびタンパク質の発現プロファイルを評価したりできます。スキャフォールドの回収条件は、実行する分析によって異なり、複数の検索方法を次のサブセクションで概説し、 図 4 に示します。
- 足場内の細胞生存率の評価
- 0.2 μmの滅菌フィルターでろ過し、層流フード内の遠心チューブに適切な細胞生存率アッセイ試薬を滅菌します。この滅菌溶液を、完全増殖培地および滅菌PBSとともに37°Cのウォーターバスで予熱します。
- 層流フードで、滅菌ピンセットを使用して、分析する足場を新しい24ウェルプレートに移します。プレートに関連するすべての詳細をラベル付けします。
注: 解析は 3 回に分けて実行してください。 - 予熱した増殖培地900 μLを各ウェルに加え、続いて100 μLの無菌細胞生存試薬を加えます。900 μLの培地と100 μLの無菌細胞生存試薬を足場のないウェルに添加して、ネガティブコントロールを含めます。プレートの蓋を元に戻し、プレートを~3分間静かに揺動させて、希釈した細胞生存率試薬をウェル全体に均等に分散させます。プレートを 37 °C、5% CO2、湿度 95% でインキュベートします。
注:インキュベーション時間は、細胞株ごとに最適化する必要があります。製造元のガイドラインを参照してください。神経芽腫細胞株の場合、4〜6時間のインキュベーションが最適と思われます。 - インキュベーション後、プレートをインキュベーターから取り出し、数秒間静かに揺動させます。
- 層流フードで、新しい半透明の 96 ウェルプレートを開きます。24ウェルプレートの各ウェルから、インキュベートした培地と試薬を96ウェルプレートの3つのウェルに100 μL/ウェルで移し、テクニカルトリプリケートを与えます。
注:この転写により、24ウェルプレートのウェルに700 μLが残ります。 - 96ウェルプレートをアルミホイルで覆い、細胞生存率試薬を光から保護します。
- 残りの700 μLのウェル内容物を、24ウェルプレートの各足場から取り出して廃棄します。各足場を1 mLの滅菌PBSで2回洗浄します。
注意: 足場からすべての色が取り除かれるわけではありません。これらのスキャフォールドは、0.1 M重炭酸ナトリウム(NaHCO3)溶液中の1 mLの1% Triton X-100に入れ、-80°Cで保存することにより、DNA定量などのさらなるアプリケーションに使用できます(セクション5.2、 図4Bを参照)。 - 層流フードから96ウェルプレートを取り外し、マイクロプレートリーダーを使用して波長570 nmおよび600 nmで各ウェルの吸光度を測定します。両方の波長の吸光度値を記録し、メーカーの指示に従って、細胞による細胞生存率試薬の減少率を計算します。
- 適切なソフトウェアを使用して、細胞生存率の結果をグラフ化し、統計的に解析します。生物学的三重値を入力してエラーバーを生成し、アッセイのばらつきを示します。
- 実験期間における細胞生存率の変化を調べるには、適切な生物統計ソフトウェアを使用して平均の多重比較を伴う一元配置分散分析(ANOVA)検定を実行します。
- グラフ上の時点間の有意差をns(P>0.05)、*(P≤0.05)、**(P≤0.01)、***(P≤0.001)、および****(P≤0.0001)として示します。
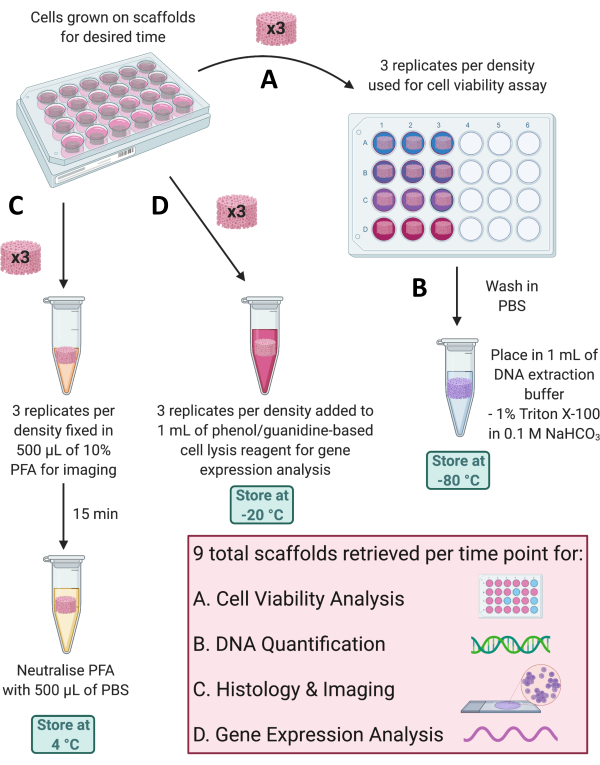
図4:各時点での異なる分析のためのスキャフォールドの検索。 (A)細胞生存率解析のために3つのスキャフォールド複製を回収します。(B)次に、これらの足場をPBSで洗浄し、0.1 M NaHCO3中の1% Triton X-100に入れ、DNA定量のために-80°Cで保存することができます。(C)さらに3回の複製を10%PFAで15分間固定し、PBSで中和し、組織学的染色およびイメージングのために4°Cで保存します。(D)最後に、フェノール/グアニジンベースの細胞溶解試薬に3回複製を添加し、遺伝子発現解析のために-20°Cで保存します。略語:PBS =リン酸緩衝生理食塩水;PFA = パラホルムアルデヒド。 この図の拡大版をご覧になるには、ここをクリックしてください。
- 足場内の細胞からのDNAの定量
注:ステップ 5.1.7 の後の注に記載されているように、DNA 定量のためのスキャフォールドの回収には、スキャフォールドを 0.1 M NaHCO3 溶液中の 1 mL の 1% Triton X-100 を含む 2 mL 遠心分離チューブに入れ、その後 -80 °C で保存します。 DNA分析を行う前に、細胞を3回の凍結融解サイクルを経て神経芽腫細胞を適切に溶解し、定量のためにDNAを放出する必要があります。- Triton X-100に保管したサンプルを-80°Cから取り出します。 サンプルを室温で1〜3時間、または解凍するまで放置します。
- サンプルを10〜20秒間ボルテックスし、サンプルを-80°Cで18〜24時間、または完全に凍結するまで戻します。このプロセスを合計3回の凍結融解サイクルで繰り返します。
- DNA収量を最大化するには、組織ライザーを使用して足場内の細胞を破壊します。
- Triton X-100の足場が入った2 mL遠心チューブに金属ビーズを入れ、チューブをアダプター内に置き、サンプルを50振動/秒で2〜3分間振とうします。
注意: 金属ビーズがテーパー底チューブに詰まる可能性があるため、丸底の遠心チューブを使用してください。
- Triton X-100の足場が入った2 mL遠心チューブに金属ビーズを入れ、チューブをアダプター内に置き、サンプルを50振動/秒で2〜3分間振とうします。
- Triton X-100 溶液中の DNA を蛍光二本鎖 DNA(dsDNA)染色試薬で定量し、マイクロプレートリーダーを使用して発光を測定します。製造元のガイドラインを参照してください。サンプルをトリスエチレンジアミン四酢酸(TE)バッファーで適切に希釈し、TE バッファーで段階希釈して 8 種類の dsDNA 標準試料を調製します(図 5)。
- 黒色不透明の 96 ウェルプレートで、各標準試料またはサンプル 100 μL を 3 回に分けてウェルに添加します。
- 蛍光 dsDNA 染色を TE バッファーで 200 倍に希釈し、マルチチャンネルピペットを使用して各標準試料/サンプルに 100 μL を加えます。プレートをアルミホイルで覆い、室温で5分間インキュベートします。
- 各ウェルの蛍光を 520 nm で測定し、記録します。メーカーのガイドラインに従って、各サンプル中のDNAの濃度を計算します。
注:使用する細胞株について、細胞当たりのDNAの平均濃度がわかっている場合、DNA濃度値は、式(4)を用いて細胞数に変換することができる。 (4)
(4) - 適切なソフトウェアを使用して、DNA定量結果をグラフ化し、統計的に解析します。生物学的三重値を入力してエラーバーを生成し、アッセイのばらつきを示します。
- 実験期間中のDNA濃度/細胞数の変化を調べるには、適切な生物統計ソフトウェアを使用して平均を多重比較する一元配置分散分析検定を実行します。
- グラフ上の時点間の有意差をns(P>0.05)、*(P≤0.05)、**(P≤0.01)、***(P≤0.001)、および****(P≤0.0001)として示します。
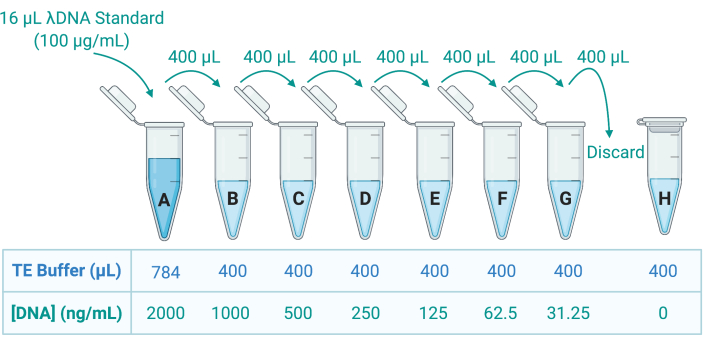
図5:検量線作成のための8種類のDNA標準試料の調製。 λDNA のストック溶液は 100 μg/mL で提供されます。これを TE バッファーで 50 倍に希釈し、2000 ng/mL で標準試料 A を調製します。次に、400 μL の A を 400 μL の TE バッファーを含むチューブ B に移します。次に、400 μL の B を C で 2 倍に希釈し、G. 標準 H は TE バッファーのみで構成されているため、DNA 濃度は 0 ng/mL になります。略語:TE = Tris-EDTA。 この図の拡大版をご覧になるには、ここをクリックしてください。
- 組織学的染色のための足場の調製
注:スキャフォールドは、免疫蛍光法(IF)用のホールスキャフォールドとして、または組織学的染色または免疫組織化学(IHC)用のホルマリン固定パラフィン包埋(FFPE)スライスとして、顕微鏡およびイメージングの目的で固定および染色できます。これにより、足場内の細胞の浸透と分布を定性的に評価することができ、タンパク質の発現を評価するために使用できます。- PBSに10%パラホルムアルデヒド(PFA)溶液を調製します。足場あたり最終容量 500 μL に十分な溶液があることを確認してください。
- この溶液を37°Cで予熱し、500 μLを標識された2 mL遠心チューブに添加して、足場回収を行います。
- 非接着性の24ウェルプレートから足場を取り外し(ステップ4.4.4)、層流フードに入れます。
- 滅菌ピンセットを使用して、足場を10%PFAを含むラベル付き遠心チューブに移します。足場を PFA 溶液で 15 分間固定します。各チューブに 500 μL の PBS を添加して PFA を中和し、4 °C で保存します。
- 自動ティッシュプロセッサー用の足場を準備するには、4°Cからスキャフォールドを取り出し、ピンセットを使用して、関連するすべての詳細を鉛筆でラベル付けしたプラスチックカセットに入れます。すべてのカセットをティッシュプロセッサーの金属製容器に入れます。
- Tissue Processorで12段階のプロトコルを開始し、足場を固定、脱水、除去し、パラフィンで一晩浸透させます。処理したサンプルが入ったカセットを回収します。
- 次に、足場をパラフィンワックスブロックに埋め込んで、足場を非常に薄いスライスに切り分けて染色します。
注:足場をカセットから取り外してワックスに埋め込むときは、画像が撮影される角度に影響を与えるため、足場の向きを考慮することが特に重要です。これは、足場への細胞浸潤を評価する際に重要です。 - ワックス埋め込み器とコールドプレートをオンにします。蓋を持ち上げてワックスのレベルを確認します。必要に応じて補充してください。
- 一度に1つのサンプルで作業し、カセットを開き、足場を取り外して、プラスチック金型の中央に配置します。
- ホットワックスをサンプルに注ぎ、正しい向きが維持されていることを確認し、ワックスが固まる前に、必要に応じて温かいピンセットで調整します。型を満たすためにさらにワックスを注ぎます。
- ラベルの付いたカセットの蓋をプラスチック型の上に置き、その上にワックスを追加します。型をコールドプレートに置き、ワックスを固めます。4°Cで一晩保存し、ミクロトミーの前にパラフィンワックスが完全に固化するようにします。
- ミクロトミーの準備をするには、35°Cのウォーターバス、乾燥プレート、およびミクロトームをオンにします。
- ホルダーに刃を差し込み、レバーを締めて固定します。
- トリムと断面の厚さを設定します (通常、足場断面の場合は 5 mm)。
- FFPE足場を金型から取り外し、ミクロトーム前面のホルダーに固定し、切片を切断する前にサンプルの端の周りの余分なワックスを慎重にトリミングします。
- ミクロトームのレバーを回転させてワックスブロックへの切り込みを開始し、スムーズな動きを確保します。
- リボン状の切片を一度に約3つの足場部分に集め、35°Cのウォーターバスにそっと入れてしわを取り除きます。ピンセットを使用して、ウォーターバス内で切片を静かに分離します。
- ポリシンコーティングされたガラス顕微鏡スライドを使用して、各切片をウォーターバスから持ち上げて、切片がスライドの中央に来るようにします。各スライドに鉛筆でラベルを付けます。
- スライドガラスを乾燥プレートの上または60°Cの乾燥オーブンに入れます。乾燥したら、4°Cで保存し、必要な組織学的染色またはIHC染色に進みます。
- 遺伝子発現解析のためのスキャフォールドの回収
- インキュベーターから非接着性の24ウェルプレートから足場を取り除き(ステップ4.4.4)、層流フードに入れます。
- 滅菌ピンセットを使用して、足場を新しいラベルの付いた2 mL遠心チューブに移します。
- ドラフト内で、フェノール/グアニジンベースの細胞溶解試薬1 mLを各チューブに添加して、足場内の細胞を溶解し、高品質のRNAを回収できるようにします。
- 適切なキットを使用してRNA抽出を行う準備ができるまで、チューブを-20°Cで保管します。標準的な逆転写定量的ポリメラーゼ連鎖反応(RT-qPCR)17を用いて、足場内の細胞における遺伝子発現を評価します。
結果
ここで説明するコラーゲンベースのスキャフォールドモデルは、神経芽腫生物学の研究から、従来の2D細胞培養よりも生理学的に天然腫瘍に類似した環境での抗がん治療薬のスクリーニングまで、多くの用途があります。特定の研究課題をテストする前に、目的の実験時間枠内で細胞の接着、増殖、および浸潤の完全な特性評価を得ることが重要です。増殖条件は、特定の各細胞株の生物学によって異なります。重要なことは、最適な条件と頑健な性能を決定するために、細胞増殖評価のいくつかの方法を実施する必要があるということです。
ここでは、足場上で増殖した神経芽腫細胞の生存率を比色細胞生存率アッセイを用いて評価した。このアッセイは、実験時間枠全体を通して必要な頻度で実行できます。記載された実験では、1日目、7日目、および14日目に、4つの異なる密度でColl-I-nHAスキャフォールド上で増殖させた2つの神経芽腫細胞株KellyLucおよびIMR32について細胞生存率評価を実施しました(図6)。1日目の生存率は、その後のすべての測定値を比較するためのベースラインとして設定されました。細胞生存率試薬の減少速度は、細胞生物学と個々の細胞株の増殖速度や代謝などの増殖特性を反映しています。足場に播種された細胞の数と減少のレベルとの間に相関関係があると予想されました。この実験では、細胞生存率試薬の減少は、予想通り、すべての密度で両方の細胞株について、各時点で概ね増加しました。
次に、各密度を両方の細胞株について個別に評価し、各時点間の減少を比較しました。テューキーの多重比較検定による一元配置分散分析を実施して、時点間の減少の有意差を検出しました(図7)。細胞株とすべての播種密度の両方について、1日目と14日目を比較すると、細胞生存率試薬の減少に有意な増加(P<0.05)が見られました。これは、足場上に存在する代謝活性細胞の有意な増加を示しました。この増加は、7日間隔(1日目と7日目、7日目と14日目)を評価すると、すべてのケースで有意ではなく、望ましい成長ウィンドウを達成するための播種密度の最適化の重要性を示しています。
細胞生存率アッセイの結果を裏付けるために、蛍光dsDNA染色剤を用いて足場から抽出したdsDNAを定量することで、足場上の細胞増殖を間接的に測定することもできます(図8A)。細胞の生存率と同様に、DNAの定量は実験のタイムライン内で必要な頻度で行うことができます。ただし、この分析では、足場の完全な回収と細胞増殖の停止が必要なため、セクション1で説明したように実験計画に織り込む必要があります。この実験では、1日目、7日目、14日目に、Coll-I-nHAスキャフォールド上で4つの異なる密度で増殖させた2つの神経芽腫細胞株、KellyLucおよびIMR32のDNAを定量しました。これらの細胞株では、細胞あたりのdsDNAの平均濃度がわかっているため、定量されたDNAからサンプルあたりの細胞数を導き出すことができました(図8B)。
DNA定量では、細胞生存率評価よりも生物学的複製間のばらつきが大きくなりましたが、一般的には各時点で増加し、14日目に最高レベルが定量されました。IMR32細胞は、DNA濃度で示されているように、Coll-I-nHAスキャフォールド上でKellyLuc細胞よりも高い細胞数に達するようです。次に、2つの細胞株について各密度を個別に評価し、各時点にわたる減少を比較しました。テューキーの多重比較検定による一元配置分散分析を実施して、時点間の減少の有意差を検出しました(図8B)。
細胞株とすべての播種密度の両方について、1日目と14日目を比較すると、播種密度4(1 × 105 細胞/足場)のKellyLucを除いて、細胞数に有意な増加(
次に、足場上の細胞増殖形態と分布の目視評価を実施し、従来のヘマトキシリンとエオシン(H&E)染色、およびIHCを網羅しました。個々の細胞株の異なる増殖パターンは、足場への浸透度の違いや細胞のクラスタリングなど、足場上のさまざまな空間配置につながると予想されます。足場をホルマリン固定し、パラフィン包埋し、5 mmの切片に切断し(図9A)、組織学的染色やIHCなどの複数の可視化技術のために足場を準備しました。
1日目、7日目、14日目にコラーゲンベースの足場で増殖したKelly、KellyCis83、およびIMR32細胞にルーチンH&E染色を適用しました(図9B)。これにより、2つのコラーゲンベースの足場上の細胞の空間配向を14日間にわたって可視化することができました。シスプラチン感受性Kelly細胞および耐性KellyCis83細胞を、Coll-I-nHA足場(図9B、i)とColl-I-GAG足場(図9B、ii)の両方で増殖させました。以前に発表されたデータと一致して、KellyCis83細胞は、侵襲性の低いKelly細胞株よりも高い速度で増殖し、両方の足場組成物に深く浸透しました。Coll-I-nHAで増殖した別の神経芽腫細胞株IMR32のH&E染色は、対照的な増殖パターンを示しています(図9B、iii)。この細胞株は、14日間にわたってコラーゲン足場上に大きく密集したクラスターで増殖しました。明視野共焦点顕微鏡は、コラーゲン繊維の自家蛍光によるコラーゲンベースの足場の多孔質構造を可視化するために使用できます(図9C)。
細胞骨格アクチンを標的とするファロイジンと核対比染色剤である4',6-ジアミジノ-2-フェニルインドール(DAPI)で細胞を染色し、実験タイムライン全体にわたって特定の細胞形質をモニターしました。この手法を用いて、Coll-I-GAGスキャフォールド上のKellyおよびKellyCis83細胞に豊富なアクチンが観察されました(図9D)。これらの結果は、このプロトコルを使用して足場上で増殖した神経芽腫細胞から空間的に分解された情報を得るために、複数のイメージング技術をどのように使用できるかを示しています。一定期間におけるコラーゲンベースの足場上の細胞増殖パターンの特性評価は、下流の生化学的アッセイの理解と解釈を改善します。
コラーゲンベースの足場で増殖した細胞によるタンパク質発現を解析して、細胞活性を in vivo シナリオと比較することができます。以前に発表されたデータでは、細胞単層で増殖したKellyLuc細胞およびKellyCis83Luc細胞、ならびにColl-I-nHAおよびColl-I-GAGスキャフォールド上で、神経芽細胞腫の代理分泌マーカーとしてのクロモグラニンA(CgA)の発現を調べました(図10)。酵素結合免疫吸着アッセイ(ELISA)を用いて、コンディショニング培地中の CgA を評価しました(図 10A)。CgAは、より侵攻性の化学療法抵抗性KellyCis83細胞株では、Kellyよりも高い速度で分泌されます(図10B、C)。これは、Coll-I-GAGおよびColl-I-nHAスキャフォールド(P<0.05)の両方で7日目に有意であったが、従来の2D培養で単層として増殖した細胞では、この時点で有意差は認められなかった。
また、これらの結果は、単層で細胞を増殖させる場合、細胞がコンフルエントに達するまでにわずか7日間の増殖しか実行できないという、実験のタイムラインが限られていることも浮き彫りにしています。足場上での細胞の増殖は、より生理学的に適切な条件でより長い期間にわたって維持できるため、この制限を克服します。細胞生存率、DNA含量、細胞形態および空間配置、および発現プロファイルに関する情報を取得するための上記の技術の組み合わせは、さまざまなコラーゲンベースの足場における神経芽腫細胞の増殖の評価を容易にします。また、このプロトコルは、特定の実験要件や目的のアプリケーションを満たすように簡単に適合させることができます。
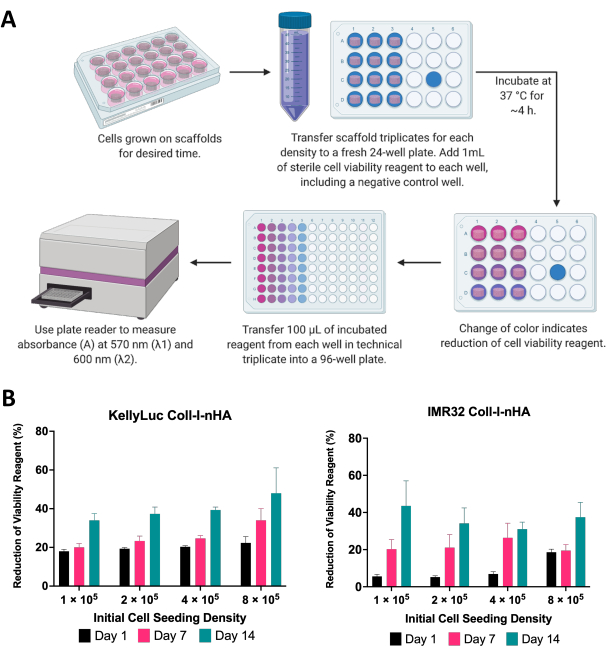
図6:細胞生存率分析。 (A)比色細胞生存率アッセイを使用して、コラーゲンベースの足場上の神経芽腫細胞の生存率を測定するための一般的な手順。インキュベーション期間は、メーカーのガイドラインを参照して、新しい細胞株ごとに最適化する必要があります。(B)1日目、7日目、14日目に測定した、4つの異なる初期播種密度でColl-I-nHAスキャフォールド上で増殖したKellyLucおよびIMR32細胞による細胞生存率試薬の減少率。サンプルは、標準偏差を表すエラーバーを使用して生物学的に3回評価されました。略語:nHA =ナノヒドロキシアパタイト;Coll-I-nHA = nHAを添加したコラーゲン足場。 この図の拡大版をご覧になるには、ここをクリックしてください。
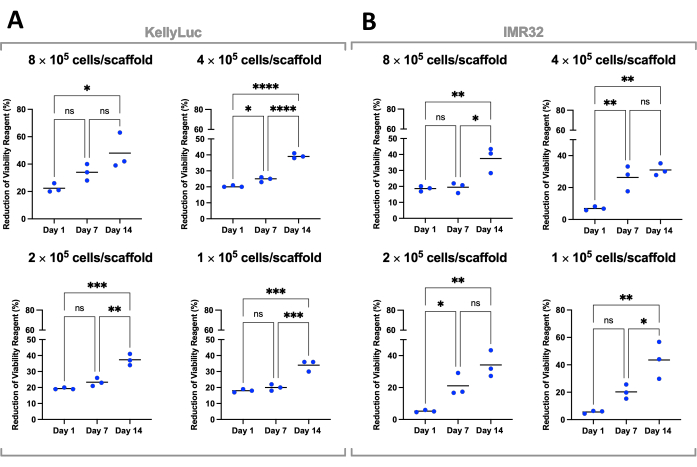
図7:Coll-I-nHAで増殖した細胞の播種密度による細胞生存率(14日間)。 (A)ケリーリュック;(B)IMR32。タイトル付きセル番号は、0日目のスキャフォールド上の初期セル播種密度を示します。サンプルは生物学的に三重で評価され、三重点で示され、棒は平均を表しています。多重比較による一元配置分散分析を使用して、グラフに示されている3つの時点における細胞生存率試薬の減少率の有意差を検出しました(ns P > 0.05、* P ≤ 0.05、** P ≤ 0.01、*** P ≤ 0.001、**** P ≤ 0.0001)。略語:nHA =ナノヒドロキシアパタイト;Coll-I-nHA = nHAを添加したコラーゲン足場。ANOVA = 分散分析ns = 有意ではない。この図の拡大版をご覧になるには、ここをクリックしてください。
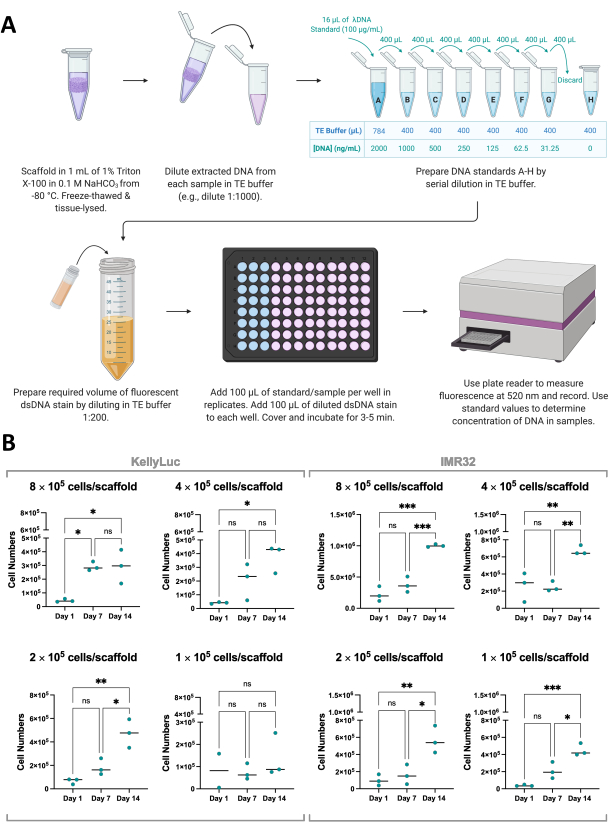
図8:足場内の細胞から抽出したDNAの定量。 (A)蛍光dsDNA染色を用いて、コラーゲンベースの足場上で増殖した細胞のdsDNAを定量するプロセス。(B)Coll-I-nHAで14日間培養したKellyLucおよびIMR32細胞の播種密度によるDNA定量分析からの細胞数。タイトルの付いた細胞番号は、0日目の足場への初期細胞播種密度を示す。サンプルは生物学的に三重で評価され、三重点で示され、棒は平均を表しています。多重比較による一元配置分散分析を使用して、グラフに示された3つの時点における細胞数の有意差を検出しました(ns P > 0.05、* P ≤ 0.05、** P ≤ 0.01、*** P ≤ 0.001、**** P ≤ 0.0001)。略語:nHA =ナノヒドロキシアパタイト;Coll-I-nHA = nHAを添加したコラーゲン足場。dsDNA = 二本鎖DNA;TE = トリス-EDTA;ANOVA = 分散分析ns = 有意ではない。 この図の拡大版をご覧になるには、ここをクリックしてください。
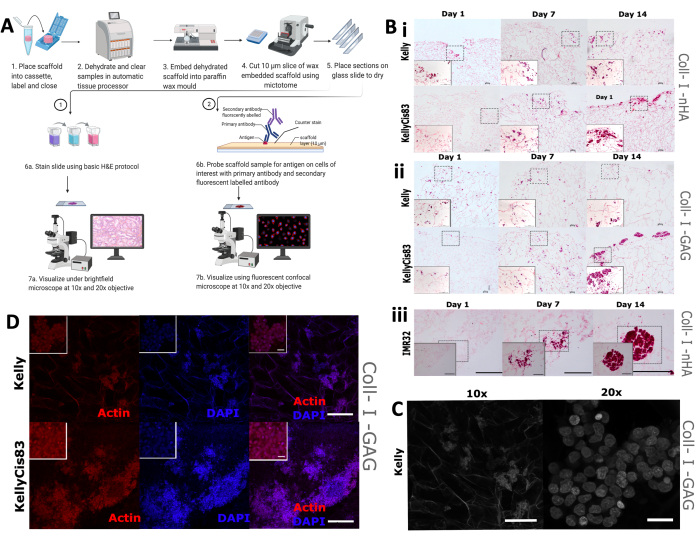
図9:足場の免疫組織化学分析のための組織処理ステップ 。 (A)画像解析用の足場を処理するためのプロトコルの概略図。このプロセスにより、一次抗体と蛍光標識二次抗体を用いたルーチンの組織学的染色と特異的抗体プロービングが可能になります。(B)H&E染色した3つの神経芽腫細胞株の代表的な画像。H&E画像は、実験の経時的な成長パターンを監視するために、1日目、7日目、14日目に撮影されます。スケールバー = 200 μm。破線の四角形は、左下端の 20 倍画像にズームインするために選択された領域を表します。スケールバー = 20 μm。 (i および ii) 2 種類のコラーゲンベースの足場上の Kelly および KellyCis83 神経芽腫細胞株 (それぞれ上段と下段) の H&E。(iii)IMR32細胞株のH&E、Coll-I-nHAスキャフォールド上のクラスター化された細胞増殖を表す。(C)明視野共焦点顕微鏡にかけたKelly細胞株の代表画像。コラーゲン自家蛍光により、多孔質の足場を可視化することができます。10倍スケールバー = 200 μm、20倍スケールバー = 20 μm。 (D)埋め込みスキャフォールドの代表画像と、ファロイジンとDAPIを用いたIHCによる分析を10倍倍の倍率で行ったもの、スケールバー=200μm。内側の小さい正方形は拡大画像(20倍)を表し、スケールバー = 20 μm。略語:nHA =ナノヒドロキシアパタイト;Coll-I-nHA = nHAを添加したコラーゲン足場。GAG = グリコサミノグリカン;Coll-I-GAG = コンドロイチン-6-硫酸を添加したコラーゲン足場。H&E = ヘマトキシリンおよびエオシン;IHC = 免疫組織化学;DAPI = 4',6-ジアミジノ-2-フェニルインドール。 この図の拡大版をご覧になるには、ここをクリックしてください。
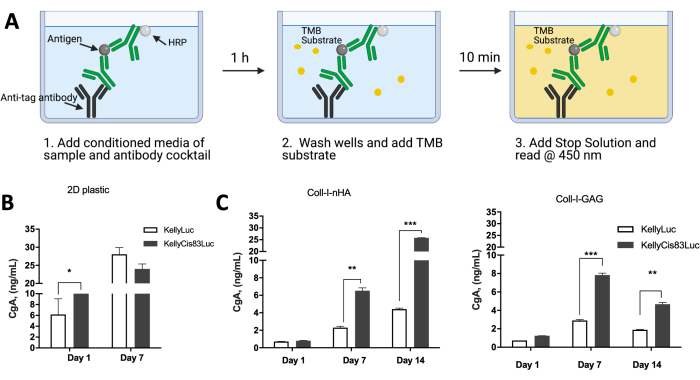
図10:3Dコラーゲンベースの足場で増殖した神経芽腫細胞によるタンパク質発現と2Dプラスチックの比較 。 (A)2Dプラスチックまたは3Dコラーゲンベースの足場で増殖した細胞の馴化培地でCgA ELISAをどのように実施したかの概略図。(B)2Dプラスチック単分子膜上で増殖した細胞の馴化培地から採取したCgAタンパク質発現レベル。細胞は7日後にコンフルエントに達したため、14日の時点は読み取れませんでした。プラスチックの7日目までに、Kelly細胞株とKellyCis83細胞株の間でCgAレベルに有意差はありませんでした。(C)コラーゲンベースの足場で増殖した細胞の馴化培地を用いて14日間連続して実施したCgA ELISA。7日目には、両方のコラーゲン足場において、より攻撃的なKellyCis83細胞株でCgAレベルが高く、2D単層と比較して3DマトリックスのCgAの生理学的関連性レベルがより高くなっています。この図は Curtin et al.17 から修正されたものです。略語:3D =3次元。2D = 2次元。CgA = クロモグラニン A;ELISA = 酵素結合免疫吸着アッセイ;nHA =ナノヒドロキシアパタイト;Coll-I-nHA = nHAを添加したコラーゲン足場。GAG = グリコサミノグリカン;Coll-I-GAG = コンドロイチン-6-硫酸を添加したコラーゲン足場。TMB = 3,3',5,5'-テトラメチルベンジジン;HRP = 西洋ワサビペルオキシダーゼ。 この図の拡大版をご覧になるには、ここをクリックしてください。
ディスカッション
3D足場がん細胞モデルは、簡略化されたTME32における神経芽細胞腫細胞の増殖、生存率、および細胞の浸潤に関するメカニズムの洞察を得るための貴重で用途の広いツールであることが証明されています。ここで説明する3D神経芽腫モデルは、最小TMEを模倣し、2D単層培養よりも生理学的に関連性のあるデータを提供します。3D細胞培養の大きな欠点は、実験が複雑になり、時間が長くなることです。ここでは、コラーゲンベースの足場上で神経芽腫細胞の播種、増殖、および維持のための最適化されたプロトコルと、それに続く下流の分析とアプリケーションについて説明し、細胞増殖の堅牢な特性評価を実現します。私たちは、足場に最適な細胞播種密度に関する洞察を得て、抗がん剤治療を評価するための予測可能で制御可能な環境を14日間の迅速な実験ウィンドウで作成することを目指しました。これらすべての記述された簡単なプロトコルの組み合わせは、足場ベースの in vitro 培養システムにおける神経芽腫細胞増殖の包括的な評価を提供します。
プロトコール設定の重要なポイントは、科学者が研究室で迅速に確立できるように強調されています。例えば、比色細胞生存率アッセイの性能を向上させるために示されたインキュベーション時間により、足場細孔への試薬のより深い浸透がすべての細胞に到達することができます。さらに、蛍光dsDNA染色技術は頑健で簡単です。しかし、足場からのDNA放出には、細胞がコラーゲン繊維内に「閉じ込められる」ため、活発な細胞溶解が必要です。
このモデルを用いて、抗がん剤スクリーニング用のコラーゲンベースの足場の対数成長期を同定することができます。記載された実験設定では、4つの初期細胞播種密度を、全体で14日間の期間と1日目、7日目、および14日目の分析時点とともに使用しました。4×105細胞/足場に播種されたKellyLuc細胞は、7日目から14日目の間に最も有意に活発な増殖ウィンドウを持つことを特定しました。この対数相増殖データにより、さまざまな細胞毒性実験の信頼性の高い解釈が可能になります。これにより、薬物の毒性ではなく、3D多孔質プラットフォーム上での増殖の抑制に起因する増殖または細胞死の低下に関する憶測が排除されます。細胞生存率は、異なる細胞タイプの増殖をサポートするための3Dプラットフォームの適合性についても広く使用されている評価です33,34。細胞生存率を測定するには、生染色/死染色、ATP測定、増殖アッセイなど数多くのアッセイがありますが、Alamar Blue比色細胞生存率アッセイの使用は、DNA定量データをサポートするためのシンプルで効果的な手法であることがわかりました。
DNA定量と細胞生存率の併用により、14日間にわたって継続的な増殖を達成するために足場に播種する細胞の最適な密度は、平均して2〜4〜×105 細胞/足場であるという補完的な証拠が得られました。ただし、このプロトコルは、さまざまな実験時間枠、分析時間ポイント、およびダウンストリームアプリケーションを満たすように簡単に適合させることができます。このプロトコルが足場のneuroblastomaのセルの単一培養のセル成長の評価を記述するが、足場は共同培養のためのプラットホームとして使用のために容易に修正可能である、傷治療の調査のケラチノサイトそして線維芽細胞を共培養するのにコラーゲンGAGの足場を利用した等Amaralを、する35。
記載された3Dモデルは、免疫蛍光法や標準H&Eなどのさまざまなよく知られた技術を使用して、細胞の増殖と浸潤を可視化することを可能にします。細胞形態や足場上の増殖パターンの多様性により、生化学的アッセイを用いて細胞を可視化し、増殖の特性評価を行うことが重要です。増殖パターンを理解することで、成長行動や抗がん剤に対する将来の反応に関する洞察を得ることができます。例えば、DNA定量を用いたIMR32の増殖はKellyと同様のパターンをもたらしますが、H&Eを用いた可視化では、IMR32はより分散した増殖を示したKellyよりも大きなクラスターで増殖します(図9)。足場内の細胞株のこれらの多様な増殖パターンは、腫瘍の不均一性の臨床シナリオを反映している。3Dスキャフォールドで形態の異なる細胞株のパネルを使用して抗がん剤の反応を調べると、同じ薬剤に対する患者の反応の予測値が高まります。
遺伝子またはタンパク質発現の検出は、目的のタンパク質が分泌される場合、RT-qPCRやELISAなどの他のアプローチを使用して行うこともできます。神経芽腫進行の代理マーカーであるクロモグラニンA(CgA)36を使用して、神経芽腫細胞増殖を3Dでさらに特徴付けました。先行研究17で説明したように、細胞が増殖するにつれてCgA分泌が増加しました(図10)。単層細胞培養ではこの増加を捉えることができませんでしたが、増殖は細胞が培養皿内で完全にコンフルエントに達したことを意味するため、3Dコラーゲンスキャフォールドを使用することで、CgA分泌の長期評価が可能になりました。
この3D in vitro モデルは、神経芽腫の生物学と治療薬への反応を研究するためのすべての研究課題に適しているとは限りません。制限の1つは、足場内の不均一な細胞浸透と、特定の細胞株に依存するさまざまなサイズの細胞クラスターの形成であり、栄養素と試験薬の制御不能な拡散につながる可能性があります。この特徴は、治療スクリーニングにおける頑健性に影響を与えます。しかしながら、この限界にもかかわらず、天然腫瘍はサイズおよびがん細胞の分布においても不均一であり、腫瘍組織内に他の多くの細胞型を含むことを考慮することが重要である。この制限を克服するために、各細胞が投入された足場を単一の微小組織として使用し、次のパラメーターを最適化することを提案します:(a)細胞生存率試薬が細胞および細胞クラスターに到達するためのインキュベーション時間、および(b)足場上の細胞を組織ライザーで前処理することにより、Triton X-100バッファーで細胞を溶解し、足場の奥深くに含まれる細胞のDNAを放出します。
このプロトコルのもう一つの技術的制限は、このモデル用に新しく製造された足場の各バッチの機械的テストの欠如です。しかし、足場の物理的および化学的特性(圧縮弾性率および引張弾性率、気孔率および視覚的細孔構造、および均質性)に関連して広範囲に特徴付けられている足場の堅牢な製造プロセスを使用することで、足場の品質がバッチ21、24、27、30、37を通じて維持されることが保証されます。
要約すると、この論文は、コラーゲンベースの足場上の細胞増殖を分析するための一連の簡単な方法を提示します。実験のタイムラインと分析ポイントは、特定の研究課題に応じて入れ替えることができます。このプロトコルは他の細胞タイプにも適応可能です。上記の結果は、この方法の編集により、さまざまな神経芽腫細胞株が14日間にわたって連続的に増殖するための最適な播種密度に関する洞察がどのように得られたかについての証拠を提供します。このプロトコルのすべての方法から得られる結果の合併は3Dコラーゲンのマトリックス内のセル成長の優秀な理解をもたらします。このモデルの将来の利用には、神経芽細胞腫TMEに特異的な共培養システムや、さまざまな新規抗がん剤の試験が含まれる可能性があります。
開示事項
著者は利益相反がないことを宣言します。
謝辞
この研究は、National Children's Research Centre(NCRC)、Irish Research Council(IRC)、およびNeuroblastoma UKの支援を受けた。イラストはBioRenderを使用して作成されました。
資料
| Name | Company | Catalog Number | Comments |
| Cells | |||
| IMR-32 | ATCC | CCL-127 | |
| Kelly | ECACC | 82110411 | |
| KellyCis83 | Made in lab – derived from Kelly (Piskareva et al., 2015) | - | Increasing exposure to cisplatin. Cross resistance acquired |
| SH-SY5Y | ATCC | CRL-2266 | |
| Disposable | |||
| 0.22 µm syringe filter | Millex | SLHP033RS | |
| 1.5 mL Eppendorf tube | Eppendorf | 0030 120.086 | |
| 100 mL sterile Pot | Starstedt | - | |
| 10 mL plastic pipette | Cellstar | 607 180 | |
| 15 mL Falcon tube | Starstedt | 62.554.502 | |
| 25 mL plastic pipette | Cellstar | 760 180 | |
| 50 mL Falcon tube | Starstedt | 62.547.254 | |
| 5 mL plastic pipette | Cellstar | 606 180 | |
| 6 mm Biopsy punches | Kai Medical | BP-60F | |
| Aluminium foil | - | - | |
| Cover Slip | Menzel-Glaser | - | |
| HYPERflask | Corning | CLS10030 | |
| Microscope slides | Thermo Scientific | J1840AMNT | |
| Opaque black 96-well plate | Costar | 3915 | |
| Sterile P10 tips | Starlab | S1121-3810 | |
| Sterile P1000 tips | Starlab | S1122-1830 | |
| Sterile P20 tips | Starlab | S1123-1810 | |
| Sterile P200 tips | Starlab | S1120-8810 | |
| T-175 (175 cm2 flask) | Sarstedt | 83.3912 | |
| T-75 (75 cm2 flask) | Sarstedt | 83.3911.302 | |
| Translucent clear 96 well plate | Cellstar | 655180 | |
| Translucent non-adherent 24 well plates | Cellstar | 83.3922.500 | |
| Equipment | |||
| Autoclave | Astell | - | |
| Automatic tissue processor | Leica | TP1020 | |
| Centrifuge 5804 | Eppendorf | - | |
| Hemocytometer | Hausser Scientific | - | |
| Incubator | ThermoScientific | - | |
| Microtome | Leica | RM2255 | |
| Oven | Memmert | Calibrated by: Cruinn diagnostics Ltd | |
| P10 pipette | Gilson | ||
| P100 pipette | Gilson | ||
| P1000 pipette | Gilson | ||
| P20 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| P200 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| Paraffin section flotation bath | Electrothermal | MH8517 | Calibrated by: Cruinn diagnostics Ltd |
| Pipette electronic dispenser | Corning | StripipetterUltra | Calibrated by: Cruinn diagnostics Ltd |
| Plate cooler | Leica | EG1140C | Calibrated by: Cruinn diagnostics Ltd |
| Refrigerator -20 °C | Liebherr | - | |
| Refrigerator -80 °C | Liebherr | - | |
| Refrigerator 4 °C | Liebherr | - | |
| Seesaw Rocker | DLAb | SK-D1807-E | |
| Spectrophotometer – Victor3V Platereader | PerkinElmer | 1420 | |
| Tissue culture hood/Laminar flow hood | GMI | 8038-30-1044 | |
| Tissue Lyser | Qiagen | TissueLyser LT | |
| Tweezers | - | - | |
| Water bath | Grant | - | |
| Wax embedder | Leica | EG1140H | |
| Materials | |||
| 1 L Water | Adrona - Biosciences | 568 | |
| 1% Triton-X | Sigma Aldrich | 9002-93-1 | |
| 10x PBS tablets | Sigma Aldrich | P4417-100TAB | |
| 37% paraformaldehyde | Sigma-Aldrich | F8775 | |
| Alamar Blue Cell Viability Reagent | Invitrogen | DAL1100 | |
| Collagen- glycosaminoglycan scaffold | Tissue engineering research group (TERG) | ||
| Collagen-nanohydroxyapatite scaffold | Tissue engineering research group (TERG) | ||
| dH20 | Adrona - Biosciences | 568 | |
| Eosin | Sigma-Aldrich | E4009 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| EtOH | Sigma-Aldrich | 1.00983.2500 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| F12 | Gibco | 21765-029 | |
| FBS | Gibco | 10270-106 | |
| Hemaytoxylin | Sigma-Aldrich | HHS32-1L | |
| L-Glutamine | Gibco | 25030-024 | |
| MEM | Gibco | 21090-022 | |
| miRNA easy Kit | Qiagen | 217004 | |
| MNEAA’s | Gibco | 11140-035 | |
| Penicillin/streptomycin | Gibco | 015140-122 | |
| Qiazol | Qiagen | 79306 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Invitrogen | P11496 | |
| RPMI | Gibco | 21875-034 | |
| Sodium bicarbonate | Sigma Aldrich | S7795-500G | |
| Tissue embedding Medium | Sigma | A6330-4LB | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Software | |||
| Excel | - | Excel 2016 | |
| ImageJ | - | - | |
| Prism | - | Version 9 |
参考文献
- Davidoff, A. M. Neuroblastoma. Seminars in Pediatric Surgery. 21 (1), 2-14 (2012).
- Matthay, K. K., et al. Neuroblastoma. Nature Reviews Disease Primers. 2, 16078 (2016).
- Costard, L. S., Hosn, R. R., Ramanayake, H., O'Brien, F. J., Curtin, C. M. Influences of the 3D microenvironment on cancer cell behaviour and treatment responsiveness: a recent update on lung, breast and prostate cancer models. Acta Biomaterialia. , (2021).
- Borriello, L., Seeger, R. C., Asgharzadeh, S., Declerck, Y. A. More than the genes, the tumor microenvironment in neuroblastoma. Cancer Letters. 380 (1), 304-318 (2016).
- Walker, C., Mojares, E., Del Río Hernández, A. Role of extracellular matrix in development and cancer progression. International Journal of Molecular Sciences. 19 (10), 3028 (2018).
- Bissell, M. J., Hall, H. G., Parry, G. How does the extracellular matrix direct gene expression. Journal of Theoretical Biology. 99 (1), 31-68 (1982).
- Schultz, G. S., Davidson, J. M., Kirsner, R. S., Bornstein, P., Herman, I. M. Dynamic reciprocity in the wound microenvironment. Wound Repair and Regeneration. 19 (2), 134-148 (2011).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6, 11 (2008).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4, 38 (2006).
- Ouellette, J. N., et al. Navigating the collagen jungle: The biomedical potential of fiber organization in cancer. Bioengineering. 8 (2), 1-19 (2021).
- Kreger, S. T., Voytik-Harbin, S. L. Hyaluronan concentration within a 3D collagen matrix modulates matrix viscoelasticity, but not fibroblast response. Matrix Biology. 28 (6), 336-346 (2009).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Hume, R. D., et al. Tumour cell invasiveness and response to chemotherapeutics in adipocyte invested 3D engineered anisotropic collagen scaffolds. Scientific Reports. 8 (1), 12658 (2018).
- Fitzgerald, K. A., et al. The use of collagen-based scaffolds to simulate prostate cancer bone metastases with potential for evaluating delivery of nanoparticulate gene therapeutics. Biomaterials. 66, 53-66 (2015).
- Sapudom, J., Pompe, T. Biomimetic tumor microenvironments based on collagen matrices. Biomaterials Science. 6 (8), 2009-2024 (2018).
- Curtin, C., et al. A physiologically relevant 3D collagen-based scaffold-neuroblastoma cell system exhibits chemosensitivity similar to orthotopic xenograft models. Acta Biomaterialia. 70, 84-97 (2018).
- Gavin, C., et al. Neuroblastoma invasion strategies are regulated by the extracellular matrix. Cancers. 13 (4), 1-23 (2021).
- Ridky, T. W., Chow, J. M., Wong, D. J., Khavari, P. A. Invasive three-dimensional organotypic neoplasia from multiple normal human epithelia. Nature Medicine. 16 (12), 1450-1456 (2010).
- Casal, e. J., Crane, J. S. . Biochemistry, Glycosaminoglycans. , (2019).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. J. The effect of pore size on cell adhesion in collagen-GAG scaffolds. Biomaterials. 26 (4), 433-441 (2005).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. Influence of freezing rate on pore structure in freeze-dried collagen-GAG scaffolds. Biomaterials. 25 (6), 1077-1086 (2004).
- Haugh, M. G., Murphy, C. M., McKiernan, R. C., Altenbuchner, C., O'Brien, F. J. Crosslinking and mechanical properties significantly influence cell attachment, proliferation, and migration within collagen glycosaminoglycan scaffolds. Tissue Engineering. Part A. 17 (9-10), 1201-1208 (2011).
- Murphy, C. M., Haugh, M. G., O'Brien, F. J. The effect of mean pore size on cell attachment, proliferation and migration in collagen-glycosaminoglycan scaffolds for bone tissue engineering. Biomaterials. 31 (3), 461-466 (2010).
- Haugh, M. G., Jaasma, M. J., O'Brien, F. J. The effect of dehydrothermal treatment on the mechanical and structural properties of collagen-GAG scaffolds. Journal of Biomedical Materials Research - Part A. 89 (2), 363-369 (2009).
- Lowe, B., Hardy, J. G., Walsh, L. J. Optimizing nanohydroxyapatite nanocomposites for bone tissue engineering. ACS Omega. 5 (1), 1-9 (2020).
- Cunniffe, G. M., Dickson, G. R., Partap, S., Stanton, K. T., O'Brien, F. J. Development and characterisation of a collagen nano-hydroxyapatite composite scaffold for bone tissue engineering. Journal of Materials Science. Materials in Medicine. 21 (8), 2293-2298 (2010).
- DuBois, S. G., et al. Metastatic sites in stage IV and IVS neuroblastoma correlate with age, tumor biology, and survival. Journal of Pediatric Hematology/Oncology. 21 (3), 181-189 (1999).
- Ryan, A. J., Gleeson, J. P., Matsiko, A., Thompson, E. M., O'Brien, F. J. Effect of different hydroxyapatite incorporation methods on the structural and biological properties of porous collagen scaffolds for bone repair. Journal of Anatomy. 227 (6), 732-745 (2015).
- Tierney, C. M., et al. The effects of collagen concentration and crosslink density on the biological, structural and mechanical properties of collagen-GAG scaffolds for bone tissue engineering. Journal of the Mechanical Behavior of Biomedical Materials. 2 (2), 202-209 (2009).
- Cox, R. F., Jenkinson, A., Pohl, K., O'Brien, F. J., Morgan, M. P. Osteomimicry of mammary adenocarcinoma cells in vitro; increased expression of bone matrix proteins and proliferation within a 3D collagen environment. PLoS One. 7 (7), 41679 (2012).
- Nolan, J. C., et al. Preclinical models for neuroblastoma: advances and challenges. Cancer Letters. 474, 53-62 (2020).
- Sirivisoot, S., Pareta, R., Harrison, B. S. Protocol and cell responses in threedimensional conductive collagen gel scaffolds with conductive polymer nanofibres for tissue regeneration. Interface Focus. 4 (1), 20130050 (2014).
- Thevenot, P., Nair, A., Dey, J., Yang, J., Tang, L. Method to analyze three-dimensional cell distribution and infiltration in degradable scaffolds. Tissue Engineering. Part C-Methods. 14 (4), 319-331 (2008).
- do Amaral, R. J. F. C., et al. Functionalising collagen-based scaffolds with platelet-rich plasma for enhanced skin wound healing potential. Frontiers in Bioengineering and Biotechnology. 7, 371 (2019).
- Gkolfinopoulos, S., Tsapakidis, K., Papadimitriou, K., Papamichael, D., Kountourakis, P. Chromogranin A as a valid marker in oncology: Clinical application or false hopes. World Journal of Methodology. 7 (1), 9-15 (2017).
- O'Brien, F. J., et al. The effect of pore size on permeability and cell attachment in collagen scaffolds for tissue engineering. Technology and Health Care. 15 (1), 3-17 (2007).
Erratum
Formal Correction: Erratum: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds
Posted by JoVE Editors on 6/23/2022. Citeable Link.
An erratum was issued for: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds. The Authors section was updated.
The Authors section was updated from:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Fergal J. O’Brien3,4,5
Olga Piskareva1,2,3,5,6
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
5Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
6National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
to:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Graeme Kelly4
Fergal J. O’Brien3,5,6
Olga Piskareva1,2,3,6,7
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Department of Chemistry, Royal College of Surgeons in Ireland (RCSI), 123 St. Stephen’s Green, Dublin 2, Ireland
5Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
6Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
7National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved