Method Article
Modelo Biomimético Tridimensional In Vitro de Neuroblastoma Utilizando Arcabouços à Base de Colágeno
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
Este artigo lista as etapas necessárias para semear linhagens celulares de neuroblastoma em arcabouços tridimensionais à base de colágeno previamente descritos, manter o crescimento celular por um período de tempo pré-determinado e recuperar arcabouços para várias análises de crescimento e comportamento celular e aplicações a jusante, adaptáveis para satisfazer uma variedade de objetivos experimentais.
Resumo
O neuroblastoma é o tumor sólido extracraniano mais comum em crianças, sendo responsável por 15% das mortes totais por câncer pediátrico. O tecido tumoral nativo é um microambiente tridimensional (3D) complexo envolvendo camadas de células cancerosas e não cancerosas circundadas por uma matriz extracelular (MEC). A MEC fornece suporte físico e biológico e contribui para a progressão da doença, prognóstico do paciente e resposta terapêutica.
Este trabalho descreve um protocolo para a montagem de um sistema baseado em arcabouço 3D para mimetizar o microambiente do neuroblastoma usando linhagens celulares de neuroblastoma e scaffolds à base de colágeno. Os scaffolds são suplementados com nanohidroxiapatita (nHA) ou glicosaminoglicanos (GAGs), naturalmente encontrados em altas concentrações no osso e na medula óssea, os sítios metastáticos mais comuns do neuroblastoma. A estrutura porosa 3D desses arcabouços permite a fixação, proliferação e migração de células do neuroblastoma e a formação de aglomerados celulares. Nessa matriz 3D, a resposta celular à terapêutica é mais reflexiva da situação in vivo .
O sistema de cultura baseado em scaffold pode manter densidades celulares mais altas do que a cultura celular bidimensional (2D) convencional. Portanto, os protocolos de otimização para números iniciais de células de semeadura são dependentes dos prazos experimentais desejados. O modelo é monitorado avaliando-se o crescimento celular via quantificação de DNA, a viabilidade celular via ensaios metabólicos e a distribuição celular dentro dos scaffolds via coloração histológica.
As aplicações deste modelo incluem a avaliação de perfis de expressão gênica e proteica, bem como testes de citotoxicidade usando drogas convencionais e miRNAs. O sistema de cultura 3D permite a manipulação precisa de componentes celulares e da MEC, criando um ambiente fisiologicamente mais semelhante ao tecido tumoral nativo. Portanto, este modelo 3D in vitro irá avançar no entendimento da patogênese da doença e melhorar a correlação entre os resultados obtidos in vitro, in vivo em modelos animais, e os seres humanos.
Introdução
O neuroblastoma é um câncer pediátrico do sistema nervoso simpático que surge durante o desenvolvimento embrionário ou início da vida pós-natal devido à transformação das células da crista neural1. É o tumor sólido extracraniano mais comum em crianças, representando 8% das neoplasias malignas diagnosticadas em pacientes menores de 15 anos e é responsável por 15% de todas as mortes por câncer infantil. A doença apresenta comportamentos clínicos altamente heterogêneos devido a alterações cromossômicas, genéticas e epigenéticas específicas, além de características histopatológicas.
Essas alterações contribuem para a agressividade do neuroblastoma e maus resultados em pacientes pediátricos. Assim, as terapias atuais mostram-se ineficazes em longo prazo para quase 80% dos pacientes com doença clinicamenteagressiva2, ressaltando o fato de que o tratamento para esse grupo de pacientes permanece desafiador. Isso provavelmente se deve aos mecanismos de heterogeneidade e metástases do neuroblastoma ainda não totalmente compreendidos. No entanto, acredita-se que o microambiente tumoral (TME) desempenhe um papel na progressão de muitos cânceres; no entanto, permanece pouco estudada noneuroblastoma3,4.
A EMT nativa é um microambiente 3D complexo envolvendo células cancerosas e não cancerosas circundadas por uma MEC. A MEC refere-se ao componente acelular de um tecido que fornece suporte estrutural e bioquímico aos seus residentes celulares e contribui para a progressão da doença, prognóstico do paciente e resposta terapêutica5. Essa promoção da progressão da doença deve-se à "reciprocidade dinâmica" ou comunicação bidirecional contínua entre as células e a MEC 6,7,8. À medida que o câncer progride, o colágeno estromal é reorganizado, muitas vezes em padrões lineares perpendiculares à interface estroma-câncer, que as células cancerosas utilizam como rota migratória para a metástase9,10,11.
Os principais componentes desse arcabouço biológico funcional nativo incluem uma rede fibrosa de colágenos tipo I e II e outras proteínas, incluindo elastina, glicoproteínas como a laminina, bem como uma gama de proteoglicanos e outros componentes solúveis12,13. Essas proteínas da MEC nativa tornaram-se biomoléculas naturais atrativas para o desenvolvimento de modelos 3D in vitro3. A aplicação de scaffolds 3D para cultura celular in vitro está aumentando em popularidade devido à sua maior representação fisiológica da TME em comparação com a cultura monocamada 2D tradicional. Os arcabouços 3D fabricados auxiliam a fixação, proliferação, migração, metabolismo e resposta celular a estímulos observados em sistemas biológicos in vivo.
O principal componente desses scaffolds 3D é o colágeno, que é peça-chave em muitos processos biológicos normais, incluindo reparo tecidual, angiogênese, morfogênese tecidual, adesão celular e migração11. Matrizes 3D baseadas em colágeno têm mostrado sua funcionalidade robusta para modelar a MEC, servindo como um microambiente biomimético in vitro, permitindo interações célula-MEC, bem como migração e invasão celular. Essas matrizes 3D também fornecem uma análise mais precisa da resposta celular a drogas quimioterápicas do que a cultura 2D tradicional ou "plana" em muitos modelos de câncer 14,15,16, incluindo o neuroblastoma 17,18. A análise genética de culturas de células 3D tem relatado maior correlação com o perfil tecidual humano, mesmo quando comparada a modelosanimais19. De modo geral, a pedra angular desses scaffolds 3D é fornecer às células um ambiente in vitro adequado, que recapitule a arquitetura do tecido nativo e facilite o crosstalk molecular bidirecional8.
Para aumentar a complexidade dos modelos à base de colágeno, outros componentes comuns da MEC são incorporados no processo de engenharia tecidual, criando modelos fisiologicamente mais relevantes para refletir TMEs de nicho de diferentes tecidos. Por exemplo, os GAGs, polissacarídeos carregados negativamente presentes em todos os tecidos de mamíferos20, facilitam a acessão, migração, proliferação e diferenciação celular. O sulfato de condroitina é um tipo específico de GAG encontrado no osso e cartilagem, que tem sido previamente utilizado em aplicações de engenharia tecidual para reparo ósseo21,22,23,24,25. A nano-hidroxiapatita (nHA) é o principal constituinte inorgânico da composição mineral do tecido ósseo humano, constituindo até 65% do osso em peso26 e, portanto, é amplamente utilizada para substituição e regeneraçãoóssea27. Assim, GAGs e nHA são compostos atraentes para reconstruir o neuroblastoma primário da MEC e modelar os sítios metastáticos mais comuns de neuroblastoma, medula óssea (70,5%) e osso (55,7%)28.
Os scaffolds incorporando esses componentes da MEC foram originalmente desenvolvidos para aplicações em engenharia de tecido ósseo com extensa análise de sua biocompatibilidade, toxicidade e características osteocondutoras e osteoindutoras29,30. São matrizes porosas, à base de colágeno, produzidas por meio de técnicas de liofilização para controle de suas propriedades físicas e biológicas. Os arcabouços de colágeno suplementados com nHA (Coll-I-nHA) ou condroitina-6-sulfato (Coll-I-GAG) demonstraram sucesso em mimetizar a EMT primária no câncer de mama31 e metástase para osso no câncer de próstata15, bem como neuroblastoma17. A técnica de liofilização utilizada na confecção desses scaffolds compósitos proporciona homogeneidade reprodutível no tamanho dos poros e porosidade no interior dos scaffolds22,23,24. Resumidamente, uma lama de colágeno (0,5% em peso%) é fabricada pela mistura de colágeno fibrilar com ácido acético 0,05 M. Para Coll-I-GAG, 0,05% em peso de sulfato de crondoitina-6 isolado da cartilagem de tubarão é adicionado à lama de colágeno durante a mistura. Para os scaffolds compósitos de Coll-I-nHA, partículas nanométricas de hidroxiapatita são sintetizadas como descrito anteriormente27 e adicionadas à lama de colágeno na proporção de 2:1 em relação ao peso do colágeno durante o processo de mistura. Todos os andaimes são fisicamente reticulados e esterilizados usando um tratamento desidrotérmico a 105 °C por 24 h25. Scaffolds cilíndricos (6 mm de diâmetro, 4 mm de altura) são obtidos por meio de um punch de biópsia e podem ser quimicamente reticulados com 3 mM de cloridrato de N-(3-dimetilaminopropil)-N'-etilcarbodiimida e 5,5 mM de N-hidroxisuccinimida (EDAC/NHS) em água destilada (dH2O) para melhorar as propriedades mecânicas dos construtos30. Este processo de fabricação bem otimizado de dois arcabouços de colágeno cria arcabouços com propriedades mecânicas reproduzíveis, incluindo tamanho de poros, porosidade e rigidez (kPa). Ambos os andaimes Coll-I-GAG e Coll-I-nHA têm propriedades físicas variadas, criando diferentes condições ambientais. As propriedades de cada andaime são exibidas na Tabela 1.
| Coll-I-GAG | Coll-I-nHA | |
| Tamanho do andaime (diâmetro [mm] x altura [mm]) | Eixo 6 x 4 17 | Eixo 6 x 4 17 |
| Concentração de colágeno (em peso) | 0,5 17 | 0,5 17 |
| Concentração de substrato (em peso) [com base no peso do colágeno] | 0,05 15,17 | 200 17 |
| Tamanho médio dos poros (mm) | 96 22 | 96 – 120 29 |
| Porosidade (%) | 99,5 23 | 98,9 – 99,4 27 |
| Rigidez (kPa) | 1,5 27 | 5,5 - 8,63 29 |
Tabela 1: Visão geral das propriedades mecânicas dos dois arcabouços adotados para o estudo da biologia do neuroblastoma.
Este trabalho descreve um protocolo de montagem de um sistema baseado em arcabouço 3D para melhor mimetizar o microambiente do neuroblastoma usando linhagens celulares de neuroblastoma e arcabouços à base de colágeno previamente descritos suplementados com nHA (Coll-I-nHA) ou condroitina-6-sulfato (Coll-I-GAG). O protocolo inclui métodos a jusante para analisar os mecanismos de crescimento das células do neuroblastoma em um ambiente fisiologicamente mais relevante, usando métodos de baixo custo previamente otimizados adaptados da cultura 2D monocamada Figura 1.
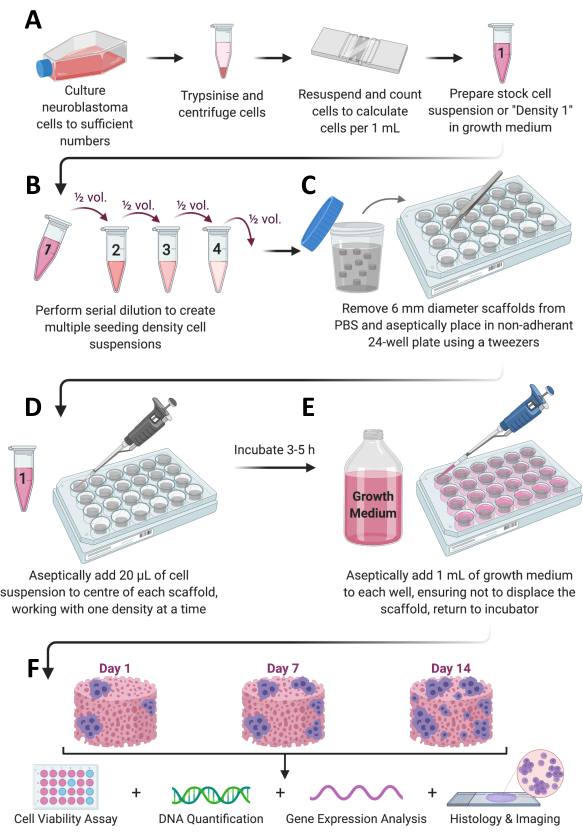
Figura 1: Fluxo de trabalho geral do protocolo. (A) As células são cultivadas em número suficiente, divididas, contadas e ressuspensas em um volume apropriado de meio. (B) Esse estoque de células passa então por diluição em série para preparar um total de 4 suspensões celulares de diferentes densidades. (C) Os arcabouços à base de colágeno são esterilizados em placas não aderentes de 24 poços, e (D) 20 μL de suspensão celular são adicionados ao centro de cada arcabouço e deixados para incubar a 37 °C, 5% de CO2 e 95% de umidade por 3-5 h. (E) O meio de crescimento completo (1 mL) é então adicionado lentamente a cada arcabouço, e as placas são colocadas de volta na incubadora para permitir o crescimento celular pelo tempo desejado. (F) Em cada momento pré-determinado, vários scaffolds são recuperados para avaliação da viabilidade e crescimento celular, análise da expressão gênica e coloração histológica. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Delineamento experimental
NOTA: O número de scaffolds e células necessários para cada experimento será dependente da escala do experimento e pode ser calculado usando as ferramentas nesta seção sobre planejamento experimental.
- Número de andaimes necessários
- Determinar o cronograma experimental geral e os intervalos de avaliação para mudanças na biologia celular (por exemplo, crescimento e metabolismo celular).
NOTA: Por exemplo, o período experimental é de 14 dias, com uma avaliação do crescimento celular a cada 7 dias. Assim, os pontos de tempo experimentais são Dias 1, 7, 14 dando um total de 3 pontos de tempo. - Em seguida, decida o número de aplicações experimentais a serem aplicadas em cada momento. Tente concluir as mesmas análises em todos os momentos para monitorar as mudanças.
NOTA: Análises típicas de crescimento celular em scaffolds incluem ensaios de viabilidade celular, quantificação de DNA, coloração histológica e imagem, e análise de expressão gênica. Estas questões serão discutidas mais aprofundadamente na secção 5. - Decidir quando utilizar o mesmo arcabouço para múltiplas aplicações a jusante, por exemplo, após a avaliação da viabilidade celular utilizando um ensaio adequado, reutilizar o arcabouço para isolamento de ADN/ARN (discutido em 5.1.11).
- Manter um mínimo de 3 réplicas biológicas para cada aplicação, por exemplo, 3 scaffolds para viabilidade celular e quantificação de DNA, 3 para histologia e 3 para análise de expressão gênica. Como isso equivale a 9 andaimes por ponto de tempo, planeje usar 27 andaimes para 3 pontos de tempo.
- Finalmente, multiplique esse número pelo número de parâmetros ou condições experimentais (por exemplo, avaliação de múltiplas linhagens celulares, densidades iniciais de semeadura, composições de andaimes).
NOTA: Neste protocolo, 4 diferentes densidades iniciais de semeadura estavam sendo avaliadas para uma linhagem celular, resultando em 108 (27 scaffolds x 4 condições) scaffolds necessários. Para contabilizar o erro humano e dar cobertura extra, adicione ~10% a esse número, por exemplo, se forem necessários 108 andaimes, prepare 120 andaimes.
- Determinar o cronograma experimental geral e os intervalos de avaliação para mudanças na biologia celular (por exemplo, crescimento e metabolismo celular).
- Número de células necessárias
NOTA: Um volume de 20 μL de suspensão celular é recomendado para semear células em um andaime cilíndrico (diâmetro 6 mm, altura 4 mm) no Dia 0. O número de células por este 20 μL de suspensão celular é ajustado de acordo com o desenho experimental, calculado no ponto 1.1. Uma densidade de semeadura inicial comum é de 2 × 105 células por 20 μL, usada como exemplo para o protocolo abaixo.- Para calcular a quantidade total de células necessárias para o experimento, multiplique as 2 × 105 células iniciais por 20 μL pelo número de arcabouços necessários.
NOTA: Por exemplo, 30 andaimes multiplicados por 20 μL dá um volume total de 600 μL. Se cada arcabouço requer 2 × 10 5 células, 600 μL de suspensão contém um total de 6 × 10 6 células (2 × 105 × 30), dando uma exigência final de 6 × 106 células em 600 μL. O número de parâmetros experimentais ditará o número total de células necessárias. Este protocolo, portanto, delineia a cultura celular usando um frasco de cultura de células multicamadas, que pode lidar com o mesmo número de células que 10 frascos tradicionais de 175 cm2.
- Para calcular a quantidade total de células necessárias para o experimento, multiplique as 2 × 105 células iniciais por 20 μL pelo número de arcabouços necessários.
2. Preparação de suportes à base de colágeno
OBS: Os scaffolds cilíndricos Coll-I-nHA e Coll-I-GAG (diâmetro 6 mm, altura 4 mm) são preparados usando métodos estabelecidos 15,21,27. Uma vez reticulados quimicamente, de acordo com métodos publicados anteriormente17, os andaimes devem ser usados dentro de 1 semana.
- Após a fabricação dos andaimes com as propriedades mecânicas desejadas, certifique-se de que os andaimes estejam totalmente hidratados e completamente lavados em solução salina tamponada com fosfato (PBS).
NOTA: Isso geralmente leva ~12 h após a reticulação dos scaffolds e pode ser realizado em recipientes de resíduos de cultura de tecidos de 100 mL a 4 °C, com um máximo de 50 scaffolds por recipiente e 2 mL de PBS por scaffold. - Conservar os andaimes em PBS a 4 °C até estarem prontos a utilizar.
3. Propagar células de neuroblastoma em frasco de cultura celular multicamadas
NOTA: A densidade de semeadura ideal para o frasco multicamadas varia. Para o frasco utilizado neste experimento, a densidade ideal de acordo com as instruções do fabricante é de 1 × 107 células. Antes de semear o balão multicamadas, propagar as células a uma densidade de 1 × 107 células ou superior num balão de cultura de tecidos adequado (por exemplo, um balão de cultura de tecidos T175 cm2 ). Para semear células no frasco multicamadas (secção 3.1), cultivá-las até 70-80% confluentes, colher e contar o número de células por ml, referindo-se aos passos 3.2.16-3.2.20 para a realização da contagem celular. Uma vez contada a suspensão celular, proceder-se imediatamente à semeadura do balão multicamadas. O trabalho de cultura de células deve ser realizado em uma capela de fluxo laminar para manter a esterilidade.
- Semeando o frasco de cultura celular multicamadas
- Pré-aquecer 550 mL de meio de crescimento completo (varia de acordo com a linhagem celular em uso) e 100 mL de PBS estéril em banho-maria a 37 °C por 20 min.
- Usando a suspensão de células colhidas, calcule o volume necessário da suspensão celular necessário para atingir a densidade de semeadura ideal de 1 × 107 células usando a equação (1), onde WANT refere-se ao número de células necessárias para semear o frasco multicamadas, e HAVE refere-se ao número de células/mL na suspensão celular:
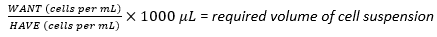 (1º)
(1º)
por exemplo,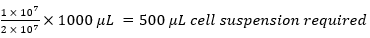
- Adicionar o volume necessário da suspensão celular a 100 mL do meio de crescimento pré-aquecido.
- Pegue um novo frasco multicamadas no capô, retire a tampa e segure o frasco em um ângulo de 60°. Pipetar lentamente todos os 100 mL da suspensão celular para o frasco, descendo pelo lado angulado do pescoço. Tampe o balão e coloque-o de lado para permitir que as células se distribuam uniformemente pelas camadas.
- Em um ângulo de 60°, adicionar 400 mL do meio de crescimento pré-aquecido ao balão multicamadas, despejando lentamente ou pipetando pelo lado angulado do pescoço. Se o pescoço ficar cheio, devolva o frasco à posição vertical ou tampe o frasco e coloque-o de lado antes de voltar a derramar.
NOTA: Despeje lentamente para evitar a formação excessiva de bolhas. Bater suavemente no balão na posição vertical para permitir que todas as bolhas subam para o topo e retire-as com uma pipeta de 10 ml. Certifique-se de que o balão está preenchido até ao fio inferior do pescoço; Adicione mais meio, se necessário, para conseguir isso. - Tampe o frasco multicamadas e incube-o com o pescoço angulado virado para baixo a 37 °C, 5% CO2 e 95% de umidade.
- Verifique o crescimento a cada 2-3 dias para confluência. Para verificar a confluência das duas camadas inferiores do frasco multicamadas, visualize-as sob uma lente objetiva 4x de um microscópio invertido.
NOTA: Quando semeado com 1 × 107 células de neuroblastoma, o frasco de 10 camadas normalmente leva uma semana para se tornar confluente - embora isso possa variar dependendo da linhagem celular usada.
- Manutenção rotineira das células do frasco multicamadas
- Pré-aquecer 550 mL de meio de crescimento completo (varia de acordo com a linhagem celular em uso), 50-100 mL de tripsina e 300 mL de PBS estéril em banho-maria a 37 °C por 20 min.
- Verifique se o frasco multicamadas tem 70-80% de confluência.
- Colocar o balão multicamadas numa capela de fluxo laminar e eliminar o meio gasto do balão por derramamento. Inicialmente, incline o frasco para que o meio seja despejado sobre a barragem de ar em um recipiente de resíduos. Durante o despejo, gire o balão em 180° até que o meio esteja fluindo pelo pescoço angulado do frasco. Gire o balão para frente e para trás ao longo deste eixo para eliminar qualquer líquido restante.
- Lave as células adicionando 100 mL de PBS estéril pré-aquecido lentamente pelo pescoço angulado. Tampe o frasco, coloque-o de lado para permitir uma distribuição uniforme do PBS e gire o frasco para trás e para a frente para lavar as células.
- Eliminar a lavagem PBS da mesma forma que o ponto 3.2.3. Repita as etapas de lavagem.
- Diluir 50 mL de tripsina pré-aquecida em 50 mL de PBS estéril pré-aquecido. Adicionar 100 ml de solução diluída de tripsina ao balão multicamadas, tampar e colocar o balão de lado para permitir uma distribuição uniforme da tripsina. Se a linhagem celular for altamente aderente, use 100 mL de tripsina não diluída.
- Incubar o frasco por 2-5 min a 37 °C, 5% CO2 e 95% de umidade, monitorando o desprendimento celular ao microscópio. Se necessário, bata firmemente no frasco para ajudar no descolamento ou coloque-o novamente na incubadora por mais um minuto.
- Colocar o frasco multicamadas em capela de fluxo laminar e neutralizar a tripsina com 100 mL de meio de cultura. Tampe o frasco, coloque-o de lado e balançar para trás e para frente para garantir a neutralização completa.
- Despeje a suspensão de células neutralizadas em tubos de centrífuga cônica de 4 x 50 mL.
NOTA: Se for necessária a colheita completa de células, lavar novamente o balão multicamadas com 100 ml de PBS estéril e deitá-lo em tubos de centrifugação de 2 x 50 ml. - Centrifugar a suspensão celular em tubos de centrífuga de 50 mL a 340 × g por 3-4 min para peletizar as células.
- Retorne os tubos da centrífuga para a capela de fluxo laminar e descarte cuidadosamente o máximo de sobrenadante possível de cada pellet.
NOTA: O pellet será grande e relativamente solto e, portanto, pode ser interrompido facilmente. - Adicione 1-5 mL de meio de crescimento a cada pellet e ressuspenda-o pipetando para cima e para baixo várias vezes.
- Junte os 4 pellets ressuspensos em um tubo de centrífuga de 50 mL, misture-os cuidadosamente com a pipeta e observe o volume total.
- Fazer uma diluição apropriada da suspensão celular para contagem de células, de modo que cada quadrado externo no hemocitômetro contenha 30-100 células.
NOTA: Uma diluição de partida adequada é de 1:100; para isso, pipetar 10 μL da suspensão celular em um tubo de centrífuga cônica de 15 mL e diluí-lo com 990 μL de PBS estéril. Pipetar a mistura para cima e para baixo várias vezes para misturar completamente. - Coloque um tubo de centrífuga de 50 mL contendo o estoque de células na incubadora durante a contagem.
- Tomando um hemocitômetro limpo, coloque uma lamínula limpa sobre a grade, como mostra a Figura 2.
- Pipetar 10 μL da suspensão celular diluída para a câmara do hemocitômetro. Se a câmara ficar cheia antes de todos os 10 μL serem dispensados, pare de pipetar.
- Coloque o hemocitômetro sob a objetiva 4x de um microscópio de luz. Ajuste o foco grosso e fino para visualizar as células.
- Conte o número de células nos quatro quadrados de canto externo da câmara, conforme destacado na Figura 2. Adicione as quatro contagens e divida por 4 para calcular a média de células por quadrado.
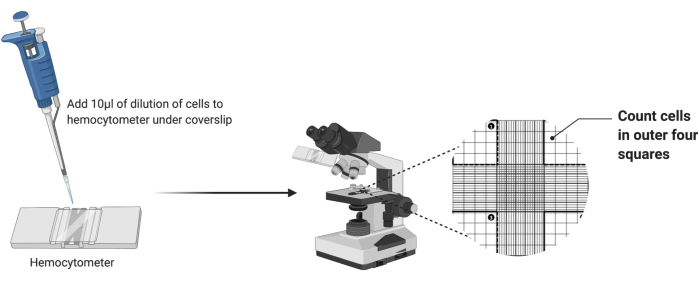
Figura 2: Contagem celular por hemocitômetro. Dez microlitros de suspensão celular são adicionados ao hemocitômetro sob a lamínula. A câmara é então colocada sob a lente objetiva 4x de um microscópio, e o número de células nos quatro cantos externos da grade é contado. Clique aqui para ver uma versão maior desta figura.
- Multiplique a contagem média pelo fator de diluição (por exemplo, 100) e multiplique esse número por 10.000 para obter o número de células por mL usando a equação (2).
 (2º)
(2º) - Calcule o número total de células na solução-estoque multiplicando pelo volume total do estoque de células (por exemplo, se os 4 pellets ressuspensos agrupados produzirem uma solução de 20 mL, multiplique células/mL por 20).
- Para manter o balão multicamadas, utilizar a equação (1) descrita no passo 3.1.2 para calcular o volume de células necessário para semear 1 ×10 7 células de volta ao balão e executar os passos 3.1.3-3.1.6 para voltar a semear o balão. Se estiver pronto para semear os andaimes, prossiga para a próxima seção.
4. Células de neuroblastoma de sementes em arcabouços
- Preparar suspensão de célula de estoque
NOTA: Este protocolo delineará as etapas para a criação de quatro diferentes densidades de semeadura de células de neuroblastoma, com um fator de multiplicação de 2 entre cada densidade. Por conseguinte, será utilizada uma diluição em série para criar mais três suspensões de células a partir da suspensão de stock. O trabalho de cultura de células deve ser realizado em uma capela de fluxo laminar para manter a esterilidade.- Utilizar a equação (3) para calcular o volume de células necessário a partir do número total de células no balão multicamadas (contado no ponto 3.2.19) para preparar a primeira densidade de semeadura ou suspensão de existências celulares.
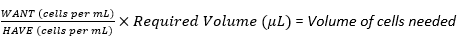 (3º)
(3º)
NOTA: Por exemplo, se a maior densidade de semeadura for de 6 × 105 células por andaime necessária para 30 andaimes, com cada andaime recebendo 20 μL de suspensão celular, a suspensão de células estoque exigiria 1,8 × 107 células (6 × 105 células × 30 andaimes) em um volume total de 600 μL (20 μL × 30 andaimes).
Como será realizada uma diluição seriada a partir dessa preparação, esses números deverão ser duplicados, ou seja, 3,6 × 107 células em um volume total de 1200 μL. Para converter isso em WANT em células por mL; dividir 3,6 × 10 7 por 1200 μL e multiplicar por 1000 μL, dando 3 × 107 células por mL.

- Adicionar o volume necessário de estoque celular a um tubo de centrífuga estéril de 2 mL ou 15 mL e levar a um volume final de 1200 μL com meio de crescimento. Rotule este tubo como Densidade 1 (Figura 3).
- Utilizar a equação (3) para calcular o volume de células necessário a partir do número total de células no balão multicamadas (contado no ponto 3.2.19) para preparar a primeira densidade de semeadura ou suspensão de existências celulares.
- Execute a diluição em série para criar várias suspensões de células de densidade de semeadura.
- A partir da densidade 1 preparada na etapa 4.1.1, preparar mais três densidades de suspensão celular através de diluição seriada, conforme Figura 3.
- Diluir cada densidade por um fator de 2 no meio de crescimento. Primeiro, adicione o volume final necessário (600 μL do exemplo anterior) de meio de crescimento a três tubos centrífugos estéreis de 2 mL ou 15 mL.
- Transfira metade da Densidade 1 (600 μL) para um dos tubos, misturando completamente a suspensão celular com o meio para diluir. Rotule este tubo como Densidade 2.
- Transfira metade da Densidade 2 (600 μL) para o próximo tubo, misturando completamente a suspensão da célula com o meio para diluir. Rotule este tubo como Densidade 3.
- Transfira metade da Densidade 3 (600 μL) para o tubo seguinte, misturando completamente a suspensão celular com o meio para diluir. Rotule este tubo como Densidade 4.
- Descarte 600 μL da Densidade 4 para que todas as quatro preparações tenham um volume final de 600 μL.
- Como controle negativo, adicionar 600 μL de meio de crescimento somente a um tubo centrífugo estéril de 2 mL. Consulte a Figura 3 para obter um esquema do processo de diluição seriada.
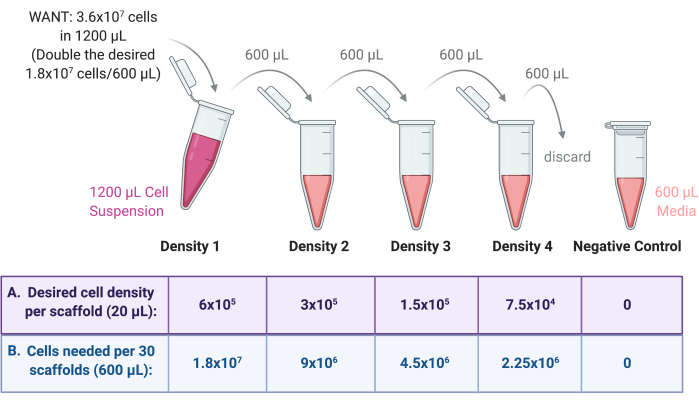
Figura 3: Diluição em série do estoque celular para preparar 4 suspensões para 4 densidades de semeadura de andaimes diferentes. (A) Os números podem ser ajustados de acordo com a densidade de semeadura desejada por andaime e (B) multiplicados pelo número total de andaimes por densidade, com cada andaime recebendo 20 μL de suspensão celular. Neste exemplo, a Densidade 1 requer 6 × 105 células por scaffold, o equivalente a 1,8 × 107 células em 600 μL para 30 scaffolds. Esse número é dobrado para iniciar a diluição seriada, pois 600 μL são então transferidos e diluídos em 600 μL de meio de crescimento no tubo seguinte. Este processo continua até que haja suspensões de 4 células com um fator de 2 entre cada uma. Um controle negativo é feito adicionando 600 μL de meio apenas a um tubo. Clique aqui para ver uma versão maior desta figura.
- Adição de suspensões de células aos andaimes
NOTA: Retire os andaimes (armazenados em PBS) do frigorífico e deixe-os chegar à temperatura ambiente (RT) antes de adicionar as células.- Traga os andaimes em PBS para a capela de fluxo laminar.
- Com pinças estéreis, colocar os andaimes em placas não aderentes de 24 poços com um andaime por poço (Figura 1C). Levante suavemente os andaimes pelos cantos e pressione-os levemente contra o lado do recipiente para remover o excesso de PBS. Adicione os andaimes no centro dos poços de lado para baixo (o lado brilhante da camada do andaime, virado para baixo nas placas de plástico de 24 poços).
- Rotule as placas de 24 poços com detalhes da linha celular, parâmetros relevantes (por exemplo, densidade de semeadura) e pontos de tempo. Trabalhar com uma densidade de semeadura de células por vez, mantendo as densidades restantes na incubadora de 37 °C até que estejam prontas para uso.
- Na capela de fluxo laminar, use uma pipeta P20 e pontas estéreis para adicionar suavemente 20 μL da suspensão celular relevante ao centro de cada andaime (Figura 1D). Mantenha as células completamente em suspensão, misturando bem enquanto adiciona as células aos andaimes. Certifique-se de que a suspensão permaneça em cima do andaime e não deslize para a base do poço, pois isso não permitirá a fixação da célula ao andaime.
- Uma vez que as células tenham sido adicionadas, incube as placas por 3-5 h (37 °C, 5% de CO2 e 95% de umidade) para permitir que a maioria das células se fixe.
- Após a incubação, adicionar lenta e suavemente 1 mL de meio de cultura pré-aquecido a cada poço (Figura 1E). Use uma pipeta P1000 para adicionar o meio para permitir um movimento mais lento e controlado, evitando o deslocamento dos andaimes. Se estiver trabalhando com um número muito alto de andaimes, use uma pipeta de 10 mL com a pistola de pipeta definida como 'drop' e 'low'.
- Incubar as placas de 24 poços durante a noite (37 °C, 5% CO2 e 95% de umidade).
- Manutenção de células nos andaimes
- Após as primeiras 24 h de fixação celular (Dia 1), transferir os arcabouços semeados para novas placas de 24 poços não aderentes e adicionar 1-2 mL de meio de crescimento fresco.
NOTA: Esta etapa remove as células que caíram no fundo das placas de plástico de 24 poços, em vez de permitir que elas cresçam nos andaimes. As réplicas de andaimes designadas como Dia 1 serão retiradas após 24 h, conforme discutido na seção 5; portanto, a manutenção não se aplica a esses andaimes. - Monitore os andaimes inicialmente a cada 2-3 dias para uma mudança na cor do meio de crescimento. À medida que o tempo avança e as células proliferam dentro dos arcabouços, alimentam-se com mais frequência.
- Com uma pistola de pipeta de 10 mL, no modo lento, retire o 1 mL do meio gasto e descarte. Se estiver realizando experimentos que exijam meio condicionado, coletar o meio gasto de réplicas biológicas em um tubo de centrífuga de 15 mL, centrifugar a 340 × g por 2 min para peletizar os detritos celulares, transferir o sobrenadante para um tubo fresco e armazenar a -80 °C.
- Adicione suavemente 2 mL de meio de crescimento pré-aquecido aos andaimes, usando a função de gotejamento novamente na pipeta, e devolva a placa de 24 poços à incubadora (37 °C, 5% CO2 e 95% de umidade). Repita sempre que o meio for gasto durante o período de crescimento desejado.
- Após as primeiras 24 h de fixação celular (Dia 1), transferir os arcabouços semeados para novas placas de 24 poços não aderentes e adicionar 1-2 mL de meio de crescimento fresco.
5. Recuperação de andaimes e aplicações
NOTA: Em cada ponto de tempo, várias aplicações podem ser usadas para monitorar o crescimento celular nos scaffolds ou avaliar perfis de expressão gênica e proteica. As condições de recuperação do andaime dependerão da análise a ser realizada, com múltiplos métodos de recuperação descritos nas subseções a seguir e demonstrados na Figura 4.
- Avaliação da viabilidade celular em scaffolds
- Esterilizar o reagente apropriado do ensaio de viabilidade celular filtrando através de um filtro estéril de 0,2 μm em um tubo de centrífuga na capela de fluxo laminar. Pré-aqueça esta solução estéril juntamente com meio de crescimento completo e PBS estéril em banho-maria a 37 °C.
- Na capela de fluxo laminar, use pinças estéreis para transferir os andaimes a serem analisados para uma placa fresca de 24 poços. Rotule a placa com todos os detalhes relevantes.
OBS: Realizar a análise em triplicata. - Adicionar 900 μL do meio de crescimento pré-aquecido a cada poço, seguido de 100 μL do reagente de viabilidade celular estéril. Incluir um controle negativo adicionando 900 μL de meio e 100 μL do reagente de viabilidade celular estéril a um poço sem arcabouço. Substitua a tampa da placa e agite suavemente a placa por ~3 min para distribuir uniformemente o reagente de viabilidade celular diluída por todo o poço. Incubar a placa a 37 °C, 5% CO2 e 95% de umidade.
NOTA: Os tempos de incubação precisarão ser otimizados para cada linhagem celular; Consulte as diretrizes do fabricante. Para linhagens celulares de neuroblastoma, a incubação de 4-6 h parece ideal. - Após a incubação, retire a placa da incubadora e agite-a suavemente por alguns segundos.
- Na capela de fluxo laminar, abra uma nova placa translúcida de 96 poços. De cada poço na placa de 24 poços, transferir o meio incubado e o reagente para três poços da placa de 96 poços com 100 μL por poço, dando triplicatas técnicas.
NOTA: Esta transferência deixará 700 μL nos poços da placa de 24 poços. - Cubra a placa de 96 poços em folha de alumínio para proteger o reagente de viabilidade celular da luz.
- Remova e elimine os restantes 700 μL do conteúdo do poço de cada andaime na placa de 24 poços. Lave cada andaime duas vezes com 1 mL de PBS estéril.
NOTA: Todas as cores não serão removidas dos andaimes. Estes arcabouços podem então ser utilizados para outras aplicações, por exemplo, quantificação de ADN, colocando-os em 1 ml de Triton X-100 a 1% em solução de bicarbonato de sódio 0,1 M (NaHCO3) e armazenando-os a -80 °C (ver secção 5.2, Figura 4B). - Remova a placa de 96 poços da capela de fluxo laminar e meça a absorbância de cada poço nos comprimentos de onda de 570 nm e 600 nm usando um leitor de microplacas. Registar os valores de absorbância em ambos os comprimentos de onda e seguir as instruções do fabricante para calcular a redução percentual do reagente de viabilidade celular pelas células.
- Analisar graficamente e estatisticamente os resultados de viabilidade celular utilizando software apropriado. Valores de triplicata biológica de entrada para produzir barras de erro e indicar a variabilidade do ensaio.
- Para examinar as mudanças na viabilidade celular ao longo do período experimental, realize um teste de análise de variância (ANOVA) one-way com comparações múltiplas das médias usando software bioestatístico apropriado.
- Indicam diferenças significativas entre os momentos nos gráficos como ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) e **** (P≤0,0001).
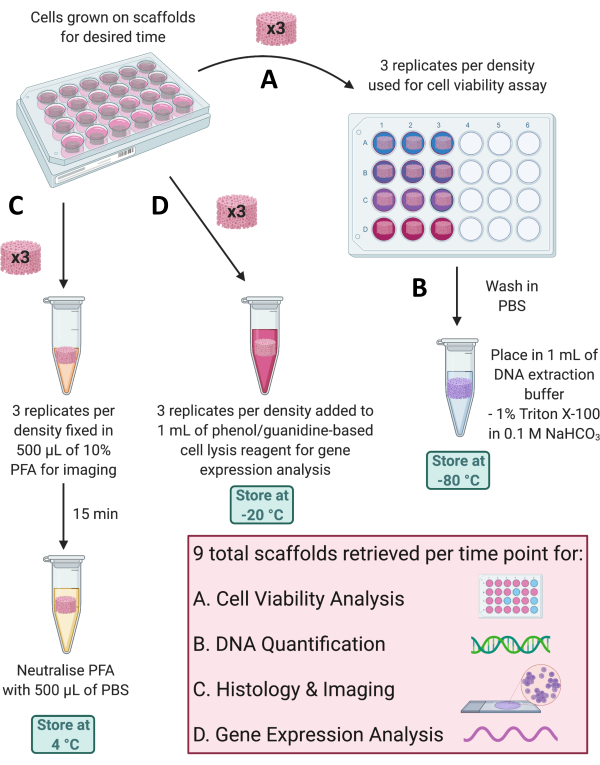
Figura 4: Recuperação de scaffolds para diferentes análises em cada ponto de tempo. (A) Três réplicas de andaimes são recuperadas para análise da viabilidade celular. (B) Esses scaffolds podem então ser lavados em PBS, colocados em Triton X-100 a 1% em NaHCO3 0,1 M e armazenados a -80 °C para quantificação de DNA. (C) Mais três réplicas são fixadas em PFA a 10% por 15 min, neutralizadas em PBS e armazenadas a 4 °C para coloração histológica e exames de imagem. (D) Finalmente, 3 réplicas são adicionadas a um reagente de lise celular à base de fenol/guanidina e armazenadas a -20 °C para análise da expressão gênica. Abreviações: PBS = solução salina tamponada com fosfato; PFA = paraformaldeído. Clique aqui para ver uma versão maior desta figura.
- Quantificação do DNA das células dos scaffolds
NOTA: Tal como referido na nota após o passo 5.1.7, a recuperação dos suportes para quantificação do ADN envolve a colocação dos suportes em tubos de centrifugação de 2 ml contendo 1 ml de Triton X-100 a 1% em solução de NaHCO3 0,1 M, seguida de armazenamento a -80 °C. Antes que a análise de DNA possa ser realizada, as células devem passar por três ciclos de congelamento-descongelamento para lisar as células do neuroblastoma adequadamente e liberar DNA para quantificação.- Retirar as amostras previamente armazenadas em Triton X-100 a partir de -80 °C. Deixe as amostras em TR por 1-3 h ou até descongelar.
- Vórtice as amostras por 10-20 s e coloque as amostras de volta a -80 °C por 18-24 h ou até congelar completamente. Repita este processo para um total de três ciclos de congelamento-descongelamento.
- Para maximizar o rendimento de DNA, use um lisador de tecido para interromper as células nos scaffolds.
- Coloque um grânulo metálico no tubo centrífugo de 2 mL contendo um andaime em Triton X-100 e coloque o tubo dentro do adaptador para agitar a amostra a 50 oscilações/segundo por 2-3 min.
NOTA: Use tubos de centrífuga de fundo redondo, pois o talão de metal pode ficar alojado em um tubo de fundo cônico.
- Coloque um grânulo metálico no tubo centrífugo de 2 mL contendo um andaime em Triton X-100 e coloque o tubo dentro do adaptador para agitar a amostra a 50 oscilações/segundo por 2-3 min.
- Quantificar o DNA na solução Triton X-100 aplicando uma coloração fluorescente de DNA de fita dupla (dsDNA) e medir a emissão usando um leitor de microplacas. Consulte as orientações do fabricante; diluir adequadamente as amostras em tampão de ácido tetracético (TE) de tris-etilenodiamina para preparar 8 padrões de dsDNA através de diluição seriada em tampão TE (Figura 5).
- Em placas pretas opacas de 96 poços, adicionar 100 μL de cada padrão ou amostra aos poços em triplicata técnica.
- Diluir a coloração fluorescente de dsDNA 200 vezes em tampão TE e adicionar 100 μL a cada padrão/amostra usando uma pipeta multicanal. Cubra a placa em folha de flandres e incube em RT por 5 min.
- Medir e registrar a fluorescência de cada poço a 520 nm. Siga as orientações do fabricante para calcular a concentração de DNA em cada amostra.
NOTA: Se a concentração média de ADN por célula for conhecida para a linhagem celular utilizada, os valores de concentração de ADN podem ser convertidos em números celulares utilizando a equação (4). (4º)
(4º) - Grafar e analisar estatisticamente os resultados da quantificação do DNA utilizando software apropriado. Valores de triplicata biológica de entrada para produzir barras de erro e indicar a variabilidade do ensaio.
- Para examinar as mudanças na concentração de DNA/número de células ao longo do período experimental, realize um teste ANOVA unidirecional com comparações múltiplas das médias usando software bioestatístico apropriado.
- Indicam diferenças significativas entre os momentos nos gráficos como ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) e **** (P≤0,0001).
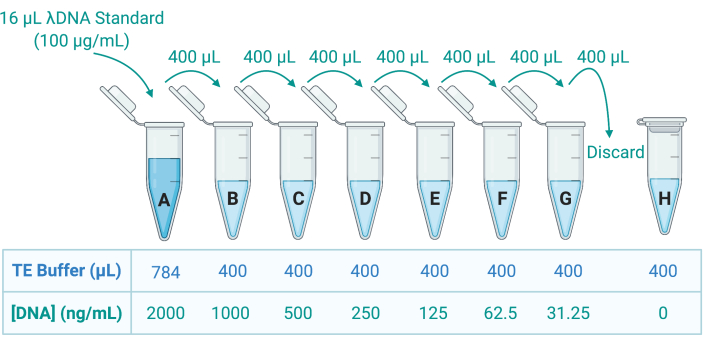
Figura 5: Elaboração de oito padrões de DNA para a geração de uma curva padrão. Uma solução-mãe de λDNA é fornecida a 100 μg/mL. Este é diluído 50 vezes em tampão TE para criar o padrão A a 2000 ng/mL; 400 μL de A são então transferidos para o tubo B, contendo 400 μL de tampão TE; 400 μL de B é então transferido e diluído 2 vezes em C, e assim por diante até G. O padrão H é composto apenas de tampão TE e, portanto, tem uma concentração de DNA de 0 ng/mL. Abreviação: TE = Tris-EDTA. Clique aqui para ver uma versão maior desta figura.
- Preparo de scaffolds para coloração histológica
NOTA: Os scaffolds podem ser fixados e corados para fins de microscopia e imagem como scaffolds inteiros para imunofluorescência (IF) ou como cortes fixados em formalina e embebidos em parafina (FFPE) para coloração histológica ou imunohistoquímica (IHQ). Isso permite a avaliação qualitativa da penetração e distribuição celular dentro de scaffolds e pode ser usado para avaliar a expressão de proteínas.- Preparar uma solução de paraformaldeído (PFA) a 10% em PBS. Certifique-se de que existe solução suficiente para um volume final de 500 μL por andaime.
- Pré-aquecer esta solução a 37 °C e adicionar 500 μL a tubos de centrífuga marcados de 2 ml para recuperação de andaimes.
- Retire os andaimes das placas não aderentes de 24 poços (passo 4.4.4) e coloque-os no capô de fluxo laminar.
- Usando pinças estéreis, transfira os suportes para os tubos de centrífuga marcados contendo 10% de PFA. Deixe os andaimes fixarem na solução de PFA por 15 min. Neutralizar o PFA adicionando 500 μL de PBS a cada tubo e armazenar a 4 °C.
- Para preparar os andaimes para o Processador Automático de Tecidos, retire-os de 4 °C e use uma pinça para colocá-los em de plástico rotulados com todos os detalhes relevantes a lápis. Coloque todas as no recipiente metálico do Processador de Tecidos.
- Inicie o protocolo de 12 estágios no processador de tecidos para fixar, desidratar, limpar os andaimes e infiltrá-los com parafina durante a noite. Recolher as contendo as amostras processadas.
- Em seguida, incorpore os scaffolds em blocos de cera de parafina para permitir a microtomia dos scaffolds em fatias muito finas para coloração.
NOTA: É especialmente importante considerar a orientação dos andaimes ao removê-los dos e incorporá-los em cera, pois isso afetará o ângulo em que as imagens são tiradas. Isso é importante ao avaliar a infiltração celular nos scaffolds. - Ligue o embutidor de cera e a placa fria; Levante a tampa para verificar o nível da cera. Reabasteça, se necessário.
- Trabalhando com uma amostra de cada vez, abra o, retire o andaime e centralize-o no molde plástico.
- Despeje cera quente sobre a amostra, garantindo que a orientação correta seja mantida e ajustando com pinças quentes, se necessário, antes que a cera se solidifique. Despeje mais cera para preencher o molde.
- Coloque a tampa do etiquetado por cima do molde de plástico e adicione cera por cima. Coloque o molde sobre a placa fria para solidificar a cera. Conservar durante a noite a 4 °C para garantir que a cera de parafina está totalmente solidificada antes da microtomia.
- Para se preparar para a microtomia, ligue um banho-maria a 35 °C, a placa de secagem e o micrótomo.
- Insira uma lâmina no suporte e aperte a alavanca para prendê-la.
- Ajuste a espessura do caimento e da seção, geralmente 5 mm para seções de andaime.
- Retire o andaime FFPE do molde, fixe-o no suporte na parte frontal do micrótomo e corte cuidadosamente o excesso de cera ao redor das bordas da amostra antes de cortar as seções.
- Comece a cortar no bloco de cera girando a alavanca do micrótomo, garantindo um movimento suave.
- Colete seções em forma de fita, cerca de 3 seções de andaime de cada vez, e coloque-as delicadamente no banho-maria a 35 °C para remover rugas. Separe suavemente as seções enquanto estiver em banho-maria usando uma pinça.
- Usando lâminas de microscópio de vidro revestidas de polisina, levante cada seção do banho-maria para que a seção fique no centro da lâmina. Rotule cada slide com um lápis.
- Coloque as lâminas de vidro na placa de secagem ou num forno de secagem a 60 °C. Depois de secos, armazená-los a 4 °C e proceder com a coloração histológica ou IHQ necessária.
- Recuperação de scaffolds para análise da expressão gênica
- Retirar os andaimes das placas não aderentes de 24 poços da incubadora (passo 4.4.4) e colocá-los na capela de fluxo laminar.
- Usando pinças estéreis, transfira os andaimes para tubos de centrífuga de 2 mL recém-marcados.
- Em um exaustor, adicione 1 mL de um reagente de lise celular à base de fenol/guanidina a cada tubo para lisar as células em arcabouços e permitir a recuperação de RNA de alta qualidade.
- Conservar os tubos a -20 °C até que estejam prontos para efectuar a extracção de ARN utilizando um kit apropriado. Usando uma reação em cadeia da polimerase quantitativa de transcrição reversa (RT-qPCR)17, avalie a expressão gênica nas células dos scaffolds.
Resultados
O modelo de arcabouço à base de colágeno descrito aqui tem muitas aplicações, desde o estudo da biologia do neuroblastoma até o rastreamento da terapêutica anticâncer em um ambiente fisiologicamente mais semelhante aos tumores nativos do que a cultura de células 2D convencional. Antes de testar uma determinada questão de pesquisa, é crucial obter uma caracterização completa da adesão, proliferação e infiltração celular dentro do prazo experimental desejado. As condições de crescimento dependerão da biologia de cada linhagem celular específica. É importante ressaltar que vários métodos de avaliação do crescimento celular devem ser implementados para determinar condições ótimas e desempenho robusto.
Aqui, a viabilidade de células de neuroblastoma cultivadas em scaffolds foi avaliada usando um ensaio colorimétrico de viabilidade celular. Este ensaio pode ser realizado com a frequência desejada durante todo o período experimental. Para o experimento descrito, a avaliação da viabilidade celular foi realizada nos dias 1, 7 e 14 para duas linhagens celulares de neuroblastoma, KellyLuc e IMR32, cultivadas em scaffolds Coll-I-nHA em 4 densidades diferentes (Figura 6). A viabilidade no Dia 1 foi definida como linha de base para comparar todas as medidas subsequentes. A taxa de redução do reagente de viabilidade celular é reflexo da biologia celular e das características de crescimento de linhagens celulares individuais, incluindo suas taxas de proliferação e metabolismo. Uma correlação entre o número de células semeadas nos scaffolds e o nível de redução era esperada. Neste experimento, a redução do reagente de viabilidade celular geralmente aumentou a cada momento para ambas as linhagens celulares em todas as densidades, como esperado.
Cada densidade foi então avaliada individualmente para ambas as linhagens celulares para comparar a redução ao longo dos tempos. ANOVA one-way com teste de comparações múltiplas de Tukey foi realizada para detectar diferenças significativas na redução entre os momentos (Figura 7). Para ambas as linhagens celulares e todas as densidades de semeadura, houve um aumento significativo (P<0,05) na redução do reagente de viabilidade celular quando comparados o dia 1 e o dia 14. Isso indicou um aumento significativo de células metabolicamente ativas presentes nos scaffolds. Esse aumento não foi significativo em todos os casos quando avaliados os intervalos de 7 dias (dia 1 vs. dia 7, dia 7 vs. dia 14), demonstrando a importância da otimização da densidade de semeadura para atingir a janela de crescimento desejada.
Para apoiar os resultados do ensaio de viabilidade celular, o crescimento celular em scaffolds também pode ser medido indiretamente através da quantificação de dsDNA extraído de scaffolds usando uma coloração fluorescente de dsDNA (Figura 8A). Assim como a viabilidade celular, a quantificação do DNA pode ser feita com a frequência desejada dentro da linha do tempo experimental. No entanto, essa análise requer a recuperação completa dos arcabouços e o término do crescimento celular e, portanto, deve ser levada em consideração no planejamento experimental, como discutido na seção 1. Para este experimento, o DNA foi quantificado nos dias 1, 7 e 14 para duas linhagens celulares de neuroblastoma, KellyLuc e IMR32, cultivadas em scaffolds Coll-I-nHA em 4 densidades diferentes. Como a concentração média de dsDNA por célula é conhecida para essas linhagens, foi possível derivar o número de células por amostra a partir do DNA quantificado (Figura 8B).
A quantificação de DNA deu origem a uma maior variabilidade entre as réplicas biológicas do que a avaliação da viabilidade celular, mas geralmente aumentou para cada ponto de tempo, com os níveis mais altos quantificados no dia 14. As células IMR32 parecem atingir maior número de células em scaffolds de Coll-I-nHA, como indicado pela concentração de DNA, do que as células KellyLuc. Cada densidade foi então avaliada individualmente para as duas linhagens celulares para comparar a redução ao longo dos tempos. ANOVA one-way com teste de comparações múltiplas de Tukey foi realizada para detectar diferenças significativas na redução entre os momentos (Figura 8B).
Para ambas as linhagens e todas as densidades de semeadura, houve um aumento significativo (P<0,05) no número de células quando comparado o dia 1 e o dia 14, com exceção de KellyLuc na densidade de semeadura 4 (1 × 105 células/scaffold), que não produziu aumentos significativos em nenhum dos momentos. Semelhante aos resultados de viabilidade celular, os aumentos não foram significativos em todos os casos quando avaliados os intervalos de 7 dias (dia 1 vs. dia 7, dia 7 vs. dia 14). Ao comparar as tendências de tempo para viabilidade celular e quantificação de DNA, houve algumas pequenas diferenças entre as duas análises. No entanto, tendências gerais semelhantes foram observadas, com valores médios aumentando entre intervalos de 7 dias para a maioria das densidades. Isso demonstra a importância do monitoramento do crescimento celular por meio de mais de um método.
Uma avaliação visual da morfologia e distribuição do crescimento celular nos scaffolds foi implementada em seguida, englobando as colorações tradicionais de hematoxilina e eosina (H&E), bem como IHQ. Espera-se que os diferentes padrões de crescimento de linhagens celulares individuais levem a arranjos espaciais variados em scaffolds, incluindo diferentes graus de penetração no arcabouço e agrupamento celular. Os scaffolds foram fixados em formalina, embebidos em parafina e cortados em cortes de 5 mm (Figura 9A), preparando-os para múltiplas técnicas de visualização, incluindo coloração histológica e IHQ.
A coloração H&E de rotina foi aplicada em células Kelly, KellyCis83 e IMR32 cultivadas em arcabouços à base de colágeno nos dias 1, 7 e 14 (Figura 9B). Isso permitiu a visualização da orientação espacial das células em dois arcabouços à base de colágeno durante um período de 14 dias. Células Kelly sensíveis à cisplatina e células KellyCis83 resistentes foram cultivadas em ambos os scaffolds Coll-I-nHA (Figura 9B, i) e Coll-I-GAG (Figura 9B, ii). Consistente com dados publicados anteriormente, as células KellyCis83 cresceram a uma taxa mais alta e se infiltraram mais profundamente em ambas as composições de andaimes do que a linhagem celular Kelly menos invasiva. A coloração H&E de outra linhagem celular de neuroblastoma, IMR32, cultivada em Coll-I-nHA demonstra um padrão de crescimento contrastante (Figura 9B, iii). Essa linhagem celular cresceu em grandes aglomerados densamente compactados nos arcabouços de colágeno durante o período de 14 dias. A microscopia confocal de campo claro pode ser usada para visualizar a arquitetura porosa dos arcabouços à base de colágeno (Figura 9C) devido à autofluorescência das fibras colágenas.
Nós coramos células com faloidina visando a actina citoesquelética e a contracoloração nuclear, 4′,6-diamidino-2-fenilindol (DAPI), para monitorar características celulares específicas ao longo da linha do tempo experimental. Uma abundância de actina foi observada em células Kelly e KellyCis83 em scaffolds Coll-I-GAG usando esta técnica (Figura 9D). Estes resultados demonstram como múltiplas técnicas de imagem podem ser usadas para derivar informações espacialmente resolvidas de células de neuroblastoma cultivadas em scaffolds usando este protocolo. Esta caracterização dos padrões de crescimento celular em arcabouços à base de colágeno durante um determinado período melhorará a compreensão e interpretação de quaisquer ensaios bioquímicos a jusante.
A expressão de proteínas por células cultivadas em arcabouços à base de colágeno pode ser analisada para comparar a atividade celular com cenários in vivo . Dados publicados anteriormente examinaram a expressão da cromogranina A (CgA) como marcador secretado substituto de neuroblastoma por células KellyLuc e KellyCis83Luc cultivadas em monocamadas celulares, bem como em scaffolds Coll-I-nHA e Coll-I-GAG (Figura 10). A CgA foi avaliada no meio condicionado por meio de ensaio imunoenzimático (ELISA) (Figura 10A). A CgA é secretada em uma taxa maior na linhagem celular KellyCis83, mais agressiva e quimio-resistente, do que na linhagem Kelly (Figura 10B,C). Isso foi significativo no dia 7 em ambos os scaffolds Coll-I-GAG e Coll-I-nHA (P<0,05), enquanto não houve diferença significativa neste momento para células cultivadas como monocamada por cultura 2D convencional.
Esses resultados também destacam o tempo restrito do crescimento de células em monocamada, com apenas 7 dias de crescimento se mostrando viável antes que as células atinjam a confluência. O crescimento de células em arcabouços supera essa limitação, pois elas podem ser mantidas por um período mais longo em condições fisiologicamente mais relevantes. A combinação de técnicas acima para adquirir informações sobre viabilidade celular, conteúdo de DNA, morfologia celular e arranjo espacial, e perfis de expressão facilita a avaliação do crescimento de células de neuroblastoma em uma variedade de arcabouços à base de colágeno. Este protocolo também pode ser facilmente adaptado para satisfazer requisitos experimentais específicos e aplicações desejadas.
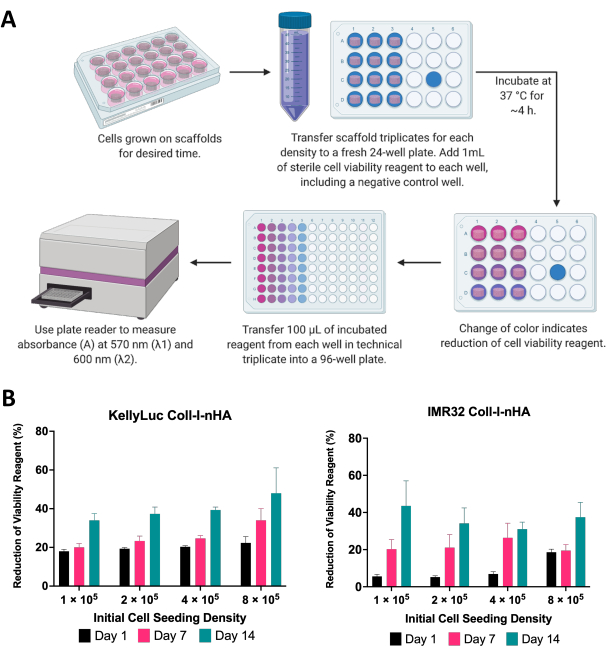
Figura 6: Análise de viabilidade celular. (A) Procedimento geral para medir a viabilidade de células de neuroblastoma em arcabouços à base de colágeno usando um ensaio colorimétrico de viabilidade celular. O período de incubação deve ser otimizado para cada nova linhagem celular, seguindo as orientações do fabricante. (B) Redução percentual do reagente de viabilidade celular por células KellyLuc e IMR32 cultivadas em scaffolds Coll-I-nHA em quatro diferentes densidades iniciais de semeadura, medidas nos dias 1, 7 e 14. As amostras foram avaliadas em triplicata biológica com barras de erro representando o desvio padrão. Abreviações: nHA = nanohidroxiapatita; Coll-I-nHA = arcabouços de colágeno suplementados com nHA. Clique aqui para ver uma versão maior desta figura.
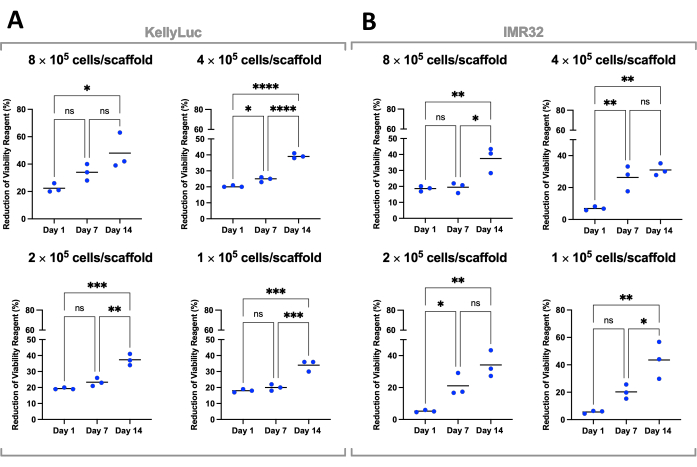
Figura 7: Viabilidade celular por densidade de semeadura para células cultivadas em Coll-I-nHA durante um período de 14 dias. (A) KellyLuc; (B) IMR32. Os números de célula intitulados referem-se à densidade inicial de semeadura de células nos andaimes no Dia 0. As amostras foram avaliadas em triplicata biológica, indicada por pontos triplicata, com barras representando a média. A ANOVA One-Way com Comparações Múltiplas foi utilizada para detectar diferenças significativas na % de redução do reagente de viabilidade celular ao longo dos três momentos, observadas nos gráficos (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abreviações: nHA = nanohidroxiapatita; Coll-I-nHA = arcabouços de colágeno suplementados com nHA; ANOVA = análise de variância; ns = não significante. Clique aqui para ver uma versão maior desta figura.
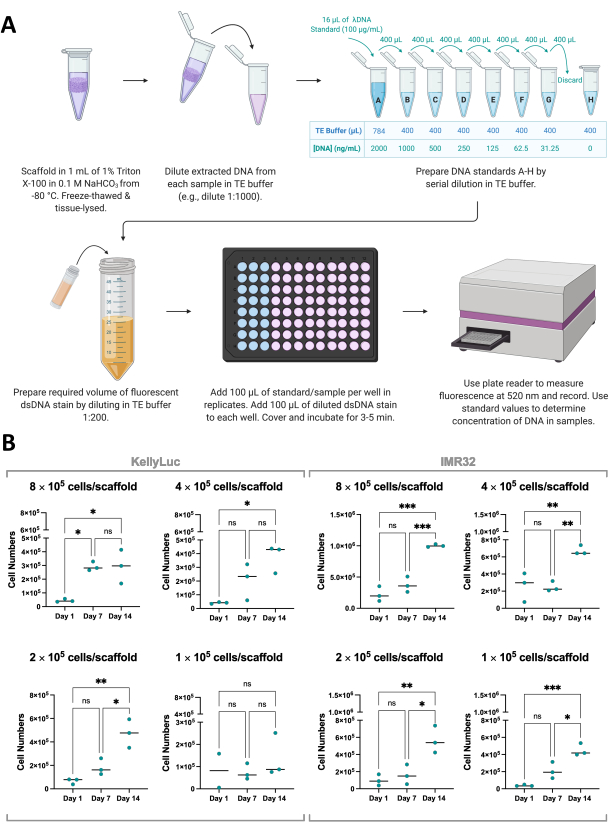
Figura 8: Quantificação do DNA extraído das células em scaffolds . (A) Processo de quantificação de dsDNA de células cultivadas em arcabouços à base de colágeno utilizando coloração fluorescente de dsDNA. (B) Números celulares da análise de quantificação de DNA por densidade de semeadura para células KellyLuc e IMR32 cultivadas em Coll-I-nHA durante um período de 14 dias. Os números de célula intitulados referem-se à densidade inicial de semeadura de células em andaimes no Dia 0. As amostras foram avaliadas em triplicata biológica, indicada por pontos triplicata, com barras representando a média. A ANOVA One-Way com Comparações Múltiplas foi utilizada para detectar diferenças significativas no número de células ao longo dos três momentos, observadas nos gráficos (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abreviações: nHA = nanohidroxiapatita; Coll-I-nHA = arcabouços de colágeno suplementados com nHA; dsDNA = DNA de fita dupla; TE = Tris-EDTA; ANOVA = análise de variância; ns = não significante. Clique aqui para ver uma versão maior desta figura.
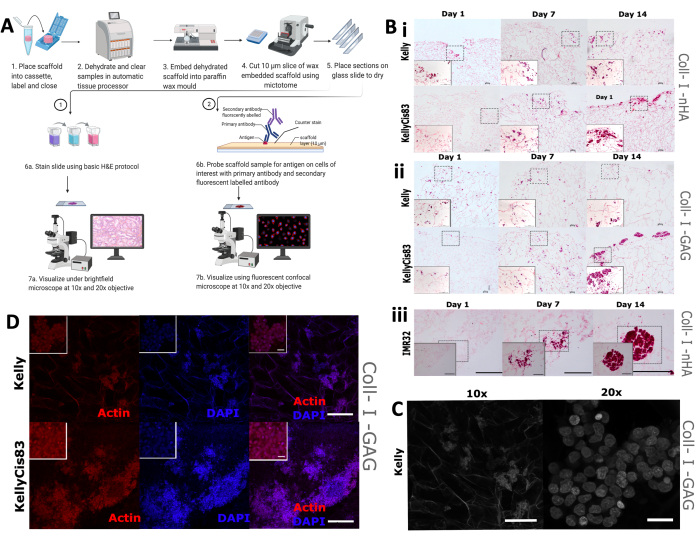
Figura 9: Etapas do processamento tecidual para análise imunoistoquímica dos scaffolds. (A) Representação esquemática do protocolo de processamento de scaffolds para análise de imagens. Esse processo permite a coloração histológica de rotina e a sondagem de anticorpos específicos usando anticorpos primários e anticorpos secundários marcados fluorescentemente. (B) Imagens representativas de três linhagens celulares de neuroblastoma submetidas à coloração H&E. As imagens H&E são tiradas nos dias 1, 7, 14 para monitorar os padrões de crescimento ao longo do tempo do experimento. Barra de escala = 200 μm. Os quadrados tracejados representam a área escolhida para imagens ampliadas em 20x na borda inferior esquerda. Barra de escala = 20 μm. (i e ii) H&E de linhagens celulares de neuroblastoma Kelly e KellyCis83 (painéis superior e inferior, respectivamente) em dois tipos de arcabouços à base de colágeno. (iii) H&E da linhagem celular IMR32, representando o crescimento celular agrupado no arcabouço Coll-I-nHA. (C) Imagem representativa da linhagem celular de Kelly, submetida à microscopia confocal de campo claro. A autofluorescência do colágeno permite a visualização do arcabouço poroso. 10x Scale bar = 200 μm, 20x scale bar = 20 μm. (D) Imagem representativa dos scaffolds embutidos seguida de análise por IHQ com faloidina e DAPI em aumento de 10x, barra de escala = 200 μm. Quadrados internos menores representam imagens ampliadas (20x), barra de escala = 20 μm. Abreviações: nHA = nanohidroxiapatita; Coll-I-nHA = arcabouços de colágeno suplementados com nHA; GAG = glicosaminoglicano; Coll-I-GAG = arcabouços de colágeno suplementados com condroitina-6-sulfato; H&E = hematoxilina e eosina; IHQ = imuno-histoquímica; DAPI = 4′,6-diamidino-2-fenilindol. Clique aqui para ver uma versão maior desta figura.
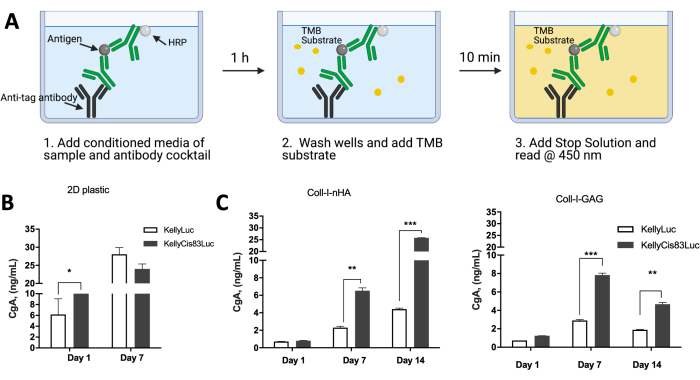
Figura 10: Expressão de proteínas por células de neuroblastoma cultivadas em arcabouços 3D à base de colágeno em comparação com o plástico 2D. (A) Um esquema de como o ELISA CgA foi realizado em meios condicionados de células cultivadas em scaffolds à base de colágeno 2D ou plástico. (B) Níveis de expressão da proteína CgA obtidos de meios condicionados de células cultivadas em monocamada plástica 2D. Como as células atingiram confluência após 7 dias, o tempo de 14 dias não foi legível. No dia 7 no plástico, não houve diferença significativa nos níveis de CgA entre as linhagens celulares Kelly e KellyCis83. (C) CgA ELISA realizada utilizando meios condicionados de células cultivadas em arcabouços à base de colágeno por 14 dias consecutivos. No dia 7, em ambos os arcabouços de colágeno, os níveis de CgA são mais elevados na linhagem celular KellyCis83, mais agressiva, destacando níveis mais relevantes fisiológicos de CgA na matriz 3D em comparação com a monocamada 2D. Esse valor foi modificado de Curtin et al.17. Abreviações: 3D = tridimensional; 2D = bidimensional; CgA = cromogranina A; ELISA = ensaio imunoenzimático; nHA = nanohidroxiapatita; Coll-I-nHA = arcabouços de colágeno suplementados com nHA; GAG = glicosaminoglicano; Coll-I-GAG = arcabouços de colágeno suplementados com condroitina-6-sulfato; TMB = 3,3',5,5'-tetrametilbenzidina; HRP = peroxidase de raiz forte. Clique aqui para ver uma versão maior desta figura.
Discussão
O modelo de células de câncer em arcabouço 3D provou ser uma ferramenta valiosa e versátil para obter informações mecanicistas sobre o crescimento, viabilidade e infiltração de células de neuroblastoma em uma TME simplificada32. O modelo de neuroblastoma 3D descrito aqui mimetiza a ETM mínima e fornece dados fisiologicamente mais relevantes do que uma cultura de monocamada 2D. Uma grande desvantagem da cultura de células 3D é o aumento da complexidade experimental e prazos mais longos. Descrito aqui é um protocolo otimizado para semeadura, crescimento e manutenção de células de neuroblastoma em arcabouços à base de colágeno, seguido por análises e aplicações a jusante, produzindo uma caracterização robusta do crescimento celular. Nosso objetivo foi obter informações sobre a densidade ideal de semeadura celular para os scaffolds para criar um ambiente previsível e controlável para avaliar tratamentos de drogas anticâncer em uma janela experimental rápida de 14 dias. A combinação de todos esses protocolos simples descritos fornece uma avaliação completa do crescimento celular de neuroblastoma no sistema de cultura in vitro baseado em scaffold.
Os pontos críticos na configuração do protocolo foram enfatizados para permitir que os cientistas estabeleçam o mesmo em seus laboratórios rapidamente. Por exemplo, os tempos de incubação indicados para melhor desempenho do ensaio de viabilidade celular colorimétrica permitem uma penetração mais profunda do reagente nos poros do arcabouço para atingir todas as células. Além disso, a técnica de coloração fluorescente de dsDNA é robusta e direta; no entanto, a liberação de DNA dos scaffolds requer lise celular vigorosa, pois as células estão "presas" dentro das fibras de colágeno.
Usando o ensaio simples de quantificação de DNA descrito, podemos identificar a fase de crescimento logarítmica em scaffolds à base de colágeno para rastreamento de drogas anticâncer usando este modelo. No cenário experimental descrito, foram utilizadas 4 densidades iniciais de semeadura celular com um período total de 14 dias e pontos de tempo de análise nos Dias 1, 7 e 14. Identificamos que as células KellyLuc semeadas em 4 × 105 células/arcabouço têm a janela de proliferação mais significativamente ativa entre os dias 7 e 14. Estes dados de crescimento em fase logarítmica permitirão uma interpretação confiável de vários experimentos de citotoxicidade celular. Ele elimina a especulação sobre qualquer declínio no crescimento ou morte celular resultante do crescimento suprimido na plataforma porosa 3D, em vez de toxicidades de drogas. A viabilidade celular também é uma avaliação amplamente utilizada para a adequação de plataformas 3D para suportar o crescimento de diferentes tipos celulares33,34. Embora existam muitos ensaios para medir a viabilidade celular, incluindo coloração viva/morta, medição de ATP, ensaios de proliferação, descobrimos que o uso do ensaio de viabilidade celular colorimétrica Alamar Blue é uma técnica simples e eficaz para apoiar dados de quantificação de DNA.
O uso combinado da quantificação do DNA e da viabilidade celular forneceu evidências complementares de que, em média, a densidade ideal para semear células no arcabouço para alcançar o crescimento contínuo durante um período de 14 dias é de 2-4 × 105 células/arcabouço. No entanto, este protocolo pode ser facilmente adaptado para satisfazer diferentes períodos de tempo experimentais, pontos de tempo de análise e aplicações a jusante. Embora este protocolo descreva a avaliação do crescimento celular em monocultura de células de neuroblastoma em scaffolds, os scaffolds são facilmente alteráveis para uso como plataforma para co-cultura, descrito por do Amaral et al., que utilizaram scaffolds de colágeno-GAG para co-cultura de queratinócitos e fibroblastos em uma investigação de cicatrização deferidas35.
O modelo 3D descrito permite a visualização do crescimento e infiltração celular utilizando diferentes técnicas bem conhecidas, como a imunofluorescência e o padrão H&E. É importante visualizar as células juntamente com a caracterização do crescimento usando ensaios bioquímicos devido à diversidade de morfologia celular e padrões de crescimento em scaffolds. Compreender o padrão de crescimento pode produzir insights sobre o comportamento de crescimento e a resposta futura a drogas anticâncer. Por exemplo, o crescimento de IMR32 usando quantificação de DNA produz padrões semelhantes aos de Kelly, embora na visualização usando H&E, IMR32 cresça em grupos maiores do que Kelly, que apresentou crescimento mais disperso (Figura 9). Esses variados padrões de crescimento de linhagens celulares em scaffolds refletem o cenário clínico de heterogeneidade tumoral. Examinar a resposta a drogas anticâncer usando um painel de linhagens celulares com diferentes morfologias em scaffolds 3D aumentará o valor preditivo para a resposta do paciente às mesmas drogas.
A detecção da expressão gênica ou proteica também pode ser realizada usando outras abordagens, como RT-qPCR ou ELISA, se a proteína de interesse for secretada. Um marcador substituto da progressão do neuroblastoma, a cromogranina A (CgA)36, foi usado para caracterizar adicionalmente o crescimento celular do neuroblastoma em 3D. Como descrito em trabalho anterior17, a secreção de CgA aumentou à medida que as células proliferavam (Figura 10). Enquanto a cultura de células monocamadas não conseguiu captar esse aumento, pois a proliferação significou que as células atingiram plena confluência nas placas de cultura, o uso dos scaffolds de colágeno 3D permitiu uma avaliação prolongada da secreção de CgA.
Este modelo 3D in vitro pode não ser adequado para todas as questões de pesquisa para estudar a biologia do neuroblastoma e a resposta à terapêutica. Uma das limitações é a penetração desigual de células dentro de arcabouços e a formação de aglomerados celulares de tamanhos variados, que dependem de uma determinada linhagem celular e podem levar à difusão incontrolável de nutrientes e fármacos. Essa característica afeta a robustez no rastreamento terapêutico. No entanto, apesar dessa limitação, é importante considerar que os tumores nativos também são heterogêneos em tamanho e distribuição de células cancerígenas e contêm muitos outros tipos celulares dentro do tecido tumoral. Para superar essa limitação, propomos o uso de cada arcabouço preenchido como um único microtecido, para o qual os seguintes parâmetros serão otimizados: (a) tempos de incubação para que o reagente de viabilidade celular atinja as células e os aglomerados celulares, e (b) lise das células em tampão Triton X-100 por pré-processamento das células em scaffolds com um lisador tecidual para liberar o DNA das células contidas profundamente no scaffold.
Outra limitação técnica deste protocolo é a falta de ensaios mecânicos de cada lote de andaimes recém-fabricados para este modelo. Entretanto, a utilização do robusto processo de fabricação dos scaffolds, que tem sido extensivamente caracterizado em relação às propriedades físicas e químicas dos scaffolds, como módulo de compressão e tração, porosidade e estrutura visual dos poros, e homogeneidade, garante que as qualidades dos andaimes sejam mantidas através de lotes 21,24,27,30,37.
Em resumo, este trabalho apresenta uma série de métodos simples para a análise do crescimento celular em arcabouços à base de colágeno. Tanto a linha do tempo experimental quanto os pontos de análise podem ser intercambiados dependendo das questões específicas de pesquisa. Este protocolo também é adaptável a outros tipos celulares. Os resultados mostrados acima fornecem evidências sobre como esta compilação de métodos forneceu informações sobre a densidade de semeadura ideal para várias linhagens celulares de neuroblastoma para criar crescimento contínuo ao longo de 14 dias. A fusão dos resultados obtidos de todos os métodos deste protocolo proporciona uma compreensão superior do crescimento celular dentro da matriz de colágeno 3D. A utilização futura deste modelo provavelmente envolverá sistemas de co-cultura específicos para o neuroblastoma TME e o teste de várias novas drogas anticâncer.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pelo National Children's Research Centre (NCRC), Irish Research Council (IRC) e Neuroblastoma UK. As ilustrações foram criadas utilizando o BioRender.
Materiais
| Name | Company | Catalog Number | Comments |
| Cells | |||
| IMR-32 | ATCC | CCL-127 | |
| Kelly | ECACC | 82110411 | |
| KellyCis83 | Made in lab – derived from Kelly (Piskareva et al., 2015) | - | Increasing exposure to cisplatin. Cross resistance acquired |
| SH-SY5Y | ATCC | CRL-2266 | |
| Disposable | |||
| 0.22 µm syringe filter | Millex | SLHP033RS | |
| 1.5 mL Eppendorf tube | Eppendorf | 0030 120.086 | |
| 100 mL sterile Pot | Starstedt | - | |
| 10 mL plastic pipette | Cellstar | 607 180 | |
| 15 mL Falcon tube | Starstedt | 62.554.502 | |
| 25 mL plastic pipette | Cellstar | 760 180 | |
| 50 mL Falcon tube | Starstedt | 62.547.254 | |
| 5 mL plastic pipette | Cellstar | 606 180 | |
| 6 mm Biopsy punches | Kai Medical | BP-60F | |
| Aluminium foil | - | - | |
| Cover Slip | Menzel-Glaser | - | |
| HYPERflask | Corning | CLS10030 | |
| Microscope slides | Thermo Scientific | J1840AMNT | |
| Opaque black 96-well plate | Costar | 3915 | |
| Sterile P10 tips | Starlab | S1121-3810 | |
| Sterile P1000 tips | Starlab | S1122-1830 | |
| Sterile P20 tips | Starlab | S1123-1810 | |
| Sterile P200 tips | Starlab | S1120-8810 | |
| T-175 (175 cm2 flask) | Sarstedt | 83.3912 | |
| T-75 (75 cm2 flask) | Sarstedt | 83.3911.302 | |
| Translucent clear 96 well plate | Cellstar | 655180 | |
| Translucent non-adherent 24 well plates | Cellstar | 83.3922.500 | |
| Equipment | |||
| Autoclave | Astell | - | |
| Automatic tissue processor | Leica | TP1020 | |
| Centrifuge 5804 | Eppendorf | - | |
| Hemocytometer | Hausser Scientific | - | |
| Incubator | ThermoScientific | - | |
| Microtome | Leica | RM2255 | |
| Oven | Memmert | Calibrated by: Cruinn diagnostics Ltd | |
| P10 pipette | Gilson | ||
| P100 pipette | Gilson | ||
| P1000 pipette | Gilson | ||
| P20 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| P200 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| Paraffin section flotation bath | Electrothermal | MH8517 | Calibrated by: Cruinn diagnostics Ltd |
| Pipette electronic dispenser | Corning | StripipetterUltra | Calibrated by: Cruinn diagnostics Ltd |
| Plate cooler | Leica | EG1140C | Calibrated by: Cruinn diagnostics Ltd |
| Refrigerator -20 °C | Liebherr | - | |
| Refrigerator -80 °C | Liebherr | - | |
| Refrigerator 4 °C | Liebherr | - | |
| Seesaw Rocker | DLAb | SK-D1807-E | |
| Spectrophotometer – Victor3V Platereader | PerkinElmer | 1420 | |
| Tissue culture hood/Laminar flow hood | GMI | 8038-30-1044 | |
| Tissue Lyser | Qiagen | TissueLyser LT | |
| Tweezers | - | - | |
| Water bath | Grant | - | |
| Wax embedder | Leica | EG1140H | |
| Materials | |||
| 1 L Water | Adrona - Biosciences | 568 | |
| 1% Triton-X | Sigma Aldrich | 9002-93-1 | |
| 10x PBS tablets | Sigma Aldrich | P4417-100TAB | |
| 37% paraformaldehyde | Sigma-Aldrich | F8775 | |
| Alamar Blue Cell Viability Reagent | Invitrogen | DAL1100 | |
| Collagen- glycosaminoglycan scaffold | Tissue engineering research group (TERG) | ||
| Collagen-nanohydroxyapatite scaffold | Tissue engineering research group (TERG) | ||
| dH20 | Adrona - Biosciences | 568 | |
| Eosin | Sigma-Aldrich | E4009 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| EtOH | Sigma-Aldrich | 1.00983.2500 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| F12 | Gibco | 21765-029 | |
| FBS | Gibco | 10270-106 | |
| Hemaytoxylin | Sigma-Aldrich | HHS32-1L | |
| L-Glutamine | Gibco | 25030-024 | |
| MEM | Gibco | 21090-022 | |
| miRNA easy Kit | Qiagen | 217004 | |
| MNEAA’s | Gibco | 11140-035 | |
| Penicillin/streptomycin | Gibco | 015140-122 | |
| Qiazol | Qiagen | 79306 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Invitrogen | P11496 | |
| RPMI | Gibco | 21875-034 | |
| Sodium bicarbonate | Sigma Aldrich | S7795-500G | |
| Tissue embedding Medium | Sigma | A6330-4LB | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Software | |||
| Excel | - | Excel 2016 | |
| ImageJ | - | - | |
| Prism | - | Version 9 |
Referências
- Davidoff, A. M. Neuroblastoma. Seminars in Pediatric Surgery. 21 (1), 2-14 (2012).
- Matthay, K. K., et al. Neuroblastoma. Nature Reviews Disease Primers. 2, 16078 (2016).
- Costard, L. S., Hosn, R. R., Ramanayake, H., O'Brien, F. J., Curtin, C. M. Influences of the 3D microenvironment on cancer cell behaviour and treatment responsiveness: a recent update on lung, breast and prostate cancer models. Acta Biomaterialia. , (2021).
- Borriello, L., Seeger, R. C., Asgharzadeh, S., Declerck, Y. A. More than the genes, the tumor microenvironment in neuroblastoma. Cancer Letters. 380 (1), 304-318 (2016).
- Walker, C., Mojares, E., Del Río Hernández, A. Role of extracellular matrix in development and cancer progression. International Journal of Molecular Sciences. 19 (10), 3028 (2018).
- Bissell, M. J., Hall, H. G., Parry, G. How does the extracellular matrix direct gene expression. Journal of Theoretical Biology. 99 (1), 31-68 (1982).
- Schultz, G. S., Davidson, J. M., Kirsner, R. S., Bornstein, P., Herman, I. M. Dynamic reciprocity in the wound microenvironment. Wound Repair and Regeneration. 19 (2), 134-148 (2011).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6, 11 (2008).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4, 38 (2006).
- Ouellette, J. N., et al. Navigating the collagen jungle: The biomedical potential of fiber organization in cancer. Bioengineering. 8 (2), 1-19 (2021).
- Kreger, S. T., Voytik-Harbin, S. L. Hyaluronan concentration within a 3D collagen matrix modulates matrix viscoelasticity, but not fibroblast response. Matrix Biology. 28 (6), 336-346 (2009).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Hume, R. D., et al. Tumour cell invasiveness and response to chemotherapeutics in adipocyte invested 3D engineered anisotropic collagen scaffolds. Scientific Reports. 8 (1), 12658 (2018).
- Fitzgerald, K. A., et al. The use of collagen-based scaffolds to simulate prostate cancer bone metastases with potential for evaluating delivery of nanoparticulate gene therapeutics. Biomaterials. 66, 53-66 (2015).
- Sapudom, J., Pompe, T. Biomimetic tumor microenvironments based on collagen matrices. Biomaterials Science. 6 (8), 2009-2024 (2018).
- Curtin, C., et al. A physiologically relevant 3D collagen-based scaffold-neuroblastoma cell system exhibits chemosensitivity similar to orthotopic xenograft models. Acta Biomaterialia. 70, 84-97 (2018).
- Gavin, C., et al. Neuroblastoma invasion strategies are regulated by the extracellular matrix. Cancers. 13 (4), 1-23 (2021).
- Ridky, T. W., Chow, J. M., Wong, D. J., Khavari, P. A. Invasive three-dimensional organotypic neoplasia from multiple normal human epithelia. Nature Medicine. 16 (12), 1450-1456 (2010).
- Casal, e. J., Crane, J. S. . Biochemistry, Glycosaminoglycans. , (2019).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. J. The effect of pore size on cell adhesion in collagen-GAG scaffolds. Biomaterials. 26 (4), 433-441 (2005).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. Influence of freezing rate on pore structure in freeze-dried collagen-GAG scaffolds. Biomaterials. 25 (6), 1077-1086 (2004).
- Haugh, M. G., Murphy, C. M., McKiernan, R. C., Altenbuchner, C., O'Brien, F. J. Crosslinking and mechanical properties significantly influence cell attachment, proliferation, and migration within collagen glycosaminoglycan scaffolds. Tissue Engineering. Part A. 17 (9-10), 1201-1208 (2011).
- Murphy, C. M., Haugh, M. G., O'Brien, F. J. The effect of mean pore size on cell attachment, proliferation and migration in collagen-glycosaminoglycan scaffolds for bone tissue engineering. Biomaterials. 31 (3), 461-466 (2010).
- Haugh, M. G., Jaasma, M. J., O'Brien, F. J. The effect of dehydrothermal treatment on the mechanical and structural properties of collagen-GAG scaffolds. Journal of Biomedical Materials Research - Part A. 89 (2), 363-369 (2009).
- Lowe, B., Hardy, J. G., Walsh, L. J. Optimizing nanohydroxyapatite nanocomposites for bone tissue engineering. ACS Omega. 5 (1), 1-9 (2020).
- Cunniffe, G. M., Dickson, G. R., Partap, S., Stanton, K. T., O'Brien, F. J. Development and characterisation of a collagen nano-hydroxyapatite composite scaffold for bone tissue engineering. Journal of Materials Science. Materials in Medicine. 21 (8), 2293-2298 (2010).
- DuBois, S. G., et al. Metastatic sites in stage IV and IVS neuroblastoma correlate with age, tumor biology, and survival. Journal of Pediatric Hematology/Oncology. 21 (3), 181-189 (1999).
- Ryan, A. J., Gleeson, J. P., Matsiko, A., Thompson, E. M., O'Brien, F. J. Effect of different hydroxyapatite incorporation methods on the structural and biological properties of porous collagen scaffolds for bone repair. Journal of Anatomy. 227 (6), 732-745 (2015).
- Tierney, C. M., et al. The effects of collagen concentration and crosslink density on the biological, structural and mechanical properties of collagen-GAG scaffolds for bone tissue engineering. Journal of the Mechanical Behavior of Biomedical Materials. 2 (2), 202-209 (2009).
- Cox, R. F., Jenkinson, A., Pohl, K., O'Brien, F. J., Morgan, M. P. Osteomimicry of mammary adenocarcinoma cells in vitro; increased expression of bone matrix proteins and proliferation within a 3D collagen environment. PLoS One. 7 (7), 41679 (2012).
- Nolan, J. C., et al. Preclinical models for neuroblastoma: advances and challenges. Cancer Letters. 474, 53-62 (2020).
- Sirivisoot, S., Pareta, R., Harrison, B. S. Protocol and cell responses in threedimensional conductive collagen gel scaffolds with conductive polymer nanofibres for tissue regeneration. Interface Focus. 4 (1), 20130050 (2014).
- Thevenot, P., Nair, A., Dey, J., Yang, J., Tang, L. Method to analyze three-dimensional cell distribution and infiltration in degradable scaffolds. Tissue Engineering. Part C-Methods. 14 (4), 319-331 (2008).
- do Amaral, R. J. F. C., et al. Functionalising collagen-based scaffolds with platelet-rich plasma for enhanced skin wound healing potential. Frontiers in Bioengineering and Biotechnology. 7, 371 (2019).
- Gkolfinopoulos, S., Tsapakidis, K., Papadimitriou, K., Papamichael, D., Kountourakis, P. Chromogranin A as a valid marker in oncology: Clinical application or false hopes. World Journal of Methodology. 7 (1), 9-15 (2017).
- O'Brien, F. J., et al. The effect of pore size on permeability and cell attachment in collagen scaffolds for tissue engineering. Technology and Health Care. 15 (1), 3-17 (2007).
Erratum
Formal Correction: Erratum: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds
Posted by JoVE Editors on 6/23/2022. Citeable Link.
An erratum was issued for: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds. The Authors section was updated.
The Authors section was updated from:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Fergal J. O’Brien3,4,5
Olga Piskareva1,2,3,5,6
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
5Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
6National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
to:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Graeme Kelly4
Fergal J. O’Brien3,5,6
Olga Piskareva1,2,3,6,7
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Department of Chemistry, Royal College of Surgeons in Ireland (RCSI), 123 St. Stephen’s Green, Dublin 2, Ireland
5Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
6Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
7National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados