Method Article
Dreidimensionales biomimetisches In-vitro-Modell des Neuroblastoms unter Verwendung von kollagenbasierten Gerüsten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Erratum Notice
Zusammenfassung
Dieser Artikel listet die Schritte auf, die erforderlich sind, um Neuroblastom-Zelllinien auf zuvor beschriebenen dreidimensionalen kollagenbasierten Gerüsten auszusäen, das Zellwachstum für einen vorbestimmten Zeitrahmen aufrechtzuerhalten und Gerüste für verschiedene Zellwachstums- und Zellverhaltensanalysen und nachgeschaltete Anwendungen zu gewinnen, die an eine Reihe von experimentellen Zielen angepasst werden können.
Zusammenfassung
Das Neuroblastom ist der häufigste extrakranielle solide Tumor bei Kindern und macht 15 % der gesamten pädiatrischen Krebstodesfälle aus. Das native Tumorgewebe ist eine komplexe dreidimensionale (3D) Mikroumgebung, die Schichten von Krebs- und Nicht-Krebszellen umfasst, die von einer extrazellulären Matrix (EZM) umgeben sind. Die ECM bietet physikalische und biologische Unterstützung und trägt zum Fortschreiten der Erkrankung, zur Prognose des Patienten und zum therapeutischen Ansprechen bei.
In diesem Artikel wird ein Protokoll für den Aufbau eines 3D-Gerüst-basierten Systems beschrieben, um die Neuroblastom-Mikroumgebung unter Verwendung von Neuroblastom-Zelllinien und kollagenbasierten Gerüsten nachzuahmen. Die Gerüste werden entweder mit Nanohydroxylapatit (nHA) oder Glykosaminoglykanen (GAGs) ergänzt, die natürlicherweise in hohen Konzentrationen im Knochen und Knochenmark, den häufigsten Metastasen des Neuroblastoms, vorkommen. Die poröse 3D-Struktur dieser Gerüste ermöglicht die Anheftung, Proliferation und Migration von Neuroblastomzellen sowie die Bildung von Zellclustern. In dieser 3D-Matrix spiegelt die Zellantwort auf Therapeutika die In-vivo-Situation besser wider.
Das gerüstbasierte Kultursystem kann höhere Zelldichten aufweisen als herkömmliche zweidimensionale (2D) Zellkulturen. Daher sind Optimierungsprotokolle für die anfängliche Aussaat der Zellzahlen von den gewünschten experimentellen Zeitrahmen abhängig. Das Modell wird überwacht, indem das Zellwachstum mittels DNA-Quantifizierung, die Zelllebensfähigkeit mittels metabolischer Assays und die Zellverteilung innerhalb der Gerüste mittels histologischer Färbung bewertet werden.
Zu den Anwendungen dieses Modells gehören die Bewertung von Gen- und Proteinexpressionsprofilen sowie Zytotoxizitätstests mit konventionellen Medikamenten und miRNAs. Das 3D-Kultursystem ermöglicht die präzise Manipulation von Zell- und EZM-Komponenten und schafft so eine Umgebung, die dem nativen Tumorgewebe physiologisch ähnlicher ist. Daher wird dieses 3D-In-vitro-Modell das Verständnis der Krankheitspathogenese verbessern und die Korrelation zwischen in vitro, in vivo in Tiermodellen und menschlichen Probanden verbessern.
Einleitung
Das Neuroblastom ist eine pädiatrische Krebserkrankung des sympathischen Nervensystems, die während der Embryonalentwicklung oder im frühen postnatalen Leben durch die Transformation von Neuralleistenzellen entsteht1. Es ist der häufigste solide extrakranielle Tumor bei Kindern, der 8 % der bei Patienten unter 15 Jahren diagnostizierten Malignome ausmacht und für 15 % aller Krebstodesfälle im Kindesalter verantwortlich ist. Die Krankheit zeigt ein sehr heterogenes klinisches Verhalten aufgrund spezifischer chromosomaler, genetischer und epigenetischer Veränderungen sowie histopathologischer Merkmale.
Diese Veränderungen tragen zur Aggressivität des Neuroblastoms und zu schlechten Ergebnissen bei pädiatrischen Patienten bei. Daher erweisen sich die derzeitigen Therapien bei fast 80 % der Patienten mit der klinisch aggressivenErkrankung als langfristig unwirksam2, was die Tatsache unterstreicht, dass die Behandlung dieser Patientengruppe nach wie vor eine Herausforderung darstellt. Dies ist wahrscheinlich darauf zurückzuführen, dass die Mechanismen der Neuroblastom-Heterogenität und der Metastasen noch nicht vollständig verstanden sind. Es wird jedoch allgemein angenommen, dass die Tumormikroumgebung (TME) eine Rolle bei der Fortschreitung vieler Krebsarten spielt. Dennoch ist es beim Neuroblastom 3,4 noch wenig untersucht.
Das native TME ist eine komplexe 3D-Mikroumgebung mit krebsartigen und nicht-krebsartigen Zellen, die von einer EZM umgeben sind. Die EZM bezieht sich auf die azelluläre Komponente eines Gewebes, die seinen zellulären Bewohnern strukturelle und biochemische Unterstützung bietet und zum Fortschreiten der Krankheit, zur Prognose des Patienten und zum therapeutischen Ansprechen beiträgt5. Diese Förderung des Fortschreitens der Krankheit ist auf eine "dynamische Reziprozität" oder eine kontinuierliche bidirektionale Kommunikation zwischen Zellen und der EZM zurückzuführen 6,7,8. Wenn der Krebs fortschreitet, wird das stromale Kollagen oft in linearen Mustern senkrecht zur Stroma-Krebs-Grenzfläche reorganisiert, die Krebszellen als Migrationsweg zur Metastasierung nutzen 9,10,11.
Zu den Hauptbestandteilen dieses nativen funktionellen biologischen Gerüsts gehören ein faseriges Netzwerk aus Kollagenen Typ I und II und anderen Proteinen, einschließlich Elastin, Glykoproteinen wie Laminin sowie eine Reihe von Proteoglykanen und anderen löslichen Komponenten12,13. Diese Proteine der nativen EZM sind nun zu attraktiven natürlichen Biomolekülen für die Entwicklung von 3D-In-vitro-Modellen geworden 3. Die Anwendung von 3D-Gerüsten für die In-vitro-Zellkultur erfreut sich zunehmender Beliebtheit, da sie im Vergleich zur herkömmlichen 2D-Monolayer-Kultur eine bessere physiologische Darstellung der TME aufweisen. Die hergestellten 3D-Gerüste unterstützen die Zelladhäsion, die Proliferation, die Migration, den Stoffwechsel und die Reaktion auf Stimuli, die in biologischen In-vivo-Systemen beobachtet werden.
Der Hauptbestandteil dieser 3D-Gerüste ist Kollagen, das eine Schlüsselrolle bei vielen normalen biologischen Prozessen spielt, darunter Gewebereparatur, Angiogenese, Gewebemorphogenese, Zelladhäsion und Migration11. Kollagenbasierte 3D-Matrizen haben ihre robuste Funktionalität zur Modellierung von EZM unter Beweis gestellt, indem sie als biomimetische In-vitro-Mikroumgebung dienen und gleichzeitig Zell-ECM-Interaktionen sowie Zellmigration und -invasion ermöglichen. Diese 3D-Matrizen bieten auch eine genauere Analyse der Zellantwort auf Chemotherapeutika als herkömmliche 2D- oder "flache" Kulturen in vielen Krebsmodellen 14,15,16, einschließlich Neuroblastom 17,18. Die genetische Analyse von 3D-Zellkulturen hat eine höhere Korrelation mit dem menschlichen Gewebeprofil gezeigt, selbst im Vergleich zu Tiermodellen19. Insgesamt besteht der Eckpfeiler dieser 3D-Gerüste darin, den Zellen eine geeignete In-vitro-Umgebung zu bieten, die die native Gewebearchitektur rekapituliert und bidirektionales molekulares Crosstalk ermöglicht8.
Um die Komplexität von kollagenbasierten Modellen zu erhöhen, werden andere gängige ECM-Komponenten in den Tissue-Engineering-Prozess integriert, wodurch physiologisch relevantere Modelle geschaffen werden, die Nischen-TMEs verschiedener Gewebe widerspiegeln. Zum Beispiel erleichtern GAGs, negativ geladene Polysaccharide, die in allen Säugetiergeweben20 vorhanden sind, die Zellanhaftung, Migration, Proliferation und Differenzierung. Chondroitinsulfat ist eine bestimmte Art von GAG, die in Knochen und Knorpel vorkommt und zuvor in Tissue-Engineering-Anwendungen zur Knochenreparatur verwendet wurde 21,22,23,24,25. Nano-Hydroxylapatit (nHA) ist der wichtigste anorganische Bestandteil der mineralischen Zusammensetzung des menschlichen Knochengewebes, der bis zu 65 Gewichtsprozent des Knochens ausmacht26 und daher häufig für den Knochenersatz und die Knochenregeneration verwendetwird 27. Daher sind GAGs und nHA attraktive Komposite für die Rekonstruktion der primären Neuroblastom-EZM und die Modellierung der häufigsten Metastasen von Neuroblastom, Knochenmark (70,5 %) und Knochen (55,7 %)28.
Scaffolds, die diese EZM-Komponenten enthalten, wurden ursprünglich für Anwendungen im Bone Tissue Engineering mit umfassender Analyse ihrer Biokompatibilität, Toxizität sowie osteokonduktiven und osteoinduktiven Eigenschaften entwickelt29,30. Es handelt sich um poröse, auf Kollagen basierende Matrizen, die mit Gefriertrocknungstechniken hergestellt werden, um ihre physikalischen und biologischen Eigenschaften zu kontrollieren. Die Kollagengerüste, die entweder mit nHA (Coll-I-nHA) oder Chondroitin-6-sulfat (Coll-I-GAG) ergänzt wurden, zeigten Erfolg bei der Nachahmung der primären TME bei Brustkrebs31 und der Metastasierung in Knochen bei Prostatakrebs15 sowie Neuroblastom17. Die Gefriertrocknungstechnik, die zur Herstellung dieser Verbundgerüste verwendet wird, führt zu einer reproduzierbaren Homogenität in Porengröße und Porosität innerhalb der Gerüste22, 23, 24. Kurz gesagt, eine Kollagenaufschlämmung (0,5 Gew.-%) wird durch Mischen von fibrillärem Kollagen mit 0,05 M Essigsäure hergestellt. Bei Coll-I-GAG werden 0,05 Gew.-% des aus Haifischknorpel isolierten Chrondoitin-6-sulfats während des Mischens der Kollagenaufschlämmung zugesetzt. Für die zusammengesetzten Coll-I-nHA-Gerüste werden Hydroxylapatitpartikel in Nanogröße, wie zuvor beschrieben27, synthetisiert und der Kollagenaufschlämmung in einem Verhältnis von 2:1 zum Gewicht des Kollagens während des Mischprozesses zugesetzt. Alle Scaffolds werden physikalisch vernetzt und mit einer dehydrothermalen Behandlung bei 105 °C für 24 h25 sterilisiert. Zylindrische Gerüste (6 mm Durchmesser, 4 mm Höhe) werden unter Verwendung einer Biopsienstanze erhalten und können mit 3 mM N-(3-Dimethylaminopropyl)-N'-ethylcarbodiimidhydrochlorid und 5,5 mM N-Hydroxysuccinimid (EDAC/NHS) in destilliertem Wasser (dH2O) chemisch vernetzt werden, um die mechanischen Eigenschaften der Konstrukte30 zu verbessern. Dieser gut optimierte Herstellungsprozess von zwei Kollagengerüsten erzeugt Gerüste mit reproduzierbaren mechanischen Eigenschaften, einschließlich Porengröße, Porosität und Steifigkeit (kPa). Sowohl Coll-I-GAG- als auch Coll-I-nHA-Gerüste haben unterschiedliche physikalische Eigenschaften, wodurch unterschiedliche Umgebungsbedingungen entstehen. Die Eigenschaften der einzelnen Gerüste sind in Tabelle 1 aufgeführt.
| Coll-I-GAG | Coll-I-nHA | |
| Größe des Gerüsts (Durchmesser [mm] x Höhe [mm]) | 6 x 4 17 | 6 x 4 17 |
| Kollagenkonzentration (Gew.-%) | 0,5 17 | 0,5 17 |
| Substratkonzentration (Gew.-%) [basierend auf dem Gewicht des Kollagens] | 0.05 15,17 | Nr. 200 17 |
| Mittlere Porengröße (mm) | 96 22 | 96 – 120 29 |
| Porosität (%) | 99,5 23 | 98,9 – 99,4 27 |
| Steifigkeit (kPa) | 1,5 27 | 5,5 - 8,63 29 |
Tabelle 1: Überblick über die mechanischen Eigenschaften der beiden Scaffolds, die für die Untersuchung der Neuroblastombiologie verwendet wurden.
In dieser Arbeit wird ein Protokoll zur Zusammenstellung eines 3D-Gerüst-basierten Systems beschrieben, um die Neuroblastom-Mikroumgebung unter Verwendung von Neuroblastom-Zelllinien und zuvor beschriebenen kollagenbasierten Gerüsten besser nachzuahmen, die entweder mit nHA (Coll-I-nHA) oder Chondroitin-6-sulfat (Coll-I-GAG) ergänzt werden. Das Protokoll umfasst nachgeschaltete Methoden zur Analyse der Wachstumsmechanismen der Neuroblastomzellen in einer physiologisch relevanteren Umgebung unter Verwendung zuvor optimierter, kostengünstiger Methoden, die aus der 2D-Monolayer-Kultur adaptiert wurden ( Abbildung 1).
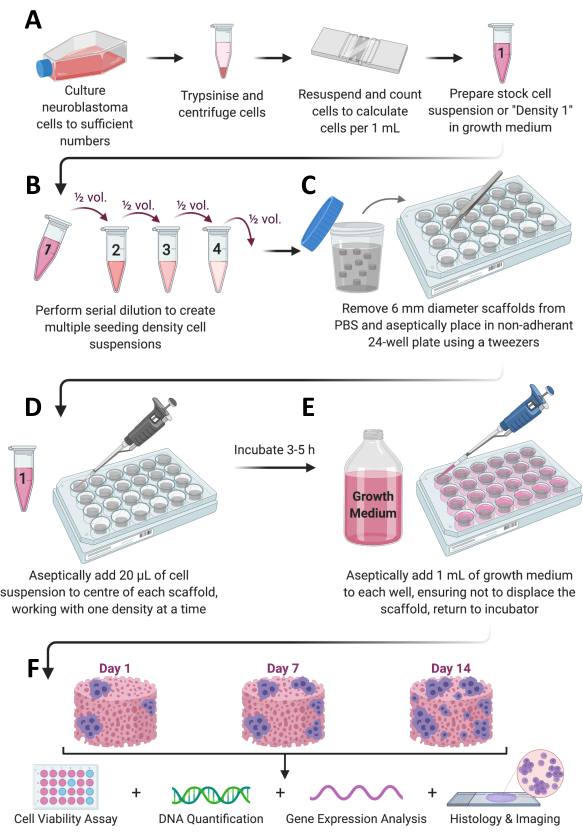
Abbildung 1: Gesamter Protokoll-Workflow. (A) Die Zellen werden in ausreichender Anzahl gezüchtet, geteilt, gezählt und in einem geeigneten Mediumvolumen resuspendiert. (B) Dieser Zellstamm wird dann einer seriellen Verdünnung unterzogen, um insgesamt 4 Zellsuspensionen unterschiedlicher Dichte herzustellen. (C) Gerüste auf Kollagenbasis werden steril in nicht haftenden 24-Well-Platten plattiert, und (D) 20 μl Zellsuspension werden in die Mitte jedes Gerüsts gegeben und 3-5 Stunden lang bei 37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit inkubiert. (E) Vollständiges Wachstumsmedium (1 ml) wird dann langsam zu jedem Gerüst gegeben, und die Platten werden wieder in den Inkubator gelegt, um das Zellwachstum für den gewünschten Zeitrahmen zu ermöglichen. (F) Zu jedem vorgegebenen Zeitpunkt werden mehrere Gerüste zur Beurteilung der Lebensfähigkeit und des Wachstums der Zellen, zur Genexpressionsanalyse und zur histologischen Färbung entnommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Versuchsplanung
HINWEIS: Die Anzahl der Gerüste und Zellen, die für jedes Experiment benötigt werden, hängt vom Umfang des Experiments ab und kann mit den Werkzeugen in diesem Abschnitt zur Versuchsplanung berechnet werden.
- Anzahl der benötigten Gerüste
- Bestimmen Sie den gesamten experimentellen Zeitplan und die Bewertungsintervalle für Veränderungen in der Zellbiologie (z. B. Zellwachstum und Stoffwechsel).
HINWEIS: Der experimentelle Zeitrahmen beträgt beispielsweise 14 Tage, wobei alle 7 Tage eine Bewertung des Zellwachstums durchgeführt wird. Die experimentellen Zeitpunkte sind also die Tage 1, 7 und 14, was insgesamt 3 Zeitpunkte ergibt. - Entscheiden Sie als Nächstes die Anzahl der experimentellen Anwendungen, die zu jedem Zeitpunkt angewendet werden sollen. Versuchen Sie, zu jedem Zeitpunkt die gleichen Analysen durchzuführen, um die Änderungen zu überwachen.
HINWEIS: Zu den typischen Analysen des Zellwachstums auf Gerüsten gehören Zellviabilitätstests, DNA-Quantifizierung, histologische Färbung und Bildgebung sowie Genexpressionsanalysen. Diese werden in Abschnitt 5 näher erläutert. - Entscheiden Sie, wann dasselbe Gerüst für mehrere nachgelagerte Anwendungen verwendet werden soll, z. B. nach der Bewertung der Zellviabilität mit einem geeigneten Assay das Gerüst für die DNA/RNA-Isolierung wiederverwenden (siehe 5.1.11).
- Halten Sie für jede Anwendung mindestens 3 biologische Replikate bereit, z. B. 3 Gerüste für die Zelllebensfähigkeit und DNA-Quantifizierung, 3 für die Histologie und 3 für die Genexpressionsanalyse. Da dies 9 Gerüsten pro Zeitpunkt entspricht, planen Sie die Verwendung von 27 Gerüsten für 3 Zeitpunkte ein.
- Multiplizieren Sie diese Zahl schließlich mit der Anzahl der experimentellen Parameter oder Bedingungen (z. B. Bewertung mehrerer Zelllinien, anfängliche Aussaatdichten, Gerüstzusammensetzungen).
HINWEIS: In diesem Protokoll wurden 4 verschiedene anfängliche Aussaatdichten für eine Zelllinie bewertet, was dazu führte, dass 108 (27 Gerüste x 4 Bedingungen) Gerüste erforderlich waren. Um menschliches Versagen zu berücksichtigen und eine zusätzliche Abdeckung zu gewährleisten, fügen Sie ~10 % zu dieser Zahl hinzu, z. B. wenn 108 Gerüste erforderlich sind, bereiten Sie 120 Gerüste vor.
- Bestimmen Sie den gesamten experimentellen Zeitplan und die Bewertungsintervalle für Veränderungen in der Zellbiologie (z. B. Zellwachstum und Stoffwechsel).
- Anzahl der benötigten Zellen
HINWEIS: Für die Aussaat von Zellen auf ein zylindrisches Gerüst (Durchmesser 6 mm, Höhe 4 mm) am Tag 0 wird ein Volumen von 20 μl Zellsuspension empfohlen. Die Anzahl der Zellen pro diesen 20 μl Zellsuspension wird entsprechend der in Abschnitt 1.1 berechneten Versuchsplanung angepasst. Eine übliche anfängliche Aussaatdichte beträgt 2 × 105 Zellen pro 20 μl, die als Beispiel für das folgende Protokoll verwendet wird.- Um die Gesamtmenge der für das Experiment benötigten Zellen zu berechnen, multiplizieren Sie die anfänglichen 2 × 105 Zellen pro 20 μl mit der Anzahl der erforderlichen Gerüste.
HINWEIS: Zum Beispiel ergeben 30 Gerüste multipliziert mit 20 μl ein Gesamtvolumen von 600 μl. Wenn für jedes Gerüst 2 × 10 5 Zellen erforderlich sind, enthalten 600 μl Suspension insgesamt 6 × 10 6 Zellen (2 × 105 × 30), was einen Endbedarf von 6 × 106 Zellen in 600 μl ergibt. Die Anzahl der experimentellen Parameter bestimmt die Gesamtzahl der benötigten Zellen. Dieses Protokoll beschreibt daher die Zellkultur mit einem mehrschichtigen Zellkulturkolben, der die gleiche Anzahl von Zellen verarbeiten kann wie 10 herkömmliche 175-cm2-Kolben.
- Um die Gesamtmenge der für das Experiment benötigten Zellen zu berechnen, multiplizieren Sie die anfänglichen 2 × 105 Zellen pro 20 μl mit der Anzahl der erforderlichen Gerüste.
2. Herstellung von Scaffolds auf Kollagenbasis
HINWEIS: Die zylindrischen Gerüste Coll-I-nHA und Coll-I-GAG (Durchmesser 6 mm, Höhe 4 mm) werden nach den etablierten Methoden 15,21,27 hergestellt. Nach der chemischen Vernetzung gemäß den zuvor veröffentlichten Verfahren17 müssen die Scaffolds innerhalb von 1 Woche verwendet werden.
- Stellen Sie nach der Herstellung der Gerüste mit den gewünschten mechanischen Eigenschaften sicher, dass die Gerüste vollständig hydratisiert und gründlich in phosphatgepufferter Kochsalzlösung (PBS) gewaschen werden.
HINWEIS: Dies dauert in der Regel ~12 h nach der Vernetzung der Gerüste und kann in 100-ml-Gewebekulturabfallbehältern bei 4 °C durchgeführt werden, mit maximal 50 Gerüsten pro Behälter und 2 ml PBS pro Gerüst. - Lagern Sie die Gerüste in PBS bei 4 °C, bis sie gebrauchsfertig sind.
3. Vermehrung von Neuroblastomzellen in einer mehrschichtigen Zellkulturflasche
HINWEIS: Die optimale Aussaatdichte für den Mehrschichtkolben variiert. Für den in diesem Versuch verwendeten Kolben beträgt die optimale Dichte gemäß den Anweisungen des Herstellers 1 × 107 Zellen. Vor der Aussaat des Mehrschichtkolbens sind die Zellen in einem geeigneten Gewebekulturkolben (z. B. einem T175 cm2 Gewebekulturkolben) auf eine Dichte von 1 ×10 7 Zellen oder höher zu vermehren. Um Zellen in den Mehrschichtkolben (Abschnitt 3.1) zu säen, züchten Sie sie, bis sie zu 70-80 % zusammenfließen, ernten und zählen Sie die Anzahl der Zellen pro ml, wobei Sie sich auf die Schritte 3.2.16-3.2.20 zur Durchführung der Zellzählung beziehen. Sobald die Zellsuspension gezählt ist, fahren Sie sofort mit der Aussaat des Mehrschichtkolbens fort. Zellkulturarbeiten müssen in einer Laminar-Flow-Haube durchgeführt werden, um die Sterilität zu gewährleisten.
- Aussaat des mehrschichtigen Zellkulturkolbens
- 550 ml vollständiges Wachstumsmedium (variiert je nach verwendeter Zelllinie) und 100 ml steriles PBS in einem 37 °C warmen Wasserbad für 20 Minuten vorwärmen.
- Berechnen Sie unter Verwendung der geernteten Zellsuspension das erforderliche Volumen der Zellsuspension, das erforderlich ist, um die optimale Aussaatdichte von 1 × 107 Zellen unter Verwendung von Gleichung (1) zu erreichen, wobei sich WANT auf die Anzahl der Zellen bezieht, die für die Aussaat des Mehrschichtkolbens erforderlich sind, und HAVE sich auf die Anzahl der Zellen/ml in der Zellsuspension bezieht:
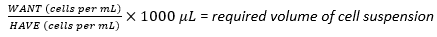 (1)
(1)
zum Beispiel.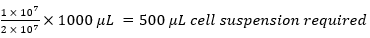
- Geben Sie das erforderliche Volumen der Zellsuspension zu 100 ml des vorgewärmten Wachstumsmediums.
- Nehmen Sie einen neuen Mehrschichtkolben in die Haube, entfernen Sie den Deckel und halten Sie den Kolben in einem Winkel von 60°. Alle 100 ml der Zellsuspension werden langsam an der abgewinkelten Seite des Halses in den Kolben pipettiert. Verschließen Sie den Kolben und legen Sie ihn auf die Seite, damit sich die Zellen gleichmäßig in den Schichten verteilen können.
- In einem Winkel von 60° werden 400 ml des vorgewärmten Wachstumsmediums in den Mehrschichtkolben gegeben, indem die abgewinkelte Seite des Halses langsam gegossen oder pipettiert wird. Wenn der Hals voll ist, bringen Sie den Kolben wieder in eine aufrechte Position oder verschließen Sie den Kolben und legen Sie ihn auf die Seite, bevor Sie wieder mit dem Ausgießen beginnen.
HINWEIS: Gießen Sie langsam, um eine übermäßige Blasenbildung zu vermeiden. Klopfen Sie vorsichtig auf den Kolben in aufrechter Position, damit alle Blasen nach oben steigen können, und entfernen Sie sie mit einer 10-ml-Pipette. Stellen Sie sicher, dass der Kolben bis zum unteren Gewinde des Halses gefüllt ist. Fügen Sie bei Bedarf mehr Medium hinzu, um dies zu erreichen. - Verschließen Sie den Mehrschichtkolben und inkubieren Sie ihn mit dem abgewinkelten Hals nach unten bei 37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit.
- Überprüfen Sie das Wachstum alle 2-3 Tage auf Konfluenz. Um die Konfluenz der unteren beiden Schichten des Mehrschichtkolbens zu überprüfen, betrachten Sie sie unter einem 4-fach-Objektiv eines inversen Mikroskops.
HINWEIS: Wenn der 10-Schicht-Kolben mit 1 × 107 Neuroblastomzellen ausgesät wird, dauert es in der Regel eine Woche, bis er konfluiert ist - obwohl dies je nach verwendeter Zelllinie variieren kann.
- Routinemäßige Wartung von Zellen im Mehrschichtkolben
- 550 ml vollständiges Wachstumsmedium (variiert je nach verwendeter Zelllinie), 50-100 ml Trypsin und 300 ml steriles PBS in einem 37 °C warmen Wasserbad für 20 Minuten vorwärmen.
- Prüfen Sie den Mehrschichtkolben auf 70-80% Konfluenz.
- Stellen Sie den Mehrschichtkolben in eine Laminar-Flow-Haube und entsorgen Sie das verbrauchte Medium durch Ausgießen aus dem Kolben. Kippen Sie den Kolben zunächst so, dass das Medium über den Luftdamm in einen Abfallbehälter fließt. Während des Gießens wird der Kolben um 180° gedreht, bis das Medium am abgewinkelten Hals des Kolbens hinunterfließt. Drehen Sie den Kolben entlang dieser Achse hin und her, um die restliche Flüssigkeit zu entfernen.
- Waschen Sie die Zellen, indem Sie 100 ml vorgewärmtes steriles PBS langsam in den abgewinkelten Hals geben. Verschließen Sie den Kolben, legen Sie ihn auf die Seite, um eine gleichmäßige Verteilung von PBS zu ermöglichen, und drehen Sie den Kolben hin und her, um die Zellen zu waschen.
- Die PBS-Wäsche ist auf die gleiche Weise wie in 3.2.3 zu verwerfen. Wiederholen Sie die Waschschritte.
- Verdünnen Sie 50 ml des vorgewärmten Trypsins in 50 ml vorgewärmtem sterilem PBS. Geben Sie 100 ml verdünnte Trypsinlösung in den Mehrschichtkolben, die Kappe und legen Sie den Kolben auf die Seite, um eine gleichmäßige Verteilung des Trypsins zu ermöglichen. Wenn die Zelllinie stark adhärent ist, verwenden Sie 100 ml unverdünntes Trypsin.
- Der Kolben wird 2-5 Minuten lang bei 37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit inkubiert und dabei die Zellablösung unter dem Mikroskop überwacht. Falls erforderlich, klopfen Sie den Kolben fest, um das Abnehmen zu erleichtern, oder legen Sie ihn für eine weitere Minute zurück in den Inkubator.
- Stellen Sie den Mehrschichtkolben in eine Laminar-Flow-Haube und neutralisieren Sie das Trypsin mit 100 ml Wachstumsmedium. Verschließen Sie den Kolben, legen Sie ihn auf die Seite und wippen Sie hin und her, um eine vollständige Neutralisierung zu gewährleisten.
- Gießen Sie die neutralisierte Zellsuspension in 4 x 50 mL konische Zentrifugenröhrchen.
HINWEIS: Wenn eine vollständige Zellernte erforderlich ist, waschen Sie den Mehrschichtkolben erneut mit 100 ml sterilem PBS und gießen Sie ihn in 2 x 50 ml Zentrifugenröhrchen. - Zentrifugieren Sie die Zellsuspension in 50-ml-Zentrifugenröhrchen bei 340 × g für 3-4 Minuten , um die Zellen zu pelletieren.
- Führen Sie die Zentrifugenröhrchen wieder in die Laminar-Flow-Haube zurück und entsorgen Sie vorsichtig so viel Überstand wie möglich aus jedem Pellet.
HINWEIS: Das Pellet ist groß und relativ locker und kann daher leicht zerbrochen werden. - Geben Sie 1-5 ml Wachstumsmedium zu jedem Pellet und resuspendieren Sie es durch mehrmaliges Auf- und Abpipettieren.
- Fassen Sie die 4 resuspendierten Pellets in einem 50-ml-Zentrifugenröhrchen zusammen, mischen Sie sie gründlich mit der Pipette und notieren Sie das Gesamtvolumen.
- Nehmen Sie eine geeignete Verdünnung der Zellsuspension für die Zellzählung vor, so dass jedes äußere Quadrat auf dem Hämozytometer 30-100 Zellen enthält.
HINWEIS: Eine geeignete Ausgangsverdünnung ist 1:100; Dazu pipettieren Sie 10 μl der Zellsuspension in ein frisches 15 ml konisches Zentrifugenröhrchen und verdünnen es mit 990 μl sterilem PBS. Pipettieren Sie die Mischung mehrmals auf und ab, um sie gründlich zu vermischen. - Legen Sie ein 50-ml-Zentrifugenröhrchen mit dem Zellvorrat in den Inkubator, während Sie zählen.
- Nehmen Sie ein sauberes Hämozytometer und legen Sie ein sauberes Deckglas über das Gitter, wie in Abbildung 2 gezeigt.
- Pipettieren Sie 10 μl der verdünnten Zellsuspension in die Hämozytometerkammer. Wenn die Kammer voll ist, bevor alle 10 μl abgegeben wurden, beenden Sie das Pipettieren.
- Platzieren Sie das Hämozytometer unter dem 4-fach-Objektiv eines Lichtmikroskops. Passen Sie den Grob- und Feinfokus an, um die Zellen zu visualisieren.
- Zählen Sie die Anzahl der Zellen in den vier äußeren Eckquadraten der Kammer, wie in Abbildung 2 dargestellt. Addieren Sie die vier Zählungen und dividieren Sie durch 4, um die durchschnittlichen Zellen pro Quadrat zu berechnen.
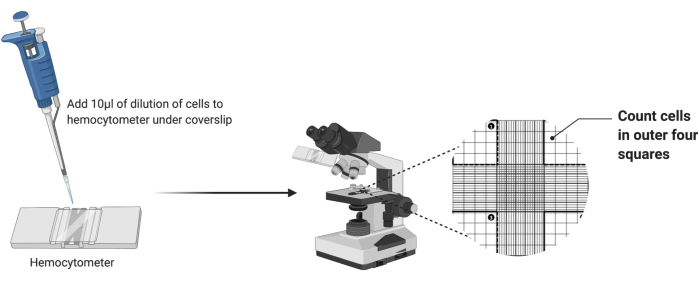
Abbildung 2: Zellzählung mit einem Hämozytometer. Zehn Mikroliter Zellsuspension werden in das Hämozytometer unter dem Deckglas gegeben. Die Kammer wird dann unter die 4-fach-Objektivlinse eines Mikroskops gestellt, und die Anzahl der Zellen in den vier äußeren Ecken des Gitters wird gezählt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Multiplizieren Sie die durchschnittliche Anzahl mit dem Verdünnungsfaktor (z. B. 100) und multiplizieren Sie diese Zahl mit 10.000, um die Anzahl der Zellen pro ml mit Gleichung (2) zu erhalten.
 (2)
(2) - Berechnen Sie die Gesamtzahl der Zellen in der Stammlösung, indem Sie sie mit dem Gesamtvolumen des Zellbestands multiplizieren (z. B. wenn die 4 resuspendierten Pellets eine 20-ml-Lösung ergeben, multiplizieren Sie die Zellen/ml mit 20).
- Um den Mehrschichtkolben instand zu halten, wird nach Gleichung (1) in Schritt 3.1.2 das Volumen des Zellmaterials berechnet, das erforderlich ist, um 1 × 107 Zellen wieder in den Kolben zu säen, und die Schritte 3.1.3 bis 3.1.6 durchzuführen, um den Kolben wieder auszusäen. Wenn Sie bereit sind, die Gerüste auszusäen, fahren Sie mit dem nächsten Abschnitt fort.
4. Neuroblastomzellen auf Gerüsten aussäen
- Vorbereiten der Stammzellensuspension
HINWEIS: In diesem Protokoll werden die Schritte zur Erstellung von vier verschiedenen Aussaatdichten von Neuroblastomzellen mit einem Multiplikationsfaktor von 2 zwischen jeder Dichte beschrieben. Eine serielle Verdünnung wird daher verwendet, um aus der Stammsuspension drei weitere Zellsuspensionen herzustellen. Zellkulturarbeiten müssen in einer Laminar-Flow-Haube durchgeführt werden, um die Sterilität zu gewährleisten.- Mit Gleichung (3) wird das benötigte Zellvolumen aus der Gesamtzahl der Zellen im Mehrschichtkolben (gezählt in Abschnitt 3.2.19) berechnet, um die erste Aussaatdichte oder Zellstocksuspension herzustellen.
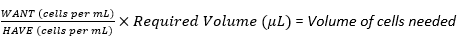 (3)
(3)
HINWEIS: Wenn beispielsweise die höchste Aussaatdichte 6 × 10 5 Zellen pro Gerüst beträgt, die für 30 Gerüste erforderlich sind, wobei jedes Gerüst 20 μl Zellsuspension erhält, würde die Stammzellensuspension 1,8 × 107 Zellen (6 × 105 Zellen × 30 Gerüste) in einem Gesamtvolumen von 600 μl (20 μl × 30 Gerüste) erfordern.
Da von diesem Präparat eine serielle Verdünnung durchgeführt wird, müssen diese Zahlen verdoppelt werden, d. h. 3,6 × 107 Zellen in einem Gesamtvolumen von 1200 μl. Um dies in WANT in Zellen pro ml umzuwandeln; 3,6 × 107 durch 1200 μl dividiert und mit 1000 μl multipliziert, so dass man 3 × 107 Zellen pro ml erhält.

- Geben Sie das erforderliche Volumen des Zellstocks in ein steriles 2-ml- oder 15-ml-Zentrifugenröhrchen und bringen Sie es mit Wachstumsmedium auf ein Endvolumen von 1200 μl. Beschriften Sie dieses Röhrchen mit Dichte 1 (Abbildung 3).
- Mit Gleichung (3) wird das benötigte Zellvolumen aus der Gesamtzahl der Zellen im Mehrschichtkolben (gezählt in Abschnitt 3.2.19) berechnet, um die erste Aussaatdichte oder Zellstocksuspension herzustellen.
- Führen Sie eine serielle Verdünnung durch, um Zellsuspensionen mit mehreren Seeding-Dichten zu erzeugen.
- Von der in Schritt 4.1.1 hergestellten Dichte 1 werden drei weitere Dichten der Zellsuspension durch serielle Verdünnung gemäß Abbildung 3 hergestellt.
- Verdünnen Sie jede Dichte im Wachstumsmedium um den Faktor 2. Geben Sie zunächst das erforderliche Endvolumen (600 μl aus dem vorherigen Beispiel) des Wachstumsmediums in drei sterile 2-ml- oder 15-ml-Zentrifugenröhrchen.
- Die Hälfte der Dichte 1 (600 μl) in eines der Röhrchen überführen und die Zellsuspension gründlich mit dem zu verdünnenden Medium vermischen. Beschriften Sie dieses Röhrchen mit Dichte 2.
- Die Hälfte der Dichte 2 (600 μl) in das nächste Röhrchen überführen und die Zellsuspension gründlich mit dem zu verdünnenden Medium vermischen. Beschriften Sie dieses Röhrchen mit Dichte 3.
- Die Hälfte der Dichte 3 (600 μl) wird in das nächste Röhrchen überführt, wobei die Zellsuspension gründlich mit dem zu verdünnenden Medium vermischt wird. Beschriften Sie dieses Röhrchen mit Dichte 4.
- Verwerfen Sie 600 μl von Dichte 4, so dass alle vier Präparate ein Endvolumen von 600 μl haben.
- Als Negativkontrolle werden 600 μl Wachstumsmedium nur in ein steriles 2-ml-Zentrifugenröhrchen gegeben. Abbildung 3 zeigt ein Schema des seriellen Verdünnungsprozesses.
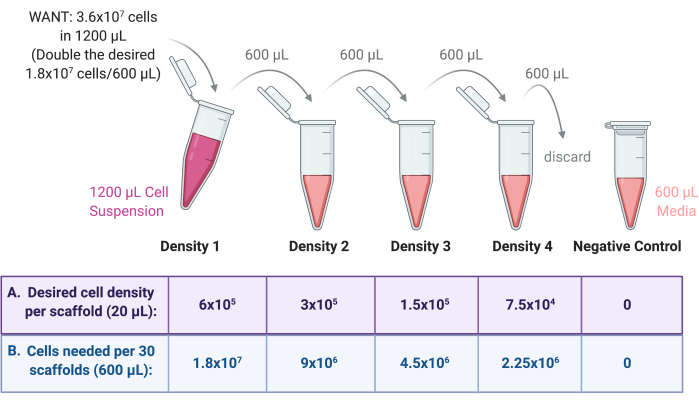
Abbildung 3: Serielle Verdünnung des Zellmaterials zur Herstellung von 4 Suspensionen für 4 verschiedene Gerüstaussaatdichten. (A) Die Zahlen können an die gewünschte Aussaatdichte pro Gerüst angepasst und (B) mit der Gesamtzahl der Gerüste pro Dichte multipliziert werden, wobei jedes Gerüst 20 μl Zellsuspension erhält. In diesem Beispiel erfordert Dichte 1 6 × 105 Zellen pro Gerüst, was 1,8 × 107 Zellen in 600 μl für 30 Gerüste entspricht. Diese Anzahl wird verdoppelt, um die serielle Verdünnung zu beginnen, da dann 600 μl übertragen und in 600 μl Wachstumsmedium im nächsten Röhrchen verdünnt werden. Dieser Prozess wird so lange fortgesetzt, bis 4 Zellsuspensionen mit einem Faktor von 2 dazwischen vorhanden sind. Eine Negativkontrolle wird durchgeführt, indem 600 μl Medium nur in ein Röhrchen gegeben werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Zugabe von Zellsuspensionen zu den Gerüsten
HINWEIS: Nehmen Sie die Gerüste (in PBS aufbewahrt) aus dem Kühlschrank und lassen Sie sie auf Raumtemperatur (RT) kommen, bevor Sie die Zellen hinzufügen.- Bringen Sie die Gerüste in PBS in die Laminar-Flow-Haube.
- Platzieren Sie die Gerüste mit einer sterilen Pinzette in nicht haftenden 24-Well-Platten mit einem Gerüst pro Wells (Abbildung 1C). Heben Sie die Gerüste vorsichtig an den Ecken an und drücken Sie sie leicht gegen die Seite des Behälters, um überschüssiges PBS zu entfernen. Fügen Sie die Gerüste mit der Hautseite nach unten in die Mitte der Vertiefungen ein (die glänzende Schichtseite des Gerüsts, mit der Vorderseite nach unten in die 24-Well-Kunststoffplatten).
- Beschriften Sie die 24-Well-Platten mit Details zur Zelllinie, relevanten Parametern (z. B. Aussaatdichte) und Zeitpunkten. Arbeiten Sie mit jeweils einer Zellaussaatdichte und lassen Sie die restlichen Dichten bis zur Verwendung im 37 °C warmen Inkubator.
- Verwenden Sie in der Laminar-Flow-Haube eine P20-Pipette und sterile Spitzen, um vorsichtig 20 μl der entsprechenden Zellsuspension in die Mitte jedes Gerüsts zu geben (Abbildung 1D). Halten Sie die Zellen gründlich in Schwebe, indem Sie sie gut mischen, während Sie die Zellen zu den Gerüsten hinzufügen. Stellen Sie sicher, dass die Aufhängung auf dem Gerüst bleibt und nicht auf den Boden des Brunnens rutscht, da dies keine Zellenbefestigung am Gerüst zulässt.
- Sobald die Zellen hinzugefügt wurden, inkubieren Sie die Platten für 3-5 Stunden (37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit), damit sich die meisten Zellen anheften können.
- Geben Sie nach der Inkubation langsam und vorsichtig 1 ml vorgewärmtes Wachstumsmedium in jede Vertiefung (Abbildung 1E). Verwenden Sie eine P1000-Pipette, um das Medium hinzuzufügen, um eine langsamere und kontrolliertere Bewegung zu ermöglichen und ein Verschieben der Gerüste zu verhindern. Wenn Sie mit einer sehr hohen Anzahl von Gerüsten arbeiten, verwenden Sie eine 10-ml-Pipette mit der Pipettenpistole auf "Tropfen" und "Niedrig".
- Inkubieren Sie die 24-Well-Platten über Nacht (37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit).
- Wartung der Zellen auf den Gerüsten
- Nach den ersten 24 Stunden der Zellanhaftung (Tag 1) werden die ausgesäten Gerüste auf neue, nicht haftende 24-Well-Platten übertragen und 1-2 ml frisches Wachstumsmedium hinzugefügt.
HINWEIS: Bei diesem Schritt werden Zellen entfernt, die auf den Boden der 24-Well-Kunststoffplatten gefallen sind, anstatt sie auf den Gerüsten wachsen zu lassen. Gerüstreplikationen, die als Tag 1 gekennzeichnet sind, werden nach 24 Stunden abgebaut, wie in Abschnitt 5 erläutert. Daher gilt die Wartung für diese Gerüste nicht. - Überwachen Sie die Scaffolds zunächst alle 2-3 Tage auf eine Farbveränderung des Nährmediums. Wenn die Zeit fortschreitet und sich die Zellen innerhalb der Gerüste vermehren, füttern Sie die Zellen häufiger.
- Entfernen Sie mit einer 10-ml-Pipettenpistole im langsamen Modus 1 ml des verbrauchten Mediums und entsorgen Sie es. Bei der Durchführung von Experimenten, die konditioniertes Medium erfordern, ist das verbrauchte Medium biologischer Replikate in einem 15-ml-Zentrifugenröhrchen aufzufangen, 2 Minuten lang bei 340 × g zu zentrifugieren, um die Zelltrümmer zu pelletieren, den Überstand in ein frisches Röhrchen zu überführen und bei -80 °C zu lagern.
- Geben Sie vorsichtig 2 ml vorgewärmtes Wachstumsmedium in die Gerüste, verwenden Sie erneut die Tropffunktion an der Pipette und legen Sie die 24-Well-Platte wieder in den Inkubator (37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit). Wiederholen Sie dies immer dann, wenn das Medium für die Dauer der gewünschten Wachstumsperiode verbraucht ist.
- Nach den ersten 24 Stunden der Zellanhaftung (Tag 1) werden die ausgesäten Gerüste auf neue, nicht haftende 24-Well-Platten übertragen und 1-2 ml frisches Wachstumsmedium hinzugefügt.
5. Gerüstabruf und Anwendungen
HINWEIS: Zu jedem Zeitpunkt können mehrere Anwendungen verwendet werden, um das Zellwachstum auf den Gerüsten zu überwachen oder Gen- und Proteinexpressionsprofile zu bewerten. Die Bedingungen für den Abruf von Gerüsten hängen von der durchzuführenden Analyse ab, wobei mehrere Abrufmethoden in den folgenden Unterabschnitten beschrieben und in Abbildung 4 demonstriert werden.
- Beurteilung der Zellviabilität innerhalb von Scaffolds
- Sterilisieren Sie das entsprechende Reagenz des Zellviabilitäts-Assays, indem Sie es durch einen 0,2-μm-Sterilfilter in ein Zentrifugenröhrchen in der Laminar-Flow-Haube filtrieren. Erwärmen Sie diese sterile Lösung zusammen mit dem vollständigen Nährmedium und dem sterilen PBS in einem 37 °C warmen Wasserbad.
- Verwenden Sie in der Laminar-Flow-Haube eine sterile Pinzette, um die zu analysierenden Scaffolds auf eine frische 24-Well-Platte zu übertragen. Beschriften Sie das Schild mit allen relevanten Details.
HINWEIS: Führen Sie die Analyse in dreifacher Ausfertigung durch. - Geben Sie 900 μl des vorgewärmten Wachstumsmediums in jede Vertiefung, gefolgt von 100 μl des sterilen Zellviabilitätsreagenzes. Schließen Sie eine Negativkontrolle ein, indem Sie 900 μl Medium und 100 μl des sterilen Zellviabilitätsreagenzes in eine Vertiefung ohne Gerüst geben. Setzen Sie den Deckel wieder auf die Platte und wiegen Sie die Platte vorsichtig ~3 Minuten lang, um das verdünnte Zellviabilitätsreagenz gleichmäßig im Well zu verteilen. Inkubieren Sie die Platte bei 37 °C, 5 % CO2 und 95 % Luftfeuchtigkeit.
HINWEIS: Die Inkubationszeiten müssen für jede Zelllinie optimiert werden. Beachten Sie die Richtlinien des Herstellers. Für Neuroblastom-Zelllinien erscheint eine Inkubation von 4-6 h optimal. - Nehmen Sie die Platte nach der Inkubation aus dem Inkubator und wiegen Sie sie einige Sekunden lang vorsichtig.
- Öffnen Sie in der Laminar-Flow-Haube eine neue, durchsichtige 96-Well-Platte. Übertragen Sie aus jeder Vertiefung in der 24-Well-Platte das inkubierte Medium und das Reagenz mit 100 μl pro Well in drei Vertiefungen der 96-Well-Platte, um technische Verdreifachungen zu erhalten.
HINWEIS: Bei diesem Transfer verbleiben 700 μl in den Vertiefungen der 24-Well-Platte. - Decken Sie die 96-Well-Platte mit Aluminiumfolie ab, um das Reagenz der Zellviabilität vor Licht zu schützen.
- Entfernen und entsorgen Sie die restlichen 700 μl des Well-Inhalts von jedem Gerüst in der 24-Well-Platte. Waschen Sie jedes Gerüst zweimal mit 1 ml sterilem PBS.
HINWEIS: Die gesamte Farbe wird nicht von den Gerüsten entfernt. Diese Gerüste können dann für weitere Anwendungen, z. B. die DNA-Quantifizierung, verwendet werden, indem sie in 1 ml 1%ige Triton X-100-Lösung in 0,1 M Natriumbicarbonat (NaHCO3)-Lösung gegeben und bei -80 °C gelagert werden (siehe Abschnitt 5.2, Abbildung 4B). - Entfernen Sie die 96-Well-Platte von der Laminar-Flow-Haube und messen Sie die Absorption jedes Wells bei Wellenlängen von 570 nm und 600 nm mit einem Mikroplatten-Reader. Zeichnen Sie die Absorptionswerte bei beiden Wellenlängen auf und befolgen Sie die Anweisungen des Herstellers, um die prozentuale Verringerung des Zellviabilitätsreagenzes durch die Zellen zu berechnen.
- Grafische und statistische Analyse der Ergebnisse der Zelllebensfähigkeit mit geeigneter Software. Geben Sie biologische Dreifachwerte ein, um Fehlerbalken zu erstellen und die Assay-Variabilität anzuzeigen.
- Um die Veränderungen der Zelllebensfähigkeit über den experimentellen Zeitraum zu untersuchen, führen Sie einen einseitigen Varianztest (ANOVA) mit mehreren Vergleichen der Mittelwerte unter Verwendung geeigneter biostatistischer Software durch.
- Geben Sie signifikante Unterschiede zwischen den Zeitpunkten in den Diagrammen als ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) und **** (P≤0,0001) an.
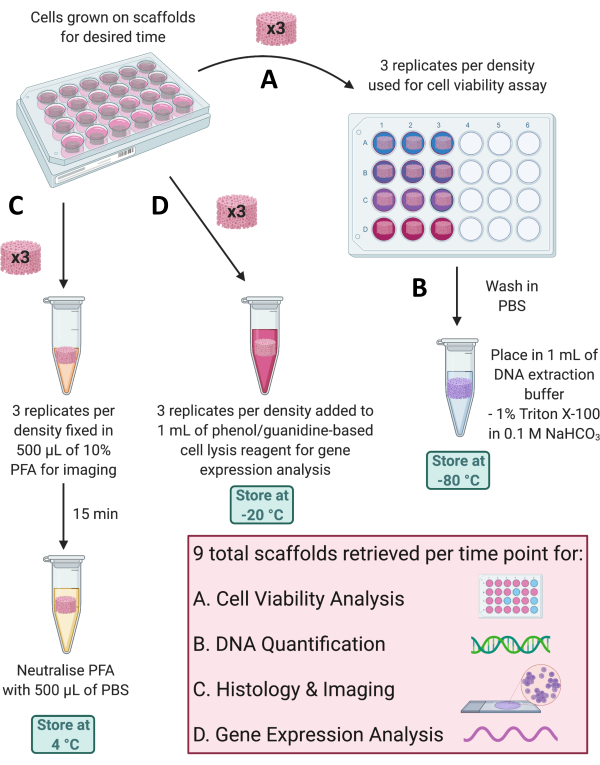
Abbildung 4: Abruf von Gerüsten für verschiedene Analysen zu jedem Zeitpunkt. (A) Für die Analyse der Zelllebensfähigkeit werden drei Gerüstreplikate entnommen. (B) Diese Gerüste können dann in PBS gewaschen, in 1% Triton X-100 in 0,1 M NaHCO3 gelegt und bei -80 °C zur DNA-Quantifizierung gelagert werden. (C) Drei weitere Replikate werden 15 min in 10%iger PFA fixiert, in PBS neutralisiert und bei 4 °C für histologische Färbung und Bildgebung gelagert. (D) Schließlich werden 3 Replikate zu einem Zelllysereagenz auf Phenol/Guanidin-Basis gegeben und bei -20 °C für die Genexpressionsanalyse gelagert. Abkürzungen: PBS = phosphatgepufferte Kochsalzlösung; PFA = Paraformaldehyd. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Quantifizierung der DNA aus den Zellen in den Scaffolds
ANMERKUNG: Wie in der Anmerkung nach Schritt 5.1.7 erwähnt, werden die Gerüste für die DNA-Quantifizierung entnommen, indem die Gerüste in 2-ml-Zentrifugenröhrchen gelegt werden, die 1 ml 1 % Triton X-100 in 0,1 M NaHCO3-Lösung enthalten, gefolgt von einer Lagerung bei -80 °C. Bevor eine DNA-Analyse durchgeführt werden kann, müssen die Zellen drei Gefrier-Auftau-Zyklen durchlaufen, um die Neuroblastomzellen entsprechend zu lysieren und DNA zur Quantifizierung freizusetzen.- Entnehmen Sie die zuvor in Triton X-100 gelagerten Proben bei -80 °C. Lassen Sie die Proben 1-3 h lang bei RT oder bis sie aufgetaut sind.
- Die Proben werden 10-20 s lang vortext und 18-24 h lang bei -80 °C oder bis sie vollständig gefroren sind. Wiederholen Sie diesen Vorgang für insgesamt drei Gefrier-Auftau-Zyklen.
- Um die DNA-Ausbeute zu maximieren, verwenden Sie einen Gewebelyser, um die Zellen in den Gerüsten aufzubrechen.
- Legen Sie eine Metallperle in das 2-ml-Zentrifugenröhrchen, das ein Gerüst in Triton X-100 enthält, und legen Sie das Röhrchen in den Adapter, um die Probe 2-3 Minuten lang mit 50 Schwingungen/Sekunde zu schütteln.
HINWEIS: Verwenden Sie Zentrifugenröhrchen mit rundem Boden, da sich die Metallperle in einem Röhrchen mit konischem Boden festsetzen kann.
- Legen Sie eine Metallperle in das 2-ml-Zentrifugenröhrchen, das ein Gerüst in Triton X-100 enthält, und legen Sie das Röhrchen in den Adapter, um die Probe 2-3 Minuten lang mit 50 Schwingungen/Sekunde zu schütteln.
- Quantifizieren Sie die DNA in der Triton X-100-Lösung durch Auftragen einer fluoreszierenden doppelsträngigen DNA-Färbung (dsDNA) und messen Sie die Emission mit einem Mikroplatten-Reader. Beachten Sie die Richtlinien des Herstellers. Verdünnen Sie die Proben entsprechend in Tris-Ethylendiamintetraessigsäure (TE)-Puffer, um 8 dsDNA-Standards durch serielle Verdünnung in TE-Puffer herzustellen (Abbildung 5).
- Bei schwarzen, undurchsichtigen 96-Well-Platten geben Sie 100 μl von jedem Standard oder jeder Probe in technischer Dreifachausführung in die Wells.
- Verdünnen Sie die fluoreszierende dsDNA-Färbung 200-fach in TE-Puffer und geben Sie mit einer Mehrkanalpipette 100 μl zu jedem Standard/jeder Probe. Die Platte in Alufolie einlegen und 5 Minuten bei RT inkubieren.
- Messen und zeichnen Sie die Fluoreszenz jedes Wells bei 520 nm auf. Befolgen Sie die Anweisungen des Herstellers, um die DNA-Konzentration in jeder Probe zu berechnen.
HINWEIS: Wenn die durchschnittliche DNA-Konzentration pro Zelle für die verwendete Zelllinie bekannt ist, können DNA-Konzentrationswerte mithilfe von Gleichung (4) in Zellzahlen umgewandelt werden. (4)
(4) - Grafische und statistische Analyse der Ergebnisse der DNA-Quantifizierung mit geeigneter Software. Geben Sie biologische Dreifachwerte ein, um Fehlerbalken zu erstellen und die Assay-Variabilität anzuzeigen.
- Um die Veränderungen der DNA-Konzentration/Zellzahl über den experimentellen Zeitraum zu untersuchen, führen Sie einen Einweg-ANOVA-Test mit mehreren Vergleichen der Mittelwerte unter Verwendung geeigneter biostatistischer Software durch.
- Geben Sie signifikante Unterschiede zwischen den Zeitpunkten in Diagrammen als ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) und **** (P≤0,0001) an.
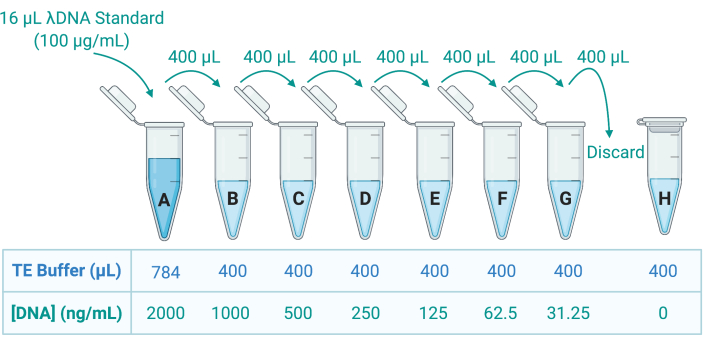
Abbildung 5: Präparation von acht DNA-Standards für die Generierung einer Standardkurve. Eine Stammlösung von λDNA wird mit 100 μg/ml bereitgestellt. Dies wird in TE-Puffer um das 50-fache verdünnt, um Standard A bei 2000 ng/ml zu erzeugen. 400 μl A werden dann in Röhrchen B überführt, das 400 μl TE-Puffer enthält; 400 μl B werden dann übertragen und 2-fach in C verdünnt, und so weiter, bis G. Standard H besteht nur aus TE-Puffer und hat daher eine DNA-Konzentration von 0 ng/ml. Abkürzung: TE = Tris-EDTA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Präparation von Scaffolds für die histologische Färbung
HINWEIS: Gerüste können für Mikroskopie- und Bildgebungszwecke entweder als ganze Gerüste für die Immunfluoreszenz (IF) oder als formalinfixierte, paraffineingebettete (FFPE) Schnitte für die histologische Färbung oder Immunhistochemie (IHC) fixiert und gefärbt werden. Dies ermöglicht die qualitative Beurteilung der Zellpenetration und -verteilung innerhalb von Scaffolds und kann zur Beurteilung der Expression von Proteinen verwendet werden.- Bereiten Sie eine 10%ige Paraformaldehyd (PFA)-Lösung in PBS vor. Stellen Sie sicher, dass genügend Lösung für ein Endvolumen von 500 μl pro Gerüst vorhanden ist.
- Erwärmen Sie diese Lösung auf 37 °C und geben Sie 500 μl in beschriftete 2-ml-Zentrifugenröhrchen für die Gerüstentnahme.
- Entfernen Sie die Gerüste von den nicht haftenden 24-Well-Platten (Schritt 4.4.4) und setzen Sie sie in die Laminar-Flow-Haube ein.
- Übertragen Sie die Gerüste mit einer sterilen Pinzette in die beschrifteten Zentrifugenröhrchen, die 10 % PFA enthalten. Lassen Sie die Gerüste 15 Minuten lang in der PFA-Lösung fixieren. Neutralisieren Sie das PFA, indem Sie 500 μl PBS in jedes Röhrchen geben und bei 4 °C lagern.
- Um die Gerüste für den Tissue Processor vorzubereiten, nehmen Sie sie aus 4 °C heraus und legen Sie sie mit einer Pinzette in Kunststoffkassetten, die mit Bleistift mit allen relevanten Angaben beschriftet sind. Legen Sie alle Kassetten in den Metallbehälter des Tissue-Prozessors.
- Beginnen Sie mit dem 12-stufigen Protokoll auf dem Gewebeprozessor, um die Gerüste zu fixieren, zu dehydrieren, zu reinigen und über Nacht mit Paraffin zu infiltrieren. Sammeln Sie die Kassetten mit den verarbeiteten Proben.
- Als nächstes betten Sie die Gerüste in Paraffinwachsblöcke ein, um die Mikrotomie der Gerüste in sehr dünne Scheiben für die Färbung zu ermöglichen.
HINWEIS: Es ist besonders wichtig, die Ausrichtung der Gerüste zu berücksichtigen, wenn sie aus den Kassetten entnommen und in Wachs eingebettet werden, da dies den Winkel beeinflusst, in dem die Bilder aufgenommen werden. Dies ist wichtig bei der Beurteilung der Zellinfiltration in die Gerüste. - Schalten Sie den Wachseinbetter und die Kühlplatte ein. Hebe den Deckel an, um den Wachsgehalt zu überprüfen. Bei Bedarf nachfüllen.
- Arbeiten Sie mit einer Probe nach der anderen, öffnen Sie die Kassette, entfernen Sie das Gerüst und zentrieren Sie es in der Kunststoffform.
- Gießen Sie heißes Wachs auf die Probe, achten Sie darauf, dass die richtige Ausrichtung beibehalten wird, und passen Sie sie bei Bedarf mit einer warmen Pinzette an, bevor das Wachs erstarrt. Gießen Sie mehr Wachs, um die Form zu füllen.
- Legen Sie den beschrifteten Kassettendeckel auf die Kunststoffform und geben Sie Wachs darauf. Lege die Form auf die Kühlplatte, um das Wachs zu verfestigen. Über Nacht bei 4 °C lagern, um sicherzustellen, dass das Paraffinwachs vor der Mikrotomie vollständig erstarrt ist.
- Zur Vorbereitung auf die Mikrotomie schalten Sie ein 35 °C warmes Wasserbad, die Trockenplatte und das Mikrotom ein.
- Setzen Sie eine Klinge in die Halterung ein und ziehen Sie den Hebel fest, um sie zu sichern.
- Stellen Sie die Beschnitt- und Querschnittsdicke ein, in der Regel 5 mm für Gerüstabschnitte.
- Entfernen Sie das FFPE-Gerüst aus der Form, befestigen Sie es in der Halterung an der Vorderseite des Mikrotoms und schneiden Sie das überschüssige Wachs vorsichtig an den Rändern der Probe ab, bevor Sie Abschnitte schneiden.
- Beginnen Sie mit dem Schneiden in den Wachsblock, indem Sie den Hebel des Mikrotoms drehen, um eine gleichmäßige Bewegung zu gewährleisten.
- Sammeln Sie bandartige Abschnitte, etwa 3 Gerüstabschnitte auf einmal, und legen Sie sie vorsichtig in das 35 °C warme Wasserbad, um Falten zu entfernen. Trennen Sie die Abschnitte vorsichtig im Wasserbad mit einer Pinzette.
- Heben Sie bei polysinbeschichteten Objektträgern aus Glas jeden Abschnitt aus dem Wasserbad, so dass der Abschnitt in der Mitte des Objektträgers sitzt. Beschriften Sie jede Folie mit einem Bleistift.
- Legen Sie die Objektträger auf die Trockenplatte oder in einen 60 °C heißen Trockenschrank. Lagern Sie sie nach dem Trocknen bei 4 °C und fahren Sie mit der erforderlichen histologischen oder IHC-Färbung fort.
- Entnahme von Scaffolds für die Genexpressionsanalyse
- Entfernen Sie die Gerüste von den nicht haftenden 24-Well-Platten aus dem Inkubator (Schritt 4.4.4) und setzen Sie sie in die Laminar-Flow-Haube ein.
- Übertragen Sie die Gerüste mit einer sterilen Pinzette in frisch beschriftete 2-ml-Zentrifugenröhrchen.
- Geben Sie in einem Abzug 1 ml eines Zelllysereagenzes auf Phenol-/Guanidin-Basis in jedes Röhrchen, um die Zellen in Gerüsten zu lysieren und die Rückgewinnung hochwertiger RNA zu ermöglichen.
- Bewahren Sie die Röhrchen bei -20 °C auf, bis Sie bereit sind, die RNA-Extraktion mit einem geeigneten Kit durchzuführen. Mit einer standardmäßigen quantitativen Reverse-Transkriptase-Kettenreaktion (RT-qPCR)17 ist die Genexpression in den Zellen in den Gerüsten zu beurteilen.
Ergebnisse
Das hier beschriebene kollagenbasierte Gerüstmodell hat viele Anwendungen, die von der Untersuchung der Biologie des Neuroblastoms bis hin zum Screening von Krebstherapeutika in einer Umgebung reichen, die nativen Tumoren physiologisch ähnlicher ist als herkömmliche 2D-Zellkulturen. Bevor eine bestimmte Forschungsfrage getestet wird, ist es entscheidend, eine vollständige Charakterisierung der Zelladhäsion, -proliferation und -infiltration innerhalb des gewünschten experimentellen Zeitrahmens zu erhalten. Die Wachstumsbedingungen hängen von der Biologie der jeweiligen Zelllinie ab. Wichtig ist, dass mehrere Methoden zur Beurteilung des Zellwachstums implementiert werden müssen, um optimale Bedingungen und eine robuste Leistung zu ermitteln.
Hier wurde die Lebensfähigkeit von Neuroblastomzellen, die auf Scaffolds gezüchtet wurden, mit einem kolorimetrischen Zellviabilitätstest untersucht. Dieser Assay kann während des gesamten experimentellen Zeitraums beliebig oft durchgeführt werden. Für das beschriebene Experiment wurde an den Tagen 1, 7 und 14 eine Bewertung der Zellviabilität für zwei Neuroblastom-Zelllinien, KellyLuc und IMR32, durchgeführt, die auf Coll-I-nHA-Gerüsten mit 4 verschiedenen Dichten gezüchtet wurden (Abbildung 6). Die Viabilität an Tag 1 wurde als Ausgangsbasis für den Vergleich aller nachfolgenden Messungen festgelegt. Die Reduktionsrate des Zellviabilitätsreagenzes spiegelt die Zellbiologie und die Wachstumseigenschaften der einzelnen Zelllinien wider, einschließlich ihrer Proliferationsraten und ihres Stoffwechsels. Es wurde eine Korrelation zwischen der Anzahl der auf den Gerüsten ausgesäten Zellen und dem Grad der Reduktion erwartet. In diesem Experiment nahm die Reduktion des Zellviabilitätsreagenzes im Allgemeinen mit jedem Zeitpunkt für beide Zelllinien bei allen Dichten zu, wie erwartet.
Jede Dichte wurde dann einzeln für beide Zelllinien bewertet, um die Reduktion über verschiedene Zeitpunkte hinweg zu vergleichen. Eine unidirektionale ANOVA mit dem Tukey-Mehrfachvergleichstest wurde durchgeführt, um signifikante Unterschiede in der Reduktion zwischen den Zeitpunkten zu erkennen (Abbildung 7). Für beide Zelllinien und alle Aussaatdichten gab es einen signifikanten Anstieg (P<0,05) in der Reduktion des Zellviabilitätsreagenzes im Vergleich von Tag 1 und Tag 14. Dies deutete auf einen signifikanten Anstieg der metabolisch aktiven Zellen hin, die auf den Gerüsten vorhanden waren. Dieser Anstieg war bei der Bewertung der 7-Tage-Intervalle (Tag 1 vs. Tag 7, Tag 7 vs. Tag 14) nicht in allen Fällen signifikant, was die Bedeutung der Optimierung der Aussaatdichte zur Erreichung des gewünschten Wachstumsfensters zeigt.
Um die Ergebnisse des Zellviabilitäts-Assays zu unterstützen, kann das Zellwachstum auf Scaffolds auch indirekt über die Quantifizierung von dsDNA gemessen werden, die aus Scaffolds mit einer fluoreszierenden dsDNA-Färbung extrahiert wurde (Abbildung 8A). Wie die Lebensfähigkeit von Zellen kann auch die DNA-Quantifizierung innerhalb der experimentellen Zeitachse beliebig oft durchgeführt werden. Diese Analyse erfordert jedoch die vollständige Entnahme von Gerüsten und die Beendigung des Zellwachstums und muss daher in die experimentelle Planung einbezogen werden, wie in Abschnitt 1 erläutert. Für dieses Experiment wurde die DNA an den Tagen 1, 7 und 14 für zwei Neuroblastom-Zelllinien, KellyLuc und IMR32, quantifiziert, die auf Coll-I-nHA-Gerüsten in 4 verschiedenen Dichten gezüchtet wurden. Da für diese Zelllinien die durchschnittliche Konzentration von dsDNA pro Zelle bekannt ist, konnte aus der quantifizierten DNA die Anzahl der Zellen pro Probe abgeleitet werden (Abbildung 8B).
Die DNA-Quantifizierung führte zu einer höheren Variabilität zwischen biologischen Replikaten als die Bewertung der Zelllebensfähigkeit, nahm aber im Allgemeinen zu jedem Zeitpunkt zu, wobei die höchsten Werte an Tag 14 quantifiziert wurden. IMR32-Zellen scheinen auf Coll-I-nHA-Gerüsten eine höhere Zellzahl zu erreichen, was durch die DNA-Konzentration angezeigt wird, als KellyLuc-Zellen. Jede Dichte wurde dann einzeln für die beiden Zelllinien bewertet, um die Reduktion über die Zeitpunkte hinweg zu vergleichen. Eine unidirektionale ANOVA mit dem Tukey-Mehrfachvergleichstest wurde durchgeführt, um signifikante Unterschiede in der Reduktion zwischen den Zeitpunkten zu erkennen (Abbildung 8B).
Für beide Zelllinien und alle Aussaatdichten gab es einen signifikanten Anstieg (P<0,05) der Zellzahlen beim Vergleich von Tag 1 und Tag 14, mit Ausnahme von KellyLuc bei der Aussaatdichte 4 (1 × 105 Zellen/Gerüst), die zu keinem der Zeitpunkte zu signifikanten Anstiegen führten. Ähnlich wie bei den Ergebnissen der Zellviabilität waren die Anstiege bei der Beurteilung der 7-Tage-Intervalle (Tag 1 vs. Tag 7, Tag 7 vs. Tag 14) nicht in allen Fällen signifikant. Beim Vergleich der Zeitpunkte für die Zelllebensfähigkeit und die DNA-Quantifizierung gab es einige leichte Unterschiede zwischen den beiden Analysen. Insgesamt wurden jedoch ähnliche Trends beobachtet, wobei die Mittelwerte zwischen den 7-Tage-Intervallen für die meisten Dichten anstiegen. Dies zeigt, wie wichtig es ist, das Zellwachstum mit mehr als einer Methode zu überwachen.
Als nächstes wurde eine visuelle Beurteilung der Zellwachstumsmorphologie und -verteilung auf den Gerüsten durchgeführt, die sowohl die traditionelle Hämatoxylin- und Eosinfärbung (H&E) als auch IHC umfasste. Es wird erwartet, dass die unterschiedlichen Wachstumsmuster einzelner Zelllinien zu unterschiedlichen räumlichen Anordnungen auf Gerüsten führen, einschließlich unterschiedlicher Penetrationsgrade in das Gerüst und Zellclustering. Die Scaffolds wurden formalinfixiert, in Paraffin eingebettet und in 5-mm-Abschnitte geschnitten (Abbildung 9A), wodurch die Scaffolds für verschiedene Visualisierungstechniken, einschließlich histologischer Färbung und IHC, vorbereitet wurden.
Die routinemäßige H&E-Färbung wurde an den Tagen 1, 7 und 14 auf Kelly-, KellyCis83- und IMR32-Zellen angewendet, die auf kollagenbasierten Gerüsten gezüchtet wurden (Abbildung 9B). Dies ermöglichte es, die räumliche Orientierung der Zellen auf zwei kollagenbasierten Gerüsten über einen Zeitraum von 14 Tagen sichtbar zu machen. Cisplatin-sensitive Kelly-Zellen und resistente KellyCis83-Zellen wurden sowohl auf Coll-I-nHA-Gerüsten (Abbildung 9B, i) als auch auf Coll-I-GAG-Gerüsten (Abbildung 9B, ii) gezüchtet. In Übereinstimmung mit zuvor veröffentlichten Daten wuchsen KellyCis83-Zellen mit einer höheren Rate und infiltrierten tiefer in beide Gerüstzusammensetzungen als die weniger invasive Kelly-Zelllinie. Die H&E-Färbung einer anderen Neuroblastom-Zelllinie, IMR32, die auf Coll-I-nHA gezüchtet wurde, zeigt ein kontrastierendes Wachstumsmuster (Abbildung 9B, iii). Diese Zelllinie wuchs über den Zeitraum von 14 Tagen in großen, dicht gepackten Clustern auf den Kollagengerüsten. Die konfokale Hellfeldmikroskopie kann verwendet werden, um die poröse Architektur von kollagenbasierten Gerüsten (Abbildung 9C) aufgrund der Autofluoreszenz von Kollagenfasern sichtbar zu machen.
Wir färbten Zellen mit Phalloidin, das auf das Zytoskelett-Aktin abzielt, und der nukleären Gegenfärbung 4′,6-Diamidino-2-phenylindol (DAPI), um spezifische Zellmerkmale während der gesamten experimentellen Zeitspanne zu überwachen. Mit dieser Technik wurde eine Fülle von Aktin in Kelly- und KellyCis83-Zellen auf Coll-I-GAG-Gerüsten beobachtet (Abbildung 9D). Diese Ergebnisse zeigen, wie mehrere bildgebende Verfahren verwendet werden können, um mit diesem Protokoll ortsaufgelöste Informationen aus Neuroblastomzellen abzuleiten, die auf Gerüsten gezüchtet wurden. Diese Charakterisierung von Zellwachstumsmustern auf kollagenbasierten Gerüsten über einen bestimmten Zeitraum wird das Verständnis und die Interpretation nachgeschalteter biochemischer Assays verbessern.
Die Proteinexpression von Zellen, die auf kollagenbasierten Gerüsten gezüchtet wurden, kann analysiert werden, um die zelluläre Aktivität mit In-vivo-Szenarien zu vergleichen. Zuvor veröffentlichte Daten untersuchten die Expression von Chromogranin A (CgA) als Surrogatmarker für Neuroblastome durch KellyLuc- und KellyCis83Luc-Zellen, die in Zellmonolayern gezüchtet wurden, sowie auf Coll-I-nHA- und Coll-I-GAG-Gerüsten (Abbildung 10). CgA wurde in den konditionierten Medien mit einem Enzyme-Linked Immunosorbent Assay (ELISA) bestimmt (Abbildung 10A). CgA wird in der aggressiveren chemoresistenten KellyCis83-Zelllinie mit einer höheren Rate sezerniert als in Kelly (Abbildung 10B,C). Dies war an Tag 7 sowohl auf Coll-I-GAG- als auch auf Coll-I-nHA-Gerüsten signifikant (P<0,05), während es zu diesem Zeitpunkt keinen signifikanten Unterschied für Zellen gab, die als Monolayer durch konventionelle 2D-Kultur gezüchtet wurden.
Diese Ergebnisse unterstreichen auch die begrenzte experimentelle Zeitspanne bei der Züchtung von Zellen in einer Monoschicht, wobei sich nur 7 Tage Wachstum als möglich erwiesen, bevor die Zellen Konfluenz erreichen. Das Wachstum von Zellen auf Gerüsten überwindet diese Einschränkung, da sie über einen längeren Zeitraum unter physiologisch relevanteren Bedingungen erhalten werden können. Die obige Kombination von Techniken zur Gewinnung von Informationen über die Lebensfähigkeit von Zellen, den DNA-Gehalt, die zelluläre Morphologie und räumliche Anordnung sowie Expressionsprofile erleichtert die Beurteilung des Wachstums von Neuroblastomzellen auf einer Reihe von kollagenbasierten Gerüsten. Dieses Protokoll kann auch leicht angepasst werden, um spezifische experimentelle Anforderungen und gewünschte Anwendungen zu erfüllen.
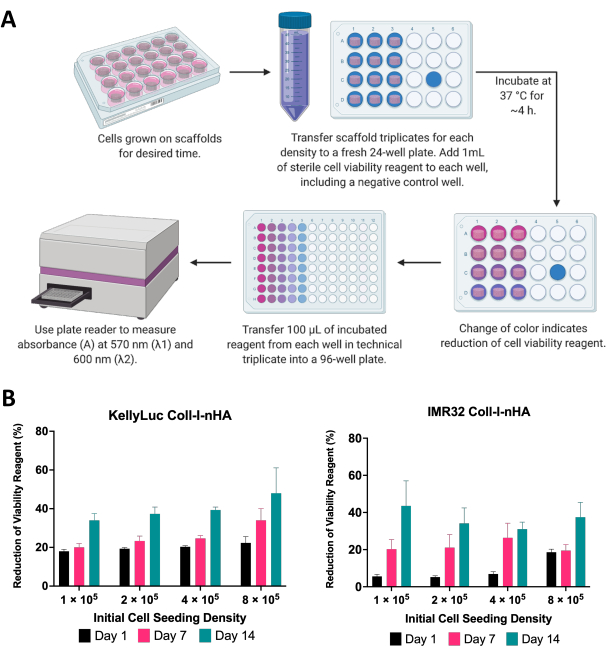
Abbildung 6: Analyse der Zellviabilität. (A) Allgemeines Verfahren zur Messung der Lebensfähigkeit von Neuroblastomzellen auf kollagenbasierten Gerüsten unter Verwendung eines kolorimetrischen Zellviabilitätsassays. Die Inkubationszeit muss für jede neue Zelllinie unter Bezugnahme auf die Richtlinien des Herstellers optimiert werden. (B) Prozentuale Reduktion des Zellviabilitätsreagenzes durch KellyLuc- und IMR32-Zellen, die auf Coll-I-nHA-Gerüsten bei vier verschiedenen anfänglichen Aussaatdichten gezüchtet wurden, gemessen an den Tagen 1, 7 und 14. Die Proben wurden in biologischer Dreifachausführung mit Fehlerbalken bewertet, die die Standardabweichung darstellten. Abkürzungen: nHA = Nanohydroxylapatit; Coll-I-nHA = Kollagengerüste, die mit nHA ergänzt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
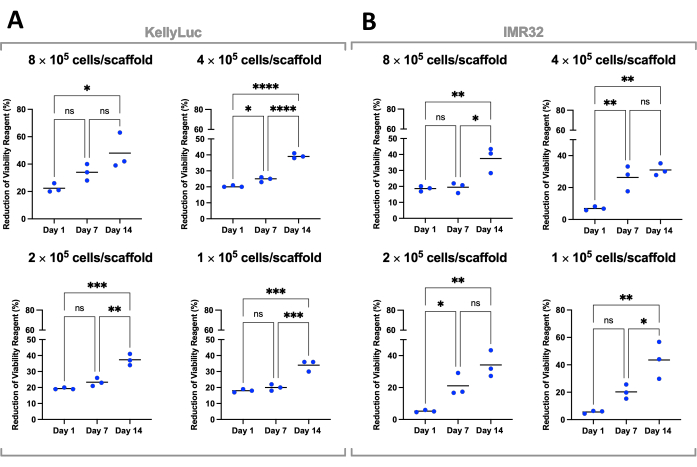
Abbildung 7: Zelllebensfähigkeit durch Aussaatdichte für Zellen, die über einen Zeitraum von 14 Tagen auf Coll-I-nHA gezüchtet wurden. (A) KellyLuc; (B) IMR32. Betitelte Zellnummern beziehen sich auf die anfängliche Zellaussaatdichte auf den Gerüsten an Tag 0. Die Proben wurden in biologischem Dreifach bewertet, das durch dreifache Punkte gekennzeichnet ist, wobei die Balken den Mittelwert darstellen. Die Einweg-ANOVA mit Mehrfachvergleichen wurde verwendet, um signifikante Unterschiede in der prozentualen Reduktion der Zellviabilität der Reagenzien über die drei Zeitpunkte zu erkennen, die in den Diagrammen vermerkt sind (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abkürzungen: nHA = Nanohydroxylapatit; Coll-I-nHA = Kollagengerüste, die mit nHA ergänzt werden; ANOVA = Varianzanalyse; ns = nicht signifikant. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
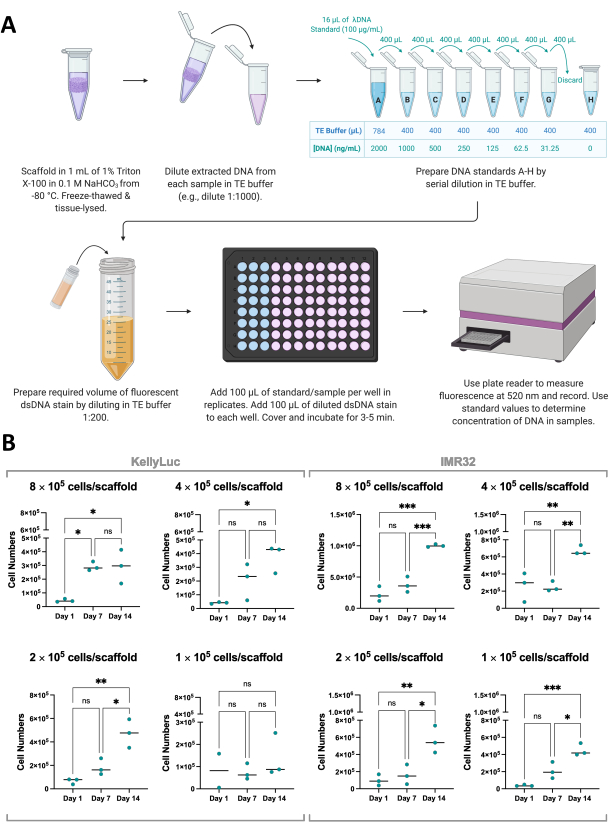
Abbildung 8: Quantifizierung von DNA, die aus Zellen in Gerüsten extrahiert wurde. (A) Verfahren zur Quantifizierung von dsDNA aus Zellen, die auf kollagenbasierten Gerüsten gezüchtet wurden, unter Verwendung einer fluoreszierenden dsDNA-Färbung. (B) Zellzahlen aus der DNA-Quantifizierungsanalyse durch Aussaatdichte für KellyLuc- und IMR32-Zellen, die über einen Zeitraum von 14 Tagen auf Coll-I-nHA gezüchtet wurden. Betitelte Zellnummern beziehen sich auf die anfängliche Zellaussaatdichte auf Gerüsten an Tag 0. Die Proben wurden in biologischem Dreifach bewertet, das durch dreifache Punkte gekennzeichnet ist, wobei die Balken den Mittelwert darstellen. Die Einweg-ANOVA mit Mehrfachvergleichen wurde verwendet, um signifikante Unterschiede in der Zellzahl über die drei Zeitpunkte hinweg zu erkennen, die in den Diagrammen vermerkt sind (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abkürzungen: nHA = Nanohydroxylapatit; Coll-I-nHA = Kollagengerüste, die mit nHA ergänzt werden; dsDNA = doppelsträngige DNA; TE = Tris-EDTA; ANOVA = Varianzanalyse; ns = nicht signifikant. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
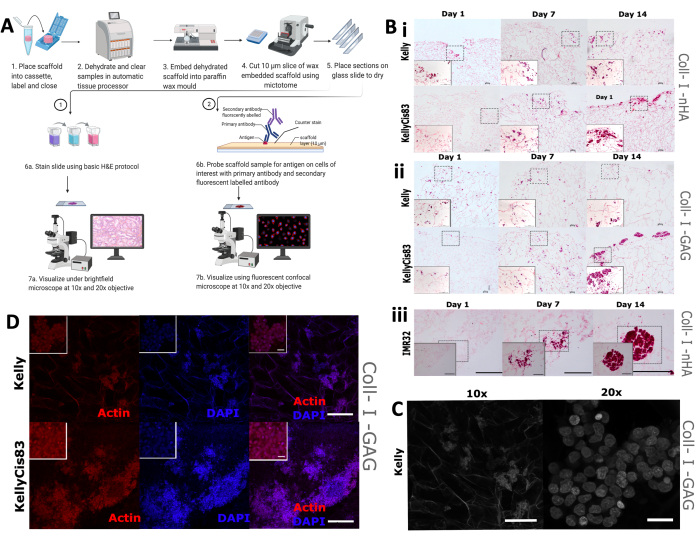
Abbildung 9: Gewebeverarbeitungsschritte für die immunhistochemische Analyse von Scaffolds. (A) Schematische Darstellung des Protokolls für die Verarbeitung von Scaffolds für die Bildanalyse. Dieses Verfahren ermöglicht die routinemäßige histologische Färbung und die spezifische Antikörpersondierung mit Primärantikörpern und fluoreszenzmarkierten Sekundärantikörpern. (B) Repräsentative Bilder von drei Neuroblastom-Zelllinien, die einer H&E-Färbung unterzogen wurden. H&E-Bilder werden an den Tagen 1, 7 und 14 aufgenommen, um die Wachstumsmuster über den Zeitraum des Experiments zu überwachen. Maßstabsleiste = 200 μm. Gestrichelte Quadrate stellen den Bereich dar, der für vergrößerte 20-fache Bilder am unteren linken Rand ausgewählt wurde. Maßstabsbalken = 20 μm. (i und ii) H&E von Kelly- und KellyCis83-Neuroblastom-Zelllinien (oberes bzw. unteres Bild) auf zwei Arten von kollagenbasierten Gerüsten. (iii) H&E der IMR32-Zelllinie, die das geclusterte Zellwachstum auf dem Coll-I-nHA-Gerüst repräsentiert. (C) Repräsentatives Bild der Kelly-Zelllinie, konfokale Hellfeldmikroskopie. Die Kollagen-Autofluoreszenz ermöglicht die Visualisierung des porösen Gerüsts. 10x Maßstabsleiste = 200 μm, 20x Maßstabsleiste = 20 μm. (D) Repräsentative Aufnahme von eingebetteten Gerüsten mit anschließender Analyse mittels IHC mit Phalloidin und DAPI bei 10-facher Vergrößerung, Maßstabsleiste = 200 μm. Kleinere Innenquadrate stellen vergrößerte Bilder dar (20x), Maßstabsleiste = 20 μm. Abkürzungen: nHA = Nanohydroxylapatit; Coll-I-nHA = Kollagengerüste, die mit nHA ergänzt werden; GAG = Glykosaminoglykan; Coll-I-GAG = Kollagengerüste, ergänzt mit Chondroitin-6-sulfat; H&E = Hämatoxylin und Eosin; IHC = Immunhistochemie; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
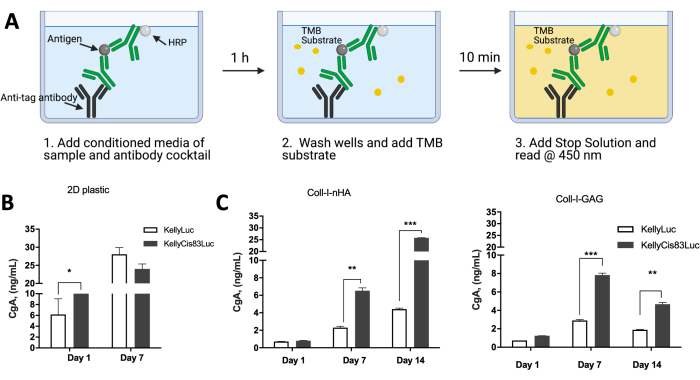
Abbildung 10: Proteinexpression von Neuroblastomzellen, die auf 3D-Kollagen-basierten Scaffolds gezüchtet wurden, im Vergleich zu 2D-Kunststoff. (A) Eine schematische Darstellung der Durchführung des CgA-ELISA auf konditionierten Medien von Zellen, die auf 2D-Kunststoff- oder 3D-Kollagen-basierten Gerüsten gezüchtet wurden. (B) CgA-Proteinexpressionsniveaus, die aus konditionierten Medien von Zellen entnommen werden, die auf einer 2D-Kunststoffmonoschicht gezüchtet wurden. Da die Zellen nach 7 Tagen die Konfluenz erreichten, war der 14-Tage-Zeitpunkt nicht lesbar. An Tag 7 auf Plastik gab es keinen signifikanten Unterschied in den CgA-Spiegeln zwischen Kelly- und KellyCis83-Zelllinien. (C) CgA-ELISA, durchgeführt mit konditionierten Medien von Zellen, die an 14 aufeinanderfolgenden Tagen auf Kollagen-basierten Gerüsten gezüchtet wurden. An Tag 7 sind die CgA-Spiegel auf beiden Kollagengerüsten in der aggressiveren KellyCis83-Zelllinie höher, was auf physiologisch relevantere CgA-Spiegel in der 3D-Matrix im Vergleich zur 2D-Monoschicht hinweist. Diese Abbildung wurde von Curtin et al.17 modifiziert. Abkürzungen: 3D = dreidimensional; 2D = zweidimensional; CgA = chromogranin A; ELISA = Enzyme-linked Immunosorbent Assay; nHA = Nanohydroxylapatit; Coll-I-nHA = Kollagengerüste, die mit nHA ergänzt werden; GAG = Glykosaminoglykan; Coll-I-GAG = Kollagengerüste, ergänzt mit Chondroitin-6-sulfat; TMB = 3,3',5,5'-Tetramethylbenzidin; HRP = Meerrettichperoxidase. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Das 3D-Gerüst-Krebszellmodell hat sich als wertvolles und vielseitiges Werkzeug erwiesen, um mechanistische Einblicke in das Wachstum, die Lebensfähigkeit und die Infiltration von Neuroblastomzellen in einem vereinfachten TME32 zu gewinnen. Das hier beschriebene 3D-Neuroblastom-Modell ahmt die minimale TME nach und liefert physiologisch relevantere Daten als eine 2D-Monolayer-Kultur. Ein großer Nachteil der 3D-Zellkultur ist die erhöhte experimentelle Komplexität und der längere Zeitrahmen. Hier wird ein optimiertes Protokoll für die Aussaat, das Wachstum und die Erhaltung von Neuroblastomzellen auf kollagenbasierten Gerüsten beschrieben, gefolgt von nachgelagerten Analysen und Anwendungen, die eine robuste Charakterisierung des Zellwachstums ermöglichen. Unser Ziel war es, Einblicke in die optimale Zellaussaatdichte für die Gerüste zu gewinnen, um eine vorhersagbare und kontrollierbare Umgebung für die Bewertung von Krebsmedikamenten in einem zügigen 14-tägigen Versuchsfenster zu schaffen. Die Kombination all dieser beschriebenen einfachen Protokolle ermöglicht eine abgerundete Beurteilung des Neuroblastom-Zellwachstums im Gerüst-basierten In-vitro-Kultursystem .
Die kritischen Punkte im Protokollaufbau wurden hervorgehoben, damit die Wissenschaftler diese in ihren Labors schnell etablieren können. Zum Beispiel ermöglichen die angegebenen Inkubationszeiten für eine bessere Leistung des kolorimetrischen Zellviabilitätstests ein tieferes Eindringen des Reagenzes in die Gerüstporen, um alle Zellen zu erreichen. Darüber hinaus ist die fluoreszierende dsDNA-Färbetechnik robust und unkompliziert; Die Freisetzung der DNA aus den Gerüsten erfordert jedoch eine starke Zelllyse, da die Zellen in Kollagenfasern "gefangen" sind.
Mit dem beschriebenen einfachen DNA-Quantifizierungsassay können wir mit diesem Modell die logarithmische Wachstumsphase auf kollagenbasierten Gerüsten für das Screening von Krebsmedikamenten identifizieren. In der beschriebenen Versuchsumgebung wurden 4 anfängliche Zellaussaatdichten mit einem Gesamtzeitraum von 14 Tagen und Analysezeitpunkten an den Tagen 1, 7 und 14 verwendet. Wir haben festgestellt, dass KellyLuc-Zellen, die bei 4 × 105 Zellen/Gerüst ausgesät wurden, das signifikanteste aktive Proliferationsfenster zwischen den Tagen 7 und 14 haben. Diese logarithmischen Wachstumsdaten ermöglichen eine zuverlässige Interpretation verschiedener Zellzytotoxizitätsexperimente. Es eliminiert Spekulationen über einen Rückgang des Wachstums oder des Zelltods, der auf unterdrücktes Wachstum auf der porösen 3D-Plattform und nicht auf Arzneimitteltoxizitäten zurückzuführen ist. Die Zellviabilität ist auch eine weit verbreitete Bewertung für die Eignung von 3D-Plattformen zur Unterstützung des Wachstums verschiedener Zelltypen33,34. Obwohl es viele Assays zur Messung der Zelllebensfähigkeit gibt, einschließlich Lebend-/Totfärbung, ATP-Messung und Proliferationsassays, haben wir festgestellt, dass die Verwendung des kolorimetrischen Zellviabilitätstests von Alamar Blue eine einfache und effektive Technik zur Unterstützung von DNA-Quantifizierungsdaten ist.
Die kombinierte Verwendung von DNA-Quantifizierung und Zellviabilität lieferte ergänzende Beweise dafür, dass die optimale Dichte zur Aussaat von Zellen auf dem Gerüst, um ein kontinuierliches Wachstum über einen Zeitraum von 14 Tagen zu erreichen, im Durchschnitt 2-4 × 105 Zellen/Gerüst beträgt. Dieses Protokoll kann jedoch leicht angepasst werden, um unterschiedliche experimentelle Zeitrahmen, Analysezeitpunkte und nachgelagerte Anwendungen zu erfüllen. Obwohl dieses Protokoll die Bewertung des Monokultur-Zellwachstums von Neuroblastomzellen auf Scaffolds beschreibt, sind die Scaffolds leicht für die Verwendung als Plattform für die Co-Kultur geeignet, beschrieben von do Amaral et al., die Kollagen-GAG-Scaffolds zur Co-Kultivierung von Keratinozyten und Fibroblasten in einer Untersuchung der Wundheilung verwendeten35.
Das beschriebene 3D-Modell ermöglicht die Visualisierung des Zellwachstums und der Infiltration mit verschiedenen bekannten Techniken, wie z.B. Immunfluoreszenz und Standard-H&E. Aufgrund der Vielfalt der Zellmorphologie und der Wachstumsmuster auf Gerüsten ist es wichtig, die Zellen zusammen mit der Charakterisierung des Wachstums mit biochemischen Assays zu visualisieren. Das Verständnis des Wachstumsmusters kann Erkenntnisse über das Wachstumsverhalten und das zukünftige Ansprechen auf Krebsmedikamente liefern. Zum Beispiel führt das Wachstum von IMR32 mittels DNA-Quantifizierung zu ähnlichen Mustern wie bei Kelly, obwohl IMR32 bei der Visualisierung mit H&E in größeren Clustern wächst als Kelly, das ein stärker verteiltes Wachstum aufwies (Abbildung 9). Diese unterschiedlichen Wachstumsmuster von Zelllinien in Scaffolds spiegeln das klinische Szenario der Tumorheterogenität wider. Die Untersuchung des Ansprechens auf Krebsmedikamente anhand eines Panels von Zelllinien mit unterschiedlichen Morphologien in 3D-Gerüsten wird den prädiktiven Wert für das Ansprechen von Patienten auf dieselben Medikamente erhöhen.
Der Nachweis der Gen- oder Proteinexpression kann auch mit anderen Ansätzen wie RT-qPCR oder ELISA durchgeführt werden, wenn das interessierende Protein sezerniert wird. Ein Surrogatmarker für die Progression des Neuroblastoms, Chromogranin A (CgA)36, wurde verwendet, um das Wachstum von Neuroblastomzellen zusätzlich in 3D zu charakterisieren. Wie in früheren Arbeiten17 beschrieben, nahm die CgA-Sekretion mit der Proliferation der Zellen zu (Abbildung 10). Während die Monolayer-Zellkultur diesen Anstieg nicht erfassen konnte, da die Proliferation dazu führte, dass die Zellen in den Kulturschalen eine vollständige Konfluenz erreichten, ermöglichte die Verwendung der 3D-Kollagengerüste eine längere Beurteilung der CgA-Sekretion.
Dieses 3D-In-vitro-Modell ist möglicherweise nicht für alle Forschungsfragen geeignet, um die Biologie des Neuroblastoms und das Ansprechen auf Therapeutika zu untersuchen. Eine der Einschränkungen ist die ungleichmäßige Zelldurchdringung innerhalb von Gerüsten und die Bildung von Zellclustern unterschiedlicher Größe, die von einer bestimmten Zelllinie abhängt und zu einer unkontrollierbaren Diffusion von Nährstoffen und Testmedikamenten führen kann. Diese Eigenschaft wirkt sich auf die Robustheit des therapeutischen Screenings aus. Trotz dieser Einschränkung ist es jedoch wichtig zu berücksichtigen, dass native Tumore auch in Größe und Krebszellverteilung heterogen sind und viele andere Zelltypen innerhalb des Tumorgewebes enthalten. Um diese Einschränkung zu überwinden, schlagen wir vor, jedes zellbesiedelte Gerüst als einzelnes Mikrogewebe zu verwenden, für das die folgenden Parameter optimiert werden: (a) Inkubationszeiten, damit das Zellviabilitätsreagenz die Zellen und Zellcluster erreicht, und (b) Lyse der Zellen im Triton X-100-Puffer durch Vorprozessierung von Zellen auf Gerüsten mit einem Gewebelyser, um die DNA der tief im Gerüst enthaltenen Zellen freizusetzen.
Eine weitere technische Einschränkung dieses Protokolls ist das Fehlen mechanischer Tests jeder Charge neu hergestellter Gerüste für dieses Modell. Die Verwendung des robusten Herstellungsprozesses der Gerüste, die in Bezug auf die physikalischen und chemischen Eigenschaften der Gerüste, wie Druck- und Zugmodul, Porosität und visuelle Porenstruktur sowie Homogenität, umfassend charakterisiert wurden, stellt jedoch sicher, dass die Gerüstqualitäten über die Chargen21, 24, 27, 30, 37 hinweg erhalten bleiben.
Zusammenfassend wird in dieser Arbeit eine Reihe einfacher Methoden zur Analyse des Zellwachstums auf kollagenbasierten Scaffolds vorgestellt. Sowohl der experimentelle Zeitplan als auch die Analysepunkte können je nach den spezifischen Forschungsfragen ausgetauscht werden. Dieses Protokoll ist auch auf andere Zelltypen anpassbar. Die oben gezeigten Ergebnisse liefern Hinweise darauf, wie diese Zusammenstellung von Methoden einen Einblick in die optimale Aussaatdichte für verschiedene Neuroblastom-Zelllinien gab, um ein kontinuierliches Wachstum über 14 Tage zu erzeugen. Die Zusammenführung der Ergebnisse aller Methoden in diesem Protokoll führt zu einem besseren Verständnis des Zellwachstums innerhalb der 3D-Kollagenmatrix. Die zukünftige Nutzung dieses Modells wird wahrscheinlich Kokultursysteme umfassen, die spezifisch für das Neuroblastom TME sind, und die Erprobung verschiedener neuartiger Krebsmedikamente.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde vom National Children's Research Centre (NCRC), dem Irish Research Council (IRC) und Neuroblastoma UK unterstützt. Die Illustrationen wurden mit BioRender erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Cells | |||
| IMR-32 | ATCC | CCL-127 | |
| Kelly | ECACC | 82110411 | |
| KellyCis83 | Made in lab – derived from Kelly (Piskareva et al., 2015) | - | Increasing exposure to cisplatin. Cross resistance acquired |
| SH-SY5Y | ATCC | CRL-2266 | |
| Disposable | |||
| 0.22 µm syringe filter | Millex | SLHP033RS | |
| 1.5 mL Eppendorf tube | Eppendorf | 0030 120.086 | |
| 100 mL sterile Pot | Starstedt | - | |
| 10 mL plastic pipette | Cellstar | 607 180 | |
| 15 mL Falcon tube | Starstedt | 62.554.502 | |
| 25 mL plastic pipette | Cellstar | 760 180 | |
| 50 mL Falcon tube | Starstedt | 62.547.254 | |
| 5 mL plastic pipette | Cellstar | 606 180 | |
| 6 mm Biopsy punches | Kai Medical | BP-60F | |
| Aluminium foil | - | - | |
| Cover Slip | Menzel-Glaser | - | |
| HYPERflask | Corning | CLS10030 | |
| Microscope slides | Thermo Scientific | J1840AMNT | |
| Opaque black 96-well plate | Costar | 3915 | |
| Sterile P10 tips | Starlab | S1121-3810 | |
| Sterile P1000 tips | Starlab | S1122-1830 | |
| Sterile P20 tips | Starlab | S1123-1810 | |
| Sterile P200 tips | Starlab | S1120-8810 | |
| T-175 (175 cm2 flask) | Sarstedt | 83.3912 | |
| T-75 (75 cm2 flask) | Sarstedt | 83.3911.302 | |
| Translucent clear 96 well plate | Cellstar | 655180 | |
| Translucent non-adherent 24 well plates | Cellstar | 83.3922.500 | |
| Equipment | |||
| Autoclave | Astell | - | |
| Automatic tissue processor | Leica | TP1020 | |
| Centrifuge 5804 | Eppendorf | - | |
| Hemocytometer | Hausser Scientific | - | |
| Incubator | ThermoScientific | - | |
| Microtome | Leica | RM2255 | |
| Oven | Memmert | Calibrated by: Cruinn diagnostics Ltd | |
| P10 pipette | Gilson | ||
| P100 pipette | Gilson | ||
| P1000 pipette | Gilson | ||
| P20 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| P200 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| Paraffin section flotation bath | Electrothermal | MH8517 | Calibrated by: Cruinn diagnostics Ltd |
| Pipette electronic dispenser | Corning | StripipetterUltra | Calibrated by: Cruinn diagnostics Ltd |
| Plate cooler | Leica | EG1140C | Calibrated by: Cruinn diagnostics Ltd |
| Refrigerator -20 °C | Liebherr | - | |
| Refrigerator -80 °C | Liebherr | - | |
| Refrigerator 4 °C | Liebherr | - | |
| Seesaw Rocker | DLAb | SK-D1807-E | |
| Spectrophotometer – Victor3V Platereader | PerkinElmer | 1420 | |
| Tissue culture hood/Laminar flow hood | GMI | 8038-30-1044 | |
| Tissue Lyser | Qiagen | TissueLyser LT | |
| Tweezers | - | - | |
| Water bath | Grant | - | |
| Wax embedder | Leica | EG1140H | |
| Materials | |||
| 1 L Water | Adrona - Biosciences | 568 | |
| 1% Triton-X | Sigma Aldrich | 9002-93-1 | |
| 10x PBS tablets | Sigma Aldrich | P4417-100TAB | |
| 37% paraformaldehyde | Sigma-Aldrich | F8775 | |
| Alamar Blue Cell Viability Reagent | Invitrogen | DAL1100 | |
| Collagen- glycosaminoglycan scaffold | Tissue engineering research group (TERG) | ||
| Collagen-nanohydroxyapatite scaffold | Tissue engineering research group (TERG) | ||
| dH20 | Adrona - Biosciences | 568 | |
| Eosin | Sigma-Aldrich | E4009 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| EtOH | Sigma-Aldrich | 1.00983.2500 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| F12 | Gibco | 21765-029 | |
| FBS | Gibco | 10270-106 | |
| Hemaytoxylin | Sigma-Aldrich | HHS32-1L | |
| L-Glutamine | Gibco | 25030-024 | |
| MEM | Gibco | 21090-022 | |
| miRNA easy Kit | Qiagen | 217004 | |
| MNEAA’s | Gibco | 11140-035 | |
| Penicillin/streptomycin | Gibco | 015140-122 | |
| Qiazol | Qiagen | 79306 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Invitrogen | P11496 | |
| RPMI | Gibco | 21875-034 | |
| Sodium bicarbonate | Sigma Aldrich | S7795-500G | |
| Tissue embedding Medium | Sigma | A6330-4LB | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Software | |||
| Excel | - | Excel 2016 | |
| ImageJ | - | - | |
| Prism | - | Version 9 |
Referenzen
- Davidoff, A. M. Neuroblastoma. Seminars in Pediatric Surgery. 21 (1), 2-14 (2012).
- Matthay, K. K., et al. Neuroblastoma. Nature Reviews Disease Primers. 2, 16078 (2016).
- Costard, L. S., Hosn, R. R., Ramanayake, H., O'Brien, F. J., Curtin, C. M. Influences of the 3D microenvironment on cancer cell behaviour and treatment responsiveness: a recent update on lung, breast and prostate cancer models. Acta Biomaterialia. , (2021).
- Borriello, L., Seeger, R. C., Asgharzadeh, S., Declerck, Y. A. More than the genes, the tumor microenvironment in neuroblastoma. Cancer Letters. 380 (1), 304-318 (2016).
- Walker, C., Mojares, E., Del Río Hernández, A. Role of extracellular matrix in development and cancer progression. International Journal of Molecular Sciences. 19 (10), 3028 (2018).
- Bissell, M. J., Hall, H. G., Parry, G. How does the extracellular matrix direct gene expression. Journal of Theoretical Biology. 99 (1), 31-68 (1982).
- Schultz, G. S., Davidson, J. M., Kirsner, R. S., Bornstein, P., Herman, I. M. Dynamic reciprocity in the wound microenvironment. Wound Repair and Regeneration. 19 (2), 134-148 (2011).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6, 11 (2008).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4, 38 (2006).
- Ouellette, J. N., et al. Navigating the collagen jungle: The biomedical potential of fiber organization in cancer. Bioengineering. 8 (2), 1-19 (2021).
- Kreger, S. T., Voytik-Harbin, S. L. Hyaluronan concentration within a 3D collagen matrix modulates matrix viscoelasticity, but not fibroblast response. Matrix Biology. 28 (6), 336-346 (2009).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Hume, R. D., et al. Tumour cell invasiveness and response to chemotherapeutics in adipocyte invested 3D engineered anisotropic collagen scaffolds. Scientific Reports. 8 (1), 12658 (2018).
- Fitzgerald, K. A., et al. The use of collagen-based scaffolds to simulate prostate cancer bone metastases with potential for evaluating delivery of nanoparticulate gene therapeutics. Biomaterials. 66, 53-66 (2015).
- Sapudom, J., Pompe, T. Biomimetic tumor microenvironments based on collagen matrices. Biomaterials Science. 6 (8), 2009-2024 (2018).
- Curtin, C., et al. A physiologically relevant 3D collagen-based scaffold-neuroblastoma cell system exhibits chemosensitivity similar to orthotopic xenograft models. Acta Biomaterialia. 70, 84-97 (2018).
- Gavin, C., et al. Neuroblastoma invasion strategies are regulated by the extracellular matrix. Cancers. 13 (4), 1-23 (2021).
- Ridky, T. W., Chow, J. M., Wong, D. J., Khavari, P. A. Invasive three-dimensional organotypic neoplasia from multiple normal human epithelia. Nature Medicine. 16 (12), 1450-1456 (2010).
- Casal, e. J., Crane, J. S. . Biochemistry, Glycosaminoglycans. , (2019).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. J. The effect of pore size on cell adhesion in collagen-GAG scaffolds. Biomaterials. 26 (4), 433-441 (2005).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. Influence of freezing rate on pore structure in freeze-dried collagen-GAG scaffolds. Biomaterials. 25 (6), 1077-1086 (2004).
- Haugh, M. G., Murphy, C. M., McKiernan, R. C., Altenbuchner, C., O'Brien, F. J. Crosslinking and mechanical properties significantly influence cell attachment, proliferation, and migration within collagen glycosaminoglycan scaffolds. Tissue Engineering. Part A. 17 (9-10), 1201-1208 (2011).
- Murphy, C. M., Haugh, M. G., O'Brien, F. J. The effect of mean pore size on cell attachment, proliferation and migration in collagen-glycosaminoglycan scaffolds for bone tissue engineering. Biomaterials. 31 (3), 461-466 (2010).
- Haugh, M. G., Jaasma, M. J., O'Brien, F. J. The effect of dehydrothermal treatment on the mechanical and structural properties of collagen-GAG scaffolds. Journal of Biomedical Materials Research - Part A. 89 (2), 363-369 (2009).
- Lowe, B., Hardy, J. G., Walsh, L. J. Optimizing nanohydroxyapatite nanocomposites for bone tissue engineering. ACS Omega. 5 (1), 1-9 (2020).
- Cunniffe, G. M., Dickson, G. R., Partap, S., Stanton, K. T., O'Brien, F. J. Development and characterisation of a collagen nano-hydroxyapatite composite scaffold for bone tissue engineering. Journal of Materials Science. Materials in Medicine. 21 (8), 2293-2298 (2010).
- DuBois, S. G., et al. Metastatic sites in stage IV and IVS neuroblastoma correlate with age, tumor biology, and survival. Journal of Pediatric Hematology/Oncology. 21 (3), 181-189 (1999).
- Ryan, A. J., Gleeson, J. P., Matsiko, A., Thompson, E. M., O'Brien, F. J. Effect of different hydroxyapatite incorporation methods on the structural and biological properties of porous collagen scaffolds for bone repair. Journal of Anatomy. 227 (6), 732-745 (2015).
- Tierney, C. M., et al. The effects of collagen concentration and crosslink density on the biological, structural and mechanical properties of collagen-GAG scaffolds for bone tissue engineering. Journal of the Mechanical Behavior of Biomedical Materials. 2 (2), 202-209 (2009).
- Cox, R. F., Jenkinson, A., Pohl, K., O'Brien, F. J., Morgan, M. P. Osteomimicry of mammary adenocarcinoma cells in vitro; increased expression of bone matrix proteins and proliferation within a 3D collagen environment. PLoS One. 7 (7), 41679 (2012).
- Nolan, J. C., et al. Preclinical models for neuroblastoma: advances and challenges. Cancer Letters. 474, 53-62 (2020).
- Sirivisoot, S., Pareta, R., Harrison, B. S. Protocol and cell responses in threedimensional conductive collagen gel scaffolds with conductive polymer nanofibres for tissue regeneration. Interface Focus. 4 (1), 20130050 (2014).
- Thevenot, P., Nair, A., Dey, J., Yang, J., Tang, L. Method to analyze three-dimensional cell distribution and infiltration in degradable scaffolds. Tissue Engineering. Part C-Methods. 14 (4), 319-331 (2008).
- do Amaral, R. J. F. C., et al. Functionalising collagen-based scaffolds with platelet-rich plasma for enhanced skin wound healing potential. Frontiers in Bioengineering and Biotechnology. 7, 371 (2019).
- Gkolfinopoulos, S., Tsapakidis, K., Papadimitriou, K., Papamichael, D., Kountourakis, P. Chromogranin A as a valid marker in oncology: Clinical application or false hopes. World Journal of Methodology. 7 (1), 9-15 (2017).
- O'Brien, F. J., et al. The effect of pore size on permeability and cell attachment in collagen scaffolds for tissue engineering. Technology and Health Care. 15 (1), 3-17 (2007).
Erratum
Formal Correction: Erratum: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds
Posted by JoVE Editors on 6/23/2022. Citeable Link.
An erratum was issued for: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds. The Authors section was updated.
The Authors section was updated from:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Fergal J. O’Brien3,4,5
Olga Piskareva1,2,3,5,6
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
5Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
6National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
to:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Graeme Kelly4
Fergal J. O’Brien3,5,6
Olga Piskareva1,2,3,6,7
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Department of Chemistry, Royal College of Surgeons in Ireland (RCSI), 123 St. Stephen’s Green, Dublin 2, Ireland
5Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
6Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
7National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten