Method Article
Modèle biomimétique in vitro tridimensionnel de neuroblastome à l’aide d’échafaudages à base de collagène
* Ces auteurs ont contribué à parts égales
Dans cet article
Erratum Notice
Résumé
Cet article énumère les étapes nécessaires pour ensemencer des lignées cellulaires de neuroblastome sur des échafaudages tridimensionnels à base de collagène décrits précédemment, maintenir la croissance cellulaire pendant une période prédéterminée et récupérer des échafaudages pour plusieurs analyses de croissance cellulaire et de comportement cellulaire et applications en aval, adaptables pour répondre à une gamme d’objectifs expérimentaux.
Résumé
Le neuroblastome est la tumeur solide extracrânienne la plus fréquente chez les enfants, représentant 15% de l’ensemble des décès par cancer pédiatrique. Le tissu tumoral natif est un microenvironnement tridimensionnel (3D) complexe impliquant des couches de cellules cancéreuses et non cancéreuses entourées d’une matrice extracellulaire (MEC). L’ECM fournit un soutien physique et biologique et contribue à la progression de la maladie, au pronostic du patient et à la réponse thérapeutique.
Cet article décrit un protocole d’assemblage d’un système 3D basé sur un échafaudage pour imiter le microenvironnement du neuroblastome à l’aide de lignées cellulaires de neuroblastome et d’échafaudages à base de collagène. Les échafaudages sont complétés par de la nanohydroxyapatite (nHA) ou des glycosaminoglycanes (GAG), naturellement présents à des concentrations élevées dans l’os et la moelle osseuse, les sites métastatiques les plus courants du neuroblastome. La structure poreuse 3D de ces échafaudages permet la fixation, la prolifération et la migration des cellules du neuroblastome, ainsi que la formation d’amas cellulaires. Dans cette matrice 3D, la réponse cellulaire à la thérapeutique reflète davantage la situation in vivo .
Le système de culture basé sur un échafaudage peut maintenir des densités cellulaires plus élevées que la culture cellulaire bidimensionnelle (2D) conventionnelle. Par conséquent, les protocoles d’optimisation du nombre initial de cellules d’ensemencement dépendent des délais expérimentaux souhaités. Le modèle est surveillé en évaluant la croissance cellulaire via la quantification de l’ADN, la viabilité cellulaire via des tests métaboliques et la distribution cellulaire dans les échafaudages via la coloration histologique.
Les applications de ce modèle comprennent l’évaluation des profils d’expression des gènes et des protéines ainsi que les tests de cytotoxicité à l’aide de médicaments conventionnels et de miARN. Le système de culture 3D permet la manipulation précise des composants cellulaires et ECM, créant ainsi un environnement plus physiologiquement similaire au tissu tumoral natif. Par conséquent, ce modèle 3D in vitro fera progresser la compréhension de la pathogenèse de la maladie et améliorera la corrélation entre les résultats obtenus in vitro, in vivo dans des modèles animaux et des sujets humains.
Introduction
Le neuroblastome est un cancer pédiatrique du système nerveux sympathique survenant au cours du développement embryonnaire ou au début de la vie postnatale en raison de la transformation des cellules de la crête neurale1. C’est la tumeur extracrânienne solide la plus fréquente chez les enfants, représentant 8 % des tumeurs malignes diagnostiquées chez les patients de moins de 15 ans et responsable de 15 % de tous les décès par cancer chez l’enfant. La maladie présente des comportements cliniques très hétérogènes en raison d’altérations chromosomiques, génétiques et épigénétiques spécifiques, et de caractéristiques histopathologiques.
Ces altérations contribuent à l’agressivité du neuroblastome et aux mauvais résultats chez les patients pédiatriques. Par conséquent, les thérapies actuelles s’avèrent inefficaces à long terme pour près de 80 % des patients atteints de la maladie cliniquement agressive2, ce qui souligne le fait que le traitement de ce groupe de patients reste difficile. Cela est probablement dû au fait que les mécanismes du neuroblastome, de l’hétérogénéité et des métastases ne sont pas encore entièrement compris. Cependant, on pense maintenant que le microenvironnement tumoral (TME) joue un rôle dans la progression de nombreux cancers ; Pourtant, il reste peu étudié dans le neuroblastome 3,4.
L’ETM natif est un microenvironnement 3D complexe impliquant des cellules cancéreuses et non cancéreuses entourées d’une MEC. L’ECM fait référence à la composante acellulaire d’un tissu qui fournit un soutien structurel et biochimique à ses résidents cellulaires et contribue à la progression de la maladie, au pronostic du patient et à la réponse thérapeutique5. Cette promotion de la progression de la maladie est due à la « réciprocité dynamique » ou à la communication bidirectionnelle continue entre les cellules et l’ECM 6,7,8. Au fur et à mesure que le cancer progresse, le collagène stromal est réorganisé souvent selon des motifs linéaires perpendiculaires à l’interface stroma-cancer, que les cellules cancéreuses utilisent comme voie migratoire vers les métastases 9,10,11.
Les principaux composants de cet échafaudage biologique fonctionnel natif comprennent un réseau fibreux de collagènes de type I et II et d’autres protéines, notamment l’élastine, des glycoprotéines telles que la laminine, ainsi qu’une gamme de protéoglycanes et d’autres composants solubles12,13. Ces protéines de l’ECM native sont aujourd’hui devenues des biomolécules naturelles attractives pour le développement de modèles 3D in vitro 3. L’application d’échafaudages 3D pour la culture cellulaire in vitro est de plus en plus populaire en raison de sa plus grande représentation physiologique de l’ETM par rapport à la culture monocouche 2D traditionnelle. Les échafaudages 3D fabriqués aident à l’attachement, à la prolifération, à la migration, au métabolisme et à la réponse aux stimuli observés dans les systèmes biologiques in vivo.
Le composant principal de ces échafaudages 3D est le collagène, qui est un acteur clé dans de nombreux processus biologiques normaux, notamment la réparation des tissus, l’angiogenèse, la morphogenèse des tissus, l’adhésion cellulaire et la migration11. Les matrices 3D à base de collagène ont montré leur fonctionnalité robuste pour modéliser l’ECM, servant de microenvironnement biomimétique in vitro tout en permettant les interactions cellule-ECM ainsi que la migration et l’invasion cellulaires. Ces matrices 3D fournissent également une analyse plus précise de la réponse cellulaire aux médicaments chimiothérapeutiques que la culture 2D traditionnelle ou « plate » dans de nombreux modèles de cancer 14,15,16, y compris le neuroblastome 17,18. L’analyse génétique des cultures cellulaires 3D a rapporté une corrélation plus élevée avec le profil des tissus humains, même par rapport aux modèles animaux19. Dans l’ensemble, la pierre angulaire de ces échafaudages 3D est de fournir aux cellules un environnement in vitro approprié, qui récapitule l’architecture tissulaire native et facilite la diaphonie moléculaire bidirectionnelle8.
Pour augmenter la complexité des modèles à base de collagène, d’autres composants courants de l’ECM sont incorporés dans le processus d’ingénierie tissulaire, créant ainsi des modèles plus pertinents sur le plan physiologique pour refléter les ETM de niche de différents tissus. Par exemple, les GAG, des polysaccharides chargés négativement présents dans tous les tissus de mammifères20, facilitent l’attachement, la migration, la prolifération et la différenciation des cellules. Le sulfate de chondroïtine est un type spécifique de GAG présent dans l’os et le cartilage, qui a déjà été utilisé dans des applications d’ingénierie tissulaire pour la réparation osseuse 21,22,23,24,25. La nano-hydroxyapatite (nHA) est le principal constituant inorganique de la composition minérale des tissus osseux humains, constituant jusqu’à 65 % de l’os en poids26 et est donc largement utilisée pour le remplacement et la régénération osseuse27. Ainsi, les GAGs et les nHA sont des composites intéressants pour la reconstruction de la MEC du neuroblastome primaire et la modélisation des sites métastatiques les plus courants du neuroblastome, de la moelle osseuse (70,5 %) et de l’os (55,7 %)28.
Les échafaudages incorporant ces composants ECM ont été développés à l’origine pour des applications d’ingénierie du tissu osseux avec une analyse approfondie de leur biocompatibilité, de leur toxicité et de leurs caractéristiques ostéoconductrices et ostéoinductives29,30. Ce sont des matrices poreuses à base de collagène produites à l’aide de techniques de lyophilisation pour contrôler leurs propriétés physiques et biologiques. Les échafaudages de collagène complétés avec du nHA (Coll-I-nHA) ou de la chondroïtine-6-sulfate (Coll-I-GAG) ont démontré leur succès dans l’imitation de l’ETM primaire dans le cancer du sein31 et des métastases osseuses dans le cancer de la prostate15 ainsi que le neuroblastome17. La technique de lyophilisation utilisée pour fabriquer ces échafaudages composites permet d’obtenir une homogénéité reproductible de la taille des pores et de la porosité à l’intérieur des échafaudages22,23,24. En bref, une suspension de collagène (0,5 % en poids) est fabriquée en mélangeant du collagène fibrillaire avec 0,05 M d’acide acétique. Pour Coll-I-GAG, 0,05 % en poids de chrondoitine-6-sulfate isolé du cartilage de requin est ajouté à la suspension de collagène lors du mélange. Pour les échafaudages composites Coll-I-nHA, des particules d’hydroxyapatite de taille nanométrique sont synthétisées comme décrit précédemment27 et ajoutées à la suspension de collagène dans un rapport de 2 :1 au poids du collagène pendant le processus de mélange. Tous les échafaudages sont réticulés physiquement et stérilisés par un traitement déhydrothermal à 105 °C pendant 24 h25. Les échafaudages cylindriques (6 mm de diamètre, 4 mm de hauteur) sont obtenus à l’aide d’un poinçon de biopsie et peuvent être réticulés chimiquement avec 3 mM de chlorhydrate de N-(3-diméthylaminopropyl)-N'-éthylcarbodiimide et 5,5 mM de N-hydroxysuccinimide (EDAC/NHS) dans de l’eau distillée (dH2O) pour améliorer les propriétés mécaniques des constructions30. Ce processus de fabrication bien optimisé de deux échafaudages de collagène crée des échafaudages avec des propriétés mécaniques reproductibles, notamment la taille des pores, la porosité et la rigidité (kPa). Les échafaudages Coll-I-GAG et Coll-I-nHA ont des propriétés physiques différentes, créant des conditions environnementales différentes. Les propriétés de chaque échafaudage sont présentées dans le tableau 1.
| Coll-I-GAG | Coll-I-nHA | |
| Taille de l’échafaudage (diamètre [mm] x hauteur [mm]) | 6 x 4 17 | 6 x 4 17 |
| Concentration de collagène (% en poids) | 0,5 17 | 0,5 17 |
| Concentration du substrat (% en poids) [basé sur le poids du collagène] | 0,05 15,17 | 200 17 |
| Taille moyenne des pores (mm) | 96 22 | 96 – 120 29 |
| Porosité (%) | 99,5 23 | 98,9 à 99,4 27 |
| Rigidité (kPa) | 1,5 27 | 5.5 à 8.63 29 |
Tableau 1 : Vue d’ensemble des propriétés mécaniques des deux échafaudages adoptés pour l’étude de la biologie du neuroblastome.
Cet article décrit un protocole d’assemblage d’un système basé sur un échafaudage 3D pour mieux imiter le microenvironnement du neuroblastome à l’aide de lignées cellulaires de neuroblastome et d’échafaudages à base de collagène précédemment décrits complétés par du nHA (Coll-I-nHA) ou du chondroïtine-6-sulfate (Coll-I-GAG). Le protocole comprend des méthodes en aval pour analyser les mécanismes de croissance des cellules de neuroblastome dans un environnement plus pertinent sur le plan physiologique en utilisant des méthodes peu coûteuses préalablement optimisées et adaptées de la culture monocouche 2D Figure 1.
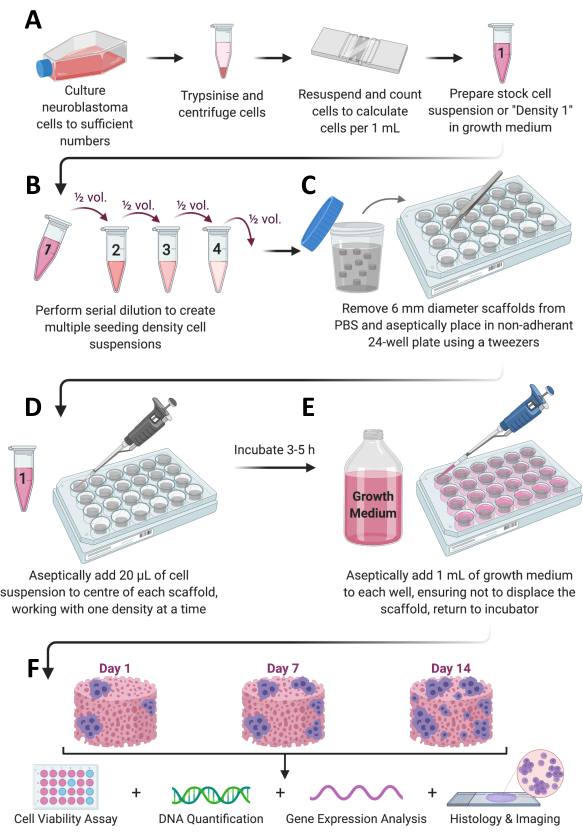
Figure 1 : Flux de travail global du protocole. (A) Les cellules sont cultivées en nombre suffisant, divisées, comptées et remises en suspension dans un volume approprié de milieu. (B) Ce stock cellulaire subit ensuite une dilution en série pour préparer un total de 4 suspensions cellulaires de densités différentes. (C) Les échafaudages à base de collagène sont plaqués de manière stérile dans des plaques non adhérentes à 24 puits, et (D) 20 μL de suspension cellulaire sont ajoutés au centre de chaque échafaudage et laissés incuber à 37 °C, 5 % de CO2 et 95 % d’humidité pendant 3 à 5 h. (E) Un milieu de croissance complet (1 mL) est ensuite ajouté lentement à chaque échafaudage, et les plaques sont replacées dans l’incubateur pour permettre la croissance cellulaire pendant la période souhaitée. (F) À chaque moment prédéterminé, plusieurs échafaudages sont récupérés pour l’évaluation de la viabilité et de la croissance cellulaires, l’analyse de l’expression génique et la coloration histologique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Plan d’expérience
REMARQUE : Le nombre d’échafaudages et de cellules nécessaires pour chaque expérience dépendra de l’échelle de l’expérience et peut être calculé à l’aide des outils de cette section sur le plan expérimental.
- Nombre d’échafaudages requis
- Déterminer l’échéancier expérimental global et les intervalles d’évaluation des changements dans la biologie cellulaire (p. ex., croissance cellulaire et métabolisme).
REMARQUE : Par exemple, la période expérimentale est de 14 jours, avec une évaluation de la croissance cellulaire tous les 7 jours. Ainsi, les points de temps expérimentaux sont les jours 1, 7, 14 donnant un total de 3 points de temps. - Ensuite, décidez du nombre d’applications expérimentales à appliquer à chaque point temporel. Essayez d’effectuer les mêmes analyses à chaque point de temps pour surveiller les changements.
REMARQUE : Les analyses typiques de la croissance cellulaire sur les échafaudages comprennent des tests de viabilité cellulaire, la quantification de l’ADN, la coloration histologique et l’imagerie, ainsi que l’analyse de l’expression génique. Ceux-ci seront examinés plus en détail dans la section 5. - Décider quand utiliser le même échafaudage pour plusieurs applications en aval, par exemple, après l’évaluation de la viabilité cellulaire à l’aide d’un test approprié, réutiliser l’échafaudage pour l’isolement de l’ADN/ARN (voir 5.1.11).
- Conservez un minimum de 3 répétitions biologiques pour chaque application, par exemple, 3 échafaudages pour la viabilité cellulaire et la quantification de l’ADN, 3 pour l’histologie et 3 pour l’analyse de l’expression génique. Comme cela équivaut à 9 échafaudages par point de temps, prévoyez d’utiliser 27 échafaudages pour 3 points de temps.
- Enfin, multipliez ce nombre par le nombre de paramètres ou de conditions expérimentales (p. ex., évaluation de plusieurs lignées cellulaires, densités initiales de semis, composition d’échafaudage).
NOTA : Dans le cadre de ce protocole, 4 densités de semis initiales différentes ont été évaluées pour une lignée cellulaire, ce qui a donné lieu à 108 échafaudages (27 échafaudages x 4 conditions) requis. Pour tenir compte de l’erreur humaine et donner une couverture supplémentaire, ajoutez ~10% à ce nombre, par exemple, si 108 échafaudages sont nécessaires, préparez 120 échafaudages.
- Déterminer l’échéancier expérimental global et les intervalles d’évaluation des changements dans la biologie cellulaire (p. ex., croissance cellulaire et métabolisme).
- Nombre de cellules requises
REMARQUE : Un volume de 20 μL de suspension cellulaire est recommandé pour l’ensemencement des cellules sur un échafaudage cylindrique (diamètre 6 mm, hauteur 4 mm) le jour 0. Le nombre de cellules par ces 20 μL de suspension cellulaire est ajusté en fonction du plan expérimental, calculé à la section 1.1. Une densité de semis initiale courante est de 2 × 105 cellules par 20 μL, utilisée à titre d’exemple pour le protocole ci-dessous.- Pour calculer le nombre total de cellules nécessaires à l’expérience, multipliez les 2 × 105 cellules initiales par 20 μL par le nombre d’échafaudages nécessaires.
REMARQUE : Par exemple, 30 échafaudages multipliés par 20 μL donnent un volume total de 600 μL. Si chaque échafaudage nécessite 2 × 10 5 cellules, 600 μL de suspension contiennent un total de 6 × 10 6 cellules (2 × 105 × 30), ce qui donne un besoin final de 6 × 106 cellules dans 600 μL. Le nombre de paramètres expérimentaux déterminera le nombre total de cellules requises. Ce protocole décrit donc la culture cellulaire à l’aide d’un flacon de culture cellulaire multicouche, qui peut traiter le même nombre de cellules que 10 flacons traditionnels de 175 cm2.
- Pour calculer le nombre total de cellules nécessaires à l’expérience, multipliez les 2 × 105 cellules initiales par 20 μL par le nombre d’échafaudages nécessaires.
2. Préparation d’échafaudages à base de collagène
NOTE : Les échafaudages cylindriques Coll-I-nHA et Coll-I-GAG (diamètre 6 mm, hauteur 4 mm) sont préparés selon les méthodes établies 15,21,27. Une fois réticulés chimiquement selon les méthodes précédemment publiées17, les échafaudages doivent être utilisés dans un délai de 1 semaine.
- Après la fabrication des échafaudages ayant les propriétés mécaniques souhaitées, assurez-vous que les échafaudages sont complètement hydratés et soigneusement lavés dans une solution saline tamponnée au phosphate (PBS).
REMARQUE : Cela prend généralement ~12 h après la réticulation des échafaudages et peut être effectué dans des conteneurs de déchets de culture tissulaire de 100 mL à 4 °C, avec un maximum de 50 échafaudages par conteneur et 2 mL de PBS par échafaudage. - Conservez les échafaudages dans du PBS à 4 °C jusqu’au moment de les utiliser.
3. Propager les cellules de neuroblastome dans un flacon de culture cellulaire multicouche
REMARQUE : La densité de semis optimale pour la fiole multicouche varie. Pour le flacon utilisé dans cette expérience, la densité optimale selon les instructions du fabricant est de 1 × 107 cellules. Avant d’ensemencer la fiole multicouche, propager les cellules à une densité de 1 × 107 cellules ou plus dans une fiole de culture tissulaire appropriée (p. ex., une fiole de culture tissulaire T175 cm2 ). Pour ensemencer des cellules dans le ballon multicouche (section 3.1), les cultiver jusqu’à ce qu’elles soient confluentes à 70-80 %, récolter et compter le nombre de cellules par mL, en se référant aux étapes 3.2.16-3.2.20 pour effectuer le comptage cellulaire. Une fois la suspension cellulaire comptée, procéder immédiatement à l’ensemencement de la fiole multicouche. Le travail de culture cellulaire doit être effectué dans une hotte à flux laminaire pour maintenir la stérilité.
- Ensemencement de la fiole de culture cellulaire multicouche
- Préchauffer 550 mL de milieu de culture complet (varie en fonction de la lignée cellulaire utilisée) et 100 mL de PBS stérile dans un bain-marie à 37 °C pendant 20 min.
- À l’aide de la suspension cellulaire récoltée, calculez le volume requis de la suspension cellulaire nécessaire pour atteindre la densité de semis optimale de 1 × 107 cellules à l’aide de l’équation (1), où WANT fait référence au nombre de cellules nécessaires à l’ensemencement de la fiole multicouche, et HAVE fait référence au nombre de cellules/mL dans la suspension cellulaire :
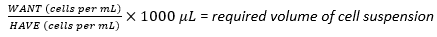 (1)
(1)
p. ex.,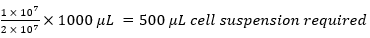
- Ajouter le volume requis de la suspension cellulaire à 100 mL du milieu de culture préchauffé.
- Prenez une nouvelle fiole multicouche dans le capot, retirez le bouchon et tenez la fiole à un angle de 60°. Pipeter lentement les 100 mL de la suspension cellulaire dans le flacon, le long du côté incliné du goulot. Fermez le flacon et placez-le sur le côté pour permettre aux cellules de se répartir uniformément dans les couches.
- À un angle de 60°, ajouter 400 mL du milieu de culture préchauffé dans la fiole multicouche en versant lentement ou en pipetant le côté incliné du goulot. Si le goulot est plein, remettez la fiole en position verticale ou bouchez la fiole et placez-la sur le côté avant de la remettre à verser.
REMARQUE : Versez lentement pour éviter la formation excessive de bulles. Tapotez doucement le flacon en position verticale pour permettre à toutes les bulles de remonter vers le haut et retirez-les à l’aide d’une pipette de 10 ml. Assurez-vous que la fiole est remplie jusqu’au filetage inférieur du goulot ; Ajoutez plus de milieu, si nécessaire, pour y parvenir. - Bouchez la fiole multicouche et incubez-la avec le col incliné vers le bas à 37 °C, 5 % de CO2 et 95 % d’humidité.
- Vérifiez la croissance tous les 2-3 jours pour la confluence. Pour vérifier la confluence des deux couches inférieures de la fiole multicouche, observez-les sous l’objectif 4x d’un microscope inversé.
REMARQUE : Lorsqu’il est ensemencé avec 1 × 107 cellules de neuroblastome, le flacon à 10 couches prendra généralement une semaine pour devenir confluent, bien que cela puisse varier en fonction de la lignée cellulaire utilisée.
- Entretien courant des cellules dans la fiole multicouche
- Préchauffer 550 mL de milieu de culture complet (varie selon la lignée cellulaire utilisée), 50 à 100 mL de trypsine et 300 mL de PBS stérile dans un bain-marie à 37 °C pendant 20 min.
- Vérifiez que la fiole multicouche a une confluence de 70 à 80 %.
- Placez la fiole multicouche dans une hotte à flux laminaire et jetez le milieu usé de la fiole en versant. Dans un premier temps, inclinez le ballon de manière à ce que le fluide se déverse sur le barrage d’air dans un conteneur à déchets. Pendant le versement, faites pivoter le flacon de 180° jusqu’à ce que le fluide s’écoule le long du col incliné du flacon. Faites pivoter le ballon d’avant en arrière le long de cet axe pour éliminer tout liquide restant.
- Lavez les cellules en ajoutant lentement 100 ml de PBS stérile préchauffé le long du goulot incliné. Bouchez le flacon, placez-le sur le côté pour permettre une distribution uniforme du PBS et faites pivoter le flacon d’avant en arrière pour laver les cellules.
- Jeter le nettoyant PBS de la même manière que 3.2.3. Répétez les étapes de lavage.
- Diluer 50 mL de trypsine préchauffée dans 50 mL de PBS stérile préchauffé. Ajouter 100 mL de solution de trypsine diluée dans la fiole multicouche, bouchon et placer la fiole sur le côté pour permettre une distribution uniforme de la trypsine. Si la lignée cellulaire est très adhérente, utilisez 100 mL de trypsine non diluée.
- Incuber le flacon pendant 2 à 5 minutes à 37 °C, 5 % de CO2 et 95 % d’humidité, en surveillant le décollement cellulaire au microscope. Si nécessaire, tapotez fermement le flacon pour faciliter le détachement ou replacez-le dans l’incubateur pendant une minute de plus.
- Placez la fiole multicouche dans une hotte à flux laminaire et neutralisez la trypsine avec 100 mL de milieu de culture. Bouchez le flacon, placez-le sur le côté et balancez-le d’avant en arrière pour assurer une neutralisation complète.
- Versez la suspension cellulaire neutralisée dans 4 tubes à centrifuger coniques de 50 ml.
REMARQUE : Si un prélèvement complet de cellules est nécessaire, laver à nouveau le ballon multicouche avec 100 mL de PBS stérile et le verser dans 2 tubes à centrifuger de 50 mL. - Centrifuger la suspension cellulaire dans des tubes à centrifuger de 50 ml à 340 × g pendant 3 à 4 min pour granuler les cellules.
- Remettez les tubes de centrifugation dans la hotte à flux laminaire et jetez soigneusement autant de surnageant que possible de chaque pastille.
REMARQUE : Le granulé sera gros et relativement lâche et, par conséquent, peut être facilement perturbé. - Ajouter 1 à 5 ml de milieu de culture à chaque pastille et la remettre en suspension en pipetant de haut en bas plusieurs fois.
- Regroupez les 4 pastilles remises en suspension dans un tube à centrifuger de 50 ml, mélangez-les soigneusement avec la pipette et notez le volume total.
- Faire une dilution appropriée de la suspension cellulaire pour le comptage cellulaire afin que chaque carré extérieur de l’hémocytomètre contienne 30 à 100 cellules.
REMARQUE : Une dilution de départ appropriée est de 1 :100 ; pour cela, pipeter 10 μL de la suspension cellulaire dans un tube à centrifuger conique frais de 15 mL et le diluer avec 990 μL de PBS stérile. Pipeter le mélange de haut en bas plusieurs fois pour bien mélanger. - Placez un tube à centrifuger de 50 ml contenant le stock de cellules dans l’incubateur pendant le comptage.
- À l’aide d’un hémocytomètre propre, placez une lamelle propre sur la grille, comme illustré à la figure 2.
- Pipeter 10 μL de la suspension cellulaire diluée dans la chambre de l’hémocytomètre. Si la chambre est pleine avant que la totalité des 10 μL ne soit distribuée, arrêtez le pipetage.
- Placez l’hémocytomètre sous l’objectif 4x d’un microscope optique. Ajustez la mise au point grossière et fine pour visualiser les cellules.
- Comptez le nombre de cellules dans les quatre carrés d’angle extérieurs de la chambre, comme indiqué à la figure 2. Additionnez les quatre comptes et divisez par 4 pour calculer la moyenne des cellules par carré.
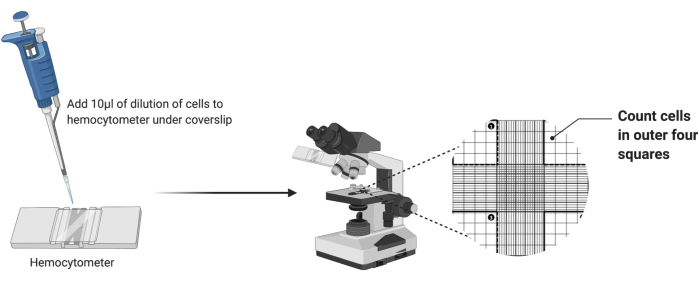
Figure 2 : Comptage cellulaire à l’aide d’un hémocytomètre. Dix microlitres de suspension cellulaire sont ajoutés à l’hémocytomètre sous la lamelle. La chambre est ensuite placée sous l’objectif 4x d’un microscope et le nombre de cellules dans les quatre coins extérieurs de la grille est compté. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Multipliez le nombre moyen par le facteur de dilution (p. ex., 100) et multipliez ce nombre par 10 000 pour obtenir le nombre de cellules par ml à l’aide de l’équation (2).
 (2)
(2) - Calculer le nombre total de cellules dans la solution mère en multipliant par le volume total de la cellule (p. ex., si les 4 pastilles remises en suspension ont donné une solution de 20 mL, multiplier le nombre de cellules/mL par 20).
- Pour maintenir la fiole multicouche, utilisez l’équation (1) décrite à l’étape 3.1.2 pour calculer le volume de cellules nécessaires pour réensemencer 1 × 107 cellules dans la fiole et effectuez les étapes 3.1.3 à 3.1.6 pour réensemencer la fiole. Si vous êtes prêt à ensemencer les échafaudages, passez à la section suivante.
4. Semer des cellules de neuroblastome sur des échafaudages
- Préparer la suspension de la cellule d’origine
REMARQUE : Ce protocole décrit les étapes de création de quatre densités d’ensemencement différentes de cellules de neuroblastome, avec un facteur de multiplication de 2 entre chaque densité. Une dilution en série sera donc utilisée pour créer trois autres suspensions cellulaires à partir de la suspension mère. Le travail de culture cellulaire doit être effectué dans une hotte à flux laminaire pour maintenir la stérilité.- Utiliser l’équation (3) pour calculer le volume de cellules nécessaires à partir du nombre total de cellules dans la fiole multicouche (compté à la section 3.2.19) pour préparer la première densité de semis ou la première suspension de cellules.
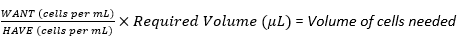 (3)
(3)
NOTA : Par exemple, si la densité de semis la plus élevée est de 6 × 10 5 cellules par échafaudage requises pour 30 échafaudages, chaque échafaudage recevant 20 μL de suspension cellulaire, la suspension de cellules d’origine nécessitera 1,8 × 107 cellules (6 × 105 cellules × 30 échafaudages) dans un volume total de 600 μL (20 μL × 30 échafaudages).
Comme une dilution en série sera effectuée à partir de cette préparation, ces nombres doivent être doublés, c’est-à-dire 3,6 × 107 cellules dans un volume total de 1200 μL. Pour convertir cela en WANT en cellules par mL ; diviser 3,6 × 10 7 par 1200 μL et multiplier par 1000 μL, ce qui donne 3 × 107 cellules par mL.

- Ajouter le volume requis de cellules dans un tube à centrifuger stérile de 2 mL ou 15 mL et porter à un volume final de 1200 μL avec le milieu de culture. Étiquetez ce tube comme étant de densité 1 (Figure 3).
- Utiliser l’équation (3) pour calculer le volume de cellules nécessaires à partir du nombre total de cellules dans la fiole multicouche (compté à la section 3.2.19) pour préparer la première densité de semis ou la première suspension de cellules.
- Effectuez une dilution en série pour créer plusieurs suspensions cellulaires de densité de semis.
- À partir de la densité 1 préparée à l’étape 4.1.1, préparez trois autres densités de suspension cellulaire par dilution en série comme indiqué à la figure 3.
- Diluer chaque densité d’un facteur 2 dans le milieu de culture. Tout d’abord, ajoutez le volume final requis (600 μL par rapport à l’exemple précédent) de milieu de culture à trois tubes à centrifuger stériles de 2 mL ou 15 mL.
- Transférer la moitié de la densité 1 (600 μL) dans l’un des tubes, en mélangeant soigneusement la suspension cellulaire avec le milieu à diluer. Étiquetez ce tube comme étant de densité 2.
- Transférer la moitié de la densité 2 (600 μL) dans le tube suivant, en mélangeant soigneusement la suspension cellulaire avec le milieu à diluer. Étiquetez ce tube comme étant de densité 3.
- Transférer la moitié de la densité 3 (600 μL) dans le tube suivant, en mélangeant soigneusement la suspension cellulaire avec le milieu à diluer. Étiquetez ce tube comme étant de densité 4.
- Éliminer 600 μL de la densité 4 afin que les quatre préparations aient un volume final de 600 μL.
- Comme témoin négatif, ajouter 600 μL de milieu de culture uniquement dans un tube à centrifuger stérile de 2 mL. Voir la figure 3 pour un schéma du processus de dilution en série.
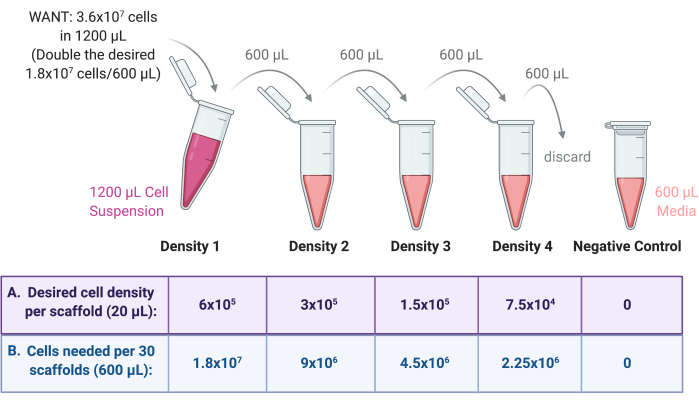
Figure 3 : Dilution en série de cellules pour préparer 4 suspensions pour 4 densités de semis d’échafaudages différentes. (A) Les nombres peuvent être ajustés en fonction de la densité de semis souhaitée par échafaudage et (B) multipliés par le nombre total d’échafaudages par densité, chaque échafaudage recevant 20 μL de suspension cellulaire. Dans cet exemple, la densité 1 nécessite 6 × 105 cellules par échafaudage, ce qui équivaut à 1,8 × 107 cellules dans 600 μL pour 30 échafaudages. Ce nombre est doublé pour commencer la dilution en série, car 600 μL sont ensuite transférés et dilués dans 600 μL de milieu de culture dans le tube suivant. Ce processus se poursuit jusqu’à ce qu’il y ait 4 suspensions cellulaires avec un facteur de 2 entre chacune. Un contrôle négatif est obtenu en ajoutant 600 μL de milieu seulement dans un tube. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Ajout de suspensions cellulaires aux échafaudages
REMARQUE : Retirez les échafaudages (stockés dans PBS) du réfrigérateur et laissez-les revenir à température ambiante (RT) avant d’ajouter les cellules.- Amenez les échafaudages en PBS dans la hotte à flux laminaire.
- À l’aide d’une pince à épiler stérile, placez les échafaudages dans des plaques non adhérentes à 24 puits avec un échafaudage par puits (figure 1C). Soulevez doucement les échafaudages par leurs coins et pressez-les légèrement contre le côté du récipient pour éliminer l’excès de PBS. Ajoutez les échafaudages au centre des puits, côté peau vers le bas (le côté de la couche brillante de l’échafaudage, face vers le bas dans les plaques en plastique à 24 puits).
- Étiquetez les plaques à 24 puits avec les détails de la lignée cellulaire, les paramètres pertinents (par exemple, la densité d’ensemencement) et les points temporels. Travaillez avec une densité d’ensemencement cellulaire à la fois, en conservant les densités restantes dans l’incubateur à 37 °C jusqu’à ce qu’elles soient prêtes à l’emploi.
- Dans la hotte à flux laminaire, utilisez une pipette P20 et des pointes stériles pour ajouter délicatement 20 μL de la suspension cellulaire correspondante au centre de chaque échafaudage (Figure 1D). Gardez les cellules bien en suspension en mélangeant bien tout en ajoutant les cellules aux échafaudages. Assurez-vous que la suspension reste sur le dessus de l’échafaudage et ne glisse pas sur la base du puits, car cela ne permettra pas la fixation de la cellule à l’échafaudage.
- Une fois les cellules ajoutées, incubez les plaques pendant 3 à 5 h (37 °C, 5 % de CO2 et 95 % d’humidité) pour permettre à la plupart des cellules de se fixer.
- Après l’incubation, ajouter lentement et doucement 1 mL de milieu de culture préchauffé dans chaque puits (figure 1E). Utilisez une pipette P1000 pour ajouter le milieu afin de permettre un mouvement plus lent et contrôlé, empêchant ainsi le déplacement des échafaudages. Si vous travaillez avec un très grand nombre d’échafaudages, utilisez une pipette de 10 ml avec le pistolet à pipette réglé sur « goutte » et « bas ».
- Incuber les plaques à 24 puits pendant une nuit (37 °C, 5 % de CO2 et 95 % d’humidité).
- Entretien des cellules sur les échafaudages
- Après les 24 premières heures de fixation des cellules (jour 1), transférez les échafaudages ensemencés sur de nouvelles plaques non adhérentes à 24 puits et ajoutez 1 à 2 ml de milieu de culture frais.
REMARQUE : Cette étape enlève les cellules qui sont tombées au fond des plaques en plastique à 24 puits plutôt que de les laisser se développer sur les échafaudages. Les répliques d’échafaudage désignées comme jour 1 seront démontées après 24 h, comme il est indiqué à la section 5 ; Par conséquent, l’entretien ne s’applique pas à ces échafaudages. - Surveillez d’abord les échafaudages tous les 2-3 jours pour un changement de couleur du milieu de culture. Au fur et à mesure que le temps passe et que les cellules prolifèrent dans les échafaudages, nourrissez les cellules plus fréquemment.
- À l’aide d’un pistolet à pipette de 10 mL, en mode lent, retirez le 1 mL du milieu usagé et jetez-le. Si l’on effectue des expériences nécessitant un milieu conditionné, recueillir le milieu usé des répétitions biologiques dans un tube à centrifuger de 15 mL, centrifuger à 340 × g pendant 2 min pour granuler les débris cellulaires, transférer le surnageant dans un tube neuf et stocker à -80 °C.
- Ajouter délicatement 2 mL de milieu de culture préchauffé dans les échafaudages, en utilisant à nouveau la fonction goutte à goutte sur la pipette, et remettre la plaque à 24 puits dans l’incubateur (37 °C, 5 % de CO2 et 95 % d’humidité). Répétez chaque fois que le milieu est dépensé pendant la durée de la période de croissance souhaitée.
- Après les 24 premières heures de fixation des cellules (jour 1), transférez les échafaudages ensemencés sur de nouvelles plaques non adhérentes à 24 puits et ajoutez 1 à 2 ml de milieu de culture frais.
5. Récupération et applications d’échafaudages
REMARQUE : À chaque instant, plusieurs applications peuvent être utilisées pour surveiller la croissance cellulaire sur les échafaudages ou évaluer les profils d’expression des gènes et des protéines. Les conditions de récupération de l’échafaudage dépendront de l’analyse à effectuer, avec plusieurs méthodes de récupération décrites dans les sous-sections suivantes et illustrées à la figure 4.
- Évaluation de la viabilité cellulaire à l’intérieur d’échafaudages
- Stériliser le réactif de test de viabilité cellulaire approprié en le filtrant à travers un filtre stérile de 0,2 μm dans un tube à centrifuger dans la hotte à flux laminaire. Préchauffer cette solution stérile avec le milieu de culture complet et le PBS stérile dans un bain-marie à 37 °C.
- Dans la hotte à flux laminaire, utilisez une pince à épiler stérile pour transférer les échafaudages à analyser sur une plaque fraîche à 24 puits. Étiquetez la plaque avec tous les détails pertinents.
REMARQUE : Effectuez l’analyse en trois exemplaires. - Ajouter 900 μL du milieu de culture préchauffé dans chaque puits, puis 100 μL du réactif stérile de viabilité cellulaire. Inclure un témoin négatif en ajoutant 900 μL de milieu et 100 μL de réactif de viabilité des cellules stériles dans un puits sans échafaudage. Replacez le couvercle sur la plaque et balancez doucement la plaque pendant ~3 min pour répartir uniformément le réactif de viabilité cellulaire dilué dans tout le puits. Incuber la plaque à 37 °C, 5 % de CO2 et 95 % d’humidité.
REMARQUE : Les temps d’incubation devront être optimisés pour chaque lignée cellulaire ; Reportez-vous aux directives du fabricant. Pour les lignées cellulaires de neuroblastome, une incubation de 4 à 6 h semble optimale. - Après l’incubation, retirez l’assiette de l’incubateur et balancez-la doucement pendant quelques secondes.
- Dans la hotte à flux laminaire, ouvrez une nouvelle plaque translucide à 96 puits. À partir de chaque puits de la plaque à 24 puits, transférer le milieu incubé et le réactif dans trois puits de la plaque à 96 puits à raison de 100 μL par puits, en obtenant des triplicats techniques.
REMARQUE : Ce transfert laissera 700 μL dans les puits de la plaque à 24 puits. - Couvrez la plaque à 96 puits d’une feuille d’aluminium pour protéger le réactif de viabilité de la cellule de la lumière.
- Retirez et jetez les 700 μL restants du contenu du puits de chaque échafaudage de la plaque à 24 puits. Lavez chaque échafaudage deux fois avec 1 mL de PBS stérile.
REMARQUE : Toutes les couleurs ne seront pas retirées des échafaudages. Ces échafaudages peuvent ensuite être utilisés pour d’autres applications, par exemple la quantification de l’ADN, en les plaçant dans 1 mL de Triton X-100 à 1 % dans une solution de bicarbonate de sodium (NaHCO3) à 0,1 M et en les stockant à -80 °C (voir rubrique 5.2, Figure 4B). - Retirez la plaque à 96 puits de la hotte à flux laminaire et mesurez l’absorbance de chaque puits à des longueurs d’onde de 570 nm et 600 nm à l’aide d’un lecteur de microplaques. Enregistrez les valeurs d’absorbance aux deux longueurs d’onde et suivez les instructions du fabricant pour calculer le pourcentage de réduction du réactif de viabilité cellulaire par les cellules.
- Représenter graphiquement et analyser statistiquement les résultats de viabilité cellulaire à l’aide d’un logiciel approprié. Entrez des valeurs biologiques triples pour produire des barres d’erreur et indiquer la variabilité du dosage.
- Pour examiner les changements dans la viabilité cellulaire au cours de la période expérimentale, effectuez un test d’analyse de variance (ANOVA) à un facteur avec de multiples comparaisons des moyennes à l’aide d’un logiciel biostatistique approprié.
- Indiquez les différences significatives entre les points temporels sur les graphiques sous la forme ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) et **** (P≤0,0001).
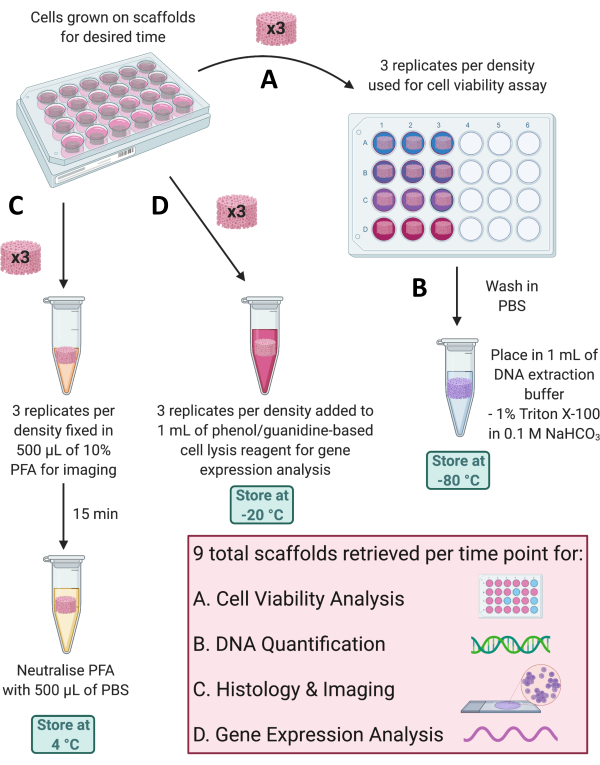
Figure 4 : Récupération d’échafaudages pour différentes analyses à chaque point temporel. (A) Trois répétitions d’échafaudage sont prélevées pour l’analyse de la viabilité cellulaire. (B) Ces échafaudages peuvent ensuite être lavés dans du PBS, placés dans du Triton X-100 à 1 % dans du NaHCO3 à 0,1 M et stockés à -80 °C pour la quantification de l’ADN. (C) Trois autres répétitions sont fixées dans du PFA à 10 % pendant 15 min, neutralisées dans du PBS et stockées à 4 °C pour coloration histologique et imagerie. (D) Enfin, 3 répétitions sont ajoutées à un réactif de lyse cellulaire à base de phénol/guanidine et stockées à -20 °C pour l’analyse de l’expression génique. Abréviations : PBS = solution saline tamponnée au phosphate ; PFA = paraformaldéhyde. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Quantification de l’ADN des cellules dans les échafaudages
NOTA : Comme mentionné dans la note après l’étape 5.1.7, la récupération des échafaudages pour la quantification de l’ADN consiste à placer les échafaudages dans des tubes à centrifuger de 2 mL contenant 1 mL de Triton X-100 à 1 % dans une solution de NaHCO3 à 0,1 M, puis à les entreposer à -80 °C. Avant que l’analyse de l’ADN puisse être effectuée, les cellules doivent subir trois cycles de gel-dégel pour lyser les cellules du neuroblastome de manière appropriée et libérer de l’ADN pour la quantification.- Retirez les échantillons précédemment stockés dans le Triton X-100 à -80 °C. Laisser les échantillons à l’état RT pendant 1 à 3 h ou jusqu’à ce qu’ils soient décongelés.
- Vaporiser les échantillons pendant 10 à 20 s et les remettre à -80 °C pendant 18 à 24 h ou jusqu’à ce qu’ils soient complètement congelés. Répétez ce processus pour un total de trois cycles de gel-dégel.
- Pour maximiser le rendement en ADN, utilisez un lyseur de tissus pour perturber les cellules dans les échafaudages.
- Placez une bille métallique dans le tube à centrifuger de 2 ml contenant un échafaudage en Triton X-100 et placez le tube dans l’adaptateur pour agiter l’échantillon à 50 oscillations/seconde pendant 2 à 3 minutes.
REMARQUE : Utilisez des tubes à centrifuger à fond rond car le cordon métallique peut se loger dans un tube à fond conique.
- Placez une bille métallique dans le tube à centrifuger de 2 ml contenant un échafaudage en Triton X-100 et placez le tube dans l’adaptateur pour agiter l’échantillon à 50 oscillations/seconde pendant 2 à 3 minutes.
- Quantifier l’ADN dans la solution Triton X-100 en appliquant une coloration fluorescente d’ADN double brin (ADNdb) et mesurer l’émission à l’aide d’un lecteur de microplaques. Reportez-vous aux directives du fabricant ; diluer les échantillons de manière appropriée dans un tampon d’acide tétraacétique (TE) Tris-éthylènediamine pour préparer 8 étalons d’ADNdb par dilution en série dans un tampon TE (Figure 5).
- Dans les plaques noires opaques à 96 puits, ajouter 100 μL de chaque étalon ou échantillon dans les puits en trois exemplaires techniques.
- Diluer la coloration fluorescente de l’ADNdb 200 fois dans un tampon TE et ajouter 100 μL à chaque étalon/échantillon à l’aide d’une pipette multicanal. Couvrir l’assiette de papier d’aluminium et incuber à RT pendant 5 min.
- Mesurez et enregistrez la fluorescence de chaque puits à 520 nm. Suivez les directives du fabricant pour calculer la concentration d’ADN dans chaque échantillon.
REMARQUE : Si la concentration moyenne d’ADN par cellule est connue pour la lignée cellulaire utilisée, les valeurs de concentration d’ADN peuvent être converties en nombre de cellules à l’aide de l’équation (4). (4)
(4) - Représenter graphiquement et analyser statistiquement les résultats de la quantification de l’ADN à l’aide d’un logiciel approprié. Entrez des valeurs biologiques triples pour produire des barres d’erreur et indiquer la variabilité du dosage.
- Pour examiner les changements dans la concentration d’ADN/nombre de cellules au cours de la période expérimentale, effectuez un test ANOVA à un facteur avec plusieurs comparaisons des moyennes à l’aide d’un logiciel biostatistique approprié.
- Indiquez les différences significatives entre les points temporels sur les graphiques sous la forme ns (P>0,05), * (P≤0,05), ** (P≤0,01), *** (P≤0,001) et **** (P≤0,0001).
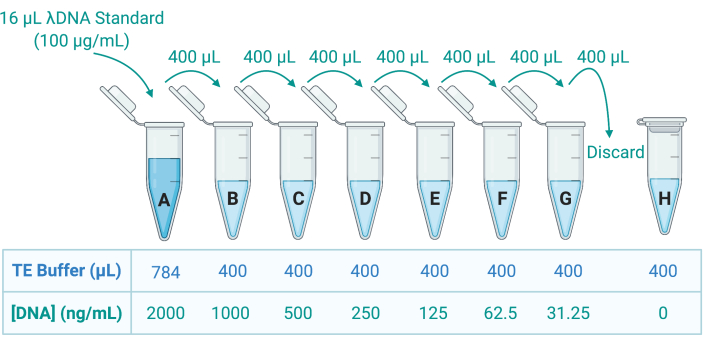
Figure 5 : Préparation de huit étalons d’ADN pour la génération d’une courbe standard. Une solution mère d’ADNλ est fournie à raison de 100 μg/mL. Celui-ci est dilué 50 fois dans un tampon TE pour créer l’étalon A à 2000 ng/mL ; 400 μL de A sont ensuite transférés dans le tube B, contenant 400 μL de tampon TE ; 400 μL de B sont ensuite transférés et dilués 2 fois dans C, et ainsi de suite jusqu’à G. La norme H est composée uniquement de tampon TE et a donc une concentration d’ADN de 0 ng/mL. Abréviation : TE = Tris-EDTA. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Préparation d’échafaudages pour la coloration histologique
REMARQUE : Les échafaudages peuvent être fixés et colorés à des fins de microscopie et d’imagerie, soit sous forme d’échafaudages entiers pour l’immunofluorescence (IF), soit sous forme de tranches fixées au formol enrobées de paraffine (FFPE) pour la coloration histologique ou l’immunohistochimie (IHC). Cela permet l’évaluation qualitative de la pénétration et de la distribution cellulaires dans les échafaudages et peut être utilisé pour évaluer l’expression des protéines.- Préparez une solution de paraformaldéhyde (PFA) à 10 % dans du PBS. Assurez-vous qu’il y a suffisamment de solution pour un volume final de 500 μL par échafaudage.
- Préchauffer cette solution à 37 °C et ajouter 500 μL dans des tubes à centrifuger de 2 mL étiquetés pour la récupération de l’échafaudage.
- Retirez les échafaudages des plaques à 24 puits non adhérentes (étape 4.4.4) et placez-les dans la hotte à flux laminaire.
- À l’aide d’une pince à épiler stérile, transférez les échafaudages dans les tubes à centrifuger étiquetés contenant 10 % de PFA. Laissez les échafaudages se fixer dans la solution de PFA pendant 15 min. Neutraliser le PFA en ajoutant 500 μL de PBS dans chaque tube et conserver à 4 °C.
- Pour préparer les échafaudages pour le préparateur automatique de tissus, retirez-les de 4 °C et placez-les au crayon à l’aide d’une pince à épiler dans des cassettes en plastique étiquetées avec tous les détails pertinents. Placez toutes les cassettes dans le récipient métallique du processeur de tissus.
- Commencez le protocole en 12 étapes sur le processeur de tissus pour fixer, déshydrater, nettoyer les échafaudages et les infiltrer avec de la paraffine pendant la nuit. Récupérez les cassettes contenant les échantillons traités.
- Ensuite, intégrez les échafaudages dans des blocs de cire de paraffine pour permettre la microtomie des échafaudages en tranches très minces pour la coloration.
REMARQUE : Il est particulièrement important de tenir compte de l’orientation des échafaudages lors de leur retrait des cassettes et de leur encastrement dans la cire, car cela affectera l’angle auquel les images sont prises. Ceci est important lors de l’évaluation de l’infiltration cellulaire dans les échafaudages. - Allumez l’embedder de cire et la plaque froide ; Soulevez le couvercle pour vérifier le niveau de la cire. Remplissez-le si nécessaire.
- En travaillant avec un échantillon à la fois, ouvrez la cassette, retirez l’échafaudage et centrez-le dans le moule en plastique.
- Versez de la cire chaude sur l’échantillon, en veillant à maintenir une orientation correcte et en ajustant avec une pince à épiler chaude, si nécessaire, avant que la cire ne se solidifie. Versez plus de cire pour remplir le moule.
- Placez le couvercle de la cassette étiquetée sur le dessus du moule en plastique et ajoutez de la cire sur le dessus. Placez le moule sur la plaque froide pour solidifier la cire. Conserver toute la nuit à 4 °C pour s’assurer que la cire de paraffine est complètement solidifiée avant la microtomie.
- Pour vous préparer à la microtomie, allumez un bain-marie à 35 °C, la plaque de séchage et le microtome.
- Insérez une lame dans le support et serrez le levier pour le fixer.
- Définissez l’épaisseur de la garniture et de la section, généralement 5 mm pour les sections d’échafaudage.
- Retirez l’échafaudage FFPE du moule, fixez-le dans le support à l’avant du microtome et coupez soigneusement l’excès de cire sur les bords de l’échantillon avant de couper les sections.
- Commencez à découper le bloc de cire en tournant le levier du microtome, en assurant un mouvement fluide.
- Rassemblez des sections en forme de ruban, environ 3 sections d’échafaudage à la fois, et placez-les délicatement dans le bain-marie à 35 °C pour éliminer les plis. Séparez délicatement les sections dans le bain-marie à l’aide d’une pince à épiler.
- À l’aide de lames de microscope en verre recouvertes de polysine, soulevez chaque section du bain-marie de manière à ce qu’elle repose au centre de la lame. Étiquetez chaque diapositive à l’aide d’un crayon.
- Placez les lames de verre sur la plaque de séchage ou dans un four de séchage à 60 °C. Une fois séchés, conservez-les à 4 °C et procédez à la coloration histologique ou IHC requise.
- Récupération d’échafaudages pour l’analyse de l’expression génique
- Retirez les échafaudages des plaques à 24 puits non adhérentes de l’incubateur (étape 4.4.4) et placez-les dans la hotte à flux laminaire.
- À l’aide d’une pince à épiler stérile, transférer les échafaudages dans des tubes à centrifuger de 2 ml fraîchement étiquetés.
- Dans une hotte, ajoutez 1 mL d’un réactif de lyse cellulaire à base de phénol/guanidine dans chaque tube pour lyser les cellules dans des échafaudages et permettre la récupération d’ARN de haute qualité.
- Conservez les tubes à -20 °C jusqu’à ce qu’ils soient prêts à effectuer l’extraction de l’ARN à l’aide d’un kit approprié. À l’aide d’une réaction en chaîne par polymérase quantitative à transcription inverse standard (RT-qPCR)17, évaluer l’expression des gènes dans les cellules des échafaudages.
Résultats
Le modèle d’échafaudage à base de collagène décrit ici a de nombreuses applications allant de l’étude de la biologie du neuroblastome au criblage de thérapies anticancéreuses dans un environnement qui est plus physiologiquement similaire aux tumeurs natives que la culture cellulaire 2D conventionnelle. Avant de tester une question de recherche donnée, il est crucial d’obtenir une caractérisation complète de l’attachement, de la prolifération et de l’infiltration cellulaires dans le délai expérimental souhaité. Les conditions de croissance dépendront de la biologie de chaque lignée cellulaire spécifique. Il est important de noter que plusieurs méthodes d’évaluation de la croissance cellulaire doivent être mises en œuvre pour déterminer les conditions optimales et les performances robustes.
Ici, la viabilité des cellules de neuroblastome cultivées sur des échafaudages a été évaluée à l’aide d’un test colorimétrique de viabilité cellulaire. Ce test peut être effectué aussi souvent que souhaité tout au long de la période expérimentale. Pour l’expérience décrite, l’évaluation de la viabilité cellulaire a été effectuée aux jours 1, 7 et 14 pour deux lignées cellulaires de neuroblastome, KellyLuc et IMR32, cultivées sur des échafaudages Coll-I-nHA à 4 densités différentes (Figure 6). La viabilité au jour 1 a été établie comme base de référence pour comparer toutes les mesures subséquentes. Le taux de réduction du réactif de viabilité cellulaire reflète la biologie cellulaire et les caractéristiques de croissance des lignées cellulaires individuelles, y compris leurs taux de prolifération et leur métabolisme. On s’attendait à une corrélation entre le nombre de cellules ensemencées sur les échafaudages et le niveau de réduction. Dans cette expérience, la réduction du réactif de viabilité cellulaire a généralement augmenté à chaque point temporel pour les deux lignées cellulaires à toutes les densités, comme prévu.
Chaque densité a ensuite été évaluée individuellement pour les deux lignées cellulaires afin de comparer la réduction à travers les points temporels. Une ANOVA à un facteur avec le test de comparaisons multiples de Tukey a été réalisée pour détecter des différences significatives de réduction entre les points temporels (Figure 7). Pour les lignées cellulaires et toutes les densités d’ensemencement, il y a eu une augmentation significative (P<0,05) de la réduction du réactif de viabilité cellulaire lorsque l’on compare le jour 1 et le jour 14. Cela indiquait une augmentation significative des cellules métaboliquement actives présentes sur les échafaudages. Cette augmentation n’a pas été significative dans tous les cas lors de l’évaluation des intervalles de 7 jours (jour 1 vs jour 7, jour 7 vs jour 14), démontrant l’importance de l’optimisation de la densité de semis pour atteindre la fenêtre de croissance souhaitée.
Pour étayer les résultats du test de viabilité cellulaire, la croissance cellulaire sur les échafaudages peut également être mesurée indirectement par la quantification de l’ADNdb extrait des échafaudages à l’aide d’une coloration fluorescente de l’ADNdb (Figure 8A). Comme la viabilité cellulaire, la quantification de l’ADN peut être effectuée aussi souvent que souhaité dans le cadre de la chronologie expérimentale. Cependant, cette analyse nécessite la récupération complète des échafaudages et l’arrêt de la croissance cellulaire et doit donc être prise en compte dans la planification expérimentale, comme nous l’avons vu à la section 1. Pour cette expérience, l’ADN a été quantifié aux jours 1, 7 et 14 pour deux lignées cellulaires de neuroblastome, KellyLuc et IMR32, cultivées sur des échafaudages Coll-I-nHA à 4 densités différentes. Comme la concentration moyenne d’ADNdb par cellule est connue pour ces lignées cellulaires, il a été possible de déduire le nombre de cellules par échantillon à partir de l’ADN quantifié (Figure 8B).
La quantification de l’ADN a donné lieu à une variabilité plus élevée entre les répétitions biologiques que l’évaluation de la viabilité cellulaire, mais a généralement augmenté pour chaque point de temps, les niveaux les plus élevés étant quantifiés au jour 14. Les cellules IMR32 semblent atteindre un nombre de cellules plus élevé sur les échafaudages Coll-I-nHA, comme l’indique la concentration d’ADN, que les cellules KellyLuc. Chaque densité a ensuite été évaluée individuellement pour les deux lignées cellulaires afin de comparer la réduction à travers les points temporels. Une ANOVA à un facteur avec le test de comparaisons multiples de Tukey a été réalisée pour détecter des différences significatives de réduction entre les points temporels (Figure 8B).
Pour les lignées cellulaires et toutes les densités d’ensemencement, on a observé une augmentation significative (P<0,05) du nombre de cellules lorsque l’on compare le jour 1 et le jour 14, à l’exception de KellyLuc à la densité de semis 4 (1 × 105 cellules/échafaudage), qui n’a donné lieu à aucune augmentation significative à aucun des points temporels. À l’instar des résultats de la viabilité cellulaire, les augmentations n’étaient pas significatives dans tous les cas lors de l’évaluation des intervalles de 7 jours (jour 1 vs jour 7, jour 7 vs jour 14). Lorsque l’on compare les tendances temporelles de la viabilité cellulaire et de la quantification de l’ADN, il y a de légères différences entre les deux analyses. Cependant, dans l’ensemble, des tendances similaires ont été observées, les valeurs moyennes augmentant entre les intervalles de 7 jours pour la plupart des densités. Cela démontre l’importance de surveiller la croissance cellulaire à l’aide de plus d’une méthode.
Une évaluation visuelle de la morphologie et de la distribution de la croissance cellulaire sur les échafaudages a ensuite été mise en œuvre, englobant la coloration traditionnelle à l’hématoxyline et à l’éosine (H&E) ainsi que l’IHC. On s’attend à ce que les différents modèles de croissance des lignées cellulaires individuelles conduisent à des arrangements spatiaux variés sur les échafaudages, y compris différents degrés de pénétration dans l’échafaudage et de regroupement cellulaire. Les échafaudages ont été fixés au formol, enrobés de paraffine et coupés en sections de 5 mm (figure 9A), préparant ainsi les échafaudages à de multiples techniques de visualisation, y compris la coloration histologique et l’IHC.
Une coloration H&E de routine a été appliquée aux cellules Kelly, KellyCis83 et IMR32 cultivées sur des échafaudages à base de collagène aux jours 1, 7 et 14 (Figure 9B). Cela a permis de visualiser l’orientation spatiale des cellules sur deux échafaudages à base de collagène sur une période de 14 jours. Des cellules de Kelly sensibles au cisplatine et des cellules de KellyCis83 résistantes ont été cultivées sur des échafaudages Coll-I-nHA (Figure 9B, i) et Coll-I-GAG (Figure 9B, ii). Conformément aux données publiées précédemment, les cellules KellyCis83 se sont développées à un rythme plus élevé et se sont infiltrées plus profondément dans les deux compositions d’échafaudage que la lignée cellulaire Kelly moins invasive. La coloration H&E d’une autre lignée cellulaire de neuroblastome, IMR32, cultivée sur Coll-I-nHA montre un schéma de croissance contrasté (Figure 9B, iii). Cette lignée cellulaire s’est développée en grandes grappes densément tassées sur les échafaudages de collagène au cours de la période de 14 jours. La microscopie confocale à fond clair peut être utilisée pour visualiser l’architecture poreuse des échafaudages à base de collagène (Figure 9C) en raison de l’autofluorescence des fibres de collagène.
Nous avons coloré des cellules avec de la phalloïdine ciblant l’actine du cytosquelette et la contre-coloration nucléaire, le 4′,6-diamidino-2-phénylindole (DAPI), afin de surveiller des traits cellulaires spécifiques tout au long de la chronologie expérimentale. Une abondance d’actine a été observée dans les cellules Kelly et KellyCis83 sur des échafaudages Coll-I-GAG en utilisant cette technique (Figure 9D). Ces résultats démontrent comment plusieurs techniques d’imagerie peuvent être utilisées pour obtenir des informations résolues spatialement à partir de cellules de neuroblastome cultivées sur des échafaudages à l’aide de ce protocole. Cette caractérisation des modèles de croissance cellulaire sur des échafaudages à base de collagène sur une période donnée permettra d’améliorer la compréhension et l’interprétation des tests biochimiques en aval.
L’expression des protéines par les cellules cultivées sur des échafaudages à base de collagène peut être analysée pour comparer l’activité cellulaire à des scénarios in vivo . Des données publiées précédemment ont examiné l’expression de la chromogranine A (CgA) en tant que marqueur sécrété de substitution du neuroblastome par les cellules KellyLuc et KellyCis83Luc cultivées dans des monocouches cellulaires ainsi que sur des échafaudages Coll-I-nHA et Coll-I-GAG (Figure 10). La CgA a été évaluée dans les milieux conditionnés à l’aide d’un test immuno-enzymatique (ELISA) (Figure 10A). Le CgA est sécrété à un taux plus élevé dans la lignée cellulaire KellyCis83, plus agressive et résistante à la chimiothérapie, que dans Kelly (Figure 10B,C). Cela était significatif au jour 7 sur les échafaudages Coll-I-GAG et Coll-I-nHA (P<0,05), alors qu’il n’y avait pas de différence significative à ce stade pour les cellules cultivées en monocouche par culture 2D conventionnelle.
Ces résultats mettent également en évidence le calendrier expérimental restreint lors de la croissance de cellules en monocouche, avec seulement 7 jours de croissance s’avérant réalisable avant que les cellules n’atteignent la confluence. La croissance des cellules sur les échafaudages permet de surmonter cette limitation car elles peuvent être maintenues sur une plus longue période dans des conditions physiologiquement plus pertinentes. La combinaison ci-dessus de techniques permettant d’acquérir des informations sur la viabilité cellulaire, le contenu en ADN, la morphologie cellulaire et l’arrangement spatial, ainsi que les profils d’expression, facilite l’évaluation de la croissance des cellules de neuroblastome sur une gamme d’échafaudages à base de collagène. Ce protocole peut également être facilement adapté pour répondre à des exigences expérimentales spécifiques et aux applications souhaitées.
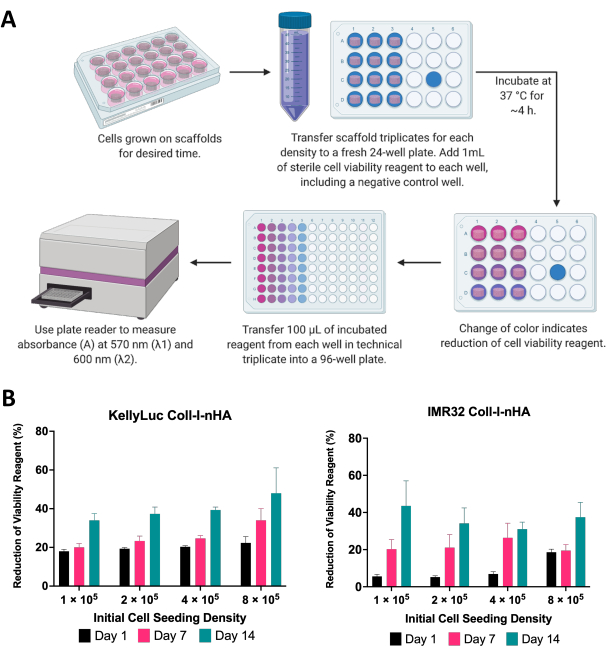
Figure 6 : Analyse de la viabilité cellulaire. (A) Procédure générale de mesure de la viabilité des cellules de neuroblastome sur des échafaudages à base de collagène à l’aide d’un test colorimétrique de viabilité cellulaire. La période d’incubation doit être optimisée pour chaque nouvelle lignée cellulaire, en se référant aux directives du fabricant. (B) Pourcentage de réduction du réactif de viabilité cellulaire par les cellules KellyLuc et IMR32 cultivées sur des échafaudages Coll-I-nHA à quatre densités initiales de semis différentes, mesurées aux jours 1, 7 et 14. Les échantillons ont été évalués en trois exemplaires biologiques, les barres d’erreur représentant l’écart-type. Abréviations : nHA = nanohydroxyapatite ; Coll-I-nHA = échafaudages de collagène complétés par du nHA. Veuillez cliquer ici pour voir une version agrandie de cette figure.
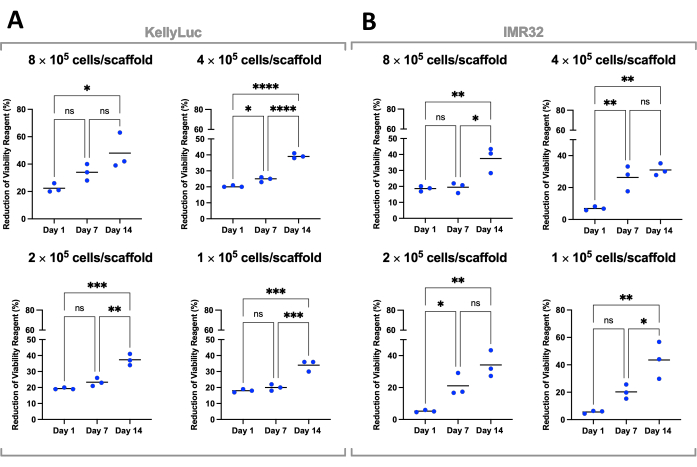
Figure 7 : Viabilité cellulaire selon la densité d’ensemencement des cellules cultivées sur Coll-I-nHA sur une période de 14 jours. a) KellyLuc ; (B) IMR32. Les numéros de cellules titrés font référence à la densité initiale d’ensemencement des cellules sur les échafaudages au jour 0. Les échantillons ont été évalués en trois exemplaires biologiques, indiqués par des points triples, avec des barres représentant la moyenne. L’ANOVA à un facteur avec comparaisons multiples a été utilisée pour détecter des différences significatives dans le pourcentage de réduction des réactifs de viabilité cellulaire entre les trois points temporels, notés sur les graphiques (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abréviations : nHA = nanohydroxyapatite ; Coll-I-nHA = échafaudages de collagène supplémentés en nHA ; ANOVA = analyse de la variance ; ns = non significatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
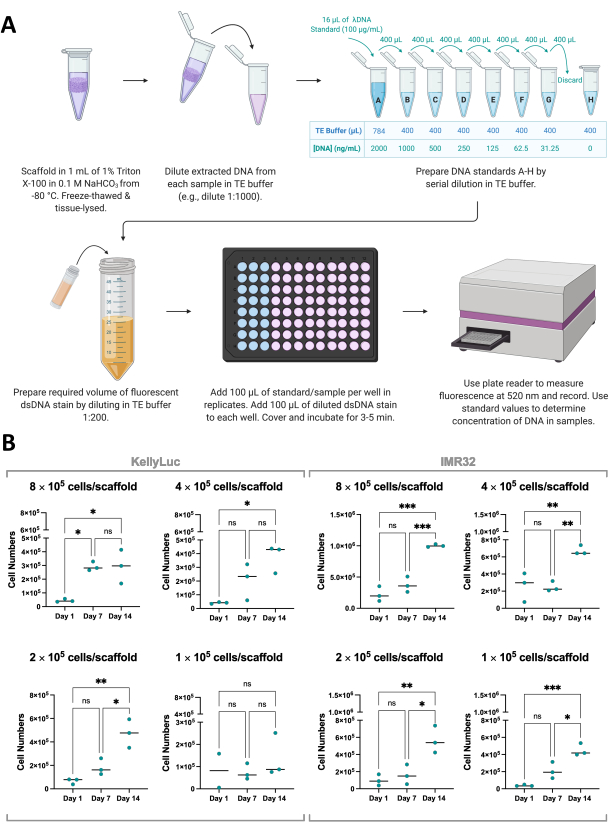
Figure 8 : Quantification de l’ADN extrait des cellules dans les échafaudages. (A) Processus de quantification de l’ADNdb à partir de cellules cultivées sur des échafaudages à base de collagène à l’aide d’une coloration fluorescente de l’ADNdb. (B) Nombre de cellules à partir de l’analyse de quantification de l’ADN par densité d’ensemencement pour les cellules KellyLuc et IMR32 cultivées sur Coll-I-nHA sur une période de 14 jours. Les numéros de cellules titrés font référence à la densité initiale d’ensemencement des cellules sur les échafaudages au jour 0. Les échantillons ont été évalués en trois exemplaires biologiques, indiqués par des points triples, avec des barres représentant la moyenne. L’ANOVA à un facteur avec comparaisons multiples a été utilisée pour détecter des différences significatives dans le nombre de cellules entre les trois points temporels, notées sur les graphiques (ns P > 0,05, * P ≤ 0,05, ** P ≤ 0,01, *** P ≤ 0,001, **** P ≤ 0,0001). Abréviations : nHA = nanohydroxyapatite ; Coll-I-nHA = échafaudages de collagène supplémentés en nHA ; ADNdb = ADN double brin ; TE = Tris-EDTA ; ANOVA = analyse de la variance ; ns = non significatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
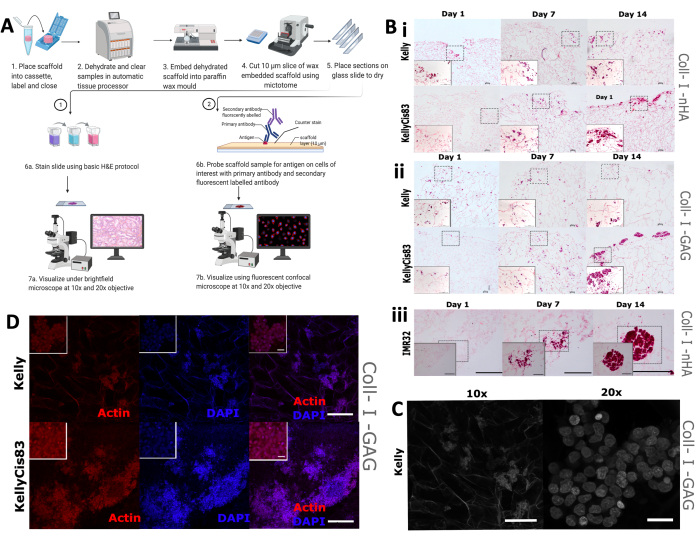
Figure 9 : Étapes de traitement tissulaire pour l’analyse immunohistochimique des échafaudages. (A) Représentation schématique du protocole de traitement des échafaudages pour l’analyse d’images. Ce procédé permet une coloration histologique de routine et un sondage d’anticorps spécifiques à l’aide d’anticorps primaires et d’anticorps secondaires marqués par fluorescence. (B) Images représentatives de trois lignées cellulaires de neuroblastome soumises à une coloration H&E. Des images H&E sont prises aux jours 1, 7 et 14 pour surveiller les modèles de croissance au cours de l’expérience. Barre d’échelle = 200 μm. Les carrés en pointillés représentent la zone qui a été choisie pour les images agrandies 20x sur le bord inférieur gauche. Barre d’échelle = 20 μm. (i et ii) H&E des lignées cellulaires de neuroblastome Kelly et KellyCis83 (panneaux supérieur et inférieur, respectivement) sur deux types d’échafaudages à base de collagène. (iii) H&E de la lignée cellulaire IMR32, représentant la croissance cellulaire groupée sur l’échafaudage Coll-I-nHA. (C) Image représentative de la lignée cellulaire de Kelly, soumise à la microscopie confocale à fond clair. L’autofluorescence du collagène permet de visualiser l’échafaudage poreux. Barre d’échelle 10x = 200 μm, barre d’échelle 20x = 20 μm. (D) Image représentative d’échafaudages encastrés suivie d’une analyse par IHC avec de la phalloïdine et du DAPI à un grossissement de 10x, Barre d’échelle = 200 μm. Les carrés intérieurs plus petits représentent les images agrandies (20x), barre d’échelle = 20 μm. Abréviations : nHA = nanohydroxyapatite ; Coll-I-nHA = échafaudages de collagène supplémentés en nHA ; GAG = glycosaminoglycane ; Coll-I-GAG = échafaudages de collagène supplémentés en chondroïtine-6-sulfate ; H&E = hématoxyline et éosine ; IHC = immunohistochimie ; DAPI = 4′,6-diamidino-2-phénylindole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
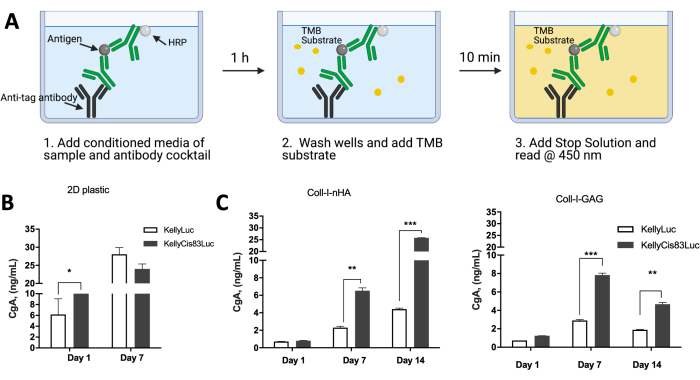
Figure 10 : Expression des protéines par les cellules de neuroblastome cultivées sur des échafaudages 3D à base de collagène par rapport au plastique 2D. (A) Un schéma de la façon dont l’ELISA CgA a été réalisée sur des milieux conditionnés de cellules cultivées sur des échafaudages 2D en plastique ou 3D à base de collagène. (B) Niveaux d’expression de la protéine CgA prélevés dans des milieux conditionnés de cellules cultivées sur une monocouche de plastique 2D. Comme les cellules ont atteint la confluence après 7 jours, le point de temps de 14 jours n’était pas lisible. Au jour 7 sur le plastique, il n’y avait pas de différence significative dans les niveaux de CgA entre les lignées cellulaires Kelly et KellyCis83. (C) L’ELISA CgA a été réalisée à l’aide de milieux conditionnés de cellules cultivées sur des échafaudages à base de collagène pendant 14 jours consécutifs. Au jour 7, sur les deux échafaudages de collagène, les niveaux de CgA sont plus élevés dans la lignée cellulaire KellyCis83 plus agressive, ce qui met en évidence des niveaux physiologiquement pertinents de CgA dans la matrice 3D par rapport à la monocouche 2D. Ce chiffre a été modifié à partir de Curtin et al.17. Abréviations : 3D = tridimensionnel ; 2D = bidimensionnel ; CgA = chromogranine A ; ELISA = dosage immuno-enzymatique ; nHA = nanohydroxyapatite ; Coll-I-nHA = échafaudages de collagène supplémentés en nHA ; GAG = glycosaminoglycane ; Coll-I-GAG = échafaudages de collagène supplémentés en chondroïtine-6-sulfate ; TMB = 3,3',5,5'-tétraméthylbenzidine ; HRP = peroxydase de raifort. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le modèle 3D de cellules cancéreuses d’échafaudage s’est avéré être un outil précieux et polyvalent pour obtenir des informations mécanistiques sur la croissance, la viabilité et l’infiltration des cellules du neuroblastome dans un TME32 simplifié. Le modèle 3D de neuroblastome décrit ici imite l’ETM minimale et fournit des données physiologiquement plus pertinentes qu’une culture monocouche 2D. L’un des principaux inconvénients de la culture cellulaire 3D est l’augmentation de la complexité expérimentale et l’allongement des délais. Nous décrivons ici un protocole optimisé pour l’ensemencement, la croissance et le maintien des cellules de neuroblastome sur des échafaudages à base de collagène, suivis d’analyses et d’applications en aval, permettant une caractérisation robuste de la croissance cellulaire. Notre objectif était d’obtenir des informations sur la densité optimale d’ensemencement cellulaire pour les échafaudages afin de créer un environnement prévisible et contrôlable pour l’évaluation des traitements médicamenteux anticancéreux dans une fenêtre expérimentale rapide de 14 jours. La combinaison de tous ces protocoles simples décrits fournit une évaluation complète de la croissance des cellules de neuroblastome dans le système de culture in vitro basé sur un échafaudage.
Les points critiques dans la mise en place du protocole ont été soulignés pour permettre aux scientifiques d’établir rapidement la même chose dans leurs laboratoires. Par exemple, les temps d’incubation indiqués pour une meilleure performance du test colorimétrique de viabilité cellulaire permettent une pénétration plus profonde du réactif dans les pores de l’échafaudage pour atteindre toutes les cellules. De plus, la technique de coloration de l’ADNdb fluorescent est robuste et simple ; cependant, la libération d’ADN à partir des échafaudages nécessite une lyse cellulaire vigoureuse car les cellules sont « piégées » dans les fibres de collagène.
À l’aide du test simple de quantification de l’ADN décrit, nous pouvons identifier la phase de croissance logarithmique sur des échafaudages à base de collagène pour le criblage de médicaments anticancéreux à l’aide de ce modèle. Dans le cadre expérimental décrit, 4 densités initiales d’ensemencement cellulaire ont été utilisées avec une période globale de 14 jours et des points de temps d’analyse aux jours 1, 7 et 14. Nous avons identifié que les cellules KellyLuc ensemencées à 4 × 105 cellules/échafaudage ont la fenêtre de prolifération la plus active entre les jours 7 et 14. Ces données de croissance en phase logarithmique permettront une interprétation fiable de diverses expériences de cytotoxicité cellulaire. Il élimine les spéculations sur un déclin de la croissance ou de la mort cellulaire résultant d’une croissance supprimée sur la plate-forme poreuse 3D plutôt que d’une toxicité médicamenteuse. La viabilité cellulaire est également une évaluation largement utilisée pour l’adéquation des plates-formes 3D à la croissance de différents types de cellules33,34. Bien qu’il existe de nombreux tests pour mesurer la viabilité des cellules, y compris la coloration vivante/morte, la mesure de l’ATP, les tests de prolifération, nous avons trouvé que l’utilisation du test colorimétrique de viabilité cellulaire Alamar Blue était une technique simple et efficace pour soutenir les données de quantification de l’ADN.
L’utilisation combinée de la quantification de l’ADN et de la viabilité cellulaire a fourni des preuves complémentaires qu’en moyenne, la densité optimale des cellules germées sur l’échafaudage pour obtenir une croissance continue sur une période de 14 jours est de 2 à 4 × 105 cellules/échafaudage. Cependant, ce protocole peut facilement être adapté pour répondre à différents délais expérimentaux, points de temps d’analyse et applications en aval. Bien que ce protocole décrive l’évaluation de la croissance cellulaire en monoculture de cellules de neuroblastome sur des échafaudages, les échafaudages sont facilement modifiables pour être utilisés comme plate-forme de co-culture, décrite par do Amaral et al., qui ont utilisé des échafaudages de collagène-GAG pour co-cultiver des kératinocytes et des fibroblastes dans une étude sur la cicatrisation des plaies35.
Le modèle 3D décrit permet de visualiser la croissance et l’infiltration cellulaires à l’aide de différentes techniques bien connues, telles que l’immunofluorescence et l’H&E standard. Il est important de visualiser les cellules ainsi que la caractérisation de la croissance à l’aide de tests biochimiques en raison de la diversité de la morphologie cellulaire et des modèles de croissance sur les échafaudages. La compréhension du modèle de croissance peut fournir des informations sur le comportement de croissance et la réponse future aux médicaments anticancéreux. Par exemple, la croissance d’IMR32 à l’aide de la quantification de l’ADN donne des modèles similaires à ceux de Kelly, bien qu’après visualisation à l’aide de H&E, IMR32 se développe en grappes plus grandes que Kelly, qui a montré une croissance plus dispersée (Figure 9). Ces modèles de croissance variés des lignées cellulaires dans les échafaudages reflètent le scénario clinique de l’hétérogénéité tumorale. L’examen de la réponse aux médicaments anticancéreux à l’aide d’un panel de lignées cellulaires de morphologies différentes dans des échafaudages 3D augmentera la valeur prédictive de la réponse des patients aux mêmes médicaments.
La détection de l’expression d’un gène ou d’une protéine peut également être réalisée à l’aide d’autres approches telles que la RT-qPCR ou l’ELISA si la protéine d’intérêt est sécrétée. Un marqueur de substitution de la progression du neuroblastome, la chromogranine A (CgA)36, a été utilisé pour caractériser en outre la croissance cellulaire du neuroblastome en 3D. Comme décrit dans les travaux précédents17, la sécrétion de CgA augmentait à mesure que les cellules proliféraient (Figure 10). Bien que la culture cellulaire monocouche n’ait pas pu capturer cette augmentation, car la prolifération signifiait que les cellules atteignaient leur pleine confluence dans les boîtes de culture, l’utilisation des échafaudages de collagène 3D a permis une évaluation prolongée de la sécrétion de CgA.
Ce modèle 3D in vitro peut ne pas convenir à toutes les questions de recherche pour étudier la biologie du neuroblastome et la réponse aux traitements. L’une des limites est la pénétration inégale des cellules dans les échafaudages et la formation d’amas cellulaires de taille variable, qui dépendent d’une lignée cellulaire donnée et peuvent conduire à une diffusion incontrôlable des nutriments et des médicaments testés. Cette caractéristique affecte la robustesse du dépistage thérapeutique. Cependant, malgré cette limitation, il est important de considérer que les tumeurs natives sont également hétérogènes en taille et en distribution des cellules cancéreuses et contiennent de nombreux autres types de cellules dans le tissu tumoral. Pour pallier cette limitation, nous proposons l’utilisation de chaque échafaudage peuplé de cellules comme un seul microtissu pour lequel les paramètres suivants seront optimisés : (a) les temps d’incubation pour que le réactif de viabilité cellulaire atteigne les cellules et les amas de cellules, et (b) la lyse des cellules dans le tampon Triton X-100 par prétraitement des cellules sur des échafaudages avec un lyseur tissulaire pour libérer l’ADN des cellules contenues en profondeur dans l’échafaudage.
Une autre limite technique de ce protocole est l’absence de tests mécaniques de chaque lot d’échafaudages nouvellement fabriqués pour ce modèle. Cependant, l’utilisation du processus de fabrication robuste des échafaudages, qui a été largement caractérisé en relation avec les propriétés physiques et chimiques des échafaudages, telles que le module de compression et de traction, la porosité et la structure visuelle des pores, et l’homogénéité, garantit que les qualités de l’échafaudage sont maintenues tout au long des lots 21,24,27,30,37.
En résumé, cet article présente une série de méthodes simples pour l’analyse de la croissance cellulaire sur des échafaudages à base de collagène. La chronologie expérimentale et les points d’analyse peuvent être intervertis en fonction des questions de recherche spécifiques. Ce protocole est également adaptable à d’autres types de cellules. Les résultats présentés ci-dessus fournissent des preuves de la façon dont cette compilation de méthodes a donné un aperçu de la densité d’ensemencement optimale pour diverses lignées cellulaires de neuroblastome afin de créer une croissance continue sur 14 jours. L’amalgame des résultats obtenus à partir de toutes les méthodes de ce protocole permet une meilleure compréhension de la croissance cellulaire dans la matrice de collagène 3D. L’utilisation future de ce modèle impliquera probablement des systèmes de co-culture spécifiques au neuroblastome TME et la mise à l’essai de divers nouveaux médicaments anticancéreux.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par le National Children’s Research Centre (NCRC), l’Irish Research Council (IRC) et Neuroblastoma UK. Les illustrations ont été créées à l’aide de BioRender.
matériels
| Name | Company | Catalog Number | Comments |
| Cells | |||
| IMR-32 | ATCC | CCL-127 | |
| Kelly | ECACC | 82110411 | |
| KellyCis83 | Made in lab – derived from Kelly (Piskareva et al., 2015) | - | Increasing exposure to cisplatin. Cross resistance acquired |
| SH-SY5Y | ATCC | CRL-2266 | |
| Disposable | |||
| 0.22 µm syringe filter | Millex | SLHP033RS | |
| 1.5 mL Eppendorf tube | Eppendorf | 0030 120.086 | |
| 100 mL sterile Pot | Starstedt | - | |
| 10 mL plastic pipette | Cellstar | 607 180 | |
| 15 mL Falcon tube | Starstedt | 62.554.502 | |
| 25 mL plastic pipette | Cellstar | 760 180 | |
| 50 mL Falcon tube | Starstedt | 62.547.254 | |
| 5 mL plastic pipette | Cellstar | 606 180 | |
| 6 mm Biopsy punches | Kai Medical | BP-60F | |
| Aluminium foil | - | - | |
| Cover Slip | Menzel-Glaser | - | |
| HYPERflask | Corning | CLS10030 | |
| Microscope slides | Thermo Scientific | J1840AMNT | |
| Opaque black 96-well plate | Costar | 3915 | |
| Sterile P10 tips | Starlab | S1121-3810 | |
| Sterile P1000 tips | Starlab | S1122-1830 | |
| Sterile P20 tips | Starlab | S1123-1810 | |
| Sterile P200 tips | Starlab | S1120-8810 | |
| T-175 (175 cm2 flask) | Sarstedt | 83.3912 | |
| T-75 (75 cm2 flask) | Sarstedt | 83.3911.302 | |
| Translucent clear 96 well plate | Cellstar | 655180 | |
| Translucent non-adherent 24 well plates | Cellstar | 83.3922.500 | |
| Equipment | |||
| Autoclave | Astell | - | |
| Automatic tissue processor | Leica | TP1020 | |
| Centrifuge 5804 | Eppendorf | - | |
| Hemocytometer | Hausser Scientific | - | |
| Incubator | ThermoScientific | - | |
| Microtome | Leica | RM2255 | |
| Oven | Memmert | Calibrated by: Cruinn diagnostics Ltd | |
| P10 pipette | Gilson | ||
| P100 pipette | Gilson | ||
| P1000 pipette | Gilson | ||
| P20 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| P200 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| Paraffin section flotation bath | Electrothermal | MH8517 | Calibrated by: Cruinn diagnostics Ltd |
| Pipette electronic dispenser | Corning | StripipetterUltra | Calibrated by: Cruinn diagnostics Ltd |
| Plate cooler | Leica | EG1140C | Calibrated by: Cruinn diagnostics Ltd |
| Refrigerator -20 °C | Liebherr | - | |
| Refrigerator -80 °C | Liebherr | - | |
| Refrigerator 4 °C | Liebherr | - | |
| Seesaw Rocker | DLAb | SK-D1807-E | |
| Spectrophotometer – Victor3V Platereader | PerkinElmer | 1420 | |
| Tissue culture hood/Laminar flow hood | GMI | 8038-30-1044 | |
| Tissue Lyser | Qiagen | TissueLyser LT | |
| Tweezers | - | - | |
| Water bath | Grant | - | |
| Wax embedder | Leica | EG1140H | |
| Materials | |||
| 1 L Water | Adrona - Biosciences | 568 | |
| 1% Triton-X | Sigma Aldrich | 9002-93-1 | |
| 10x PBS tablets | Sigma Aldrich | P4417-100TAB | |
| 37% paraformaldehyde | Sigma-Aldrich | F8775 | |
| Alamar Blue Cell Viability Reagent | Invitrogen | DAL1100 | |
| Collagen- glycosaminoglycan scaffold | Tissue engineering research group (TERG) | ||
| Collagen-nanohydroxyapatite scaffold | Tissue engineering research group (TERG) | ||
| dH20 | Adrona - Biosciences | 568 | |
| Eosin | Sigma-Aldrich | E4009 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| EtOH | Sigma-Aldrich | 1.00983.2500 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| F12 | Gibco | 21765-029 | |
| FBS | Gibco | 10270-106 | |
| Hemaytoxylin | Sigma-Aldrich | HHS32-1L | |
| L-Glutamine | Gibco | 25030-024 | |
| MEM | Gibco | 21090-022 | |
| miRNA easy Kit | Qiagen | 217004 | |
| MNEAA’s | Gibco | 11140-035 | |
| Penicillin/streptomycin | Gibco | 015140-122 | |
| Qiazol | Qiagen | 79306 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Invitrogen | P11496 | |
| RPMI | Gibco | 21875-034 | |
| Sodium bicarbonate | Sigma Aldrich | S7795-500G | |
| Tissue embedding Medium | Sigma | A6330-4LB | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Software | |||
| Excel | - | Excel 2016 | |
| ImageJ | - | - | |
| Prism | - | Version 9 |
Références
- Davidoff, A. M. Neuroblastoma. Seminars in Pediatric Surgery. 21 (1), 2-14 (2012).
- Matthay, K. K., et al. Neuroblastoma. Nature Reviews Disease Primers. 2, 16078 (2016).
- Costard, L. S., Hosn, R. R., Ramanayake, H., O'Brien, F. J., Curtin, C. M. Influences of the 3D microenvironment on cancer cell behaviour and treatment responsiveness: a recent update on lung, breast and prostate cancer models. Acta Biomaterialia. , (2021).
- Borriello, L., Seeger, R. C., Asgharzadeh, S., Declerck, Y. A. More than the genes, the tumor microenvironment in neuroblastoma. Cancer Letters. 380 (1), 304-318 (2016).
- Walker, C., Mojares, E., Del Río Hernández, A. Role of extracellular matrix in development and cancer progression. International Journal of Molecular Sciences. 19 (10), 3028 (2018).
- Bissell, M. J., Hall, H. G., Parry, G. How does the extracellular matrix direct gene expression. Journal of Theoretical Biology. 99 (1), 31-68 (1982).
- Schultz, G. S., Davidson, J. M., Kirsner, R. S., Bornstein, P., Herman, I. M. Dynamic reciprocity in the wound microenvironment. Wound Repair and Regeneration. 19 (2), 134-148 (2011).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6, 11 (2008).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4, 38 (2006).
- Ouellette, J. N., et al. Navigating the collagen jungle: The biomedical potential of fiber organization in cancer. Bioengineering. 8 (2), 1-19 (2021).
- Kreger, S. T., Voytik-Harbin, S. L. Hyaluronan concentration within a 3D collagen matrix modulates matrix viscoelasticity, but not fibroblast response. Matrix Biology. 28 (6), 336-346 (2009).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Hume, R. D., et al. Tumour cell invasiveness and response to chemotherapeutics in adipocyte invested 3D engineered anisotropic collagen scaffolds. Scientific Reports. 8 (1), 12658 (2018).
- Fitzgerald, K. A., et al. The use of collagen-based scaffolds to simulate prostate cancer bone metastases with potential for evaluating delivery of nanoparticulate gene therapeutics. Biomaterials. 66, 53-66 (2015).
- Sapudom, J., Pompe, T. Biomimetic tumor microenvironments based on collagen matrices. Biomaterials Science. 6 (8), 2009-2024 (2018).
- Curtin, C., et al. A physiologically relevant 3D collagen-based scaffold-neuroblastoma cell system exhibits chemosensitivity similar to orthotopic xenograft models. Acta Biomaterialia. 70, 84-97 (2018).
- Gavin, C., et al. Neuroblastoma invasion strategies are regulated by the extracellular matrix. Cancers. 13 (4), 1-23 (2021).
- Ridky, T. W., Chow, J. M., Wong, D. J., Khavari, P. A. Invasive three-dimensional organotypic neoplasia from multiple normal human epithelia. Nature Medicine. 16 (12), 1450-1456 (2010).
- Casal, e. J., Crane, J. S. . Biochemistry, Glycosaminoglycans. , (2019).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. J. The effect of pore size on cell adhesion in collagen-GAG scaffolds. Biomaterials. 26 (4), 433-441 (2005).
- O'Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. Influence of freezing rate on pore structure in freeze-dried collagen-GAG scaffolds. Biomaterials. 25 (6), 1077-1086 (2004).
- Haugh, M. G., Murphy, C. M., McKiernan, R. C., Altenbuchner, C., O'Brien, F. J. Crosslinking and mechanical properties significantly influence cell attachment, proliferation, and migration within collagen glycosaminoglycan scaffolds. Tissue Engineering. Part A. 17 (9-10), 1201-1208 (2011).
- Murphy, C. M., Haugh, M. G., O'Brien, F. J. The effect of mean pore size on cell attachment, proliferation and migration in collagen-glycosaminoglycan scaffolds for bone tissue engineering. Biomaterials. 31 (3), 461-466 (2010).
- Haugh, M. G., Jaasma, M. J., O'Brien, F. J. The effect of dehydrothermal treatment on the mechanical and structural properties of collagen-GAG scaffolds. Journal of Biomedical Materials Research - Part A. 89 (2), 363-369 (2009).
- Lowe, B., Hardy, J. G., Walsh, L. J. Optimizing nanohydroxyapatite nanocomposites for bone tissue engineering. ACS Omega. 5 (1), 1-9 (2020).
- Cunniffe, G. M., Dickson, G. R., Partap, S., Stanton, K. T., O'Brien, F. J. Development and characterisation of a collagen nano-hydroxyapatite composite scaffold for bone tissue engineering. Journal of Materials Science. Materials in Medicine. 21 (8), 2293-2298 (2010).
- DuBois, S. G., et al. Metastatic sites in stage IV and IVS neuroblastoma correlate with age, tumor biology, and survival. Journal of Pediatric Hematology/Oncology. 21 (3), 181-189 (1999).
- Ryan, A. J., Gleeson, J. P., Matsiko, A., Thompson, E. M., O'Brien, F. J. Effect of different hydroxyapatite incorporation methods on the structural and biological properties of porous collagen scaffolds for bone repair. Journal of Anatomy. 227 (6), 732-745 (2015).
- Tierney, C. M., et al. The effects of collagen concentration and crosslink density on the biological, structural and mechanical properties of collagen-GAG scaffolds for bone tissue engineering. Journal of the Mechanical Behavior of Biomedical Materials. 2 (2), 202-209 (2009).
- Cox, R. F., Jenkinson, A., Pohl, K., O'Brien, F. J., Morgan, M. P. Osteomimicry of mammary adenocarcinoma cells in vitro; increased expression of bone matrix proteins and proliferation within a 3D collagen environment. PLoS One. 7 (7), 41679 (2012).
- Nolan, J. C., et al. Preclinical models for neuroblastoma: advances and challenges. Cancer Letters. 474, 53-62 (2020).
- Sirivisoot, S., Pareta, R., Harrison, B. S. Protocol and cell responses in threedimensional conductive collagen gel scaffolds with conductive polymer nanofibres for tissue regeneration. Interface Focus. 4 (1), 20130050 (2014).
- Thevenot, P., Nair, A., Dey, J., Yang, J., Tang, L. Method to analyze three-dimensional cell distribution and infiltration in degradable scaffolds. Tissue Engineering. Part C-Methods. 14 (4), 319-331 (2008).
- do Amaral, R. J. F. C., et al. Functionalising collagen-based scaffolds with platelet-rich plasma for enhanced skin wound healing potential. Frontiers in Bioengineering and Biotechnology. 7, 371 (2019).
- Gkolfinopoulos, S., Tsapakidis, K., Papadimitriou, K., Papamichael, D., Kountourakis, P. Chromogranin A as a valid marker in oncology: Clinical application or false hopes. World Journal of Methodology. 7 (1), 9-15 (2017).
- O'Brien, F. J., et al. The effect of pore size on permeability and cell attachment in collagen scaffolds for tissue engineering. Technology and Health Care. 15 (1), 3-17 (2007).
Erratum
Formal Correction: Erratum: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds
Posted by JoVE Editors on 6/23/2022. Citeable Link.
An erratum was issued for: Three-dimensional In Vitro Biomimetic Model of Neuroblastoma using Collagen-based Scaffolds. The Authors section was updated.
The Authors section was updated from:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Fergal J. O’Brien3,4,5
Olga Piskareva1,2,3,5,6
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
5Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
6National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
to:
Ciara Gallagher*1,2,3
Catherine Murphy*1,2,3
Graeme Kelly4
Fergal J. O’Brien3,5,6
Olga Piskareva1,2,3,6,7
1Cancer Bioengineering Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
2School of Pharmacy and Biomolecular Sciences, RCSI University of Medicine and Health Sciences, Dublin, Ireland
3Tissue Engineering Research Group, Department of Anatomy and Regenerative Medicine, RCSI University of Medicine and Health Sciences, Dublin, Ireland
4Department of Chemistry, Royal College of Surgeons in Ireland (RCSI), 123 St. Stephen’s Green, Dublin 2, Ireland
5Trinity Centre for Bioengineering, Trinity College Dublin, Dublin, Ireland
6Advanced Materials and Bioengineering Research Centre (AMBER), RCSI and TCD, Dublin, Ireland
7National Children’s Research Centre, Our Lady's Children's Hospital Crumlin, Dublin, Ireland
* These authors contributed equally
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon