このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
DNAメチルトランスフェラーゼ阻害剤をスクリーニングするための連続蛍光ベースのエンドヌクレアーゼ共役DNAメチル化アッセイ(英語)
要約
DNAメチルトランスフェラーゼは、潜在的な抗がん剤標的です。ここでは、DNAメチルトランスフェラーゼ阻害について低分子を評価するためのプロトコルが提示されています。このアッセイでは、エンドヌクレアーゼを利用してDNAメチル化と蛍光生成を結合させ、酵素活性をリアルタイムでモニタリングすることができます。
要約
エピジェネティックな遺伝子調節の一形態であるDNAメチル化は、正常な細胞機能にとって重要です。細胞内では、DNAメチルトランスフェラーゼ(DNMT)と呼ばれるタンパク質がDNAメチル化パターンを確立し、維持します。正常なDNAメチル化パターンの変化は、がんの発生と進行に関連しており、DNMTは潜在的な抗がん剤標的となっています。したがって、これらの酵素の新規低分子阻害剤を同定し、特徴付けることは非常に重要です。この論文は、DNAメチルトランスフェラーゼ阻害剤のスクリーニングに使用できるプロトコルを提示します。連続共役動態アッセイにより、潜在的な低分子阻害剤の有無にかかわらず、DNAメチル化の初期速度を決定することができます。このアッセイでは、メチル感受性エンドヌクレアーゼGla Iを使用して、ヘミメチル化DNA基質のメチル化を蛍光生成に結合させます。
この連続アッセイにより、酵素活性をリアルタイムでモニタリングすることができます。マイクロタイタープレートで少量アッセイを行うことで、試薬のコストを削減できます。このアッセイを用いて、ヒトにおいて最も豊富なDNMTアイソザイムであるDNMT1の阻害剤について、小さな例のスクリーニングを行った。高度に置換されたアントラキノン天然物であるラッカイン酸Aは、DNMT1の強力なDNA競合阻害剤です。ここでは、アッセイプロトコルを説明するために、3つの潜在的な低分子阻害剤(アントラキノンまたは1〜3個の置換基を持つアントラキノン様分子)を2つの濃度で調べます。初期速度は、各分子の存在下で観察される活性パーセントを計算するために使用される。調べた3つの化合物のうちの1つは、DNMT1活性の濃度依存的な阻害を示し、それがDNMT1の潜在的な阻害剤であることを示しています。
概要
DNAメチル化は、遺伝子発現とクロマチン構造を調節する重要なエピジェネティックマークです。メチル化は主にCpGジヌクレオチド - シトシン、続いてグアノシンで起こる。メチル基はシトシンの5位に付加される。適切な細胞の発生と機能には、正しいDNAメチル化パターン、したがって適切な遺伝子発現が必要です。多くの疾患状態は、正常なメチル化パターンの変化と関連している1,2,3。たとえば、がんの開始と進行、およびDNAメチル化パターンの変化との間には関連性があります。通常、癌細胞はメチルシトシンの全体的なレベルが低く、これがゲノム不安定性の一因となります。同時に、ゲノム中に存在するメチルシトシンは、腫瘍抑制遺伝子のプロモーター領域に集中しており、これらの重要なタンパク質の遺伝子サイレンシングをもたらす。特に、エピジェネティックな変化は、腫瘍形成に関連するDNA変異とは異なり、動的で可逆的です。これにより、エピジェネティックな遺伝子制御に関与するタンパク質が興味深い薬物標的になりました2,4。
DNAメチルトランスフェラーゼ(DNMT)は、DNAメチル化パターンの生成と維持に関与するタンパク質です。3つの触媒活性アイソザイム、DNMT1、DNMT3a、およびDNMT3bがヒトに存在します。発生および分化の過程で、de novoメチルトランスフェラーゼであるDNMT3aおよびDNMT3bはメチル化パターンを確立します。両方の酵素は、触媒的に不活性なDNMT3Lタンパク質に結合して、増加した活性を示す複合体を形成することができます1,5。細胞分裂後、娘細胞にはヘミメチル化DNA(二重鎖の1本鎖のみにメチルシトシンを含むDNA)が含まれていますが、これは新しく合成されたDNAにメチル化マークがないためです。DNMT1の主な機能は、このヘミメチル化DNAをメチル化し、完全なメチル化パターンを再確立することです1,5。
DNMT活性とがんとの関連は十分に確立されています。転写機構または翻訳後機構のいずれかによるDNMT1の過剰発現は、いくつかの一般的な発癌経路の結果である6、7、8、9。低形態対立遺伝子を用いたDNMT1活性を低下させる遺伝的アプローチは、Apc(Min)マウスにおける腫瘍形成の減少をもたらす10。DNMT1をノックダウンするアンチセンスオリゴヌクレオチドは、細胞培養およびマウス腫瘍モデルにおける新生物を阻害する11、12。したがって、DNMT1活性を阻害することは、有望な癌治療アプローチのように思われます。しかし、DNMT3アイソザイムが果たす役割はそれほど単純ではありません。DNMT3a変異は、急性骨髄性白血病13および骨髄異形成症候群14に見られます。同定された変異の少なくとも1つは、酵素15のDNAメチル化活性を低下させることが示されている。しかし、DNMT3bは乳癌16および結腸直腸癌17において過剰発現している。さまざまなDNMTアイソザイムが発がんにおいて異なる役割を果たしているため、アイソザイム特異的阻害剤の同定が重要になります。これらの化合物は治療薬の開発に役立つだけでなく、アイソザイム特異的阻害剤は、がんの病因における各DNMTアイソザイムの役割を分析するための非常に貴重なツールにもなります。
いくつかのDNMT阻害剤が文献で報告されています。既知のDNMT阻害剤は、ヌクレオシドと非ヌクレオシドの2つのクラスに分けることができます。ヌクレオシド阻害剤は、典型的にはシチジン類似体である。これらの化合物はDNAに組み込まれ、DNMTを共有結合的にトラップします。 5-アザシチジンおよび5-アザ-2'-デオキシシチジンは、骨髄異形成症候群および急性骨髄性白血病の治療薬として承認されています4,18。これらの化合物の高い毒性、低いバイオアベイラビリティ、および化学的不安定性は問題を提示します。進行中の研究は、次世代のヌクレオシド阻害剤の有効性を調べることです。SGI−110は、5−アザ−2'−デオキシシチジンから誘導される、19、20の一例である。ヌクレオシド阻害剤はアイソザイム特異的ではなく、遭遇したDNMTアイソザイムを不活性化します。したがって、ヌクレオシド脱メチル化剤で処理すると、すべてのDNMTアイソザイムが枯渇します4,18。非ヌクレオシド阻害剤は、その阻害効果を発揮するためにDNAに組み込む必要はありません。代わりに、これらの分子はDNMTに直接結合し、アイソザイム特異的阻害の可能性をもたらします。SGI-1027 21、ヒドララジン22、プロカインアミド23、RG108および誘導体24、ならびに天然物、(−)−エピガロカテキン3−ガレート(EGCG)25およびラッカイン酸A26,27を含む、いくつかの非ヌクレオシド阻害剤がこれまでに発見されている。これまでに発見された非ヌクレオシド阻害剤のほとんどは、アイソザイム選択的ではないか、1つのDNMTアイソザイムに対して弱い選好を示します。さらに、これらの分子の効力は、特に細胞4,18において改善される必要がある。したがって、より強力なアイソザイム選択的DNMT阻害剤を発見または開発する必要があります。
DNMTの新しい低分子阻害剤を発見するためのハードルは、DNMT活性を調べるために伝統的に使用されている骨の折れるアッセイです28。アッセイは通常、複数のステップで不連続です。DNMTの酵素活性は、放射性S-アデノシルメチオニン(SAM)29,30,31,32,33,34を用いて日常的にアッセイされています。DNAメチル化のための非放射性アッセイも開発されています。例えば、消化産物を分離するためにメチル感受性制限エンドヌクレアーゼおよび電気泳動を利用するアッセイが記載されている35、36。これらのタイプの不連続な多段階アッセイは、創薬に容易に適していません。2000年代半ば以降、より高いスループットを有するいくつかのDNAメチル化アッセイが開発されてきた28。シンチレーション近接アッセイを用いて、DNMT1阻害剤37をスクリーニングした。メチル感受性制限エンドヌクレアーゼを利用する別のアッセイを、DNMT3a阻害剤のスクリーニングに使用した25、38。どちらのアッセイも従来のDNAメチル化アッセイよりも高いスループットを可能にしましたが、アッセイは複数のステップを必要とし、メチル化活性をリアルタイムで観察することはできません。最近では、メチル化反応の1つの生成物であるS-アデノシルホモシステイン(SAH)の形成を、NADPH酸化に関連する340 nmでの分光学的変化に結合する連続速度論アッセイが記載されている39。このアッセイでは、3つのカップリング酵素を利用して分光シグナルを生成します。
我々は、市販の単一のカップリング酵素を利用し、リアルタイムでデータを生成できる蛍光ベースのエンドヌクレアーゼ共役DNAメチル化アッセイを開発しました(図1)。3つのメチルシトシンを含むヘアピンオリゴヌクレオチドを基質として用いる。基質DNAには、5'末端に蛍光色素、3'末端に消光物質が含まれています。ヘミメチル化CpGサイトのメチル化は、エンドヌクレアーゼGla I - 完全メチル化GCGCの切断部位を生成する。生成物オリゴヌクレオチドのGla I切断は、クエンチャーから蛍光色素を放出し、リアルタイムで蛍光を生成します。このアッセイは、DNMTの任意のアイソフォームの活性を調べるために使用できます。しかし、このアイソザイムがヘミメチル化DNA1,5を優先的にメチル化するため、DNMT1ではより高い活性が観察されます。自己抑制性複製病巣標的配列(RFTS)ドメインがDNMT1から除去されると、さらに堅牢な活性が観察されます。N末端調節領域に見られるこのドメインは、触媒部位に結合し、DNA結合を妨げます。最初の~600個のアミノ酸を除去すると、完全長酵素よりも有意に活性の高い切断酵素が得られます(kcat/Kmの~640倍の増加)40。RFTS欠損DNMT1(アミノ酸621〜1616)と呼ばれるこの活性化型の酵素は、触媒力の増加により、阻害剤の同定を容易にします。この論文は、潜在的な低分子阻害剤をスクリーニングするためのアッセイでRFTS欠損DNMT1を利用するためのプロトコルを提示します。エンドヌクレアーゼ共役連続アッセイを用いて、数個の低分子の存在下および非存在下で初速度を決定する。各潜在的な阻害剤を2つの濃度で調べて、濃度依存的なDNMT1阻害を探します。小分子の存在下で観察された活性パーセントを、それぞれの場合において計算した。

図1:DNAメチル化アッセイ。 5'末端に蛍光色素、3'末端に消光物質を有するヘミメチル化ヘアピンDNAを基質として使用します。DNMT1は、 S-アデノシルメチオニンから非メチル化CpG部位へのメチル基の転移を触媒し、 S-アデノシルホモシステインおよび完全メチル化DNAを生成します。DNA産物には、完全にメチル化されたGCGC部位を切断するエンドヌクレアーゼGla Iの切断部位が含まれています。製品DNAが切断されると、3'クエンチャーから5'蛍光色素が放出され、蛍光が発生します。略語:Fl =フルオロフォア;Q =クエンチャー;DNMT1 = DNAメチルトランスフェラーゼ1;SAM = S-アデノシルメチオニン;SAH = S-アデノシルホモシステイン。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
1. スクリーニング用のアッセイ溶液を調製する
注:このアッセイで使用される基質の濃度は調整できます。RFTSが存在しないDNMT1の場合、ヘアピンDNA基質とSAMについて実験的に決定されたKm値は、それぞれ1〜2 nMおよび2 μMであり、それぞれ26,40です。
- 氷上のマイクロ遠心チューブで試験する各アッセイ条件を600 μL準備します。
注:必要なアッセイ溶液の総量は、実施する反復数によって異なります。各アッセイ条件は、このプロトコルについて3連で実行した。- ddH 2Oおよび5xメチル化バッファー(250 mM Tris pH 7.5、5 mM MgCl2、500 mM グルタミン酸カリウム、5 mMジチオスレイトール(DTT)、25%グリセロール)を各チューブに追加して、最終濃度1xバッファーを達成します。20 μM化合物を含むアッセイの場合は、472 μLのddH2Oと120 μL 5xバッファーを追加します。50 μM化合物を含むアッセイの場合は、470 μLのddH2Oと120 μLの5xバッファーを追加します。
- 3.15 μLの20 mg/mLウシ血清アルブミン(BSA)を各サンプルに加えます。
- 各サンプルに2 μLの3.15 μMヘアピンDNA基質と1.33 μLの4.75 mM SAMを追加します。
注:アッセイ溶液混合物中の基質の濃度は、所望の最終濃度の1.05倍です。 - 適切なサンプルに、最終濃度21 μM(10 mMの1.26 μL)または52.5 μM(10 mMの3.15 μL)の阻害剤を追加します。等量のジメチルスルホキシド(DMSO)を対照サンプルに加えます。
注:スクリーンで使用される阻害剤の濃度は変えることができます。最大最終DMSO濃度は≤5%(v / v)である必要があります。5%(v/v)までのDMSO濃度は、アッセイ26で観察される活性に影響を及ぼさない。
- ボルテックス(3,000 rpm)で3秒間溶液を混合します。卓上ミニ遠心分離機(1,200 × g)でサンプルを数秒間回転させ、溶液がチューブの底部に確実に収集されるようにします。
- 各アッセイ溶液95 μLを、黒い半面積96ウェルプレート内の6つの連続したウェルに分注します(図2)。
注:これらのスクリーニングアッセイは2つのバッチで実施されました。まず、20 μM阻害剤サンプル(合計4条件)を調べ、続いて50 μM阻害剤サンプル(合計4条件)を調べました。
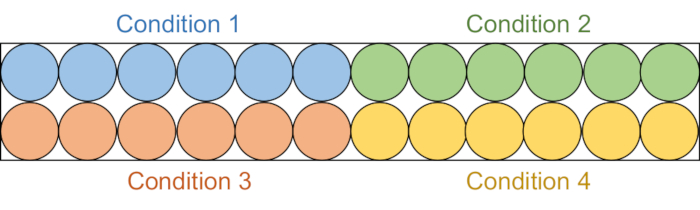
図2:アッセイプレートのセットアップ。 各アッセイ溶液は、黒色の96ウェルプレート内の6つのウェルに分注されます:DMSOコントロール(青)、化合物1(緑)、化合物2(赤)、および化合物3(黄色)。RFTSを欠くDNMT1とGla Iの両方が3つのウェルに追加されます。対照として、Gla Iのみが他の3つのウェルに添加されるであろう。略語: RFTS = レプリケーション フォーシー ターゲティング シーケンス;DNMT1 = DNAメチルトランスフェラーゼ1;DMSO = ジメチルスルホキシド。 この図の拡大版を表示するには、ここをクリックしてください。
2.スクリーニング用の酵素溶液を調製します
注:RFTS欠損DNMT1は 大腸菌 で発現し、均質に精製することができます。RFTS欠損DNMT1の発現および精製手順は、以前に記載されている41。必要な酵素の量は、実施するアッセイの数によって異なります。ここでは、各セットで4つの異なるアッセイが実行されます。各アッセイは3回で完了する。
- 氷上でマイクロ遠心チューブに各酵素溶液75 μLを調製します。
注:2つの酵素溶液が必要になります。1つの溶液にはDNMT1とGla Iが含まれます。もう一方にはGla Iのみが含まれます。- ddH 2Oおよび5xメチル化バッファー(250 mM Tris pH 7.5、5 mM MgCl2、500 mM グルタミン酸カリウム、5 mM DTT、25%グリセロール)を各チューブに追加して、最終濃度1xバッファーを達成します。Gla I酵素溶液には、15 μLの5xバッファーと58.8 μL ddH2Oを加えます。DNMT1+Gla I 酵素溶液に、15 μL の 5x バッファーと 58.2 μL ddH2O を加えます。
- 1.2 μLの10 U/μL Gla Iを各溶液に加え、最終濃度は0.16 U/μLです。
- DNMT1+Gla I溶液に、最終濃度40 nMの5 μM RFTS欠損DNMT1を0.6 μL加えます。
- 軽くたたいて溶液を混ぜます。卓上ミニ遠心分離機(1,200 × g)でサンプルを数秒間回転させ、溶液がチューブの底に確実に収集されるようにします。
- 各酵素溶液12 μLを円錐底96ウェルプレートの6ウェルに分注します(図3)。

図3:酵素プレートのセットアップ。 Gla I(灰色)およびDNMT1+Gla I(青色)溶液は、それぞれ96ウェルプレートの6つのウェルに分注されます。マルチチャンネルピペットを使用して、酵素をアッセイ溶液の列に同時に添加することができます。各アッセイ条件(6ウェル)について、3つのウェルはDNMT1 + Gla Iを受け取り、3つのウェルはGla Iのみを受け取ります。略称:DNMT1 = DNAメチルトランスフェラーゼ1。 この図の拡大版を表示するには、ここをクリックしてください。
3. アッセイを実行し、データを分析します。
- プレートリーダーを37°Cに予熱します。 アッセイ溶液が入った黒いプレートを挿入します。プレートを振って(425cpmで5秒間二重軌道)、励起波長485nm、発光波長528nmの蛍光を読み取ります。プレートを37°Cで5分間インキュベートします。
注:または、適切な波長カットオフを備えたフィルターを蛍光測定値に使用できます。 - アッセイプレートを取り外します。各ウェルに5 μLの酵素溶液を加え、上下にピペッティングして混合します。
注意: マルチチャンネルピペットを使用して、サンプルの列を同時に開始できるようにします。 - プレートをプレートリーダーに挿入します。53秒ごとに30分間蛍光を記録します。各読み取りの前に、プレートを振ってください(425 cpmで4秒間二重軌道)。
- 各条件について平均三連GlaIコントロールアッセイ。RFTS欠損DNMT1の存在下で得られた三重の微量から平均Gla I含有対照反応痕跡を差し引く。補正された反復の平均と標準誤差を求めます。
- 平均補正反応トレースをプロットし、初期線形部分を線に当てはめて初速度を決定します。
- 化合物の存在下で観察された速度をDMSO含有対照反応で観察された速度で割り、100を掛けることにより、活性率を決定します。
4. 追加のアッセイ制御 — 酵素の逐次添加
- 氷上でマイクロ遠心チューブに550 μLのアッセイ溶液を調製します。110 μLの5xメチル化バッファー、3.88 μLの3.15 μMヘアピンDNA基質、6.43 μLの47.5 mM SAM、3.06 μLの20 mg/mL BSA、および426.6 μLのddH2Oを追加します。
- ボルテックス(3,000 rpm)で3秒間溶液を混合します。卓上ミニ遠心分離機(1,200 × g)でサンプルを数秒間回転させ、溶液がチューブの底に確実に収集されるようにします。
- 90 μLのアッセイ溶液を、黒色半面積96ウェルプレート内の6つの連続したウェルに分注します。
- 氷上でDNMT1酵素溶液を調製します。4 μLの5xメチル化バッファー、0.76 μLの5 μM RFTS欠損DNMT1、および15.24 μLのddH2Oを1本のチューブに加えます。4 μLの5xメチル化バッファーと16 μLのddH2Oを別のチューブに加えます。
- 軽くたたいて溶液を混ぜます。卓上ミニ遠心分離機(1,200 × g)でサンプルを数秒間回転させ、溶液がチューブの底部に確実に収集されるようにします。
- 5 μLのDNMT1含有溶液を、黒色の半面積96ウェルプレートの3つのウェルに加えます。5 μLのバッファーコントロール溶液を他の3つのウェルに加えます。
- マイクロタイタープレートを37°Cに予熱したプレートリーダーに入れます。 プレートを振って(425cpmで3秒間二重軌道)、励起波長485nm、発光波長528nmの蛍光を読み取ります。シェイクを繰り返し、60秒ごとに30分間読みます。
- アッセイプレートをプレートリーダーに入れた状態で、氷上でGla I溶液を調製します。8 μLの5xメチル化バッファー、0.64 μLの10 U/μL Gla I、および31.4 μLのddH2Oをマイクロ遠心チューブに加えます。軽くたたいて溶液を混ぜます。卓上ミニ遠心分離機(1,200 × g)でサンプルを数秒間回転させ、溶液がチューブの底部に確実に収集されるようにします。
- 6 μLのGla I溶液を円錐底の96ウェルプレートの6ウェルに分注します。
- 最初の30分の読み取りに続いて、プレートリーダーから黒いプレートを取り外します。5 μLのGla I溶液を6つのウェルすべてに加え、上下にピペッティングして混合します。
注意: マルチチャンネルピペットを使用して、すべてのウェルを同時に開始できるようにします。 - 黒いプレートをプレートリーダーに戻します。35秒ごとに35分間蛍光を記録します。各読み取りの前にプレートを振ってください(425cpmで3秒間二重軌道)。
結果
アクティブ DNMT1 は、この分析の前提条件です。RFTS欠損DNMT1を 大腸菌 で発現させ、以前に公表された手順41に従って均質に精製した。精製酵素が活性であることを確認するために、不連続エンドヌクレアーゼ共役アッセイを使用してDNAメチル化活性を調べた36。このアッセイでは、Sau3A1切断部位に配置された単一のヘミメチル化CpGを有する32塩基対の?...
ディスカッション
DNAメチルトランスフェラーゼの阻害剤を同定し、特徴付けるには、酵素の活性を測定する必要があります。DNAメチルトランスフェラーゼ活性を調べるためのいくつかの方法が存在します。活動は一般的に放射能を使用して監視されます。SAMの標識メチル基の転移は、29、30、31、32、33、
開示事項
著者は開示する利益相反を持っていません。
謝辞
著者らは、この研究を支援してくれたバックネル大学と化学科に感謝する。
資料
| Name | Company | Catalog Number | Comments |
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
参考文献
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3'-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what's next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2'-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved