É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaio de metilação de DNA acoplado à endonuclease à base de fluorescência contínua para triagem de inibidores da DNA metiltransferase
Neste Artigo
Resumo
As metiltransferases de DNA são potenciais alvos de drogas contra o câncer. Aqui, um protocolo é apresentado para avaliar pequenas moléculas para inibição da DNA metiltransferase. Este ensaio utiliza uma endonuclease para acoplar a metilação do DNA à geração de fluorescência e permite que a atividade enzimática seja monitorada em tempo real.
Resumo
A metilação do DNA, uma forma de regulação genética epigenética, é importante para a função celular normal. Nas células, as proteínas chamadas DNA metiltransferases (DNMTs) estabelecem e mantêm o padrão de metilação do DNA. Mudanças no padrão normal de metilação do DNA estão ligadas ao desenvolvimento e progressão do câncer, tornando os DNMTs potenciais alvos de medicamentos contra o câncer. Assim, identificar e caracterizar novos inibidores de pequenas moléculas dessas enzimas é de grande importância. Este trabalho apresenta um protocolo que pode ser utilizado para o rastreamento de inibidores da DNA metiltransferase. O ensaio de cinética acoplada contínua permite que as velocidades iniciais de metilação do DNA sejam determinadas na presença e ausência de potenciais inibidores de moléculas pequenas. O ensaio usa a endonuclease sensível à metila Gla I para acoplar a metilação de um substrato de DNA hemimetilado à geração de fluorescência.
Este ensaio contínuo permite que a atividade enzimática seja monitorada em tempo real. A realização do ensaio em pequenos volumes em placas de microtitulação reduz o custo dos reagentes. Usando este ensaio, uma pequena tela de exemplo foi conduzida para inibidores de DNMT1, a isoenzima DNMT mais abundante em humanos. O produto natural de antraquinona altamente substituído, o ácido lacáico A, é um potente inibidor competitivo de DNA da DNMT1. Aqui, examinamos três potenciais inibidores de moléculas pequenas - antraquinonas ou moléculas semelhantes à antraquinona com um a três substituintes - em duas concentrações para descrever o protocolo de ensaio. As velocidades iniciais são usadas para calcular a porcentagem de atividade observada na presença de cada molécula. Um dos três compostos examinados exibe inibição dependente da concentração da atividade DNMT1, indicando que é um inibidor potencial da DNMT1.
Introdução
A metilação do DNA é uma importante marca epigenética que regula a expressão gênica e a estrutura da cromatina. A metilação ocorre predominantemente nos dinucleotídeos CpG — citosina seguida de guanosina; o grupo metilo é adicionado à posição 5 da citosina. Padrões corretos de metilação do DNA e, portanto, a expressão gênica adequada, são necessários para o desenvolvimento e a função celular apropriados. Muitos estados patológicos têm sido associados a alterações no padrão normal de metilação 1,2,3. Por exemplo, existe uma ligação entre o início e a progressão do câncer e alterações no padrão de metilação do DNA. Normalmente, as células cancerosas exibem níveis gerais mais baixos de metilcitosina, o que contribui para a instabilidade do genoma. Ao mesmo tempo, a metilcitosina que está presente no genoma está concentrada nas regiões promotoras dos genes supressores de tumor, o que leva ao silenciamento genético dessas proteínas importantes. Notavelmente, as alterações epigenéticas são dinâmicas e reversíveis, ao contrário das mutações no DNA associadas à tumorigênese. Isso tornou as proteínas envolvidas na regulação epigenética gênica alvos de drogas interessantes 2,4.
As metiltransferases de DNA (DNMTs) são as proteínas responsáveis por gerar e manter os padrões de metilação do DNA. Três isozimas cataliticamente ativas, DNMT1, DNMT3a e DNMT3b, existem em humanos. Durante o desenvolvimento e diferenciação, as metiltransferases de novo, DNMT3a e DNMT3b, estabelecem padrões de metilação. Ambas as enzimas podem se ligar à proteína DNMT3L cataliticamente inativa para formar complexos que exibem atividade aumentada 1,5. Após a divisão celular, as células filhas contêm DNA hemimetilado - DNA contendo metilcitosina em apenas uma fita do duplex - porque o DNA recém-sintetizado é desprovido de marcas de metilação. A principal função do DNMT1 é metilar esse DNA hemimetilado, restabelecendo assim o padrão completo de metilação 1,5.
As ligações entre a atividade da DNMT e o câncer estão bem estabelecidas. A superexpressão de DNMT1, seja por mecanismos transcricionais ou pós-traducionais, é consequência de várias vias oncogênicas comuns 6,7,8,9. Abordagens genéticas para menor atividade da DNMT1 usando alelos hipomórficos resultam em diminuição da formação tumoral em camundongos Apc(Min)10. Os oligonucleotídeos antisense que derrubam a DNMT1 inibem a neoplasia em modelos de cultura celular e tumores de camundongos11,12. Assim, inibir a atividade da DNMT1 parece ser uma abordagem promissora de terapia contra o câncer. No entanto, os papéis que as isozimas DNMT3 desempenham não são tão simples. As mutações DNMT3a são encontradas na leucemia mieloide aguda13 e na síndrome mielodisplásica14. Pelo menos uma das mutações identificadas demonstrou diminuir a atividade de metilação do DNA da enzima15. No entanto, o DNMT3b é superexpresso no câncer de mama16 e no câncer colorretal17. Com as várias isozimas DNMT desempenhando papéis diferentes na carcinogênese, a identificação de inibidores específicos de isoenzimas será crítica. Não só esses compostos serão úteis para o desenvolvimento de terapias, mas os inibidores específicos da isoenzima também seriam uma ferramenta inestimável para dissecar o papel de cada isoenzima DNMT na etiologia do câncer.
Vários inibidores de DNMT têm sido relatados na literatura. Os inibidores conhecidos da DNMT podem ser divididos em duas classes: nucleosídeo e não nucleosídeo. Os inibidores de nucleosídeos são tipicamente análogos da citidina. Esses compostos são incorporados ao DNA e retêm covalentemente DNMTs. 5-azacitidina e 5-aza-2'-desoxicitidina foram aprovados para o tratamento da síndrome mielodisplásica e leucemia mieloide aguda 4,18. A alta toxicidade, a baixa biodisponibilidade e a instabilidade química desses compostos apresentam problemas. O trabalho em andamento está examinando a eficácia da próxima geração de inibidores de nucleosídeos; SGI-110, derivado de 5-aza-2'-desoxicitidina, é um exemplo19,20. Os inibidores de nucleosídeos não são específicos da isoenzima e inativarão qualquer isoenzima DNMT encontrada. Portanto, o tratamento com um agente nucleosídeo-desmetilante resulta na depleção de todas as isozimas DNMT 4,18. Os inibidores não nucleosídeos não precisam ser incorporados ao DNA para exercer seus efeitos inibitórios. Em vez disso, essas moléculas se ligam diretamente aos DNMTs, introduzindo a possibilidade de inibição específica da isozima. Vários inibidores não nucleosídeos foram descobertos até o momento, incluindo SGI-102721, hidralazina 22, procainamida 23, RG108 e derivados 24, e produtos naturais, (−)-epigalocatequina 3-galato (EGCG)25 e ácido lacáico A 26,27. A maioria dos inibidores não-nucleosídeos descobertos até o momento não são seletivos de isoenzima ou exibem preferências fracas para uma isoenzima DNMT. Além disso, a potência dessas moléculas precisa ser melhorada, principalmente nas células 4,18. Assim, há uma necessidade de descobrir ou desenvolver inibidores de DNMT mais potentes e seletivos de isozima.
Um obstáculo para a descoberta de novos inibidores de pequenas moléculas de DNMTs são os ensaios laboriosos tradicionalmente usados para examinar a atividade da DNMT28. Os ensaios geralmente são descontínuos com várias etapas. A atividade enzimática dos DNMTs ainda é rotineiramente ensaiada utilizando S-adenosilmetionina radioativa (SAM)29,30,31,32,33,34. Ensaios não radioativos para metilação do DNA também foram desenvolvidos. Por exemplo, ensaios utilizando endonucleases de restrição sensíveis à metila e eletroforese para separar os produtos da digestão têm sido descritos35,36. Esses tipos de ensaios descontínuos e em várias etapas não são facilmente passíveis de descoberta de medicamentos. Desde meados dos anos 2000, vários ensaios de metilação de DNA com maior rendimento foram desenvolvidos28. Um ensaio de proximidade de cintilação foi utilizado para triagem de inibidores de DNMT137. Outro ensaio utilizando endonuclease de restrição sensível à metila foi utilizado para rastrear inibidores de DNMT3a25,38. Embora ambos os ensaios tenham permitido um rendimento mais elevado do que os ensaios tradicionais de metilação do ADN, os ensaios requerem várias etapas e não permitem a observação da atividade de metilação em tempo real. Mais recentemente, um ensaio de cinética contínua tem sido descrito que acopla a formação de S-adenosilhomocisteína (HAS), um produto da reação de metilação, à alteração espectroscópica a 340 nm associada à oxidação do NADPH39. Este ensaio utiliza três enzimas de acoplamento para gerar um sinal espectroscópico.
Desenvolvemos um ensaio de metilação de DNA acoplado à endonuclease baseado em fluorescência que utiliza uma única enzima de acoplamento comercialmente disponível e pode gerar dados em tempo real (Figura 1). Um oligonucleotídeo hairpin contendo três metilcitosinas é usado como substrato. O DNA do substrato contém um fluoróforo na extremidade 5' e um quencher na extremidade 3'. A metilação do sítio CpG hemimetilado gera o sítio de clivagem para a endonuclease Gla I — GCGC totalmente metilado. A clivagem Gla I do oligonucleotídeo do produto libera o fluoróforo do quencher e gera fluorescência em tempo real. O ensaio pode ser usado para examinar a atividade de qualquer isoforma de DNMT; no entanto, observa-se maior atividade com DNMT1, pois esta isoenzima preferencialmente metila o DNA hemimetilado 1,5. Atividade ainda mais robusta é observada se o domínio RFTS (Replication Foci Targeting Sequence) autoinibitório for removido da DNMT1. Este domínio, encontrado na região reguladora N-terminal, liga-se ao sítio catalítico e impede a ligação do DNA. A remoção dos primeiros ~600 aminoácidos resulta em uma enzima truncada que é significativamente mais ativa do que a enzima de comprimento total (~640 vezes maior em kcat/Km)40. Esta forma ativada da enzima, referida como DNMT1 sem RFTS (aminoácidos 621-1616), permite a identificação mais fácil de inibidores devido ao seu aumento do poder catalítico. Este artigo apresenta um protocolo para utilizar DNMT1 sem RFTS em ensaios para triagem de potenciais inibidores de moléculas pequenas. Usando o ensaio contínuo acoplado à endonuclease, a velocidade inicial é determinada na presença e ausência de algumas moléculas pequenas. Cada inibidor potencial é examinado em duas concentrações para procurar inibição DNMT1 dependente da concentração. A porcentagem de atividade observada na presença das pequenas moléculas foi calculada em cada caso.

Figura 1: Ensaio de metilação do DNA. Um DNA de grampo de cabelo hemimetilado com um fluoróforo na extremidade 5' e um quencher na extremidade 3' é usado como substrato. DNMT1 catalisa a transferência do grupo metilo da S-adenosilmetionina para o sítio CpG não metilado, gerando S-adenosilhomocisteína e DNA totalmente metilado. O produto de DNA contém o local de clivagem para a endonuclease Gla I, que cliva os sítios GCGC totalmente metilados. A clivagem do DNA do produto libera o fluoróforo de 5' do quencher de 3', gerando fluorescência. Abreviaturas: Fl = fluoróforo; Q = quencher; DNMT1 = DNA metiltransferase 1; SAM = S-adenosilmetionina; HAS = S-adenosilhomocisteína. Por favor, clique aqui para ver uma versão maior desta figura.
Protocolo
1. Prepare soluções de ensaio para a tela
NOTA: As concentrações de substratos utilizadas neste ensaio podem ser adaptadas. Para DNMT1 sem RFTS, os valores de Km determinados experimentalmente para o substrato de DNA do hairpin e SAM são 1–2 nM e 2 μM, respectivamente26,40.
- Preparar 600 μL de cada condição de ensaio que está sendo testada em tubos de microcentrífuga sobre gelo.
NOTA: O volume total de solução de ensaio necessário depende do número de replicações que estão sendo conduzidas. Cada condição de ensaio foi executada em triplicado para este protocolo.- Adicione ddH 2O e tampão de metilação 5x (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM glutamato de potássio, 5 mM ditiotreitol (DTT), 25% de glicerol) a cada tubo para atingir uma concentração final de 1x tampão. Para ensaios contendo composto de 20 μM, adicionar 472 μL de ddH2O e 120 μL 5x tampão. Para ensaios contendo 50 μM de composto, adicionar 470 μL de ddH2O e 120 μL de tampão 5x.
- Adicionar 3,15 μL de 20 mg/mL de albumina sérica bovina (ASC) a cada amostra.
- Adicionar 2 μL de substrato de DNA hairpin de 3,15 μM e 1,33 μL de SAM de 4,75 mM a cada amostra.
NOTA: A concentração de substratos na mistura de solução de ensaio é de 1,05x as concentrações finais desejadas. - Adicionar inibidor a uma concentração final de 21 μM (1,26 μL de 10 mM) ou 52,5 μM (3,15 μL de 10 mM) às amostras adequadas. Adicionar uma quantidade equivalente de dimetilsulfóxido (DMSO) a uma amostra de controlo.
NOTA: A concentração do inibidor utilizado no ecrã pode ser variada. A concentração final máxima de DMSO deve ser de ≤5% (v/v). Concentrações de DMSO até 5% (v/v) não têm efeito sobre a atividade observada no ensaio26.
- Misture as soluções por vórtice (3.000 rpm) por 3 s. Gire as amostras por alguns segundos em uma minicentrífuga de mesa (1.200 × g) para garantir que a solução seja coletada no fundo do tubo.
- Aliquota 95 μL de cada solução de ensaio em 6 poços consecutivos em uma placa preta de 96 poços de meia área (Figura 2).
NOTA: Estes ensaios de triagem foram realizados em dois lotes. Primeiro, 20 amostras de inibidores de μM (4 condições totais) foram examinadas, seguidas pelas amostras de inibidores de 50 μM (4 condições totais).
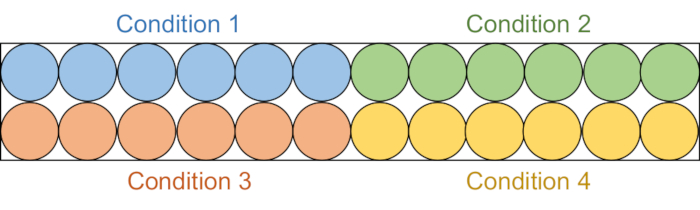
Figura 2: Configuração da placa de ensaio. Cada solução de ensaio é aliquotada em seis poços na placa preta de 96 poços: controle DMSO (azul), composto 1 (verde), composto 2 (vermelho) e composto 3 (amarelo). Tanto o DNMT1 com falta de RFTS quanto o Gla I serão adicionados a três poços. Como controle, só Gla I será adicionada aos outros três poços. Abreviaturas: RFTS = Replication Foci Targeting Sequence; DNMT1 = DNA metiltransferase 1; DMSO = dimetilsulfóxido. Por favor, clique aqui para ver uma versão maior desta figura.
2. Preparar soluções enzimáticas para triagem
NOTA: DNMT1 sem RFTS pode ser expresso em E. coli e purificado até homogeneidade. Procedimentos de expressão e purificação para DNMT1 sem RFTS foram descritos anteriormente41. O volume de enzima necessário depende do número de ensaios que estão sendo conduzidos. Aqui, quatro ensaios diferentes estão sendo realizados em cada conjunto; cada ensaio é completado em triplicata.
- Preparar 75 μL de cada solução enzimática em tubos de microcentrífuga sobre gelo.
NOTA: Serão necessárias duas soluções enzimáticas. Uma solução conterá DNMT1 e Gla I; o outro conterá apenas Gla I.- Adicionar ddH 2O e tampão de metilação 5x (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM glutamato de potássio, 5 mM DTT, 25% de glicerol) a cada tubo para atingir uma concentração final de 1x tampão. Para a solução da enzima Gla I, adicionar 15 μL de tampão 5x e 58,8 μL ddH2O. Para a solução enzimática DNMT1+Gla I, adicionar 15 μL de tampão 5x e 58,2 μL ddH2O.
- Adicionar 1,2 μL de 10 U/μL Gla I a cada solução para uma concentração final de 0,16 U/μL.
- Adicionar 0,6 μL de DNMT1 com falta de RFTS de 5 μM para uma concentração final de 40 nM à solução DNMT1+Gla I.
- Bata suavemente para misturar as soluções. Gire as amostras por alguns segundos em uma minicentrífuga de mesa (1.200 × g) para garantir que a solução seja coletada no fundo do tubo.
- Aliquota 12 μL de cada solução enzimática em 6 poços em uma placa de 96 poços de fundo cônico (Figura 3).

Figura 3: Configuração da placa enzimática. As soluções Gla I (cinza) e DNMT1+Gla I (azul) são alíquotas em seis poços na placa de 96 poços. Usando uma pipeta multicanal, a enzima pode ser adicionada a uma linha de soluções de ensaio simultaneamente. Para cada condição de ensaio (seis poços), três poços receberão DNMT1+Gla I e três poços receberão apenas Gla I. Abreviatura: DNMT1 = DNA metiltransferase 1. Por favor, clique aqui para ver uma versão maior desta figura.
3. Execute o ensaio e analise os dados.
- Pré-aqueça o leitor de placas a 37 °C. Insira a placa preta que contém as soluções de ensaio. Agitar a placa (orbital duplo a 425 cpm durante 5 s) e ler a fluorescência com um comprimento de onda de excitação de 485 nm e um comprimento de onda de emissão de 528 nm. Incubar a placa a 37 °C durante 5 min.
NOTA: Alternativamente, filtros com cortes de comprimento de onda adequados podem ser usados para as leituras de fluorescência. - Remova a placa de ensaio. Adicionar 5 μL de solução enzimática a cada poço e misturar pipetando para cima e para baixo.
Observação : use pipes multicanal para que uma linha de amostras pode ser iniciada simultaneamente. - Insira a placa no leitor de placas. Grave a fluorescência a cada 53 s por 30 min. Agite a placa (orbital duplo a 425 cpm durante 4 s) antes de cada leitura.
- Ensaios de controle de Gla I triplicados médios para cada condição. Subtraia o traço médio da reação de controle contendo Gla I dos traços triplicados obtidos na presença de DNMT1 sem RFTS. Determine o erro médio e padrão da média para as replicações corrigidas.
- Plote o traço médio de reação corrigida e ajuste a porção linear inicial a uma linha para determinar a velocidade inicial.
- Determine a porcentagem de atividade dividindo a velocidade observada na presença de um composto pela velocidade observada na reação de controle contendo DMSO e multiplicando por 100.
4. Controlo adicional do ensaio — adição sequencial de enzimas
- Preparar 550 μL de solução de ensaio num tubo de microcentrífuga sobre gelo. Adicionar 110 μL de tampão de metilação 5x, 3,88 μL de substrato de DNA hairpin de 3,15 μM, 6,43 μL de SAM de 47,5 mM, 3,06 μL de BSA de 20 mg/mL e 426,6 μL de ddH2O.
- Misture a solução por vórtice (3.000 rpm) por 3 s. Gire as amostras por alguns segundos em uma minicentrífuga de mesa (1.200 × g) para garantir que a solução seja coletada no fundo do tubo.
- Aliquota 90 μL da solução de ensaio em 6 poços consecutivos em uma placa preta de 96 poços de meia área.
- Preparar as soluções enzimáticas DNMT1 no gelo. Adicione 4 μL de 5x tampão de metilação, 0,76 μL de 5 μM de DNMT1 sem RFTS e 15,24 μL de ddH2O a um tubo. Adicionar 4 μL de tampão de metilação 5x e 16 μL de ddH2O a outro tubo.
- Bata suavemente para misturar as soluções. Gire as amostras por alguns segundos em uma minicentrífuga de mesa (1.200 × g) para garantir que a solução seja coletada no fundo do tubo.
- Adicionar 5 μL da solução contendo DNMT1 a três poços na placa preta de 96 poços de meia área. Adicionar 5 μL da solução de controle tampão aos outros três poços.
- Colocar a placa de microtitulação no leitor de placas pré-aquecida a 37 °C. Agitar a placa (orbital duplo a 425 cpm durante 3 s) e ler a fluorescência com um comprimento de onda de excitação de 485 nm e um comprimento de onda de emissão de 528 nm. Repita o shake e leia a cada 60 s por 30 min.
- Enquanto a placa de ensaio estiver no leitor de placas, prepare a solução Gla I no gelo. Adicionar 8 μL de tampão de metilação 5x, 0,64 μL de 10 U/μL Gla I e 31,4 μL de ddH2O a um tubo de microcentrífuga. Bata suavemente para misturar a solução. Gire a amostra por alguns segundos em uma minicentrífuga de mesa (1.200 × g) para garantir que a solução seja coletada no fundo do tubo.
- Aliquota 6 μL da solução de Gla I em 6 poços em uma placa de fundo cônico de 96 poços.
- Após a leitura inicial de 30 minutos, remova a placa preta do leitor de placas. Adicione 5 μL de solução Gla I a todos os 6 poços e misture pipetando para cima e para baixo.
NOTA: Use um pipete multicanal para que todos os poços possam ser iniciados simultaneamente. - Coloque a placa preta de volta no leitor de placas. Grave a fluorescência a cada 35 s por 35 min. Agite a placa (orbital duplo a 425 cpm durante 3 s) antes de cada leitura.
Resultados
DNMT1 ativo é um pré-requisito para esta análise. A DNMT1 sem RFTS foi expressa em E.coli e purificada até a homogeneidade seguindo procedimentos previamente publicados41. Para garantir que a enzima purificada estivesse ativa, um ensaio descontínuo acoplado à endonuclease foi usado para examinar a atividade de metilação do DNA36. Este ensaio utiliza um DNA duplex de 32 pares de bases com um único CpG hemimetilado posicionado em um local de clivagem Sau3A1....
Discussão
Para identificar e caracterizar inibidores de DNA metiltransferases, a atividade da enzima deve ser medida. Existem vários métodos para examinar a atividade da DNA metiltransferase. A atividade é comumente monitorada usando radioatividade; a transferência do grupo metilo marcado da SAM pode ser quantificada 29,30,31,32,33,34
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores agradecem à Universidade de Bucknell e ao Departamento de Química por seu apoio a este trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
Referências
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3'-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what's next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2'-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados