Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Essai de méthylation de l’ADN couplé à l’endonucléase en continu basé sur la fluorescence pour dépister les inhibiteurs de l’ADN méthyltransférase
Dans cet article
Résumé
Les ADN méthyltransférases sont des cibles potentielles de médicaments anticancéreux. Ici, un protocole est présenté pour évaluer les petites molécules pour l’inhibition de l’ADN méthyltransférase. Ce test utilise une endonucléase pour coupler la méthylation de l’ADN à la génération de fluorescence et permet de surveiller l’activité enzymatique en temps réel.
Résumé
La méthylation de l’ADN, une forme de régulation épigénétique des gènes, est importante pour la fonction cellulaire normale. Dans les cellules, des protéines appelées ADN méthyltransférases (DNMT) établissent et maintiennent le schéma de méthylation de l’ADN. Les changements dans le schéma normal de méthylation de l’ADN sont liés au développement et à la progression du cancer, ce qui fait des DNMT des cibles potentielles pour les médicaments anticancéreux. Ainsi, l’identification et la caractérisation de nouvelles petites molécules inhibitrices de ces enzymes sont d’une grande importance. Cet article présente un protocole qui peut être utilisé pour dépister les inhibiteurs de l’ADN méthyltransférase. Le test cinétique couplé continu permet de déterminer les vitesses initiales de méthylation de l’ADN en présence et en l’absence d’inhibiteurs potentiels de petites molécules. Le test utilise l’endonucléase méthyl-sensible Gla I pour coupler la méthylation d’un substrat d’ADN hémiméthylé à la génération de fluorescence.
Ce test continu permet de surveiller l’activité enzymatique en temps réel. La réalisation du test en petits volumes dans des plaques de microtitrage réduit le coût des réactifs. À l’aide de ce test, un petit exemple de dépistage a été effectué pour les inhibiteurs de DNMT1, l’isozyme DNMT le plus abondant chez l’homme. Le produit naturel anthraquinone hautement substitué, l’acide laccaïque A, est un puissant inhibiteur de DNMT1 compétitif pour l’ADN. Ici, nous examinons trois inhibiteurs potentiels de petites molécules – anthraquinones ou molécules semblables à l’anthraquinone avec un à trois substituants – à deux concentrations pour décrire le protocole de dosage. Les vitesses initiales sont utilisées pour calculer le pourcentage d’activité observé en présence de chaque molécule. L’un des trois composés examinés présente une inhibition dépendante de la concentration de l’activité DNMT1, ce qui indique qu’il s’agit d’un inhibiteur potentiel de DNMT1.
Introduction
La méthylation de l’ADN est une marque épigénétique importante qui régule l’expression des gènes et la structure de la chromatine. La méthylation se produit principalement dans les dinucléotides CpG - cytosine suivie de guanosine; Le groupe méthyle est ajouté à la position 5 de la cytosine. Des schémas de méthylation corrects de l’ADN, et donc une expression génique appropriée, sont nécessaires pour un développement et une fonction cellulaires appropriés. De nombreux états pathologiques ont été associés à des modifications du schéma normal de méthylation 1,2,3. Par exemple, il existe un lien entre l’initiation et la progression du cancer et les altérations du schéma de méthylation de l’ADN. En règle générale, les cellules cancéreuses présentent des niveaux globaux plus faibles de méthylcytosine, ce qui contribue à l’instabilité du génome. Dans le même temps, la méthylcytosine présente dans le génome est concentrée dans les régions promotrices des gènes suppresseurs de tumeurs, ce qui conduit au silençage génique de ces protéines importantes. Notamment, les changements épigénétiques sont dynamiques et réversibles, contrairement aux mutations de l’ADN associées à la tumorigenèse. Cela a rendu les protéines impliquées dans la régulation des gènes épigénétiques intéressantescibles médicamenteuses 2,4.
Les ADN méthyltransférases (DNMT) sont les protéines responsables de la génération et du maintien des schémas de méthylation de l’ADN. Trois isoenzymes catalytiquement actifs, DNMT1, DNMT3a et DNMT3b, existent chez l’homme. Au cours du développement et de la différenciation, les méthyltransférases de novo, DNMT3a et DNMT3b, établissent des schémas de méthylation. Les deux enzymes peuvent lier la protéine DNMT3L catalytiquement inactive pour former des complexes qui présentent une activité accrue 1,5. Après la division cellulaire, les cellules filles contiennent de l’ADN hémiméthylé – de l’ADN contenant de la méthylcytosine dans un seul brin du duplex – parce que l’ADN nouvellement synthétisé est dépourvu de marques de méthylation. La fonction principale de DNMT1 est de méthyler cet ADN hémiméthylé, rétablissant ainsi le schéma de méthylation complet 1,5.
Les liens entre l’activité DNMT et le cancer sont bien établis. La surexpression de DNMT1, que ce soit par des mécanismes transcriptionnels ou post-traductionnels, est une conséquence de plusieurs voies oncogéniques courantes 6,7,8,9. Les approches génétiques visant à réduire l’activité de DNMT1 à l’aide d’allèles hypomorphiques entraînent une diminution de la formation tumorale chez les souris Apc(Min)10. Les oligonucléotides antisens qui neutralisent DNMT1 inhibent la néoplasie dans la culture cellulaire et les modèles tumorauxde souris 11,12. Ainsi, l’inhibition de l’activité DNMT1 semble être une approche prometteuse de traitement du cancer. Cependant, les rôles que jouent les isoenzymes DNMT3 ne sont pas si simples. Les mutations DNMT3a sont retrouvées dans la leucémie myéloïde aiguë13 et le syndrome myélodysplasique14. Il a été démontré qu’au moins une des mutations identifiées diminue l’activité de méthylation de l’ADN de l’enzyme15. Cependant, DNMT3b est surexprimé dans le cancer du sein16 et le cancer colorectal17. Les différents isoenzymes DNMT jouant différents rôles dans la cancérogenèse, l’identification des inhibiteurs spécifiques de l’isozyme sera essentielle. Non seulement ces composés seront utiles pour le développement de produits thérapeutiques, mais les inhibiteurs spécifiques à l’isozyme seraient également un outil précieux pour disséquer le rôle de chaque isozyme DNMT dans l’étiologie du cancer.
Plusieurs inhibiteurs de DNMT ont été rapportés dans la littérature. Les inhibiteurs connus de la DNMT peuvent être divisés en deux classes : nucléosidiques et non nucléosidiques. Les inhibiteurs nucléosidiques sont généralement des analogues de la cytidine. Ces composés sont incorporés dans l’ADN et piègent par covalence les DNMT. La 5-azacytidine et la 5-aza-2'-désoxycytidine ont été approuvées pour le traitement du syndrome myélodysplasique et de la leucémie myéloïde aiguë 4,18. La toxicité élevée, la faible biodisponibilité et l’instabilité chimique de ces composés posent des problèmes. Les travaux en cours examinent l’efficacité de la prochaine génération d’inhibiteurs nucléosidiques; SGI-110, dérivé de la 5-aza-2'-désoxycytidine, en est un exemple19,20. Les inhibiteurs nucléosidiques ne sont pas spécifiques à l’isozyme et inactiveront tout isozyme DNMT rencontré. Par conséquent, le traitement avec un agent déméthylant des nucléosides entraîne l’épuisement de tous les isoenzymes DNMT 4,18. Les inhibiteurs non nucléosidiques n’ont pas besoin d’être incorporés dans l’ADN pour exercer leurs effets inhibiteurs. Au lieu de cela, ces molécules se lient directement aux DNMT, introduisant la possibilité d’une inhibition spécifique de l’isozyme. Plusieurs inhibiteurs non nucléosidiques ont été découverts à ce jour, notamment SGI-1027 21, hydralazine22, procaïnamide 23, RG108 et dérivés 24, et des produits naturels, (−)-épigallocatéchine 3-gallate (EGCG)25 et acide laccaïque A26,27. La plupart des inhibiteurs non nucléosidiques découverts à ce jour ne sont pas isozymes-sélectifs ou présentent de faibles préférences pour un isozyme DNMT. De plus, la puissance de ces molécules doit être améliorée, en particulier dans les cellules 4,18. Il est donc nécessaire de découvrir ou de développer des inhibiteurs de DNMT isozymes-sélectifs plus puissants.
Un obstacle à la découverte de nouvelles petites molécules inhibitrices de DNMT est les tests laborieux traditionnellement utilisés pour examiner l’activité DNMT28. Les tests sont généralement discontinus avec plusieurs étapes. L’activité enzymatique des DNMT est encore systématiquement dosée à l’aide de S-adénosylméthionine (SAM) radioactive29,30,31,32,33,34. Des tests non radioactifs pour la méthylation de l’ADN ont également été développés. Par exemple, des essais utilisant des endonucléases de restriction méthylsensibles et l’électrophorèse pour séparer les produits de digestion ont été décrits35,36. Ces types de tests discontinus en plusieurs étapes ne se prêtent pas facilement à la découverte de médicaments. Depuis le milieu des années 2000, plusieurs tests de méthylation de l’ADN avec un débit plus élevé ont été développés28. Un test de proximité par scintillation a été utilisé pour dépister les inhibiteurs de DNMT137. Un autre essai utilisant une endonucléase de restriction méthylsensible a été utilisé pour dépister les inhibiteurs de DNMT3a25,38. Bien que les deux essais aient permis un débit plus élevé que les tests traditionnels de méthylation de l’ADN, les essais nécessitent plusieurs étapes et ne permettent pas d’observer l’activité de méthylation en temps réel. Plus récemment, un essai cinétique continu a été décrit qui couple la formation de S-adénosylhomocystéine (SAH), un produit de la réaction de méthylation, au changement spectroscopique à 340 nm associé à l’oxydation du NADPH39. Ce test utilise trois enzymes de couplage pour générer un signal spectroscopique.
Nous avons développé un test de méthylation de l’ADN couplé à l’endonucléase basé sur la fluorescence qui utilise une seule enzyme de couplage disponible dans le commerce et peut générer des données en temps réel (Figure 1). Un oligonucléotide en épingle à cheveux contenant trois méthylcytosines est utilisé comme substrat. L’ADN du substrat contient un fluorophore à l’extrémité 5' et un quencher à l’extrémité 3'. La méthylation du site CpG hémiméthylé génère le site de clivage de l’endonucléase Gla I — GCGC entièrement méthylée. Le clivage Gla I de l’oligonucléotide produit libère le fluorophore de l’anti-quencher et génère une fluorescence en temps réel. Le test peut être utilisé pour examiner l’activité de toute isoforme de DNMT; cependant, une activité plus élevée est observée avec DNMT1 car cet isozyme méthyle préférentiellement l’ADN hémiméthylé 1,5. Une activité encore plus robuste est observée si le domaine autoinhibiteur de la séquence de ciblage des foyers de réplication (RFTS) est supprimé de DNMT1. Ce domaine, trouvé dans la région régulatrice N-terminale, se lie au site catalytique et empêche la liaison à l’ADN. L’élimination des premiers ~600 acides aminés donne une enzyme tronquée qui est significativement plus active que l’enzyme pleine longueur (~640 fois plus de kcat/Km)40. Cette forme activée de l’enzyme, appelée DNMT1 dépourvu de RFTS (acides aminés 621-1616), permet d’identifier plus facilement les inhibiteurs en raison de son pouvoir catalytique accru. Cet article présente un protocole d’utilisation du DNMT1 dépourvu de RFTS dans les tests de dépistage des inhibiteurs potentiels de petites molécules. À l’aide du test continu couplé à l’endonucléase, la vitesse initiale est déterminée en présence et en l’absence de quelques petites molécules. Chaque inhibiteur potentiel est examiné à deux concentrations pour rechercher une inhibition du DNMT1 dépendante de la concentration. Le pourcentage d’activité observé en présence des petites molécules a été calculé dans chaque cas.

Figure 1 : Essai de méthylation de l’ADN. Un ADN d’épingle à cheveux hémiméthylé avec un fluorophore à l’extrémité 5' et un désincant à l’extrémité 3' est utilisé comme substrat. DNMT1 catalyse le transfert du groupe méthyle de la S-adénosylméthionine au site CpG non méthylé, générant de la S-adénosylhomocystéine et de l’ADN entièrement méthylé. Le produit d’ADN contient le site de clivage de l’endonucléase Gla I, qui clive les sites GCGC entièrement méthylés. Le clivage de l’ADN du produit libère le fluorophore 5' de l’anti-quencher 3', générant une fluorescence. Abréviations : Fl = fluorophore; Q = quencher; DNMT1 = ADN méthyltransférase 1; SAM = S-adénosylméthionine; HSA = S-adénosylhomocystéine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparer les solutions de dosage pour le criblage
NOTE: Les concentrations de substrats utilisés dans ce test peuvent être adaptées. Pour le DNMT1 dépourvu de RFTS, les valeurs Km déterminées expérimentalement pour le substrat d’ADN en épingle à cheveux et SAM sont de 1–2 nM et 2 μM, respectivement26,40.
- Préparer 600 μL de chaque condition d’essai testée dans des tubes à microcentrifugeuses sur de la glace.
REMARQUE : Le volume total de solution de dosage nécessaire dépend du nombre de répétitions effectuées. Chaque condition de test a été exécutée en trois exemplaires pour ce protocole.- Ajouter du tampon de méthylation ddH2O2 et 5x (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM glutamate depotassium, 5 mM dithiothréitol (DTT), 25% glycérol) à chaque tube pour obtenir une concentration finale de 1x tampon. Pour les dosages contenant 20 μM composé, ajouter 472 μL deddH2Oet 120 μL de tampon 5x. Pour les dosages contenant 50 μM composé, ajouter 470 μL deddH2Oet 120 μL de tampon 5x.
- Ajouter 3,15 μL de 20 mg/mL d’albumine sérique bovine (BSA) à chaque échantillon.
- Ajouter 2 μL de substrat d’ADN en épingle à cheveux de 3,15 μM et 1,33 μL de SAM de 4,75 mM à chaque échantillon.
NOTE: La concentration des substrats dans le mélange de solution d’essai est de 1,05 fois les concentrations finales souhaitées. - Ajouter l’inhibiteur à une concentration finale de 21 μM (1,26 μL de 10 mM) ou 52,5 μM (3,15 μL de 10 mM) aux échantillons appropriés. Ajouter une quantité équivalente de diméthylsulfoxyde (DMSO) à un échantillon témoin.
REMARQUE: La concentration d’inhibiteur utilisé dans le dépistage peut varier. La concentration finale maximale de DMSO devrait être de ≤5 % (v/v). Des concentrations de DMSO allant jusqu’à 5 % (v/v) n’ont aucun effet sur l’activité observée dans le test26.
- Mélanger les solutions en tourbillonnant (3 000 tr/min) pendant 3 s. Faites tourner les échantillons pendant quelques secondes dans une mini-centrifugeuse de table (1 200 × g) pour vous assurer que la solution est recueillie au fond du tube.
- Aliquote 95 μL de chaque solution d’essai dans 6 puits consécutifs dans une plaque noire de 96 puits en demi-zone (figure 2).
REMARQUE : Ces tests de dépistage ont été effectués en deux lots. Tout d’abord, 20 échantillons d’inhibiteurs μM (4 conditions totales) ont été examinés, suivis des échantillons d’inhibiteurs de 50 μM (4 conditions totales).
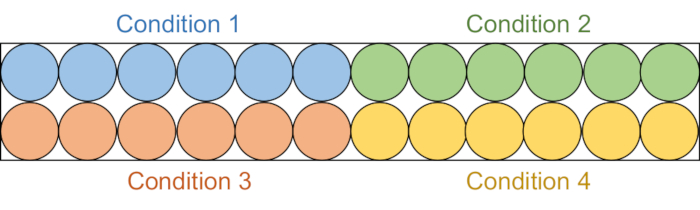
Figure 2 : Configuration de la plaque de dosage. Chaque solution d’essai est aliquote dans six puits dans la plaque noire de 96 puits : témoin DMSO (bleu), composé 1 (vert), composé 2 (rouge) et composé 3 (jaune). DNMT1 et Gla I dépourvus de RFTS seront ajoutés à trois puits. En tant que contrôle, seul Gla I sera ajouté aux trois autres puits. Abréviations : RFTS = Replication Foci Targeting Sequence; DNMT1 = ADN méthyltransférase 1; DMSO = diméthylsulfoxyde. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Préparer des solutions enzymatiques pour le dépistage
REMARQUE : Le DNMT1 dépourvu de RFTS peut être exprimé dans E. coli et purifié jusqu’à homogénéité. Les procédures d’expression et de purification du DNMT1 dépourvu de RFTS ont été décrites précédemment41. Le volume d’enzyme nécessaire dépend du nombre de tests effectués. Ici, quatre tests différents sont effectués dans chaque série; Chaque essai est complété en trois exemplaires.
- Préparer 75 μL de chaque solution enzymatique dans des tubes à microcentrifugation sur de la glace.
NOTE: Deux solutions enzymatiques seront nécessaires. Une solution contiendra DNMT1 et Gla I; l’autre ne contiendra que Gla I.- Ajouter du tampon de méthylation ddH2O2 et 5x (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM glutamate de potassium, 5 mMDTT, 25% glycérol) à chaque tube pour obtenir une concentration finale de 1x tampon. Pour la solution enzymatique Gla I, ajouter 15 μL de tampon 5x et 58,8 μL deddH2O. Pour la solution enzymatique DNMT1+Gla I, ajouter 15 μL de tampon 5x et 58,2 μL deddH2O.
- Ajouter 1,2 μL de 10 U/μL Gla I à chaque solution pour une concentration finale de 0,16 U/μL.
- Ajouter 0,6 μL de DNMT1 dépourvu de RFTS 5 μM pour une concentration finale de 40 nM à la solution DNMT1+Gla I.
- Tapotez doucement pour mélanger les solutions. Faites tourner les échantillons pendant quelques secondes dans une mini-centrifugeuse de table (1 200 × g) pour vous assurer que la solution est recueillie au fond du tube.
- Aliquote 12 μL de chaque solution enzymatique dans 6 puits dans une plaque conique de 96 puits (Figure 3).

Figure 3 : Configuration de la plaque enzymatique. Les solutions Gla I (gris) et DNMT1+Gla I (bleu) sont chacune aliquotes dans six puits dans la plaque de 96 puits. À l’aide d’une pipette multicanal, l’enzyme peut être ajoutée simultanément à une rangée de solutions de dosage. Pour chaque condition d’essai (six puits), trois puits recevront DNMT1+Gla I, et trois puits recevront Gla I seul. Abréviation : DNMT1 = ADN méthyltransférase 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Exécutez le test et analysez les données.
- Préchauffez le lecteur de plaques à 37 °C. Insérez la plaque noire contenant les solutions de dosage. Agiter la plaque (double orbitale à 425 cpm pendant 5 s) et lire la fluorescence avec une longueur d’onde d’excitation de 485 nm et une longueur d’onde d’émission de 528 nm. Incuber la plaque à 37 °C pendant 5 min.
REMARQUE: Alternativement, des filtres avec des coupures de longueur d’onde appropriées peuvent être utilisés pour les lectures de fluorescence. - Retirez la plaque de dosage. Ajouter 5 μL de solution enzymatique à chaque puits et mélanger en pipetant de haut en bas.
Remarque : Utilisez des pipets multicanaux afin qu’une rangée d’échantillons puisse être lancée simultanément. - Insérez la plaque dans le lecteur de plaques. Enregistrer la fluorescence toutes les 53 s pendant 30 min. Agiter la plaque (double orbitale à 425 cpm pendant 4 s) avant chaque lecture.
- Tests moyens de contrôle Gla I triplicate pour chaque condition. Soustrayez la trace moyenne de réaction de contrôle contenant du Gla I des traces triplicates obtenues en présence de DNMT1 dépourvu de RFTS. Déterminer l’erreur moyenne et l’erreur-type de la moyenne pour les répétitions corrigées.
- Tracez la trace de réaction corrigée moyenne et ajustez la partie linéaire initiale à une ligne pour déterminer la vitesse initiale.
- Déterminer le pourcentage d’activité en divisant la vitesse observée en présence d’un composé par la vitesse observée dans la réaction de contrôle contenant du DMSO et en multipliant par 100.
4. Contrôle supplémentaire du dosage — addition séquentielle d’enzymes
- Préparer 550 μL de solution d’essai dans un tube à microcentrifugation sur de la glace. Ajouter 110 μL de tampon de méthylation 5x, 3,88 μL de substrat d’ADN en épingle à cheveux 3,15 μM, 6,43 μL de 47,5 mM SAM, 3,06 μL de BSA 20 mg/mL et 426,6 μL de ddH2O.
- Mélanger la solution en tourbillonnant (3 000 tr/min) pendant 3 s. Faites tourner les échantillons pendant quelques secondes dans une mini-centrifugeuse de table (1 200 × g) pour vous assurer que la solution est recueillie au fond du tube.
- Aliquote 90 μL de la solution d’essai dans 6 puits consécutifs dans une plaque noire de 96 puits en demi-zone.
- Préparer les solutions enzymatiques DNMT1 sur glace. Ajouter 4 μL de tampon de méthylation 5x, 0,76 μL de DNMT1 dépourvu de RFTS 5 μM et 15,24 μL deddH2Odans un tube. Ajouter 4 μL de tampon de méthylation 5x et 16 μL deddH2Odans un autre tube.
- Tapotez doucement pour mélanger les solutions. Faites tourner les échantillons pendant quelques secondes dans une mini-centrifugeuse de table (1 200 × g) pour vous assurer que la solution est recueillie au fond du tube.
- Ajouter 5 μL de la solution contenant du DNMT1 à trois puits dans la plaque noire de 96 puits. Ajouter 5 μL de la solution de contrôle tampon aux trois autres puits.
- Placer la plaque de microtitrage dans le lecteur de plaque préchauffé à 37 °C. Agiter la plaque (double orbitale à 425 cpm pendant 3 s) et lire la fluorescence avec une longueur d’onde d’excitation de 485 nm et une longueur d’onde d’émission de 528 nm. Répétez le shake et lisez toutes les 60 s pendant 30 minutes.
- Pendant que la plaque d’essai est dans le lecteur de plaque, préparer la solution de Gla I sur de la glace. Ajouter 8 μL de tampon de méthylation 5x, 0,64 μL de Gla I 10 U/μL et 31,4 μL deddH2Odans un tube microcentrifugé. Tapotez doucement pour mélanger la solution. Faites tourner l’échantillon pendant quelques secondes dans une mini-centrifugeuse de table (1 200 × g) pour vous assurer que la solution est recueillie au fond du tube.
- Aliquote 6 μL de la solution de Gla I dans 6 puits dans une plaque conique de 96 puits.
- Après la lecture initiale de 30 minutes, retirez la plaque noire du lecteur de plaques. Ajouter 5 μL de solution de Gla I aux 6 puits et mélanger en pipetant de haut en bas.
REMARQUE: Utilisez une pipette multicanal afin que tous les puits puissent être démarrés simultanément. - Replacez la plaque noire dans le lecteur de plaques. Enregistrer la fluorescence toutes les 35 s pendant 35 min. Agiter la plaque (double orbitale à 425 cpm pendant 3 s) avant chaque lecture.
Access restricted. Please log in or start a trial to view this content.
Résultats
Le DNMT1 actif est une condition préalable à cette analyse. Le DNMT1 dépourvu de RFTS a été exprimé dans E. coli et purifié à l’homogénéité selon les procédures précédemment publiées41. Pour s’assurer que l’enzyme purifiée était active, un test discontinu couplé à l’endonucléase a été utilisé pour examiner l’activité de méthylation de l’ADN36. Ce test utilise un ADN duplex de 32 paires de bases avec un seul CpG hémiméthylé po...
Access restricted. Please log in or start a trial to view this content.
Discussion
Pour identifier et caractériser les inhibiteurs de l’ADN méthyltransférases, l’activité de l’enzyme doit être mesurée. Il existe plusieurs méthodes d’examen de l’activité de l’ADN méthyltransférase. L’activité est généralement surveillée à l’aide de la radioactivité; Le transfert du groupe méthyle marqué de SAM peut être quantifié 29,30,31,32,33,34...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs remercient l’Université Bucknell et le Département de chimie pour leur soutien à ce travail.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
Références
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369(2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3'-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15(2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what's next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2'-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44(2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752(2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56(2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830(2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon