Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kontinuierlicher fluoreszenzbasierter Endonuklease-gekoppelter DNA-Methylierungsassay zum Screening auf DNA-Methyltransferase-Inhibitoren
In diesem Artikel
Zusammenfassung
DNA-Methyltransferasen sind potenzielle Ziele für Krebsmedikamente. Hier wird ein Protokoll vorgestellt, um kleine Moleküle auf DNA-Methyltransferase-Hemmung zu untersuchen. Dieser Assay verwendet eine Endonuklease, um die DNA-Methylierung an die Fluoreszenzerzeugung zu koppeln und ermöglicht die Überwachung der Enzymaktivität in Echtzeit.
Zusammenfassung
DNA-Methylierung, eine Form der epigenetischen Genregulation, ist wichtig für die normale Zellfunktion. In Zellen etablieren und erhalten Proteine, die DNA-Methyltransferasen (DNMTs) genannt werden, das DNA-Methylierungsmuster. Veränderungen des normalen DNA-Methylierungsmusters sind mit der Entwicklung und Progression von Krebs verbunden, was DNMTs zu potenziellen Zielen für Krebsmedikamente macht. Daher ist die Identifizierung und Charakterisierung neuartiger niedermolekularer Inhibitoren dieser Enzyme von großer Bedeutung. Dieses Papier stellt ein Protokoll vor, das verwendet werden kann, um nach DNA-Methyltransferase-Inhibitoren zu suchen. Der Test der kontinuierlichen gekoppelten Kinetik ermöglicht es, die Anfangsgeschwindigkeiten der DNA-Methylierung in Gegenwart und Abwesenheit potenzieller niedermolekularer Inhibitoren zu bestimmen. Der Assay verwendet die methylsensitive Endonuklease Gla I, um die Methylierung eines hemimethylierten DNA-Substrats an die Fluoreszenzerzeugung zu koppeln.
Dieser kontinuierliche Assay ermöglicht die Überwachung der Enzymaktivität in Echtzeit. Die Durchführung des Assays in kleinen Volumina in Mikrotiterplatten reduziert die Kosten für Reagenzien. Mit diesem Assay wurde ein kleines Beispiel-Screening für Inhibitoren von DNMT1, dem am häufigsten vorkommenden DNMT-Isoenzym beim Menschen, durchgeführt. Das hochsubstituierte Anthrachinon-Naturprodukt Milchsäure A ist ein potenter, DNA-kompetitiver Inhibitor von DNMT1. Hier untersuchen wir drei potenzielle niedermolekulare Inhibitoren - Anthrachinone oder Anthrachinon-ähnliche Moleküle mit ein bis drei Substituenten - in zwei Konzentrationen, um das Assay-Protokoll zu beschreiben. Anfangsgeschwindigkeiten werden verwendet, um die prozentuale Aktivität zu berechnen, die in Gegenwart jedes Moleküls beobachtet wird. Eine der drei untersuchten Verbindungen weist eine konzentrationsabhängige Hemmung der DNMT1-Aktivität auf, was darauf hindeutet, dass es sich um einen potenziellen Inhibitor von DNMT1 handelt.
Einleitung
DNA-Methylierung ist eine wichtige epigenetische Markierung, die die Genexpression und die Chromatinstruktur reguliert. Die Methylierung erfolgt überwiegend in CpG-Dinukleotiden - Cytosin gefolgt von Guanosin; Die Methylgruppe wird zur 5-Position von Cytosin hinzugefügt. Korrekte DNA-Methylierungsmuster und damit die richtige Genexpression sind für eine angemessene zelluläre Entwicklung und Funktion erforderlich. Viele Krankheitszustände wurden mit Veränderungen des normalen Methylierungsmusters 1,2,3 in Verbindung gebracht. Zum Beispiel gibt es einen Zusammenhang zwischen Krebsentstehung und -progression und Veränderungen des DNA-Methylierungsmusters. Typischerweise weisen Krebszellen insgesamt niedrigere Methylcytosinspiegel auf, was zur Instabilität des Genoms beiträgt. Gleichzeitig konzentriert sich das im Erbgut vorhandene Methylcytosin in den Promotorregionen von Tumorsuppressorgenen, was zu einer Gen-Silencierung dieser wichtigen Proteine führt. Insbesondere epigenetische Veränderungen sind dynamisch und reversibel, im Gegensatz zu den DNA-Mutationen, die mit der Tumorgenese assoziiert sind. Dies hat die Proteine, die an der epigenetischen Genregulation beteiligt sind, zu interessantenWirkstoffzielen 2,4 gemacht.
DNA-Methyltransferasen (DNMTs) sind die Proteine, die für die Erzeugung und Aufrechterhaltung von DNA-Methylierungsmustern verantwortlich sind. Drei katalytisch aktive Isoenzyme, DNMT1, DNMT3a und DNMT3b, existieren beim Menschen. Während der Entwicklung und Differenzierung etablieren die de novo Methyltransferasen DNMT3a und DNMT3b Methylierungsmuster. Beide Enzyme können das katalytisch inaktive DNMT3L-Protein zu Komplexen binden, die eine erhöhte Aktivitätaufweisen 1,5. Nach der Zellteilung enthalten Tochterzellen hemimethylierte DNA - DNA, die Methylcytosin in nur einem Strang des Duplex enthält -, weil die neu synthetisierte DNA frei von Methylierungsmarkierungen ist. Die Hauptfunktion von DNMT1 besteht darin, diese hemimethylierte DNA zu methylieren, wodurch das vollständige Methylierungsmuster 1,5 wiederhergestellt wird.
Zusammenhänge zwischen DNMT-Aktivität und Krebs sind gut etabliert. Die Überexpression von DNMT1, entweder durch transkriptionelle oder posttranslationale Mechanismen, ist eine Folge mehrerer gemeinsamer onkogener Signalwege 6,7,8,9. Genetische Ansätze zur Senkung der DNMT1-Aktivität unter Verwendung hypomorpher Allele führen zu einer verminderten Tumorbildung bei Apc(Min)Mäusen10. Antisense-Oligonukleotide, die DNMT1 niederschlagen, hemmen Neoplasien in Zellkultur- undMaustumormodellen 11,12. Daher scheint die Hemmung der DNMT1-Aktivität ein vielversprechender Krebstherapieansatz zu sein. Die Rollen, die die DNMT3-Isoenzyme spielen, sind jedoch nicht so einfach. DNMT3a-Mutationen finden sich bei akuter myeloischer Leukämie13 und myelodysplastischem Syndrom14. Es wurde gezeigt, dass mindestens eine der identifizierten Mutationen die DNA-Methylierungsaktivität des Enzyms15 verringert. DNMT3b ist jedoch bei Brustkrebs16 und Darmkrebs17 überexprimiert. Da die verschiedenen DNMT-Isoenzyme unterschiedliche Rollen bei der Karzinogenese spielen, wird die Identifizierung isoenzymspezifischer Inhibitoren entscheidend sein. Diese Verbindungen werden nicht nur für die Entwicklung von Therapeutika nützlich sein, sondern isoenzymspezifische Inhibitoren wären auch ein unschätzbares Werkzeug, um die Rolle jedes DNMT-Isoenzyms in der Krebsätiologie zu analysieren.
Mehrere DNMT-Inhibitoren wurden in der Literatur beschrieben. Bekannte DNMT-Inhibitoren können in zwei Klassen unterteilt werden: Nukleosid und Nicht-Nukleosid. Nukleosidinhibitoren sind typischerweise Cytidinanaloga. Diese Verbindungen werden in die DNA eingebaut und fangen DNMTs kovalent ein. 5-Azacytidin und 5-Aza-2'-Desoxycytidin sind für die Behandlung des myelodysplastischen Syndroms und der akuten myeloischen Leukämie zugelassen 4,18. Die hohe Toxizität, geringe Bioverfügbarkeit und chemische Instabilität dieser Verbindungen stellen Probleme dar. Laufende Arbeiten untersuchen die Wirksamkeit der nächsten Generation von Nukleosidinhibitoren; SGI-110, abgeleitet von 5-Aza-2'-Desoxycytidin, ist ein Beispiel19,20. Nukleosid-Inhibitoren sind nicht isoenzymspezifisch und inaktivieren jedes auftretende DNMT-Isoenzym. Daher führt die Behandlung mit einem nukleosiddemethylierenden Mittel zur Erschöpfung aller DNMT-Isoenzyme 4,18. Nicht-Nukleosid-Inhibitoren müssen nicht in die DNA eingebaut werden, um ihre hemmende Wirkung auszuüben. Stattdessen binden diese Moleküle direkt an DNMTs, was die Möglichkeit einer isoenzymspezifischen Hemmung einführt. Bisher wurden mehrere Nicht-Nukleosid-Inhibitoren entdeckt, darunter SGI-1027 21, Hydralazin-22, Procainamid23, RG108 und Derivate 24 sowie die Naturstoffe (−)-Epigallocateching-3-Gallat (EGCG)25 und Milchsäure A 26,27. Die meisten der bisher entdeckten Nicht-Nukleosid-Inhibitoren sind nicht isoenzymselektiv oder zeigen schwache Präferenzen für ein DNMT-Isoenzym. Darüber hinaus muss die Wirksamkeit dieser Moleküle verbessert werden, insbesondere in Zellen 4,18. Daher besteht die Notwendigkeit, potentere, isoenzymselektive DNMT-Inhibitoren zu entdecken oder zu entwickeln.
Eine Hürde bei der Entdeckung neuer niedermolekularer Inhibitoren von DNMTs sind die aufwendigen Assays, die traditionell zur Untersuchung der DNMT-Aktivität verwendetwerden 28. Assays sind in der Regel diskontinuierlich mit mehreren Schritten. Die enzymatische Aktivität von DNMTs wird immer noch routinemäßig mit radioaktivem S-Adenosylmethionin (SAM) 29,30,31,32,33,34 untersucht. Nicht-radioaktive Assays für die DNA-Methylierung wurden ebenfalls entwickelt. Zum Beispiel wurden Assays beschrieben, die methylsensitive Restriktionsendonukleasen und Elektrophorese zur Trennung der Verdauungsprodukte verwenden35,36. Diese Arten von diskontinuierlichen, mehrstufigen Assays sind für die Wirkstoffforschung nicht ohne weiteres zugänglich. Seit Mitte der 2000er Jahre wurden mehrere DNA-Methylierungsassays mit höherem Durchsatz entwickelt28. Ein Szintillations-Proximity-Assay wurde verwendet, um nach DNMT1-Inhibitoren zu suchen37. Ein weiterer Assay unter Verwendung einer methylsensitiven Restriktionsendonuklease wurde verwendet, um nach DNMT3a-Inhibitoren25,38 zu suchen. Während beide Assays einen höheren Durchsatz als herkömmliche DNA-Methylierungsassays ermöglichten, erfordern die Assays mehrere Schritte und erlauben nicht die Beobachtung der Methylierungsaktivität in Echtzeit. In jüngerer Zeit wurde ein kontinuierlicher Kinetik beschrieben, der die Bildung von S-Adenosylhomocystein (SAH), einem Produkt der Methylierungsreaktion, an die spektroskopische Veränderung bei 340 nm koppelt, die mit der NADPH-Oxidation39 assoziiert ist. Dieser Assay verwendet drei Kopplungsenzyme, um ein spektroskopisches Signal zu erzeugen.
Wir entwickelten einen fluoreszenzbasierten Endonuklease-gekoppelten DNA-Methylierungsassay, der ein einzelnes kommerziell verfügbares Kopplungsenzym verwendet und Daten in Echtzeit generieren kann (Abbildung 1). Als Substrat wird ein Haarnadel-Oligonukleotid verwendet, das drei Methylcytosine enthält. Die Substrat-DNA enthält einen Fluorophor am 5'-Ende und einen Quencher am 3'-Ende. Die Methylierung der hemimethylierten CpG-Stelle erzeugt die Spaltstelle für die Endonuklease Gla I - vollständig methyliertes GCGC. Die Gla-I-Spaltung des Produktoligonukleotids setzt den Fluorophor aus dem Quencher frei und erzeugt Fluoreszenz in Echtzeit. Der Assay kann verwendet werden, um die Aktivität jeder Isoform von DNMT zu untersuchen; jedoch wird eine höhere Aktivität mit DNMT1 beobachtet, da dieses Isoenzym bevorzugt hemimethylierte DNA 1,5 methyliert. Eine noch robustere Aktivität wird beobachtet, wenn die autoinhibitorische RFTS-Domäne (Replication Foci Targeting Sequence) aus DNMT1 entfernt wird. Diese Domäne, die sich in der N-terminalen regulatorischen Region befindet, bindet an die katalytische Stelle und verhindert die DNA-Bindung. Die Entfernung der ersten ~600 Aminosäuren führt zu einem verkürzten Enzym, das signifikant aktiver ist als das Enzym in voller Länge (~640-facher Anstieg in kKatze / Km)40. Diese aktivierte Form des Enzyms, die als RFTS-fehlendes DNMT1 (Aminosäuren 621–1616) bezeichnet wird, ermöglicht aufgrund ihrer erhöhten katalytischen Kraft die leichtere Identifizierung von Inhibitoren. Dieses Papier stellt ein Protokoll vor, um RFTS-fehlendes DNMT1 in Assays zu verwenden, um nach potenziellen niedermolekularen Inhibitoren zu suchen. Mit dem Endonuklease-gekoppelten kontinuierlichen Assay wird die Anfangsgeschwindigkeit in Gegenwart und Abwesenheit einiger kleiner Moleküle bestimmt. Jeder potenzielle Inhibitor wird bei zwei Konzentrationen untersucht, um nach konzentrationsabhängiger DNMT1-Hemmung zu suchen. Die prozentuale Aktivität, die in Gegenwart der kleinen Moleküle beobachtet wurde, wurde jeweils berechnet.

Abbildung 1: DNA-Methylierungstest. Als Substrat wird eine hemimethylierte Haarnadel-DNA mit einem Fluorophor am 5'-Ende und einem Quencher am 3'-Ende verwendet. DNMT1 katalysiert den Transfer der Methylgruppe von S-Adenosylmethionin zur nicht methylierten CpG-Stelle und erzeugt S-Adenosylhomocystein und vollständig methylierte DNA. Das DNA-Produkt enthält die Spaltstelle für die Endonuklease Gla I, die vollständig methylierte GCGC-Stellen spaltet. Die Spaltung der Produkt-DNA setzt den 5'-Fluorophor aus dem 3'-Quencher frei und erzeugt Fluoreszenz. Abkürzungen: Fl = Fluorophor; Q =Quencher; DNMT1 = DNA-Methyltransferase 1; SAM = S-Adenosylmethionin; SAH = S-Adenosylhomocystein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Bereiten Sie Assay-Lösungen für den Bildschirm vor
HINWEIS: Die Konzentrationen der in diesem Assay verwendeten Substrate können angepasst werden. Für RFTS-fehlendes DNMT1 betragen die experimentell ermitteltenK-m-Werte für das Haarnadel-DNA-Substrat und SAM 1–2 nM bzw. 2 μM bzw.26,40.
- Bereiten Sie 600 μL jeder Testbedingung vor, die in Mikrozentrifugenröhrchen auf Eis getestet wird.
HINWEIS: Das Gesamtvolumen der benötigten Assaylösung hängt von der Anzahl der durchgeführten Replikate ab. Jede Assay-Bedingung wurde für dieses Protokoll in dreifacher Ausfertigung durchgeführt.- Fügen Sie ddH2 O und 5x Methylierungspuffer (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM Kaliumglutamat, 5 mM Dithiothreitol (DTT), 25% Glycerin) zu jedem Röhrchen hinzu, um eine Endkonzentration von 1x Puffer zu erreichen. Für Assays, die 20 μM Verbindung enthalten, fügen Sie 472 μLddH2Ound 120 μL 5x Puffer hinzu. Für Assays, die eine 50-μM-Verbindung enthalten, werden 470 μLddH2Ound 120 μL 5x-Puffer hinzugefügt.
- Zu jeder Probe werden 3,15 μL 20 mg/ml Rinderserumalbumin (BSA) gegeben.
- Fügen Sie jeder Probe 2 μL 3,15 μM Haarnadel-DNA-Substrat und 1,33 μL 4,75 mM SAM hinzu.
HINWEIS: Die Konzentration der Substrate in der Assaylösungsmischung beträgt das 1,05-fache der gewünschten Endkonzentrationen. - Inhibitor zu einer Endkonzentration von 21 μM (1,26 μL von 10 mM) oder 52,5 μM (3,15 μL von 10 mM) in die entsprechenden Proben geben. Geben Sie eine äquivalente Menge Dimethylsulfoxid (DMSO) zu einer Kontrollprobe.
HINWEIS: Die Konzentration des im Bildschirm verwendeten Inhibitors kann variiert werden. Die maximale Endkonzentration von DMSO sollte ≤5% (v/v) betragen. DMSO-Konzentrationen bis zu 5% (v/v) haben keinen Einfluss auf die im Assay26 beobachtete Aktivität.
- Mischen Sie die Lösungen durch Wirbeln (3.000 U/min) für 3 s. Drehen Sie die Proben einige Sekunden lang in einer Tisch-Minizentrifuge (1.200 × g), um sicherzustellen, dass die Lösung am Boden des Röhrchens gesammelt wird.
- Aliquot 95 μL jeder Assaylösung in 6 aufeinanderfolgende Vertiefungen in einer schwarzen Halbflächenplatte mit 96 Vertiefungen (Abbildung 2).
HINWEIS: Diese Screening-Assays wurden in zwei Chargen durchgeführt. Zuerst wurden 20 μM-Inhibitorproben (4 Gesamtbedingungen) untersucht, gefolgt von den 50 μM-Inhibitorproben (4 Gesamtbedingungen).
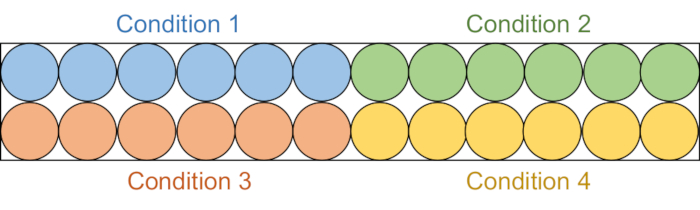
Abbildung 2: Aufbau der Assay-Platte. Jede Assay-Lösung wird in sechs Vertiefungen in der schwarzen 96-Well-Platte aliquotiert: DMSO-Kontrolle (blau), Verbindung 1 (grün), Verbindung 2 (rot) und Verbindung 3 (gelb). Sowohl RFTS-fehlende DNMT1 als auch Gla I werden zu drei Bohrungen hinzugefügt. Als Kontrolle wird Gla I allein zu den anderen drei Brunnen hinzugefügt. Abkürzungen: RFTS = Replication Foci Targeting Sequence; DNMT1 = DNA-Methyltransferase 1; DMSO = Dimethylsulfoxid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Bereiten Sie Enzymlösungen für das Screening vor
HINWEIS: RFTS-fehlendes DNMT1 kann in E. coli exprimiert und auf Homogenität gereinigt werden. Expressions- und Reinigungsverfahren für RFTS-fehlendes DNMT1 wurden zuvorbeschrieben 41. Das Volumen des benötigten Enzyms hängt von der Anzahl der durchgeführten Assays ab. Hier werden in jedem Set vier verschiedene Assays durchgeführt; Jeder Assay wird in dreifacher Ausfertigung durchgeführt.
- Bereiten Sie 75 μL jeder Enzymlösung in Mikrozentrifugenröhrchen auf Eis vor.
HINWEIS: Es werden zwei Enzymlösungen benötigt. Eine Lösung enthält DNMT1 und Gla I; der andere wird nur Gla I enthalten.- Fügen Sie ddH2 O und 5x Methylierungspuffer (250 mM Tris pH 7,5, 5 mM MgCl2, 500 mM Kaliumglutamat, 5 mM DTT, 25% Glycerin) zu jedem Röhrchen hinzu, um eine Endkonzentration von 1x Puffer zu erreichen. Für die Gla I-Enzymlösung fügen Sie 15 μL 5x Puffer und 58,8 μLddH2Ohinzu. Für die DNMT1+Gla I-Enzymlösung werden 15 μL 5x Puffer und 58,2 μLddH2Ohinzugefügt.
- Zu jeder Lösung werden 1,2 μl 10 U/μL Gla I für eine Endkonzentration von 0,16 U/μL gegeben.
- 0,6 μL 5 μM RFTS-fehlendes DNMT1 für eine Endkonzentration von 40 nM in die DNMT1+Gla I-Lösung geben.
- Klopfen Sie vorsichtig, um die Lösungen zu mischen. Drehen Sie die Proben einige Sekunden lang in einer Tisch-Minizentrifuge (1.200 × g), um sicherzustellen, dass die Lösung am Boden des Röhrchens gesammelt wird.
- Aliquot 12 μL jeder Enzymlösung in 6 Vertiefungen in einer konischen 96-Well-Platte (Abbildung 3).

Abbildung 3: Aufbau der Enzymplatte. Die Lösungen Gla I (grau) und DNMT1+Gla I (blau) sind jeweils in sechs Vertiefungen in der 96-Well-Platte aliquotiert. Mit einer Mehrkanalpipette kann das Enzym gleichzeitig zu einer Reihe von Assaylösungen hinzugefügt werden. Für jede Untersuchungsbedingung (sechs Bohrungen) erhalten drei Bohrlöcher DNMT1+Gla I und drei Bohrungen nur Gla I. Abkürzung: DNMT1 = DNA-Methyltransferase 1. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
3. Führen Sie den Assay durch und analysieren Sie die Daten.
- Plattenleser auf 37 °C vorheizen. Legen Sie die schwarze Platte mit den Assaylösungen ein. Schütteln Sie die Platte (Doppelorbital bei 425 cpm für 5 s) und lesen Sie die Fluoreszenz mit einer Anregungswellenlänge von 485 nm und einer Emissionswellenlänge von 528 nm ab. Die Platte bei 37 °C 5 min inkubieren.
HINWEIS: Alternativ können Filter mit geeigneten Wellenlängengrenzwerten für die Fluoreszenzmessungen verwendet werden. - Entfernen Sie die Assay-Platte. Fügen Sie 5 μL Enzymlösung in jede Vertiefung und mischen Sie durch Pipettieren auf und ab.
HINWEIS: Verwenden Sie Mehrkanalpipetten, damit eine Reihe von Proben gleichzeitig initiiert werden kann. - Setzen Sie die Platte in das Plattenlesegerät ein. Fluoreszenz alle 53 s für 30 min aufzeichnen. Schütteln Sie die Platte (Doppelorbital bei 425 cpm für 4 s) vor jeder Messung.
- Durchschnittliche dreifache Gla I Kontrollassays für jede Bedingung. Subtrahieren Sie die durchschnittliche Gla I-haltige Kontrollreaktionsspur von den dreifachen Spuren, die in Gegenwart von RFTS-fehlendem DNMT1 erhalten wurden. Bestimmen Sie den Mittelwert und den Standardfehler des Mittelwerts für die korrigierten Replikate.
- Zeichnen Sie die durchschnittlich korrigierte Reaktionsspur und passen Sie den anfänglichen linearen Abschnitt an eine Linie an, um die Anfangsgeschwindigkeit zu bestimmen.
- Bestimmen Sie die prozentuale Aktivität, indem Sie die in Gegenwart einer Verbindung beobachtete Geschwindigkeit durch die in der DMSO-haltigen Kontrollreaktion beobachtete Geschwindigkeit dividieren und mit 100 multiplizieren.
4. Zusätzliche Assay-Kontrolle — sequentielle Zugabe von Enzymen
- Bereiten Sie 550 μL Assaylösung in einem Mikrozentrifugenröhrchen auf Eis vor. Fügen Sie 110 μL 5x Methylierungspuffer, 3,88 μL 3,15 μM Haarnadel-DNA-Substrat, 6,43 μL 47,5 mM SAM, 3,06 μL 20 mg/ml BSA und 426,6 μLddH2Ohinzu.
- Mischen Sie die Lösung durch Wirbeln (3.000 U/min) für 3 s. Drehen Sie die Proben einige Sekunden lang in einer Tisch-Minizentrifuge (1.200 × g), um sicherzustellen, dass die Lösung am Boden des Röhrchens gesammelt wird.
- Aliquot 90 μL der Assaylösung in 6 aufeinanderfolgende Vertiefungen in einer schwarzen Halbflächenplatte mit 96 Vertiefungen.
- Bereiten Sie die DNMT1-Enzymlösungen auf Eis vor. Fügen Sie 4 μL 5x Methylierungspuffer, 0,76 μL 5 μM RFTS-fehlendes DNMT1 und 15,24 μLddH2Ozu einem Röhrchen hinzu. 4 μL 5x Methylierungspuffer und 16 μLddH2Oin ein anderes Röhrchen geben.
- Klopfen Sie vorsichtig, um die Lösungen zu mischen. Drehen Sie die Proben einige Sekunden lang in einer Tisch-Minizentrifuge (1.200 × g), um sicherzustellen, dass die Lösung am Boden des Röhrchens gesammelt wird.
- 5 μL der DNMT1-haltigen Lösung in drei Vertiefungen in die schwarze Halbflächenplatte mit 96 Vertiefungen geben. Geben Sie 5 μL der Pufferkontrolllösung in die anderen drei Vertiefungen.
- Legen Sie die Mikrotiterplatte in den auf 37 °C vorgeheizten Plattenleser. Schütteln Sie die Platte (Doppelorbital bei 425 cpm für 3 s) und lesen Sie die Fluoreszenz mit einer Anregungswellenlänge von 485 nm und einer Emissionswellenlänge von 528 nm. Wiederholen Sie den Shake und lesen Sie alle 60 s für 30 Minuten.
- Während sich die Testplatte im Plattenleser befindet, bereiten Sie die Gla I-Lösung auf Eis vor. 8 μL 5x Methylierungspuffer, 0,64 μL 10 U/μL Gla I und 31,4 μLddH2Oin ein Mikrozentrifugenröhrchen geben. Klopfen Sie vorsichtig, um die Lösung zu mischen. Drehen Sie die Probe einige Sekunden lang in einer Tisch-Minizentrifuge (1.200 × g), um sicherzustellen, dass die Lösung am Boden des Röhrchens gesammelt wird.
- Aliquot 6 μL der Gla I-Lösung in 6 Vertiefungen in einer konischen 96-Well-Platte.
- Entfernen Sie nach den ersten 30 Minuten Lesezeit die schwarze Platte aus dem Plattenleser. 5 μL Gla I Lösung in alle 6 Vertiefungen geben und durch Pipettieren auf und ab mischen.
HINWEIS: Verwenden Sie eine Mehrkanalpipette, damit alle Vertiefungen gleichzeitig eingeleitet werden können. - Setzen Sie die schwarze Platte wieder in das Plattenlesegerät ein. Fluoreszenz alle 35 s für 35 min aufzeichnen. Schütteln Sie die Platte (Doppelorbital bei 425 cpm für 3 s) vor jeder Messung.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Aktives DNMT1 ist Voraussetzung für diese Analyse. RFTS-fehlendes DNMT1 wurde in E. coli exprimiert und nach zuvor veröffentlichten Verfahren41 auf Homogenität gereinigt. Um sicherzustellen, dass das gereinigte Enzym aktiv war, wurde ein diskontinuierlicher Endonuklease-gekoppelter Assay verwendet, um die DNA-Methylierungsaktivität36 zu untersuchen. Dieser Assay verwendet eine 32-Basenpaar-Duplex-DNA mit einem einzelnen hemimethylierten CpG, das in einer Sau3A1...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Um Inhibitoren von DNA-Methyltransferasen zu identifizieren und zu charakterisieren, muss die Aktivität des Enzyms gemessen werden. Es gibt mehrere Methoden zur Untersuchung der DNA-Methyltransferase-Aktivität. Die Aktivität wird üblicherweise mit Radioaktivität überwacht; Der Transfer der markierten Methylgruppe von SAM kann quantifiziert werden 29,30,31,32,33,34.
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken der Bucknell University und dem Department of Chemistry für ihre Unterstützung dieser Arbeit.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
Referenzen
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369(2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3'-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15(2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what's next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2'-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44(2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752(2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56(2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830(2019).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten