このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
遺伝的にコードされた電圧インジケーターを用いたミトコンドリア膜電位 のin vivo 測定
要約
このプロトコルは、ミトコンドリアを標的とする遺伝的にコードされた電圧インジケータ(GEVI)のアプリケーションについて説明しています。これらのGEVIは、ミトコンドリア膜電位の特異的、 in vivo、およびリアルタイムのモニタリングを可能にすることにより、従来のミトコンドリア膜電位色素に比べて大きな利点を提供します。
要約
ミトコンドリア膜電位(MMP、ΔΨm)は、ATP合成、イオン輸送、活性酸素種(ROS)の生成、核にコードされるタンパク質の輸入など、ミトコンドリアの機能にとって重要です。ΔΨm を測定する既存の方法は、通常、ローダミン800やテトラメチルローダミンメチルエステル(TMRM)などの親油性カチオン色素を使用しますが、これらは特異性が低いため制限があり、 in vivo アプリケーションにはあまり適していません。これらの制限に対処するために、遺伝的にコードされた電圧インジケータ(GEVI)を利用した新しいプロトコルを開発しました。膜電位の変化に応答して蛍光シグナルを生成する遺伝的にコードされた電圧指標(GEVI)は、原形質膜およびニューロン電位のモニタリングに大きな可能性を示しています。しかし、ミトコンドリア膜への応用はまだ未開拓です。本研究では、生きた動物の細胞や運動野のΔΨm揺らぎを検出できるタンパク質をベースとしたミトコンドリア標的GEVIを開発しました。ミトコンドリア電位指標(MPI)は、ΔΨm ダイナミクスをリアルタイムで研究するための非侵襲的アプローチを提供し、正常条件と病理学的条件の両方でミトコンドリア機能を調査する方法を提供します。
概要
ミトコンドリアは真核細胞の必須細胞小器官であり、アデノシン三リン酸(ATP)の生成を通じて主要なエネルギー供給者として機能するとともに、代謝物合成、カルシウムイオンの緩衝、熱産生、細胞生存の調節など、さまざまな重要な機能も果たしています1。その役割は、脳や心臓などの代謝性の高い組織で特に重要であり、細胞の恒常性を維持するのに役立ちます。ミトコンドリア膜電位(MMP、Ψm)は、酸化的リン酸化によるATP合成の促進、ミトコンドリア膜を介した代謝物とイオンの輸送の促進、活性酸素種(ROS)の生成に寄与するなど、これらのプロセスの中心です2,3。MMPは、マイトファジー(ミトコンドリアの選択的分解)5やアポトーシス(プログラムされた細胞死)6など、ミトコンドリアの形態と動態4にも影響を与えます。適切なΨmを維持することは、細胞機能にとって不可欠です。その調節不全は、神経変性疾患、心不全、癌など、多くの病状に関連しています。現在のΨm測定法は、TMRM(テトラメチルローダミンメチルエステル)、TMRE(テトラメチルローダミンエチルエステル)、ローダミン123、サフラニンO、ローダミン800、DiOC6、JC-1などの親油性カチオン性色素の使用を主としていました7。ただし、これらの蛍光分子にはいくつかの制限があります。これらの色素は細胞特異性を欠き、消光の影響を受けやすく、一部は有毒です。さらに、それらは時間の経過とともに拡散することがあり、ミトコンドリアΔΨが失われると漏れ出し、脱分極したミトコンドリアの膜電位を示すことができなくなります。さらに、TMRMやTMREなどのロダミンベースの色素は温度感受性があり8、特に細胞の熱発生に関与する生理活性中のミトコンドリア膜電位を測定する場合は、色素の蛍光に対する温度の影響を慎重に検討する必要があります。
遺伝的にコードされた電位指標(GEVI)は、蛍光シグナル9,10を通じて膜電位の変化を検出できるタンパク質であり、さまざまな細胞状況で膜電位を監視するための強力なツールとして浮上している11。GEVIは原形質膜の研究に広く適用されてきましたが、特にミトコンドリアの細胞内膜電位を測定するためのGEVIの適応はほとんど進んでいません。このプロトコルは、in vitro および in vivo でミトコンドリア膜電位を監視できるミトコンドリア標的 GEVI を使用して、このギャップに対処しようとしています。既存のGEVIにミトコンドリアシグナル配列を付加することにより、適切なGEVIをミトコンドリア12に標的化することができる。これらのミトコンドリア電位指標(MPI)は、ミトコンドリアの生理学に関する新たな洞察を提供し、in vivoでのさまざまな病態におけるミトコンドリアの機能を探索するための大きな可能性を提供し、ミトコンドリアのダイナミクスが正常な細胞プロセスと病理学的な細胞プロセスの両方にどのように寄与するかについての理解を深めます。
プロトコル
すべての動物の世話と実験は、鄭州大学の施設動物管理および使用委員会のガイドラインに従って行われました。使用前にすべての手術器具を滅菌してください。感染を防ぐために、無菌技術に従ってください。すべてのデータが取得された後、動物は吸入麻酔の過剰投与とそれに続く斬首を使用して安楽死させられました。
1. in vitro アプリケーション
- プラスミド構造
- Addgeneから、または配列から合成する活動電位1(ASAP1)およびASAP3遺伝子の加速センサーを取得します。(ASAP1、NCBIアクセッションID:AHV90412.1、Addgene ID:52519;ASAP3、Addgene ID:132331)。
- プラスミド構造には 、表1 にリストされているプライマーを使用してください。
- 細胞での発現には、EGFPN1ベクターを構築に使用します。
- 最初に、Cox8遺伝子配列(NP_004065、アミノ酸1〜29)を、NheIおよびXhoI制限部位に隣接する4つのタンデムリピート(4cox8)で合成します。合成した4cox8フラグメントとEGFPN1ベクターの両方をNheIおよびXhoIで消化し、続いてライゲーションして4cox8-EGFPベクターを生成します。
- 続いて、ASAP1配列のPCR増幅をプライマー1およびプライマー2で行います。ASAP1フラグメントと4cox8-EGFPベクターをSalIおよびNotIで消化し、続いてライゲーションして最終的なMPI-1ベクターを生成します(MPIはミトコンドリア電位インジケーターの略です)。
注:CMV-MPI-1のベクトルマップを 図1に示します。詳細な配列はNCBI(アクセッションID:PQ678920)からアクセスできます。
- ニューロンでの特異的発現のためには、AAV-hSyn-EGFPプラスミド(NCBIアクセッションID:MH458079、Addgene ID:50465)のhSynプロモーター配列のPCR増幅をプライマー3およびプライマー4を用いて行いましょう。
- hSyn配列と4cox8-EGFPベクターの両方をAseIおよびNheIで消化し、続いてライゲーションを行い、hSyn-4cox8-EGFPベクターを生成します。続いて、ASAP3配列のPCR増幅をプライマー1およびプライマー2で行います。
- hSyn-4cox8-EGFPベクターとASAP3配列をSalIおよびNotIで消化し、続いてライゲーションを行い、hSyn-MPI-2ベクターを生成します。
注:PCR条件は、特定の酵素とプライマー融解温度(Tm)によって異なります。一般的なPCR反応では、95°Cで30秒間の初期変性ステップを行い、その後、25〜35サイクルの変性(95°C、10秒)、アニーリング(55°C、30秒)、および伸長(72°C、30秒/kb)を行います。72°Cで5分間の最終的な伸長ステップにより、完全な製品合成が保証され、その後4°Cで保持されます。 アニーリング温度は、プライマーのTmより約5°C低くする必要があります。最適なPCRパラメータについては、製造元の指示を参照してください。ASAP1とASAP3のプライマーは同じです。
- 生細胞でのイメージング
- 10%ウシ胎児血清と100 mg/Lペニシリン/ストレプトマイシンを培地に添加して、DMEM培地を調製します。
- Ca2+ トランスフェクション試薬を調製します: 2.5 mol/L CaCl2、2x HEBS (274 mmol/L NaCl、10 mmol/L KCl、1.4 mmol/L Na2HPO4、15 mmol/L D-グルコース、42 mmol/L HEPES、pH 7.07)。
- イメージング中に細胞を維持するためのTyrode緩衝液を調製します(145 mmol/L NaCl、3 mmol/L KCl、10 mmol/L HEPES、10 mmol/L Glucose、pH 7.4)。
- 35 mmの培養皿にDMEM培養培地を投入し、温度37°C、雰囲気5% CO2/95%空気のインキュベーターでHela細胞を培養します。皿ごとに2〜3枚のカバースリップを追加して、細胞が成長できるようにします。
- リン酸カルシウム沈殿法によりHela細胞をトランスフェクションします。
- 細胞をトランスフェクションするには、まず古い培地を2 mLの無血清DMEMに交換します。35分後、10 μgのDNA、4 μLのCaCl2、およびddH2Oを微量遠心チューブ内で最終容量40 μLに混合して、DNA/Ca2+溶液を調製します。
- 40 μLの2x HEBSをDNA/Ca2+ 溶液に滴下しながら、激しく攪拌します。ピペットで混合物に空気を泡立てて、リン酸カルシウムの沈殿を促進します。
- 25分後、この沈殿液80μLを細胞と一緒に皿に加え、さらに1〜2時間インキュベーターに戻します。次に、非血清DMEMを取り出し、15%グリセロール(PBSに溶解)で細胞を2分間すすぎます。
- グリセロールを吸引した後、10%FBSを含むDMEMを2mL加え、細胞を16〜24時間培養し続けます。
- 細胞の入ったカバースリップを2 mLのTyrodeバッファーを入れた35 mmの培養皿に入れ、20 μmol/Lのローダミン800(ddH2Oに溶解)を皿に添加して最終濃度50 nmol/Lまでローダミン800色素で細胞を染色します。
- 蛍光顕微鏡を準備し、適切なフィルターと光源を使用してセットアップし、488 nmの光源でタンパク質を励起し、490〜540 nmの発光を収集します。ローダミン800色素を633 nmの光源で励起し、650〜720 nmの発光を収集します。
- シアン化カルボニルm-クロロフェニルヒドラゾン(CCCP)溶液を500 μM(ストック)で調製し、イメージング中に最終濃度5 μMまで細胞に添加します。
注:2x HEBS溶液のpHは、トランスフェクション効率にとって非常に重要です。pHは、固体の水酸化ナトリウムで7.07に慎重に調整する必要があります。pHは7.01未満または7.12より高くすることはできません。
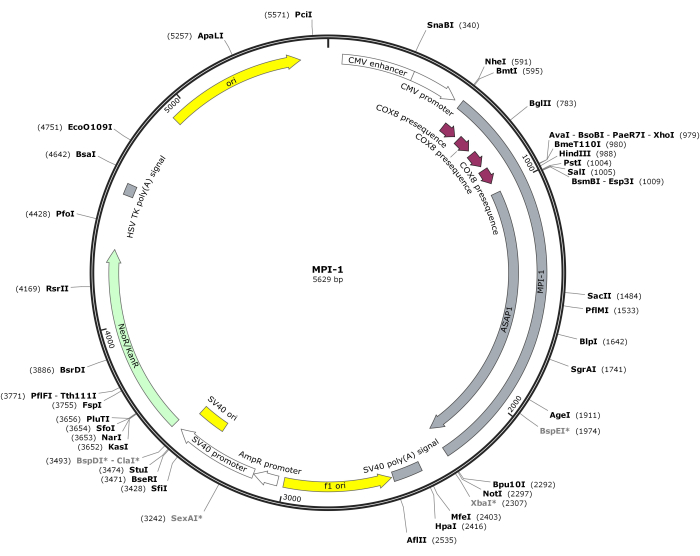
図1:CMV-MPI-1のベクトルマップ。この図の拡大版を表示するには、ここをクリックしてください。
2. in vivo アプリケーション
- プラスミドコンストラクト
- in vivoでの発現には、形質導入にAAVウイルスを使用します。AAVウイルスパッケージング用の逆末端反復(ITR)配列を含むAAVパッケージング用の発現バックボーン(Addgene ID:46954または26968)を取得します。
- hSyn-MPI-2ベクター(ステップ1.1.4から)のPCR増幅を 、表1のプライマー5および7を用いて行います。
- 表1の同じテンプレートとプライマー6および8を使用して、2回目のPCR増幅を行います。
- 第1および第2のPCRの産物(ステップ2.1.2および2.1.3)をテンプレートおよび 表1のプライマー5および8として使用して最終PCRを実施し、最終的なhSyn-MPI-2配列を生成します。
- hSyn-MPI-2配列とpAAV骨格をMluIおよびEcoRIで消化し、続いてライゲーションを行い、最終的なpAAV-hSyn-MPI-2コンストラクトを生成します。pAAV-hSyn-MPI-2のベクトルマップを 図2に示します。詳細な配列はNCBI(アクセッションID:PQ678919)からアクセスできます。
注:hSyn-MPI-2ベクターには、元のEGFPN1骨格に由来する内部EcoRI部位が含まれており、これはオーバーラップPCR戦略(ステップ2.1.2-2.1.4)で修飾プライマーを使用して除去されます。
- ウイルスの調製と形質導入
- 温度37°C、雰囲気5%CO、2/95%空気のインキュベーターで10% FBS DMEM培地を給餌した100 mm培養皿でHEK293t細胞を培養し、70%のコンフルエンスに到達します。
- リン酸カルシウム沈殿法を用いて、HEK293t細胞にAAV遺伝子プラスミド、キャプシド(pAAV-DJまたはpAAV9)、およびヘルパープラスミド(pHelper)を同時導入します。各プラスミドのDNA量は、遺伝子:10μg、pHelper:11μg、キャプシド:9μgでした。トランスフェクション試薬量:12 μL の CaCl2、120 μL の 2x HEBS
- トランスフェクションした細胞を72時間インキュベートして、ウイルス産生を可能にします。
- インキュベーション期間後、400 gの遠心分離機で細胞を回収します。細胞をPBSで2回すすぎ、細胞を400μLのPBSに再懸濁します。
- 細胞懸濁液からウイルスを4サイクルの凍結融解法(37°Cおよび-80°C)で回収します。ライセートを13,500 × g 、4°Cで遠心分離し、ペレットを廃棄します。上清にはAAVウイルスが含まれています。
- リアルタイムPCRを実施して、調製したウイルスの滴定を決定します。AAV滴定測定の詳細なプロトコルは、https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/ に記載されています。
- 調製したAAVウイルスを分注し、-80°Cで保存します。
- 定位ウイルス注射
- 体重 24-26 g の成体 C57/BL 雄マウスに 3% ペントバルビタールナトリウムを 30 mg/kg の腹腔内注射で麻酔します。麻酔をかけたら、マウスを定位固定装置に固定して、処置全体を通して安定した位置を維持します。
- 獣医用眼科用軟膏を塗布して、光による損傷から目を保護し、目を湿らせます。.
- 脱毛クリームを使用して、マウスの頭の毛を取り除き、頭皮を露出させます。
- 手術用ハサミで頭皮を開き、頭蓋骨を露出させます。頭蓋骨を3%過酸化水素で処理し、ブレグマやラムダサイトなどの主要なランドマークを滅菌して視覚化します。
- マウスの頭の位置を調整して、頭蓋骨が水平になるようにします。
- プログラム可能なマイクロピペットプーラーを使用して、ガラスピペットを希望の形状とサイズに引っ張り、正確な注入を実現します。ガラスマイクロピペットにパラフィンオイルを入れます。
- マイクロピペットをターゲットサイトに配置します。このプロトコルでは、ターゲットは右側のM2領域であり、次の座標を使用します:AP(前後)、+ 1.94 mm;ML(中外側)、+ 0.75 mm;DV(背腹側)、-1.5mm。
- 電動ドリルを使用して、目的の部位の頭蓋骨に小さな穴を開けます。
- マイクロピペットの先端から1.5 x 1011 ゲノムコピー/ミリリットル(GC/mL)の力価で500 nLのウイルスを熱望します。注入ポンプを使用して、100 nL/minの流量で500 nLのウイルスを標的部位に注入します。
- 注入後、ピペットを約5分間そのままにして拡散させ、慎重に引っ込めます。
- 注入部位に光ファイバー(直径200μm、開口数0.37、長さ2mm)を注入します。
- デンタルセメントを使用して光ファイバーを所定の位置に固定します。
- マウスの頭皮を5-0ナイロン糸(審美針Δ1/2 4x12)で縫合し、ホームケージに戻します。
- ホームケージを加熱パッドに置き、マウスが麻酔から完全に回復するまでマウスの体温を維持します。
- 動物が動き始めたら、加熱パッドをオフにしてください。マウスをハウジング施設に移す前に、室温(RT)にさらに2時間順応させます。
- 22°Cで12時間のライト/12時間のダークサイクルでハツネズミを2〜3週間餌と水に自由にアクセスできるようにして、ウイルスの発現を可能にします。
- ファイバー測光
- マウスに埋め込まれた光ファイバーを光ケーブルに接続します。
- 光ケーブルのもう一方の端を、イメージングデータのキャプチャに使用する検出器に接続します。
- イメージングには、光源を使用して488nmの波長でタンパク質を励起します。490 nmから540 nmの範囲の波長で放出された光を収集するように検出器を設定します。
- 励起光の強度を調整して、脳組織に損傷を与えることなく信号を最適化します。
- 蛍光シグナルのゲインを変更して、検出器がクリアで強いシグナルを捕捉できるようにします。
- イメージングの品質を低下させる蛍光の急速な漂白を避けるために、必要に応じて光強度と蛍光シグナルのゲインを微調整します。
- マウスの動作テストを実施します。
- 動作テスト中のイメージングプロセスを継続的に監視します。
- データ分析
- PythonとGNUplotを使用して、データを視覚的に表現するための実験的試行のヒートマップを作成します。ソースコードは 補足ファイル1に記載されています。
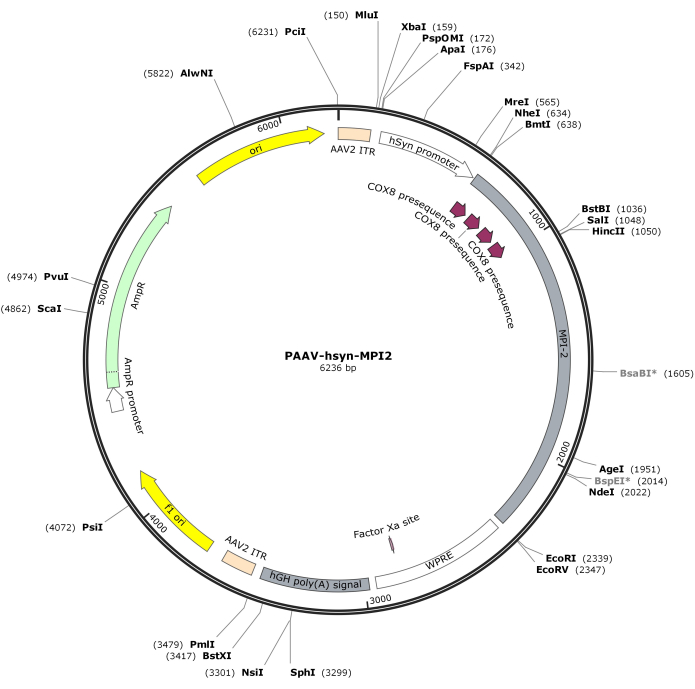
図2:AAV-hSyn-MPI-2のベクトルマップ。この図の拡大版を表示するには、ここをクリックしてください。
結果
CMV-MPI-1プラスミドを構築した後、ミトコンドリアを標的とするその能力を、染色用のミトコンドリアマーカーRhodamine 800を使用してHela細胞でテストしました。共局在実験では、MPI-1の蛍光シグナルとローダミン800からのシグナルとの間には高い重なりが見られ、MPI-1がミトコンドリアに首尾よく局在していることが示されました(図3)。
ディスカッション
ミトコンドリア膜の電圧は、休止条件下では-120-180mVに維持され、代謝状態の変化に伴って変動します。現在、ミトコンドリア膜電位の測定は、電気生理学的方法や蛍光色素法を用いて行うことができます。ミトコンドリアパッチのクランプには、ミトコンドリアの単離と細胞構造の破壊が必要です13。このアプローチでは、生理学的条件から逸脱し...
開示事項
著者は何も開示していません。
謝辞
中国国家自然科学基金会(NSF)のJSK(32071137および92054103)の支援と、鄭州大学第一付属病院の科学研究イノベーションチームへの資金提供(ZYCXTD2023014)に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
参考文献
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -. Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431 (2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977 (2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911 (2018).
- Yang, R. -. Z., Wang, D. -. D., Li, S. -. M., Liu, P. -. P., Kang, J. -. S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347 (2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -. F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541 (2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678 (2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved