Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung des mitochondrialen Membranpotentials in vivo mit Hilfe eines genetisch kodierten Spannungsindikators
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Anwendung von mitochondriengesteuerten genetisch kodierten Spannungsindikatoren (GEVIs). Diese GEVIs bieten einen signifikanten Vorteil gegenüber herkömmlichen mitochondrialen Membranpotentialfarbstoffen, indem sie eine spezifische In-vivo- und Echtzeitüberwachung des mitochondrialen Membranpotenzials ermöglichen.
Zusammenfassung
Das mitochondriale Membranpotential (MMP, ΔΨm) ist entscheidend für die mitochondrialen Funktionen, einschließlich der ATP-Synthese, des Ionentransports, der Erzeugung reaktiver Sauerstoffspezies (ROS) und des Imports von Proteinen, die vom Zellkern kodiert werden. Bestehende Methoden zur Messung von ΔΨm verwenden typischerweise lipophile Kationenfarbstoffe wie Rhodamin 800 und Tetramethylrhodaminmethylester (TMRM), aber diese sind durch eine geringe Spezifität begrenzt und eignen sich nicht gut für In-vivo-Anwendungen . Um diese Einschränkungen zu beheben, haben wir ein neuartiges Protokoll entwickelt, das genetisch kodierte Spannungsindikatoren (GEVIs) verwendet. Genetisch kodierte Spannungsindikatoren (GEVIs), die Fluoreszenzsignale als Reaktion auf Membranpotentialänderungen erzeugen, haben ein erhebliches Potenzial für die Überwachung von Plasmamembran- und neuronalen Potenzialen gezeigt. Ihre Anwendung auf mitochondriale Membranen ist jedoch noch unerforscht. Hier haben wir proteinbasierte mitochondriale GEVIs entwickelt, die in der Lage sind, ΔΨm-Fluktuationen in Zellen und im motorischen Kortex lebender Tiere zu erkennen. Der mitochondriale Potentialindikator (MPI) bietet einen nicht-invasiven Ansatz zur Untersuchung der ΔΨm-Dynamik in Echtzeit und bietet eine Methode zur Untersuchung der mitochondrialen Funktion sowohl unter normalen als auch unter pathologischen Bedingungen.
Einleitung
Mitochondrien sind essentielle Organellen in eukaryotischen Zellen, die durch die Bildung von Adenosintriphosphat (ATP) als primäre Energielieferanten dienen und gleichzeitig eine Vielzahl anderer wichtiger Funktionen erfüllen, wie z. B. die Metabolitensynthese, die Pufferung von Kalziumionen, die Wärmeproduktion und die Regulierung des Zellüberlebens1. Ihre Rolle ist besonders wichtig in stark metabolischen Geweben wie Gehirn und Herz, wo sie zur Aufrechterhaltung der zellulären Homöostase beitragen. Das mitochondriale Membranpotential (MMP, Ψm) ist für diese Prozesse von zentraler Bedeutung, einschließlich der Förderung der ATP-Synthese durch oxidative Phosphorylierung, der Erleichterung des Transports von Metaboliten und Ionen durch die Mitochondrienmembranen und des Beitrags zur Bildung reaktiver Sauerstoffspezies (ROS)2,3. MMP beeinflusst auch die mitochondriale Morphologie und Dynamik4, einschließlich der Mitophagie (dem selektiven Abbau von Mitochondrien)5und der Apoptose (programmierter Zelltod)6. Die Aufrechterhaltung eines angemessenen Ψm ist für die Zellfunktion unerlässlich; Seine Dysregulation ist mit zahlreichen Krankheiten verbunden, darunter neurodegenerative Erkrankungen, Herzinsuffizienz und Krebs. Derzeitige Methoden zur Messung von Ψm beruhten in erster Linie auf der Verwendung lipophiler kationischer Farbstoffe, einschließlich TMRM (Tetramethylrhodaminmethylester), TMRE (Tetramethylrhodaminethylester), Rhodamin 123, Safranin O, Rhodamin 800, DiOC6, JC-1 usw.7. Diese fluoreszierenden Moleküle haben jedoch mehrere Einschränkungen. Diesen Farbstoffen mangelt es an Zellspezifität, sie sind anfällig für Quenching und einige sind giftig. Darüber hinaus können sie im Laufe der Zeit diffundieren, und wenn mitochondriales ΔΨ verloren geht, treten sie aus, wodurch sie nicht mehr in der Lage sind, das Membranpotenzial depolarisierter Mitochondrien anzuzeigen. Darüber hinaus sind Rhodamin-basierte Farbstoffe wie TMRM und TMRE temperaturempfindlich8, was eine sorgfältige Berücksichtigung der Temperatureffekte auf die Farbstofffluoreszenz erfordert, insbesondere bei der Messung der mitochondrialen Membranspannung während physiologischer Aktivitäten, die die zelluläre Thermogenese betreffen.
Genetisch kodierte Spannungsindikatoren (GEVIs), Proteine, die in der Lage sind, Membranpotentialänderungen durch Fluoreszenzsignale zu erkennen 9,10, haben sich als leistungsfähige Werkzeuge zur Überwachung von Membranpotentialen in einer Vielzahl von zellulären Kontexten erwiesen11. Während GEVIs in großem Umfang zur Untersuchung von Plasmamembranen eingesetzt wurden, gab es nur geringe Fortschritte bei der Anpassung an sie zur Messung intrazellulärer Membranpotentiale, insbesondere für Mitochondrien. Dieses Protokoll versucht, diese Lücke zu schließen, indem mitochondriale zielgerichtete GEVIs verwendet werden, die das mitochondriale Membranpotenzial in vitro und in vivo überwachen können. Durch Hinzufügen einer mitochondrialen Signalsequenz zu den vorhandenen GEVIs kann eine geeignete GEVI auf die Mitochondrienausgerichtet werden 12. Diese mitochondrialen Potentialindikatoren (MPI) würden neue Einblicke in die mitochondriale Physiologie liefern und ein erhebliches Potenzial für die Erforschung der mitochondrialen Funktion in verschiedenen Krankheitszuständen in vivo bieten, um unser Verständnis darüber zu verbessern, wie die mitochondriale Dynamik sowohl zu normalen als auch zu pathologischen zellulären Prozessen beiträgt.
Protokoll
Alle Tierpflegemaßnahmen und -versuche wurden in Übereinstimmung mit den Richtlinien des Institutional Animal Care and Use Committee der Universität Zhengzhou durchgeführt. Sterilisieren Sie alle chirurgischen Instrumente vor dem Gebrauch. Befolgen Sie aseptische Techniken, um Infektionen zu verhindern. Nachdem alle Daten erfasst worden waren, wurden die Tiere mit einer Überdosis Inhalationsanästhesie euthanasiert, gefolgt von einer Enthauptung.
1. In-vitro-Anwendungen
- Aufbau von Plasmiden
- Den beschleunigten Sensor der Aktionspotentiale 1 (ASAP1) und ASAP3-Gene erhalten Sie aus Addgene oder synthetisieren aus der Sequenz. (ASAP1, NCBI-Beitritts-ID: AHV90412.1, Addgene-ID: 52519; ASAP3, Addgene-ID: 132331).
- Verwenden Sie die in Tabelle 1 aufgeführten Primer für Plasmidkonstruktionen.
- Für die Expression in Zellen verwenden Sie den EGFPN1-Vektor für die Konstruktion.
- Zunächst wird die cox8-Gensequenz (NP_004065, Aminosäuren 1-29) mit vier Tandem-Repeats (4cox8) synthetisiert, die von NheI- und XhoI-Restriktionsstellen flankiert werden. Verdau sowohl des synthetisierten 4cox8-Fragments als auch des EGFPN1-Vektors mit NheI und XhoI, gefolgt von einer Ligation zur Erzeugung des 4cox8-EGFP-Vektors.
- Führen Sie anschließend eine PCR-Amplifikation der ASAP1-Sequenz mit Primer 1 und Primer 2 durch. Verdau des ASAP1-Fragments und des 4cox8-EGFP-Vektors mit SalI und NotI, gefolgt von einer Ligation, um den endgültigen MPI-1-Vektor zu generieren (MPI steht für mitochondrial potential indicator).
HINWEIS: Die Vektorkarte von CMV-MPI-1 ist in Abbildung 1 dargestellt. Die detaillierte Sequenz kann über NCBI (Beitritts-ID: PQ678920) abgerufen werden.
- Für eine spezifische Expression in Neuronen führen Sie eine PCR-Amplifikation der hSyn-Promotorsequenz aus dem AAV-hSyn-EGFP-Plasmid (NCBI Accession ID: MH458079, Addgene ID: 50465) unter Verwendung von Primer 3 und Primer 4 durch.
- Verdau sowohl der hSyn-Sequenz als auch des 4cox8-EGFP-Vektors mit AseI und NheI, gefolgt von einer Ligation zur Erzeugung des hSyn-4cox8-EGFP-Vektors. Führen Sie anschließend eine PCR-Amplifikation der ASAP3-Sequenz mit Primer 1 und Primer 2 durch.
- Verdau des hSyn-4cox8-EGFP-Vektors und der ASAP3-Sequenz mit SalI und NotI, gefolgt von einer Ligation zur Generierung des hSyn-MPI-2-Vektors.
HINWEIS: Die PCR-Bedingungen variieren je nach den spezifischen Enzymen und den Schmelztemperaturen des Primers (Tm). Eine typische PCR-Reaktion umfasst einen ersten Denaturierungsschritt bei 95 °C für 30 s, gefolgt von 25-35 Zyklen der Denaturierung (95 °C, 10 s), des Glühens (55 °C, 30 s) und der Verlängerung (72 °C, 30 s/kb). Ein letzter Verlängerungsschritt bei 72 °C für 5 min sorgt für eine vollständige Produktsynthese, gefolgt von einem Halten bei 4 °C. Die Glühtemperatur sollte ca. 5 °C unter der Tm der Grundierungen liegen. Beziehen Sie sich auf die Anweisungen des Herstellers für optimale PCR-Parameter. Die Primer für ASAP1 und ASAP3 sind identisch.
- Bildgebung in lebenden Zellen
- Bereiten Sie das DMEM-Kulturmedium vor, indem Sie dem Medium 10 % fötales Rinderserum und 100 mg/l Penicillin/Streptomycin zusetzen.
- Bereiten Sie Ca2+ Transfektionsreagenzien vor: 2,5 mol/L CaCl2, 2x HEBS (274 mmol/L NaCl, 10 mmol/L KCl, 1,4 mmol/L Na2HPO4, 15 mmol/L D-Glucose, 42 mmol/L HEPES, pH 7,07).
- Bereiten Sie den Tyrode-Puffer für die Erhaltung der Zellen während der Bildgebung vor (145 mmol/l NaCl, 3 mmol/l KCl, 10 mmol/l HEPES, 10 mmol/l Glukose, pH 7,4).
- Kultivieren Sie Hela-Zellen in einer 35-mm-Kulturschale, die mit DMEM-Kulturmedium in einem Inkubator mit einer Temperatur von 37 °C und einer Atmosphäre von 5 % CO2/95 % Luft gefüttert wird. Fügen Sie 2-3 Deckgläser pro Schale hinzu, damit die Zellen darauf wachsen können.
- Transfizieren Sie die Hela-Zellen mit der Calciumphosphat-Fällungsmethode.
- Um Zellen zu transfizieren, ersetzen Sie zunächst das alte Kulturmedium durch 2 ml serumfreies DMEM. Bereiten Sie nach 35 Minuten die DNA/Ca2+ -Lösung vor, indem Sie 10 μg DNA, 4 μl CaCl2 und ddH2O in einem Mikrozentrifugenröhrchen zu einem Endvolumen von 40 μl mischen.
- Geben Sie 40 μL 2x HEBS tropfenweise unter kräftigem Rühren in die DNA/Ca2+ Lösung. Blasen Sie Luft mit einer Pipette in die Mischung, um die Calciumphosphatfällung zu erleichtern.
- Nach 25 min geben Sie 80 μl dieser Fällungslösung in die Schale mit den Zellen und stellen Sie sie für weitere 1-2 h in den Inkubator. Entfernen Sie dann das Nicht-Serum-DMEM und spülen Sie die Zellen 2 Minuten lang mit 15% Glycerin (gelöst in PBS).
- Nach dem Aspirieren des Glycerins fügen Sie 2 ml DMEM mit 10% FBS hinzu und kultivieren Sie die Zellen für 16-24 Stunden.
- Legen Sie Deckgläser mit Zellen in eine 35-mm-Kulturschale mit 2 mL Tyrode-Puffer und färben Sie die Zellen mit Rhodamin-800-Farbstoff, indem Sie 20 μmol/L Rhodamin 800 (gelöst in ddH2O) bis zu einer Endkonzentration von 50 nmol/l in die Schale geben.
- Bereiten Sie ein Fluoreszenzmikroskop vor und richten Sie es mit den entsprechenden Filtern und der Lichtquelle ein, um das Protein mit einer 488-nm-Lichtquelle anzuregen und die Emission zwischen 490-540 nm zu sammeln. Stimulieren Sie den Farbstoff Rhodamin 800 mit einer 633 nm Lichtquelle und sammeln Sie die Emission zwischen 650-720 nm.
- Bereiten Sie eine Carbonylcyanid-m-Chlorphenylhydrazon (CCCP)-Lösung bei 500 μM (Stamm) vor und geben Sie sie während der Bildgebung bis zu einer Endkonzentration von 5 μM in die Zellen.
HINWEIS: Der pH-Wert der 2x HEBS-Lösung ist entscheidend für die Wirksamkeit der Transfektion. Der pH-Wert sollte vorsichtig mit festem Natriumhydroxid auf 7,07 eingestellt werden. Der pH-Wert darf nicht niedriger als 7,01 oder höher als 7,12 sein.
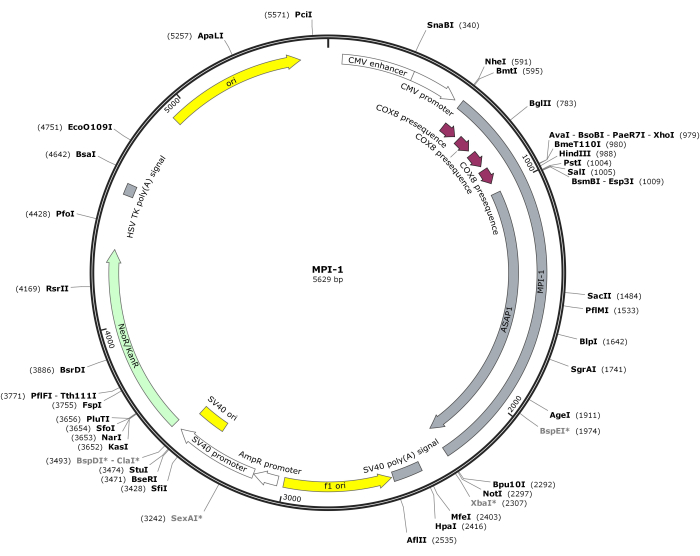
Abbildung 1: Vektorkarte von CMV-MPI-1. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. In-vivo-Anwendungen
- Plasmid-Konstrukt
- Für die In-vivo-Expression verwenden Sie das AAV-Virus für die Transduktion. Besorgen Sie sich das exprimierende Rückgrat für die AAV-Verpackung (Addgene-ID: 46954 oder 26968), das ITR-Sequenzen (Inverted Terminal Repeats) für die AAV-Virusverpackung enthält.
- Führen Sie die PCR-Amplifikation des hSyn-MPI-2-Vektors (aus Schritt 1.1.4) mit den Primern 5 und 7 in Tabelle 1 durch.
- Führen Sie eine zweite PCR-Amplifikation mit demselben Template und denselben Primern 6 und 8 in Tabelle 1 durch.
- Führen Sie eine abschließende PCR durch, bei der das Produkt aus der ersten und der zweiten PCR (Schritte 2.1.2 und 2.1.3) als Template und die Primer 5 und 8 in Tabelle 1 verwendet werden, wobei die endgültige hSyn-MPI-2-Sequenz generiert wird.
- Verdauen Sie die hSyn-MPI-2-Sequenz und das pAAV-Rückgrat mit MluI und EcoRI, gefolgt von einer Ligation, um das endgültige pAAV-hSyn-MPI-2-Konstrukt zu generieren. Die Vektorkarte von pAAV-hSyn-MPI-2 ist in Abbildung 2 dargestellt. Die detaillierte Sequenz kann über NCBI (Beitritts-ID: PQ678919) abgerufen werden.
HINWEIS: Der hSyn-MPI-2-Vektor enthält eine interne EcoRI-Stelle, die vom ursprünglichen EGFPN1-Rückgrat stammt und mit modifizierten Primern in einer überlappenden PCR-Strategie entfernt wird (Schritte 2.1.2-2.1.4).
- Viruspräparation und -transduktion
- Kultivieren Sie HEK293t-Zellen in einer 100-mm-Kulturschale, die mit 10 % FBS DMEM-Kulturmedium in einem Inkubator mit einer Temperatur von 37 °C und einer Atmosphäre von 5 % CO2 /95 % Luft gefüttert wurde, um eine Konfluenz von 70 % zu erreichen.
- Co-transfiziert die HEK293t-Zellen mit AAV-Genplasmid, Kapsid (pAAV-DJ oder pAAV9) und Helferplasmiden (pHelper) unter Verwendung der Calciumphosphat-Fällungsmethode. Die DNA-Menge für jedes Plasmid war wie folgt: Gen: 10 μg, pHelper: 11 μg, Kapsid: 9 μg. Die Menge des Transfektionsreagenzes: 12 μl CaCl2, 120 μl 2x HEBS.
- Inkubieren Sie die transfizierten Zellen für einen Zeitraum von 72 Stunden, um die Virusproduktion zu ermöglichen.
- Nach der Inkubationszeit werden die Zellen mit einer Zentrifuge von 400 g geerntet. Spülen Sie die Zellen 2 Mal mit PBS und resuspendieren Sie die Zelle in 400 μl PBS.
- Ernten Sie Viren aus Zellsuspensionen mit vier Zyklen von Gefrier-Auftau-Methoden (37 °C und -80 °C). Das Lysat wird bei 13.500 × g bei 4 °C zentrifugiert und das Pellet verworfen. Der Überstand enthält das AAV-Virus.
- Führen Sie eine Echtzeit-PCR durch, um die Titration des vorbereiteten Virus zu bestimmen. Ein detailliertes Protokoll zur Bestimmung der AAV-Titration finden Sie unter https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/.
- Aliquotieren und lagern Sie das vorbereitete AAV-Virus bei -80 °C.
- Stereotaktische Virusinjektion
- Anästhesieren Sie eine erwachsene männliche C57/BL-Maus mit einem Gewicht von 24-26 g mit 3% Pentobarbital-Natrium durch intraperitoneale Injektion in einer Dosierung von 30 mg/kg. Befestigen Sie die Mäuse nach der Betäubung auf einem stereotaktischen Gerät, um während des gesamten Eingriffs eine stabile Position beizubehalten.
- Tragen Sie veterinärmedizinische Augensalbe auf, um die Augen vor Lichtschäden zu schützen und die Augen feucht zu halten.
- Verwenden Sie Enthaarungscreme, um Haare auf dem Kopf der Maus zu entfernen und die Kopfhaut freizulegen.
- Öffnen Sie die Kopfhaut mit einer Operationsschere und legen Sie den Schädel frei. Behandeln Sie den Schädel mit 3 % Wasserstoffperoxid, um wichtige Orientierungspunkte wie die Bregma- und Lambda-Stellen zu sterilisieren und sichtbar zu machen.
- Passen Sie die Position des Mauskopfes an, um sicherzustellen, dass der Schädel horizontal ist.
- Verwenden Sie einen programmierbaren Mikropipettenabzieher, um Glaspipetten in die gewünschte Form und Größe zu ziehen, um präzise Injektionen zu ermöglichen. Füllen Sie die Glasmikropipette mit Paraffinöl.
- Positionieren Sie die Mikropipette an der Zielstelle. In diesem Protokoll ist das Ziel die M2-Region auf der rechten Seite unter Verwendung der folgenden Koordinaten: AP (anterior-posterior), +1,94 mm; ML (mediolateral), +0,75 mm; DV (dorsoventral), -1,5 mm.
- Verwende eine elektrische Bohrmaschine, um an der Zielstelle ein kleines Loch in den Schädel zu bohren.
- Aspirieren Sie 500 nL Virus bei einem Titer von 1,5 x 1011 Genomkopien pro Milliliter (GC/ml) durch die Spitze der Mikropipette. Injizieren Sie 500 nL Virus mit einer Injektionspumpe mit einer Durchflussrate von 100 nL/min in die Zielstelle.
- Lassen Sie die Pipette nach der Injektion ca. 5 Minuten lang an Ort und Stelle, um eine Diffusion zu ermöglichen, bevor Sie sie vorsichtig zurückziehen.
- Implantieren Sie eine optische Faser (200 μm Durchmesser, 0,37 numerische Apertur, 2 mm Länge) in die Injektionsstelle.
- Befestigen Sie die optische Faser mit Zahnzement.
- Vernähen Sie die Kopfhaut der Maus mit 5-0 Nylonfaden (ästhetische Nadel Δ1/2 4x12) und bringen Sie sie in ihren Heimatkäfig zurück.
- Stellen Sie den Heimkäfig auf ein Heizkissen, um die Körpertemperatur der Maus zu halten, bis sie sich vollständig von der Narkose erholt hat.
- Schalten Sie das Heizkissen aus, sobald sich das Tier zu bewegen beginnt. Warten Sie weitere 2 Stunden Eingewöhnung an die Raumtemperatur (RT), bevor Sie die Maus in die Haltungseinrichtung bringen.
- Hausmaus in einem 12-stündigen Licht-/12-stündigen Dunkelzyklus bei 22 °C mit ad libitum Zugang zu Nahrung und Wasser für 2-3 Wochen, um die Virusexpression zu ermöglichen.
- Faser-Photometrie
- Verbinden Sie die in der Maus implantierte Glasfaser mit einem Glasfaserkabel.
- Schließen Sie das andere Ende des optischen Kabels an einen Detektor an, der zur Erfassung der Bilddaten verwendet wird.
- Verwenden Sie für die Bildgebung eine Lichtquelle, um die Proteine bei einer Wellenlänge von 488 nm anzuregen. Richten Sie den Detektor so ein, dass er das emittierte Licht bei Wellenlängen von 490 nm bis 540 nm sammelt.
- Passen Sie die Intensität des Anregungslichts an, um das Signal zu optimieren, ohne das Hirngewebe zu schädigen.
- Ändern Sie die Verstärkung des Fluoreszenzsignals, um sicherzustellen, dass ein klares und starkes Signal vom Detektor erfasst wird.
- Um ein schnelles Ausbleichen der Fluoreszenz zu vermeiden, das die Qualität der Bildgebung beeinträchtigen würde, sollten die Lichtintensität und die Verstärkung des Fluoreszenzsignals nach Bedarf fein abgestimmt werden.
- Führen Sie einen Test des Mausverhaltens durch.
- Überwachen Sie den Bildgebungsprozess während des Verhaltenstests kontinuierlich.
- Datenanalyse
- Verwenden Sie Python und GNUplot, um eine Heatmap von experimentellen Versuchen für eine visuelle Darstellung der Daten zu erstellen. Die Quellcodes sind in der Zusatzdatei 1 enthalten.
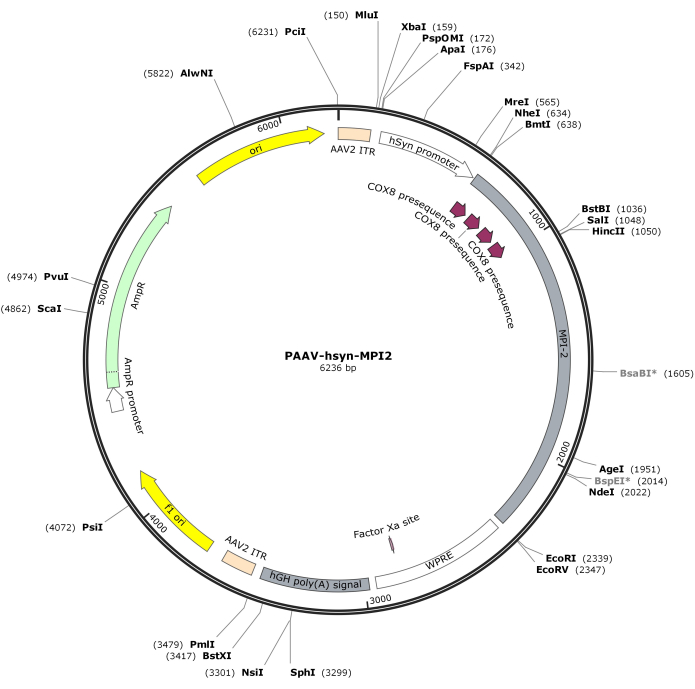
Abbildung 2: Vektorkarte von AAV-hSyn-MPI-2. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Nach der Konstruktion des CMV-MPI-1-Plasmids wurde seine Fähigkeit, auf Mitochondrien abzuzielen, in Hela-Zellen unter Verwendung des mitochondrialen Markers Rhodamin 800 zur Färbung getestet. Kolokalisationsexperimente zeigten ein hohes Maß an Überlappung zwischen dem Fluoreszenzsignal von MPI-1 und dem Signal von Rhodamin 800, was darauf hindeutet, dass MPI-1 erfolgreich in den Mitochondrien lokalisiert wurde (Abbildung 3).
Diskussion
Die mitochondriale Membranspannung wird unter Ruhebedingungen bei -120--180 mV gehalten und schwankt mit Änderungen des Stoffwechselzustands. Derzeit kann die Messung des mitochondrialen Membranpotentials mit elektrophysiologischen Methoden und Fluoreszenzfarbstoffmethoden durchgeführt werden. Die mitochondriale Patch-Klemmung erfordert die Isolierung der Mitochondrien und die Zerstörung zellulärer Strukturen13. Dieser Ansatz kann zu Messungen führen, die von...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken der National Natural Science Foundation (NSF) Chinas: JSK (32071137 und 92054103) und der Finanzierung des wissenschaftlichen Forschungs- und Innovationsteams des ersten angeschlossenen Krankenhauses der Universität Zhengzhou: JSK (ZYCXTD2023014).
Materialien
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
Referenzen
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -. Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431 (2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977 (2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911 (2018).
- Yang, R. -. Z., Wang, D. -. D., Li, S. -. M., Liu, P. -. P., Kang, J. -. S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347 (2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -. F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541 (2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678 (2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten