É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medição do Potencial de Membrana Mitocondrial In Vivo usando um Indicador de Voltagem Geneticamente Codificado
Neste Artigo
Resumo
Este protocolo descreve a aplicação de indicadores de voltagem geneticamente codificados (GEVIs) direcionados às mitocôndrias. Esses GEVIs oferecem uma vantagem significativa sobre os corantes tradicionais de potencial de membrana mitocondrial, permitindo o monitoramento específico, in vivo e em tempo real do potencial de membrana mitocondrial.
Resumo
O potencial de membrana mitocondrial (MMP, ΔΨm) é crítico para as funções mitocondriais, incluindo síntese de ATP, transporte de íons, geração de espécies reativas de oxigênio (ROS) e importação de proteínas codificadas pelo núcleo. Os métodos existentes para medir ΔΨm normalmente usam corantes catiônicos lipofílicos, como rodamina 800 e éster metílico de tetrametilrodamina (TMRM), mas estes são limitados pela baixa especificidade e não são adequados para aplicações in vivo . Para resolver essas limitações, desenvolvemos um novo protocolo utilizando indicadores de tensão geneticamente codificados (GEVIs). Indicadores de voltagem geneticamente codificados (GEVIs), que geram sinais fluorescentes em resposta a mudanças no potencial da membrana, demonstraram um potencial significativo para monitorar a membrana plasmática e os potenciais neuronais. No entanto, sua aplicação às membranas mitocondriais permanece inexplorada. Aqui, desenvolvemos GEVIs direcionados a mitocôndrias baseados em proteínas, capazes de detectar flutuações de ΔΨm nas células e no córtex motor de animais vivos. O indicador de potencial mitocondrial (MPI) oferece uma abordagem não invasiva para estudar a dinâmica do ΔΨm em tempo real, fornecendo um método para investigar a função mitocondrial em condições normais e patológicas.
Introdução
As mitocôndrias são organelas essenciais nas células eucarióticas, servindo como fornecedoras primárias de energia por meio da geração de trifosfato de adenosina (ATP), ao mesmo tempo em que desempenham uma variedade de outras funções cruciais, como síntese de metabólitos, tamponamento de íons de cálcio, produção de calor e regulação da sobrevivência celular1. Seus papéis são particularmente críticos em tecidos altamente metabólicos, como o cérebro e o coração, onde ajudam a manter a homeostase celular. O potencial de membrana mitocondrial (MMP, Ψm) é central para esses processos, incluindo a condução da síntese de ATP via fosforilação oxidativa, facilitando o transporte de metabólitos e íons através das membranas mitocondriais e contribuindo para a geração de espécies reativas de oxigênio (ROS) 2 , 3 . A MMP também influencia a morfologia e a dinâmica mitocondrial4, incluindo a mitofagia (a degradação seletiva das mitocôndrias)5e a apoptose (morte celular programada)6. Manter um Ψm apropriado é essencial para a função celular; Sua desregulação está ligada a inúmeras patologias, incluindo doenças neurodegenerativas, insuficiência cardíaca e câncer. Os métodos atuais para medir Ψm foram baseados principalmente no uso de corantes catiônicos lipofílicos, incluindo TMRM (éster metílico de tetrametilrodamina), TMRE (éster etílico de tetrametilrodamina), Rodamina 123, Safranina O, Rodamina 800, DiOC6, JC-1, etc.7. No entanto, essas moléculas fluorescentes têm várias limitações. Esses corantes carecem de especificidade celular, são suscetíveis à extinção e alguns são tóxicos. Além disso, eles podem se difundir com o tempo e, quando o ΔΨ mitocondrial é perdido, eles vazam, tornando-os incapazes de indicar o potencial de membrana das mitocôndrias despolarizadas. Além disso, corantes à base de rodamina como TMRM e TMRE são sensíveis à temperatura8, necessitando de consideração cuidadosa dos efeitos da temperatura na fluorescência do corante, particularmente ao medir a voltagem da membrana mitocondrial durante atividades fisiológicas envolvendo termogênese celular.
Indicadores de voltagem geneticamente codificados (GEVIs), proteínas capazes de detectar alterações no potencial da membrana por meio de sinais fluorescentes 9,10, surgiram como ferramentas poderosas para monitorar potenciais de membrana em uma variedade de contextos celulares11. Embora os GEVIs tenham sido aplicados extensivamente para estudar as membranas plasmáticas, houve pouco progresso em adaptá-los para medir os potenciais da membrana intracelular, particularmente para as mitocôndrias. Este protocolo procura preencher essa lacuna usando GEVIs direcionados a mitocôndrias que podem monitorar o potencial da membrana mitocondrial in vitro e in vivo. Ao adicionar a sequência de sinal mitocondrial aos GEVIs existentes, os GEVI apropriados podem ser direcionados para as mitocôndrias12. Esses indicadores de potencial mitocondrial (MPI) forneceriam novos insights sobre a fisiologia mitocondrial e ofereceriam um potencial significativo para explorar a função mitocondrial em vários estados de doença in vivo, aprimorando nossa compreensão de como a dinâmica mitocondrial contribui para processos celulares normais e patológicos.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os cuidados e experimentos com animais foram realizados de acordo com as diretrizes do Comitê Institucional de Cuidados e Uso de Animais da Universidade de Zhengzhou. Esterilize todos os instrumentos cirúrgicos antes de usar. Siga técnicas assépticas para prevenir infecções. Após a aquisição de todos os dados, os animais foram eutanasiados com overdose de anestesia inalatória seguida de decapitação.
1. Aplicações in vitro
- Construção de plasmídeo
- Obtenha o sensor acelerado dos genes dos potenciais de ação 1 (ASAP1) e ASAP3 de Addgene ou sintetizando a partir da sequência. (ASAP1, ID de acesso do NCBI: AHV90412.1, ID Addgene: 52519; ASAP3, ID Addgene: 132331).
- Use os primers listados na Tabela 1 para construções de plasmídeo.
- Para expressão em células, use o vetor EGFPN1 para construção.
- Inicialmente, sintetizar a sequência do gene cox8 (NP_004065, aminoácidos 1-29) com quatro repetições em tandem (4cox8) ladeadas por sítios de restrição NheI e XhoI. Digerir o fragmento 4cox8 sintetizado e o vetor EGFPN1 com NheI e XhoI, seguido de ligação para gerar o vetor 4cox8-EGFP.
- Posteriormente, realizar a amplificação por PCR da sequência ASAP1 com primer 1 e primer 2. Digerir o fragmento ASAP1 e o vetor 4cox8-EGFP com SalI e NotI, seguido de ligação para gerar o vetor MPI-1 final (MPI significa indicador de potencial mitocondrial).
NOTA: O mapa vetorial do CMV-MPI-1 é representado na Figura 1. A sequência detalhada pode ser acessada no NCBI (ID de acesso: PQ678920).
- Para expressão específica em neurônios, execute a amplificação por PCR da sequência do promotor hSyn do plasmídeo AAV-hSyn-EGFP (NCBI Accession ID: MH458079, Addgene ID: 50465) usando o primer 3 e o primer 4.
- Digerir a sequência hSyn e o vetor 4cox8-EGFP com AseI e NheI, seguido de ligação para gerar o vetor hSyn-4cox8-EGFP. Posteriormente, realizar amplificação por PCR da sequência ASAP3 com primer 1 e primer 2.
- Digerir o vetor hSyn-4cox8-EGFP e a sequência ASAP3 com SalI e NotI, seguido de ligação para gerar o vetor hSyn-MPI-2.
NOTA: As condições de PCR variam de acordo com as enzimas específicas e as temperaturas de fusão do primer (Tm). Uma reação de PCR típica envolve uma etapa inicial de desnaturação a 95 ° C por 30 s, seguida por 25-35 ciclos de desnaturação (95 ° C, 10 s), recozimento (55 ° C, 30 s) e extensão (72 ° C, 30 s / kb). Uma etapa final de extensão a 72 °C por 5 min garante a síntese completa do produto, seguida por uma retenção a 4 °C. A temperatura de recozimento deve ser aproximadamente 5 °C inferior ao Tm dos primers. Consulte as instruções do fabricante para obter os parâmetros ideais de PCR. Os primers para ASAP1 e ASAP3 são idênticos.
- Aquisição de imagens em células vivas
- Prepare o meio de cultura DMEM adicionando 10% de soro fetal bovino e 100 mg/L de penicilina/estreptomicina ao meio.
- Prepare reagentes de transfecção de Ca2+ : 2,5 mol/L CaCl2, 2x HEBS (274 mmol/L NaCl, 10 mmol/L KCl, 1,4 mmol/L Na2HPO4, 15 mmol/L D-glicose, 42 mmol/L HEPES, pH 7,07).
- Prepare o tampão de Tyrode para manter as células durante a imagem (145 mmol / L NaCl, 3 mmol / L KCl, 10 mmol / L HEPES, 10 mmol / L Glicose, pH 7,4).
- Cultivar células Hela numa placa de cultura de 35 mm alimentada com meio de cultura DMEM numa incubadora com uma temperatura de 37 °C e uma atmosfera de 5% de CO2/95% de ar. Adicione 2-3 lamínulas por prato para permitir que as células cresçam nelas.
- Transfectar as células Hela através do método de precipitação de fosfato de cálcio.
- Para transfectar as células, comece substituindo o meio de cultura antigo por 2 mL de DMEM sem soro. Após 35 min, prepare a solução de DNA/Ca2+ misturando 10 μg de DNA, 4 μL de CaCl2 e ddH2O até um volume final de 40 μL em um tubo de microcentrífuga.
- Adicione 40 μL de 2x HEBS à solução de DNA / Ca2+ gota a gota, mexendo vigorosamente. Borbulhe ar com uma pipeta na mistura para facilitar a precipitação de fosfato de cálcio.
- Após 25 min, adicione 80 μL desta solução precipitada ao prato com as células e devolva-o à incubadora por mais 1-2 h. Em seguida, remova o DMEM não sérico e enxágue as células com glicerol a 15% (dissolvido em PBS) por 2 min.
- Depois de aspirar o glicerol, adicione 2 mL de DMEM contendo 10% de FBS e continue cultivando as células por 16-24 h.
- Coloque lamínulas com células em uma placa de cultura de 35 mm com 2 mL de tampão de Tyrode e core as células com corante rodamina 800 adicionando 20 μmol / L rodamina 800 (dissolvida em ddH2O) à placa até uma concentração final de 50 nmol / L.
- Preparar um microscópio de fluorescência e configurá-lo com os filtros e a fonte de luz adequados para excitar a proteína com uma fonte de luz de 488 nm e recolher a emissão entre 490-540 nm. Excite o corante rodamina 800 com uma fonte de luz de 633 nm e colete a emissão entre 650-720 nm.
- Preparar uma solução de cianeto de carbonilo m-clorofenil-hidrazona (CCCP) a 500 μM (reserva) e adicionar às células até uma concentração final de 5 μM durante a obtenção de imagens.
NOTA: O pH da solução 2x HEBS é crucial para a eficiência da transfecção. O pH deve ser cuidadosamente ajustado para 7,07 com hidróxido de sódio sólido. O pH não pode ser inferior a 7,01 ou superior a 7,12.
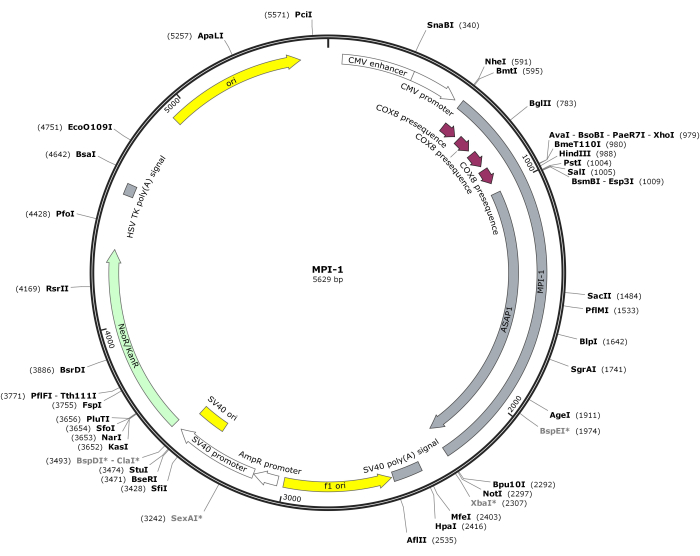
Figura 1: Mapa vetorial do CMV-MPI-1. Clique aqui para ver uma versão maior desta figura.
2. Aplicações in vivo
- Construção de plasmídeo
- Para expressão in vivo , use o vírus AAV para transdução. Obtenha o backbone de expressão para empacotamento de AAV (Addgene ID: 46954 ou 26968), que contém sequências de repetições de terminal invertido (ITR) para empacotamento de vírus AAV.
- Realize a amplificação por PCR do vetor hSyn-MPI-2 (da etapa 1.1.4) usando os primers 5 e 7 na Tabela 1.
- Realize uma segunda amplificação de PCR usando o mesmo modelo e os primers 6 e 8 na Tabela 1.
- Realizar uma PCR final utilizando o produto da primeira e da segunda PCR (passos 2.1.2 e 2.1.3) como molde e os iniciadores 5 e 8 do quadro 1, gerando a sequência final de hSyn-MPI-2.
- Digerir a sequência hSyn-MPI-2 e o backbone pAAV com MluI e EcoRI, seguido de ligação para gerar a construção final pAAV-hSyn-MPI-2. O mapa vetorial de pAAV-hSyn-MPI-2 é ilustrado na Figura 2. A sequência detalhada pode ser acessada no NCBI (ID de acesso: PQ678919).
NOTA: O vetor hSyn-MPI-2 contém um sítio EcoRI interno originado do backbone EGFPN1 original, que é removido usando primers modificados em uma estratégia de PCR sobreposta (etapas 2.1.2-2.1.4).
- Preparação e transdução de vírus
- Cultura de células HEK293t em uma placa de cultura de 100 mm alimentada com meio de cultura FBS DMEM a 10% em uma incubadora com temperatura de 37 °C e atmosfera de 5% de CO2/95% de ar para atingir uma confluência de 70%.
- Co-transfectar as células HEK293t com plasmídeo do gene AAV, capsídeo (pAAV-DJ ou pAAV9) e plasmídeos auxiliares (pHelper) usando o método de precipitação de fosfato de cálcio. A quantidade de DNA para cada plasmídeo foi a seguinte: gene: 10 μg, pHelper: 11 μg, capsídeo: 9 μg. A quantidade de reagente de transfecção: 12 μL de CaCl2, 120 μL de 2x HEBS.
- Incubar as células transfectadas por um período de 72 h para permitir a produção do vírus.
- Após o período de incubação, colher as células por centrifugação a 400 g. Enxágue as células com PBS 2 vezes e ressuspenda a célula em 400 μL de PBS.
- Colher vírus da suspensão celular com quatro ciclos de métodos de congelamento e descongelamento (37 °C e -80 °C). Centrifugar o lisado a 13,500 × g a 4 °C e rejeitar o sedimento. O sobrenadante contém o vírus AAV.
- Realize PCR em tempo real para determinar a titulação do vírus preparado. O protocolo detalhado para a determinação da titulação do AAV pode ser encontrado em https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/.
- Aliquotar e armazenar o vírus AAV preparado a -80 °C.
- Injeção de vírus estereotáxica
- Anestesiar um camundongo macho C57 / BL adulto pesando 24-26 g com pentobarbital sódico a 3% por meio de injeção intraperitoneal na dosagem de 30 mg / kg. Uma vez anestesiados, fixe os camundongos em um aparelho estereotáxico para manter uma posição estável durante todo o procedimento.
- Aplique pomada oftálmica veterinária para proteger os olhos dos danos causados pela luz e para mantê-los úmidos.
- Use creme depilatório para remover os pelos da cabeça do rato e expor o couro cabeludo.
- Abra o couro cabeludo usando uma tesoura cirúrgica e exponha o crânio. Trate o crânio com peróxido de hidrogênio a 3% para esterilizar e visualizar os principais pontos de referência, como os locais bregma e lambda.
- Ajuste a posição da cabeça do mouse para garantir que o crânio esteja na horizontal.
- Use um extrator de micropipeta programável para puxar as pipetas de vidro para a forma e tamanho desejados para injeções precisas. Encha a micropipeta de vidro com óleo de parafina.
- Posicione a micropipeta no local alvo. Neste protocolo, o alvo é a região M2 do lado direito, utilizando as seguintes coordenadas: AP (ântero-posterior), +1,94 mm; ML (mediolateral), +0,75 mm; VD (dorsoventral), -1,5 mm.
- Use uma furadeira elétrica para fazer um pequeno orifício no crânio no local alvo.
- Aspire 500 nL de vírus a um título de 1,5 x 1011 cópias do genoma por mililitro (GC / mL) através da ponta da micropipeta. Injete 500 nL de vírus no local alvo usando uma bomba injetora a uma taxa de fluxo de 100 nL / min.
- Após a injeção, deixe a pipeta no lugar por aproximadamente 5 minutos para permitir a difusão antes de retraí-la cuidadosamente.
- Implante fibra óptica (200 μm de diâmetro, abertura numérica de 0,37, comprimento de 2 mm) no local da injeção.
- Prenda a fibra óptica no lugar usando cimento dental.
- Suturar o couro cabeludo do camundongo com fio de náilon 5-0 (agulha estética Δ1/2 4x12) e devolvê-lo à sua gaiola de origem.
- Coloque a gaiola doméstica em uma almofada de aquecimento para manter a temperatura corporal do mouse até que ele se recupere totalmente da anestesia.
- Desligue a almofada de aquecimento assim que o animal começar a se mover. Deixe mais 2 h de aclimatação à temperatura ambiente (RT) antes de transferir o mouse para a instalação de alojamento.
- Rato doméstico em um ciclo de 12 horas de luz / 12 horas de escuridão a 22 ° C com acesso ad libitum a comida e água por 2-3 semanas para permitir a expressão viral.
- Fotometria de fibra
- Conecte a fibra óptica implantada no mouse a um cabo óptico.
- Conecte a outra extremidade do cabo óptico a um detector que será usado para capturar os dados de imagem.
- Para obter imagens, use uma fonte de luz para excitar as proteínas em um comprimento de onda de 488 nm. Configure o detector para coletar a luz emitida em comprimentos de onda que variam de 490 nm a 540 nm.
- Ajuste a intensidade da luz de excitação para otimizar o sinal sem causar danos ao tecido cerebral.
- Modifique os ganhos do sinal fluorescente para garantir que um sinal claro e forte seja capturado pelo detector.
- Para evitar o branqueamento rápido da fluorescência, o que reduziria a qualidade da imagem, ajuste a intensidade da luz e o ganho do sinal fluorescente conforme necessário.
- Realize o teste de comportamento do mouse.
- Monitore continuamente o processo de imagem durante o teste de comportamento.
- Análise de dados
- Use Python e GNUplot para criar um mapa de calor de testes experimentais para uma representação visual dos dados. Os códigos-fonte são fornecidos no Arquivo Suplementar 1.
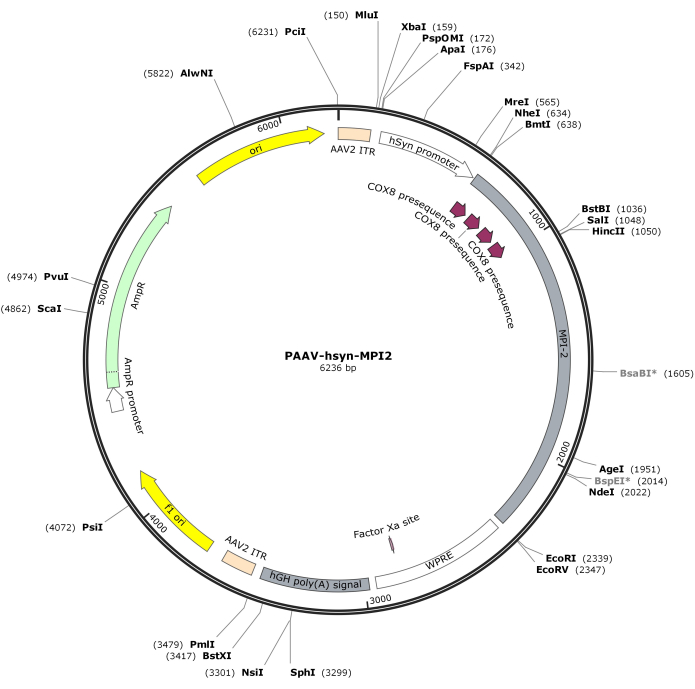
Figura 2: Mapa vetorial de AAV-hSyn-MPI-2. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Após a construção do plasmídeo CMV-MPI-1, sua capacidade de atingir as mitocôndrias foi testada em células Hela usando o marcador mitocondrial Rodamina 800 para coloração. Experimentos de colocalização mostraram um alto grau de sobreposição entre o sinal de fluorescência do MPI-1 e o sinal da Rodamina 800, indicando que o MPI-1 foi localizado com sucesso nas mitocôndrias (Figura 3).
Access restricted. Please log in or start a trial to view this content.
Discussão
A voltagem da membrana mitocondrial é mantida em -120--180 mV em condições de repouso e flutua com as mudanças no estado metabólico. Atualmente, a medição do potencial de membrana mitocondrial pode ser realizada usando métodos eletrofisiológicos e métodos de corante de fluorescência. O clampeamento mitocondrial requer o isolamento das mitocôndrias e a destruição das estruturas celulares13. Essa abordagem pode levar a medições que se desviam das con...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Agradecemos o apoio da Fundação Nacional de Ciências Naturais (NSF) da China: JSK (32071137 e 92054103) e o Financiamento para a Equipe de Pesquisa Científica e Inovação do Primeiro Hospital Afiliado da Universidade de Zhengzhou: JSK (ZYCXTD2023014).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
Referências
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431(2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977(2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911(2018).
- Yang, R. -Z., Wang, D. -D., Li, S. -M., Liu, P. -P., Kang, J. -S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347(2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541(2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678(2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados