JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
유전적으로 인코딩된 전압 표시기를 사용한 생체 내 미토콘드리아 막 전위 측정
요약
이 프로토콜은 미토콘드리아 표적 유전자 인코딩 전압 지표(GEVI)의 적용을 설명합니다. 이러한 GEVI는 미토콘드리아 막 전위의 특이적, 생체 내 및 실시간 모니터링을 가능하게 함으로써 기존 미토콘드리아 막 전위 염료에 비해 상당한 이점을 제공합니다.
초록
미토콘드리아 막 전위(MMP, ΔΨm)는 ATP 합성, 이온 수송, 반응성 산소종(ROS) 생성, 핵에 의해 암호화된 단백질 가져오기를 포함한 미토콘드리아 기능에 매우 중요합니다. ΔΨm 을 측정하기 위한 기존 방법은 일반적으로 로다민 800 및 테트라메틸로다민 메틸 에스테르(TMRM)와 같은 친유성 양이온 염료를 사용하지만, 이는 특이성이 낮아 제한적이며 in vivo 응용 분야에 적합하지 않습니다. 이러한 한계를 해결하기 위해 당사는 유전적으로 인코딩된 전압 표시기(GEVI)를 활용하는 새로운 프로토콜을 개발했습니다. 막 전위 변화에 대한 반응으로 형광 신호를 생성하는 GEVI(Genetically encoded voltage indicator)는 원형질막 및 신경 전위를 모니터링할 수 있는 상당한 잠재력을 보여주었습니다. 그러나 미토콘드리아 막에 대한 적용은 아직 연구되지 않았습니다. 여기에서 우리는 살아있는 동물의 세포와 운동 피질에서 ΔΨm 변동을 감지할 수 있는 단백질 기반 미토콘드리아 표적 GEVI를 개발했습니다. 미토콘드리아 전위 지표(MPI)는ΔΨM 역학을 실시간으로 연구하기 위한 비침습적 접근 방식을 제공하여 정상 및 병리학적 조건 모두에서 미토콘드리아 기능을 조사할 수 있는 방법을 제공합니다.
서문
미토콘드리아는 진핵 세포의 필수 소기관으로, 아데노신 삼인산(ATP) 생성을 통해 주요 에너지 공급자 역할을 하는 동시에 대사 산물 합성, 칼슘 이온 완충, 열 생성 및 세포 생존 조절과 같은 다양한 기타 중요한 기능을 수행합니다1. 이들의 역할은 뇌와 심장과 같은 고도의 신진대사 조직에서 특히 중요하며, 세포 항상성을 유지하는 데 도움이 됩니다. 미토콘드리아 막 전위(MMP, Ψm)는 산화적 인산화를 통한 ATP 합성 촉진, 미토콘드리아 막을 통한 대사 산물 및 이온 수송 촉진, 활성 산소 종(ROS) 생성에 기여하는 등 이러한 과정의 핵심입니다.2,3. MMP는 또한 미토파지(mitophagy, 미토콘드리아의 선택적 분해)5및 세포사멸(apoptosis, 프로그래밍된 세포 사멸)6을 포함한 미토콘드리아 형태 및 역학4에 영향을 미칩니다. 적절한 Ψm을 유지하는 것은 세포 기능에 필수적입니다. 조절 장애는 신경 퇴행성 질환, 심부전 및 암을 포함한 수많은 병리학과 관련이 있습니다. Ψm을 측정하는 현재의 방법은 주로 TMRM (테트라 메틸 로다민 메틸 에스테르), TMRE (테트라 메틸 로다민 에틸 에스테르), 로다 민 123, 사프라닌 O, 로다 민 800, DiOC6, JC-1 등을 포함한 친유 성 양이온 염료의 사용을 기반으로했습니다 7. 그러나 이러한 형광 분자에는 몇 가지 제한 사항이 있습니다. 이러한 염료는 세포 특이성이 부족하고 담금질에 취약하며 일부는 독성이 있습니다. 또한 시간이 지남에 따라 확산될 수 있으며 미토콘드리아 ΔΨ가 손실되면 누출되어 탈분극된 미토콘드리아의 막 전위를 나타낼 수 없게 됩니다. 또한, TMRM 및 TMRE와 같은 로다민 기반 염료는 온도에 민감하기 때문에8 특히 세포 열발생과 관련된 생리학적 활동 중 미토콘드리아 막 전압을 측정할 때 염료 형광에 대한 온도 영향을 신중하게 고려해야 합니다.
형광 신호 9,10를 통해 막 전위 변화를 감지할 수 있는 단백질인 GEVI(Genetically encoded voltage indicator)는 다양한 세포 맥락에서 막 전위를 모니터링하기 위한 강력한 도구로 부상했습니다11. GEVI는 원형질막을 연구하기 위해 광범위하게 적용되었지만, 특히 미토콘드리아에 대한 세포 내 막 전위를 측정하기 위해 GEVI를 적용하는 데는 거의 진전이 없었습니다. 이 프로토콜은 in vitro 및 in vivo에서 미토콘드리아 막 전위를 모니터링할 수 있는 미토콘드리아 표적 GEVI를 사용하여 이러한 격차를 해소하고자 합니다. 기존 GEVI에 미토콘드리아 신호 서열을 추가함으로써 적절한 GEVI를 미토콘드리아12에 표적화할 수 있습니다. 이러한 미토콘드리아 전위 지표(MPI)는 미토콘드리아 생리학에 대한 새로운 통찰력을 제공하고 생체 내 다양한 질병 상태에서 미토콘드리아 기능을 탐구할 수 있는 상당한 잠재력을 제공하여 미토콘드리아 역학이 정상 및 병리학적 세포 과정에 어떻게 기여하는지에 대한 이해를 높일 것입니다.
프로토콜
모든 동물 관리 및 실험은 정저우 대학의 기관 동물 관리 및 사용 위원회 지침에 따라 수행되었습니다. 사용하기 전에 모든 수술 기구를 소독하십시오. 감염을 예방하기 위해 무균 기술을 따르십시오. 모든 데이터를 수집한 후, 동물들은 흡입제 마취를 과다 투여한 후 참수형을 집행하여 안락사시켰다.
1. In vitro 적용
- 플라스미드 구조
- Addgene에서 활동전위 1(ASAP1) 및 ASAP3 유전자의 가속 센서를 얻거나 염기서열에서 합성합니다. (ASAP1, NCBI 등록 ID: AHV90412.1, 애드진 ID: 52519; ASAP3, Addgene ID: 132331).
- 플라스미드 구조를 위해 표 1 에 나열된 프라이머를 사용하십시오.
- 세포에서 발현하려면 EGFPN1 벡터를 구성에 사용합니다.
- 처음에는 NheI 및 XhoI 제한 부위 옆에 있는 4개의 탠덤 반복(4cox8)으로 cox8 유전자 서열(NP_004065, 아미노산 1-29)을 합성합니다. 합성된 4cox8 단편과 EGFPN1 벡터를 모두 NheI 및 XhoI으로 분해한 다음 결찰하여 4cox8-EGFP 벡터를 생성합니다.
- 그 후, primer 1 및 primer 2를 사용하여 ASAP1 염기서열의 PCR 증폭을 수행합니다. ASAP1 단편과 4cox8-EGFP 벡터를 SalI 및 NotI로 분해한 다음 결찰하여 최종 MPI-1 벡터(MPI는 미토콘드리아 전위 지표)를 생성합니다.
참고: CMV-MPI-1의 벡터 맵은 그림 1에 나와 있습니다. 자세한 순서는 NCBI(등록 ID: PQ678920)에서 액세스할 수 있습니다.
- 뉴런에서의 특이적 발현을 위해 primer 3 및 primer 4를 사용하여 AAV-hSyn-EGFP plasmid(NCBI Accession ID: MH458079, Addgene ID: 50465)에서 hSyn promoter sequence의 PCR 증폭을 수행합니다.
- hSyn 염기서열과 4cox8-EGFP 벡터를 모두 AseI 및 NheI으로 분해한 다음 접합하여 hSyn-4cox8-EGFP 벡터를 생성합니다. 그 후, primer 1 및 primer 2를 사용하여 ASAP3 염기서열의 PCR 증폭을 수행합니다.
- hSyn-4cox8-EGFP 벡터와 ASAP3 염기서열을 SalI 및 NotI로 분해한 다음 ligation을 수행하여 hSyn-MPI-2 벡터를 생성합니다.
참고: PCR 조건은 특정 효소 및 프라이머 용융 온도(Tm)에 따라 달라집니다. 일반적인 PCR 반응은 95°C에서 30초 동안 초기 변성 단계를 거친 다음 변성(95°C, 10초), 어닐링(55°C, 30초) 및 확장(72°C, 30초/kb)의 25-35 사이클을 포함합니다. 72°C에서 5분 동안 최종 확장 단계를 수행하면 완전한 제품 합성이 보장된 후 4°C에서 유지됩니다. 어닐링 온도는 프라이머의 Tm보다 약 5°C 낮아야 합니다. 최적의 PCR 매개변수에 대해서는 제조업체의 지침을 참조하십시오. ASAP1 및 ASAP3의 프라이머는 동일합니다.
- 살아있는 세포에서의 이미징
- 배지에 10% 소 태아 혈청과 100mg/L 페니실린/스트렙토마이신을 첨가하여 DMEM 배양 배지를 준비합니다.
- Ca2+ 형질주입 시약을 준비합니다: 2.5 mol/L CaCl2, 2x HEBS (274 mmol/L NaCl, 10 mmol/L KCl, 1.4 mmol/L Na2HPO4, 15 mmol/L D-glucose, 42 mmol/L HEPES, pH 7.07).
- 이미징 중 세포를 유지하기 위한 Tyrode의 완충액을 준비합니다(145mmol/L NaCl, 3mmol/L KCl, 10mmol/L HEPES, 10mmol/L 포도당, pH 7.4).
- 37°C의 온도와 5% CO2/95% 공기의 분위기의 인큐베이터에서 DMEM 배양 배지를 공급한 35mm 배양 접시에서 헬라 세포를 배양합니다. 접시 당 2-3 개의 커버 슬립을 추가하여 세포가 자랄 수 있도록합니다.
- calcium phosphate 침전법을 통해 Hela 세포를 transfection합니다.
- 세포를 transfection하려면 먼저 기존 배양 배지를 2mL의 무혈청 DMEM으로 교체합니다. 35분 후, 마이크로 원심분리 튜브에서 DNA 10μg, CaCl2 4μL 및 ddH2O를 최종 부피 40μL까지 혼합하여 DNA/Ca2+ 용액을 준비합니다.
- 40μL의 2x HEBS를 DNA/Ca2+ 용액에 적가하면서 격렬하게 저어줍니다. 인산칼슘 침전을 촉진하기 위해 피펫으로 혼합물에 공기를 거품을 냅니다.
- 25분 후, 이 침전액 용액 80μL를 세포가 있는 접시에 넣고 1-2시간 더 인큐베이터에 다시 넣습니다. 그런 다음 비혈청 DMEM을 제거하고 15% 글리세롤(PBS에 용해됨)로 세포를 2분 동안 헹굽니다.
- 글리세롤을 흡인시킨 후 10% FBS가 함유된 DMEM 2mL를 첨가하고 16-24시간 동안 세포를 계속 배양합니다.
- 2mL의 Tyrode 완충액이 있는 35mm 배양 접시에 세포가 있는 커버슬립을 놓고 접시에 20μmol/L 로다민 800(ddH2O에 용해됨)을 최종 농도 50nmol/L로 첨가하여 세포를 로다민 800 염료로 염색합니다.
- 형광 현미경을 준비하고 적절한 필터와 광원을 설정하여 488nm 광원으로 단백질을 여기시키고 490-540nm 사이의 방출을 수집합니다. 633nm 광원으로 rhodamine 800 염료를 여기시키고 650-720nm 사이의 방출을 수집합니다.
- 500μM(스톡)에서 카르보닐 시안화물 m-클로로페닐 히드라존(CCCP) 용액을 준비하고 이미징 중에 최종 농도 5μM까지 세포에 추가합니다.
참고: 2x HEBS 용액의 pH는 transfection 효율에 매우 중요합니다. pH는 고체 수산화나트륨으로 7.07로 조심스럽게 조정해야 합니다. pH는 7.01보다 낮거나 7.12보다 높을 수 없습니다.
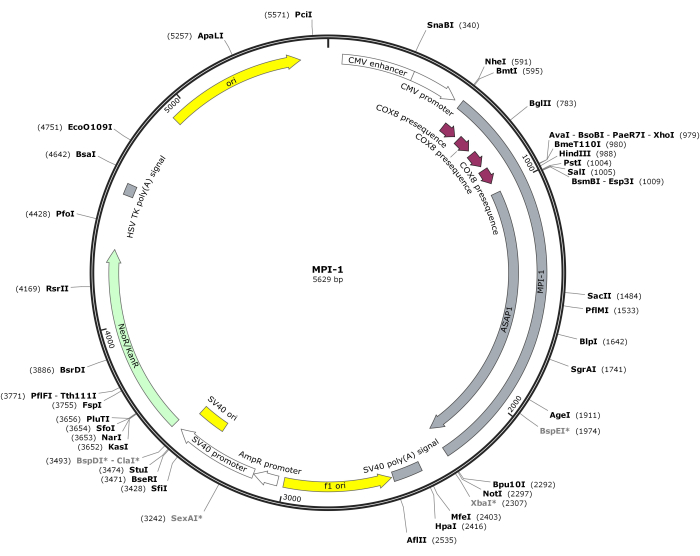
그림 1: CMV-MPI-1의 벡터 맵. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. In vivo 응용 프로그램
- 플라스미드 구조체
- in vivo 발현의 경우 형질도입을 위해 AAV 바이러스를 사용합니다. AAV 바이러스 패키징을 위한 ITR(Inverted Terminal Repeats) 염기서열을 포함하는 AAV 패키징(Addgene ID: 46954 또는 26968)의 발현 백본을 얻습니다.
- 표 1의 프라이머 5 및 7을 사용하여 hSyn-MPI-2 벡터의 PCR 증폭을 수행합니다(단계 1.1.4에서).
- 표 1에서 동일한 템플렛 및 뇌관 6 및 8을 사용하여 두번째 PCR 확대를 실행한다.
- 첫 번째 및 두 번째 PCR(단계 2.1.2 및 2.1.3)의 생성물을 템플릿으로 사용하고 표 1의 프라이머 5 및 8을 사용하여 최종 PCR을 수행하여 최종 hSyn-MPI-2 서열을 생성합니다.
- MluI 및 EcoRI를 사용하여 hSyn-MPI-2 염기서열과 pAAV 백본을 분해한 다음 결찰을 수행하여 최종 pAAV-hSyn-MPI-2 구조를 생성합니다. pAAV-hSyn-MPI-2의 벡터 맵은 그림 2에 나와 있습니다. 자세한 시퀀스는 NCBI(등록 ID: PQ678919)에서 액세스할 수 있습니다.
참고: hSyn-MPI-2 벡터에는 원래 EGFPN1 백본에서 유래한 내부 EcoRI 부위가 포함되어 있으며, 이는 중첩 PCR 전략에서 변형된 프라이머를 사용하여 제거됩니다(단계 2.1.2-2.1.4).
- 바이러스 준비 및 형질전환
- 37°C의 온도와 5% CO2/95% 공기의 분위기로 인큐베이터에서 10% FBS DMEM 배양 배지를 공급한 100mm 배양 접시에서 HEK293t 세포를 배양하여 70% 합류점에 도달합니다.
- 인산칼슘 침전법을 사용하여 HEK293t 세포를 AAV 유전자 플라스미드, 캡시드(pAAV-DJ 또는 pAAV9) 및 헬퍼 플라스미드(pHelper)와 함께 transfection합니다. 각 플라스미드에 대한 DNA 양은 다음과 같다: gene: 10 μg, pHelper: 11 μg, capsid: 9 μg. 형질주입 시약량: 12 μL의 CaCl2, 120 μL의 2x HEBS.
- 형질주입된 세포를 72시간 동안 배양하여 바이러스가 생성될 수 있도록 합니다.
- 잠복기가 끝나면 400g에서 원심 분리기로 세포를 수확합니다. PBS로 세포를 2회 헹구고 PBS 400μL에 세포를 재현탁합니다.
- 4주기의 동결-해동 방법(37°C 및 -80°C)으로 세포 현탁액에서 바이러스를 수확합니다. 용해물을 4°C에서 13,500 × g 으로 원심분리하고 펠릿을 버립니다. 상등액에는 AAV 바이러스가 포함되어 있습니다.
- Real-Time PCR을 수행하여 준비된 바이러스의 적정을 측정합니다. AAV 적정 측정을 위한 자세한 프로토콜은 https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/ 에서 확인할 수 있습니다.
- 제조된 AAV 바이러스를 분취하여 -80°C에서 보관합니다.
- 정위 바이러스 주입
- 30 mg/kg의 복강내 주사를 통해 3% 펜토바르비탈 나트륨으로 24-26g 무게의 성인 C57/BL 수컷 마우스를 마취합니다. 마취가 끝나면 시술 내내 안정적인 위치를 유지하기 위해 마우스를 입체 장치에 고정합니다.
- 수의학 안과 연고를 바르면 빛 손상으로부터 눈을 보호하고 눈을 촉촉하게 유지할 수 있습니다.
- 제모 크림을 사용하여 쥐 머리의 털을 제거하고 두피를 노출시킵니다.
- 수술용 가위를 사용하여 두피를 열고 두개골을 드러냅니다. 두개골을 3% 과산화수소로 처리하여 브레그마(bregma) 및 람다(lambda) 사이트와 같은 주요 랜드마크를 살균하고 시각화합니다.
- 두개골이 수평이 되도록 마우스 머리의 위치를 조정합니다.
- 프로그래밍 가능한 마이크로피펫 풀러를 사용하여 정확한 주입을 위해 유리 피펫을 원하는 모양과 크기로 당깁니다. 유리 마이크로피펫에 파라핀 오일을 채웁니다.
- 마이크로피펫을 대상 부위에 놓습니다. 이 프로토콜에서 대상은 다음 좌표를 사용하여 오른쪽의 M2 영역입니다: AP(전방-후), +1.94mm; ML (내측), +0.75 mm; DV (배복부), -1.5mm.
- 전기 드릴을 사용하여 대상 부위의 두개골에 작은 구멍을 뚫습니다.
- 마이크로피펫 끝을 통해 1.5 x 1011 Genome Copies per milliliter(GC/mL)의 역가에서 500nL의 바이러스를 열망합니다. 주입 펌프를 사용하여 100nL/min의 유속으로 500nL의 바이러스를 표적 부위에 주입합니다.
- 주입 후 피펫을 조심스럽게 집어넣기 전에 확산을 허용하기 위해 약 5분 동안 피펫을 제자리에 두십시오.
- 주사 부위에 광섬유(직경 200μm, 개구수 0.37, 길이 2mm)를 주입합니다.
- 치과용 시멘트를 사용하여 광섬유를 제자리에 고정합니다.
- 5-0 나일론 실(미적 바늘 Δ1/2 4x12)로 마우스의 두피를 봉합하고 집에 있는 케이지로 되돌립니다.
- 마취에서 완전히 회복될 때까지 마우스의 체온을 유지하기 위해 가정용 케이지를 가열 패드에 놓습니다.
- 동물이 움직이기 시작하면 발열 패드를 끕니다. 마우스를 하우징 시설로 옮기기 전에 실내 온도(RT)에 2시간 더 적응할 수 있습니다.
- 22°C에서 12시간 조명/12시간 어두운 주기의 하우스 마우스에서 바이러스 발현을 허용하기 위해 2-3주 동안 음식과 물에 자유롭게 접근할 수 있습니다.
- 섬유 측광
- 마우스에 이식된 광섬유를 광 케이블에 연결합니다.
- 광 케이블의 다른 쪽 끝을 이미징 데이터를 캡처하는 데 사용할 감지기에 연결합니다.
- 이미징을 위해 광원을 사용하여 488nm의 파장에서 단백질을 여기시킵니다. 490nm에서 540nm 범위의 파장에서 방출된 빛을 수집하도록 검출기를 설정합니다.
- 여기광의 강도를 조정하여 뇌 조직에 손상을 주지 않고 신호를 최적화합니다.
- 형광 신호의 게인을 수정하여 선명하고 강한 신호가 검출기에 의해 캡처되도록 합니다.
- 이미징의 품질을 저하시킬 수 있는 형광의 급격한 표백을 방지하려면 필요에 따라 광도와 형광 신호의 이득을 미세 조정하십시오.
- 마우스 동작 테스트를 수행합니다.
- 동작 테스트 중에 이미징 프로세스를 지속적으로 모니터링합니다.
- 데이터 분석
- Python 및 GNUplot을 사용하여 데이터를 시각적으로 표현하기 위한 실험 시행의 히트맵을 만듭니다. 소스 코드는 보충 파일 1에 제공됩니다.
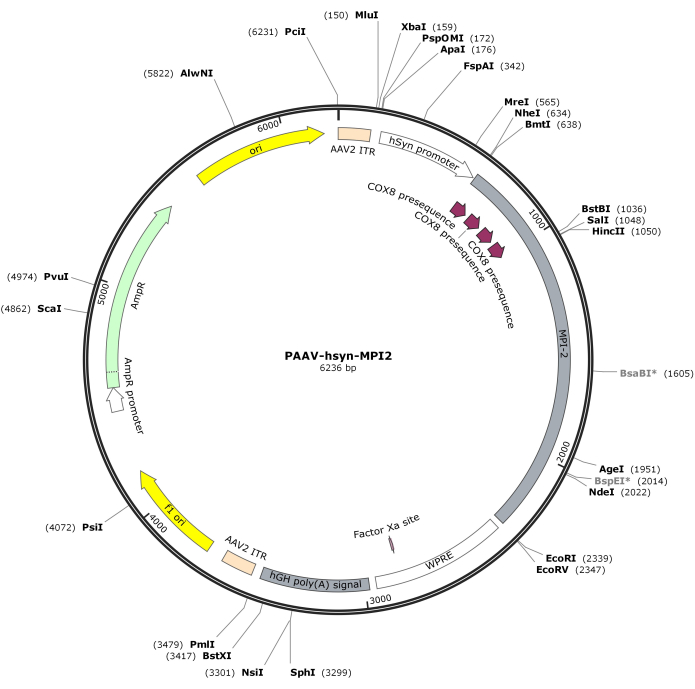
그림 2: AAV-hSyn-MPI-2의 벡터 맵. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
CMV-MPI-1 플라스미드를 구축한 후, 염색을 위해 미토콘드리아 마커 Rhodamine 800을 사용하여 Hela 세포에서 미토콘드리아를 표적으로 하는 능력을 테스트했습니다. 공동 국소화 실험은 MPI-1의 형광 신호와 Rhodamine 800의 신호 사이에 높은 수준의 중복을 보여주었으며, 이는 MPI-1이 미토콘드리아에 성공적으로 국소화되었음을 나타냅니다(그림 3).
토론
미토콘드리아 막 전압은 휴지 상태에서 -120--180mV로 유지되며 대사 상태의 변화에 따라 변동합니다. 현재 미토콘드리아 막 전위의 측정은 전기생리학적 방법과 형광 염료 방법을 사용하여 수행할 수 있습니다. 미토콘드리아 패치 클램핑은 미토콘드리아의 분리와 세포 구조의 파괴를 필요로 합니다13. 이 접근 방식은 생리학적 조건에서 벗어난 측정으로 ?...
공개
저자는 공개할 내용이 없습니다.
감사의 말
중국 국립자연과학재단(National Natural Science Foundation, NSF)의 지원인 JSK(32071137 및 92054103)와 정저우대학교 제1부속병원(The First Affiliated Hospital of Zhengzhou University: JSK, ZYCXTD2023014)의 과학 연구 및 혁신팀에 대한 자금 지원에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
참고문헌
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -. Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431 (2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977 (2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911 (2018).
- Yang, R. -. Z., Wang, D. -. D., Li, S. -. M., Liu, P. -. P., Kang, J. -. S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347 (2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -. F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541 (2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678 (2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유