Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure du potentiel de la membrane mitochondriale in vivo à l’aide d’un indicateur de tension génétiquement codé
Dans cet article
Résumé
Ce protocole décrit l’application d’indicateurs de tension génétiquement codés (GEVI) ciblant les mitochondries. Ces GEVI offrent un avantage significatif par rapport aux colorants de potentiel de membrane mitochondriale traditionnels en permettant une surveillance spécifique, in vivo et en temps réel du potentiel de membrane mitochondriale.
Résumé
Le potentiel membranaire mitochondrial (MMP, ΔΨm) est essentiel pour les fonctions mitochondriales, notamment la synthèse de l’ATP, le transport d’ions, la génération d’espèces réactives de l’oxygène (ROS) et l’importation de protéines codées par le noyau. Les méthodes existantes de mesure de ΔΨm utilisent généralement des colorants cationiques lipophiles, tels que la rhodamine 800 et l’ester méthylique de tétraméthylrhodamine (TMRM), mais ceux-ci sont limités par leur faible spécificité et ne sont pas bien adaptés aux applications in vivo . Pour remédier à ces limitations, nous avons développé un nouveau protocole utilisant des indicateurs de tension génétiquement codés (GEVI). Les indicateurs de tension génétiquement codés (GEVI), qui génèrent des signaux fluorescents en réponse aux changements de potentiel membranaire, ont démontré un potentiel significatif pour la surveillance de la membrane plasmique et des potentiels neuronaux. Cependant, leur application aux membranes mitochondriales reste inexplorée. Ici, nous avons développé des GEVI mitochondriaux à base de protéines capables de détecter les fluctuations de ΔΨm dans les cellules et le cortex moteur des animaux vivants. L’indicateur de potentiel mitochondrial (MPI) offre une approche non invasive pour étudier la dynamique de ΔΨm en temps réel, fournissant une méthode pour étudier la fonction mitochondriale dans des conditions normales et pathologiques.
Introduction
Les mitochondries sont des organites essentiels dans les cellules eucaryotes, servant de principales sources d’énergie par la génération d’adénosine triphosphate (ATP) tout en remplissant une variété d’autres fonctions cruciales, telles que la synthèse des métabolites, le tampon des ions calcium, la production de chaleur et la régulation de la survie cellulaire1. Leurs rôles sont particulièrement critiques dans les tissus hautement métaboliques comme le cerveau et le cœur, où ils aident à maintenir l’homéostasie cellulaire. Le potentiel de membrane mitochondriale (MMP, Ψm) est au cœur de ces processus, notamment en pilotant la synthèse de l’ATP par phosphorylation oxydative, en facilitant le transport des métabolites et des ions à travers les membranes mitochondriales et en contribuant à la génération d’espèces réactives de l’oxygène (ROS)2,3. La MMP influence également la morphologie etla dynamique mitochondriales4, y compris la mitophagie (la dégradation sélective des mitochondries)5et l’apoptose (mort cellulaire programmée)6. Le maintien d’un Ψm approprié est essentiel pour la fonction cellulaire ; Sa dérégulation est liée à de nombreuses pathologies, notamment les maladies neurodégénératives, l’insuffisance cardiaque et le cancer. Les méthodes actuelles de mesure de Ψm reposent principalement sur l’utilisation de colorants cationiques lipophiles, notamment TMRM (ester méthylique de tétraméthylrhodamine), TMRE (ester éthylique de tétraméthylrhodamine), Rhodamine 123, Safranine O, Rhodamine 800, DiOC6, JC-1, etc.7. Cependant, ces molécules fluorescentes présentent plusieurs limites. Ces colorants manquent de spécificité cellulaire, sont sensibles à la trempe et certains sont toxiques. De plus, ils peuvent diffuser au fil du temps, et lorsque ΔΨ mitochondrial est perdu, ils s’échappent, ce qui les rend incapables d’indiquer le potentiel membranaire des mitochondries dépolarisées. De plus, les colorants à base de rhodamine comme le TMRM et le TMRE sont sensibles à la température8, ce qui nécessite un examen attentif des effets de la température sur la fluorescence du colorant, en particulier lors de la mesure de la tension de la membrane mitochondriale pendant les activités physiologiques impliquant la thermogenèse cellulaire.
Les indicateurs de tension génétiquement codés (GEVI), des protéines capables de détecter les changements de potentiel membranaire par des signaux fluorescents 9,10, sont devenus des outils puissants pour surveiller les potentiels membranaires dans divers contextes cellulaires11. Bien que les GEVI aient été largement appliqués à l’étude des membranes plasmiques, il y a eu peu de progrès dans leur adaptation pour mesurer les potentiels de membrane intracellulaire, en particulier pour les mitochondries. Ce protocole cherche à combler cette lacune en utilisant des GEVI ciblés sur les mitochondries qui pourraient surveiller le potentiel de la membrane mitochondriale in vitro et in vivo. En ajoutant une séquence de signal mitochondrial aux GEVI existants, il est possible de cibler les mitochondries12. Ces indicateurs de potentiel mitochondrial (MPI) fourniraient de nouvelles informations sur la physiologie mitochondriale et offriraient un potentiel important pour l’exploration de la fonction mitochondriale dans divers états pathologiques in vivo, améliorant ainsi notre compréhension de la façon dont la dynamique mitochondriale contribue aux processus cellulaires normaux et pathologiques.
Access restricted. Please log in or start a trial to view this content.
Protocole
Tous les soins et expériences sur les animaux ont été effectués conformément aux directives du Comité institutionnel de soin et d’utilisation des animaux de l’Université de Zhengzhou. Stérilisez tous les instruments chirurgicaux avant utilisation. Suivez des techniques d’asepsie pour prévenir l’infection. Après que toutes les données aient été acquises, les animaux ont été euthanasiés à l’aide d’une surdose d’anesthésie inhalée suivie d’une décapitation.
1. Applications in vitro
- Construction plasmidique
- Obtenir les gènes du capteur accéléré de potentiels d’action 1 (ASAP1) et ASAP3 à partir d’Addgene ou en synthétisant à partir de la séquence. (ASAP1, N° d’accession NCBI : AHV90412.1, N° Addgene : 52519 ; ASAP3, Addgene ID : 132331).
- Utilisez les amorces répertoriées dans le tableau 1 pour les constructions plasmidiques.
- Pour l’expression dans les cellules, utilisez le vecteur EGFPN1 pour la construction.
- Initialement, synthétisez la séquence du gène cox8 (NP_004065, acides aminés 1-29) avec quatre répétitions en tandem (4cox8) flanquées de sites de restriction NheI et XhoI. Digérer à la fois le fragment synthétisé de 4cox8 et le vecteur EGFPN1 avec NheI et XhoI, suivi d’une ligature pour générer le vecteur 4cox8-EGFP.
- Par la suite, effectuez une amplification PCR de la séquence ASAP1 avec l’amorce 1 et l’amorce 2. Digérer le fragment ASAP1 et le vecteur 4cox8-EGFP avec SalI et NotI, suivi d’une ligature pour générer le vecteur MPI-1 final (MPI signifie indicateur de potentiel mitochondrial).
REMARQUE : La carte vectorielle du CMV-MPI-1 est illustrée à la figure 1. La séquence détaillée peut être consultée à partir du NCBI (ID d’accession : PQ678920).
- Pour une expression spécifique dans les neurones, effectuez une amplification PCR de la séquence du promoteur hSyn à partir du plasmide AAV-hSyn-EGFP (NCBI Accession ID : MH458079, Addgene ID : 50465) à l’aide de l’amorce 3 et de l’amorce 4.
- Digérer à la fois la séquence hSyn et le vecteur 4cox8-EGFP avec AseI et NheI, suivi d’une ligature pour générer le vecteur hSyn-4cox8-EGFP. Par la suite, effectuez une amplification PCR de la séquence ASAP3 avec l’amorce 1 et l’amorce 2.
- Digérer le vecteur hSyn-4cox8-EGFP et la séquence ASAP3 avec SalI et NotI, suivis d’une ligature pour générer le vecteur hSyn-MPI-2.
REMARQUE : Les conditions de PCR varient en fonction des enzymes spécifiques et des températures de fusion de l’amorce (Tm). Une réaction PCR typique implique une étape initiale de dénaturation à 95 °C pendant 30 s, suivie de 25 à 35 cycles de dénaturation (95 °C, 10 s), de recuit (55 °C, 30 s) et d’extension (72 °C, 30 s/kb). Une dernière étape d’extension à 72 °C pendant 5 min assure une synthèse complète du produit, suivie d’un maintien à 4 °C. La température de recuit doit être d’environ 5 °C en dessous de la Tm des amorces. Reportez-vous aux instructions du fabricant pour connaître les paramètres PCR optimaux. Les amorces pour ASAP1 et ASAP3 sont identiques.
- Imagerie dans les cellules vivantes
- Préparez le milieu de culture DMEM en ajoutant 10 % de sérum fœtal bovin et 100 mg/L de pénicilline/streptomycine au milieu.
- Préparez des réactifs de transfection Ca2+ : 2,5 mol/L de CaCl2, 2x HEBS (274 mmol/L de NaCl, 10 mmol/L de KCl, 1,4 mmol/L de Na2HPO4, 15 mmol/L de D-glucose, 42 mmol/L d’HEPES, pH 7,07).
- Préparez le tampon de Tyrode pour le maintien des cellules pendant l’imagerie (145 mmol/L de NaCl, 3 mmol/L de KCl, 10 mmol/L de HEPES, 10 mmol/L de glucose, pH 7,4).
- Cellules de culture Hela dans une boîte de culture de 35 mm alimentée avec un milieu de culture DMEM dans un incubateur à une température de 37 °C et une atmosphère de 5 % CO2/95 % air. Ajoutez 2-3 lamelles par boîte pour permettre aux cellules de se développer dessus.
- Transfectez les cellules Hela via la méthode de précipitation au phosphate de calcium.
- Pour transfecter des cellules, commencez par remplacer l’ancien milieu de culture par 2 ml de DMEM sans sérum. Après 35 min, préparez la solution d’ADN/Ca2+ en mélangeant 10 μg d’ADN, 4 μL de CaCl2 et du ddH2O jusqu’à un volume final de 40 μL dans un tube de microcentrifugation.
- Ajouter 40 μL de 2x HEBS à la solution d’ADN/Ca2+ goutte à goutte tout en remuant vigoureusement. Faites des bulles d’air avec une pipette dans le mélange pour faciliter la précipitation du phosphate de calcium.
- Après 25 min, ajoutez 80 μL de cette solution précipitée dans la boîte avec les cellules et remettez-la dans l’incubateur pendant encore 1 à 2 h. Ensuite, retirez le DMEM non sérique et rincez les cellules avec du glycérol à 15 % (dissous dans du PBS) pendant 2 min.
- Après avoir aspiré le glycérol, ajoutez 2 mL de DMEM contenant 10 % de FBS et poursuivez la culture des cellules pendant 16 à 24 h.
- Placer les lamelles avec les cellules dans une boîte de culture de 35 mm avec 2 mL de tampon Tyrode et colorer les cellules avec un colorant à la rhodamine 800 en ajoutant 20 μmol/L de rhodamine 800 (dissoute dans ddH2O) dans la boîte jusqu’à une concentration finale de 50 nmol/L.
- Préparez un microscope à fluorescence et installez-le avec les filtres et la source de lumière appropriés pour exciter la protéine avec une source lumineuse de 488 nm et collecter l’émission entre 490 et 540 nm. Excitez le colorant rhodamine 800 avec une source lumineuse de 633 nm et collectez l’émission entre 650 et 720 nm.
- Préparer une solution de cyanure de carbonyle m-chlorophénylhydrazone (CCCP) à 500 μM (stock) et l’ajouter aux cellules jusqu’à une concentration finale de 5 μM pendant l’imagerie.
REMARQUE : Le pH de la solution HEBS 2x est crucial pour l’efficacité de la transfection. Le pH doit être soigneusement ajusté à 7,07 avec de l’hydroxyde de sodium solide. Le pH ne peut pas être inférieur à 7,01 ou supérieur à 7,12.
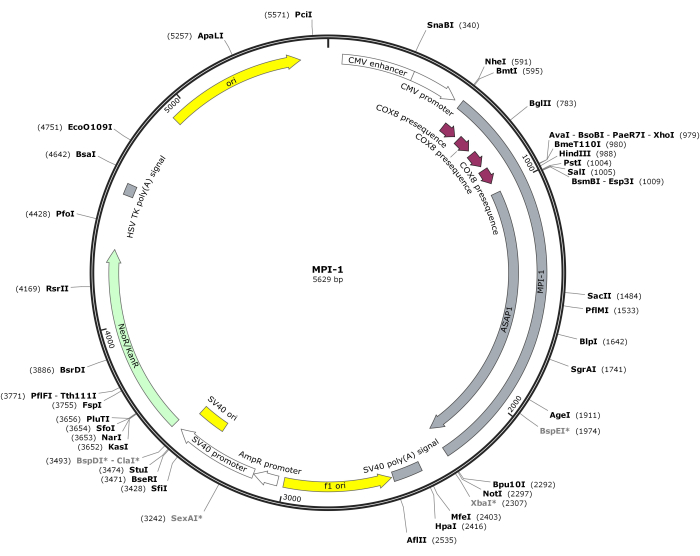
Figure 1 : Carte vectorielle du CMV-MPI-1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Applications in vivo
- Construction plasmidique
- Pour l’expression in vivo , utilisez le virus AAV pour la transduction. Procurez-vous le squelette d’expression pour l’emballage AAV (ID Addgene : 46954 ou 26968), qui contient des séquences de répétitions terminales inversées (ITR) pour l’emballage du virus AAV.
- Effectuez l’amplification par PCR du vecteur hSyn-MPI-2 (à partir de l’étape 1.1.4) à l’aide des amorces 5 et 7 du tableau 1.
- Effectuez une deuxième amplification par PCR à l’aide du même gabarit et des amorces 6 et 8 du tableau 1.
- Effectuer une PCR finale en utilisant le produit de la première et de la deuxième PCR (étapes 2.1.2 et 2.1.3) comme matrice et amorces 5 et 8 du tableau 1, générant la séquence finale de hSyn-MPI-2.
- Digérer la séquence hSyn-MPI-2 et le squelette pAAV avec MluI et EcoRI, suivi d’une ligature pour générer la construction finale de pAAV-hSyn-MPI-2. La carte vectorielle de pAAV-hSyn-MPI-2 est illustrée à la figure 2. La séquence détaillée peut être consultée à partir du NCBI (ID d’accession : PQ678919).
REMARQUE : Le vecteur hSyn-MPI-2 contient un site EcoRI interne provenant du squelette EGFPN1 d’origine, qui est éliminé à l’aide d’amorces modifiées dans le cadre d’une stratégie de PCR qui se chevauche (étapes 2.1.2 à 2.1.4).
- Préparation et transduction du virus
- Cultivez des cellules HEK293t dans une boîte de culture de 100 mm alimentée avec un milieu de culture 10 % FBS DMEM dans un incubateur à une température de 37 °C et une atmosphère de 5 % CO2/95 % d’air pour atteindre une confluence de 70 %.
- Co-transfectez les cellules HEK293t avec le plasmide du gène AAV, la capside (pAAV-DJ ou pAAV9) et les plasmides auxiliaires (pHelper) en utilisant la méthode de précipitation du phosphate de calcium. La quantité d’ADN pour chaque plasmide était la suivante : gène : 10 μg, pHelper : 11 μg, capside : 9 μg. La quantité de réactif de transfection : 12 μL de CaCl2, 120 μL de 2x HEBS.
- Incuber les cellules transfectées pendant une période de 72 h pour permettre la production du virus.
- Après la période d’incubation, récoltez les cellules par centrifugation à 400 g. Rincez les cellules avec du PBS 2 fois et remettez la cellule en suspension dans 400 μL de PBS.
- Récoltez les virus à partir de cellules en suspension à l’aide de quatre cycles de méthodes de congélation-décongélation (37 °C et -80 °C). Centrifugez le lysat à 13 500 × g à 4 °C et jetez la pastille. Le surnageant contient le virus AAV.
- Effectuez une PCR en temps réel pour déterminer le titrage du virus préparé. Le protocole détaillé pour la détermination du titrage AAV peut être trouvé à l’https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/.
- Aliquote et conservez le virus AAV préparé à -80 °C.
- Injection stéréotaxique de virus
- Anesthésier une souris mâle adulte C57/BL pesant de 24 à 26 g avec 3 % de pentobarbital sodique par injection intrapéritonéale à une dose de 30 mg/kg. Une fois anesthésiées, fixez les souris sur un appareil stéréotaxique pour maintenir une position stable tout au long de la procédure.
- Appliquez une pommade ophtalmique vétérinaire pour protéger les yeux des dommages causés par la lumière et pour garder les yeux humides.
- Utilisez une crème dépilatoire pour enlever les poils sur la tête de la souris et exposer le cuir chevelu.
- Ouvrez le cuir chevelu à l’aide de ciseaux chirurgicaux et exposez le crâne. Traitez le crâne avec du peroxyde d’hydrogène à 3 % pour stériliser et visualiser les points de repère clés tels que les sites bregma et lambda.
- Ajustez la position de la tête de la souris pour vous assurer que le crâne est horizontal.
- Utilisez un extracteur de micropipette programmable pour tirer les pipettes en verre à la forme et à la taille souhaitées pour des injections précises. Remplissez la micropipette en verre d’huile de paraffine.
- Positionnez la micropipette sur le site cible. Dans ce protocole, la cible est la région M2 du côté droit, en utilisant les coordonnées suivantes : AP (antéro-postérieur), +1,94 mm ; ML (médiolatéral), +0,75 mm ; DV (dorsoventral), -1,5 mm.
- Utilisez une perceuse électrique pour faire un petit trou dans le crâne à l’endroit cible.
- Aspire 500 nL de virus à un titre de 1,5 x10 11 copies du génome par millilitre (GC/mL) à travers la pointe de la micropipette. Injecter 500 nL de virus dans le site cible à l’aide d’une pompe d’injection à un débit de 100 nL/min.
- Après l’injection, laissez la pipette en place pendant environ 5 minutes pour permettre la diffusion avant de la rétracter soigneusement.
- Implantez la fibre optique (200 μm de diamètre, ouverture numérique de 0,37, longueur de 2 mm) dans le site d’injection.
- Fixez la fibre optique en place à l’aide de ciment dentaire.
- Suturez le cuir chevelu de la souris avec du fil de nylon 5-0 (aiguille esthétique Δ1/2 4x12) et renvoyez-la dans sa cage d’origine.
- Placez la cage domestique sur un coussin chauffant pour maintenir la température corporelle de la souris jusqu’à ce qu’elle se remette complètement de l’anesthésie.
- Éteignez le coussin chauffant une fois que l’animal commence à se déplacer. Prévoyez encore 2 h d’acclimatation à la température ambiante (RT) avant de transférer la souris dans le logement.
- Souris domestique dans un cycle de 12 h à la lumière et 12 h à l’obscurité à 22 °C avec accès à volonté à la nourriture et à l’eau pendant 2 à 3 semaines pour permettre l’expression virale.
- Photométrie sur fibre
- Connectez la fibre optique implantée dans la souris à un câble optique.
- Fixez l’autre extrémité du câble optique à un détecteur qui sera utilisé pour capturer les données d’imagerie.
- Pour l’imagerie, utilisez une source lumineuse pour exciter les protéines à une longueur d’onde de 488 nm. Configurez le détecteur pour qu’il capte la lumière émise à des longueurs d’onde allant de 490 nm à 540 nm.
- Ajustez l’intensité de la lumière d’excitation pour optimiser le signal sans endommager le tissu cérébral.
- Modifiez les gains du signal fluorescent pour vous assurer qu’un signal clair et fort est capturé par le détecteur.
- Pour éviter un blanchiment rapide de la fluorescence, qui réduirait la qualité de l’imagerie, ajustez l’intensité lumineuse et le gain du signal fluorescent si nécessaire.
- Effectuez un test de comportement de souris.
- Surveillez en permanence le processus d’imagerie pendant le test de comportement.
- Analyse des données
- Utilisez Python et GNUplot pour créer une carte thermique des essais expérimentaux afin d’obtenir une représentation visuelle des données. Les codes sources sont fournis dans le Fichier supplémentaire 1.
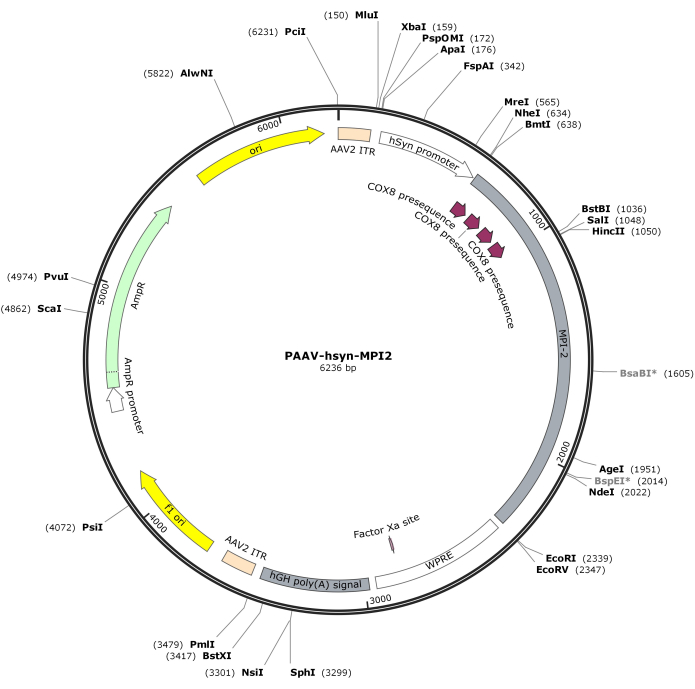
Figure 2 : Carte vectorielle d’AAV-hSyn-MPI-2. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Après la construction du plasmide CMV-MPI-1, sa capacité à cibler les mitochondries a été testée dans des cellules Hela à l’aide du marqueur mitochondrial Rhodamine 800 pour la coloration. Les expériences de colocalisation ont montré un degré élevé de chevauchement entre le signal de fluorescence de MPI-1 et le signal de Rhodamine 800, indiquant que MPI-1 a été localisé avec succès dans les mitochondries (Figure 3).
Access restricted. Please log in or start a trial to view this content.
Discussion
La tension de la membrane mitochondriale est maintenue à -120--180 mV dans des conditions de repos et fluctue avec les changements d’état métabolique. Actuellement, la mesure du potentiel de membrane mitochondriale peut être effectuée à l’aide de méthodes électrophysiologiques et de méthodes de colorant de fluorescence. Le clampage du patch mitochondrial nécessite l’isolement des mitochondries et la destruction des structures cellulaires13. Cette a...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions le soutien de la Fondation nationale des sciences naturelles (NSF) de Chine : JSK (32071137 et 92054103) et le financement de l’équipe de recherche scientifique et d’innovation du premier hôpital affilié de l’Université de Zhengzhou : JSK (ZYCXTD2023014).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
Références
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431(2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977(2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911(2018).
- Yang, R. -Z., Wang, D. -D., Li, S. -M., Liu, P. -P., Kang, J. -S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347(2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541(2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678(2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon