È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione del potenziale della membrana mitocondriale in vivo utilizzando un indicatore di tensione geneticamente codificato
In questo articolo
Riepilogo
Questo protocollo descrive l'applicazione di indicatori di tensione geneticamente codificati (GEVI) mirati ai mitocondri. Questi GEVI offrono un vantaggio significativo rispetto ai tradizionali coloranti con potenziale di membrana mitocondriale, consentendo il monitoraggio specifico, in vivo e in tempo reale del potenziale di membrana mitocondriale.
Abstract
Il potenziale di membrana mitocondriale (MMP, ΔΨm) è fondamentale per le funzioni mitocondriali, tra cui la sintesi di ATP, il trasporto ionico, la generazione di specie reattive dell'ossigeno (ROS) e l'importazione di proteine codificate dal nucleo. I metodi esistenti per misurare il ΔΨm utilizzano tipicamente coloranti cationici lipofili, come la rodamina 800 e l'estere metilico tetrametilrodamina (TMRM), ma questi sono limitati dalla bassa specificità e non sono adatti per applicazioni in vivo . Per affrontare queste limitazioni, abbiamo sviluppato un nuovo protocollo che utilizza indicatori di tensione geneticamente codificati (GEVI). Gli indicatori di tensione geneticamente codificati (GEVI), che generano segnali fluorescenti in risposta a variazioni del potenziale di membrana, hanno dimostrato un potenziale significativo per il monitoraggio della membrana plasmatica e dei potenziali neuronali. Tuttavia, la loro applicazione alle membrane mitocondriali rimane inesplorata. Qui, abbiamo sviluppato GEVI mitocondriali a bersaglio mitocondriale basati su proteine in grado di rilevare le fluttuazioni ΔΨm nelle cellule e nella corteccia motoria degli animali viventi. L'indicatore del potenziale mitocondriale (MPI) offre un approccio non invasivo per studiare la dinamica ΔΨm in tempo reale, fornendo un metodo per studiare la funzione mitocondriale sia in condizioni normali che patologiche.
Introduzione
I mitocondri sono organelli essenziali nelle cellule eucariotiche, fungendo da fornitori primari di energia attraverso la generazione di adenosina trifosfato (ATP) e svolgendo anche una varietà di altre funzioni cruciali, come la sintesi dei metaboliti, il tamponamento degli ioni calcio, la produzione di calore e la regolazionedella sopravvivenza cellulare. I loro ruoli sono particolarmente critici nei tessuti altamente metabolici come il cervello e il cuore, dove aiutano a mantenere l'omeostasi cellulare. Il potenziale di membrana mitocondriale (MMP, Ψm) è fondamentale per questi processi, tra cui la guida della sintesi di ATP attraverso la fosforilazione ossidativa, facilitando il trasporto di metaboliti e ioni attraverso le membrane mitocondriali e contribuendo alla generazione di specie reattive dell'ossigeno (ROS)2,3. La MMP influenza anche la morfologia e la dinamica mitocondriale4, tra cui la mitofagia (la degradazione selettiva dei mitocondri)5e l'apoptosi (morte cellulare programmata)6. Il mantenimento di un Ψm appropriato è essenziale per la funzione cellulare; La sua disregolazione è legata a numerose patologie, tra cui malattie neurodegenerative, insufficienza cardiaca e cancro. Gli attuali metodi per misurare Ψm si basavano principalmente sull'uso di coloranti cationici lipofili, tra cui TMRM (estere metilmetilrodamina), TMRE (estere etilico tetrametilrodammina), rodamina 123, safranina O, rodamina 800, DiOC6, JC-1, ecc.7. Tuttavia, queste molecole fluorescenti hanno diverse limitazioni. Questi coloranti mancano di specificità cellulare, sono suscettibili di estinzione e alcuni sono tossici. Inoltre, possono diffondersi nel tempo e, quando la ΔΨ mitocondriale viene persa, fuoriescono, rendendoli incapaci di indicare il potenziale di membrana dei mitocondri depolarizzati. Inoltre, i coloranti a base di rodamina come TMRM e TMRE sono sensibili alla temperatura8, il che richiede un'attenta considerazione degli effetti della temperatura sulla fluorescenza del colorante, in particolare quando si misura la tensione della membrana mitocondriale durante le attività fisiologiche che coinvolgono la termogenesi cellulare.
Gli indicatori di tensione geneticamente codificati (GEVI), proteine in grado di rilevare i cambiamenti del potenziale di membrana attraverso segnali fluorescenti 9,10, sono emersi come potenti strumenti per monitorare i potenziali di membrana in una varietà di contesti cellulari11. Mentre i GEVI sono stati ampiamente applicati per studiare le membrane plasmatiche, ci sono stati pochi progressi nell'adattarli per misurare i potenziali di membrana intracellulare, in particolare per i mitocondri. Questo protocollo cerca di colmare questa lacuna utilizzando GEVI mirati ai mitocondri in grado di monitorare il potenziale di membrana mitocondriale in vitro e in vivo. Aggiungendo la sequenza del segnale mitocondriale ai GEVI esistenti, i GEVI appropriati possono essere indirizzati ai mitocondri12. Questi indicatori del potenziale mitocondriale (MPI) fornirebbero nuove informazioni sulla fisiologia mitocondriale e offrirebbero un potenziale significativo per esplorare la funzione mitocondriale in vari stati patologici in vivo, migliorando la nostra comprensione di come le dinamiche mitocondriali contribuiscano ai processi cellulari sia normali che patologici.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutte le cure e gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida del Comitato Istituzionale per la Cura e l'Uso degli Animali dell'Università di Zhengzhou. Sterilizzare tutti gli strumenti chirurgici prima dell'uso. Seguire tecniche asettiche per prevenire l'infezione. Dopo che tutti i dati sono stati acquisiti, gli animali sono stati soppressi utilizzando un'overdose di anestesia inalante seguita da decapitazione.
1. Applicazioni in vitro
- Costruzione plasmidico
- Ottenere il sensore accelerato dei potenziali d'azione 1 (ASAP1) e i geni ASAP3 da Addgene o sintetizzando dalla sequenza. (ASAP1, ID di accesso NCBI: AHV90412.1, ID Addgene: 52519; ASAP3, Addgene ID: 132331).
- Utilizzare i primer elencati nella Tabella 1 per le costruzioni plasmidiche.
- Per l'espressione nelle cellule, utilizzare il vettore EGFPN1 per la costruzione.
- Inizialmente, sintetizzare la sequenza genica cox8 (NP_004065, amminoacidi 1-29) con quattro ripetizioni tandem (4cox8) affiancate da siti di restrizione NheI e XhoI. Digerire sia il frammento 4cox8 sintetizzato che il vettore EGFPN1 con NheI e XhoI, seguito da legatura per generare il vettore 4cox8-EGFP.
- Successivamente, eseguire l'amplificazione PCR della sequenza ASAP1 con il primer 1 e il primer 2. Digerire il frammento ASAP1 e il vettore 4cox8-EGFP con SalI e NotI, seguito da legatura per generare il vettore MPI-1 finale (MPI sta per indicatore di potenziale mitocondriale).
NOTA: La mappa vettoriale di CMV-MPI-1 è illustrata nella Figura 1. La sequenza dettagliata è accessibile da NCBI (Accession ID: PQ678920).
- Per l'espressione specifica nei neuroni, eseguire l'amplificazione PCR della sequenza del promotore hSyn dal plasmide AAV-hSyn-EGFP (NCBI Accession ID: MH458079, Addgene ID: 50465) utilizzando il primer 3 e il primer 4.
- Digerire sia la sequenza hSyn che il vettore 4cox8-EGFP con AseI e NheI, seguito da legatura per generare il vettore hSyn-4cox8-EGFP. Successivamente, eseguire l'amplificazione PCR della sequenza ASAP3 con il primer 1 e il primer 2.
- Digerire il vettore hSyn-4cox8-EGFP e la sequenza ASAP3 con SalI e NotI, seguiti da legatura per generare il vettore hSyn-MPI-2.
NOTA: Le condizioni della PCR variano in base agli enzimi specifici e alle temperature di fusione del primer (Tm). Una tipica reazione PCR prevede una fase iniziale di denaturazione a 95 °C per 30 s, seguita da 25-35 cicli di denaturazione (95 °C, 10 s), ricottura (55 °C, 30 s) ed estensione (72 °C, 30 s/kb). Un'ultima fase di estensione a 72 °C per 5 minuti garantisce la sintesi completa del prodotto, seguita da una tenuta a 4 °C. La temperatura di ricottura deve essere di circa 5 °C inferiore alla Tm degli inneschi. Fare riferimento alle istruzioni del produttore per i parametri PCR ottimali. I primer per ASAP1 e ASAP3 sono identici.
- Imaging in cellule viventi
- Preparare il terreno di coltura DMEM aggiungendo al terreno il 10% di siero fetale bovino e 100 mg/L di penicillina/streptomicina.
- Preparare i reagenti di trasfezione Ca2+ : 2,5 mol/L CaCl2, 2x HEBS (274 mmol/L NaCl, 10 mmol/L KCl, 1,4 mmol/L Na2HPO4, 15 mmol/L D-glucosio, 42 mmol/L HEPES, pH 7,07).
- Preparare il tampone di Tyrode per il mantenimento delle cellule durante l'imaging (145 mmol/L NaCl, 3 mmol/L KCl, 10 mmol/L HEPES, 10 mmol/L glucosio, pH 7,4).
- Coltura di cellule Hela in una piastra di coltura da 35 mm alimentata con terreno di coltura DMEM in un incubatore con una temperatura di 37 °C e un'atmosfera di 5% CO2/95% aria. Aggiungi 2-3 vetrini coprioggetti per piatto per consentire alle cellule di crescere su di essi.
- Trasfettare le cellule Hela con il metodo della precipitazione con fosfato di calcio.
- Per trasfettare le cellule, iniziare sostituendo il vecchio terreno di coltura con 2 ml di DMEM privo di siero. Dopo 35 minuti, preparare la soluzione di DNA/Ca2+ mescolando 10 μg di DNA, 4 μL di CaCl2 e ddH2O fino a un volume finale di 40 μL in una provetta da microcentrifuga.
- Aggiungere 40 μl di 2x HEBS alla soluzione di DNA/Ca2+ goccia a goccia, mescolando energicamente. Far bollire l'aria con una pipetta nella miscela per facilitare la precipitazione del fosfato di calcio.
- Dopo 25 minuti, aggiungere 80 μL di questa soluzione precipitata alla piastra con le cellule e rimetterla nell'incubatore per altre 1-2 ore. Quindi, rimuovere il DMEM non sierico e sciacquare le cellule con glicerolo al 15% (disciolto in PBS) per 2 minuti.
- Dopo aver aspirato il glicerolo, aggiungere 2 mL di DMEM contenente il 10% di FBS e continuare a coltivare le cellule per 16-24 ore.
- Posizionare i vetrini coprioggetti con le cellule in una piastra di coltura da 35 mm con 2 mL di tampone di Tyrode e colorare le cellule con il colorante rodamina 800 aggiungendo 20 μmol/L di rodamina 800 (disciolta in ddH2O) alla piastra fino a una concentrazione finale di 50 nmol/L.
- Preparare un microscopio a fluorescenza e impostarlo con i filtri e la sorgente luminosa appropriati per eccitare la proteina con una sorgente luminosa a 488 nm e raccogliere l'emissione tra 490-540 nm. Eccita il colorante rodamina 800 con una sorgente luminosa a 633 nm e raccogli l'emissione tra 650-720 nm.
- Preparare una soluzione di cianuro di carbonile m-clorofenil idrazone (CCCP) a 500 μM (stock) e aggiungere alle cellule fino a una concentrazione finale di 5 μM durante l'imaging.
NOTA: Il pH della soluzione 2x HEBS è fondamentale per l'efficienza della trasfezione. Il pH deve essere accuratamente regolato a 7,07 con idrossido di sodio solido. Il pH non può essere inferiore a 7,01 o superiore a 7,12.
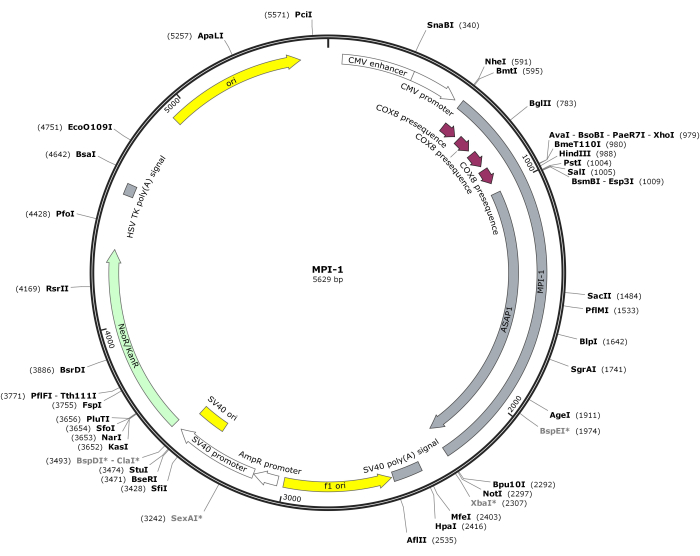
Figura 1: Mappa vettoriale di CMV-MPI-1. Clicca qui per visualizzare una versione più grande di questa figura.
2. Applicazioni in vivo
- Costrutto plasmidico
- Per l'espressione in vivo , utilizzare il virus AAV per la trasduzione. Ottenere la spina dorsale di espressione per il packaging AAV (Addgene ID: 46954 o 26968), che contiene sequenze ITR (Inverted Terminal Repeats) per il packaging del virus AAV.
- Eseguire l'amplificazione PCR del vettore hSyn-MPI-2 (dal passaggio 1.1.4) utilizzando i primer 5 e 7 nella Tabella 1.
- Eseguire una seconda amplificazione PCR utilizzando lo stesso modello e i primer 6 e 8 nella Tabella 1.
- Eseguire una PCR finale utilizzando il prodotto della prima e della seconda PCR (passaggi 2.1.2 e 2.1.3) come modello e primer 5 e 8 nella Tabella 1, generando la sequenza finale hSyn-MPI-2.
- Digerire la sequenza hSyn-MPI-2 e la spina dorsale pAAV con MluI ed EcoRI, seguita dalla legatura per generare il costrutto finale pAAV-hSyn-MPI-2. La mappa vettoriale di pAAV-hSyn-MPI-2 è illustrata nella Figura 2. La sequenza dettagliata è accessibile da NCBI (Accession ID: PQ678919).
NOTA: Il vettore hSyn-MPI-2 contiene un sito EcoRI interno originato dalla dorsale EGFPN1 originale, che viene rimosso utilizzando primer modificati in una strategia di PCR sovrapposta (passaggi 2.1.2-2.1.4).
- Preparazione e trasduzione del virus
- Coltura di cellule HEK293t in una piastra di coltura da 100 mm alimentata con il 10% di terreno di coltura FBS DMEM in un incubatore con una temperatura di 37 °C e un'atmosfera di CO2/95% di aria per raggiungere una confluenza del 70%.
- Co-trasfettare le cellule HEK293t con il plasmide del gene AAV, il capside (pAAV-DJ o pAAV9) e i plasmidi helper (pHelper) utilizzando il metodo di precipitazione del fosfato di calcio. La quantità di DNA per ciascun plasmide era la seguente: gene: 10 μg, pHelper: 11 μg, capside: 9 μg. La quantità di reagente di trasfezione: 12 μL di CaCl2, 120 μL di 2x HEBS.
- Incubare le cellule trasfettate per un periodo di 72 ore per consentire la produzione del virus.
- Trascorso il periodo di incubazione, raccogliere le cellule mediante centrifuga a 400 g. Sciacquare le cellule con PBS 2 volte e risospendere la cella in 400 μL di PBS.
- Raccogli i virus dalla sospensione cellulare con quattro cicli di metodi di congelamento-scongelamento (37 °C e -80 °C). Centrifugare il lisato a 13.500 × g a 4 °C ed eliminare il pellet. Il surnatante contiene il virus AAV.
- Eseguire la PCR in tempo reale per determinare la titolazione del virus preparato. Il protocollo dettagliato per la determinazione della titolazione AAV è disponibile all'https://www.addgene.org/protocols/aav-titration-qpcr-using-sybr-green-technology/.
- Aliquotare e conservare il virus AAV preparato a -80 °C.
- Iniezione stereotassica di virus
- Anestetizzare un topo maschio adulto C57/BL del peso di 24-26 g con il 3% di pentobarbital sodico tramite iniezione intraperitoneale alla dose di 30 mg/kg. Una volta anestetizzati, fissare i topi su un apparecchio stereotassico per mantenere una posizione stabile durante tutta la procedura.
- Applicare un unguento oftalmico veterinario per proteggere gli occhi dai danni della luce e per mantenere gli occhi umidi.
- Usa la crema depilatoria per rimuovere i peli sulla testa del topo ed esporre il cuoio capelluto.
- Apri il cuoio capelluto usando le forbici chirurgiche ed esponi il cranio. Trattare il cranio con perossido di idrogeno al 3% per sterilizzare e visualizzare i punti di riferimento chiave come i siti di bregma e lambda.
- Regola la posizione della testa del mouse per assicurarti che il teschio sia orizzontale.
- Utilizzare un estrattore per micropipette programmabile per estrarre le pipette di vetro alla forma e alle dimensioni desiderate per iniezioni precise. Riempire la micropipetta di vetro con olio di paraffina.
- Posizionare la micropipetta nel sito di destinazione. In questo protocollo, il bersaglio è la regione M2 sul lato destro, utilizzando le seguenti coordinate: AP (antero-posteriore), +1,94 mm; ML (mediolaterale), +0,75 mm; DV (dorso-ventrale), -1,5 mm.
- Usa un trapano elettrico per fare un piccolo foro nel cranio nel sito bersaglio.
- Aspire 500 nL di virus a un titolo di 1,5 x 1011 copie del genoma per millilitro (GC/mL) attraverso la punta della micropipetta. Iniettare 500 nL di virus nel sito bersaglio utilizzando una pompa di iniezione a una velocità di flusso di 100 nL/min.
- Dopo l'iniezione, lasciare la pipetta in posizione per circa 5 minuti per consentire la diffusione prima di ritrarla con cautela.
- Impianto di fibra ottica (diametro 200 μm, apertura numerica 0,37, lunghezza 2 mm) nel sito di iniezione.
- Fissare la fibra ottica in posizione utilizzando il cemento dentale.
- Sutura il cuoio capelluto del topo con filo di nylon 5-0 (ago estetico Δ1/2 4x12) e riportalo nella sua gabbia di casa.
- Posiziona la gabbia domestica su un termoforo per mantenere la temperatura corporea del mouse fino a quando non si riprende completamente dall'anestesia.
- Spegni il termoforo una volta che l'animale inizia a muoversi. Attendere altre 2 ore di acclimatazione alla temperatura ambiente (RT) prima di trasferire il mouse nella struttura di stabulazione.
- Topo domestico in un ciclo di 12 ore luce/12 ore buio a 22 °C con accesso ad libitum a cibo e acqua per 2-3 settimane per consentire l'espressione virale.
- Fotometria in fibra
- Collegare la fibra ottica impiantata nel mouse a un cavo ottico.
- Collegare l'altra estremità del cavo ottico a un rivelatore che verrà utilizzato per acquisire i dati di imaging.
- Per l'imaging, utilizzare una sorgente luminosa per eccitare le proteine a una lunghezza d'onda di 488 nm. Impostare il rivelatore per raccogliere la luce emessa a lunghezze d'onda comprese tra 490 nm e 540 nm.
- Regola l'intensità della luce di eccitazione per ottimizzare il segnale senza causare danni al tessuto cerebrale.
- Modificare i guadagni del segnale fluorescente per garantire che il rivelatore catturi un segnale chiaro e forte.
- Per evitare un rapido sbiancamento della fluorescenza, che ridurrebbe la qualità dell'imaging, regolare con precisione l'intensità della luce e il guadagno del segnale fluorescente, se necessario.
- Condurre un test del comportamento del mouse.
- Monitorare continuamente il processo di imaging durante il test comportamentale.
- Analisi dei dati
- Usa Python e GNUplot per creare una mappa termica delle prove sperimentali per una rappresentazione visiva dei dati. I codici sorgente sono forniti nel File supplementare 1.
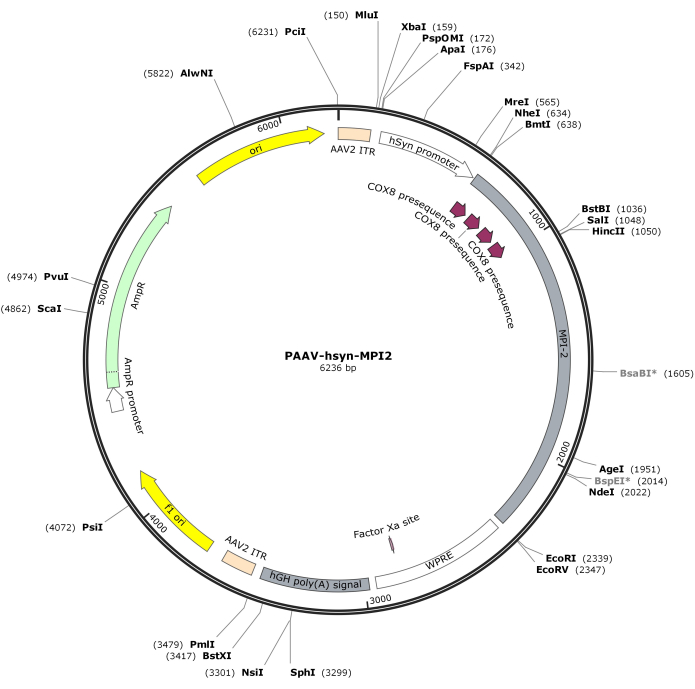
Figura 2: Mappa vettoriale di AAV-hSyn-MPI-2. Clicca qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Dopo aver costruito il plasmide CMV-MPI-1, la sua capacità di colpire i mitocondri è stata testata nelle cellule Hela utilizzando il marcatore mitocondriale Rhodamine 800 per la colorazione. Gli esperimenti di colocalizzazione hanno mostrato un alto grado di sovrapposizione tra il segnale di fluorescenza di MPI-1 e il segnale di Rhodamine 800, indicando che MPI-1 è stato localizzato con successo nei mitocondri (Figura 3).
Access restricted. Please log in or start a trial to view this content.
Discussione
La tensione della membrana mitocondriale viene mantenuta a -120-180 mV in condizioni di riposo e fluttua con i cambiamenti dello stato metabolico. Attualmente, la misurazione del potenziale di membrana mitocondriale può essere eseguita utilizzando metodi elettrofisiologici e metodi di coloranti a fluorescenza. Il clampaggio del cerotto mitocondriale richiede l'isolamento dei mitocondri e la distruzione delle strutture cellulari13. Questo approccio può portare a ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo il sostegno della National Natural Science Foundation (NSF) della Cina: JSK (32071137 e 92054103) e il finanziamento del team di ricerca scientifica e innovazione del primo ospedale affiliato dell'Università di Zhengzhou: JSK (ZYCXTD2023014).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| BamHI | Thermo | FD0054 | |
| Calcium chloride | Sigma | C4901 | |
| CCCP | Sigma | C2759 | |
| Centrifuge | eppendorf | 5430R | |
| Centrifuge (cell culture) | eppendorf | 5810R | |
| CO2 cell incubator | ESCO | 170L IR Sensor | |
| Coverslips | Glaswarenfabrik Karl Hecht GmbH & Co.KG | 92100100030 | |
| Dental adhensive resin cement | Sun medical company, LTD. | Super-Bond C&B Kit | |
| D-glucose | Sigma | G7021 | |
| DNA Ligation Kit Ver. 2.1 | Takara | 6022 | |
| Dulbecco's modified Eagle medium | Gibco | 11965092 | |
| Electric drill | RWD Instruments | 78001 | |
| Fetal bovine serum | Gibco | A5670701 | |
| Fiber optic cannula | RWD Instruments | R-FOC-L200C-39NA | |
| Fiber photometry detector | Thinker | QAXK_FPS-TC-MC-LED | |
| Fluorescence microscope | Olympus | IX83 | |
| Glass pipette (for injection) | Drummond Scientific company | 3.5" Drummond # 3-000-203-G/X | |
| HEK293t | ATCC | Cat# CRL-3216 | |
| Hela cells | ATCC | Cat# CCL-2 | |
| HEPES | Sigma | H3375 | |
| Injection pump | Drummond Scientific company | 3-000-207 | |
| Isoflurane | RWD Instruments | R510-22 | |
| Laser scanning confocal microscope | Zeiss | LSM980 | |
| MluI | Thermo | FD0564 | |
| NheI | Thermo | FD0974 | |
| Optical fibers | RWD Instruments | R-FC-L-N3-200-L1 | |
| Paraffin oil | Sangon | B500301 | |
| PCR thermal Cycler | analytik jena | Biometra Tone 96G | |
| Pentobarbital sodium | Sinopharm Chemical Reagent Co.LTD | 57-33-0 | |
| Potassium chloride | Sigma | P5405 | |
| PrimeSTAR HS DNA Polymerase | Takara | R010A | |
| Programmable micropipette puller | Sutter Instruments | P2000 | |
| Quick self-curing acrylic resin | Yamahachi | V-PINK | |
| Real-time PCR thermal Cycler | analytik jena | qTOWER³ auto | |
| Rhodamine 800 | Sigma | 83701 | |
| SalI | Thermo | FD0644 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium phosphate dibasic | Sigma | S9763 | |
| Stereotaxic apparatus | RWD Instruments | E06354 | |
| Veterinary ophthalmic ointment | Puralube | NA | |
| XhoI | Thermo | FD0694 |
Riferimenti
- Vyas, S., Zaganjor, E., Haigis, M. C. Mitochondria and cancer. Cell. 166 (3), 555-566 (2016).
- Dzbek, J., Korzeniewski, B. Control over the contribution of the mitochondrial membrane potential (ΔΨ) and proton gradient (ΔpH) to the protonmotive force (Δp): IN SILICO STUDIES. J Biol Chem. 283 (48), 33232-33239 (2008).
- O'Rourke, B., Cortassa, S., Aon, M. A. Mitochondrial ion channels: Gatekeepers of life and death. Physiology. 20 (5), 303-315 (2005).
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu Rev Cell Dev Biol. 22 (1), 79-99 (2006).
- Jin, S. M., Lazarou, M., Wang, C., Kane, L. A., Narendra, D. P., Youle, R. J. Mitochondrial membrane potential regulates PINK1 import and proteolytic destabilization by PARL. J Cell Biol. 191 (5), 933-942 (2010).
- Ly, J. D., Grubb, D. R., Lawen, A. The mitochondrial membrane potential (deltapsi(m)) in apoptosis; an update. Apoptosis. 8 (2), 115-128 (2003).
- Perry, S. W., Norman, J. P., Barbieri, J., Brown, E. B., Gelbard, H. A. Mitochondrial membrane potential probes and the proton gradient: a practical usage guide. Biotechniques. 50 (2), 98-115 (2011).
- Meng, X. -Y., et al. A sensitive mitochondrial thermometry 2.0 and the availability of thermogenic capacity of brown adipocyte. Front Physiol. 13, 977431(2022).
- Kaestner, L., et al. Genetically encoded voltage indicators in circulation research. Int J Mol Sci. 16 (9), 21626-21642 (2015).
- Yang, H. H., St-Pierre, F. Genetically encoded voltage indicators: Opportunities and challenges. J Neurosci. 36 (39), 9977(2016).
- Sepehri Rad, M., Cohen, L. B., Braubach, O., Baker, B. J. Monitoring voltage fluctuations of intracellular membranes. Sci Rep. 8 (1), 6911(2018).
- Yang, R. -Z., Wang, D. -D., Li, S. -M., Liu, P. -P., Kang, J. -S. Development and application of a mitochondrial genetically encoded voltage indicator in narcosis. Neurosci Bull. 40 (10), 1529-1544 (2024).
- Kumari, A., Nguyen, D. M., Garg, V. Patch-clamp technique to study mitochondrial membrane biophysics. J Gen Physiol. 155 (8), e202313347(2023).
- Bayne, A. N., Dong, J., Amiri, S., Farhan, S. M. K., Trempe, J. -F. MTSviewer: A database to visualize mitochondrial targeting sequences, cleavage sites, and mutations on protein structures. PLoS One. 18 (4), e0284541(2023).
- Kim, B. B., et al. A red fluorescent protein with improved monomericity enables ratiometric voltage imaging with ASAP3. Sci Rep. 12 (1), 3678(2022).
- Evans, S. W., et al. A positively tuned voltage indicator for extended electrical recordings in the brain. Nat Methods. 20 (7), 1104-1113 (2023).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon