JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
특정 인간과 동물 바이러스를 사용하여 미생물 소스 추적을위한 비용 효율적인 방법
요약
연구는 산악 표준시 도구로 제안 인간 / 돼지의 / 소 DNA 바이러스, adenoviruses과 polyomaviruses의 특정 부량에 대한 qPCR을 사용하여 물속에서 nitrates에 의해 배설물 / 소변 오염 또는 오염의 소스의 식별을위한 비용 효율적인 방법을 설명합니다.
초록
환경의 미생물 오염 중요한 건강 위험을 나타냅니다. 고전 세균 배설물 지표는 상당한 한계를 가지고 나타났습니다, 바이러스 오염의 원인에 대한 정보를 제공하지 않는 많은 불활 성화 과정 및 표준 배설물 지표에 더 저항하고 있습니다. 물 분자 assays에서 바이러스의 농도에 대한 비용 효율적인 방법의 개발은 배설물 오염의 지표와 같은 미생물의 소스 추적 (MST) 도구와 같은 바이러스의 적용을 용이하게합니다. Adenoviruses 및 polyomaviruses 인간을 포함한 척추 특정 수종을 감염 DNA 바이러스이며 지속적으로 배설물 및 / 또는 공부를 모든 분야에서 지역 소변에서 배설됩니다. 이전 연구에서 우리는 인간 adenoviruses (HAdV)과 인간의 배설물로 오염의 인덱스로 정량 PCR (qPCR)에 의해 JC의 polyomaviruses (JCPyV)의 부량을 제안했습니다. 최근, 우리는 포의 특정 부량에 대한 qPCR의 assays을 개발했습니다cine의 adenoviruses (PAdV) 및 테스트 튜브 당 10-10 게놈 사본 감성과 오염의 동물 배설물 마커로 소 polyomaviruses (BPyV). 본 연구에서는, 우리는 이러한 도구를 사용하여 물 샘플의 오염의 원인을 확인하기 위해 따라야하는 절차를 제시한다. 대표 결과 예를 들어, nitrates의 높은 수준을 보여주고 지하수의 바이러스의 분석이 나타납니다.
낮은 또는 적당히 오염된 바다에서 바이러스가 탐지되면 훨씬 작은 볼륨, 보통 시리즈의 두 농도 단계를 포함하는 프로 시저에 물을 적어도 몇 리터에서 바이러스의 농도를 필요로합니다. 이 다소 성가신 과정 및 바이러스 recoveries에서 관찰 다양성은 크게 물 샘플을 다수의 동시 처리를 방해.
우리가 이전 studie에서 개발한 한 단계 프로토콜을 적용 두 단계 절차로 인해 발생하는 병목 현상을 제거하기 위해의 및 수상 매트릭스의 다양한 적용. 열 리터 물 샘플의 산성화, 무지방 우유에 의해 응집, flocculated 물질의 중력 침강, 침전과 원심 분리를 수집, 10 ML 인산 버퍼에있는 침전물의 resuspension를 : 프로 시저가 포함됩니다. 바이러스 집중은 바이러스 핵산과 특정 adenoviruses과 관심 polyomaviruses qPCR에 의한 계량 아르의 추출에 사용됩니다. 샘플 높은 번호는 동시에이 저렴한 비용으로 집중 방법을 사용하여 분석 수 있습니다.
절차는 입욕 물, 해수 및 하천 물의 분석에 적용이 연구에서 우리는 지하수 샘플을 분석 결과를 제시했습니다. 이 높은 처리량 정량 방법, 신뢰할 수있는 간단하고, 비용 효과적입니다.
프로토콜
1. 바이러스 입자의 농도는 물 샘플에 존재
- 수집 및 물 샘플의 컨디셔닝
- 2의 최소 프로세스 제어와 같은 평면 하단에 하나 추가 샘플과 플라스틱 용기에 시료 당 10 L의 복제 수집합니다. 마지막 예제는 바이러스 입자의 알려진 양의 마약을하고 컨트롤로 사용됩니다.
참고 :이 아군 샘플 특별한 별도의 자료를 (병, 튜브 등)을 권장합니다. - conductimeter의 교정을 확인하고 필요한 경우 재조정. 한 개는 플라스틱 10 L 컨테이너에서 이전에 충분한 전도성으로 조정 수돗물을 (1.6 아래 참조)를 사용하여 부정적인 컨트롤을 준비합니다.
- 아군 샘플 : 샘플로 제어 바이러스의 표준 볼륨 (물 10 L 당 약 10 5 게놈 사본) 추가합니다. 피하 splashing 및 에어로졸에 감동하여 섞는다. 긍정적인 컨트롤은 아데노 바이러스 s의 드문 변형으로 이루어져 수HAdV - 35 또는 MS2 같은 박테리오 파지로 uch.
- 샘플 정지 재료 (모래 또는 기타 자료)의 높은 수량을 제시하는 경우 15 분 동안 퇴적물을하자. 새 용기에 물을 전송합니다.
- 1 N HCL의 추가에 의해 3.5 (± 0.1)로 물 시료의 산도를 조정합니다. 이 단계는 바이러스의 농도 중요하므로 산도가 제대로 조정되어 있는지 확인하십시오. HCL을 추가하는 동안 활발한 교반에 의해 철저하게 물을 섞는다. (참고 : 산도 3.5 추가 1 M NaOH보다 낮은 경우).
- 전도율을 조정하십시오. 예제 1,500 μS / cm 이상의 전도성이있는 경우이 단계가 필요하지 않습니다. 전도율이 1,500 μS / cm flocculated 재료의 형성 (flocs)가 보증하지 않습니다보다 낮은 경우 있도록 인공 바다 소금 (시그마)의 추가로 1,500 μS / cm로 전도율을 조정합니다. 바다 소금을 추가하는 동안 교반하여 적극적으로 섞는다.
- 뿐만 아니라 이전과 컨디셔닝 후 샘플의 산도를 기록S는 HCL의 볼륨 사용. 전도율도 산도 조정 후 기록한다. 항상 신선한 HCL 솔루션 산도 미터 및 conductimeter 전극을 소독, 10 % 나트륨 tiosulphate 솔루션 dechlorinate 그리고 마지막으로 증류수로 씻어.
- 2의 최소 프로세스 제어와 같은 평면 하단에 하나 추가 샘플과 플라스틱 용기에 시료 당 10 L의 복제 수집합니다. 마지막 예제는 바이러스 입자의 알려진 양의 마약을하고 컨트롤로 사용됩니다.
- 사전 flocculated 1% 무지방 우유의 준비 (PSM)
- 산도 미터 및 conductimeter의 교정을 확인하고 필요한 경우 재조정.
- 1 L 인공 해수에 10g 무지방 우유 분말 (Difco)를 용해하여 사전 flocculated 무지방 우유 솔루션 (1 % PSM, W / V)를 준비 (dechlorinated 수돗물과 압력솥 1 L에 인공 바다 소금의 33.3 g을 용해)과 신중하게 1N HCL로 3.5 산도를 조정. flocs가 표시되어야합니다. 24 H. 4에서 사용되거나 저장되는 직전 솔루션 ° C를 준비 dechlorination 물 100 ML 당 10 % tiosulphate 솔루션 1 ML을 사용합니다.
- 바이러스 입자의 응집 물 샘플에 존재
- 10 - L 물 샘플 1 %의 PSM 100 ML을 추가합니다.
- 바이러스가 flocs에 adsorb 수 있도록 80-10 H에 대한 샘플을 저어. 80-10 H. 후 스위치 - 오프 감동에 타이머를 사용하여
- 교반을 중지하고 80-10 H.에 대한 중력에 의해 flocs 퇴적물을하자
- flocs를 수집하고 다시 용해. 원심 분리
- 연동 펌프 플라스틱 튜브에 연결된 플라스틱 피펫을 사용하여 표면에 뜨는을 제거합니다. 아군 샘플 뜨는는 병으로 수집되어야하며 내부 절차에 따라 소독. 모든 경우에서 펠릿를 수집하지 않도록 조심해.
- 원심 분리기 병에 flocs (약 500 ML)와 침전물를 수집합니다.
- PSM의 산도 3.5의 추가에 의해 냄비 균형을.
- 4 30 분 8,000 XG에서 고속 원심 분리기에 냄비를 원심 ° C. 즉시 원심이 중지로 조심스럽게 원심에서 원심의 냄비를 제거합니다.
- 매우 gently에서 기름을 붓고 뜨는 폐기하십시오. 전염성 물질에 대한 적절한 조치를 따르십시오.
- 각각의 원심 분리기 병의 펠렛을 해산하기 위해 인산 버퍼 7 ML을 추가합니다.
- flocs가 해산되고 나면, 측정 및 10 ML의 총 볼륨에 도달 인산 버퍼를 추가합니다.
- vortexing하여 바이러스 집중을 Homogenize와 -80에서 냉동해야 깨끗한 microtubes로 10 ML을 배포 ° C 더 분석이 필요까지.
2. 핵산 추출
- 제조 업체 지침에 따라 QIAamp 바이러스성 RNA 미니 키트와 핵산 추출을 수행합니다. 이 키트는 자동 플랫폼 (예 : Qiacube, Qiagen 등)의 사용이 가능합니다.
3. 인간 adenoviruses (HAdV), JC의 polyomaviruses (JCPyV), 돼지의의 adenoviruses (PAdV)와 소 polyomaviruses의 양적 PCR (BPyV)
- 샘플의 게놈 사본 부량
- 준비TaqMan 환경 PCR 마스터 믹스 2X을 (Biosystems 응용)를 사용하여 깨끗한 별도의 영역에 qPCR 믹스. 반응은 광학 접착제 커버로 덮여 96 잘 광 반응 플레이트에서 열립니다. 마스터 믹스의 농도는 primers와 프로브는 표 1에 설명되어 있습니다.
- 일단 믹스가 나누어지는의 15μl 잘 (3.2 참조) 컨트롤을 포함하여 각에, 준비되었습니다. 타겟 또한 이후에 반응에 대한 전체 볼륨 (15μl 믹스 + 10μl 샘플 또는 표준) 25μl 것입니다.
- 별도의 영역에 샘플 (10μl)에서 핵산 extractions을 추가합니다. 중복된 각 샘플의 물을 정화에 직접 10 배로 희석 실행합니다. 접착제 커버의 일부로 샘플을 포함하고있는 우물을 커버, 다음 단계에 대한 커버의 다른 부분을 유지.
- 세중의 10 0-10 6 GC/10μl에서 DNA 표준 정지 (10μl)의 dilutions를 추가하고 전용 스탠 사용 micropipette를 사용하여dard의 DNA. 플라스미드 DNA를 파괴하고 각 사용 후에 micropipette를 청소하는 UV 방사선을 갖춘 지역의 표준을 추가하는 것이 좋습니다 좋습니다. 사전 컷 접착제 커버와 기준을 포함하고있는 우물을 커버.
참고 : 게놈 사본의 부량에 사용되는 표준 정지를 준비하기 위해서는 대상 지역은 DNA 플라스미드 및 선형에 복제해야합니다. 다음 주소에서 당신은 qPCR에 사용되는 플라스미드 DNA를 템플릿을 사용하여 표준 곡선을 만드는 방법에 대한 자세한 절차를 발견할 것이다 :
http://www.appliedbiosystems.com/support/tutorials/pdf/quant_pcr.pdf - 적절한 매개 변수를 (접착제 커버하고 각 잘 등에서 전체 볼륨의 사용을 고려) 선택 적절한 시스템에 qPCR을 수행합니다. 95시 10 분 대한 AmpliTaq 골드의 다음 활성화 ° C, 증폭의 40주기 다음과 같이 수행됩니다 : 95 ° C 15의그리고 HAdV, JCPyV 및 BPyV, 및 95에 15의 60 ° C에서 1 분 ° C, 55에서 20 ° C와 60 ° 20 PAdV를위한 C.
- 반응이 완료되면, 같은 장비의 사용자 매뉴얼에 설명되어있는 저장 데이터와 결과를 사용합니다. DNA의 금액은 필요할 때 희석 계수를 수정 후 얻은 데이터의 평균으로 정의됩니다.
- 컨트롤
- 긍정과 부정 컨트롤을 사용합니다. 검정은 혼합이 형광을 생산하지 않는 증명하기 위해 하나 이상의 비 템플릿 컨트롤 (NTC)를 포함해야합니다. 공부 샘플에 존재 억제제에 의한 잠재 효소 억제를 평가하기 위해 긍정적인 공정 제어를 실행하는 것이 좋습니다 좋습니다.
- 표준 DNA와 프로세스 제어의 두 가지 dilutions의 qPCR assays의 기록 결과를. assays의 감도와 효율성에 관한 품질 관리 (QC) 프로그램에 대한 제어 차트를 준비하는 결과를 사용합니다.
- 의 확인결과
- 긍정적인 결과는 더욱 긴장의 뉴클레오 티드 시퀀스에 대한 추가 데이터를 생산, amplicons의 중첩된 - PCR 및 염기 순서를 1,5,6,7,9,12를 감지를 사용하여 확인할 수 있습니다.
절차 설명에 따라 인간과 동물의 바이러스가 감지되어 있고 해수욕장 바다, 해수와 강물 2,3의 계량. 대표 예를 들어, nitrates의 높은 수준을 보여주고 지역에서 지하수 샘플이 오염의 근원을 정의하는 평가했다. 만 리터 물 샘플은 스페인에서 동북아 지역의 시골 지역에 4 개의 우물에서 수집되었다. 다섯 공정 제어로 사용 인간 아데노 바이러스 2 씨앗을 각각 잘되는 한 복제에 수집된 복제합니다. 샘플은 그림 1에 나타내는 프로토콜에 따라 처리했다. 넷 (값 7.7을 의미 PAdV에 대한 긍정적인 결과를 보여주 공부 네 개의 사이트 중 하나에서 분석 복제샘플링 사이트를 둘러싸고있는 지역에서 돼지 slurries의 존재과 관련이있을 것이며 지하수에 nitrates (표 2)의 소스로 지저분한 돼지의 오염을 지원 4x10이 GC / L).
4. 대표 결과 :
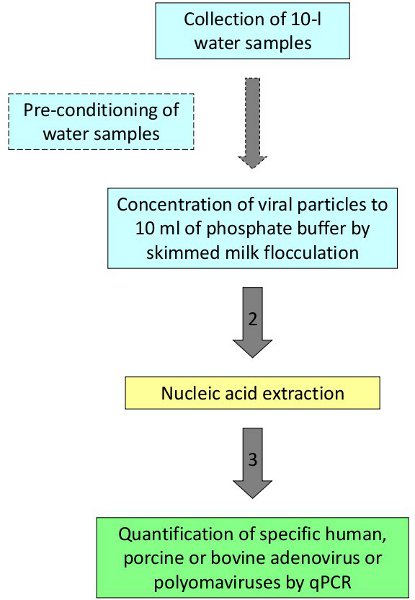
그림 1. 물 속에 바이러스의 감지 및 부량 절차.
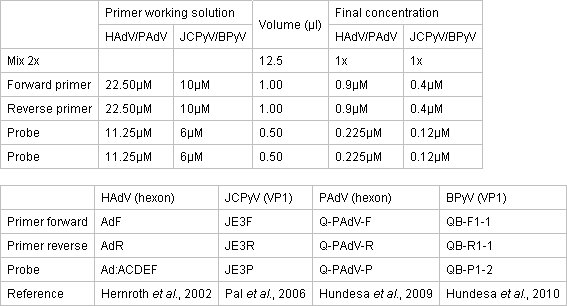
primers와 qPCR의 assays에 대한 프로브의 표 1. 농도.

표 2. 감지 및 지하수 샘플에서 동물과 인간의 adenoviruses과 polyomaviruses의 부량.
의 N 번호 분석 복제
긍정의 % 비율 복제
(-) 이외의 발견
토론
재현성, 신뢰성, 간단하고 비용 효율 : 설명 절차는 일상적인 환경과 공중 보건 실험실의 피팅 방법에 대한 조건을 충족합니다. 프로토콜은 간단하다, 그러나 그것은 신중하게 따라야합니다. 응집에 대한 교반 시간이 크게 (예를 들어 미만 5 시간) 감소 경우가대로 인공 해수 염분의 요청 농도를 추가하지 않고 샘플의 낮은 전도성이 크게 바이러스의 회복을 줄일 것입니다.
공개
두 개의 특허 출원이 PAdV과 BPyV의 부량을위한 프로토콜의 지적 재산권을 보호하기 위해 2009 년 제기했다.
감사의 말
이 작품은 부분적으로 유럽 연합 (EU) 연구 프레임 워크 7 투자 프로젝트 (계약 번호 513648) VIROBATHE, VIROCLIME (계약 번호 243923로, 스페인 정부 "정부의 드 Educación Y Ciencia"(프로젝트 AGL2008-05275-C01/ALI)에 의해 지원되었다 )과 물의 카탈루냐어 기관, Agència Catalana DE L' Aigua (ACA), Departament 드 제어 내가 Millora dels Ecosistemes 수상하여. 개발 연구 기간 동안 마르타 Rusiñol는 카탈루냐어 정부 "AGAUR"(FI - DGR)의 동료했다.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 |
| 고속 원심 분리기 (8,000 XG) | Berckman 보습 바로 앞에 달린 풀베는 날 | 아벤티 J - 20XP | |
| 산도 미터, 온도계 및 conductimeter | Afora | LPPC3003 | |
| 플라스틱 튜브 1백-2백센티미터 길이 | Deltalab | 350,059 | |
| 멸균 일회용 pipettes 졸업 | Labclinics | PN10E1 | |
| 1.5 10-15 ML (Eppendorf, 팔콘, 등) 살균 플라스틱 튜브 | Afora | KA298/00 | |
| 원심 분리기의 냄비 (500 ML) | 피셔 과학 | SE5753512 | |
| 자기 stirrers 및 자석 (당샘플) | 피셔 과학 | 10,510 | |
| 자기 stirrers의 사용을 허용하는 평면 바닥 데 유리 또는 플라스틱 용기 | Deltalab | 191,642 | |
| 뜨는 (또는 물 제트 진공 펌프)를 제거 연동 펌프 | 왓슨 - Marlow | 323E / D | |
| 타이머 스위치 - 오프 80-10시간 후 감동에 | Deltalab | 900,400 | |
| 염산 (1N과 0.1N) | Panreac | 141020.1611 | |
| 수산화 나트륨 (1N) | Panreac | 131687.1211 | |
| 인공 해수 바다 소금 | 시그마 | S9883 | |
| 무지방 우유 (SM) | Difco | 232,100 | |
| 인산 버퍼 산도 75 | 1시 2분 V / V 살균 나 2 HPO 4 0,2 M과 산도 7.5에 아니 2 PO 4 0,2 M | ||
| Thiosulphate | Panreac | 121879.1209 | 물에 10 % 용액을 만들어 |
| QIAamp 바이러스성 RNA 미니 키트 | Qiagen | 52,904 | |
| 96 - 웰 광 반응 플레이트 (500 세대) | 응용 Biosystems | 43426659 | |
| 광학 접착제 커버 (100 세대) | 응용 Biosystems | 4,311,971 | |
| TaqMan 환경 PCR 마스터 믹스 2 배 | 응용 Biosystems | 4,396,838 |
참고문헌
- Bofill-Mas, S., Clemente-Casares, P., Major, E. O., Curfman, B., Girones, R. Analysis of the excreted JC virus strains and their potential oral transmission. J. Neurovirol. 9 (4), 498-507 (2003).
- Bofill-Mas, S., Albinana-Gimenez, N., Clemente-Casares, P., Hundesa, A., Rodriguez-Manzano, J., Allard, A., Calvo, M., Girones, R. Quantification and stability of human adenoviruses and polyomavirus JCPyV in wastewater matrices. Appl. Environ. Microbiol. 72 (12), 7894-7896 (2006).
- Calgua, B., Mengewein, A., Grunert, A., Bofill-Mas, S., Clemente-Casares, P., Hundesa, A., Wyn-Jones, A. P., López-Pila, J. M., Girones, R. Development and application of a one-step low cost procedure to concentrate viruses from seawater samples. J. Virol. Methods. 153 (2), 79-83 (2008).
- Hernroth, B. E., Conden-Hansson, A. C., Rehnstam-Holm, A. S., Girones, R., Allard, A. K. Environmental factors influencing human viral pathogens and their potential indicator organisms in the blue mussel, Mytilus edulis: the first Scandinavian report. Appl. Environ. Microbiol. 68, 4523-4533 (2002).
- Hundesa, A., Bofill-Mas, S., de Motes, M. a. l. u. q. u. e. r., Rodriguez-Manzano, C., Bach, J., Casas, A., M, ., Girones, R. Development of a quantitative PCR assay for the quantitation of bovine polyomavirus as a microbial source-tracking tool. J. Virol. Methods. 163 (2), 385-389 (2010).
- Hundesa, A., de Motes, M. a. l. u. q. u. e. r., Albinana-Gimenez, C., Rodriguez-Manzano, N., Bofill-Mas, J., Suñen, S., E, ., Girones, R. Development of a qPCR assay for the quantification of porcine adenoviruses as an MST tool for swine fecal contamination in the environment. J. Virol. Methods. 158 (1-2), 130-135 (2009).
- Hundesa, A., de Motes, M. a. l. u. q. u. e. r., Bofill-Mas, C., Albinana-Gimenez, S., N, ., Girones, R. Identification of human and animal adenoviruses and polyomaviruses for determination of sources of fecal contamination in the environment. Appl. Environ. Microbiol. 72 (12), 7886-7893 (2006).
- Layton, B. A., Walters, S. P., Lam, L. H., Boehm, A. B. Enterococcus species distribution among human and animal hosts using multiplex PCR. J. Appl. Microbiol. 109, 539-547 (2010).
- de Motes, M. a. l. u. q. u. e. r., Clemente-Casares, C., Hundesa, P., Martín, M., Girones, R. Detection of bovine and porcine adenoviruses for tracing the source of fecal contamination. Appl. Environ. Microbiol. 70 (3), 1448-1454 (2004).
- Pal, A., Sirota, L., Maudru, T., Peden, K., Lewis, A. M. Real-time PCR assays for the detection of virus-specific DNA in simples with mixed populations of polyomaviruses. J. Virol. Methods. 135 (1), 32-42 (2006).
- Stapleton, C. M., Kay, D., Wyer, D. a. v. i. e. s., Watkins, C., Kay, J., McDonald, C., Porter, A. T., J, ., Gawler, A. Evaluating the operational utility of a Bacteroidales quantitative PCR-based MST approach in determining the source of faecal indicator organisms at a UK bathing water. Water Res. 43, 4888-4899 (2010).
- Wang, J., Horner, G. W., Keef, O., S, J. Detection and molecular characterization of bovine polyomavirus in bovine sera in New Zealand. N. Z. Vet. J. 53, 26-30 (2005).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유