Method Article
특이적이고 효율적인 단백질 고갈을 달성하기 위한 튜닝 분해
요약
여기서, β-est AID 시스템을 사용하여 효모 사카로마이세스 세레비시아에 관심 있는 단백질을 효과적이고 구체적으로 고갈시키기 위한 프로토콜을 제시한다.
초록
식물 보조 인 결합 수용체, TIR1, 오신의 존재에 특정 보조 유도 성 degron (AID) 모티프를 포함하는 단백질을 인식, 분해를 대상으로. 이 시스템은 많은 비 식물 진핵생물에서 악용되며, 따라서 AID 모티브로 태그된 표적 단백질이 auxin 첨가시 저하된다. TIR1 표현식의 수준은 매우 중요합니다. 과도한 발현은 보조가 없는 경우에도 AID 태그가 지정된 단백질의 분해를 유도하는 반면, 낮은 발현은 느린 고갈로 이어집니다. β-estradiol 유도성 AID 시스템은 β-estradiol 유도성 프로모터의 제어 하에 TIR1의 발현과 함께 생성되었다. TIR1의 수준은 보조 추가하기 전에 β-estradiol로 배양 시간을 변경하여 튜닝할 수 있습니다. 이 프로토콜은 AID 시스템을 사용하여 표적 단백질을 빠르게 고갈하는 방법을 설명합니다. 적절한 β-estradiol 인큐베이션 시간은 표적 단백질의 풍부에 달려 있다. 따라서 효율적인 고갈은 보조 와 독립적 인 고갈을 최소화하는 최적의 타이밍에 달려 있습니다.
서문
온도에 민감한 돌연변이와 같은 조건부 돌연변이는 필수 단백질 연구를 위한 강력한 도구로, 허용되는 조건하에서 세포 성장을 허용하지만 비관적 조건하에서 기능 의 손실을 일으킵니다. 그러나, 세포 대사는 결함을 유도하는 데 필요한 성장 조건의 변화에 의해 심각하게 교란 될 수 있으며 또한 오프 타겟 효과를 만들 수 있습니다. 관심 있는 단백질이 조건부 1또는 그 발현이 작은 분자의 첨가에 의해2,3조절되는 여러 가지 방법이 개발되었다. 이 프로토콜은 보조 및 보조 유도성 다그론(AID) 시스템을 사용하여 표적 단백질을 효율적으로 고갈시킵니다.
AID 시스템은 식물에서 기원을 가지고 있는데, 여기서 보조(이 프로토콜인돌-3-아세트산(IAA)가 사용됨)은 SCF U3 유비퀴틴 리가제 복합체의 일원인 TIR1과 보조/IAA 단백질의 상호작용을 자극한다4. SCF 복합 상호 작용은 aux/IAA 패밀리 단백질의 폴리유비퀴틴화를 일으켜 프로테아좀5,6에의한 분해를 초래합니다.
이 시스템은 이전에 효모 세포에서 오리자 사티바 (osTIR)에서 TIR1 단백질을 발현하여 효모 사카로마이스 세레비시아7,8에 사용하기 위해 적응되었으며, 여기서 내인성 효모와 상호 작용할 수 있습니다. SCF 복합체. 관심 있는 단백질은 Aux/IAA 단백질 IAA17의 모티브로 태그가 지정되어 분해를 목표로 합니다. IAA17의 기능성 잘림은 나중에 개발되었다,와 같은 AID * 8,9,10,애기장대에서 43 아미노산 auxin 에민감한 모티프를 포함 탈리아절 IAA17, 에피토프 태그를 가능하게 검색.
이 시스템은 초기에 효모7,8에 사용하기 위해 효모 GAL 프로모터로부터 osTIR1 단백질을 발현시켰다. 발현은 단독 탄소 공급원으로 갈락토제와 함께 성장 배지로 이동해야 하며, 이는 불행히도 세포 대사에 대한 광범위한 변화와 함께 디오틱 한 변화를 초래한다11. 한편, TIR1의 구성발현은 발현 수준이 높은 경우 보조/IAA12가 없는 상태에서 표적 단백질의 분해를 유발할 수 있는 반면, 낮은 TIR1 발현은 비효율적인 고갈을 야기하는 것으로 보고되었다. β-est AID라는 개선된 AID 시스템은 osTIR이 세포 대사에 최소한의 영향을 미치면서 표적 단백질에 맞게 조정할 수 있는 유도성 프로모터의 통제하에 있는 개발되었습니다. 이를 달성하기 위해, VP16 바이러스 전사 활성제가 에스트로겐 수용체 및 4개의 Zn 핑거 DNA 결합 도메인(DBD)에 융합되는 인공 전사 인자(ATF)가 구성되었다. β-estradiol(에스트로겐)이 존재할 때, ATF는 핵에 진입하여 프로모터(Z4EVpr)에 결합하여 osTIR 전사를 유도할 수 있다(Z4EVpr)13,12.
osTIR 발현은 일반적으로 β-estradiol12를첨가한 후 약 20분 후에 검출할 수 있다. 그러나, auxin첨가 전에 고갈을 피하면서, 태그된 단백질의 효율적인 고갈을 달성하기 위해 osTIR 발현의 최적 지속 기간은 각 표적 단백질에 대해 경험적으로 결정될 필요가 있다. 이 사전 인큐베이션에 대한 대략적인 시간은 사카로마이세스 게놈 데이터베이스(SGD https://www.yeastgenome.org/)의 풍부한 값으로부터 추정될 수 있다. 도1에서 볼 수 있듯이, 풍부한 단백질인 Dcp1(2880 내지 4189 분자/세포)은 β-estradiol을 사용하여 40분 동안 사전 배양이 필요하며, 보조-독립적인 고갈이 관찰되지 않는다. 훨씬 덜 풍부한 단백질, Prp2 (172 받는 사람 211 분자/세포), 강하게 사전 배양의 단지 20 분 후 고갈. 이 초기 예상 시간 전후10~20분 후의 두 개의 추가 사전 배양 시간을 테스트하는 것이 좋습니다(20분은 권장되는 최소 시간임). 최적의 사전 인큐베이션 시간은 보조를 첨가하기 전에 표적 단백질이 고갈되지 않은 시간이며, 일단 auxin이 첨가되면 고갈이 허용가능하거나 단백질 수준이 가능한 최소에 근접하는 시간입니다. 그래서, 도1b에서, Prp22에 대한 사전 배양의 30 분, 수준은 auxin 추가 후 많은 10 분 감소하지 않았습니다. 추가 고갈이 거의 없는 IAA로 40분 간 배양하고 15분 동안 배양하는 경우, 특히 비보조물의 증거가 있기 때문에 10분 이상 인큐베이션하거나 30분 이상 사전 인큐베이션하는 데 아무런 이점이 없습니다. 40 분에서 의존 고갈. 사전 배양 40분(단백질 수준이 auxin 첨가 전에 약 100%인 마지막 지점)이 있는 Dcp1의 경우, 15-20분의 고갈이 허용됩니다. 세포 대사에 대한 이차적 효과를 줄이기 위해 고갈 시간을 가능한 한 짧게 유지하는 것이 좋습니다14.
이 논문은 auxin을 추가하기 전에 고갈없이 IAA 첨가 시 빠른 표적 단백질 고갈을 달성하기 위해 osTIR 발현을 위한 β-estradiol 인큐베이션의 타이밍을 최적화하여 β-est AID 시스템을 사용하는 방법을 보여줍니다.
프로토콜
참고: 그래픽 요약은 그림 2를 참조하십시오.
1. 변형 준비
-
ura3-strain을 사용하여, β-est AID 시스템(즉, β-estradiol 반응성 전사 인자(ATF) 및 osTIR을 코딩하는 유전자)를 도입하고 AID*가 표적 단백질에 태그를 지정합니다(절차의 요약을 위한 도 3 및 표 1 참조).
- 15 pZTRK (G418 저항 마커) 또는 pZTRL(LEU2 마커) 플라스미드 (효모 유전 자원 센터에서 사용 가능) ura3- 효모 균주로 변환하거나 플라스미드를 템플릿으로 사용하여 게놈용 PCR 제품을 생산합니다. 통합.
- PCR은 플라스미드 pZTRK 또는 pZTRL 중 어느 곳에서고 고충실도 폴리머라제를 사용하여 ATF(플라스미드 맵에 Z4EVATF 표시)와 osTIR을 증폭시다. 유전체 영역에 대한 상동성과 함께 50~ 60 염기 3' 확장을 가진 프라이머를사용하여, 상동 재조합(16)에 의해 직접 통합한다. 두 성분의 게놈 통합은 별도로 또는 함께, 프라이머 및 조건에 대한 표 1을 참조하십시오.
참고: 균주 pZ4EV-NTR1은 이미 게놈에 통합 된 구성 요소를 가지고 있습니다 (효모 유전 자원 센터에서 사용할 수, 일본). - 표적 단백질이 Longtine 절차17을 사용하여 태그가 지정된지 확인합니다(도 3b 및 표 1참조).
- Β-estradiol 및 IAA가 없는 균주에 대한 성장 분석을 수행하여 AID* 태그가 성장에 영향을 미치는지 확인하고 1.5단계에서 사용하기 위한 성장 속도를 예측합니다.
2. 고갈에 대한 일반적인 절차
- 모든 샘플을 수집하는 데 필요한 배양량 정도를 계산합니다. 예를 들어, 0.8의 OD600에서 의 배양 10 mL은 단일 샘플에 대한 단백질, RNA 및 DNA 추출에 충분하므로 6개의 샘플에 대해, 적어도 60 mL의 배양이 필요하다.
- 야간 배양으로부터, OD600 0.1 에서 0.1 에서 0.2에 충분한 새로운 배양을 설정하고 30°C에서 성장하도록 방치한다. 다른 성장 조건을 사용할 수 있지만 YPDA와 같은 풍부한 매체를 사용하는 것이 좋습니다.
효모 추출물 10g 펩톤 (것)과 같은 20 g 포도 당 20 g 아데닌 황산염 40 mg H2O ~ 1L
참고: 오토클레이브 또는 필터 멸균; 필터 살균은 샘플 수집에 사용되는 메탄올에서 침전물 오토클레이브에 의해 생성 된 펩티드 / 설탕 복합체로 바람직하다. - 샘플을 받을 준비를 합니다.
- 메탄올의 의도된 샘플 부피의 30~50%를 튜브에 넣습니다. 예를 들어, 10 mL 샘플을 채취해야 하는 경우, 메탄올 5 mL을 15 mL 팔콘 튜브에 넣고 튜브를 단단히 닫습니다. 일단 닫히면 튜브에 라벨을 붙이고 드라이 아이스 또는 -80 °C에서 식힙니다.
주의 사항: 메탄올을 연기 후드에 분배합니다. - 샘플을 장기간 보관하기 위해 1.5 mL 튜브에 라벨을 부착하고 얼음에 두어 식힙니다.
- 얼음에충분히 H2 O (샘플 당 적어도 1 mL)를 식힙니다.
- 메탄올의 의도된 샘플 부피의 30~50%를 튜브에 넣습니다. 예를 들어, 10 mL 샘플을 채취해야 하는 경우, 메탄올 5 mL을 15 mL 팔콘 튜브에 넣고 튜브를 단단히 닫습니다. 일단 닫히면 튜브에 라벨을 붙이고 드라이 아이스 또는 -80 °C에서 식힙니다.
- 문화의 성장을 예측합니다. 시료를 수집하기 위한 표적 OD는 약 0.7~0.8이지만, 사전 배양 단계(osTIR를 유도하기 위한 β-estradiol을 사용하는 배양)는 시료가 될 때까지 배양이 대략 적정 OD에 도달할 수 있도록 조기에 시작해야 합니다. 수집.
참고: 이러한 시작 OD를 추정할 수 있도록 실험에 사용되는 조건에서 성장 곡선을 수행하는 것이 바람직하다. - 사전 배양의 시작을 위한 표적 OD에 도달하면, 샘플(보통 10 mL)을 차가운 메탄올을 함유하는 미리 준비된 튜브로 채취한다. 잠시 뒤집어 서 드라이 아이스에 섞어서 다시 놓습니다.
참고: 시료는 약 5분 후에 물 얼음으로 옮겨질 수 있으며, 편리한 경우 얼음으로 옮길 수 있다. - 즉시 β-estradiol, 배양의 1 μL/mL (10 μM의 최종 농도)를 추가하십시오; 샘플을 수집하고 β-estradiol을 추가하는 데 걸린 시간을 줄이기 위해 사용하기 위해 파이펫에서 미리 측정된 β-estradiol을 갖습니다. 활발하게 소용돌이에 의해 빠르게 혼합.
- 전과 같이 배양을 계속 성장시키다(단계 2.2), 배양(이것은 "사전 배양 단계") 최적의 시간 동안 β-estradiol을 사용한다(최적의 사전 배양 시간의 측정을 위해 도 1참조).
- IAA(보조)를 추가할 준비를 합니다. 2.10단계(즉, 배양 mL당 IAA의 0.5 μL)에 필요한 IAA의 부피를 취한다. 이렇게 하면 단계 2.20속도가 빨라집니다.
- 2.5단계로 샘플을 수집합니다.
- 즉시 2.8단계에서 제조된 바와 같이 배양물의 IAA 0.5 μL/mL을 최종 농도750 μM에 첨가한다. 활발하게 소용돌이에 의해 빠르게 혼합.
- 실험 설계에 따라 2.5단계로 샘플을 수집합니다. 단백질이 안정적으로 고갈될 것으로 예상되는 시점에서 단일 샘플, 또는 고갈의 시간 과정에서 여러 샘플. 예를 들어, 5분 간격은 타이밍에 편리하며 다양한 단백질 수준을 제공합니다. 그림1과 같이 최적화 전략은 적절한 시간을 나타냅니다.
- 샘플을 처리합니다.
- 샘플을 얼음 위에 놓습니다. 샘플이 고정되지 않았는지 확인합니다. 그들이있는 경우, 부드럽게 온도가 로컬로 상승하지 않도록 지속적으로 반전, 손에 따뜻하게.
참고: 이것은 시료의 온도를 평가 할 수 있으므로 손에서 가장 잘 수행되며 항상 차가운 느낌을 가져야합니다. 얼음 위에 놓습니다. 이것은 일시 정지 지점이 아닙니다 - 모든 샘플이 액체가되면 다음 단계로 진행하십시오. - 모든 시료가 수집되고 더 이상 동결되지 않으면 3,500 x g에서 2분 동안 회전합니다(가능하면 4°C에서).
- 메탄올/미디엄 믹스를 붓고 얼음 위에 다시 놓습니다. 모든 액체가 제거되지 않은 경우 걱정하지 마십시오.
- 1 mL의 얼음 차가운 H2 O(단계 2.3.3에서)에서 세포 펠릿을 다시 중단하고 라벨이 붙은 1.5 mL 튜브 (단계 2.3.2에서 제조)로 얼음상에 전달합니다.
- >15,000 x g에서 짧게 돌리면 세포를 다시 펠렛하고 얼음 위에 다시 놓고 액체를 제거합니다.
- 포부로H2 O를 제거합니다. 세포 펠릿은 장기 저장을 위해 -20°C, 또는 -80°C에서 보관될 수 있다.
- 샘플을 얼음 위에 놓습니다. 샘플이 고정되지 않았는지 확인합니다. 그들이있는 경우, 부드럽게 온도가 로컬로 상승하지 않도록 지속적으로 반전, 손에 따뜻하게.
- 서양 얼룩 분석18에의해 단백질이 고갈된 수준을 확인합니다.
참고: 희소한 RNA 종은 더 많은 견본 양을 요구할 지도 모르지만 충분한 단백질19 및 핵산은 대부분의 목적을 위해 단 하나 세포 펠릿에서 추출될 수 있습니다.
결과
고갈의 대표적인 예는 그림1에 표시됩니다. 이 그림에 제시된 3개의 실험은 Prp2, Prp22 및 Dcp1 단백질의 고갈을 위한 최적화 실험이었다. 낮은 풍부도, 스플리케소말 Prp2 및 Prp22 단백질은 β-estradiol을 가진 40 분 사전 배양 후에 20% 이하로 고갈되고 15 분 auxin으로. 배양 전 시간이 길수록 더 빠른 고갈을 초래할 뿐만 아니라 보조를 첨가하기 전에 바람직하지 않은 단백질 고갈을 보일 수도 있습니다. 이에 비해, 더 풍부한 Dcp1은 동일한 치료로 약 30%로 고갈되었지만, 60분의 사전 배양으로 인해 동일한 보조제 처리로 13%까지 고갈되었고, 보조제가 첨가되기 전에 고갈되는 대가를 치렀다. β-estradiol을 가진 사전 배양의 50 분 및 auxin를 가진 15 분은 더 짧은 시점에서 유사한 결과를 달성했을 것이고 그래서 더 최적이었을 것입니다.
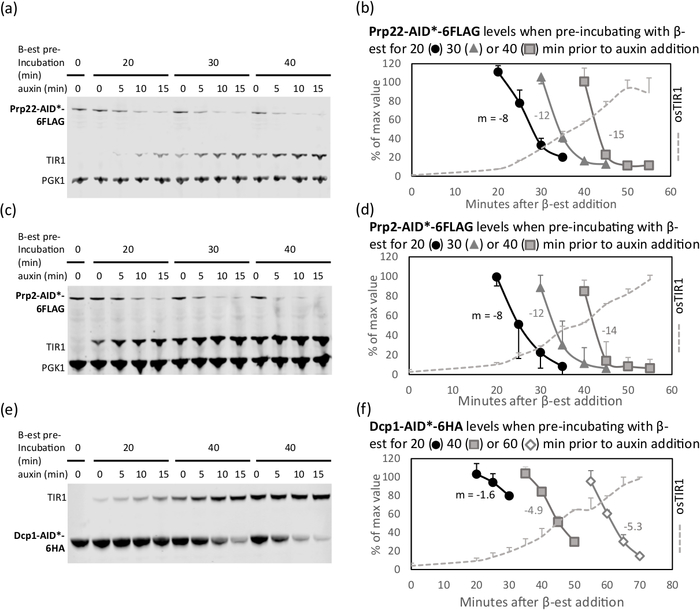
도 1: 고갈속도는 β-estradiol 사전 배양 의 지속 시간을 조절하여 조정할 수 있다. 대상 단백질의 서쪽 얼룩18: (a 및 b)Prp22-AID*-6FLAG, (c 및 d)Prp2-AID*-6FLAG, 및 (e 및 f)Dcp1-AID*-6HA, β-est(β-est)로 미리 배양된 배양물에서 20, 30, 40 또는 60 분 전에 auxin 추가12. 동일한 양의 총 단백질을 각 레인에 적재하했습니다. Pgk1은 Pgk1 및 Dcp1이 공동 마이그레이션하는 패널 e를 제외하고 시각적 로딩 컨트롤로 감지됩니다. 패널에서단백질 밴드의 정량화 a, c 및 e는 각각 패널 b, d및 f에 도시된다. 고갈률의 척도로서, 경사(m)는 각 곡선의 선형 단면(초기 값의 100%에서 30%까지)에 대해 계산되었습니다. 최적의 사전 배양 시간은 단백질 수준이 여전히 유도되지 않은 수준 (100%)에 가까운 시간입니다. 그리고 고갈의 후속 속도는 빠릅니다. Dcp1 (f)의 경우, 단백질이 보조제의 부재에서 분해되기 시작했기 때문에 사전 배양 60 분은 너무 길고, 단백질이이 시간 과정에서 현저하게 고갈되지 않기 때문에 20 분은 너무 짧습니다. 40분 간 배양 후, 15분 동안 단백질이 약 70% 고갈되어 사용할 수 있으며, 20분은 추가 고갈을 초래할 수 있지만, 이차 효과도 발생할 수 있습니다. 오차 막대는 두 생물학적 복제물의 표준 편차를 나타냅니다. 각 실험에 대해 하나의 대표적인 얼룩이 도시됩니다. 이 그림은 이전 발행물9에서 파생됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
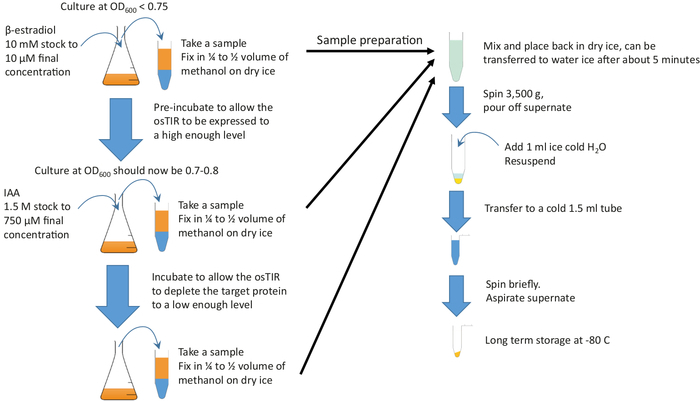
그림 2: 그래픽 요약. 사전 배양을 시작하기 위해 풍부한 배지와 필요한 온도에서 성장하는 충분한 배양물에 β-estradiol을 추가합니다. 고갈을 시작하기 위해 IAA (보조)를 추가하기 전에 필요한 사전 배양 시간 동안 성장을 계속하십시오. 사전 배양 및 고갈 시간은 고갈되는 단백질에 따라 달라지지만, 사전 배양 시간은 종종 20-60 분의 범위이며 고갈 시간은 일반적으로 10 내지 20 분의 순서로 진행됩니다. g 고갈. 이 견본은 펠릿하고 저장하기 전에 차가운 메탄올에서 급속하게 고정됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
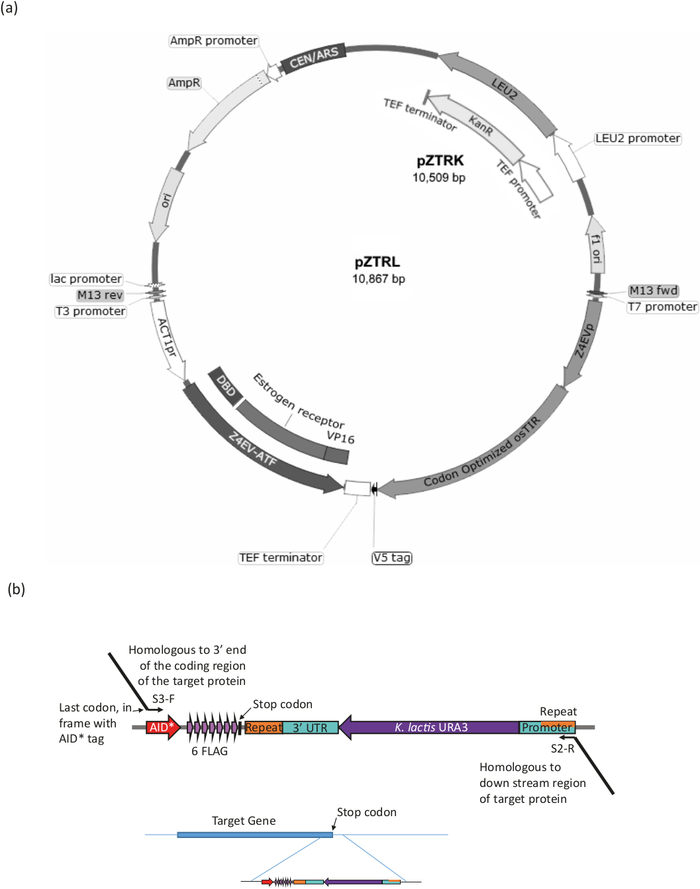
그림 3: B-est 시스템의 스트레인 생성. (a) AID* 시스템으로 효모 균주를 생성하기 위해, pZTRL(LEU2) 또는 pZTRK(카나마이신(G418) 저항성) 중 하나, 플라스미드가 균주에 도입되어야 하거나, 대안적으로, ATF 및 osTIR을 게놈내로 삽입할 수 있다. 3엔드 프라이머에서 PCR에 의해 생성된 단편의 상동 재조합(그림 3b 및 표 1참조). (b) 표적 단백질의 C 말단 태깅은 플라스미드 pURA3-AID*-6FLAG(pURA3_AID*-6HA는 태그에서만 다르며 정확히 동일한 방식으로 취급될 수 있음)의 적절한 부위의 PCR 증폭에 의해 달성되며, 롱틴 프라이머 S3-F및 S2-R은 표적 단백질의 3' 말단까지 3' 확장이 상동한다. 정방향 프라이머 확장은 AID* 태그가 시작되는 프레임에 마지막 아미노산 코돈이 포함되어야 하며 정지 코동을 포함해서는 안 됩니다. 역방향 프라이머 확장은 코딩 영역의 다운스트림 영역이어야 합니다. 일단 게놈으로 삽입되면, URA3 마커를 분실한 세포 (마커의 양 단부에서 발견되는 동일한 영역 사이 상동 재조합에 의해)는 5-FOA로 성장에 의해 선택될 수 있으며, 이는 URA3 세포를 반대선택한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| a. 프라이머 시퀀스 | |||||||
| 대상 | 위치 | 뇌관 | 이름 | 시퀀스 | Tm(°C) | ||
| pZTRL | 516 | F | pZTRL_F | <-지역 ->GCGACAGCATCACCGACTTCG | 61.23 | ||
| 7897년 | R | pZTR_R | CGCCGCCTACCTTGCAGA & 상동성의 지역 (RC)-> | 61.30 | |||
| pZTRK | 9154 | F | pZTRK_F | <-지역 ->ACGTTGAGATATTATTGCTTGCACC | 59.40 | ||
| 5897년 | R | pZTR_R | CGCCGCCTACCTTGCAGA & 상동성의 지역 (RC)-> | 61.30 | |||
| pURA3-AID*-6FLAG 또는 pURA3_AID*-6HA | F | S3-F | 상동성의 지역 ->CGTACGCTGCAggTCGAC | 59.21 | |||
| R | S2-R | ATCGATATTCGAGCTCG & 상동성의 지역 (RC)-> | 52.76 | ||||
| pZRTL/K는 β-est AID 시스템을 증폭시키는 것입니다. | |||||||
| 대상 단백질에 태그를 지정하는 HD3-AID*-6FLAG/6HA로 AID*와 에피토프 태그를 증폭합니다(론틴 시술) | |||||||
| 상동성의 지역 -> | 시스템을 삽입할 측면 영역과 상동 영역입니다. 이 영역이 길수록 수정이 성공할 가능성이 높습니다. 50 - 100 염기 권장. | ||||||
| 상동성 (RC)->> | 시스템이 삽입될 측면 영역과 상동영역은 역보수를 사용해야 합니다. 위와 같이 이 영역이 길수록 좋습니다. | ||||||
| Tm(°C) | 50 mM NaCl을 가진 %GC 방법을 사용하여 Tm | ||||||
| b. PCR 믹스 | |||||||
| 구성 요소 | 볼륨(μl) | ||||||
| 템플릿 | <10 | ||||||
| NEB 푸시온 HF 버퍼 (5x)* | 100개 | ||||||
| 포워드 프라이머 100 μM | 2.5 | ||||||
| 리버스 프라이머 100 μM | 2.5 | ||||||
| dNTPs 각 10 mM | 10개 | ||||||
| H2O | 최대 500 | ||||||
| * NEB 푸시온 GC 버퍼 (5x)도 사용할 수 있지만 바람직하지 않습니다. | |||||||
| 이 혼합물을 50 μl의 10 튜브로 나누어 각각 혼합하고 표 1 c로 PCR을 수행합니다. | |||||||
| 아가로즈 젤을 실행하여 PCR이 작동했는지 확인 | |||||||
| 모든 성공적인 반응을 하나의 튜브와 에탄올 침전물로 결합 | |||||||
| PCR에서 생산한 모든 재료로 효모를 변형시킵니다. | |||||||
| c. PCR 조건 | |||||||
| 단계 | 온도(°C) | 시간 | |||||
| 초기 변성 | 98세 | 30s | |||||
| 25-35 사이클 | 변성 | 98세 | 10s | ||||
| 어닐 (것)과 같은 | 45-60 | 20s | |||||
| 확장 | 72세 | 30 s/kb | |||||
| 최종 확장 | 72세 | 약 10분 | |||||
| 개최 | 8개 | ||||||
| 론틴 프라이머 세트(S3-F 및 S2-R)의 경우 45°C에서 어닐및 pZTRL/K 프라이머의 경우 60°C | |||||||
| 론틴 PCR의 경우 3분, pZTRL/K의 경우 3분 연장 | |||||||
표 1: 프라이머 서열, PCR 혼합 및 PCR 조건.
토론
잘 최적화된 프로토콜은 표적 단백질의 신속하고 효율적인 고갈을 생성할 수 있다. β-estradiol을 가진 대략적인 사전 배양 시간을 결정하는 것은 중요합니다, 이것은 고갈의 재현성을 증가하기 때문에, 그러나 사전 배양 시간에 있는 작은 변이는 허용될 수 있습니다. 다른 한편으로는, 단백질 수준이 아주 급속하게 감소하기 때문에, 주의는 auxin 추가 후에 타이밍으로 주의해야 합니다.
이 접근법의 장점은 β-estradiol 및 IAA 인큐베이션 시간과 사전 배양 시간의 다양한 조합에 의해 조정된 고갈이 달성될 수 있다는 것입니다. 예를 들어, 원하는 경우, 표적 단백질은 배양 전 시간을 감소시킴으로써 더 느리게 고갈될 수 있다.
β-est AID 시스템은 OsTIR이 구성적으로 표현되는 시스템에 비해 특정 한 이점을 제공합니다. 예를 들어, 표적 단백질이 생존에 필수적인 경우, osTIR의 조절된 발현은 표적 단백질의 조기 고갈을 피할 수 있다. 더욱이, osTIR의 발현은 표적 단백질의 풍부성과 그 저화에 대한 감수성에 맞게 조정될 수 있으며, 고갈은 빠르거나 느릴 수 있다. 두 개의 작은 분자 이펙터, β-estradiol 및 auxin, 여기에 사용되는 조건하에서 효모 대사를 교란하지 않으며, 라파마이신과는 달리, 앵커 어웨이 시스템에 사용되는1.
일부 단백질을 태그하면 기능이 저하되며, 이는 표적 고갈 시스템에 문제가 됩니다. 이 경우 C-터미널 태그가 작동하지 않을 때 N-터미널 태그가 작동할 수 있습니다. 또한, 모든 단백질이 효율적으로 고갈되는 것은 아닙니다. 예를 들어, 표적 단백질에 대한 AID 태그는 osTIR 단백질에 접근할 수 없을 수 있다. 따라서, AID 태깅 후, 각 표적 단백질은 성장에 대한 태그의 임의의 영향에 대해 시험되어야 하며, 고갈이 효과적인지 여부를 결정하기 위해, β-estradiol 사전 배양 및 보조치료의 타이밍이 최적화되기 전에.
이 AID* 시스템은 매우 간단하며 단백질, DNA 또는 RNA 분석 또는 현미경 검사법과 같은 추가 성장을 수반하지 않는 후속 실험 절차와 호환됩니다. 또한, 시스템은 초기 RNA20을정화하기 위해 티오라벨링과 결합될 때 잘 작동한다.
이 시스템은 효모 세포의 대사에 영향을 미치지 않으면서 단백질을 고갈시키는 신속하고 구체적이며 재현 가능한 수단을 제공합니다.
공개
저자는 공개 할 것이 없다.
감사의 말
이 프로그램을 시작하고, 개발을위한 바바라 Terlouw, "우라 루퍼"구조에 대한 바히드 아슬란자데와 많은 유용한 토론을위한 수사나 드 루카스 덕분에. 이 작품은 Consejo Nacional 드 Ciencia y Tecnología, 멕시코 (CONACYT) 및 에든버러 생물 과학 대학, IEM에 웰컴 박사 과정 학생학 [105256]과 웰컴 자금 [104648]에서 GIMO에 장학금에 의해 지원되었다 [104648] JD 베그에 . 세포 생물학을 위한 웰컴 센터에서 일은 Wellcome 코어 자금 조달 [092076]에 의해 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| Adenine sulphate | Formedium | DOC0230 | |
| Agar | Formedium | AGA03 | |
| Β-estradiol | Sigma Aldrich | E2758-1G | 10 mM solution in ethanol. Store at -20 °C |
| DMSO | Alfa Aesar | 42780 | DMSO should be solid at 4 °C |
| Glucose | Fisher Scientific | G/0500/60 | |
| IAA 1H-Indole-3-acetic acid | Across Orgainics | 122150100 | Auxin analogue. 1.5 M in DMSO. The solution will be a russet colour and darken as time goes on; a deep red solution should be discarded and a new one made. Store at -20 °C. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530 | |
| Peptone | Formedium | PEP03 | |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Yeast Extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 |
참고문헌
- Haruki, H., Nishikawa, J., Laemmli, U. K. The Anchor-Away Technique: Rapid, Conditional Establishment of Yeast Mutant Phenotypes. Molecular Cell. 31, 925-932 (2008).
- Bellí, G., Garí, E., Piedrafita, L., Aldea, M., Herrero, E. An activator/repressor dual system allows tight tetracycline-regulated gene expression in budding yeast. Nucleic Acids Research. 26, 942-947 (1998).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Deshaies, R. J., Joazeiro, C. A. P. RING Domain E3 Ubiquitin Ligases. Annual Review of Biochemistry. 78, 399-434 (2009).
- Tan, X., et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase. Nature. 446, 640-645 (2007).
- Teale, W. D., Paponov, I. A., Palme, K. Auxin in action: signalling, transport and the control of plant growth and development. Nature Reviews Molecular Cell Biology. 7, 847-859 (2006).
- Nishimura, K., Fukagawa, T., Takisawa, H., Kakimoto, T., Kanemaki, M. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nature Methods. 6, 917-922 (2009).
- Morawska, M., Ulrich, H. D. An expanded tool kit for the auxin-inducible degron system in budding yeast. Yeast. 30, 341-351 (2013).
- Kubota, T., Nishimura, K., Kanemaki, M. T., Donaldson, A. D. The Elg1 Replication Factor C-like Complex Functions in PCNA Unloading during DNA Replication. Molecular Cell. 50, 273-280 (2013).
- Brosh, R., et al. A dual molecular analogue tuner for dissecting protein function in mammalian cells. Nature Communications. 7, 11742 (2016).
- DeRisi, J. L., Iyer, V. R., Brown, P. O. Exploring the metabolic and genetic control of gene expression on a genomic scale. Science. 278, 680-686 (1997).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding. Yeast. , (2018).
- McIsaac, R. S., et al. Synthetic gene expression perturbation systems with rapid, tunable, single-gene specificity in yeast. Nucleic Acids Res. 41, e57 (2013).
- Prusty, R., Grisafi, P., Fink, G. R. The plant hormone indoleacetic acid induces invasive growth in Saccharomyces cerevisiae. PNAS. 101, 4153-4157 (2004).
- Geitz, D., St Jean, A., Woods, R. A., Schiest, R. H. Improved method for high efficiency transformation of intact yeast cells. Nucleic Acids Research. 20, 1425 (1992).
- Widlund, P. O., Davis, T. N. A high-efficiency method to replace essential genes with mutant alleles in yeast. Yeast. 22, 769-774 (2005).
- Longtine, M. S., et al. Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae. Yeast. 14, 953-961 (1998).
- Eaton, S. L., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. , e52099 (2014).
- Volland, C., Urban-Grimal, D., Géraud, G., Haguenauer-Tsapis, R. Endocytosis and degradation of the yeast uracil permease under adverse conditions. Journal of Biological Chemistry. 269, 9833-9841 (1994).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유