Method Article
Ajustando a degradação para conseguir a depleção de proteína específica e eficiente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para efetivamente e especificamente esgotar uma proteína de interesse na levedura Saccharomyces cerevisiae usando o sistema β-est Aid.
Resumo
O receptor de ligação de auxina da planta, TIR1, reconhece as proteínas que contêm um motivo de degron auxina-inducible específico (AID) na presença de auxina, visando-os para a degradação. Este sistema é explorado em muitos eucariontes não vegetais, de tal forma que uma proteína alvo, marcada com o motivo AID, é degradada na adição de auxina. O nível de expressão TIR1 é crítico; a expressão excessiva conduz à degradação da proteína AID-etiquetada mesmo na ausência de auxin, visto que a baixa expressão conduz à prostração lenta. Um sistema de auxílio β-estradiol-induzível foi criado, com expressão de TIR1 o controle de um promotor induzível do β-estradiol. O nível de TIR1 é sintonável alterando o tempo de incubação com β-estradiol antes da adição de auxina. Este protocolo descreve como esgotar rapidamente uma proteína alvo usando o sistema AID. O tempo adequado de incubação de β-estradiol depende da abundância da proteína alvo. Portanto, a depleção eficiente depende do tempo ideal que também minimiza a depleção independente de auxina.
Introdução
Mutações condicionais, como mutantes sensíveis à temperatura, são uma ferramenta poderosa para o estudo de proteínas essenciais, permitindo o crescimento celular a condição permissiva, mas causando perda de função condições não permissivas. No entanto, o metabolismo celular pode ser seriamente perturbado pela mudança nas condições de crescimento necessárias para induzir o defeito e também pode criar efeitos fora do alvo. Vários métodos foram desenvolvidos, em que a proteína de interesse é condicionalmente seqüestrado1 ou sua expressão é controlada2,3 por adição de uma pequena molécula. Este protocolo utiliza auxina e o sistema de degron (AID) auxina-inducible para esgotar eficientemente uma proteína alvo.
O sistema AID tem sua origem em plantas, onde uma auxina (neste protocolo indol-3-ácido acético (IAA) é usada), estimula a interação da proteína aux/IAA com TIR1, um membro do complexo de ligase de ubiquitina SCF U34. A interação complexa do SCF causa a poliubiquitinação das proteínas da família aux/IAA, o que resulta em sua degradação pelo Proteassoma5,6.
Este sistema foi previamente adaptado para uso na levedura Saccharomyces cerevisiae7,8 expressando a proteína TIR1 de Oriza Sativa (Ostir) em células de levedura, onde é capaz de interagir com o fermento endógeno Complexo SCF. A proteína de interesse foi marcada com um motivo da proteína aux/IAA IAA17 para direcioná-la para a degradação. Os truncamentos funcionais de IAA17 foram desenvolvidos mais tarde, tais como o auxílio *8,9,10, contendo o 43 de aminoácido auxina-motivo sensível do IAA17 de Arabidopsis Arabidopsis thaliana, junto com um Tag do epítopo para permitir Detecção.
O sistema inicialmente adaptado para uso em brotamento de levedura7,8 expressou a proteína osTIR1 de um promotor de galão de levedura. A expressão requer a mudança para o meio de crescimento com galactose como a única fonte de carbono, que, infelizmente, resulta em uma mudança diauxica com mudanças de amplo alcance para o metabolismo celular11. Por outro lado, tem sido relatado que a expressão constitutiva de TIR1 pode levar à degradação da proteína alvo na ausência de auxina/IAA12 se o nível de expressão for alto, enquanto a baixa expressão de TIR1 provoca depleção ineficiente. Foi desenvolvido um sistema de auxílios melhorado denominado β-est Aid em que o Ostir está o controlo de um promotor induzível que é sintonável para se adequar à proteína alvo, com efeito mínimo no metabolismo celular. Para isso, foi construído um fator de transcrição artificial (ATF) no qual o ativador de transcrição viral VP16 é fundido a um receptor de estrogênio e um domínio de ligação de DNA de quatro dedos de Zn (DBD). Quando o β-estradiol (um estrogénio) está presente, a ATF pode entrar no núcleo e induzir a transcrição do Ostir ligando ao seu promotor (Z4EVpr)13,14.
a expressão de osTIR é geralmente detectável aproximadamente 20 minutos após a adição de β-estradiol12. Entretanto, a duração óptima da expressão de Ostir para conseguir o esgotamento eficiente da proteína etiquetada com auxina, ao evitar a prostração antes da adição do auxina, precisa de ser determinada empiricamente para cada proteína do alvo. Um tempo aproximado para esta pré-incubação pode ser estimado a partir de valores de abundância no banco de dados do genoma de Saccharomyces (SGD https://www.yeastgenome.org/). Como pode ser observado na Figura 1, a proteína abundante, Dcp1 (2880 a 4189 moléculas/célula), requer 40 min de pré-incubação com β-estradiol, sem depleção de auxina independente observada. A proteína muito menos abundante, prp2 (172 a 211 moléculas/pilha), é empobrecido fortemente após somente 20 minutos do pre-incubação. É aconselhável testar dois tempos de pré-incubação adicionais, 10 a 20 minutos antes ou depois deste tempo estimado inicial (20 min é o tempo mínimo recomendado). O tempo de pré-incubação ideal é o tempo em que a proteína alvo não se esgotou antes de adicionar auxina e uma vez que a auxina é adicionada a depleção é aceitável ou os níveis de proteína se aproximam do mínimo possível. Assim, a partir da Figura 1b, para Prp22 com 30 min de pré-incubação, os níveis não diminuíram muito 10 min após a adição de auxina. Comparando-se com 40 min de pré-incubação e 15 min com IAA, onde há pouca depleção adicional, não há benefício na incubação com auxina maior que 10 min ou pré-incubando por mais de 30 min, particularmente porque há evidência de não-auxina depleção dependente em 40 min. Para Dcp1 com 40 min de pré-incubação (o último ponto em que o nível de proteína é aproximadamente 100% antes da adição de auxina), 15 a 20 min de depleção com auxina é aceitável. Recomenda-se manter o tempo de depleção o mais curto possível para reduzir os efeitos secundários no metabolismo celular14.
Este artigo demonstra como usar o sistema de β-est AID otimizando o sincronismo da incubação do β-estradiol para a expressão de osTIR para conseguir a prostração rápida da proteína do alvo em cima da adição de IAA sem prostração antes de adicionar o auxin.
Protocolo
Nota: Consulte a Figura 2 para obter um resumo gráfico.
1. preparação de estirpe
-
Utilizando uma estirpe ura3, introduza o sistema β-est Aid (ou seja, os genes que codificam o factor de transcrição responsivo β-estradiol (ATF) e o Ostir) e a tag Aid * a proteína alvo (ver Figura 3 e tabela 1 para um resumo do procedimento).
- Transforme15 pZTRK (marcador de resistência G418) ou pZTRL (marcadorLEU2 ) plasmídeo (disponível a partir do centro de recursos genéticos de levedura) na cepa ura3- levedura ou use o plasmídeo como modelo para produzir o produto PCR para Integração.
- O PCR amplifica o ATF (marcado Z4EVATF no mapa do plasmídeo) e Ostir usando um polymerase da alta fidelidade de um do plasmídeos pztrk ou de pztrl. Use primers com 50 a 60 base 3 ' extensões com homologia para a região genômica, para direcionar a integração por recombinação homóloga16. Para a integração genômica dos dois componentes separadamente ou em conjunto, ver tabela 1 para primers e condições.
Nota: A estirpe pZ4EV-NTR1 tem os componentes já integrados no genoma (disponível a partir do centro de recursos genéticos de levedura, Japão). - Certifique-se de que a proteína alvo é AID * etiquetada utilizando o procedimento de longtine17 (ver Figura 3B e tabela 1).
- Realize uma análise de crescimento da cepa sem β-estradiol e IAA presentes para determinar se a tag AID * afeta o crescimento e prever a taxa de crescimento para uso na etapa 1,5.
2. procedimento geral de esgotamento
- Calcule quanta cultura é necessária para que todas as amostras sejam coletadas; por exemplo, 10 mL de cultura em OD600 de 0,8 é suficiente para a extração de proteínas, RNA e DNA para uma única amostra, portanto, para 6 amostras, pelo menos 60 ml de cultura é necessária.
- De uma cultura overnight, configurar uma cultura nova suficiente em OD600 0,1 para 0,2 e deixar de crescer a 30 ° c. Um meio rico como o YPDA é recomendado, embora outras condições de crescimento possam ser usadas:
Extrato de levedura 10 g de Peptona 20 g de Glicose 20 g de Sulfato de adenina 40 mg H2o a 1 litro
Nota: Autoclave ou filtro esterilizar; a esterilização do filtro é preferida como os complexos do peptide/açúcar produzidos pelo precipitado autoclavagem no metanol usado na coleção da amostra. - Prepare-se para receber as amostras.
- Coloque 30 a 50% do volume amostral pretendido de metanol em um tubo. Por exemplo, se uma amostra de 10 mL for tomada, coloque 5 mL de metanol em um tubo Falcon de 15 mL e feche o tubo firmemente. Uma vez que fechado, Rotule o tubo e põr sobre o gelo seco ou em-80 ° c para refrigerar.
Atenção: Dispense o metanol em uma capa de fumaça. - Rotule 1,5 tubos de mL para o armazenamento a longo prazo das amostras e coloc no gelo para refrigerar.
- Arrefecer o suficiente H2o (pelo menos 1 ml por amostra) no gelo.
- Coloque 30 a 50% do volume amostral pretendido de metanol em um tubo. Por exemplo, se uma amostra de 10 mL for tomada, coloque 5 mL de metanol em um tubo Falcon de 15 mL e feche o tubo firmemente. Uma vez que fechado, Rotule o tubo e põr sobre o gelo seco ou em-80 ° c para refrigerar.
- Antecipar o crescimento da cultura. A OD alvo para a coleta das amostras é de aproximadamente 0,7 a 0,8, mas a etapa de pré-incubação (a incubação com β-estradiol para induzir o osTIR), precisa ser iniciada mais cedo para que a cultura atinja aproximadamente a OD direita no momento em que as amostras são Coletados.
Nota: É aconselhável realizar uma curva de crescimento nas condições a serem utilizadas no experimento para que esta OD inicial possa ser estimada. - Uma vez atingida a OD alvo para o início da pré-incubação, tomar uma amostra (geralmente 10 mL), no tubo pré-preparado contendo metanol frio. Inverta brevemente para misturar e colocar de volta no gelo seco.
Nota: A amostra pode ser movida para o gelo de água após cerca de 5 min, se conveniente para fazê-lo. - Adicione imediatamente o β-estradiol, 1 μL/mL de cultura (concentração final de 10 μM); ter o β-estradiol pré-medido em uma pipeta pronta para uso, a fim de reduzir o tempo necessário entre a coleta da amostra e a adição do β-estradiol. Misture rapidamente girando vigorosamente.
- Continuar a cultivar a cultura como antes (passo 2,2), incubar (esta é a etapa de "pré-incubação") com β-estradiol para o tempo ideal (para a determinação do tempo de pré-incubação ideal ver Figura 1).
- Prepare-se para adicionar IAA (auxina). Tome acima o volume de IAA necessário para a etapa 2,10 (isto é, 0,5 μL de IAA por o mL da cultura). Isso torna o passo 2,20 mais rápido.
- Colete uma amostra como etapa 2,5.
- Adicione imediatamente IAA 0,5 μL/mL de cultura a uma concentração final de 750 μM conforme preparado na etapa 2,8. Misture rapidamente girando vigorosamente.
- Colete amostras, como etapa 2,5, de acordo com seu projeto experimental. Ou uma única amostra, em um momento em que se espera que a proteína será seguramente esgotada, ou várias amostras em um curso de tempo de depleção. Por exemplo, os intervalos de 5 minutos são convenientes para o sincronismo e fornecem uma escala de níveis da proteína. A estratégia de otimização, como mostrado na Figura 1, dará uma indicação de horários adequados.
- Processe as amostras.
- Coloque as amostras no gelo, se não for feito já. Assegure-se de que nenhuma das amostras tenha congelado; Se eles têm, suavemente quente na mão, invertendo constantemente para que a temperatura não sobe localmente.
Nota: Isto é feito melhor na mão como a temperatura da amostra pode ser avaliada, ele deve sempre sentir frio. Coloque no gelo. Este não é um ponto de pausa-uma vez que todas as amostras são fluidas, prossiga para a próxima etapa. - Uma vez que todas as amostras foram coletadas e não estão mais congeladas, gire em 3.500 x g por 2 min (a 4 ° c, se possível).
- Despeje o metanol/mistura média e coloque de volta no gelo; Não se preocupe se nem todo o líquido foi removido.
- Ressuscita o pellet de células em 1 mL de gelo frio H2o (a partir do passo 2.3.3) e transfere para um tubo rotulado de 1,5 ml (preparado no passo 2.3.2) no gelo.
- Gire brevemente (por exemplo, 10 s de tempo total) em > 15000 x g para re-pellet as células, coloque de volta no gelo e retire o líquido.
- Retire o H2o por aspiração. As pelotas da pilha podem ser armazenadas em-20 ° c, ou-80 ° c para o armazenamento a longo prazo.
- Coloque as amostras no gelo, se não for feito já. Assegure-se de que nenhuma das amostras tenha congelado; Se eles têm, suavemente quente na mão, invertendo constantemente para que a temperatura não sobe localmente.
- Verifique o nível para o qual a proteína foi esgotada pela análise Western blot18.
Nota: A proteína suficiente19 e/ou o ácido nucleico podem ser extraídos de uma única pelota da pilha para a maioria de finalidades, embora as espécies raras do RNA possam exigir mais volume da amostra.
Resultados
Exemplos representativos de depleção são exibidos na Figura 1. Os três experimentos apresentados nesta figura foram experimentos de otimização para depleção das proteínas prp2, Prp22 e Dcp1. A baixa abundância, as proteínas prp2 e Prp22 de spliceosomal esgotou-se a menos de 20% após o 40 min pre-incubação com β-estradiol seguido por 15 minutos com auxin. Tempos de pré-incubação mais longos levam a depleção mais rápida, mas também mostram depleção de proteínas indesejáveis antes da adição de auxina. Em comparação, o Dcp1 mais abundante foi apenas esgotado para aproximadamente 30% com o mesmo tratamento, mas 60 min de pré-incubação resultou em depleção para 13% com o mesmo tratamento de auxina, ao custo de depleção antes da adição da auxina. É possível que 50 min de pré-incubação com β-estradiol e 15 min com auxina teria conseguido resultados semelhantes em um ponto de tempo mais curto e assim teria sido mais ideal.
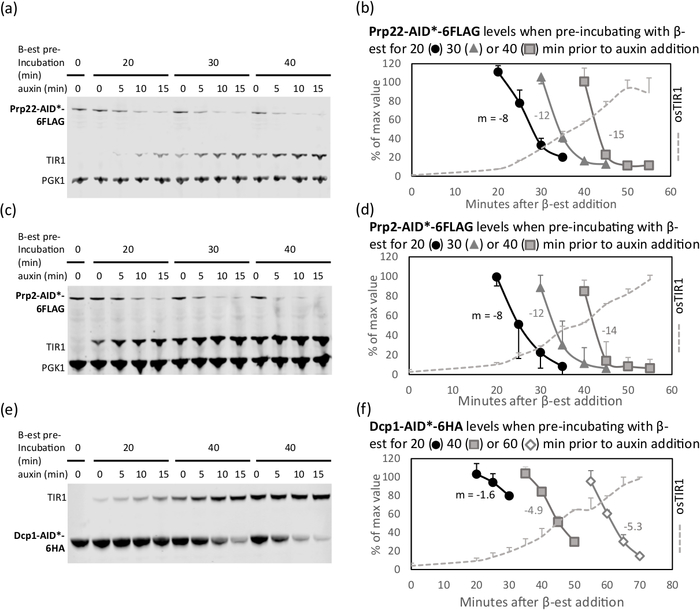
Figura 1: a taxa de depleção pode ser ajustada modulando a duração da pré-incubação de β-estradiol. Western blot18 de proteínas alvo: (a e b) PRP22-Aid *-6 Flag, (c e d) prp2-Aid *-6 Flag, e ( e e f) Dcp1-Aid *-6ha, a partir de culturas pré-incubadas com β-estradiol (β-est) para 20, 30, 40, ou 60 min antes da auxina adição12. Quantidades iguais de proteína total foram carregadas em cada pista. Pgk1 é detectado como um controle de carregamento Visual, exceto para o painel e, onde Pgk1 e Dcp1 comigrar. A quantificação das bandas proteicas nos painéis a, c e e é mostrada nos painéis b, de f, respectivamente. Como medida de taxa de depleção, calculou-se a inclinação (m) para a seção linear (de 100% para 30% dos valores iniciais) de cada curva. O tempo de pré-incubação ideal é o tempo em que os níveis proteicos ainda estão próximos dos níveis não induzidos (100%) e a taxa subsequente de depleção é rápida. Para Dcp1 (f), 60 min de pré-incubação é muito longo, como a proteína começou a degradar na ausência de auxina, enquanto 20 min é muito curto, como a proteína não se esgota de forma apreciável neste curso de tempo. Após 40 min de pré-incubação, 15 min com auxina pode ser usado como a proteína é de aproximadamente 70% empobrecido e, embora 20 min resultaria em mais depleção, também poderia resultar em efeitos secundários. As barras de erro representam o desvio padrão de duas replicações biológicas. Para cada experimento, um borrão representativo é mostrado. Esse valor é derivado da publicação anterior9. Por favor clique aqui para ver uma versão maior desta figura.
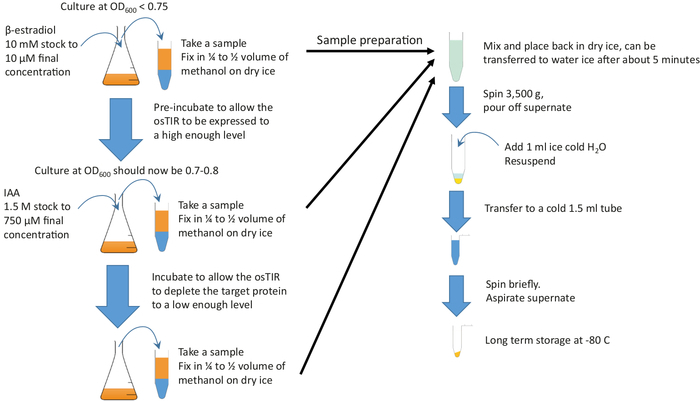
Figura 2: Resumo gráfico. Adicionar β-estradiol a cultura suficiente crescendo em meio rico e na temperatura desejada, a fim de iniciar a pré-incubação. Continue o crescimento para o tempo de pré-incubação necessário antes de adicionar IAA (auxina) para iniciar a depleção. Os tempos de pré-incubação e depleção dependem da proteína a ser esgotada, mas a pré-incubação é muitas vezes na faixa de 20-60 min e o tempo de depleção é tipicamente na ordem de 10 a 20 min. 10 mL de amostras devem ser tomadas no início e no fim da pré-incubação e durina g o esgotamento. Estas amostras são rapidamente fixadas em metanol frio antes de peletização e armazenamento. Por favor clique aqui para ver uma versão maior desta figura.
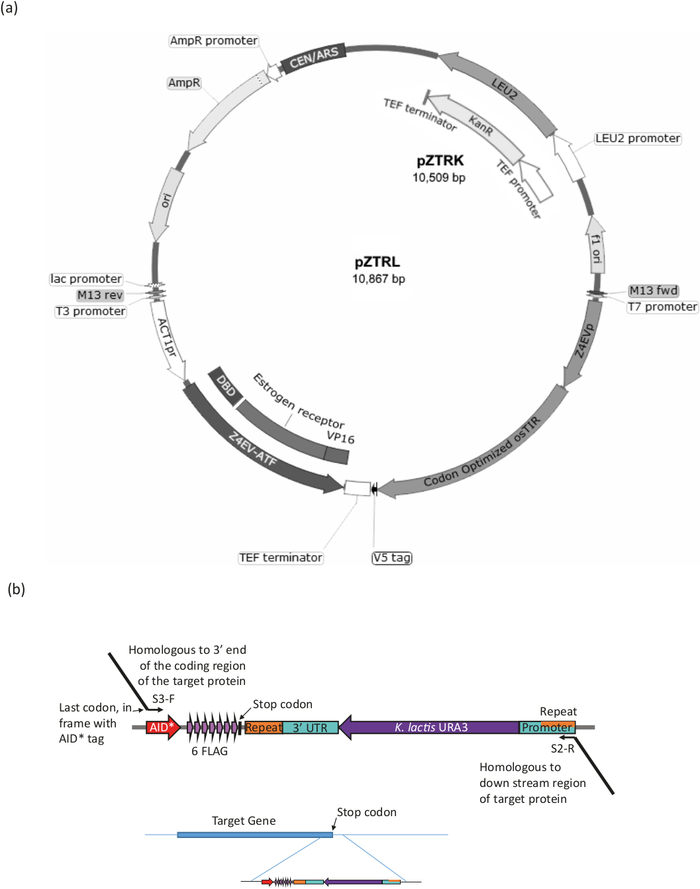
Figura 3: geração de estirpe para o sistema B-est. (a) para gerar uma estirpe de levedura com o sistema Aid *, tanto o pZTRL (LEU2) ou o pztrk (kanamycin (G418) resistência), plasmídeo deve ser introduzido na estirpe ou, alternativamente, o ATF e Ostir pode ser inserido no genoma por recombinação homous de um fragmento gerado pelo PCR de 3 ' primers finais (veja a Figura 3B e a tabela 1). (b) C-a etiquetagem terminal da proteína alvo é conseguida pela amplificação do PCR da região apropriada do PURA3-Aid do plasmídeo *-6flag (pURA3_AID *-6ha difere somente no Tag e pode ser tratada exatamente da mesma maneira), usando os primers S3-F de longtine e S2-R com 3 ' extensões homólogo à extremidade 3 ' da proteína do alvo. A extensão da cartilha para a frente deve incluir o último Codon do aminoácido no quadro com o início da etiqueta AID * e não deve incluir o Codon de batente. A extensão de primer reverso deve ser para uma região a jusante da região de codificação. Uma vez inseridos no genoma, as células que perderam o marcador URA3 (por recombinação homous entre as regiões idênticas encontradas em ambas as extremidades do marcador) podem ser selecionadas pelo crescimento com 5-Foa, que contraria URA3 células. Por favor clique aqui para ver uma versão maior desta figura.
| a. sequências de primer | |||||||
| Alvo | Localização | Primer | Nome | Seqüência | TM (° c) | ||
| o pZTRL | 516 | F | pZTRL_F | <-região de homologia-> GCGACAGCATCACCGACTTCG | 61,23 | ||
| 7897 | R | pZTR_R | CGCCGCCTCTACCTTGCAGA <-região de homologia (RC)-> | 61,30 | |||
| o pZTRK | 9154 | F | pZTRK_F | <-região de homologia-> ACGTTGAGCCATTAGTATCAATTTGCTTACC | 59,40 | ||
| 5897 | R | pZTR_R | CGCCGCCTCTACCTTGCAGA <-região de homologia (RC)-> | 61,30 | |||
| pURA3-AID *-6BANDEIRA ou pURA3_AID *-6HA | F | S3-F | <-região de homologia-> CGTACGCTGCAGGTCGAC | 59,21 | |||
| R | S2-R | ATCGATGAATTCGAGCTCG <-região de homologia (RC)-> | 52,76 | ||||
| pZRTL/K é amplificar o sistema β-est AID | |||||||
| pURA3-Aid *-6 Flag/6ha para amplificar o auxílio * e epítopo mapeado tag para etiquetar a proteína alvo (procedimento lontine) | |||||||
| <-região de homologia-> | Região homólogo às regiões flanqueando onde o sistema deve ser introduzido. Quanto mais essa região for mais provável, a modificação será bem-sucedida; 50-100 bases é recomendado. | ||||||
| <-região de homologia (RC)-> | Região homólogo às regiões flanqueando onde o sistema deve ser introduzido, recorde usar o complemento reverso. Como acima, quanto mais tempo esta região é melhor. | ||||||
| TM (° c) | TM utilizando o método% GC com 50 mM NaCl | ||||||
| b. PCR Mix | |||||||
| Componente | Volume (μL) | ||||||
| Modelo | < 10 | ||||||
| NEB Phusion HF buffer (5x) * | 100 | ||||||
| Primer forward 100 μM | 2,5 | ||||||
| Primer reverso 100 μM | 2,5 | ||||||
| dNTPs 10 mM cada | 10 | ||||||
| H2O | a 500 | ||||||
| * O NEB Phusion GC buffer (5x) também pode ser usado, mas não é preferível | |||||||
| Fazer esta mistura, dividida em 10 tubos de 50 μL misturar cada um e executar o PCR como tabela 1 c. | |||||||
| Verifique o PCR funcionou executando em um gel do agarose | |||||||
| Combine todas as reações bem-sucedidas em um tubo e etanol precipitado | |||||||
| Transforme o fermento com todo o material produzido pelo PCR | |||||||
| c. condições de PCR | |||||||
| Passo | Temp (° c) | Tempo | |||||
| Desnaturação inicial | 98 | de 30 s | |||||
| 25-35 ciclos | Desnaturar | 98 | 10 de s | ||||
| Anneal | 45 – 60 | 20 de s | |||||
| Extensão | 72 | 30 s/KB | |||||
| Extensão final | 72 | 10 minutos | |||||
| Segurar | 8 | ||||||
| Anneal a 45 ° c para o conjunto primário Lontine (S3-F e S2-R) e 60 ° c para os primers pZTRL/K | |||||||
| Estender por 3 minutos para o PCR Lontine e 3 minutos para pZTRL/K | |||||||
Tabela 1: sequências da cartilha, mistura de PCR e condições de PCR.
Discussão
Um protocolo bem otimizado pode produzir depleção rápida e eficiente da proteína alvo. Determinar o tempo de pré-incubação aproximado com β-estradiol é importante, pois isso aumenta a reprodutibilidade do esgotamento, mas pequenas variações no tempo de pré-incubação podem ser toleradas. Por outro lado, deve-se tomar cuidado com o tempo após a adição de auxina, pois o nível de proteína diminui muito rapidamente.
Uma vantagem dessa abordagem é que a depleção ajustada pode ser alcançada por diferentes combinações de tempo de pré-incubação com o tempo de incubação de β-estradiol e IAA. Por exemplo, se desejado, a proteína alvo pode ser mais lentamente esgotada, reduzindo o tempo de pré-incubação.
O sistema de ajuda β-est oferece certas vantagens em relação aos sistemas em que o OsTIR é expresso constitutivamente. Por exemplo, se a proteína alvo for essencial para a viabilidade, a expressão regulada de osTIR pode evitar a depleção prematura da proteína alvo. Além disso, a expressão de osTIR pode ser ajustada para se adequar à abundância da proteína alvo e sua susceptibilidade à degradação, e a depleção pode ser rápida ou lenta. Os dois efetores de moléculas pequenas, β-estradiol e AUXINA, não perturbam o metabolismo das leveduras nas condições aqui utilizadas, diferentemente da rapamicina, utilizadas no sistema âncora-afastado1.
Deve-se notar que a marcação de algumas proteínas interrompe a sua função, o que é um problema com qualquer sistema de esgotamento alvo. Nesse caso, uma marca N-terminal pode funcionar quando uma marca de terminal C não. Além disso, nem todas as proteínas serão esgotadas eficientemente; por exemplo, a tag-AID na proteína alvo pode ser inacessível à proteína osTIR. Portanto, após a etiquetagem de ajuda, cada proteína alvo deve ser testada para qualquer efeito da marca no crescimento, e para determinar se a depleção é efetiva, antes que os intervalos de pré-incubação de β-estradiol e o tratamento de auxina sejam otimizados.
Este sistema AID * é muito simples e é compatível com qualquer procedimento experimental subsequente que não envolva um crescimento adicional, como a análise de proteínas, DNA ou RNA ou microscopia. Além, o sistema trabalha bem quando combinado com o thiolabelling para purificar o RNA nascente20.
Este sistema fornece um rápido, específico, e reprodutível meios de esgotar uma proteína sem de outra forma afetando o metabolismo da célula de levedura.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Graças a Jane Reid para iniciar este programa, Barbara Terlouw para o desenvolvimento, Vahid Aslanzadeh para o "ura Looper" construções e Susana de Lucas para muitas discussões úteis. Este trabalho foi apoiado por uma bolsa de estudos para a GIMO do Consejo Nacional de Ciencia y Tecnología, México (CONACYT) e da Universidade de Edimburgo escola de ciências biológicas, um Wellcome PhD Studentship para IEM [105256] e por Wellcome financiamento [104648] para JD Beggs . O trabalho no centro Wellcome para a biologia de pilha é apoiado pelo financiamento do núcleo de Wellcome [092076].
Materiais
| Name | Company | Catalog Number | Comments |
| Adenine sulphate | Formedium | DOC0230 | |
| Agar | Formedium | AGA03 | |
| Β-estradiol | Sigma Aldrich | E2758-1G | 10 mM solution in ethanol. Store at -20 °C |
| DMSO | Alfa Aesar | 42780 | DMSO should be solid at 4 °C |
| Glucose | Fisher Scientific | G/0500/60 | |
| IAA 1H-Indole-3-acetic acid | Across Orgainics | 122150100 | Auxin analogue. 1.5 M in DMSO. The solution will be a russet colour and darken as time goes on; a deep red solution should be discarded and a new one made. Store at -20 °C. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530 | |
| Peptone | Formedium | PEP03 | |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Yeast Extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 |
Referências
- Haruki, H., Nishikawa, J., Laemmli, U. K. The Anchor-Away Technique: Rapid, Conditional Establishment of Yeast Mutant Phenotypes. Molecular Cell. 31, 925-932 (2008).
- Bellí, G., Garí, E., Piedrafita, L., Aldea, M., Herrero, E. An activator/repressor dual system allows tight tetracycline-regulated gene expression in budding yeast. Nucleic Acids Research. 26, 942-947 (1998).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Deshaies, R. J., Joazeiro, C. A. P. RING Domain E3 Ubiquitin Ligases. Annual Review of Biochemistry. 78, 399-434 (2009).
- Tan, X., et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase. Nature. 446, 640-645 (2007).
- Teale, W. D., Paponov, I. A., Palme, K. Auxin in action: signalling, transport and the control of plant growth and development. Nature Reviews Molecular Cell Biology. 7, 847-859 (2006).
- Nishimura, K., Fukagawa, T., Takisawa, H., Kakimoto, T., Kanemaki, M. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nature Methods. 6, 917-922 (2009).
- Morawska, M., Ulrich, H. D. An expanded tool kit for the auxin-inducible degron system in budding yeast. Yeast. 30, 341-351 (2013).
- Kubota, T., Nishimura, K., Kanemaki, M. T., Donaldson, A. D. The Elg1 Replication Factor C-like Complex Functions in PCNA Unloading during DNA Replication. Molecular Cell. 50, 273-280 (2013).
- Brosh, R., et al. A dual molecular analogue tuner for dissecting protein function in mammalian cells. Nature Communications. 7, 11742(2016).
- DeRisi, J. L., Iyer, V. R., Brown, P. O. Exploring the metabolic and genetic control of gene expression on a genomic scale. Science. 278, 680-686 (1997).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding. Yeast. , (2018).
- McIsaac, R. S., et al. Synthetic gene expression perturbation systems with rapid, tunable, single-gene specificity in yeast. Nucleic Acids Res. 41, e57(2013).
- Prusty, R., Grisafi, P., Fink, G. R. The plant hormone indoleacetic acid induces invasive growth in Saccharomyces cerevisiae. PNAS. 101, 4153-4157 (2004).
- Geitz, D., St Jean, A., Woods, R. A., Schiest, R. H. Improved method for high efficiency transformation of intact yeast cells. Nucleic Acids Research. 20, 1425(1992).
- Widlund, P. O., Davis, T. N. A high-efficiency method to replace essential genes with mutant alleles in yeast. Yeast. 22, 769-774 (2005).
- Longtine, M. S., et al. Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae. Yeast. 14, 953-961 (1998).
- Eaton, S. L., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. , e52099(2014).
- Volland, C., Urban-Grimal, D., Géraud, G., Haguenauer-Tsapis, R. Endocytosis and degradation of the yeast uracil permease under adverse conditions. Journal of Biological Chemistry. 269, 9833-9841 (1994).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados