É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma abordagem rápida para alta resolução de imagem de fluorescência em fatias grossas Semi-Cérebro
Neste Artigo
Resumo
Aqui nós descrevemos um método rápido e simples de células de imagem fluorescente etiquetado em fatias grossas semi-cérebro. Através da fixação, corte, limpeza e opticamente tecido cerebral descrevemos como imagens de epifluorescência ou confocal padrão pode ser usado para visualizar as células individuais e redes neuronal no tecido nervoso intacto.
Resumo
Um objetivo fundamental tanto para a neurociência básica e clínica é compreender melhor a identidade, composição molecular e padrões de conectividade que são característicos para os neurônios do cérebro em ambos os normais e doentes. Para isso, um grande esforço foi colocado na construção de mapas de alta resolução neuroanatômicas 1-3. Com a expansão da genética molecular e os avanços em microscopia de luz veio a capacidade de consulta não só morfologias neuronal, mas também a composição molecular e celular dos neurônios individuais e suas redes associadas 4. Grandes avanços na capacidade de marcar e manipular neurônios através gene transgênico e tecnologias de direcionamento no roedor agora permitir que os pesquisadores subconjuntos neuronal "programa" à vontade 5-6. Indiscutivelmente, uma das contribuições mais influentes para a neurociência contemporânea foi a descoberta e clonagem de genes que codificam proteínas fluorescentes (PQ) em invertebrados marinhos 7-8, ao lado de seus subseqüentes de engenharia para produzir uma caixa de ferramentas cada vez maior de repórteres vital 9. Exploração de atividade do promotor de células de tipo específico para conduzir direcionados expressão FP em discretos populações neuronais agora oferece investigações neuroanatômicas com precisão genética.
Engenharia expressão FP em neurônios melhorou muito nossa compreensão da estrutura e função cerebral. No entanto, os neurônios individuais de imagem e suas redes associadas em tecidos cerebral profunda, ou em três dimensões, manteve-se um desafio. Devido ao alto teor lipídico, o tecido nervoso é bastante opaco e apresenta fluorescência auto. Estas propriedades inerentes biofísicos dificultam a visualização e imagem neurônios fluorescente etiquetado em alta resolução através de microscopia confocal de epifluorescência ou padrão profundidades além de dezenas de microns. Para contornar este desafio investigadores muitas vezes empregam imagens seção fina de série e métodos de reconstrução 10, ou 2-fótons de microscopia de varredura a laser 11. Inconvenientes atuais para essas abordagens são a preparação do tecido associado de trabalho intensivo, ou de custo proibitivo de instrumentação, respectivamente.
Aqui, apresentamos um método relativamente rápido e simples de visualizar células fluorescente etiquetado na fixa semi-grossas fatias de cérebro de rato, limpando óptica e de imagem. No protocolo anexo descrevemos os métodos de: 1) o tecido cerebral fixação in situ, através de perfusão intracardíaca, 2) dissecção e remoção do cérebro inteiro, 3) a incorporação do cérebro estacionário em agarose, 4) elaboração de precisão fatia semi-grossa usando instrumentação vibratome nova , 5) o tecido cerebral de compensação através de um gradiente de glicerol, e 6) de montagem em lâminas de vidro para microscopia de luz e z-stack reconstrução (Figura 1).
Para preparar fatias de cérebro implementamos um pedaço relativamente novo de instrumentação chamado de "Compresstome" VF-200 (http://www.precisionary.com/products_vf200.html). Este instrumento é um micrótomo semi-automatizado, equipado com um avanço motorizados e sistema de vibração da lâmina com características semelhantes em função de vibratomes outros. Ao contrário de outras vibratomes, o tecido a ser cortado é montado em um plugue de agarose dentro de um cilindro de aço inoxidável. O tecido é extrudado em espessuras desejadas do cilindro, e corte pela lâmina para a frente avançando vibrando. A ficha de agarose / sistema de cilindro permite tecido reprodutíveis de montagem, alinhamento e corte de precisão. Em nossas mãos, o "Compresstome 'rende fatias de alta qualidade de tecidos para eletrofisiologia, imuno-histoquímica, e dirigir-tecido fixo de montagem e de imagem. Combinados com a óptica, aqui demonstrar a preparação de semi-grossas fatias de cérebro fixa de alta resolução de imagem fluorescente.
Protocolo
1. Na fixação do cérebro situ
* Prepare uma seringa de 10 ml (28 agulha) preenchido com tampão fosfato salino (PBS).
* Prepare uma seringa de 10 ml (28 agulha) preenchido com paraformaldeído a 4% (PFA) em PBS. Reserve um adicional de 50-10 ml de PFA / PBS para fixação post.
- Injetar por via intraperitoneal rato experimental com uma dose letal de Avertin ou Nembutal.
- Uma vez que o mouse está sedado, molhar o abdômen com etanol e segura o mouse para a parte inferior de uma bandeja em camadas com a cortiça, a cera, ou elastômero de silicone utilizando os pinos de dissecação. Com abdômen virado para cima, segura as quatro patas para a superfície - espalhando-os o mais amplo possível.
- Agarrar a pele com uma pinça ao nível do esterno, e cortado transversalmente para expor o fígado. Corte lateral e depois para cima através das costelas e diafragma. Remover o retalho de tecido costela e continue cortando até que o coração está acessível.
- Furar ou cortar o átrio direito para drenar o sangue circular.
- Lave sangue residual do sistema circulatório por perfusão de PBS através do ventrículo esquerdo.
- Acessando o buraco mesma agulha, corrigir rato inteiro por perfusão subsequente de PBS / PFA através do ventrículo esquerdo.
2. Dissecção e extração de cérebro
- Retire a pele da cabeça, pescoço e crânio, reduzindo em torno de canal auditivo e as órbitas dos olhos, seguida puxando a pele anteriormente para expor o crânio.
* Veja a Figura 2 para uma ilustração de passos 2.2) a 2.5) - Usando uma tesoura osso, cortado transversalmente do olho do soquete através do corneto nasal ossos. Faça isso para cada lado. O crosscut deve ser anterior aos olhos e bulbos olfatórios.
- Corte longitudinal de cada canal de orelha a cavidade ocular.
- Usando uma tesoura pequena dissecção, corte a placa de osso occipital que cobre o cerebelo de um canal do ouvido para o outro.
- Com a tesoura mesmo dissecção, corte anteriormente ao longo da linha média ao nível dos bulbos olfativos.
- Usando pinça, retire cada placa óssea a partir do cérebro, tendo o cuidado de remover qualquer tecido conjuntivo para que o cérebro permanece intacto ao levantar fora do osso.
- Nervos cranianos e Snip óptica, corte as vértebras cervical superior e remova o cérebro em fixador.
- Pós-fix por 1-2 horas a 4 ° C. Lavar 3 vezes 15 min cada em PBS.
3. Incorporação de agarose
- Derreta 2% de alta resistência, com baixo ponto de gel de agarose tipo IB (Sigma número de catálogo: A0576) em PBS salina com forno de microondas.
- Transferência de agarose fundida a um tubo de ensaio. Coloque o tubo de ensaio em um banho de água morna ou incubadora (40 ° -41 ° C) até a temperatura de equilíbrio.
- Cola tecido cerebral fixa para o êmbolo da seringa amostra com cola de cianoacrilato (Figura 3A). Pincel uma fina camada de solução de sacarose-PBS saturada na superfície do tecido cerebral para facilitar a liberação posterior do anel de agarose as fatias de cérebro.
- Sacar o êmbolo com o tecido do cérebro dentro do alojamento de êmbolo (Figura 3B).
- Pipeta ou derramar agarose quente dentro da caixa do êmbolo, cobrindo o tecido cerebral. Evite bolhas de interceptação no agarose. Bolhas de ar no agarose irá comprometer a qualidade fatia.
- Grampo da seringa com uma amostra de frio (° C a -25 ° C 0) bloco de refrigeração por 1 minuto para definir a agarose (Figura 3C).
4. Seccionamento fatia do cérebro com o Compresstome
- Monte a seringa contendo a amostra de tecido cerebral no Compresstome (Figura 3D).
- Alinhe a extremidade da lâmina de barbear em estreita colaboração com a saída da seringa de amostra (Figura 3E).
- Encher o tanque com a solução tampão PBS.
- Gire o micrômetro de espessura uma fatia para a frente a extrusão do bloco de tecido cerebral agarose-out da seringa espécime.
- Pressione o botão "iniciar" na unidade de controle do Compresstome para iniciar o processo de corte.
- Repita os passos 4.4) a 4.5) até fatias desejados são obtidos em determinar a espessura.
- Colheita fatias de cérebro com pincel ou pipeta para frascos ou poços coloração preenchido com PBS (Figura 3F).
5. Clearing óptica
- Prepare um 75% vol: vol solução de glicerol: PBS.
- Pour (ou pipeta) off metade do volume de PBS de lavagem dos fatias de cérebro coletados.
- Adicionar novamente o volume removido com glicerol / PBS.
- Deixar equilibrar com a mistura suave a 4 ° C até a pia fatias.
- Repita os passos 6.2) a 6,4) até uma concentração de glicerol final atinja os 75%.
- Substituir solução com 90% de glicerol em PBS e deixar equilibrar.
* Nota: Tome cuidado para não introduzir bolhas de ar, enquanto a adição de soluções de glicerol. Despeje lentamente soluções e misture delicadamente, uma vez que as bolhas de ar irá interferir com uniforme equilibração e será transferido para montagem de carros.
6. Fatia de montagem para imagens
- Corte de dupla face adesiva de vedação (SA-S-1L, Grace Bio-Labs) em um retângulo aberto para se ajustar à área visível de um slide micro (Superfrost Plus, 25 X 75 X 1 gato mm,. # 48311-703, VWR ) com lados 2-3 mm de largura.
- Coloque fatias de cérebro dentro do centro do retângulo de fita adesiva, utilizando uma pipeta ou pincel. Remover o glicerol em excesso com a aspiração suave.
- Arrume as fatias de cérebro em slide na ordem desejada com um pincel.
- Tampa de vidro delicadamente mais baixo (24 X 60 mm, gato. # 48383-139, VWR) sobre a fita adesiva, tomando cuidado para não introduzir bolhas de ar. Aplique uma suave pressão sobre tampa de vidro para garantir o contato com fita adesiva.
- Aspirar glicerol em excesso das bordas entre lâmina e lamínula.
- Selo de slides e lamela utilizando unha polonês claro.
- Deixar secar antes de imagem, ou armazenamento de longo prazo em caixas de slides, a 4 ° C.
7. Resultados representativos:
Processamento, de imagem, e analisar o tecido cerebral fluorescente etiquetado tornaram-se indispensáveis para o estudo da neurobiologia. Muitas dessas investigações requerem sofisticados manipulações genéticas para obter expressão repórter em subconjuntos direcionados neuronal, seguido por ambas as análises de imagem de baixa e alta resolução. Muitas vezes as limitações técnicas experimentais e torná-la impossível obter estes tipos de dados a partir do mesmo animal, ou de uma forma rápida. Preparação de opticamente-desmarcada, seções semi-grossa cérebro para lâmpadas fluorescentes imagem aids neste desafio. Um exemplo de uma fatia do cérebro intacto espessura rotulados com um vírus geneticamente modificados para expressar raiva engenharia EGFP, e fotografada por microscopia de epifluorescência e confocal é mostrado na Figura 4 e Figura 5, respectivamente. Para geração de imagens de epifluorescência usamos uma Leica M205 FA, e para aquisição de imagem confocal foi utilizada uma Zeiss LSM 510. Devido à relativa simplicidade do protocolo anexo, este método é capaz de produzir dados de imagem úteis a partir do tecido expressar repórteres fluorescente com um tempo de retorno de menos de um dia, e é compatível tanto com a microscopia de luz baixa e alta resolução.
Se o pesquisador escolhe para incorporar métodos postmortem adicionais rotulagem, coloração imuno-histoquímica, ou fazer cortes mais finos, o protocolo alonga em conformidade. No entanto, o método descrito acima representa um método simples e relativamente high-throughput screening para os padrões de expressão repórter vital no cérebro intacto.
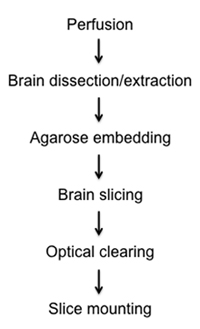
Figura 1. Fluxograma diagramação a fixação, a dissecção, corte, limpeza e procedimento de montagem de fatias de espessura semi-cérebro.
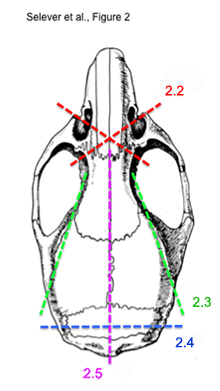
Figura 2. Diagrama de passos seqüenciais de corte para extrair o cérebro de camundongos intactos.

Figura 3. Passos para montar e cortar o tecido cerebral usando o Compresstome. A) Colocação de tecido do cérebro em corte êmbolo com supercola. B) Desenho baixo do tecido cerebral montada no êmbolo para a incorporação de agarose. C) Solidificando uma ficha cérebro agarose usando um bloco de compressão refrigerados. D) Inserção de agarose ficha cérebro e êmbolo em Compresstome câmara de corte. E) O alinhamento da lâmina de barbear para o dispositivo de êmbolo. F) Corte e coleta de fatias de cérebro em Compresstome câmara de buffer.
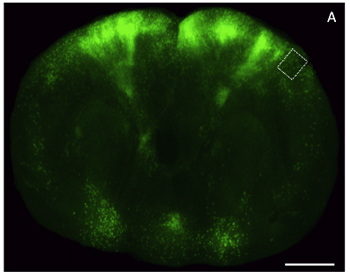
Figura 4. Imagens de microscopia de luz de uma grossa fatia de glicerol limpo tecido cerebral através do córtex frontal expressando a proteína verde fluorescente melhorada (EGFP). Uma fatia) Coronal através de um cérebro de camundongo (200 um de espessura) marcada com um vetor viral EGFP expressar e fotografada em baixa resolução usando um estereoscópio de epifluorescência. Escala de bar, 2 mm.

Figura 5. Ampliação de imagem de alta da camada fluorescente etiquetado 06/05 neurônios corticais em uma fatia do cérebro limpo de espessura. A) Imagem de alta resolução confocal de uma projeção máxima Z-stack (150 hm de espessura) através da região em destaque na (Figura 4). Barra de escala, 25 um.
Discussão
Dada a aplicação generalizada do uso de proteínas fluorescentes para atingir subconjuntos neuronal para investigação por microscopia óptica, a necessidade de rapidamente de tela, imagem, e analisar as redes neurais dentro do tecido cerebral intacto tem valor inestimável.
Avanços tecnológicos no desenvolvimento de user-friendly vetores virais, em técnicas de eletroporação in vivo, e linhagens de camundongos geneticamente modificados agora fornece uma fon...
Divulgações
Jian-Qiang Kong é um empregado de Precisionary Instruments, Inc., que fabrica um produto utilizado neste artigo.
Agradecimentos
Este trabalho foi financiado pelo apoio através da Fundação McNair, NARSAD, e conceder NINDS R00NS064171-03.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do item | Número de catálogo | Comments (opcional) | |
|---|---|---|---|
| Osso tesoura | FST | 16044-10 | Ou equivalente |
| Tesouras de dissecação | FST | 14084-08 | Ou equivalente |
| Tipo IB agarose | Sigma | A0576 | |
| Compresstome | Instrumentos Precisionary | VF-200 | -Outros vibratomes são compatíveis |
| Adesiva dupla face | Grace Bio-Labs | SA-S-1L | |
| Além disso Superfrost slides | VWR | 48311-703 | |
| Tampa de vidro | VWR | 48383-139 | |
| Glicerol | EMD Chemicals Inc. | GX0185-6 | Ou equivalente |
Referências
- Pfister, H., Lichtman, J., Reid, C. . The Connectome Project. , (2009).
- Briggman, K. L., Denk, W. Towards neural circuit reconstruction with volume electron microscopy techniques. Curr Opin Neurobiol. 16, 562-570 (2006).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Arenkiel, B. R., Ehlers, . Molecular genetic and imaging technologies for circuit based neuroanatomy. Nature. 461, 900-907 (2009).
- Capecchi, M. R. Altering the genome by homologous recombination. Science. 244, 1288-1292 (1989).
- Luo, L., Callaway, E. M., Svoboda, K. Genetic dissection of neural circuits. Neuron. 57, 634-660 (2008).
- Shimomura, O., Johnson, F., Saiga, Y. Extraction, purification, and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. J. Cell Comp Physiol. 59, 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W., Prasher, D. Gene fluorescent protein as a marker for gene expression. Science. 263, 802-805 (1994).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Svoboda, K., Yasuda, R. Principles of two-photon excitation microscopy and its applications to neuroscience. Neuron. 50, 823-839 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados