È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un metodo rapido per High-Resolution Imaging di fluorescenza in semi-spessa fettine di cervello
In questo articolo
Riepilogo
Qui si descrive un metodo rapido e semplice per celle di immagine fluorescente in semi-densi fettine di cervello. Fissando, affettare, e otticamente compensazione tessuto cerebrale si descrivono le modalità standard di imaging epifluorescente o confocale può essere utilizzato per visualizzare le singole cellule e reti neuronali all'interno intatto tessuto nervoso.
Abstract
Un obiettivo fondamentale sia di base e clinica delle neuroscienze è quello di comprendere meglio le identità, trucco molecolare, e modelli di connettività che sono caratteristici di neuroni nel cervello sia normale e malato. In questo senso, un grande sforzo è stato posto sulla costruzione di mappe ad alta risoluzione neuroanatomici 1-3. Con l'espansione della genetica molecolare e progressi nella microscopia luce è venuta la possibilità di interrogare non solo morfologie neuronale, ma anche la composizione molecolare e cellulare dei singoli neuroni e delle loro reti associate 4. Importanti progressi nella capacità di segnare e manipolare i neuroni attraverso le tecnologie transgeniche e gene targeting nei roditori ora consentire agli investigatori di sottoinsiemi neuronale 'programma' a volontà 5-6. Probabilmente, uno dei contributi più influenti di neuroscienza contemporanea è stata la scoperta e la clonazione di geni che codificano per proteine fluorescenti (PQ) in invertebrati marini 7-8, accanto ai loro ingegneria successive per produrre una sempre maggiore cassetta degli attrezzi di giornalisti vitale 9. Sfruttando cellula tipo-specifici promotore di attività mirate a guidare l'espressione FP in discrete popolazioni neuronali offre ora indagine neuroanatomici con precisione genetica.
Ingegneria espressione FP nei neuroni ha migliorato notevolmente la nostra comprensione della struttura del cervello e funzione. Tuttavia, i singoli neuroni imaging e le loro reti associate nei tessuti cerebrali profondi, o in tre dimensioni, è rimasta una sfida. A causa di alto contenuto di lipidi, tessuto nervoso è piuttosto opaco e auto presenta fluorescenza. Queste proprietà intrinseche biofisiche rendono difficile la visualizzazione e l'immagine neuroni fluorescente ad alta risoluzione con un microscopio confocale epifluorescente o al di là profondità di decine di micron. Per aggirare questo problema i ricercatori spesso utilizzano seriale sottile sezione imaging e metodi di ricostruzione 10, o 2-fotone microscopia a scansione laser 11. Svantaggi attuali a questi approcci sono associati alta intensità di lavoro di preparazione dei tessuti, o troppo costose strumentazioni rispettivamente.
Qui, vi presentiamo un metodo relativamente rapido e semplice per visualizzare le cellule fluorescente a fette topo fisso semi-densi cervello deselezionando ottici e di imaging. Nel protocollo allegato si descrivono i metodi di: 1) tessuto cerebrale di fissaggio in situ tramite perfusione intracardial, 2) la dissezione e la rimozione di intero cervello, 3) incorporando cervello stazionario in agarosio, 4) di precisione semi-densi preparazione fetta utilizzando nuovi strumenti vibratome compensazione, 5) il tessuto cerebrale da un gradiente di glicerolo, e 6) il montaggio su vetrini per microscopia ottica e z-stack ricostruzione (Figura 1).
Per la preparazione di fettine di cervello abbiamo implementato un pezzo relativamente nuovo di strumenti chiamato 'Compresstome' VF-200 (http://www.precisionary.com/products_vf200.html). Questo strumento è un semi-automatico microtomo dotato di un anticipo motorizzato e vibrazioni sistema blade con caratteristiche simili in funzione di vibratomes altri. A differenza di altri vibratomes, il tessuto da tagliare è montato in una spina agarosio all'interno di un cilindro di acciaio inox. Il tessuto viene estruso a spessori desiderati dal cilindro, e tagliare con la lama in avanti avanzando vibrante. La spina agarosio / sistema cilindro permette di tessuto riproducibile montaggio, allineamento e il taglio di precisione. Nelle nostre mani, il 'Compresstome' alte rese fette qualità dei tessuti per elettrofisiologia, immunoistochimica, e diretto fissa tessuto di montaggio e di imaging. In combinazione con compensazione ottica, qui abbiamo dimostrato la preparazione di semi-densi fettine di cervello fissata per imaging ad alta risoluzione fluorescenti.
Protocollo
1. Nella fissazione del cervello in situ
* Preparare una siringa da 10 ml (28 gauge) riempite con tampone fosfato (PBS).
* Preparare una siringa da 10 ml (28 gauge), riempito con il 4% paraformaldeide (PFA) in PBS. Riservare un ulteriore 5-10 ml di PFA / PBS per il fissaggio post.
- Iniettare intraperitoneale sperimentale del mouse con una dose letale di Avertin o Nembutal.
- Una volta che il mouse è sedato, bagnato l'addome con l'etanolo e sicuro il mouse fino al fondo di un vassoio a strati con sughero, cera, o elastomero di silicone con perni di dissezione. Con addome rivolto verso l'alto, sicuro il quattro zampe alla superficie - la diffusione di loro il più ampio possibile.
- Afferra la pelle con una pinza a livello dello sterno, e tagliato trasversalmente per esporre il fegato. Taglio lateralmente e poi attraverso le costole e il diaframma. Rimuovere il lembo costola e continuare a tagliare fino a quando il cuore è accessibile.
- Forare o tagliare l'atrio destro di drenare il sangue in circolazione.
- Filo di sangue residuo sistema circolatorio per perfusione di PBS attraverso ventricolo sinistro.
- Accesso al foro stesso ago, fissare tutto con il mouse perfusione successive di PBS / PFA attraverso ventricolo sinistro.
2. Dissezione e l'estrazione del cervello
- Togliere la pelle dalla testa, del collo e cranio, tagliando intorno canale uditivo e le orbite degli occhi, seguito da tirando la pelle anteriormente per esporre il cranio.
* Vedere la Figura 2 per una illustrazione delle fasi 2.2) a 2.5) - Utilizzando un paio di forbici osso, tagliato trasversalmente dall'occhio presa attraverso il turbinati nasali. Fate questo per ogni lato. Il taglio trasversale dovrebbe essere anteriore agli occhi e bulbi olfattivi.
- Tagliati longitudinalmente da ogni canale uditivo cavità oculare.
- Utilizzando un paio di forbici dissezione piccolo, tagliare la piastra occipitale osso sovrastante il cervelletto da un canale orecchio all'altro.
- Con le forbici dissezione stessa, tagliare anteriormente lungo la linea mediana al livello dei bulbi olfattivi.
- Utilizzando pinze, rimuovere ogni piatto osso dal cervello, avendo cura di rimuovere qualsiasi tessuto connettivo in modo che il cervello rimane intatto quando si solleva l'osso.
- Snip nervi ottici e del cranio, tagliare la parte superiore vertebre cervicali, e rimuovere il cervello in fissativo.
- Post-fix per 1-2 ore a 4 ° C. Lavare 3 volte 15 minuti ciascuno in PBS.
3. Agarosio embedding
- Fate sciogliere il 2% ad alta resistenza, basso punto di gel di agarosio di tipo IB (numero di catalogo Sigma: A0576) in PBS salina con forno a microonde.
- Trasferimento agarosio fuso in una provetta. Luogo provetta in un bagno caldo incubatore o acqua (40 ° -41 ° C) fino al raggiungimento dell'equilibrio di temperatura.
- Colla tessuto cerebrale fissata su lo stantuffo della siringa campione con colla cianoacrilato (Figura 3A). Pennello un sottile strato di satura di saccarosio-PBS soluzione sulla superficie del tessuto cerebrale per facilitare il successivo rilascio del ring agarosio dalle sezioni di cervello.
- Disegna lo stantuffo con tessuto cerebrale nella sede pistone (Figura 3B).
- Pipetta o versare calde agarosio nella sede stantuffo, che copre il tessuto cerebrale. Evitare di inglobare bolle d'aria nel agarosio. Le bolle d'aria nel agarosio comprometterà la qualità fetta.
- Bloccare il campione con una siringa freddo (0 ° a -25 ° C) blocco agghiacciante per 1 minuto per impostare l'agarosio (Figura 3C).
4. Sezionamento fetta cervello con la Compresstome
- Assemblare la siringa contenente il campione di tessuto cerebrale in Compresstome (Fig. 3D).
- Allineare il bordo della lama di un rasoio a stretto contatto con la presa della siringa campione (Fig. 3E).
- Riempire il serbatoio con la soluzione tampone PBS.
- Ruotate di uno spessore della fetta micrometro avanti per estrudere l'agarosio-cervello blocco di tessuto dalla siringa campione.
- Premere il pulsante "start" sulla centralina del Compresstome di avviare il processo di sezionamento.
- Ripetere i passaggi 4.4) a 4,5) fino a fettine desiderati sono ottenuti a determinare lo spessore.
- Cervello fette raccolto con pennello o una pipetta di fiale o pozzi colorazione piena di PBS (Figura 3F).
5. Cancellazione ottico
- Preparare un 75% vol: vol soluzione di glicerolo: PBS.
- Versare (o pipetta) via la metà del volume di PBS di lavaggio dai fettine di cervello raccolti.
- Aggiungere nuovamente il volume rimosso con glicerolo / PBS.
- Lasciare equilibrare con miscelazione delicata a 4 ° C fino a quando il lavandino fette.
- Ripetere i passaggi 6.2) a 6.4) fino a una concentrazione di glicerolo finale raggiunge il 75%.
- Sostituire la soluzione con il 90% glicerolo in PBS e lasciare equilibrare.
* Nota: fare attenzione a non introdurre bolle d'aria mentre l'aggiunta di soluzioni di glicerolo. Versare soluzioni lentamente e mescolare delicatamente, dal momento che bolle d'aria interferisce con uniforme Equilibrazione e saranno riportati a scivolare di montaggio.
6. Fetta di montaggio per l'imaging
- Taglio biadesivo sigillo (SA-S-1L, Grace Bio-Labs) in un rettangolo aperto per soddisfare l'area visibile di una diapositiva micro (Superfrost Plus, 25 X 75 X 1 mm, cat. # 48311-703, VWR ) con lati di 2-3 mm di larghezza.
- Luogo fettine di cervello all'interno del centro del rettangolo di nastro adesivo con una pipetta o pennello. Rimuovere l'eccesso di glicerolo con aspirazione delicata.
- Disporre le fette del cervello su diapositive, in sequenza desiderata con un pennello.
- Vetro di copertura Abbassare delicatamente (24 X 60 mm, cat. # 48383-139, VWR) sul nastro adesivo, facendo attenzione a non introdurre bolle d'aria. Esercitare una leggera pressione sul vetro di copertura per assicurare un contatto con del nastro adesivo.
- Aspirare il glicerolo in eccesso dai bordi tra diapositive e coprioggetti.
- Sigillo di diapositive e coprioggetto utilizzando smalto trasparente per unghie.
- Lasciare asciugare prima di imaging, o conservare a lungo termine in scatole di diapositive a 4 ° C.
7. Rappresentante dei risultati:
L'elaborazione, l'imaging, e analizzando il tessuto cerebrale fluorescente sono diventate indispensabili per lo studio della neurobiologia. Molte di queste indagini richiedono sofisticate manipolazioni genetiche per ottenere l'espressione giornalista in sottoinsiemi mirati neuronale, seguita da entrambe le analisi di immagini a bassa e ad alta risoluzione. Spesso limitazioni sperimentali e tecniche rendono impossibile ottenere questi tipi di dati dello stesso animale, o in maniera rapida. Preparazione di ottica-ripulito, semi-densi sezioni di cervello per l'imaging fluorescente aiuti in questa sfida. Un esempio di una fetta intatta del cervello di spessore etichettato con un virus geneticamente modificato per esprimere rabbia progettato EGFP, e ripreso usando la microscopia confocale e epifluorescente è mostrata in Figura 4 e Figura 5, rispettivamente. Per l'imaging epifluorescente abbiamo usato una Leica M205 FA, e per l'acquisizione di immagini confocale abbiamo usato un Zeiss LSM 510. A causa della relativa semplicità del protocollo allegato, questo metodo è in grado di produrre dati di immagini utili da esprimere reporters tessuto fluorescente con un tempo di risposta di meno di un giorno, ed è compatibile sia con microscopia ottica a bassa e ad alta risoluzione.
Se il ricercatore sceglie di incorporare ulteriori modalità di etichettatura post-mortem, immunoistochimica, o fare sezioni più sottili, il protocollo si allunga di conseguenza. Tuttavia, il metodo di cui sopra rappresenta un metodo semplice e relativamente high-throughput per lo screening modelli di espressione giornalista vitale nel cervello intatto.
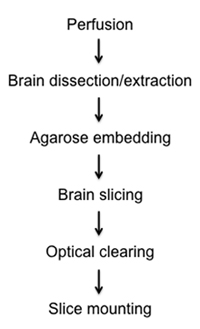
Figura 1. Diagramma di flusso diagraming la fissazione, dissezione, affettare, radura, e la procedura di montaggio semi-densi fettine di cervello.
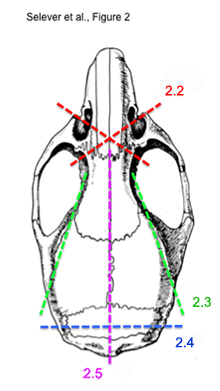
Figura 2. Schema di passi sequenziali taglio per estrarre il cervello intatto mouse.

Figura 3. Passaggi per montare e tagliare tessuti cerebrali usando la Compresstome. A) Posizionamento del tessuto cerebrale sul pistone di taglio con supercolla. B) Disegno del tessuto cerebrale montato nel pistone per l'incorporamento agarosio. C) Consolidare una spina del cervello agarosio utilizzando un blocco raffreddato compressione. D) Inserimento di plug cervello agarosio e stantuffo in camera di taglio Compresstome. E) L'allineamento di lametta al dispositivo stantuffo. F) Taglio e raccolta di fette di cervello in camera di buffer di Compresstome.
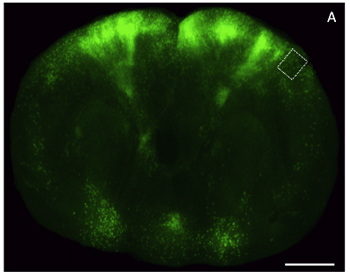
Figura 4. Immagini di microscopia luce di una spessa fetta di glicerolo-ripulito tessuto cerebrale attraverso la corteccia frontale che esprime maggiore proteina fluorescente verde (EGFP). A) fetta coronale attraverso un cervello di topo (200 um di spessore) etichettato con un vettore virale EGFP esprimere e ripreso a bassa risoluzione con un stereoscopio epifluorescente. Scala grafica, 2 mm.

Figura 5. Immagine Alto ingrandimento fluorescente strato di 5 / 6 neuroni corticali in una fetta spessa cancellata cervello. A) immagine confocale ad alta risoluzione di una proiezione massima Z-stack (150 um di spessore) attraverso la regione evidenziata in (Figura 4). Scala grafica, 25 um.
Discussione
Dato l'applicazione diffusa di utilizzare proteine fluorescenti a bersaglio sottoinsiemi neuronale per le indagini tramite microscopia ottica, la necessità di rapido dello schermo, l'immagine, e analizzare le reti neurali all'interno del tessuto cerebrale intatto è diventata insostituibile.
I progressi tecnici nello sviluppo di vettori virali user-friendly, nelle tecniche di elettroporazione in vivo, e ceppi di topi geneticamente modificati, off...
Divulgazioni
Jian-Qiang Kong è un dipendente di Precisionary Instruments, Inc., che produce un prodotto utilizzato in questo articolo.
Riconoscimenti
Questo lavoro è stato finanziato con il sostegno attraverso la Fondazione McNair, NARSAD e NINDS concedere R00NS064171-03.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome della voce | Azienda | Numero di catalogo | Commenti (opzionale) |
|---|---|---|---|
| Osso forbici | FST | 16044-10 | -O equivalente |
| Dissezione forbici | FST | 14084-08 | -O equivalente |
| Tipo IB agarosio | Sigma | A0576 | |
| Compresstome | Strumenti Precisionary | VF-200 | -Altri vibratomes sono compatibili |
| Biadesivo | Grace Bio-Labs | SA-S-1L | |
| Superfrost Inoltre diapositive | VWR | 48311-703 | |
| Copertura in vetro | VWR | 48383-139 | |
| Glicerolo | EMD Chemicals Inc. | GX0185-6 | -O equivalente |
Riferimenti
- Pfister, H., Lichtman, J., Reid, C. . The Connectome Project. , (2009).
- Briggman, K. L., Denk, W. Towards neural circuit reconstruction with volume electron microscopy techniques. Curr Opin Neurobiol. 16, 562-570 (2006).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Arenkiel, B. R., Ehlers, . Molecular genetic and imaging technologies for circuit based neuroanatomy. Nature. 461, 900-907 (2009).
- Capecchi, M. R. Altering the genome by homologous recombination. Science. 244, 1288-1292 (1989).
- Luo, L., Callaway, E. M., Svoboda, K. Genetic dissection of neural circuits. Neuron. 57, 634-660 (2008).
- Shimomura, O., Johnson, F., Saiga, Y. Extraction, purification, and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. J. Cell Comp Physiol. 59, 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W., Prasher, D. Gene fluorescent protein as a marker for gene expression. Science. 263, 802-805 (1994).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-224 (2006).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55, 25-36 (2007).
- Svoboda, K., Yasuda, R. Principles of two-photon excitation microscopy and its applications to neuroscience. Neuron. 50, 823-839 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon