Method Article
De alta capacidade de Titulação de luciferase expressando recombinantes Vírus
Neste Artigo
Resumo
This article presents a high-throughput luciferase expression-based method of titrating various RNA and DNA viruses using automated and manual liquid handlers.
Resumo
Standard plaque assays to determine infectious viral titers can be time consuming, are not amenable to a high volume of samples, and cannot be done with viruses that do not form plaques. As an alternative to plaque assays, we have developed a high-throughput titration method that allows for the simultaneous titration of a high volume of samples in a single day. This approach involves infection of the samples with a Firefly luciferase tagged virus, transfer of the infected samples onto an appropriate permissive cell line, subsequent addition of luciferin, reading of plates in order to obtain luminescence readings, and finally the conversion from luminescence to viral titers. The assessment of cytotoxicity using a metabolic viability dye can be easily incorporated in the workflow in parallel and provide valuable information in the context of a drug screen. This technique provides a reliable, high-throughput method to determine viral titers as an alternative to a standard plaque assay.
Introdução
Classical viral plaque assays continue to be a mainstay in virus research even though they can be notoriously time-consuming and constitute a significant bottleneck to obtaining results from experiments. More rapid indirect virus quantification methods have emerged including quantitative polymerase chain reaction (qPCR), ELISA, and flow cytometry1-4. Recent innovations such as the Virocyt virus counter can directly count viruses using advanced flow sorting technology and through a combination of protein and DNA/RNA dyes5. While all of these methods have undoubtedly quickened the pace of virus research, each method has its advantages and drawbacks. For example, qPCR can allow for quantification of specific viral genome sequence but cannot effectively discriminate infectious from defective virions6. ELISAs can be very specific however require a suitable antibody against the desired target viral protein and can be very expensive. While flow cytometry technology offers many advantages and has improved significantly, throughput and accessibility to highly specialized equipment nonetheless remains a hurdle. Importantly, all of these techniques are not ideally suited for high-throughput screening, for which the ease and time requirement of the virus quantification step is of critical importance.
Here we describe a high-throughput and easily automatable technique to titer viruses that express a Firefly luciferase (Fluc) transgene. This method generates approximate viral titers in a test sample based on luminescence signal reads through the parallel use of a standard curve of known amounts of virus. Samples containing unknown quantities of luciferase-expressing virus are transferred on to a permissive “plaquing” cell line in parallel with the standard virus dilution curve and virus-associated luminescence is read after a few hours incubation time. This allows for rapid, quantitative, often same-day generation of results, unlike classic plaque assay protocols which typically require several days of incubation in order to manually count visible plaques7-9.
The protocol outlines the steps of our titration method using oncolytic Vesicular Stomatitis Virus encoding a Fluc transgene (VSV∆51-Fluc) as an example and provides an overview of 1. Sample preparation 2. The plating of a permissive cell line for virus titration using an automated dispenser 3. The preparation of the viral standard curve 4. The transfer of the sample supernatants onto the permissive cell line using a 96-well manual pipettor 5. The assessment of sample cytotoxicity using a cell viability reagent 6. The preparation of the luciferin substrate 7. Reading of bioluminescence and 8. Data analysis.
Protocolo
Preparação 1 Amostra
- Obtenção de amostras contendo vírus que expressam a luciferase (aqui VSVΔ51-Fluc como exemplo) para a titulação e a transferência para placas de 96 poços. Alternativamente, realizar infecções em placas de 96 poços de cultura de tecidos e os sobrenadantes usar diretamente.
- Deixe duas colunas não tratadas para a inclusão de curvas padrão. Por experiências feitas directamente nas placas de 96 poços, de título no final da experiência (40 horas pós-infecção) ou armazenar a -80 ° C e titulação em data posterior.
2: Preparação de células permissivas para o vírus da Titulação
- 24 h antes da titulação, preparar uma suspensão de células Vero, a uma concentração de 2,5 x 10 5 células / ml em meio de Eagle modificado por Dulbecco (DMEM) contendo 10% de soro fetal bovino (FBS), HEPES 30 mM, e 1% de penicilina / estreptomicina. NOTA: Apesar de este protocolo utiliza células Vero, qualquer linha celular permissiva adequado para uma infecção VSV poderia be usado.
- Semente de 2,5 x 10 4 células (100 ul) em placas de fundo plano de 96 poços sólidos brancos, utilizando um dispensador de microplacas.
- Preparar 12 ml de suspensão de células por placa de mais 5 ml de preparação.
- Limpo microplaca cassete dispensador, através de lavagem com o tubo de 50 ml de água estéril.
- Encha as linhas de microplacas dispensador com suspensão de células e deixou 5 ml de suspensão de células fluir.
- Selecione um programa que irá dispensar 100 mL em cada poço de uma placa de 96 poços de paredes brancas. Dispensar, e repetir se necessário para placas adicionais. Sementes também alguns poços numa placa de 96 poços de fundo plano transparente se placas de paredes brancas opacas de fundo são usados para verificação da saúde das células e densidade.
- Quando terminar, lave as células de volta para o recipiente original. Limpar o cartucho através da execução de 50 ml de etanol a 70% seguido por 50 ml de água esterilizada quente através do tubo.
- Certifique-cassete e tubos são apdamente limpo entre os usos.
- Incubar as células durante 24 horas a 37 ° C numa atmosfera humidificada com 5% de CO 2 incubadora.
3 Preparação da curva padrão viral
- Prepara-se uma curva padrão de VSVΔ51-Fluc em DMEM isento de soro, tal que a concentração final de unidades formadoras de placas (pfu) por ml depois de se transferir para as células Vero, é como se segue: 10 8 pfu / ml, de 10 7 pfu / ml, 10 6 pfu / ml, de 10 5 pfu / mL, 10 4 ufp / mL, 10 3 ufp / mL, 2 10 pfu / ml, e 10 1 ufc / ml. Prepare 50 mL de cada concentração por placa de células Vero, acrescido de um adicional de 10%.
NOTA: Titer do estoque de vírus luciferase terá de ser avaliado de uma forma clássica de 10, a fim de gerar uma curva padrão que permita a quantificação absoluta. Caso contrário, a quantificação relativa pode ser obtida sem informação precisa titulação ajustando arbitrariamente título viral baseadoem passos de diluição. Por exemplo, a primeira diluição da curva padrão pode ser definido como 10 8 unidades virais e a diluição 1/10 a 10 seguintes e assim por diante 7. Neste contexto, deve-se expressar valores como um fold-change em comparação com uma amostra pré-determinada como a quantificação absoluta não será preciso.
4. Transferência de sobrenadantes de exemplo para células permissivas
- Verificar as células Vero plaqueadas no fundo claro-placa de 96 poços sob um microscópio de luz para confirmar que as monocamadas são pelo menos 95% confluentes.
- Transferir 25 mL de amostra de sobrenadante sobre as células Vero semeados em placas de paredes brancas. Não transferir sobrenadantes nas duas colunas designadas por curvas padrão. NOTA: Este pode ser realizado simultaneamente em todos os poços de uma única chapa através de um manipulador de líquidos de 96 canais.
- Usando uma pipeta de multi-canal 8 ou 12 canais, adicionar 25 ul de cada diluição da curva padrão preparada no Passo 3 para as células Veroas duas colunas designadas.
- Centrifugar as placas durante 5 min a 430 xg à temperatura ambiente.
- Incubar durante 5 horas a 37 ° C numa atmosfera humidificada com 5% de CO 2 incubadora.
5. avaliação da viabilidade celular
- NOTA: Ao iniciar a partir de sobrenadantes obtidos a partir de experiências de infecção Realizados em claras placas de 96 poços, a viabilidade da amostra pode ser avaliada antes da quantificação com um corante indicador de viabilidade celular, tais como resazurina.
- Adicionar resazurina em uma quantidade igual a 10% do volume, em cada poço da placa de 96 poços de amostras que contêm vírus e as células. Incluir celular só controla bem como o controle apenas pela mídia para determinar os valores de 100% e 0% de viabilidade, respectivamente.
- Depois de 2-4 horas (tempo de incubação pode variar, dependendo do tipo de célula e da concentração de pó de corante comercialmente disponível ou reconstituído), ler e gravar o sinal utilizando um leitor de fluorescência de placas (530-560 excitação, 590 de emissão). Relatório viabili celularty para uma amostra de acordo com a fórmula de Atividade = ((sinal da amostra de teste - sinal de controle negativo) / (Cell só sinal de controle - sinal de controle negativo)) Relativa Metabólica x 100%
6 Preparação do substrato luciferina
- 30 minutos antes do final do período de incubação de 5 horas, a luciferina preparar para se obter uma solução / ml 2 mg em tampão fosfato salino estéril (PBS). Prepare 2,5 ml por placa, mais um extra de 2 ml. Proteger a solução da luz. NOTA: A luciferina pode também ser preparado no início do dia e armazenadas a 4 ° C até à sua utilização.
7 Leitura Bioluminescência
- Após o período de incubação de 5 h, adicionar 25 ul de luciferina para cada poço de células Vero em placas de sólidos brancos. Adicionar luciferina manualmente ou com um distribuidor automático integrados no luminómetro. NOTA: O uso de um instrumento com apenas um único ponto e no final lê pode exigir a adição de um buf lise compatível luciferasefer antes da adição do substrato luciferina para melhorar a consistência.
- Ler as placas com os seguintes parâmetros:
- Agitar durante 5 seg.
- Espere 30 segundos.
- Leia luminescência em um valor de sensibilidade / exposição fixa apropriado. Se a opção estiver disponível no instrumento, use multi-ponto lê (por exemplo, 3 x 3 matriz) para melhorar a precisão.
- Gravar e salvar a intensidade bioluminescente quantificado.
- Se um distribuidor automático foi usado para adicionar luciferina purgar a luciferina e injetar as linhas com água esterilizada morna.
Análise de Dados 8.
- Para resultados precisos, resolver a regressão não-linear para gerar uma equação de Hill a partir das curvas padrão. Aplicar esta equação para as amostras tituladas para calcular unidades de expressão virais. Alguns vírus ou situações podem produzir uma curva padrão com uma relação linear; neste caso, para resolver uma equação linear.
Resultados
Um resumo do fluxo que descreve o método de alto rendimento é ilustrada na Figura 1. Figura 2 mostra os resultados de uma curva padrão típica ofVSVΔ51-Fluc. Quatro parâmetros de análise de regressão não-linear gerada uma trama monte a partir do qual desconhecido pfu de entrada (estimativa do título viral) podem ser interpolados. Estes títulos são estimados denominado unidades de expressão viral (VEU). Figura 3A mostra Veus e os títulos obtidos através da realização de um ensaio de placas padrão com as mesmas amostras 10. As amostras provenientes de um experimento em que foram utilizados vários produtos químicos para aumentar a replicação e disseminação de VSVΔ51-Fluc em 786-0 células. Veus interpolado a partir da curva padrão tem de ser multiplicado por um factor que é baseado na diluição de sobrenadantes das amostras serem transferidos para células Vero (no presente caso, o factor de diluição é 5). A correlação linear entre os títulos de VEU e é mostrado na Figura 3B Com um R 2 de 0,8083 e pontuação r de Pearson de 0,899 (p <0,0001). Na Figura 4, os dados típicos de citotoxicidade de células obtidas a partir de um ensaio metabólico é mostrado para 786-0 células tratadas com produtos químicos antes da infecção com VSVΔ51-Fluc. Finalmente, a Figura 5 mostra curvas típicas padrão obtidas com vários vírus (vírus herpes simplex (HSV), vírus vaccinia e vírus adeno-associado (AAV), todos expressando a luciferase do pirilampo) e descreve os tempos de incubação e dos ciclos de congelamento e descongelamento, tal como requerido.

Figura 1 Fluxo de trabalho de titulação de vírus de alto rendimento e ensaio de citotoxicidade utilizando VSV Δ 51 Fluc. (A) 2,5 x 10 Semente 4 células Vero por poço (100 ul) e incubar a 37 ° C. (B) 24hr depois de transferência 25 ul de curva padrão sobre a células Vero (2 colunas por placa). (C) Transferir 25 uL de amostras para ser titulado em células Vero às restantes. Centrifugar as placas e incuba-se a 37 ° C. (D) numa quantidade igual a 10% do volume da cavidade, adicionar o reagente de resazurina a placa contendo as amostras originais. Incubar as placas a 37 ° C (E). Depois de 3 horas, ler e registar a fluorescência e avaliar a citotoxicidade. (F) Depois de 5 h, adicionar 25 ul de uma solução 2 mg / ml de luciferina para cada poço de células Vero. Leia luminescência e calcular unidades de expressão virais.
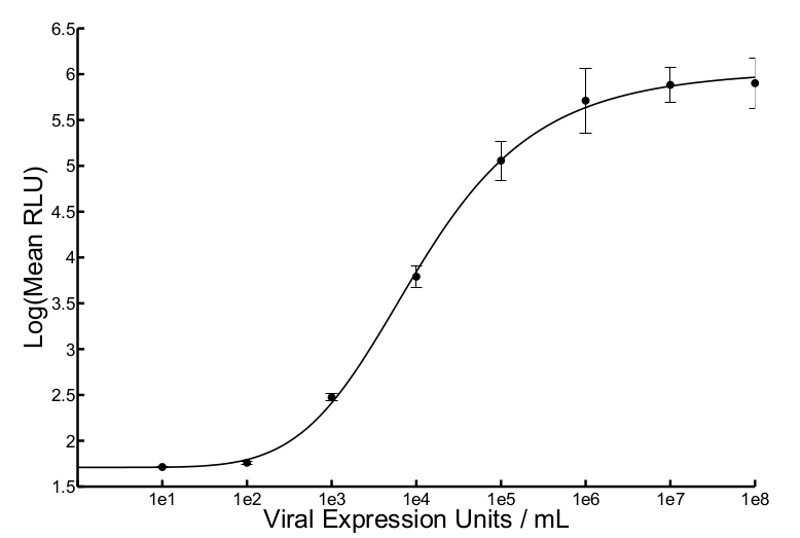
Figura 2. esperado curva padrão de VSV Δ 51-Fluc. Expressão da luciferase era measured 5 hr transferência pós sobrenadante em cinco pontos diferentes dentro de um poço usando uma luminometer e bioluminescência foi expresso em médias unidades relativas de luz (RLU). A média de RLU foi representada graficamente contra a entrada conhecida pfu / ml para resolver a regressão não-linear e gerar uma equação de Hill. A média de duas réplicas de curvas e as barras de erro padrão são apresentados (r2 = 0,9993).

Figura 3 Comparação de títulos de ensaio de placa padrão com os obtidos pelo método de alto rendimento. (A) Os títulos virais em pfu / ml obtido por ensaio de placas em células Vero padrão foram comparados com os títulos virais calculadas (VEU / ml) a partir das mesmas amostras tituladas utilizando o ensaio de elevado débito da luciferase. (B) a relação linear entre VEU / ml e através de titulação padrão de pensaio laque. Curva de regressão linear e coeficiente de determinação (R 2) são mostrados.

Figura 4 Amostra viabilidade. Viabilidade amostra antes da transferência de sobrenadante de células Vero foi determinada avaliando a actividade metabólica celular, utilizando uma solução resazurina disponível comercialmente. Os valores de fluorescência em bruto foram normalizados para a de não tratadas não infectadas, poços.
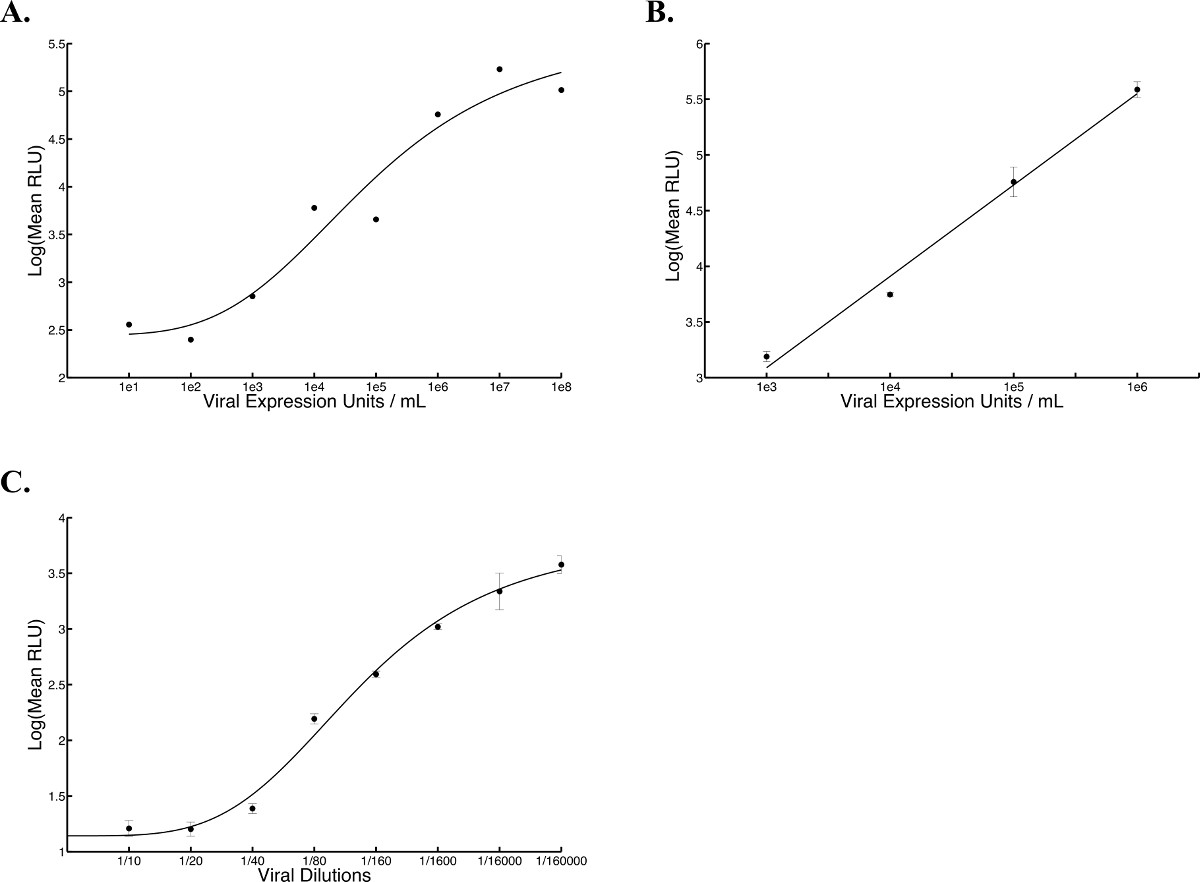
Figura 5 Esperado curvas padrão com HSV, vaccinia e vírus AAV. Expressão da luciferase foi medida em cinco pontos diferentes dentro de um poço, utilizando um luminómetro e bioluminescência foi expressa em rela médiosunidades de luz tiva (RLU). (A) HSV curva padrão foi adicionado em células Vero plaqueadas 24 horas antes a uma densidade de 2,5 x 10 4 culas por po (100 ul) e medição da luciferase foi feita 17 horas pós transferência sobrenadante (R2 = 0,9489, n = 1). Uma equação de Hill foi gerada pela solução da regressão não-linear. (B) Vaccinia curva padrão foi adicionado em células Vero chapeado 24 h antes de uma densidade de 2,5 x 10 4 culas por po (100 ul) e incubou-se durante 2,5 horas a 37 ° C, após o que foram tomadas medidas de luciferase (R2 = 0,9892). Uma equação linear foi gerado por resolver para a regressão linear. (C) de AAV curva padrão foi adicionado em células de carcinoma de pulmão humano (A549) chapeado 24 horas antes a uma densidade de 2,5 x 10 4 culas por po (100 ul) e medição da luciferase foi feito 24 horas após a infecção. Uma equação de Hill foi gerada pela solução da regressão não-linear. A médiade cinco curvas em duplicado eo barras de erro padrão são mostrados (R 2 = 0,9926).
Discussão
A abordagem aqui descrita luciferase oferece um número de vantagens sobre os outros métodos existentes, incluindo a sua facilidade, rapidez, um equipamento mínimo necessário, e de custo relativamente baixo. Um contribuidor chave para isso é a prevenção de uma etapa de diluição serial. No entanto, os derivados à base de diluição em série deste protocolo são certamente viável e recentemente foram utilizados para avaliar expressando luciferase títulos de Ebola em uma tela antiviral de alto rendimento 11. Enquanto inerentemente mais demorado e mais caro, essas adaptações podem proporcionar uma maior gama dinâmica para quantificação viral quando necessário. Além de ser particularmente adequado para avaliar os títulos virais no contexto de telas de alto rendimento, o nosso método de quantificação viral a partir de uma etapa de luciferase gera estimativas precisas dos viriões infecciosos, no caso de se replicar os vírus. Além disso, as leituras de luminescência ao longo de citotoxicidade com dados a partir do mesmo experimento dar um maisimagem completa do efeito das condições experimentais em células alvo, o que é especialmente útil no contexto de vírus oncolí ticos e telas de drogas.
O exemplo mostrado aqui utiliza um vírus RNA fita única replicando negativo; No entanto, este protocolo pode ser adaptado a um número de replicação e não replicação de vírus com alguns ajustamentos menores do protocolo. Isto inclui os vírus de ADN tais como Vaccinia, o HSV, e de AAV (ver Figura 5). As amostras infectadas com os vírus intracelulares, tais como vírus vaccinia, por exemplo, requerem um passo de libertação antes da quantificação do vírus (por exemplo, pelo menos um ciclo de congelação-descongelação). Quando se aplica esta técnica para outros vírus, o que é necessário para optimizar o tempo de incubação da transferência do sobrenadante viral ou lisado para a leitura das placas pelo luminómetro. Este parâmetro depende principalmente no ciclo de replicação do vírus na linha celular permissiva e a força do promoter dirigir a expressão da luciferase. Este é o melhor feito usando a curva padrão completo na etapa de otimização. Para fazer isso, é necessário infectar a linha celular permissiva apropriado com várias repetições da curva padrão preparada e ler cada replicar num momento diferente de pontos de pós-transferência. Idealmente, um ponto de tempo de incubação é escolhido de que conduz a uma relação linear entre LOG (RLU) e LOG (título) que abrange o intervalo de amostra título esperado. Para VSVΔ51-Fluc, este é tipicamente a partir de 10 4 pfu / ml -10 7 pfu / ml para um tempo de incubação de 5 h. Se os títulos mais baixos ou mais elevados são esperados a partir de amostras, pode-se simplesmente aumentar ou reduzir o tempo de incubação, respectivamente. Como alternativa, as amostras podem ser diluídas para cair dentro da gama muito como é feito tipicamente por ELISA.
Como mencionado acima, este método é também adequado para realizar telas de alto rendimento de drogas usando bibliotecas de drogas como a maior parte dos passos pode ser automatizado. As células podem ser plaqueadas efficiently utilizando um dispensador automático de microplacas, a biblioteca de fármacos pode ser adicionado utilizando um manipulador de líquidos de 96 canais, o vírus pode ser adicionado utilizando um dispensador de microplacas e placas lida utilizando um luminómetro automatizado. Em teoria, este também pode ser adaptado para 384 poços ou formatos mais pequenos; no entanto, a limitação para este fim é o número de células que podem ser banhados, dadas menos células leva a uma faixa mais estreita na linearidade do LOG (RLU) a relação log (Titer). Finalmente, avaliação da viabilidade celular utilizando resazurina ou outros corantes metabólicos podem ser facilmente incorporados no fluxo de trabalho, permitindo a discriminação de compostos citotóxicos em telas antivirais ou identificação de compostos que levam à morte sinérgica em combinação com o vírus 12. No entanto, as limitações deste método incluem a necessidade de um vírus que expressa transgene da luciferase, o que nem sempre é possível, e a disponibilidade de uma linha celular suficientemente permissiva. No entanto, é provável possível adaptaro método para ser utilizado com outros genes repórter (por exemplo, GFP), desde que o método de quantificação repórter tem uma linearidade adequada e a relação sinal-ruído. No geral, o método de alto rendimento descrito pode ser modificado para atender muitos vírus diferentes e adaptadas às diversas aplicações.
Divulgações
The authors have nothing to disclose.
Agradecimentos
Vanessa Garcia is funded by a Queen Elizabeth II Ontario Graduate Scholarship in Science and Technology and Cory Batenchuk by a Natural Sciences and Engineering Research Council fellowship.
Materiais
| Name | Company | Catalog Number | Comments |
| Dulbeccos' Modified Eagle's Medium (DMEM) | Corning | SH30243.01 | |
| Fetal bovine serum | NorthBio Inc. | NBSF-701 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| HEPES | Fisher Scientific | BP310-1 | Prepare a 1 mM solution, pH 7.3 |
| alamarBlue | AbD Serotec | BUF012B | |
| D-Luciferin potassium salt | Biotium | 10101-2 | |
| 96-well solid white flat bottom polystyrene TC-treated microplates | Corning | 3917 | 384-well plates can also be used for higher throughput |
| Synergy Mx | BioTek | SMTBL | Monochromator microplate reader |
| Liquidator96 | Mettler Toledo | LIQ-96-200 | 96 tip manual pipetting system |
| Liquidator96 LTS Tips sterilized with filters | Mettler Toledo | LQR-200F | Any sterile filtered tips compatible with pipettors of choice are appropriate |
| Microflo | BioTek | 111-206-21 | Used to plate cells in a 96-well plate |
| Fluoroskan Ascent FL | Thermo Scientific | 5210450 | Microplate fluorometer |
Referências
- Grigorov, B., Rabilloud, J., Lawrence, P., Gerlier, D. Rapid titration of measles and other viruses: optimization with determination of replication cycle length. PLoS One. 6 (e24135), (2011).
- Lizee, G., et al. Real-time quantitative reverse transcriptase-polymerase chain reaction as a method for determining lentiviral vector titers and measuring transgene expression. Hum Gene Ther. 14, 497-507 (2003).
- McSharry, J. J. Uses of flow cytometry in virology. Clin Microbiol Rev. 7, 576-604 (1994).
- Watcharatanyatip, K., et al. Multispecies detection of antibodies to influenza A viruses by a double-antigen sandwich ELISA. J Virol Methods. 163, 238-243 (2010).
- Dawson, E. Rapid, Direct Quantification of Viruses in Solution Using the ViroCyt Virus Counter. Journal of Biomolecular Techniques. 23 (S10), (2012).
- Snyder, R. O. AAV and RT-PCR: true or false. Mol Ther. 1, 389-390 (2000).
- Diallo, J. S., Roy, D., Abdelbary, H., De Silva, N., Bell, J. C. Ex vivo infection of live tissue with oncolytic viruses. J Vis Exp. 52, (2011).
- Gaush, C. R., Smith, T. F. Replication and plaque assay of influenza virus in an established line of canine kidney cells. Appl Microbiol. 16, 588-594 (1968).
- Green, M., Loewenstein, P. M. Human adenoviruses: propagation, purification, quantification, and storage. Curr Protoc Microbiol. 14 (14C 11), (2006).
- Diallo, J. S., Vaha-Koskela, M., Le Boeuf, F., Bell, J. Propagation, purification, and in vivo testing of oncolytic vesicular stomatitis virus strains. Methods Mol Biol. 797, 127-140 (2012).
- Hoenen, T., Groseth, A., Callison, J., Takada, A., Feldmann, H. A novel Ebola virus expressing luciferase allows for rapid and quantitative testing of antivirals. Antiviral Res. 99, 207-213 (2013).
- Diallo, J. S., et al. A high-throughput pharmacoviral approach identifies novel oncolytic virus sensitizers. Mol Ther. 18, 1123-1129 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados