Method Article
High-throughput Titolazione della luciferasi che esprimono ricombinanti virus
In questo articolo
Riepilogo
This article presents a high-throughput luciferase expression-based method of titrating various RNA and DNA viruses using automated and manual liquid handlers.
Abstract
Standard plaque assays to determine infectious viral titers can be time consuming, are not amenable to a high volume of samples, and cannot be done with viruses that do not form plaques. As an alternative to plaque assays, we have developed a high-throughput titration method that allows for the simultaneous titration of a high volume of samples in a single day. This approach involves infection of the samples with a Firefly luciferase tagged virus, transfer of the infected samples onto an appropriate permissive cell line, subsequent addition of luciferin, reading of plates in order to obtain luminescence readings, and finally the conversion from luminescence to viral titers. The assessment of cytotoxicity using a metabolic viability dye can be easily incorporated in the workflow in parallel and provide valuable information in the context of a drug screen. This technique provides a reliable, high-throughput method to determine viral titers as an alternative to a standard plaque assay.
Introduzione
Classical viral plaque assays continue to be a mainstay in virus research even though they can be notoriously time-consuming and constitute a significant bottleneck to obtaining results from experiments. More rapid indirect virus quantification methods have emerged including quantitative polymerase chain reaction (qPCR), ELISA, and flow cytometry1-4. Recent innovations such as the Virocyt virus counter can directly count viruses using advanced flow sorting technology and through a combination of protein and DNA/RNA dyes5. While all of these methods have undoubtedly quickened the pace of virus research, each method has its advantages and drawbacks. For example, qPCR can allow for quantification of specific viral genome sequence but cannot effectively discriminate infectious from defective virions6. ELISAs can be very specific however require a suitable antibody against the desired target viral protein and can be very expensive. While flow cytometry technology offers many advantages and has improved significantly, throughput and accessibility to highly specialized equipment nonetheless remains a hurdle. Importantly, all of these techniques are not ideally suited for high-throughput screening, for which the ease and time requirement of the virus quantification step is of critical importance.
Here we describe a high-throughput and easily automatable technique to titer viruses that express a Firefly luciferase (Fluc) transgene. This method generates approximate viral titers in a test sample based on luminescence signal reads through the parallel use of a standard curve of known amounts of virus. Samples containing unknown quantities of luciferase-expressing virus are transferred on to a permissive “plaquing” cell line in parallel with the standard virus dilution curve and virus-associated luminescence is read after a few hours incubation time. This allows for rapid, quantitative, often same-day generation of results, unlike classic plaque assay protocols which typically require several days of incubation in order to manually count visible plaques7-9.
The protocol outlines the steps of our titration method using oncolytic Vesicular Stomatitis Virus encoding a Fluc transgene (VSV∆51-Fluc) as an example and provides an overview of 1. Sample preparation 2. The plating of a permissive cell line for virus titration using an automated dispenser 3. The preparation of the viral standard curve 4. The transfer of the sample supernatants onto the permissive cell line using a 96-well manual pipettor 5. The assessment of sample cytotoxicity using a cell viability reagent 6. The preparation of the luciferin substrate 7. Reading of bioluminescence and 8. Data analysis.
Protocollo
Preparazione del campione 1
- Ottenere campioni contenenti-luciferasi che esprimono virus (qui VSVΔ51-Fluc come esempio) per la titolazione e il trasferimento in piastre da 96 pozzetti. In alternativa, eseguire infezioni in 96 pozzetti piastre di coltura di tessuti e uso surnatanti direttamente.
- Lascia due colonne non trattati per l'inserimento di curve standard. Per gli esperimenti fatti direttamente in piastre a 96 pozzetti, titolo alla fine dell'esperimento (40 ore post-infezione) o conservare a -80 ° C e titolo in una data successiva.
2 Preparazione di cellule permissive per Virus Titolazione
- 24 ore prima della titolazione, preparare una sospensione di cellule Vero ad una concentrazione di 2,5 x 10 5 cellule / ml in mezzo di Eagle modificato di Dulbecco (DMEM) contenente siero bovino fetale al 10% (FBS), HEPES 30 mM e 1% di penicillina / streptomicina. NOTA: Anche se questo protocollo utilizza cellule Vero, ogni linea cellulare permissiva adatto per l'infezione da VSV potrebbe be utilizzato.
- Seed 2,5 x 10 4 cellule (100 microlitri) in piastre a fondo piatto solido 96 pozzetti bianchi con un dispenser di micropiastre.
- Preparare 12 ml di sospensione cellulare per piastra più 5 ml per adescamento.
- Pulire micropiastre cassette dispenser dal lavaggio del tubo con 50 ml di acqua sterile.
- Riempire le linee dispenser micropiastra con sospensione cellulare e lasciare 5 ml di sospensione cellulare fluire attraverso.
- Selezionare un programma che dispensare 100 ml in ciascun pozzetto di una piastra a 96 pozzetti bianco-murato. Dispensare, e ripetere se necessario per piastre supplementari. Sementi anche alcuni pozzi in una chiara piatto fondo piatto da 96 pozzetti, se si utilizzano le piastre bianche pareti opache-fondo per la verifica della salute delle cellule e la densità.
- Al termine, le celle a filo nel contenitore originale. Pulire la cassetta eseguendo 50 ml di etanolo al 70% seguita da 50 ml di acqua sterile calda attraverso il tubo.
- Assicurarsi cassette e tubi sono aptamente puliti tra usi.
- Incubare le cellule per 24 ore a 37 ° C in un umidificata al 5% di CO 2 incubatore.
3 Preparazione di Viral curva standard
- Preparare una curva standard di VSVΔ51-Fluc in DMEM senza siero tale che la concentrazione finale delle unità formanti placca (PFU) per ml dopo il trasferimento su cellule Vero è la seguente: 10 8 pfu / ml, 10 7 pfu / ml, 10 6 pfu / ml, 10 5 pfu / ml, 10 4 pfu / ml, 10 3 pfu / ml, 10 2 pfu / ml, e 10 1 pfu / ml. Preparare 50 ml di ciascuna concentrazione per piastra di cellule Vero, più un ulteriore 10%.
NOTA: Titer del virus magazzino luciferasi dovrà essere valutata in modo classico 10, al fine di generare una curva standard che permetterà per la quantificazione assoluta. Altrimenti relativa quantificazione può essere raggiunto senza informazioni precise titolo fissando arbitrariamente titolo virale basatasui gradini di diluizione. Ad esempio la prima diluizione della curva standard può essere impostato su 10 8 unità virali e la diluizione 1/10 testo seguente 10 7 e così via. In questo contesto, si dovrebbe esprimere i valori come un fold-variazione rispetto ad un campione predeterminato come quantificazione assoluta non sarà preciso.
4 Trasferimento di surnatante del campione su celle di consenso
- Controllare cellule Vero placcato nella piastra a 96 pozzetti con fondo trasparente sotto un microscopio ottico per confermare che monostrati sono almeno il 95% confluenti.
- Trasferire 25 ml di campione di surnatante su cellule Vero seminate in piastre bianche pareti. Non trasferire surnatanti nelle 2 colonne designate per le curve standard. NOTA: Questo può essere fatto contemporaneamente per tutti i pozzetti su un unico piatto utilizzando un gestore di liquido a 96 canali.
- Utilizzando una pipetta multicanale a 8 o 12 canali, aggiungere 25 ml di ogni diluizione della curva standard preparata al punto 3 per le cellule Vero inle 2 colonne designate.
- Piastre di centrifugazione per 5 min a 430 xg a temperatura ambiente.
- Incubare per 5 ore a 37 ° C in un umidificata al 5% di CO 2 incubatore.
5 Valutazione della vitalità cellulare
- NOTA: Quando si avvia da surnatanti ottenuti da esperimenti di infezione fatto in piastre a 96 pozzetti chiari, la redditività del campione può essere valutata prima di quantificazione con un indicatore di vitalità cellulare colorante come resazurina.
- Aggiungere resazurina in quantità pari al 10% del volume in ciascun pozzetto della piastra a 96 pozzetti di campioni contenenti virus e cellule. Includi cellulare solo controlla e di controllo solo media per determinare rispettivamente i valori per il 100% e 0% vitalità.
- Dopo 2-4 ore (tempo di incubazione varia a seconda del tipo di cellula e sulla concentrazione di disponibili in commercio o in polvere ricostituito del colorante), leggere e registrare il segnale utilizzando un lettore di piastre a fluorescenza (eccitazione 530-560, 590 emissione). Relazione viabili cellularety per un campione in base alla formula relativa attività metabolica = ((segnale campione di prova - segnale di controllo negativo) / (cellulare segnale di controllo solo - segnale di controllo negativo)) x 100%
6 Preparazione del substrato luciferina
- 30 minuti prima della fine dell'incubazione 5 ore, preparare la luciferina per ottenere una soluzione / ml 2 mg sterile tampone fosfato salino (PBS). Preparare 2,5 ml per piastra più un extra di 2 ml. Proteggere la soluzione dalla luce. NOTA: La luciferina può anche essere preparato in precedenza in giornata e conservato a 4 ° C fino al momento dell'uso.
7 Lettura bioluminescenza
- Dopo il periodo di incubazione di 5 ore, aggiungere 25 ml di luciferina in ciascun pozzetto di cellule Vero nei piatti solidi bianchi. Aggiungere luciferina manualmente o con un dosatore automatico integrato nel luminometro. NOTA: L'uso di uno strumento con unico punto singolo e end legge può richiedere l'aggiunta di una luciferasi lisi BUF compatibilefer prima di aggiungere il substrato luciferina di migliorare la coerenza.
- Leggere le piastre con i seguenti parametri:
- Agitare per 5 sec.
- Attendere 30 sec.
- Leggi luminescenza ad un appropriato valore di sensibilità / esposizione fisso. Se l'opzione è disponibile sullo strumento, utilizzare multi-point legge (ad esempio, matrice 3 x 3) per migliorare la precisione.
- Registrare e salvare l'intensità bioluminescente quantificato.
- Se un distributore automatico è stato utilizzato per aggiungere luciferina spurgare la luciferina e adescare le linee con acqua sterile tiepida.
Analisi dei dati 8.
- Per ottenere risultati accurati, risolvere la regressione non lineare per generare un'equazione Hill dalle curve standard. Applicare questa equazione per i campioni titolato al fine di calcolare le unità di espressione virale. Alcuni virus o situazioni possono produrre una curva standard con una relazione lineare; in questo caso per risolvere una equazione lineare.
Risultati
Una sintesi del flusso che descrive il metodo ad alta produttività è illustrato in Figura 1. Figura 2 mostra i risultati di una tipica curva standard ofVSVΔ51-Fluc. Quattro parametro analisi di regressione non lineare generato una trama collina da cui ingresso sconosciuto pfu (stima del titolo virale) possono essere interpolati. Questi titoli stimati sono denominate unità di espressione virali (VEU). Figura 3A mostra VEUS e titoli ottenuti eseguendo un saggio di targa di serie con gli stessi campioni 10. I campioni provenivano da un esperimento in cui sono stati utilizzati vari prodotti chimici per migliorare la replicazione e la diffusione di VSVΔ51-Fluc in 786-0 cellule. VEUS interpolato dalla curva standard deve essere moltiplicato per un fattore che è basato sulla diluizione del campione surnatanti stati trasferiti a cellule Vero (in questo caso, il fattore di diluizione è 5). La correlazione lineare tra titoli e VEU è mostrato in Figura 3B Con un R 2 di 0,8083 e r punteggio di un Pearson di 0,899 (p <0,0001). Nella Figura 4, i dati tipici citotossicità cellulo ottenuti da un test metabolico è indicata per 786-0 cellule trattate con sostanze chimiche prima di infezione da VSVΔ51-Fluc. Infine, la Figura 5 mostra le curve tipiche standard ottenute con vari virus (Herpes Simplex Virus (HSV), il virus vaccinico, e virus adeno-associato (AAV), tutti esprimono luciferasi Firefly) e descrive i tempi di incubazione e cicli di gelo e disgelo, come richiesto.

Figura 1 Flusso di lavoro di high-throughput titolazione virus e test di citotossicità utilizzando VSV Δ 51-Fluc. (A) Seed 2,5 x 10 4 cellule Vero per pozzetto (100 ml) e incubare a 37 ° C. (B) 24hr trasferimento successivamente 25 ml di curva standard su cellule Vero (2 colonne per piastra). (C) Trasferire 25 ml di campioni per essere titolati a rimanenti cellule Vero. Piastre di centrifuga e incubare a 37 ° C. (D) per un importo pari al 10% del volume del bene, aggiungere il reagente resazurina alla piastra contenente i campioni originali. Incubare le piastre a 37 ° C. (E) Dopo 3 ore, leggere e registrare la fluorescenza e valutare la citotossicità. (F) Dopo 5 ore, aggiungere 25 ml di soluzione di 2 mg / ml di luciferina in ogni pozzetto di cellule Vero. Leggi luminescenza e calcolare Unità Espressione virali.
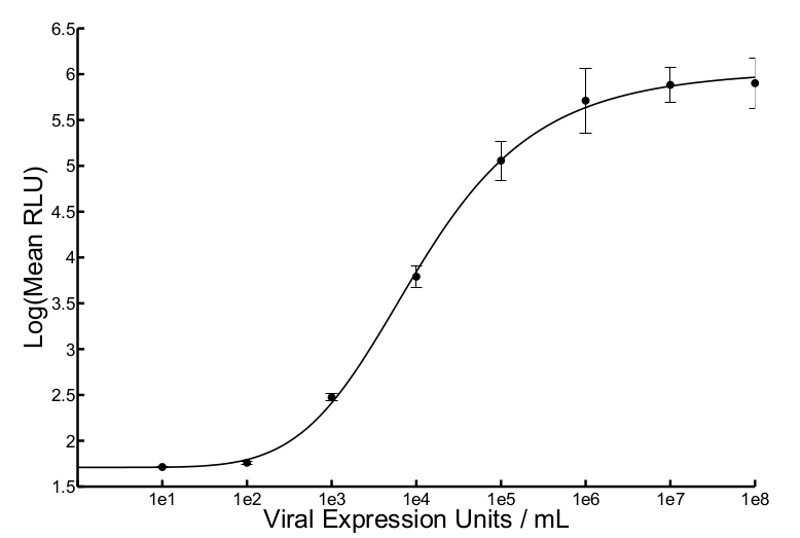
Figura 2 Previsto curva standard di VSV Δ 51-Fluc. Espressione luciferasi me eraasured 5 hr trasferimento, dopo il surnatante in cinque punti diversi all'interno di un pozzo con un luminometro e bioluminescenza è stato espresso in media unità di luce relativa (RLU). RLU media è stata rilevata in entrata noto pfu / ml per risolvere la regressione non lineare e generare una equazione di Hill. La media delle due curve ripetute e barre di errore standard sono riportati (R 2 = 0,9993).

Figura 3 Confronto di titoli test placca standard con quelli ottenuti con il metodo alto throughput. (A) titoli virali in pfu / ml ottenuti dal saggio di placca standard su cellule Vero sono stati confrontati con titoli virali calcolati (VEU / ml) dagli stessi campioni titolati utilizzando il saggio high-throughput luciferasi. (B) relazione lineare tra VEU / ml e titolo ottenuto tramite p di serietest Laque. Sono mostrati curva di regressione lineare e coefficiente di determinazione (R 2).

Figura 4 Esempio di vitalità. Redditività del campione prima del trasferimento surnatante su cellule Vero è stata determinata valutando l'attività metabolica cellulare usando una soluzione resazurina disponibile in commercio. Valori di fluorescenza Raw sono stati normalizzati a quella di non trattati, pozzi non infetti.
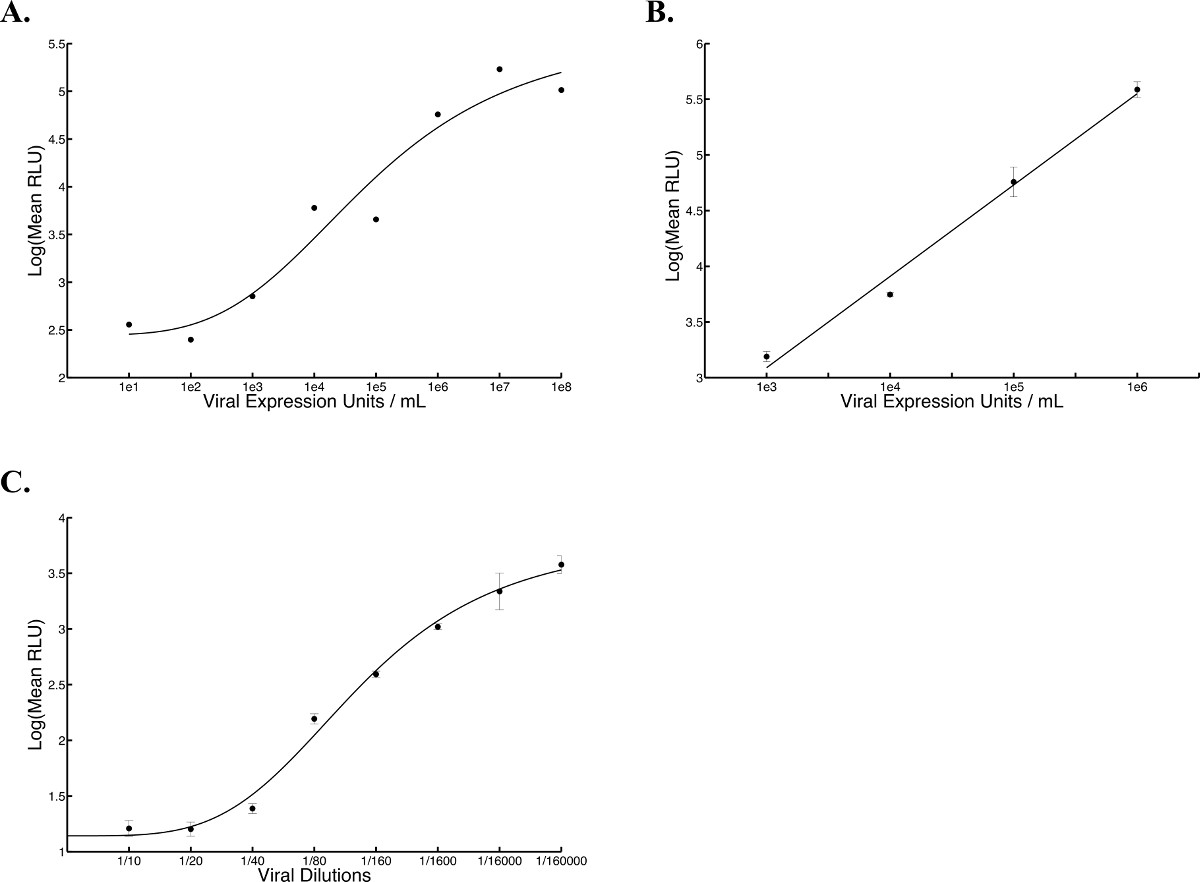
Figura 5 Previsto curve standard con HSV, Vaccinia, e virus AAV. Espressione luciferasi è stata misurata in cinque punti diversi all'interno di un pozzo con un luminometro e bioluminescenza è stato espresso in rela medigruppi ottici tivi (RLU). (A) curva standard HSV è stato aggiunto su cellule Vero placcato 24 ore prima ad una densità di 2,5 x 10 4 cellule per pozzetto (100 ml) e la misurazione della luciferasi è stata fatta 17 ore dopo il trasferimento surnatante (R 2 = 0,9489, n = 1). Un'equazione Hill è stata generata risolvendo la regressione non lineare. Curva standard (B) vaccinico stato aggiunto su cellule Vero placcato 24 h prima una densità di 2,5 x 10 4 cellule per pozzetto (100 microlitri) e incubate per 2,5 ore a 37 ° C, dopo di che sono state prese le misure luciferasi (R 2 = 0,9892). Un'equazione lineare è stata generata risolvendo per la regressione lineare. (C) AAV curva standard è stato aggiunto su cellule di carcinoma polmonare umano (A549) placcato 24 ore prima ad una densità di 2,5 x 10 4 cellule per pozzetto (100 microlitri) e la misurazione luciferasi è stata fatta 24 ore dopo l'infezione. Un'equazione Hill è stata generata risolvendo la regressione non lineare. La mediadi sono mostrati cinque curve ripetute e barre di errore standard (R 2 = 0,9926).
Discussione
L'approccio basato luciferasi-qui descritta fornisce una serie di vantaggi rispetto ad altri metodi esistenti, compresa la sua facilità, rapidità, minima necessità di attrezzature, e relativamente a basso costo. Un contributo fondamentale a questo è quello di evitare una fase di diluizione seriale. Tuttavia, i derivati di serie a base di diluizione, di questo protocollo sono certamente fattibile e sono stati recentemente utilizzati per valutare-luciferasi che esprimono Ebola titoli in uno schermo antivirale high-throughput 11. Mentre intrinsecamente più tempo e più costoso, tali adattamenti possono fornire una maggiore gamma dinamica per la quantificazione virale quando necessario. Oltre ad essere particolarmente adatto per la valutazione titoli virali nel contesto degli schermi high-throughput, il nostro metodo di quantificazione virale base di uno stadio luciferasi genera stime accurate di virioni infettivi nel caso di replicare virus. Inoltre, letture di luminescenza insieme ai dati di citotossicità dello stesso esperimento danno una piùquadro completo l'effetto delle condizioni sperimentali su cellule bersaglio, che è particolarmente utile nel contesto di virus oncolitici e schermi di droga.
L'esempio qui illustrato utilizza un replicante virus RNA a singolo filamento negativo; Tuttavia, questo protocollo può essere adattato ad un numero di replicare e non replicanti virus con pochi adattamenti protocollo minori. Questo include virus a DNA come vaccinico, HSV, e AAV (vedi Figura 5). I campioni infettati da virus intracellulari, come il virus vaccinico, ad esempio, richiedono un passo virus-release prima di quantificazione (ad esempio, almeno un ciclo gelo-disgelo). Quando si applica questa tecnica ad altri virus, è necessario ottimizzare il tempo di incubazione dal trasferimento del surnatante virale o lisato alla lettura delle piastre dal luminometro. Questo parametro dipende principalmente dal ciclo di replicazione del virus nella linea cellulare permissiva e la forza del promoter guida espressione luciferasi. Questo è fatto meglio usando la curva standard completa nella fase di ottimizzazione. Per fare questo, si deve infettare la linea cellulare permissiva appropriata con varie repliche della curva standard preparata e leggere ogni replica in un momento diverso punti post-trasferimento. Idealmente, un punto di tempo di incubazione è scelto che conduce ad una relazione lineare tra LOG (RLU) e LOG (titolo) che attraversa il campionario titolo atteso. Per VSVΔ51-Fluc, questo è in genere da 10 a 4 pfu / ml -10 7 pfu / ml per un tempo di incubazione 5 ore. Se titoli inferiori o superiori sono attesi da campioni, si può semplicemente aumentare o ridurre il tempo di incubazione, rispettivamente. In alternativa, i campioni possono essere diluiti per rientrare nel campo di quanto avviene tipicamente per ELISA.
Come accennato in precedenza, questo metodo è adatto per eseguire droga schermi high-throughput utilizzando librerie di droga, come la maggior parte delle operazioni possono essere automatizzate. Le cellule possono essere placcati efficiently utilizzando un dispensatore di micropiastre automatico, la biblioteca farmaco può essere aggiunto utilizzando un gestore di liquido a 96 canali, virus può essere aggiunto utilizzando un dispenser di micropiastre e piastre leggere utilizzando un luminometro automatico. In teoria, questo può anche essere adattato a 384 pozzetti o formati più piccoli; tuttavia, la limitazione a tal fine è il numero di cellule che possono essere placcato, dato minor numero di cellule porta ad una gamma più ristretta nella linearità del LOG (RLU) a LOG (Titer) rapporto. Infine, la valutazione della vitalità cellulare mediante resazurina o altri coloranti metabolici può essere facilmente integrato nel flusso di lavoro, consentendo la discriminazione di composti citotossici in schermi antivirali o identificazione di composti che portano alla uccisione sinergica in combinazione con virus 12. Tuttavia, le limitazioni di questo metodo includono il requisito di un virus transgene che esprime la luciferasi, che non è sempre possibile, e la disponibilità di una linea cellulare permissiva sufficientemente. Tuttavia, è probabile possibile adattareil metodo usato con altri geni reporter (per esempio, GFP), se il metodo di quantificazione giornalista ha una linearità adatto e rapporto segnale rumore. In generale, il metodo high-throughput descritto può essere modificato per soddisfare molti virus diversi e su misura per diverse applicazioni.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Vanessa Garcia is funded by a Queen Elizabeth II Ontario Graduate Scholarship in Science and Technology and Cory Batenchuk by a Natural Sciences and Engineering Research Council fellowship.
Materiali
| Name | Company | Catalog Number | Comments |
| Dulbeccos' Modified Eagle's Medium (DMEM) | Corning | SH30243.01 | |
| Fetal bovine serum | NorthBio Inc. | NBSF-701 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| HEPES | Fisher Scientific | BP310-1 | Prepare a 1 mM solution, pH 7.3 |
| alamarBlue | AbD Serotec | BUF012B | |
| D-Luciferin potassium salt | Biotium | 10101-2 | |
| 96-well solid white flat bottom polystyrene TC-treated microplates | Corning | 3917 | 384-well plates can also be used for higher throughput |
| Synergy Mx | BioTek | SMTBL | Monochromator microplate reader |
| Liquidator96 | Mettler Toledo | LIQ-96-200 | 96 tip manual pipetting system |
| Liquidator96 LTS Tips sterilized with filters | Mettler Toledo | LQR-200F | Any sterile filtered tips compatible with pipettors of choice are appropriate |
| Microflo | BioTek | 111-206-21 | Used to plate cells in a 96-well plate |
| Fluoroskan Ascent FL | Thermo Scientific | 5210450 | Microplate fluorometer |
Riferimenti
- Grigorov, B., Rabilloud, J., Lawrence, P., Gerlier, D. Rapid titration of measles and other viruses: optimization with determination of replication cycle length. PLoS One. 6 (e24135), (2011).
- Lizee, G., et al. Real-time quantitative reverse transcriptase-polymerase chain reaction as a method for determining lentiviral vector titers and measuring transgene expression. Hum Gene Ther. 14, 497-507 (2003).
- McSharry, J. J. Uses of flow cytometry in virology. Clin Microbiol Rev. 7, 576-604 (1994).
- Watcharatanyatip, K., et al. Multispecies detection of antibodies to influenza A viruses by a double-antigen sandwich ELISA. J Virol Methods. 163, 238-243 (2010).
- Dawson, E. Rapid, Direct Quantification of Viruses in Solution Using the ViroCyt Virus Counter. Journal of Biomolecular Techniques. 23 (S10), (2012).
- Snyder, R. O. AAV and RT-PCR: true or false. Mol Ther. 1, 389-390 (2000).
- Diallo, J. S., Roy, D., Abdelbary, H., De Silva, N., Bell, J. C. Ex vivo infection of live tissue with oncolytic viruses. J Vis Exp. 52, (2011).
- Gaush, C. R., Smith, T. F. Replication and plaque assay of influenza virus in an established line of canine kidney cells. Appl Microbiol. 16, 588-594 (1968).
- Green, M., Loewenstein, P. M. Human adenoviruses: propagation, purification, quantification, and storage. Curr Protoc Microbiol. 14 (14C 11), (2006).
- Diallo, J. S., Vaha-Koskela, M., Le Boeuf, F., Bell, J. Propagation, purification, and in vivo testing of oncolytic vesicular stomatitis virus strains. Methods Mol Biol. 797, 127-140 (2012).
- Hoenen, T., Groseth, A., Callison, J., Takada, A., Feldmann, H. A novel Ebola virus expressing luciferase allows for rapid and quantitative testing of antivirals. Antiviral Res. 99, 207-213 (2013).
- Diallo, J. S., et al. A high-throughput pharmacoviral approach identifies novel oncolytic virus sensitizers. Mol Ther. 18, 1123-1129 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon