É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medição de alto rendimento da membrana plasmática, efectuou a eficiência em células de mamíferos
Neste Artigo
Resumo
Aqui nós descrevemos um alta produtividade baseada em fluorescência do ensaio que mede a membrana plasmática, efectuou a eficiência por meio de análises fluorométrica e da imagem latente em células vivas. Este ensaio pode ser usado para triagem de drogas ou genes-alvo que regulam a membrana plasmática de fechamento em células de mamíferos.
Resumo
Em seu ambiente fisiológico, células de mamíferos são frequentemente sujeitas a tensões mecânicas e bioquímicas que resultam em danos de membrana plasmática. Em resposta a esses danos, máquinas moleculares complexas rapidamente selar novamente a membrana plasmática para restaurar a sua função de barreira e manter a sobrevivência da pilha. Apesar de 60 anos de investigação neste domínio, ainda falta uma compreensão completa da célula máquinas de fechamento. Com o objetivo de identificar componentes celulares que controle de fechamento de membrana plasmática ou drogas que podem melhorar a recelagem, nós desenvolvemos um ensaio de alto rendimento baseada em fluorescência que mede a membrana plasmática recelagem eficiência em células de mamíferos cultivadas em microplacas. Como um sistema modelo para danos de membrana plasmática, as células estão expostas à bactérias formadoras de poros toxina listeriolisina O (LLO), que forma grandes 30-50 nm de diâmetro proteicas poros em colesterol, contendo membranas. O uso de um leitor de microplacas de modo multi temperatura controlada permite medições de spectrofluorometric rápida e sensível, em combinação com brightfield e imagem de microscopia de fluorescência das células vivas. Análise cinética da intensidade da fluorescência emitida pelo fluorocromo impermeant de ácido nucleico-ligação membrana reflete a extensão da membrana, ferindo e efectuou no nível da população de célula, permitindo o cálculo da célula eficiência de fechamento . Imagem de microscopia de fluorescência permite a enumeração de células, que constitutivamente expressa uma quimera fluorescente de 2B de histona a proteína nuclear, em cada poço do microplate em conta possíveis variações em seu número e permite a eventual identificação de populações de células distintas. Este ensaio de alto rendimento é uma ferramenta poderosa, espera-se expandir a nossa compreensão dos mecanismos de reparação de membrana através de triagem para genes do hospedeiro ou exogenamente adicionados compostos que efectuou de membrana plasmática de controle.
Introdução
Células de mamíferos estão sujeitos a estresse mecânico, osmótico e bioquímico, resultando na perda da integridade da membrana plasmática. Sem rápida e eficiente de fechamento, células danificadas rapidamente iria sucumbir à morte programada ou necrótica. Desde a década de 1960, os esforços para compreender a membrana de plasma nesse processo tem sido motivados pelas consequências devastadoras associadas com suas disfunções. Na verdade, doenças como Distrofia Muscular de membro-cinta, diabetes e síndrome de Chediak-Higashi têm sido associadas a reparação da membrana de plasma deficiente devido a mutações no gene dysferlin codificação, produção de produtos finais de glicação avançada e defeitos em o regulador de tráfico dos lisossomos CHS1, respectivamente,1,2,3,4,5,6. No entanto, até à data, nossa compreensão da membrana de fechamento é ainda limitado7. Os estudos iniciais demonstraram que a membrana recelagem é iniciada pelo influxo de extracelular Ca2 + através da membrana de plasma danificado8,9,10. Desde então, vários não mutuamente exclusivas Ca2 +-dependentes mecanismos têm sido propostos para selar as células. A hipótese de remendo propõe que no próximo a ferida, intracelulares vesículas fundem-se com os outros e a membrana plasmática danificada para atuar como um patch11,12,13,14. Um segundo modelo propõe que exocitose de cálcio-dependente de lisossomos para a ferida local libera a enzima lisossomal sphingomyelinase ácido, que converte esfingomielina, a ceramida no folheto externo da membrana plasmática. Esta súbita mudança na composição de lipídios resulta em endocitose ceramida-conduzido a região danificada15,16,17. Por último, o terceiro mecanismo proposto envolve um papel para o CDDP classificação complexa necessária para o transporte (ESCRT) promover a formação de vesículas virados que brote fora da membrana plasmática18. Somente um conjunto limitado de proteínas foi identificado nestes modelos, e suas máquinas devem ser ainda mais elucidada.
Aqui descrevemos um ensaio de alto rendimento que medidas a membrana plasmática recelagem eficiência em células de mamíferos aderentes sujeitos a danos mediadas por recombinação listeriolisina O (LLO)19. LLO é uma toxina formadoras de poros (PFT) secretada pelo patógeno intracelular facultativo Listeria monocytogenes20,21,22 e pertence a MACPF/CDC (complexo de ataque de membrana, perforina, e Superfamília Citolisina dependente de colesterol). MACPF são mamíferos poro-formando proteínas envolvidas em defesas imunitárias, Considerando que os conjuntos são toxinas bacterianas principalmente produziram por patógenos gram-positivos que danificam as células do hospedeiro para promover sua patogenicidade, estilos de vida23. Conjuntos são sintetizados como monômeros Water-soluble ou dímeros que se ligam ao colesterol presente na membrana plasmática e oligomerize em um complexo prepore de até 50 subunidades. O complexo de prepore então se reorganiza para inserir β-vertentes através da bicamada lipídica, formando um poro do β-tambor que se estende por 30-50 nm de diâmetro24,25,26,27. Estes poros dilatados permitam fluxos de íons e pequenos componentes celulares dentro e fora da célula; no entanto, alguns estudos têm proposto que os poros de tamanhos menores também são formados28,29,30. Entre os conjuntos, LLO exibe propriedades exclusivas, incluindo agregação irreversível e temperatura-dependente do pH, que é propício para análises de alta produtividade31,32. LLO pode ser adicionado ao meio de cultura de células no 4 ˚ c, uma temperatura permissiva a sua ligação às células, mas não para a formação do complexo do poro. Iniciação de formação de poros então pode ser sincronizada, elevando a temperatura a 37 ˚ c, permitindo que pela difusão de moléculas de toxina no plano da membrana oligómeros de forma eficiente e para a remodelação conformacional envolvidos na geração de poros. Portanto, seguir o interruptor de temperatura, a cinética de danos celulares dependerá da quantidade de toxina ligada a membrana plasmática. Importante, LLO solúvel (não ligado a membrana plasmática) rápida e irreversivelmente agrega quando a temperatura atinge 37 ˚ c, que alivia a necessidade de lavar as moléculas da toxina não acoplado e limita a extensão dos danos de membrana ao longo do tempo. Por último, porque LLO vincula-se ao colesterol e forma poros nas membranas celulares ricos em colesterol, este ensaio é passível de uma ampla variedade de células de mamíferos. É importante ter em mente que LLO afeta a célula hospedeira sinalização principalmente através de formação de poros, com poucas exceções em qual célula independente de poro sinalização pode ocorrer33,34,35,36 ,37,38,39. Portanto, não pode ser excluído que LLO sinalização atividades podem influenciar o processo de reparação da membrana.
Este ensaio avalia diretamente a extensão da célula ferindo medindo-se a incorporação de uma célula impermeant fluorocromo (por exemplo, iodeto de propidium) que passivamente entra as células feridas e torna-se altamente fluorescente, uma vez que associa os ácidos nucleicos . Daí, o fluorocromo pode ser mantido em meio de cultura celular durante todo o experimento, permitindo análises em tempo real da célula ferindo. A intensidade da fluorescência do corante ácido nucleico-ligação aumentará a concentração de toxina e, para uma dada concentração de toxina, irá aumentar ao longo do tempo até que todos os poros são formados, e as células são totalmente reparadas ou até saturação é alcançada. O afluxo de extracelular Ca2 + através da membrana poros é um evento de condição sine qua non para selar. Portanto, a eficiência de fechamento pode ser evidenciada indiretamente comparando a célula ferindo em meio de cultura contendo Ca2 + (condição permissiva do reparo) para ferir em um Ca2 +-livre médio (condição restritiva de reparação). Porque a intensidade da fluorescência do corante ácido nucleico-vinculação é diretamente proporcional à concentração de células em cada poço, é importante para as células de semente na mesma concentração em todos os poços. Também é importante enumerar as células em cada poço antes e após o ensaio para garantir que desprendimento celular não ocorre, como flutuante, agregados de células podem obscurecer as leituras de fluorescência que podem complicar a interpretação dos dados. Para enumerar as células, as células expressando nuclear-localizada histona 2B-GFP (H2B-GFP) foram utilizadas neste ensaio. Temperatura controlada, multi-modo, leitores de microplacas combinam rápidas medições, de alto rendimento (usando um formato de placa de 96 ou 384 poços) das intensidades de fluorescência com imagem de microscopia de células vivas, a 37 ° C. Este último pode ser usado para enumerar o número de telemóvel e observar a eventual formação de populações de células distintas.
Em última análise, este ensaio fornece aos usuários a capacidade de expandir seus conhecimentos sobre a complexidade dos mecanismos de reparação da membrana por triagem para moléculas do anfitrião ou reparar de forma exógena adicionados compostos que podem controlar a membrana. O protocolo a seguir descreve as etapas experimentais para medir a eficiência de fechamento de células expostas a LLO e avaliar os efeitos de uma determinada droga ou tratamento celular na eficiência de fechamento.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. preparação
- Chapeamento de célula
Nota: Células epiteliais do colo do útero humanas, HeLa e HeLa expressando histona 2B-GFP (H2B-GFP), foram utilizadas neste protocolo, mas este ensaio pode ser adaptado a outras células de mamíferos,19.- Separe células aderentes de um frasco de cultura de células de2 75cm lavando as células com 2 mL de EDTA-tripsina 0,25%. Substitua a tripsina usada com 2 mL de fresco do trypsin-EDTA 0,25%.
- Incube as celulas em 37 ˚ c por 5 min até as células arredondadas e retirado o balão.
- Ressuspender as células em 8 mL de meio de cultura (DMEM contendo 10% inactivadas pelo calor fetal de soro bovino, 100 U/mL penicilina e estreptomicina 100 de µ g/mL).
- Determine a concentração de células usando um hemocytometer e 10 µ l de suspensão de células.
- Dilua as células em um meio para uma concentração de 2,5 x 105 células/mL.
- Despeje a suspensão de células em uma bacia de pipeta estéril e misturar cuidadosamente a suspensão usando uma pipeta sorológica 10ml.
- Utilizando uma micropipeta multicanal-12 e 200 µ l dicas, distribua células HeLa (2,5 x 104 células/100 µ l/poço) em triplicado (ou quatro exemplares) em uma placa de cultura de tecidos tratados poliestireno inferior plana de 96 poços, claro, preto.
Nota: Um arranjo de chapeamento é apresentado como um exemplo na Figura 1. - Cultura de células para 24 h em uma incubadora de cultura celular umidificado no 37 ˚ c e 5% CO2.
- Preparação da solução-mãe
- Preparar 1 L de um estoque de 10 x de buffer M (usado para preparar a M1 e M2) pela adição de 95 g de Hanks equilibrada solução salina, 0,476 g de MgCl2 (5 mM) e 23,83 g de HEPES (100 mM) de 900 mL de água. Ajustar o pH para 7,4 e aumentar o volume de 1 L. Filter esterilizar.
- Prepare-se 50 mL de um 50 x (1.25 M) estoque de glicose, adicionando 11,26 g de D-(+)-glicose para um total de 50 mL de água. Filtro de esterilizar a solução.
- Prepare-se 50 mL de um 100 x estoque (120 mM) de cálcio, adicionando 0,666 g de CaCl2 para um total de 50 mL de água. Filtro de esterilizar a solução.
- Preparar 50 mL de um 10 x estoque (50 mM) de etileno glicol-bis(2-aminoethylether)-N, N, N', N', ácido etilenodiaminotetracético (EGTA) adicionando 0,951 g de EGTA a 40 mL de água. Aumentar o pH a 8 usando NaOH para dissolver o EGTA e, em seguida, aumentar o volume de 50 mL. Filtro de esterilizar a solução.
- Para uma única placa de 96 poços, preparar 50 mL de meio 1 (M1, contém Ca2 +), 50 mL de meio 2 (M2, Ca2 +-livre) e 15 mL de 2 meio suplementado com EGTA, consequentemente:
- Para M1, adicione 5 mL de 10x Buffer M, 0,5 mL de 100 x CaCl2e 1 mL de glicose de x 50 a 43,5 mL de água.
- Para M2, adicione 5 mL de 10x Buffer M e 1 mL de glicose 50 x a 44 mL de água.
- Para M2/EGTA, adicionar 1,5 mL de 10 x Buffer M e 1,5 mL de 10 x EGTA a 12 mL de água.
Nota: Todas as soluções contendo iodeto de propidium (PI) devem ser preparadas diretamente antes da adição para as células.
- Placa leitor/imagens citômetro configurações
Nota: Use um leitor de placa multi-modo, equipado com duas unidades de detecção: um spectrofluorometer e um citômetro de imagem. Limite a exposição de fluorescência para evitar fotobranqueamento o fluorophores.- Pré-aquecer o leitor a 37 ° C antes de realizar o ensaio.
- Configurar os parâmetros para o ensaio cinético adequadamente dentro do modo de configurações :
- Escolher o monocromador, FL (fluorescência)e cinética para a configuração óptica, modos de ler e ler tipo, respectivamente.
- Em Configurações de comprimento de onda, selecione um 9 e 15 nm excitação e emissão bandpass, respectivamente. Para ensaios usando iodeto de propidium (PI), definir os comprimentos de onda de excitação e emissão de 535 e 617 nm, respectivamente.
- Em Tipo de placa, selecione 96 poços para o formato da placa e uma configuração de placa de pre-ajuste correspondente para um prato fundo transparente preto-parede.
- Sob a Área de leitura, destaca os poços que serão analisados em toda a cinética.
- Sob a óptica e PGTO, preset os flashes por leitura de 6 e a caixa de seleção para a leitura da parte inferior.
- Sob o Timing, insira 00:30:00 na caixa do Tempo Total de execução de um ensaio cinético de 30 min e insira 00:05:00 para o intervalo.
Nota: Para cada vez que aponte e um comprimento de onda, o tempo de leitura de uma placa de 96 poços completo é de 30 s. - Confirme as configurações especificadas nas Informações de configurações para a direita e selecione Okey. Imprensa leitura para iniciar a cinética executar.
- Configurar os parâmetros de imagem adequadamente dentro do modo de configurações:
- Escolher o Minimax, Imaginge ponto de extremidade para a configuração óptica, modos de ler e ler tipo, respectivamente.
- Em comprimentos de onda, selecione transmitida a luze uma ou ambas as caixas de fluorescência correspondente a excitação e emissão de comprimentos de onda de 456/541 nm (GFP) e 713/625 nm (PI).
- Use as mesmas opções para o Tipo de placa e Área de leitura , conforme definido nas etapas 1.3.2.3 e 1.3.2.4.
- Em Bem a área de configuração, selecione o número de sites dentro de um poço para ser fotografada.
Nota: 12 locais correspondem a uma imagem bem cheia. - Sob as Configurações de aquisição de imagem, selecione os tempos de exposição para luz transmitida, 541 (GFP) e 713 (PI). Para as boas práticas agrícolas, todo o bem com um tempo de exposição de 20 ms/imagem de imagem. Para transmitir luz (TL) e fluorescência de PI, adquirir uma única imagem do centro de cada poço com tempos de exposição de 8 e 20 ms, respectivamente.
- Confirme as configurações especificadas nas informações de configurações para a direita e selecione Okey. O tempo de aquisição de imagem de toda a superfície de cada poço (12 imagens/poço) de uma placa de 96 poços e para um comprimento de onda é ~ 15min. imprensa leitura para iniciar a geração de imagens.
Nota: O tempo de aquisição de um único imagem/bem de uma placa de 96 poços requer ~2.5 min/placa para um comprimento de onda. Os parâmetros descritos acima correspondem ao equipamento específico em nosso laboratório. Spectrofluorometric medições: uma lâmpada de flash de xénon exibindo 1,0 nm incremento da excitação comprimentos de onda (250-850 nm) com um ajustável 9 ou 15 nm passa-banda, um detector de tubo fotomultiplicador com um 6 > log gama dinâmica e uma emissão de nm 15 ou 25 ajustável bandpass. Citômetro de imagem: uma fonte de luz iluminação capaz de luz branca, 460 nm e 625 nm excitação comprimentos de onda com um 20 nm bandpass, filtros de emissão centrados em 541 nm (108 nm bandpass) e 713 nm (123 nm bandpass), respectivamente, e um objectivo X 4 acoplado a um 1.25 megapixel câmera de 12 bits dispositivo de carga acoplada.
2. ensaio
Nota: Na época do ensaio, as células devem ser confluente de 70-90%. Durante as etapas de lavagem, o meio deve ser retirado e aplicado para a parede lateral do poço (não diretamente acima das células). Manter a temperatura de LLO no < 4 ˚ c para evitar a sua agregação até passo 3.1.5.
- Preparar um estoque de 30 µM PI em M1 e um estoque de 30 µM PI em M2 pré aquecido a 37 ˚ c.
- Lave delicadamente as células na placa 1 uso uma micropipeta multicanal-12 e 200 µ l conselhos, como segue:
- Para condições de reparação-permissivo, remova o meio de crescimento e lavagem que das células duas vezes com 200 µ l/poço M1 pré aquecidas a 37 ˚ c. Substitua o medium com 100 µ l/poço de M1 quente contendo 30 µM PI.
- Para reparar a condições restritivas, remover o meio de crescimento e lavar as células uma vez com 200 µ l/poço quente M2 contendo 5 mM EGTA de quelato Ca2 +, seguido por uma lavagem com 200 µ l/poço M2. Substitua o medium com 100 µ l/poço quente M2 contendo 30 µM PI.
- Depois que o meio de crescimento foi lavado e substituído com meio contendo iodeto de propidium, directamente mova para passo 2.1.3.
- Placa 1 sob luz transmitida, GFP e PI como detalhado sob 1.3.3 (pré-cinético). Esta etapa leva 15-20 min.
- Durante o período de 15 min na etapa 2.1.3, prepare o prato 2 usando uma micropipeta multicanal-12 e 200 µ l dicas da seguinte maneira:
- Coloque uma microplaca de polipropileno de fundo redondo de 96 poços no gelo. Configure a placa usando um delineamento experimental correspondente para placa 1 (Figura 1).
- Para condições de reparação-permissivo, adicionar 100 µ l/poço de M1 gelada contendo 60 µM PI, seguido pela adição de 100 µ l/poço de M1 gelada contendo 4 x LLO ou não para o controle.
- Para condições restritivas para reparação, adicionar 100 µ l/poço de gelada M2 contendo 60 µM PI, seguido pela adição de 100 µ l/poço de gelada M2 contendo 4 x LLO ou não para o controle.
- Imagem placa 1 (passo 2.1.3), imediatamente depois de colocá-lo no gelo, usando papel alumínio para separar a placa de contato direto com gelo. Permitir que a placa 1 arrefecer por 5 min.
- Utilizando uma micropipeta multicanal-12 e 200 µ l dicas, transferi 100 µ l de cada poço para a placa 2 (passo 2.1.4) aos poços correspondentes na placa 1. Para distribuir adequadamente a toxina na mídia da chapa 1, insira as dicas abaixo o menisco e ejetar suavemente o volume sem introduzir bolhas.
Nota: Não pipete acima e para baixo, como isso pode, inadvertidamente, separar as células. - Deixe o prato para um 1 min adicional permitir que a toxina a ligar para as células e transferir imediatamente chapa 1 para o leitor para o ensaio cinético usando o modo de spectrofluorometer (passo 1.3.2).
- No final do ensaio cinético, imediatamente imagem placa 1 (pós-cinético) usando passo 1.3.3.
3. análise: Célula enumeração
- Determine a contagem de células baseada em fluorescência nuclear usando o software de enumeração de célula de microplacas.
- Dentro de configurações, selecione Re-análisee sob a seção categoria dentro as Configurações de análise de imagem , selecione Discreto objeto análise usando 541 como o comprimento de onda para encontrar objetos.
- Dentro da opção de encontrar objetos, usando a desenhar nas imagens encontrar método, selecione núcleos sob a guia de configurações e pressione aplicar.
- Pressione Okey e leitura para iniciar a célula algoritmo de contagem.
- Alternativamente, se tal ferramenta não estiver disponível, use um software de análise de imagem tais como ImageJ para enumerar as células.
- No ImageJ, abra o arquivo de imagem como uma pilha.
- Converter imagens em tons de cinza de 8 bits clicando em imagem na barra de menu, passe o mouse sobre o tipo, a pilha e selecione 8-bit.
- Subtrair o plano de fundo: clique na imagem na barra de menus, focalize em ajustee selecione Brightness/Contrast. Ajuste o valor mínimo para remover o ruído de fundo e selecione Apply.
- Limiar para criar imagens binárias: clique na imagem na barra de menus, focalize em ajustee selecione o limiar. Selecione o fundo escuro, ajustar os valores de limiar mínimo e máximo e clique em aplicar.
- Em caso de cumulação de núcleos, uma ferramenta de bacia hidrográfica pode ser usado para núcleos do segmento. Clique em processo no menu, passe o mouse sobre o binário e selecione o divisor de águas.
Nota: Isto separará automaticamente núcleos conectados. - Analise as imagens mascaradas aplicando critérios especificados pelo usuário (tamanho e circularidade) para aperfeiçoar a identificação dos núcleos e excluir os restos celulares.
- Clique em Analyze no menu e em seguida analisar partículas. Definir o tamanho desejado (pixel ^ 2) e circularidade (um valor de 1 é um círculo perfeito) intervalos que são suficientes para incluir células/núcleos individuais.
- Na caixa de lista suspensa mostrar, selecione as opções desejadas, verificar resumire clique em Okey para obter a contagem de células.
4. análise: Curvas de cinéticas
- Transferi dados cinéticos a partir do software de leitor de placa para um software de dados analíticos.
- Para cada condição experimental, média as intensidades de fluorescência das repetições em cada commit, juntamente com o correspondente desvio padrão e erro padrão da média para cada condição experimental.
- Para cada condição experimental, traçar a curva correspondente-cinética: intensidade de PI (eixo y) versus tempo (eixo x).
- Para calcular a eficiência de fechamento de uma condição de determinado tratamento, calcular a área sob a curva (AUC) da + LLO em M1 (AUC(M1)) e + LLO em M2 (AUC(M2)). Use a abordagem sugerida abaixo para avaliar a eficiência (E) efectuou:
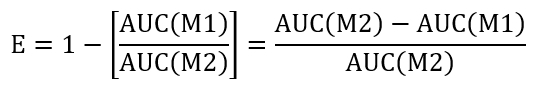
- Realize uma comparação entre o tratamento de controle e teste, determinando-se o rácio de eficiência (RFEP) indicado abaixo:
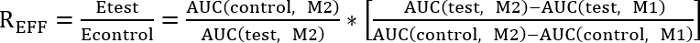
REFF = 1, tratamento de teste não tem efeito sobre reparação
RFEP < 1, teste tratamento inibe reparação
RFEP > 1, teste tratamento melhora a reparação - Calcule a área sob a curva utilizando a seguinte equação:
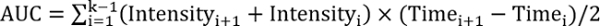 , onde k é o número total de follow-ups.
, onde k é o número total de follow-ups.
Access restricted. Please log in or start a trial to view this content.
Resultados
Célula, contando com precisão: as células HeLa são usadas frequentemente como uma linha de células de mamíferos do modelo para explorar mecanismos de reparação da membrana. Ao avaliar a reparação da membrana no nível da população de célula, é importante para as células da placa na mesma concentração em todos os poços para interpretação de dados apropriados. Também é importante verificar no momento do ensaio que celulares sejam equivalentes em poços. Células HeLa q...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este ensaio mede a eficiência da membrana efectuou a nível de população de células com capacidade de alta produtividade. Pode ser usada para triagem de bibliotecas de drogas que poderiam afetar a reparação da membrana ou componentes celulares. O ensaio descrito usado um formato de placa de 96 poços, mas pode ser adaptado para placas boas 384 para maior rendimento. Uma vantagem deste teste é sua capacidade de obter medidas de fluorescência das células vivas aderentes em tempo real sem a necessidade de célula e...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Reconhecemos o Dr. Jesse Kwiek (The Ohio State University) por gentilmente nos terem permitido usar sua plataforma de detecção modo multi para alguns experimentos preliminares. Pesquisa relatada neste artigo foi apoiada pelo Instituto Nacional de alergia e doenças infecciosas dos institutos nacionais de saúde, sob número de prêmio RO1AI107250 para Stephanie Seveau. O conteúdo é exclusivamente da responsabilidade dos autores e não representa necessariamente a opinião oficial do institutos nacionais da saúde.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| SpectraMax i3x Multi-Mode Microplate Reader | Molecular Devices | i3x | |
| MiniMax 300 Imaging cytometer | Molecular Devices | 5024062 | |
| TO-PRO-3 | ThermoFisher Scientific | T3605 | |
| Propidium Iodide | ThermoFisher Scientific | P3566 | |
| HeLa | ATCC | CCL2 | |
| HeLa H2B-GFP | Millipore | SCC117 | |
| Trypsin-EDTA 0.25% | ThermoFisher Scientific | 25200056 | |
| 96-well Corning flat bottom black polystyrene tissue culture treated plate | Corning | 3603 | |
| Hanks' balanced Salts | Sigma-Aldrich | H4891 | |
| EGTA | ISC BioExpress | 0732-100G | |
| HEPES | Fisher Scientific | BP310-500 | |
| D-(+)-Glucose, HybriMax | Sigma-Aldrich | G5146-1KG |
Referências
- Demonbreun, A. R., McNally, E. M. Plasma Membrane Repair in Health and Disease. Current Topics in Membranes. 77, 67-96 (2016).
- Howard, A. C., McNeil, A. K., McNeil, P. L. Promotion of plasma membrane repair by vitamin E. Nature Communications. 2, 597(2011).
- Howard, A. C., et al. A novel cellular defect in diabetes: membrane repair failure. Diabetes. 60 (11), 3034-3043 (2011).
- Lozano, M. L., et al. Towards the targeted management of Chediak-Higashi syndrome. Orphanet Journal of Rare Diseases. 9, 132(2014).
- Vainzof, M., et al. Dysferlin protein analysis in limb-girdle muscular dystrophies. Journal of Molecular Neuroscience. 17 (1), 71-80 (2001).
- Huynh, C., et al. Defective lysosomal exocytosis and plasma membrane repair in Chediak-Higashi/beige cells. Proceeding of the National Academy of Sciences of the United States of America. 101 (48), 16795-16800 (2004).
- Cooper, S. T., McNeil, P. L. Membrane Repair: Mechanisms and Pathophysiology. Physiological Reviews. 95 (4), 1205-1240 (2015).
- Steinhardt, R. A., Bi, G., Alderton, J. M. J. M. Cell membrane resealing by a vesicular mechanism similar to neurotransmitter release. Science. 263 (5145), 390-393 (1994).
- De Mello, W. C. Membrane sealing in frog skeletal-muscle fibers. Proceedings of the National Academy of Sciences of the United States of America. 70 (4), 982-984 (1973).
- Fishman, H. M., Tewari, K. P., Stein, P. G. Injury-induced vesiculation and membrane redistribution in squid giant axon. Biochimica et Biophysica Acta. 1023 (3), 421-435 (1990).
- Davenport, N. R., Bement, W. M. Cell repair: Revisiting the patch hypothesis. Communicative & Integrative Biology. 9 (6), 1253643(2016).
- McNeil, P. L., et al. Patching plasma membrane disruptions with cytoplasmic membrane. Journal of Cell Science. 113 (11), 1891-1902 (2000).
- Terasaki, M., Miyake, K., McNeil, P. L. Large plasma membrane disruptions are rapidly resealed by Ca2+-dependent vesicle-vesicle fusion events. Journal of Cell Biology. 139 (1), 63-74 (1997).
- Bi, G. Q., Alderton, J. M., Steinhardt, R. A. Calcium-regulated exocytosis is required for cell membrane resealing. Journal of Cell Biology. 131 (6 Pt. 2), 1747-1758 (1995).
- Tam, C., et al. Exocytosis of acid sphingomyelinase by wounded cells promotes endocytosis and plasma membrane repair. Journal of Cell Biology. 189 (6), 1027-1038 (2010).
- Rodriguez, A., et al. Lysosomes behave as Ca2+-regulated exocytic vesicles in fibroblasts and epithelial cells. Journal of Cell Biology. 137 (1), 93-104 (1997).
- Reddy, A., Caler, E. V., Andrews, N. W. Plasma membrane repair is mediated by Ca(2+)-regulated exocytosis of lysosomes. Cell. 106 (2), 157-169 (2001).
- Jimenez, A. J., et al. ESCRT machinery is required for plasma membrane repair. Science. 343 (6174), 1247136(2014).
- Pathak-Sharma, S., et al. High-Throughput Microplate-Based Assay to Monitor Plasma Membrane Wounding and Repair. Frontiers in Cellular and Infection Microbiology. 7, 305(2017).
- Hamon, M. A., et al. Listeriolysin O: the Swiss army knife of Listeria. Trends in Microbiology. 20 (8), 360-368 (2012).
- Seveau, S. Multifaceted activity of listeriolysin O, the cholesterol-dependent cytolysin of Listeria monocytogenes. Subcellular Biochemistry. 80, 161-195 (2014).
- Osborne, S. E., Brumell, J. H. Listeriolysin O: from bazooka to Swiss army knife. Philosophical Transactions of the Royal Society of London B: Biological Sciences. 372 (1726), (2017).
- Lukoyanova, N., Hoogenboom, B. W., Saibil, H. R. The membrane attack complex, perforin and cholesterol-dependent cytolysin superfamily of pore-forming proteins. Journal of Cell Science. 129 (11), 2125-2133 (2016).
- Tweten, R. K. Cholesterol-dependent cytolysins, a family of versatile pore-forming toxins. Infection and Immunity. 73 (10), 6199-6209 (2005).
- Koster, S., et al. Crystal structure of listeriolysin O reveals molecular details of oligomerization and pore formation. Nature Communications. 5, 3690(2014).
- Duncan, J. L., Schlegel, R. Effect of streptolysin O on erythrocyte membranes, liposomes, and lipid dispersions. A protein-cholesterol interaction. Journal of Cell Biology. 67 (1), 160-174 (1975).
- Morgan, P. J., et al. Subunit organisation and symmetry of pore-forming, oligomeric pneumolysin. FEBS Letters. 371 (1), 77-80 (1995).
- Leung, C., et al. Stepwise visualization of membrane pore formation by suilysin, a bacterial cholesterol-dependent cytolysin. eLife. 3, (2014).
- Marchioretto, M., et al. What planar lipid membranes tell us about the pore-forming activity of cholesterol-dependent cytolysins. Biophysical Chemistry. 182, 64-70 (2013).
- Palmer, M., et al. Assembly mechanism of the oligomeric streptolysin O pore: the early membrane lesion is lined by a free edge of the lipid membrane and is extended gradually during oligomerization. European Molecular Biology Organization Journal. 17 (6), 1598-1605 (1998).
- Bavdek, A., et al. pH dependence of listeriolysin O aggregation and pore-forming ability. Federation of European Biochemical Society Journal. 279 (1), 126-141 (2012).
- Schuerch, D. W., Wilson-Kubalek, E. M., Tweten, R. K. Molecular basis of listeriolysin O pH dependence. Proceeding of the National Academy of Sciences of the United States of America. 102 (35), 12537-12542 (2005).
- Cassidy, S. K., O'Riordan, M. X. More than a pore: the cellular response to cholesterol-dependent cytolysins. Toxins (Basel). 5 (4), 618-636 (2013).
- Lam, J., et al. Host cell perforation by listeriolysin O (LLO) activates a Ca(2+)-dependent cPKC/Rac1/Arp2/3 signaling pathway that promotes L. monocytogenes internalization independently of membrane resealing. Molecular Biology of the Cell. , (2017).
- Gekara, N. O., Weiss, S. Lipid rafts clustering and signalling by listeriolysin O. Biochemical Society Transactions. 32 (Pt 5), 712-714 (2004).
- Magassa, N., Chandrasekaran, S., Caparon, M. G. Streptococcus pyogenes cytolysin-mediated translocation does not require pore formation by streptolysin O. European Molecular Biology Organization Reports. 11 (5), 400-405 (2010).
- Baba, H., et al. Induction of gamma interferon and nitric oxide by truncated pneumolysin that lacks pore-forming activity. Infection and Immunity. 70 (1), 107-113 (2002).
- Carrero, J. A., Vivanco-Cid, H., Unanue, E. R. Listeriolysin o is strongly immunogenic independently of its cytotoxic activity. Public Library of Science One. 7 (3), e32310(2012).
- Coconnier, M. H., et al. Listeriolysin O-induced stimulation of mucin exocytosis in polarized intestinal mucin-secreting cells: evidence for toxin recognition of membrane-associated lipids and subsequent toxin internalization through caveolae. Cell Microbiology. 2 (6), 487-504 (2000).
- Suzuki, T., et al. DNA staining for fluorescence and laser confocal microscopy. Journal of Histochemistry and Cytochemistry. 45 (1), 49-53 (1997).
- Bink, K., et al. TO-PRO-3 is an optimal fluorescent dye for nuclear counterstaining in dual-colour FISH on paraffin sections. Histochemistry and Cell Biology. 115 (4), 293-299 (2001).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nature Methods. 6 (8), 569-575 (2009).
- Zhang, X. D. A pair of new statistical parameters for quality control in RNA interference high-throughput screening assays. Genomics. 89 (4), 552-561 (2007).
- Zhang, X. D. A new method with flexible and balanced control of false negatives and false positives for hit selection in RNA interference high-throughput screening assays. Journal of Biomolecular Screening. 12 (5), 645-655 (2007).
- Idone, V., et al. Repair of injured plasma membrane by rapid Ca2+-dependent endocytosis. Journal of Cell Biology. 180 (5), 905-914 (2008).
- Davenport, N. R., et al. Membrane dynamics during cellular wound repair. Molecular Biology of the Cell. 27 (14), 2272-2285 (2016).
- Defour, A., Sreetama, S. C., Jaiswal, J. K. Imaging cell membrane injury and subcellular processes involved in repair. Journal of Visualized Experiments. (85), (2014).
- Lee, J. J. A., et al. Cell Membrane Repair Assay Using a Two-photon Laser Microscope. Journal of Visualized Experiments. (131), (2018).
- Weisleder, N., et al. Visualization of MG53-mediated cell membrane repair using in vivo and in vitro systems. Journal of Visualized Experiments. (52), (2011).
- Corrotte, M., et al. Toxin pores endocytosed during plasma membrane repair traffic into the lumen of MVBs for degradation. Traffic. 13 (3), 483-494 (2012).
- Kuismanen, E., Saraste, J. Low temperature-induced transport blocks as tools to manipulate membrane traffic. Methods in Cell Biology. 32, 257-274 (1989).
- Togo, T., et al. The mechanism of facilitated cell membrane resealing. Journal of Cell Science. 112, 719-731 (1999).
- Johnson, S. A., et al. Temperature-dependent phase behavior and protein partitioning in giant plasma membrane vesicles. Biochimica et Biophysica Acta. 1798 (7), 1427-1435 (2010).
- Lam, J. G. T., et al. Host cell perforation by listeriolysin O (LLO) activates a Ca(2+)-dependent cPKC/Rac1/Arp2/3 signaling pathway that promotes Listeria monocytogenes internalization independently of membrane resealing. Molecular Biology of the Cell. 29 (3), 270-284 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados