É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudando o tráfico de proteínas de membrana em células fotorreceptoras de Drosophila usando proteínas marcadas pelo eGFP
Neste Artigo
Resumo
Aqui, métodos não invasivos são descritos para localização de proteínas de membrana fotorreceptora e avaliação da degeneração da retina no olho composto de Drosophila usando fluorescência eGFP.
Resumo
O tráfico de proteínas de membrana regula a incorporação e remoção de receptores e canais de íons na membrana plasmática. Esse processo é fundamentalmente importante para a função celular e a integridade celular dos neurônios. As células fotorreceptoras de drosophila tornaram-se um modelo para estudar o tráfico de proteínas de membrana. Além da rodopsina, que após a iluminação torna-se internalizada a partir da membrana fotorreceptora e é degradada, o canal de íons de íons potencial-semelhante ao receptor transitório (TRPL) em Drosophila exibe uma translocação dependente da luz entre a membrana fotorreceptora rabdomeral (onde está localizada no escuro) e o corpo celular fotorreceptor (para o qual é transportado após a iluminação). Este transporte intracelular de TRPL pode ser estudado de forma simples e não invasiva expressando TRPL marcado pelo EGFP em células fotorreceptoras. A fluorescência eGFP pode então ser observada tanto no pseudopupil profundo quanto por microscopia de imersão hídrica. Esses métodos permitem a detecção de fluorescência no olho intacto e, portanto, são úteis para ensaios de alto rendimento e telas genéticas para mutantes de Drosophila defeituosos na translocação TRPL. Aqui, a preparação de moscas, as técnicas microscópicas, bem como os métodos de quantificação utilizados para estudar esta translocação acionada pela luz de TRPL são explicados em detalhes. Esses métodos podem ser aplicados também para estudos de tráfico em outras proteínas fotorreceptoras de Drosophila , por exemplo, rhodopsina. Além disso, utilizando proteínas rabdomerais marcadas pelo eGFP, esses métodos podem ser usados para avaliar a degeneração de células fotorreceptoras.
Introdução
Ao fornecer e remover proteínas de e para a membrana plasmática, o tráfico de proteínas de membrana nos neurônios controla o equipamento de membrana plasmática com receptores, bem como canais de íons e, como resultado, regula a função neuronal. A má regulação ou defeitos no tráfico de proteínas normalmente têm efeitos prejudiciais sobre as células e resultam em degeneração neuronal. Em humanos, isso pode causar doenças neurodegenerativas como Alzheimer e Mal de Parkinson ou Retinite pigmentosa1. Fotorreceptores no olho composto de Drosophila melanogaster tornaram-se um sistema modelo in vivo para estudar o tráfico de proteínas de membrana2. Isso não se deve apenas à versatilidade genética da Drosophila que permite telas genéticas eficazes, mas também porque todos os componentes essenciais da membrana fotorreceptora absorvente da luz são caracterizados em grande detalhe e técnicas microscópicas eficientes estão disponíveis que podem ser aplicadas ao olho mosca. Essas técnicas são o foco deste artigo.
Nas células fotorreceptoras de Drosophila, a membrana plasmática apical forma uma pilha densamente embalada de microvilli ao longo de um lado da célula, chamada rabdomero. Os rabdomeres das células fotorreceptoras R1-6 são dispostos em um padrão trapezoidal característico, enquanto as células fotorreceptoras R7 e R8 formam um único rabdomero no centro deste trapezoide3. O tráfico de proteínas de membrana é necessário para uma rotatividade regulamentada de proteínas de membrana rabdomeral, como a rodopsina e os canais de íons TRP ativados pela luz (potencial do receptor transitório) e TRPL (semelhante ao TRP) para garantir a quantidade adequada dessas proteínas de fototransdução no rabdomere. As proteínas da membrana fotorreceptora são sintetizadas no ânticulo endoplasmático e transportadas através do aparelho Golgi para o rabdomiô. Após a ativação da rodopsina pela luz, uma molécula de rodopsina pode ficar inativada pela absorção de um segundo fóton ou pode ser removida do rabdomero por endocitose mediada por clathrin. A rodopsina endocitada ou se degrada no lysosome ou é reciclada de volta ao rabdomere 4,5. O canal de íonS TRPL também é internalizado após a ativação da cascata de fototransdução e passa por uma translocação dependente da luz entre o rhabdomere (onde está localizado quando as moscas são mantidas no escuro) e um compartimento de armazenamento enriquecido em ER no corpo celular (para o qual é transportado dentro de várias horas após a iluminação)6,7, 8, 9,10 . Em contraste com a rodopsina endocitada, apenas pequenas quantidades de TRPL são degradadas através da via endolíssóica, e a maioria é armazenada intracelularmente em vez disso e reciclada de volta ao rabdomero após a adaptação escura6. Assim, o TRPL pode ser usado para analisar o tráfico de proteínas de membrana plasmática. As células fotorreceptoras de drosophila também são empregadas para estudar a degeneração neuronal. A degeneração celular fotorreceptora é frequentemente determinada pela avaliação da estrutura dos rabdomiados, que se desintegram como resultado dos processos degenerativos5.
Para estudar a localização subcelular de TRPL e rhodopsina em células fotorreceptoras ou degeneração celular fotorreceptora, dois métodos de microscopia de fluorescência que diferem em relação à velocidade e resolução da análise foram aplicados aqui. Um método muito rápido e não invasivo que pode ser usado para telas genéticas, mas com uma resolução espacial limitada é a detecção de fluorescência no pseudopupil profundo (DPP). O DPP é um fenômeno óptico de olhos compostos artrópodes cuja origem geométrica foi explicada em detalhes por Franceschini e Kirschfeld em 197111. Em suma, em vários planos ópticos abaixo da sobreposição de retina imagens de rabdomeros de ommatidia adjacentes podem ser observados. Em um plano focal através do centro da curvatura do olho, essas projeções sobrepostas formam uma imagem que se assemelha ao layout trapezoidal de rabdomeres em um único ommatidium apenas ordens de magnitude maior. Este fenômeno também pode ser observado independentemente da expressão exógena de proteínas de fluorescência (por exemplo, TRPL:::eGFP8), que, no entanto, tornam o DPP mais fácil de detectar (Figura 1A-A'')12. Um segundo método não invasivo é a microscopia de imersão hídrica que se baseia em proteínas fluorescentes marcadas por imagens após neutralizar o aparelho dicótrico dos olhos com água (Figura 1B-C'')12. Utilizando o método de imersão em água, a quantidade relativa de TRPL::eGFP nos rabdomers ou corpo celular pode ser avaliada quantitativamente para células fotorreceptoras individuais. Além disso, proteínas não translocantes marcadas por fluorescência podem ser utilizadas para avaliar a integridade rabdomeral e determinar o curso temporal de uma potencial degeneração de forma quantitativa, conforme descrito aqui.
Embora as gravações do DPP sejam de longe os métodos mais fáceis e rápidos de executar, a resolução espacial dos dados que eles geram é limitada. Além disso, existem inúmeras razões pelas quais um DPP pode estar ausente, que não são necessariamente perceptíveis pela própria imagem DPP. Uma vez que o DPP representa uma soma de várias ommatidia, as informações sobre células individuais são perdidas. Assim, a imagem DPP de baixa resolução serve a uma função importante na triagem de um grande número de moscas, mas geralmente deve ser seguida por gravações de maior resolução por meio de microscopia de imersão hídrica. Micrografos de imersão hídrica permitem interpretações sobre células individuais, defeitos de desenvolvimento, morfologia ocular, mislocalização proteica ou degeneração da retina, bem como quantificação desses efeitos. Este Protocolo descreve essas duas técnicas em detalhes.
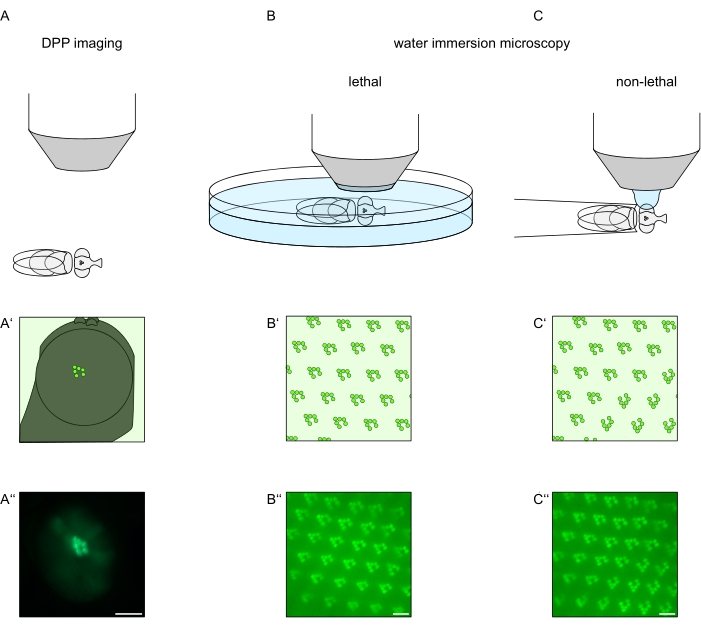
Figura 1: Visão geral das variações de microscopia para o olho de Drosophila apresentadas neste Protocolo. Representações esquemáticas e micrografias exemplares de (A-A'') imagem de pseudopupil fluorescente (DPP), (B-B'') microscopia letal de imersão em água de rabdominais fluorescentes, e (C-C'') microscopia de gota de água não letal de rabdomeros fluorescentes. Barra de escala (A''): 100 μm. Barras de escala (B''-C''): 10 μm. O valor foi modificado a partir da referência13. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Considerações gerais
- Use estoques de Drosophila expressando uma proteína de fluorescência localizada permanentemente para análise morfológica (por exemplo, TRP:::eGFP, eGFP::NINAC) e proteínas translocantes para análises sobre tráfico de proteínas (por exemplo, TRPL:::eGFP, Arr2:::eGFP).
- Condições de exposição à luz pré-dedeterminadas de moscas selecionadas para a abordagem experimental.
- Para adaptação escura, mantenha as moscas em caixas escuras pelo período desejado a 25 °C. Para iluminação de até 16 h em experimentos de translocação (por exemplo, TRPL::eGFP expressando moscas), mantenha moscas sob um tubo fluorescente à temperatura ambiente.
- Em experimentos que avaliam a degeneração do fotorreceptor (por exemplo, moscas expressas TRP::eGFP), mantenha as moscas sob um tubo fluorescente a 25 °C em uma luz de 12 h / ciclo escuro de 12 h para iluminação de longo prazo de até 28 dias com luz branca.
- Para iluminar moscas com luz colorida, use diferentes caixas de plástico transparentes coloridas junto com o tubo fluorescente.
- Se os estoques de moscas com olhos pigmentados são usados, os animais de idade precisamente para análise comparativa, uma vez que a pigmentação dos olhos pode aumentar significativamente com a idade.
NOTA: Para a interpretação dos dados, é importante notar que existe um viés de sinal devido ao efeito óptico de orientação de ondas da estrutura rabdomeral. Assim, o sinal de fluorescência do rabdomero será sempre amplificado até certo ponto em imagens DPP e microscopia de imersão de água em relação aos sinais obtidos do corpo celular. Isso é mais proeminente observado em olhos pigmentados, onde a fluorescência de fora dos rabdomers é absorvida por esses pigmentos e é de particular importância quando proteínas de fusão intracelularmente translocantes devem ser detectadas. Assim, no que diz respeito a passos críticos, este estudo considera separadamente as moscas brancas e de olhos vermelhos. - Quanto à microscopia de imersão hídrica, são descritas duas variações. Uma variação letal mais rápida, bem como uma variação não letal permitindo a recuperação para estudos subsequentes.
2. Imagem DPP
- Prepare o espaço de trabalho com os equipamentos e reagentes necessários, como mostrado na Figura 2. Moscas de anestesizante (1-3 dias de idade) de um genótipo que expressa uma proteína de fluorescência em células fotorreceptoras com CO2 em um flypad. Selecione animais para imagem sob um microscópio estéreo com uma fonte de luz convencional e baixa ampliação (por exemplo, 10x).

Figura 2: Espaço de trabalho de imagem DPP. Os materiais necessários são (A) aparelho de anestesia CO2 , (B) microscópio estéreo com uma lâmpada UV e conjunto de filtro de fluorescência, (C) fonte de luz, (D) câmera montada no microscópio com (E) software, (F) pincel de tinta, (G) papelão preto e (H) frasco de mosca. Clique aqui para ver uma versão maior desta figura.
- Para a imagem DPP, mantenha as moscas selecionadas anestesiadas e posicione uma delas no centro do objetivo do microscópio em seu lado para que o olho esquerdo ou direito esteja voltado precisamente para o objetivo (Figura 3A).
NOTA: Uma vez que o layout ommatidial das células fotorreceptoras exibe simetria espelhada na linha média dorsoventral, o DPP é melhor observado ligeiramente acima ou abaixo do equador do olho (Figura 3B).
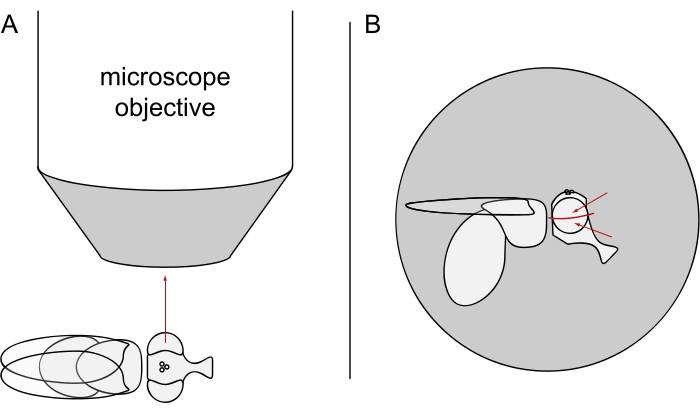
Figura 3: Posicionamento da mosca sob o estereóquio para imagem DPP. (A) Ilustração da mosca de lado com um olho voltado para o microscópio objetivo radialmente. (B) A cabeça de mosca precisa ser virada ligeiramente para cima ou para baixo de modo que o objetivo se concentre em um ponto ligeiramente acima ou abaixo do equador do olho, conforme indicado pelas setas vermelhas. Clique aqui para ver uma versão maior desta figura.
- Aumente a ampliação para caber o olho inteiro (por exemplo, 100x) e centralizar a ommatidia central do olho. Diminua a profundidade de campo do microscópio, por exemplo, ajustando o diafragma de íris dupla para um ajuste raso (Figura 4A-B').
- Desligue a fonte de luz convencional e ligue a lâmpada UV do microscópio com intensidade máxima e selecione o conjunto do filtro de fluorescência do microscópio de acordo com a proteína de fluorescência expressa nos olhos (Figura 4C-E). Coloque o caminho da luz em direção à câmera montada no microscópio.
- Use o recurso de imagem ao vivo dentro do software para ajustar o brilho da imagem a uma configuração que detecta apenas sinais específicos do olho, ajustando o tempo de exposição e o valor de ganho (por exemplo, 80 ms e 12x, respectivamente). Reajustar o foco do microscópio "em" o olho (abaixo da córnea) para gerar a imagem sobreposta da DPP (Figura 4C-E').
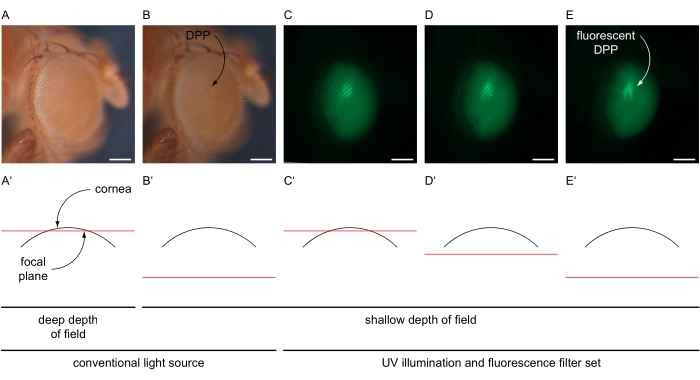
Figura 4: Ilustração de DPP e imagem fluorescente DPP. Imagens exemplares de olhos de Drosophila sob iluminação convencional e UV com conjunto de filtro GFP, tiradas com diferentes planos focais ilustrados em seções transversais esquemáticas através do olho. (A) Micrografia registrada com configurações brilhantes de uma fonte de luz convencional, tempo de exposição de 30 ms, ganho de 1x, profundidade profunda de campo e plano focal perto da superfície da córnea como ilustrado em (A'). (B) Micrografia registrada com configurações brilhantes de uma fonte de luz convencional, tempo de exposição de 30 ms, ganho de 1x, profundidade rasa de campo e plano focal aproximadamente 180 μm abaixo da superfície da córnea como ilustrado em (B'). DPP indicado. (C-E) Micrografo registrado com configurações de alta intensidade de fonte de luz UV e conjunto de filtro GFP, tempo de exposição de 80 ms, ganho de 12x, profundidade rasa de campo e o plano focal (C') perto, (D') ligeiramente abaixo, ou (E') aproximadamente 180 μm abaixo da superfície da córnea. DPP fluorescente é indicado com uma seta curva. Barra de escala 100 μm. Clique aqui para ver uma versão maior desta figura.
- Tire uma foto do DPP fluorescente. Mude o microscópio de volta para a luz visível e o caminho de luz de volta para os oculares. Recupere o animal imaged em um frasco de mosca para procedimentos adicionais de acordo com seu fenótipo DPP (por exemplo, cruzes). Continue com o próximo animal na etapa 2.2.
3. Microscopia de imersão hídrica
- Preparação de moscas
- Prepare o espaço de trabalho com os equipamentos e reagentes necessários, conforme mostrado na Figura 5. Transfira moscas com idade predeterminada e condições de iluminação em um tubo de centrífugas pré-resfriado de 15 mL e anestesiar incubando-as no gelo por 15 a 30 minutos.
NOTA: Leve ao longo de 1 dia de idade, moscas de adaptação escura como referência. Geralmente, moscas adaptadas à escuridão devem ser transferidas para uma geladeira com uma tampa no escuro. Moscas adaptadas à luz podem ser transferidas para o gelo à luz da sala.
- Prepare o espaço de trabalho com os equipamentos e reagentes necessários, conforme mostrado na Figura 5. Transfira moscas com idade predeterminada e condições de iluminação em um tubo de centrífugas pré-resfriado de 15 mL e anestesiar incubando-as no gelo por 15 a 30 minutos.

Figura 5: Espaço de trabalho de microscopia de imersão de água. Os materiais necessários são: (A) tubo de centrífuga de 15 mL, (B) flocos de gelo, (C) água destilada resfriada, (D) estereomicroscópio, (E) placa de Petri, (F) plasticina, (G) slide de objeto, (H) pinos de inseto ou pontas de pipeta e bisturi, (I) microscópio de fluorescência com (J) software. Clique aqui para ver uma versão maior desta figura.
- Selecione a preparação adequada dos dois descritos abaixo (variação letal de 3.1.3 ou variação não letal de 3,1,7) e sempre que a diferenciação entre olhos pigmentados e não pigmentados for feita, siga os respectivos passos.
- Prepare moscas para a variação letal da seguinte forma.
- Adere a um pedaço de plasticine em um slide de objeto e outra peça no centro de uma placa de Petri (por exemplo, 94 mm Ø) e mantenha-os separados por enquanto. Encha a placa de Petri com água destilada gelada e alguns flocos de gelo (Figura 6A).
- Coloque uma mosca anestésica sob um estereómico em cima do slide de objeto revestido de plasticina. Vire a mosca de costas e perfure um pino de inseto pelo centro do tórax (Figura 6B). Fixar o pino horizontalmente no slide do objeto revestido de plasticina e orientar o olho esquerdo ou direito da mosca para cima (Figura 6C).
- Fixar cuidadosamente o slide do objeto, com seu lado sem plasticina voltado para baixo, na placa de Petri impedindo a rotação da mosca. Certifique-se de que o olho de mosca está coberto de água (Figura 6D). Use uma agulha de preparação para remover cuidadosamente quaisquer bolhas de ar que possam ter se formado ao redor do olho, e proceda imediatamente à aquisição de imagens para melhores resultados.
NOTA: Atraso significativo na aquisição de imagens resulta em despertar e movimentos da mosca que podem levar a imagens embaçadas.
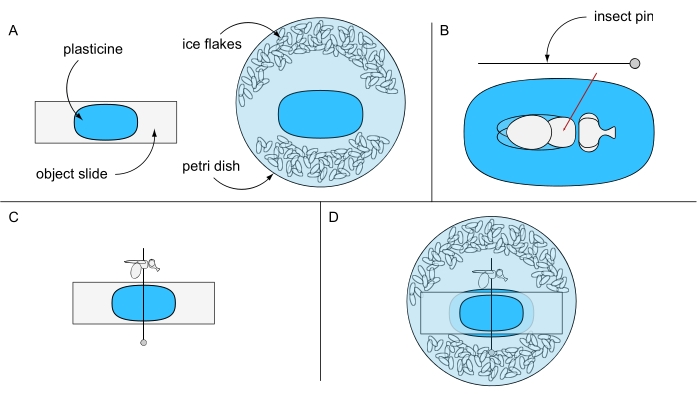
Figura 6: Preparação para microscopia letal de imersão em água. Ilustração de (A) slide de objeto revestido de plasticine e placa de Petri, (B) fixando uma mosca através do tórax em solo de plasticina, (C) orientação de mosca no slide do objeto revestido de plasticine e (D) configuração experimental final. Clique aqui para ver uma versão maior desta figura.
- Prepare moscas para a variação não letal da seguinte forma.
- Transfira uma mosca anestriizada de gelo de cabeça para uma ponta de pipeta de 200 μL e empurre a mosca em direção à ponta cuidadosamente com ar comprimido.
- Corte a ponta da pipeta bem na frente da cabeça usando um bisturi. Usando pinças, empurre cuidadosamente a mosca a poucos milímetros da ponta. Corte a ponta da pipeta novamente e empurre a mosca de volta para a ponta com ar comprimido para que apenas a cabeça da mosca se saliente da ponta da pipeta.
- Adere um pedaço de plasticine em um slide de objeto e pressione a ponta da pipeta nele de modo que o olho esquerdo ou direito da mosca fique para cima (Figura 7A). Logo antes da aquisição da imagem, use uma pipeta de laboratório para aderir a uma grande gota de água gelada na parte inferior de um objetivo de imersão em água (Figura 7B). Imediatamente prossiga para aquisição de imagem para melhores resultados.
NOTA: Atraso significativo na aquisição de imagens resulta em despertar e movimentos da mosca que podem levar a imagens embaçadas.
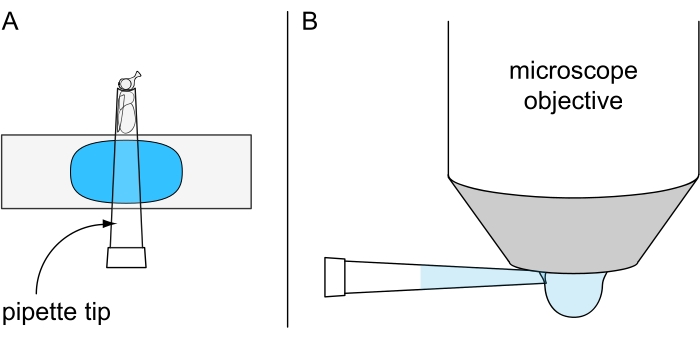
Figura 7: Preparação para microscopia de gota de água não letal. Ilustração de (A) uma mosca anestinada a frio fixada dentro de uma ponta de pipeta de 200 μL montada no slide do objeto revestido de plasticina e (B) a aplicação de gota de água gelada na parte inferior do objetivo de imersão de água. Clique aqui para ver uma versão maior desta figura.
- Aquisição de imagens
- Coloque cuidadosamente a placa de Petri (etapa 3.1.3) ou o slide do objeto (passo 3.1.7) com a mosca preparada para o estágio do microscópio e selecione um objetivo de imersão na água.
- Abaixe o objetivo de imersão de água manualmente até que entre em contato com a superfície da água (passo 3.1.3) ou o olho da mosca toque a gota (passo 3.1.7) (Figura 8A,B).
- Ligue a lâmpada UV do microscópio e selecione o conjunto de filtros apropriado. Use as oculares para posicionar a mosca sob o objetivo e concentrar o microscópio na superfície do olho.
- Alterne o caminho da luz em direção à câmera do microscópio e gere uma imagem ao vivo no software correspondente. Reajuste o foco para a câmera e avalie a orientação do olho, considerando que o olho tem que enfrentar o microscópio objetivo radialmente como ilustrado em mais detalhes na Figura 8C-E.
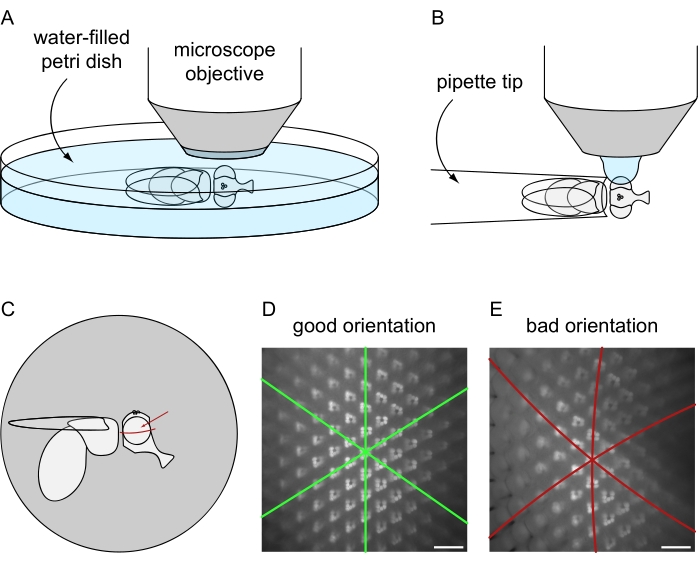
Figura 8: Posicionamento da mosca sob o microscópio de fluorescência para imagens de imersão hídrica. Configuração e orientação final para aquisição de imagens usando os protocolos (A) letais ou (B) de preparação de moscas não letais. (C) Ilustração da orientação de moscas para obtenção dos melhores resultados de imagens de microscopia de imersão em água. O ponto ideal para focar no olho não é o centro exato em relação aos eixos anterior/posterior e dorsal/ventral, mas está ligeiramente acima do equador do olho, como indicado pela seta vermelha. (D) Exemplo de imagem de imersão em água para um olho perfeitamente posicionado. Todos os três eixos de simetria do revestimento ommatidial hexagonal aparecem como linhas retas e a quantidade máxima de ommatidia pode estar em foco ao mesmo tempo. (E) Exemplo de imagem de imersão em água de um olho mal posicionado. A imagem contém eixos curvos e uma profundidade rasa de campo. Barra de escala: 20 μm. Clique aqui para ver uma versão maior desta figura.
- Use o LUT apropriado (procure a tabela) dentro do software de imagem para detectar a supersaturação (indicada como pixels vermelhos).
- No caso de moscas não pigmentadas, ajuste o tempo de exposição de modo que os pixels mais brilhantes estejam logo abaixo do limite de saturação para cada imagem.
- No caso das moscas pigmentadas e da variação letal, ajuste o tempo de exposição de modo que todos os pixels mais brilhantes estejam logo abaixo do limite de saturação em pelo menos cinco moscas individuais de 1 dia de idade, adaptadas à escuridão. Aplique o tempo médio de exposição calculado a todas as outras condições experimentais (genótipos, condições de iluminação, pontos de tempo, etc.).
- No caso de moscas pigmentadas (por exemplo, olhos vermelhos) e variação não letal, ajuste o tempo de exposição de modo que todos os pixels mais brilhantes estejam logo abaixo do limite de saturação para cada mosca de 1 dia de idade, adaptada à escuridão individualmente. Aplique este tempo de exposição a todas as outras condições experimentais (condições de iluminação, pontos de tempo, etc.) desta mosca.
- Grave uma imagem e salve-a como um arquivo bruto para arquivar todos os metadados correspondentes da gravação. Exporte a imagem em um formato .tif para a seguinte quantificação.
NOTA: No caso da variação não letal, ilumine as moscas por 5 minutos com luz vermelha (por exemplo, 630 nm) imediatamente após a aquisição da imagem, se elas forem destinadas a serem usadas para outros experimentos. A luz vermelha desativa a cascata de fototransdução que foi ativada excessivamente pela intensa luz de ondas curtas durante a aquisição de imagens.
- Análise de dados e quantificação da fluorescência relativa eGFP nos rabdomers de micrografos de imersão hídrica
- Baixe, instale e execute o software ImageJ/Fiji.
- Ajuste as configurações do ImageJ clicando em Analisar > definir medidas... e verifique apenas a caixa para O Valor Cinza Médio. Importe uma imagem .tif clicando no Arquivo > Aberto... ou arrastando e caindo. Escolha uma região representativa da imagem que esteja em foco e amplie-a para 200%-300% pressionando repetidamente Ctrl e + juntos .
- Selecione a ferramenta Oval e ao pressionar a tecla Shift gere uma seleção circular na imagem significativamente menor que um rabdomero fluorescente. Antes de soltar o botão do mouse, procure o tamanho exato exibido abaixo da barra de ferramentas na janela principal imageJ. Use o mesmo tamanho de seleção circular para todas as análises.
NOTA: O tamanho exato da seleção circular em pixels ou mícrons depende da configuração específica. Use um círculo de aproximadamente 1/3 ou 1/4 do diâmetro rabdomeral de moscas de controle de 1 dia de idade, adaptadas à escuridão. - Mova a seleção circular clicando nele e arrastando ou pressionando as Teclas de Seta no teclado.
- Para medir as intensidades de fluorescência dentro da seleção circular, mova o círculo para o primeiro rabdomero (r1) e clique em Analisar > Medida ou use o atalho Ctrl + M. Uma janela resultando listando o valor cinza medido aparecerá.
- Continue com medições de r2-r6 como medições repetidas e uma medição do sinal de fundo (b). No caso de moscas não pigmentadas, faça medições adicionais das áreas correspondentes do corpo celular (c1-c6) (Figura 9).
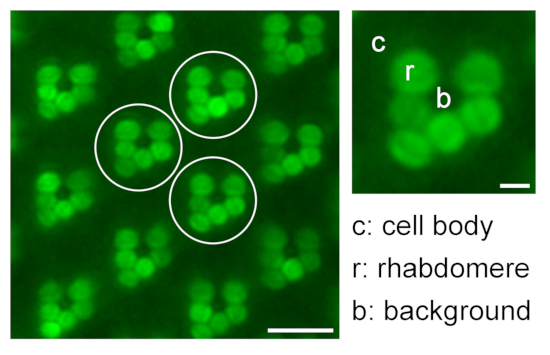
Figura 9: Quantificação da fluorescência rhabdomeral relativa para estudos de translocação. Ilustração sobre quantificação da fluorescência relativa do eGFP nos rabdomers medindo a intensidade de fluorescência de rabdomere (r), corpo celular (c) e fundo (b) de três ommatidia representativas diferentes (círculos brancos) em uma imagem de microscopia de imersão de água; barra de escala: 10 μm. Um ommatidium ampliado é mostrado à direita; barra de escala: 2 μm. Clique aqui para ver uma versão maior desta figura.
- Repetir as etapas 3.3.5 e 3.3.6 para mais duas ommatidia, resultando em três réplicas técnicas. Marque a ommatidia analisada usando a ferramenta Lápis e salve esta imagem para documentação.
- Selecione e copie os valores de cinza medidos da janela Resultado e cole-os em software de planilha para cálculos adicionais. Classificar os valores da intensidade da fluorescência de acordo com sua origem nas categorias rabdomere (r), corpo celular (c) e fundo (b). Calcular a intensidade média de cada categoria (Ir, Ic, Ib).
- Calcule a quantidade relativa de eGFP presente no rabdomere (R), utilizando a seguinte fórmula (1) para não pigmentado e fórmula (2) para olhos pigmentados:
 (1)
(1) (2)
(2) - Continue com a próxima imagem na etapa 3.3.3. Recomenda-se o uso de imagens de pelo menos cinco indivíduos de cada grupo experimental como réplicas biológicas para obter uma medição confiável.
- Análise de dados e quantificação da morfologia ocular por fluorescência eGFP em micrografos de imersão hídrica
- Baixe, instale e execute o software ImageJ/Fiji. Importe uma imagem .tif clicando no Arquivo > Aberto... ou arrastando e soltando. Escolha três ommatidia adjacentes em uma região representativa da imagem que está em foco.
- Avalie os 18 rabdomers da seleção individualmente de acordo com sua intensidade eGFP, nitidez de borda e contraste com relação ao sinal de fundo circundante para gerar um índice de degeneração. Pontuação de rabdomeres claramente visíveis com um valor de 2, rabdomeres fracamente visíveis com um valor de 1, e rabdomeres ausentes com um valor de 0 (Figura 10).
NOTA: Esta forma de quantificação resulta em uma pontuação de 36 para olhos totalmente intactos e uma pontuação de 0 para olhos totalmente degenerados. Recomenda-se definir a pontuação de 36 a 100% no índice de degeneração.

Figura 10: Quantificação via avaliação de rabdomere para estudos de degeneração. Uma ilustração sobre quantificação da morfologia ocular marcando os rabdomeros de três ommatidia representativas diferentes (círculos brancos) em uma imagem de microscopia de imersão de água com valores de 2 (claramente visível; círculo azul), 1 (fracamente visível; círculo laranja) ou 0 (ausente; círculo vermelho). Barra de escala: 10 μm. Um ommatidium ampliado é mostrado à direita; barra de escala: 2 μm. Clique aqui para ver uma versão maior desta figura.
- Abra a próxima imagem e continue com o passo 3.4.2. Recomenda-se o uso de imagens de pelo menos oito indivíduos de cada grupo experimental como réplicas biológicas para obter uma medição confiável.
NOTA: Uma vez que este método de quantificação é menos objetivo do que o método de quantificação da translocação por intensidade de fluorescência, o número recomendado de réplicas é maior.
Resultados
Foram geradas moscas transgênicas de Drosophila expressando uma proteína de fusão TRPL::eGFP sob o controle do promotor de rodopsina 1. Nestas moscas, TRPL::eGFP é expresso em células fotorreceptoras R1-6 do olho composto e exibe uma localização dependente da iluminação. Quando as moscas são mantidas no escuro, TRPL::eGFP é incorporada aos rabdomiôs externos. Após a iluminação por várias horas, o TRPL transloca-se no corpo celular onde é armazenado em um compartimento enriquecido com ER.
Discussão
A aplicabilidade das proteínas de fluorescência e a simplicidade da triagem por imagem DPP e microscopia de imersão em água da retina tem sido comprovadamente bem sucedida por muitos grupos12. Estratégias semelhantes às apresentadas aqui têm sido utilizadas em diversas telas genéticas para detectar defeitos nos níveis de expressão de rodopsina, homeostase, organização da retina ou integridade celular com a ajuda de Rh1::eGFP 17,18,19,20,21.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Gostaríamos de agradecer aos nossos pesquisadores estudantes ao longo dos anos. Em particular, Nina Meyer, Sibylle Mayer, Juliane Kaim e Laura Jaggy, cujos dados foram utilizados neste protocolo como resultados representativos. A pesquisa do nosso grupo aqui apresentada foi financiada por subvenções da Deutsche Forschungsgemeinschaft (Hu 839/2-4, Hu 839/7-1) para Armin Huber.
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Greiner Bio-One | 188271 | |
| CO2 anaesthesia fly pad | Flystuff | 59-172 | |
| Cold light lamp (KL 1500 LCD) | Zeiss | ||
| Fiji/ImageJ | NIH | ||
| Fluorescence microscope with UV lamp, camera, filter set and software (AxioImager.Z1m, Axiocam 530 mono, 38 HE, ZEN2 blue edition) | Zeiss | ||
| Fluorescent tube (Lumilux T8, L 30W/840, 4000 K, G13) [1750 Lux, Ee470nm = 298 µW cm-2, Ee590nm = 215 µW cm-2] and [760 Lux, Ee470nm = 173 µW cm-2, Ee590nm = 147 µW cm-2] | Osram | 4050300518039 | |
| Laboratory pipette (20-200 µL) | Eppendorf | ||
| Object slide | Roth | 0656.1 | |
| Petri dish (94 mm) | Greiner Bio-One | 633102 | |
| Pipette tips (200 µL) | Labsolute | 7695844 | |
| Plasticine (Blu-Tack) | Bostik | 30811745 | |
| Stereo microscope (SMZ445) | Nikon | ||
| Stereo microscope with UV lamp, camera, filer set and software (MZ16F, MC170 HD, GFP3, LAS 4.12) | Leica |
Referências
- Wang, X., Huang, T., Bu, G., Xu, H. Dysregulation of protein trafficking in neurodegeneration. Molecular Neurodegeneration. 9. 9, 31 (2014).
- Schopf, K., Huber, A. Membrane protein trafficking in Drosophila photoreceptor cells. European Journal of Cell Biology. 96 (5), 391-401 (2017).
- Hardie, R. C. The photoreceptor array of the dipteran retina. Trends in Neurosciences. 9, 419-423 (1986).
- Wang, T., Montell, C. Phototransduction and retinal degeneration in Drosophila. Pflügers Archiv - European Journal of Physiology. 454 (5), 821-847 (2007).
- Xiong, B., Bellen, H. J. Rhodopsin homeostasis and retinal degeneration: lessons from the fly. Trends in Neurosciences. 36 (11), 652-660 (2013).
- Bähner, M., et al. Light-regulated subcellular translocation of Drosophila TRPL channels induces long-term adaptation and modifies the light-induced current. Neuron. 34 (1), 83-93 (2002).
- Cronin, M. A., Lieu, M. -. H., Tsunoda, S. Two stages of light-dependent TRPL-channel translocation in Drosophila photoreceptors. Journal of Cell Science. 119, 2935-2944 (2006).
- Meyer, N., Joel-Almagor, T., Frechter, S., Minke, B., Huber, A. Subcellular translocation of the eGFP-tagged TRPL channel in Drosophila photoreceptors requires activation of the phototransduction cascade. Journal of Cell Science. 119, 2592-2603 (2006).
- Oberegelsbacher, C., Schneidler, C., Voolstra, O., Cerny, A., Huber, A. The Drosophila TRPL ion channel shares a Rab-dependent translocation pathway with rhodopsin. European Journal of Cell Biology. 90 (8), 620-630 (2011).
- Wagner, K., Smylla, T. K., Lampe, M., Krieg, J., Huber, A. Phospholipase D and retromer promote recycling of TRPL ion channel via the endoplasmic reticulum. Traffic. , (2021).
- Franceschini, N., Kirschfeld, K. Les phénoménes de pseudopupille dans l'oeil compose de Drosophila. Kybernetik. 9 (5), 159-182 (1971).
- Pichaud, F., Desplan, C. A new visualization approach for identifying mutations that affect differentiation and organization of the Drosophila ommatidia. Development. 128 (6), 815-826 (2001).
- Smylla, T. K., Wagner, K., Huber, A. Application of fluorescent proteins for functional dissection of the Drosophila visual system. International Journal of Molecular Sciences. 22 (16), (2021).
- Meyer, N. Mechanisms of the light-dependent translocation of the ion channel TRPL in the photoreceptors of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer. nat. University of Karlsruhe (TH). , (2005).
- Meyer, N., Oberegelsbacher, C., Dürr, T. D., Schäfer, A., Huber, A. An eGFP-based genetic screen for defects in light-triggered subcelluar translocation of the Drosophila photoreceptor channel TRPL. Fly. 2 (1), 36-46 (2008).
- Zheng, L., Carthew, R. W. Lola regulates cell fate by antagonizing Notch induction in the Drosophila eye. Mechanisms of Development. 125 (1-2), 18-29 (2008).
- Hibbard, K. L., O'Tousa, J. E. A role for the cytoplasmic DEAD box helicase Dbp21E2 in rhodopsin maturation and photoreceptor viability. Journal of Neurogenetics. 26 (2), 177-188 (2012).
- Huang, Y., Xie, J., Wang, T. A Fluorescence-based genetic screen to study retinal degeneration in Drosophila. PloS One. 10 (12), 0144925 (2015).
- Zhao, H., Wang, J., Wang, T. The V-ATPase V1 subunit A1 is required for rhodopsin anterograde trafficking in Drosophila. Molecular Biology of the Cell. 29 (13), 1640-1651 (2018).
- Xiong, L., et al. ER complex proteins are required for rhodopsin biosynthesis and photoreceptor survival in Drosophila and mice. Cell Death and Differentiation. 27 (2), 646-661 (2020).
- Zhao, H., Wang, T. PE homeostasis rebalanced through mitochondria-ER lipid exchange prevents retinal degeneration in Drosophila. PLoS Genetics. 16 (10), 1009070 (2020).
- Cerny, A. C., et al. The GTP- and phospholipid-binding protein TTD14 regulates trafficking of the TRPL ion channel in Drosophila photoreceptor cells. PLoS Genetics. 11 (10), 1005578 (2015).
- Richter, D. Structural and functional analyses of TRP ion channels in the photoreceptor cells of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer, nat. University of Karlsruhe (TH). , (2007).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Developmental Biology. 351 (1), 128-134 (2011).
- Hardie, R. C., Liu, C. -. H., Randall, A. S., Sengupta, S. In vivo tracking of phosphoinositides in Drosophila photoreceptors. Journal of Cell Science. 128 (23), 4328-4340 (2015).
- Chakrabarti, P., et al. A dPIP5K dependent pool of phosphatidylinositol 4,5 bisphosphate (PIP2) is required for G-protein coupled signal transduction in Drosophila photoreceptors. PLoS Genetics. 11 (1), 1004948 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados