È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio del traffico proteico di membrana nelle cellule fotorecettrici di Drosophila utilizzando proteine con tag eGFP
In questo articolo
Riepilogo
Qui, vengono descritti metodi non invasivi per la localizzazione delle proteine di membrana dei fotorecettori e la valutazione della degenerazione retinica nell'occhio composto Drosophila utilizzando la fluorescenza eGFP.
Abstract
Il traffico proteico di membrana regola l'incorporazione e la rimozione di recettori e canali ionici nella membrana plasmatica. Questo processo è di fondamentale importanza per la funzione cellulare e l'integrità cellulare dei neuroni. Le cellule fotorecettori di Drosophila sono diventate un modello per lo studio del traffico proteico di membrana. Oltre alla rodopsina, che dopo l'illuminazione viene internalizzata dalla membrana dei fotorecettori e viene degradata, il canale ionico potenziale del recettore transitorio (TRPL) in Drosophila mostra una traslocazione dipendente dalla luce tra la membrana del fotorecettore rabdomerale (dove si trova al buio) e il corpo cellulare del fotorecettore (a cui viene trasportato all'illuminazione). Questo trasporto intracellulare di TRPL può essere studiato in modo semplice e non invasivo esprimendo TRPL marcato con eGFP nelle cellule dei fotorecettori. La fluorescenza eGFP può quindi essere osservata sia nello pseudopupil profondo che mediante microscopia ad immersione in acqua. Questi metodi consentono il rilevamento della fluorescenza nell'occhio intatto e sono quindi utili per saggi ad alto rendimento e screening genetici per mutanti di Drosophila difettosi nella traslocazione TRPL. Qui, la preparazione delle mosche, le tecniche microscopiche e i metodi di quantificazione utilizzati per studiare questa traslocazione innescata dalla luce di TRPL sono spiegati in dettaglio. Questi metodi possono essere applicati anche per studi di traffico su altre proteine fotorecettrici di Drosophila , ad esempio la rodopsina. Inoltre, utilizzando proteine rabdomerali marcate con eGFP, questi metodi possono essere utilizzati per valutare la degenerazione delle cellule fotorecettrici.
Introduzione
Fornendo e rimuovendo proteine da e verso la membrana plasmatica, il traffico di proteine di membrana nei neuroni controlla l'apparecchiatura della membrana plasmatica con recettori e canali ionici e, di conseguenza, regola la funzione neuronale. La cattiva regolazione o i difetti nel traffico proteico hanno in genere effetti dannosi sulle cellule e provocano la degenerazione neuronale. Nell'uomo, questo può causare malattie neurodegenerative come il morbo di Alzheimer e Parkinson o la retinite pigmentosa1. I fotorecettori nell'occhio composto di Drosophila melanogaster sono diventati un sistema modello in vivo per lo studio del traffico proteico di membrana2. Ciò non è dovuto solo alla versatilità genetica di Drosophila che consente screening genetici efficaci, ma anche perché tutti i componenti essenziali della membrana fotorecettore che assorbe la luce sono caratterizzati in grande dettaglio e sono disponibili tecniche microscopiche efficienti che possono essere applicate all'occhio della mosca. Queste tecniche sono al centro di questo articolo.
Nelle cellule fotorecettori di Drosophila, la membrana plasmatica apicale forma una pila densamente imballata di microvilli lungo un lato della cellula, chiamata rabdomere. I rabdomeri delle cellule fotorecettrici R1-6 sono disposti in un caratteristico schema trapezoidale mentre le cellule fotorecettrici R7 e R8 formano un unico rabdomere al centro di questo trapezio3. Il traffico proteico di membrana è necessario per un turnover regolato delle proteine di membrana rabdomerali come la rodopsina e i canali ionici TRP (potenziale recettore transitorio) e TRPL (TRP-like) attivati dalla luce per assicurare la giusta quantità di queste proteine di fototrasduzione nel rabdomere. Le proteine di membrana dei fotorecettori sono sintetizzate nel reticolo endoplasmatico e trasportate attraverso l'apparato di Golgi al rabdomero. Dopo l'attivazione della rodopsina da parte della luce, una molecola di rodopsina può essere inattivata dall'assorbimento di un secondo fotone o può essere rimossa dal rabdomere mediante endocitosi mediata dalla clatrina. La rodopsina endocitosa viene degradata nel lisosoma o viene riciclata nel rabdomero 4,5. Anche il canale ionico TRPL viene internalizzato in seguito all'attivazione della cascata di fototrasduzione e subisce una traslocazione dipendente dalla luce tra il rabdomere (dove si trova quando le mosche sono tenute al buio) e un vano di stoccaggio arricchito di ER nel corpo cellulare (a cui viene trasportato entro diverse ore dall'illuminazione)6,7,8,9,10 . A differenza della rodopsina endocitosa, solo piccole quantità di TRPL vengono degradate attraverso la via endolisosomiale e la maggior parte viene invece immagazzinata intracellulare e riciclata nel rabdomere dopo l'adattamento oscuro6. TRPL può quindi essere utilizzato per analizzare il traffico innescato dalla luce delle proteine della membrana plasmatica. Le cellule fotorecettrici di Drosophila sono anche impiegate per studiare la degenerazione neuronale. La degenerazione delle cellule fotorecettrici è spesso determinata valutando la struttura dei rabdomeri, che si disintegrano a seguito di processi degenerativi5.
Al fine di studiare la localizzazione subcellulare di TRPL e rodopsina nelle cellule fotorecettrici o la degenerazione cellulare dei fotorecettori, sono stati applicati qui due metodi di microscopia a fluorescenza che differiscono per quanto riguarda la velocità e la risoluzione dell'analisi. Un metodo molto veloce e non invasivo che può essere utilizzato per gli screening genetici ma con una risoluzione spaziale limitata è il rilevamento della fluorescenza nello pseudopupil profondo (DPP). Il DPP è un fenomeno ottico di occhi composti di artropodi la cui origine geometrica è stata spiegata in dettaglio da Franceschini e Kirschfeld nel 197111. In breve, su diversi piani ottici al di sotto della retina si possono osservare immagini di rabdomere da ommatidi adiacenti. Su un piano focale attraverso il centro della curvatura dell'occhio, queste proiezioni sovrapposte formano un'immagine che assomiglia al layout trapezoidale dei rabdomeri in un singolo ommatidio solo ordini di grandezza più grande. Questo fenomeno può essere osservato anche indipendentemente dall'espressione esogena delle proteine di fluorescenza (ad esempio TRPL::eGFP8), che tuttavia rendono il DPP più facile da rilevare (Figura 1A-A'')12. Un secondo metodo non invasivo è la microscopia ad immersione in acqua che si basa sull'imaging di proteine marcate fluorescentemente dopo aver neutralizzato otticamente l'apparato diottrico degli occhi con acqua (Figura 1B-C'')12. Utilizzando il metodo dell'immersione in acqua, la quantità relativa di TRPL::eGFP nei rabdomeri o nel corpo cellulare può essere valutata quantitativamente per le singole cellule fotorecettrici. Inoltre, le proteine non traslocanti marcate con fluorescenza possono essere utilizzate per valutare l'integrità rabdomerale e per determinare il decorso temporale di una potenziale degenerazione in modo quantitativo, come descritto qui.
Mentre le registrazioni del DPP sono di gran lunga il più semplice e veloce di questi metodi da eseguire, la risoluzione spaziale dei dati che generano è limitata. Inoltre, ci sono numerose ragioni per cui un DPP può essere assente, che non sono necessariamente distinguibili dall'imaging DPP stesso. Poiché il DPP rappresenta una somma di diversi ommatidi, le informazioni sulle singole cellule vengono perse. Pertanto, l'imaging DPP a bassa risoluzione svolge una funzione importante nello screening di un gran numero di mosche, ma dovrebbe generalmente essere seguito da registrazioni a risoluzione più elevata mediante microscopia ad immersione in acqua. Le micrografie ad immersione in acqua consentono interpretazioni su singole cellule, difetti dello sviluppo, morfologia oculare, errata localizzazione proteica o degenerazione retinica, nonché la quantificazione di questi effetti. Questo protocollo descrive in dettaglio queste due tecniche.
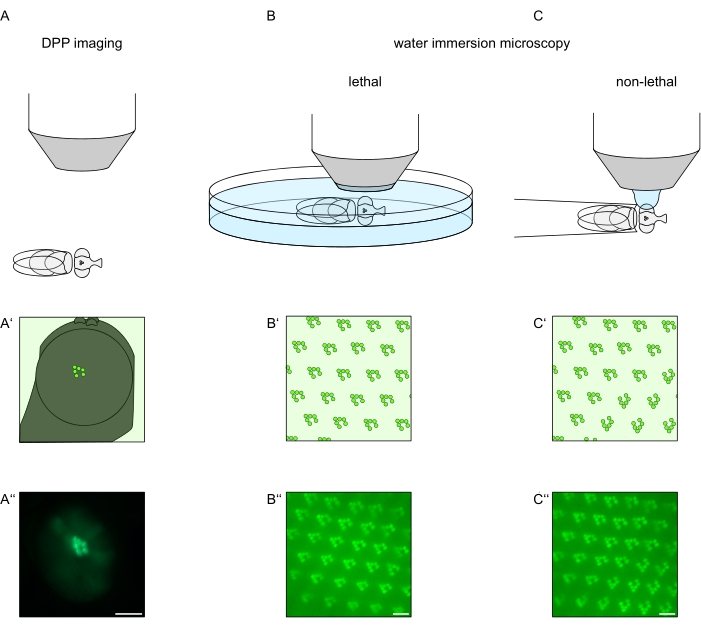
Figura 1: Panoramica delle variazioni di microscopia per l'occhio Drosophila presentate in questo protocollo. Rappresentazioni schematiche e micrografie esemplari di (A-A'') imaging fluorescente a pseudopupila profonda (DPP), (B-B'') microscopia ad immersione in acqua letale di rabdomere fluorescenti e (C-C'') microscopia a goccia d'acqua non letale di rabdomere fluorescenti. Barra di scala (A''): 100 μm. Barre di scala (B''-C''): 10 μm. La figura è stata modificata dal riferimento13. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Considerazioni generali
- Utilizzare stock di Drosophila che esprimono una proteina di fluorescenza permanentemente localizzata in modo rachdomerico per l'analisi morfologica (ad esempio, TRP::eGFP, eGFP::NINAC) e la traslocazione delle proteine per le analisi relative al traffico proteico (ad esempio, TRPL::eGFP, Arr2::eGFP).
- Predeterminare le condizioni di esposizione alla luce delle mosche selezionate per l'approccio sperimentale.
- Per l'adattamento al buio, tenere le mosche in scatole scure per il periodo desiderato a 25 °C. Per un'illuminazione fino a 16 ore negli esperimenti di traslocazione (ad esempio, trPL::eGFP che esprimono mosche), tenere le mosche sotto un tubo fluorescente a temperatura ambiente.
- Negli esperimenti che valutano la degenerazione dei fotorecettori (ad esempio, TRP::eGFP che esprimono mosche), tenere le mosche sotto un tubo fluorescente a 25 °C in un ciclo di luce di 12 ore / 12 ore di buio per un'illuminazione a lungo termine fino a 28 giorni con luce bianca.
- Per illuminare le mosche con luce colorata, utilizzare scatole di plastica trasparente di colore diverso insieme al tubo fluorescente.
- Se vengono utilizzati stock di mosche con occhi pigmentati, invecchiare gli animali precisamente per l'analisi comparativa, poiché la pigmentazione degli occhi può aumentare significativamente con l'età.
NOTA: per l'interpretazione dei dati, è importante notare che esiste una distorsione del segnale dovuta all'effetto ottico di guida d'onda della struttura rabdomerale. Di conseguenza, il segnale di fluorescenza dal rabdomere sarà sempre amplificato in una certa misura nell'imaging DPP e nella microscopia ad immersione in acqua rispetto ai segnali ottenuti dal corpo cellulare. Ciò è osservato in modo più evidente negli occhi pigmentati, dove la fluorescenza dall'esterno dei rabdomere viene assorbita da questi pigmenti ed è di particolare importanza quando si devono rilevare proteine di fusione traslocanti intracellulari. Pertanto, per quanto riguarda i passaggi critici, questo studio considera separatamente le mosche dagli occhi bianchi e rossi. - Per quanto riguarda la microscopia ad immersione in acqua, vengono descritte due varianti. Una variazione letale più veloce e una variazione non letale che consente il recupero per studi successivi.
2. Imaging DPP
- Preparare lo spazio di lavoro con le attrezzature e i reagenti necessari, come mostrato nella Figura 2. Anestetizzare mosche (1-3 giorni) di un genotipo che esprime una proteina di fluorescenza in cellule fotorecettrici con CO2 su un flypad. Selezionare gli animali per l'imaging al microscopio stereo con una sorgente luminosa convenzionale e un basso ingrandimento (ad esempio, 10x).

Figura 2: Area di lavoro di imaging DPP. I materiali necessari sono (A) apparecchio per anestesia CO2 , (B) stereomicroscopio con lampada UV e set di filtri a fluorescenza, (C) sorgente luminosa, (D) telecamera montata al microscopio con (E) software, (F) pennello, (G) cartone nero e (H) fiala volante. Fare clic qui per visualizzare una versione più grande di questa figura.
- Per l'imaging DPP, tenere le mosche selezionate anestetizzate e posizionarne una al centro dell'obiettivo del microscopio su un lato in modo che l'occhio sinistro o destro sia rivolto verso l'obiettivo esattamente radialmente (Figura 3A).
NOTA: Poiché la disposizione ommatidiale delle cellule dei fotorecettori mostra una simmetria speculare sulla linea mediana dorsoventrale, il DPP è meglio osservato leggermente sopra o sotto l'equatore dell'occhio (Figura 3B).
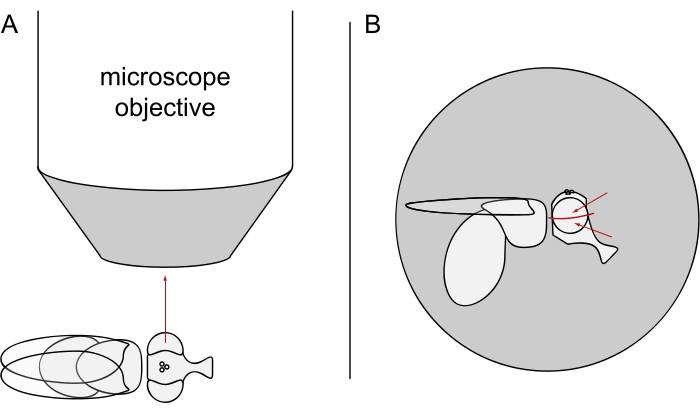
Figura 3: Posizionamento della mosca sotto lo stereomicroscopio per l'imaging DPP. (A) Illustrazione della mosca su un lato con un occhio rivolto verso l'obiettivo del microscopio radialmente. (B) La testa della mosca deve essere leggermente rivolta verso l'alto o verso il basso in modo tale che l'obiettivo si concentri su un punto leggermente sopra o sotto l'equatore dell'occhio come indicato dalle frecce rosse. Fare clic qui per visualizzare una versione più grande di questa figura.
- Aumentare l'ingrandimento per adattarsi all'intero occhio (ad esempio, 100x) e centrare l'ommatidi centrale dell'occhio. Diminuire la profondità di campo del microscopio, ad esempio regolando il diaframma a doppio diaframma su un'impostazione poco profonda (Figura 4A-B').
- Spegnere la sorgente luminosa convenzionale e accendere la lampada UV del microscopio alla massima intensità e selezionare il set di filtri di fluorescenza del microscopio in base alla proteina di fluorescenza espressa negli occhi (Figura 4C-E). Impostare il percorso della luce verso la fotocamera montata al microscopio.
- Utilizzare la funzione di imaging dal vivo all'interno del software per regolare la luminosità dell'immagine su un'impostazione che rileva solo segnali specifici dall'occhio regolando il tempo di esposizione e il valore di guadagno (ad esempio, rispettivamente 80 ms e 12x). Riadattare la messa a fuoco del microscopio "in" l'occhio (sotto la cornea) per generare l'immagine sovrapposta del DPP (Figura 4C-E').
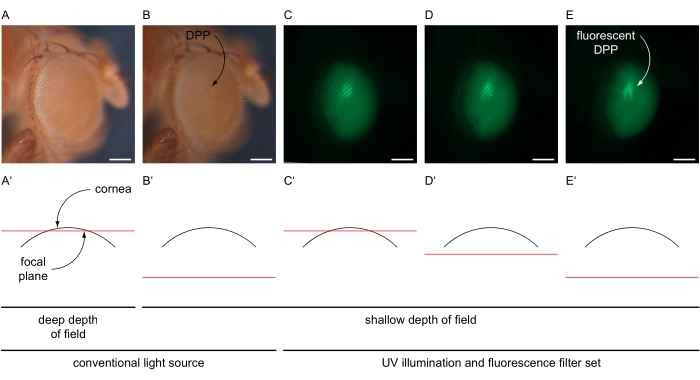
Figura 4: Illustrazione dell'imaging DPP e DPP fluorescente. Immagini esemplari di occhi di Drosophila sotto illuminazione convenzionale e UV con set di filtri GFP, scattate con piani focali variabili illustrati in sezioni trasversali schematiche attraverso l'occhio. (A) Micrografia registrata con impostazioni luminose di una sorgente luminosa convenzionale, tempo di esposizione di 30 ms, guadagno 1x, profondità di campo profonda e piano focale vicino alla superficie della cornea come illustrato in (A'). (B) Micrografia registrata con impostazioni luminose di una sorgente luminosa convenzionale, tempo di esposizione di 30 ms, guadagno 1x, profondità di campo ridotta e piano focale di circa 180 μm sotto la superficie della cornea come illustrato in (B'). DPP indicato. (C-E) Micrografia registrata con impostazioni ad alta intensità della sorgente di luce UV e del set di filtri GFP, tempo di esposizione di 80 ms, guadagno 12x, profondità di campo ridotta e piano focale (C') vicino, (D') leggermente inferiore o (E') circa 180 μm sotto la superficie della cornea. Il DPP fluorescente è indicato con una freccia curva. Barra della scala 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
- Scatta un'istantanea del DPP fluorescente. Riportare il microscopio alla luce visibile e il percorso della luce verso gli oculari. Recuperare l'animale ripreso in una fiala di mosca per ulteriori procedimenti in base al suo fenotipo DPP (ad esempio, croci). Continuare con l'animale successivo nel passaggio 2.2.
3. Microscopia ad immersione in acqua
- Preparazione della mosca
- Preparare lo spazio di lavoro con le attrezzature e i reagenti necessari come mostrato nella Figura 5. Trasferire le mosche con età e condizioni di illuminazione predeterminate in un tubo centrifugo da 15 ml pre-raffreddato e anestetizzare incubandole sul ghiaccio per 15-30 minuti.
NOTA: portare con sé mosche di 1 giorno, adattate al buio come riferimento. Generalmente, le mosche adattate al buio dovrebbero essere trasferite in una ghiacciaia con un coperchio al buio. Le mosche adattate alla luce possono essere trasferite sul ghiaccio nella luce della stanza.
- Preparare lo spazio di lavoro con le attrezzature e i reagenti necessari come mostrato nella Figura 5. Trasferire le mosche con età e condizioni di illuminazione predeterminate in un tubo centrifugo da 15 ml pre-raffreddato e anestetizzare incubandole sul ghiaccio per 15-30 minuti.

Figura 5: Spazio di lavoro per microscopia ad immersione in acqua. I materiali necessari sono: (A) tubo centrifugo da 15 ml, (B) fiocchi di ghiaccio, (C) acqua distillata refrigerata, (D) stereomicroscopio, (E) capsula di Petri, (F) plastilina, (G) vetrino per oggetti, (H) spille per insetti o punte di pipette e bisturi, (I) microscopio a fluorescenza con (J) software. Fare clic qui per visualizzare una versione più grande di questa figura.
- Selezionare la preparazione appropriata tra le due descritte di seguito (3.1.3 variazione letale o 3.1.7 variazione non letale) e ogni volta che viene effettuata la differenziazione tra occhi pigmentati e non pigmentati, seguire i rispettivi passaggi.
- Prepara le mosche per la variazione letale come segue.
- Far aderire un pezzo di plastilina su una diapositiva di un oggetto e un altro pezzo al centro di una capsula di Petri (ad esempio, 94 mm Ø) e tenerli separati per ora. Riempire la capsula di Petri con acqua distillata ghiacciata e alcuni fiocchi di ghiaccio (Figura 6A).
- Metti una mosca anestetizzata con ghiaccio sotto uno stereomicroscopio sopra il vetrino dell'oggetto rivestito di plastilina. Gira la mosca sulla schiena e perfora una spilla da insetto attraverso il centro del torace (Figura 6B). Fissare il perno orizzontalmente sulla diapositiva dell'oggetto rivestito di plastilina e orientare l'occhio sinistro o destro della mosca verso l'alto (Figura 6C).
- Fissare con cura la diapositiva dell'oggetto, con il suo lato privo di plastilina rivolto verso il basso, nella capsula di Petri impedendo la rotazione della mosca. Assicurarsi che l'occhio della mosca sia coperto d'acqua (Figura 6D). Utilizzare un ago di preparazione per rimuovere con cura eventuali bolle d'aria che potrebbero essersi formate intorno all'occhio e procedere immediatamente all'acquisizione delle immagini per ottenere i migliori risultati.
NOTA: un ritardo significativo nell'acquisizione delle immagini provoca il risveglio e i movimenti della mosca che possono portare a immagini sfocate.
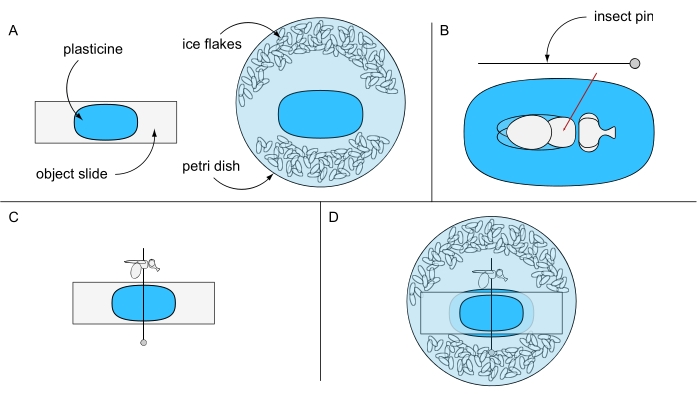
Figura 6: Preparazione per la microscopia ad immersione in acqua letale. Illustrazione di (A) vetrino di oggetti rivestiti di plastilina e piatto di Petri, (B) appuntamento di una mosca attraverso il torace su terreno di plastilina, (C) orientamento della mosca sulla diapositiva dell'oggetto rivestito di plastilina e (D) configurazione sperimentale finale. Fare clic qui per visualizzare una versione più grande di questa figura.
- Preparare le mosche per la variazione non letale come segue.
- Trasferire una mosca anestetizzata con ghiaccio a testa in giù in una punta di pipetta da 200 μL e spingere la mosca verso la punta con attenzione con aria compressa.
- Tagliare la punta della pipetta proprio davanti alla testa usando un bisturi. Usando una pinzetta, spingere con attenzione la mosca a pochi millimetri di distanza dalla punta. Tagliare di nuovo la punta della pipetta e spingere la mosca indietro verso la punta con aria compressa in modo che solo la testa della mosca sporga dalla punta della pipetta.
- Far aderire un pezzo di plastilina su una diapositiva di un oggetto e premere la punta della pipetta in modo che l'occhio sinistro o destro della mosca sia rivolto verso l'alto (Figura 7A). Subito prima dell'acquisizione dell'immagine, utilizzare una pipetta da laboratorio per far aderire una grande goccia di acqua refrigerata sul lato inferiore di un obiettivo di immersione in acqua (Figura 7B). Procedere immediatamente all'acquisizione delle immagini per ottenere i migliori risultati.
NOTA: un ritardo significativo nell'acquisizione delle immagini provoca il risveglio e i movimenti della mosca che possono portare a immagini sfocate.
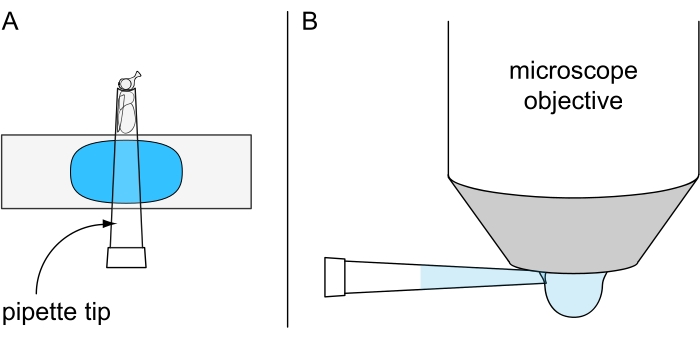
Figura 7: Preparazione per la microscopia a goccia d'acqua non letale. Illustrazione di (A) una mosca anestetizzata a freddo fissata all'interno di una punta di pipetta da 200 μL montata su un vetrino rivestito di plastilina e (B) l'applicazione di goccia d'acqua refrigerata sul lato inferiore dell'obiettivo di immersione in acqua. Fare clic qui per visualizzare una versione più grande di questa figura.
- Acquisizione di immagini
- Posizionare con attenzione la capsula di Petri (punto 3.1.3) o il vetrino dell'oggetto (punto 3.1.7) con la mosca preparata sullo stadio del microscopio e selezionare un obiettivo di immersione in acqua.
- Abbassare manualmente l'obiettivo di immersione in acqua fino a quando non entra in contatto con la superficie dell'acqua (punto 3.1.3) o l'occhio della mosca tocca la goccia (punto 3.1.7) (Figura 8A,B).
- Accendere la lampada UV del microscopio e selezionare il set di filtri appropriato. Usa gli oculari per posizionare la mosca sotto l'obiettivo e focalizzare il microscopio sulla superficie dell'occhio.
- Cambiare il percorso della luce verso la fotocamera del microscopio e generare un'immagine dal vivo nel software corrispondente. Riregolare la messa a fuoco per la fotocamera e valutare l'orientamento dell'occhio, considerando che l'occhio deve affrontare l'obiettivo del microscopio radialmente come illustrato in modo più dettagliato nella Figura 8C-E.
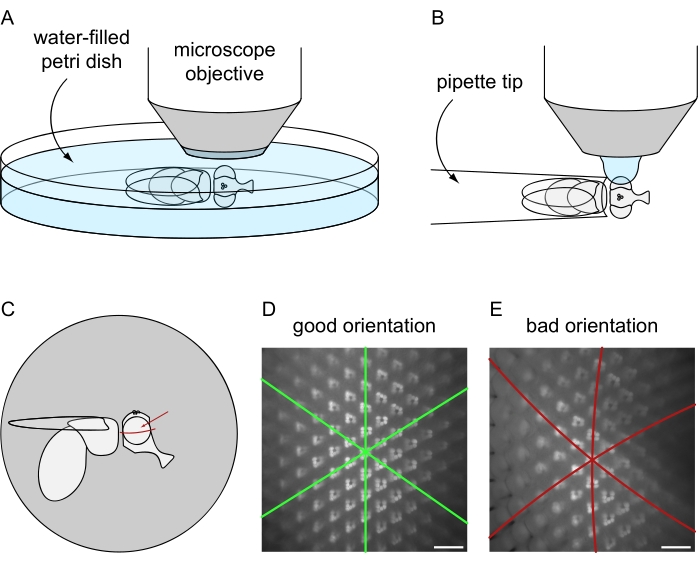
Figura 8: Posizionamento della mosca sotto il microscopio a fluorescenza per l'imaging ad immersione in acqua. Impostazione e orientamento finale per l'acquisizione delle immagini utilizzando i protocolli di preparazione alla mosca (A) letale o (B) non letale. (C) Illustrazione dell'orientamento della mosca per ottenere i migliori risultati delle immagini al microscopio ad immersione in acqua. Il punto ideale per mettere a fuoco l'occhio non è il centro esatto rispetto agli assi anteriore/posteriore e dorsale/ventrale ma è leggermente al di sopra dell'equatore dell'occhio, come indicato dalla freccia rossa. (D) Esempio di immagine di immersione in acqua per un occhio perfettamente posizionato. Tutti e tre gli assi di simmetria della piastrellatura ommatidiale esagonale appaiono come linee rette e la quantità massima di ommatidi può essere messa a fuoco allo stesso tempo. (E) Esempio di immagine di immersione in acqua di un occhio posizionato in modo improprio. L'immagine contiene assi curvi e una profondità di campo ridotta. Barra della scala: 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
- Utilizzare la LUT appropriata (tabella di ricerca) all'interno del software di imaging per rilevare la sovrasaturazione (indicata come pixel rossi).
- Nel caso di mosche non pigmentate, regolare il tempo di esposizione in modo che i pixel più luminosi siano appena al di sotto del limite di saturazione per ogni immagine.
- Nel caso di mosche pigmentate e della variazione letale, regolare il tempo di esposizione in modo tale che tutti i pixel più luminosi siano appena al di sotto del limite di saturazione in almeno cinque singole mosche di 1 giorno, adattate al buio. Applicare il tempo medio di esposizione calcolato a tutte le altre condizioni sperimentali (genotipi, condizioni di illuminazione, punti temporali, ecc.).
- Nel caso di mosche pigmentate (ad esempio, occhi rossi) e la variazione non letale, regolare il tempo di esposizione in modo tale che tutti i pixel più luminosi siano appena al di sotto del limite di saturazione per ogni mosca di 1 giorno, adattata al buio individualmente. Applicare questo tempo di esposizione a tutte le altre condizioni sperimentali (condizioni di illuminazione, punti temporali, ecc.) di questa mosca.
- Registrare un'immagine e salvarla come file raw per archiviare tutti i metadati corrispondenti della registrazione. Esportare l'immagine in un formato .tif per la seguente quantificazione.
NOTA: Nel caso della variazione non letale, illuminare le mosche per 5 minuti con luce rossa (ad esempio, 630 nm) immediatamente dopo l'acquisizione dell'immagine, se sono destinate ad essere utilizzate per ulteriori esperimenti. La luce rossa disattiva la cascata di fototrasduzione che è stata attivata eccessivamente da un'intensa luce a onde corte durante l'acquisizione dell'immagine.
- Analisi dei dati e quantificazione della fluorescenza relativa eGFP nei rabdomere delle micrografie ad immersione in acqua
- Scarica, installa ed esegui il software ImageJ/Fiji.
- Regola le impostazioni di ImageJ facendo clic su Analizza > Imposta misurazioni... e seleziona solo la casella Valore grigio medio. Importa un'immagine .tif cliccando su File > Apri... o trascinando e rilasciando. Scegli una regione rappresentativa dell'immagine a fuoco e ingrandiscila al 200%-300% premendo ripetutamente Ctrl e + insieme .
- Selezionate lo strumento Ovale e, mentre premete il tasto Maiusc , generate una selezione circolare nell'immagine significativamente più piccola di un rabdomere fluorescente. Prima di rilasciare il pulsante del mouse, cerca la dimensione esatta visualizzata sotto la barra degli strumenti nella finestra principale di ImageJ. Utilizzare la stessa dimensione della selezione circolare per tutte le analisi.
NOTA: la dimensione esatta della selezione circolare in pixel o micron dipende dalla configurazione specifica. Usa un cerchio di circa 1/3 o 1/4 del diametro rabdomerale delle mosche di controllo di 1 giorno, adattate al buio. - Spostare la selezione circolare facendo clic con il mouse su di essa e trascinandola oppure premendo i tasti freccia sulla tastiera.
- Per misurare le intensità di fluorescenza all'interno della selezione circolare, spostare il cerchio sul primo rabdomere (r1) e fare clic su Analizza > Misura o utilizzare la scorciatoia Ctrl + M. Verrà visualizzata una finestra Dei risultati che elenca il valore grigio misurato.
- Continuare con le misurazioni di r2-r6 come misurazioni ripetute e una misurazione del segnale di fondo (b). Nel caso di mosche non pigmentate, effettuare ulteriori misurazioni delle corrispondenti aree del corpo cellulare (c1-c6) (Figura 9).
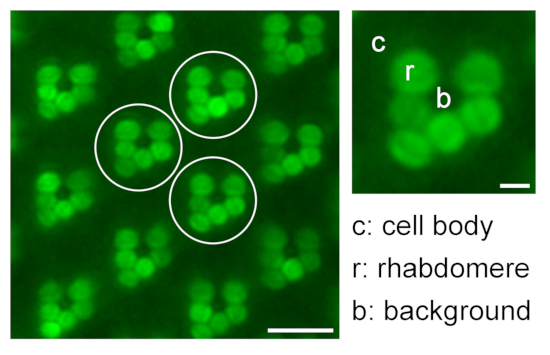
Figura 9: Quantificazione della fluorescenza rabdomerale relativa per studi di traslocazione. Un'illustrazione riguardante la quantificazione della fluorescenza relativa eGFP nei rabdomere misurando l'intensità di fluorescenza del rabdomere (r), del corpo cellulare (c) e dello sfondo (b) di tre diverse ommatidi rappresentative (cerchi bianchi) in un'immagine di microscopia ad immersione in acqua; barra della scala: 10 μm. Un ommatidium ingrandito è mostrato a destra; barra della scala: 2 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
- Ripetere i passaggi 3.3.5 e 3.3.6 per altri due ommatidi, ottenendo tre repliche tecniche. Contrassegnare l'ommatidi analizzata utilizzando lo strumento Matita e salvare questa immagine per la documentazione.
- Selezionare e copiare i valori di grigio misurati dalla finestra Risultato e incollarli nel software del foglio di calcolo per ulteriori calcoli. Ordina i valori di intensità di fluorescenza in base alla loro origine nelle categorie rabdomere (r), corpo cellulare (c) e sfondo (b). Calcola l'intensità media di ogni categoria (Ir, Ic, Ib).
- Calcolare la quantità relativa di eGFP presente nel rabdomere (R), utilizzando la seguente formula (1) per i non pigmentati e formula (2) per gli occhi pigmentati:
 (1)
(1) (2)
(2) - Continuare con l'immagine successiva nel passaggio 3.3.3. Si consiglia di utilizzare le immagini di almeno cinque individui di ciascun gruppo sperimentale come repliche biologiche per ottenere una misurazione affidabile.
- Analisi dei dati e quantificazione della morfologia oculare mediante fluorescenza eGFP in micrografie ad immersione in acqua
- Scarica, installa ed esegui il software ImageJ/Fiji. Importa un'immagine .tif facendo clic su File > Apri... o trascinando e rilasciando. Scegli tre ommatidi adiacenti in una regione rappresentativa dell'immagine a fuoco.
- Valutare i 18 rhabdomere della selezione individualmente in base alla loro intensità eGFP, nitidezza dei bordi e contrasto rispetto al segnale di sfondo circostante per generare un indice di degenerazione. Punteggiare i rabdomeri chiaramente visibili con un valore di 2, i rabdomeri debolmente visibili con un valore di 1 e i rabdomeri assenti con un valore di 0 (Figura 10).
NOTA: Questo modo di quantificazione si traduce in un punteggio di 36 per gli occhi completamente intatti e un punteggio di 0 per gli occhi completamente degenerati. Si consiglia di impostare il punteggio da 36 a 100% sull'indice di degenerazione.

Figura 10: Quantificazione tramite valutazione del rabdomere per studi di degenerazione. Un'illustrazione riguardante la quantificazione della morfologia oculare segnando i rabdomere di tre diversi ommatidi rappresentativi (cerchi bianchi) in un'immagine al microscopio ad immersione in acqua con valori di 2 (chiaramente visibile; cerchio blu), 1 (debolmente visibile; cerchio arancione) o 0 (assente; cerchio rosso). Barra di scala: 10 μm. Un ommatidium ingrandito è mostrato a destra; barra della scala: 2 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
- Aprire l'immagine successiva e continuare con il passaggio 3.4.2. Si raccomanda di utilizzare le immagini di almeno otto individui di ciascun gruppo sperimentale come repliche biologiche per ottenere una misurazione affidabile.
NOTA: poiché questo metodo di quantificazione è meno oggettivo del metodo per quantificare la traslocazione in base all'intensità di fluorescenza, il numero raccomandato di repliche è più alto.
Access restricted. Please log in or start a trial to view this content.
Risultati
Sono state generate mosche transgeniche drosophila che esprimono una proteina di fusione TRPL::eGFP sotto il controllo del promotore della rodopsina 1. In queste mosche, TRPL::eGFP è espresso nelle cellule fotorecettrici R1-6 dell'occhio composto e mostra una localizzazione dipendente dall'illuminazione. Quando le mosche sono tenute al buio, TRPL::eGFP è incorporato nei rabdomeri esterni. Dopo l'illuminazione per diverse ore, TRPL trasloca nel corpo cellulare dove viene immagazzinato in un compartimento arricc...
Access restricted. Please log in or start a trial to view this content.
Discussione
L'applicabilità delle proteine di fluorescenza e la semplicità dello screening mediante imaging DPP e microscopia ad immersione in acqua retinica hanno dimostrato di avere successo in molti gruppi12. Strategie simili a quelle qui presentate sono state utilizzate in diversi screening genetici per rilevare difetti nei livelli di espressione della rodopsina, nell'omeostasi, nell'organizzazione retinica o nell'integrità cellulare con l'aiuto di Rh1::eGFP 17,18,19,20,21....
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Vorremmo ringraziare i nostri studenti ricercatori nel corso degli anni. In particolare, Nina Meyer, Sibylle Mayer, Juliane Kaim e Laura Jaggy, i cui dati sono stati utilizzati in questo protocollo come risultati rappresentativi. La ricerca del nostro gruppo qui presentata è stata finanziata da sovvenzioni della Deutsche Forschungsgemeinschaft (Hu 839/2-4, Hu 839/7-1) ad Armin Huber.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Greiner Bio-One | 188271 | |
| CO2 anaesthesia fly pad | Flystuff | 59-172 | |
| Cold light lamp (KL 1500 LCD) | Zeiss | ||
| Fiji/ImageJ | NIH | ||
| Fluorescence microscope with UV lamp, camera, filter set and software (AxioImager.Z1m, Axiocam 530 mono, 38 HE, ZEN2 blue edition) | Zeiss | ||
| Fluorescent tube (Lumilux T8, L 30W/840, 4000 K, G13) [1750 Lux, Ee470nm = 298 µW cm-2, Ee590nm = 215 µW cm-2] and [760 Lux, Ee470nm = 173 µW cm-2, Ee590nm = 147 µW cm-2] | Osram | 4050300518039 | |
| Laboratory pipette (20-200 µL) | Eppendorf | ||
| Object slide | Roth | 0656.1 | |
| Petri dish (94 mm) | Greiner Bio-One | 633102 | |
| Pipette tips (200 µL) | Labsolute | 7695844 | |
| Plasticine (Blu-Tack) | Bostik | 30811745 | |
| Stereo microscope (SMZ445) | Nikon | ||
| Stereo microscope with UV lamp, camera, filer set and software (MZ16F, MC170 HD, GFP3, LAS 4.12) | Leica |
Riferimenti
- Wang, X., Huang, T., Bu, G., Xu, H. Dysregulation of protein trafficking in neurodegeneration. Molecular Neurodegeneration. 9. 9, 31(2014).
- Schopf, K., Huber, A. Membrane protein trafficking in Drosophila photoreceptor cells. European Journal of Cell Biology. 96 (5), 391-401 (2017).
- Hardie, R. C. The photoreceptor array of the dipteran retina. Trends in Neurosciences. 9, 419-423 (1986).
- Wang, T., Montell, C. Phototransduction and retinal degeneration in Drosophila. Pflügers Archiv - European Journal of Physiology. 454 (5), 821-847 (2007).
- Xiong, B., Bellen, H. J. Rhodopsin homeostasis and retinal degeneration: lessons from the fly. Trends in Neurosciences. 36 (11), 652-660 (2013).
- Bähner, M., et al. Light-regulated subcellular translocation of Drosophila TRPL channels induces long-term adaptation and modifies the light-induced current. Neuron. 34 (1), 83-93 (2002).
- Cronin, M. A., Lieu, M. -H., Tsunoda, S. Two stages of light-dependent TRPL-channel translocation in Drosophila photoreceptors. Journal of Cell Science. 119, 2935-2944 (2006).
- Meyer, N., Joel-Almagor, T., Frechter, S., Minke, B., Huber, A. Subcellular translocation of the eGFP-tagged TRPL channel in Drosophila photoreceptors requires activation of the phototransduction cascade. Journal of Cell Science. 119, Pt 12 2592-2603 (2006).
- Oberegelsbacher, C., Schneidler, C., Voolstra, O., Cerny, A., Huber, A. The Drosophila TRPL ion channel shares a Rab-dependent translocation pathway with rhodopsin. European Journal of Cell Biology. 90 (8), 620-630 (2011).
- Wagner, K., Smylla, T. K., Lampe, M., Krieg, J., Huber, A. Phospholipase D and retromer promote recycling of TRPL ion channel via the endoplasmic reticulum. Traffic. , (2021).
- Franceschini, N., Kirschfeld, K. Les phénoménes de pseudopupille dans l'oeil compose de Drosophila. Kybernetik. 9 (5), 159-182 (1971).
- Pichaud, F., Desplan, C. A new visualization approach for identifying mutations that affect differentiation and organization of the Drosophila ommatidia. Development. 128 (6), 815-826 (2001).
- Smylla, T. K., Wagner, K., Huber, A. Application of fluorescent proteins for functional dissection of the Drosophila visual system. International Journal of Molecular Sciences. 22 (16), (2021).
- Meyer, N. Mechanisms of the light-dependent translocation of the ion channel TRPL in the photoreceptors of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer. nat. University of Karlsruhe (TH). , Karlsruhe. (2005).
- Meyer, N., Oberegelsbacher, C., Dürr, T. D., Schäfer, A., Huber, A. An eGFP-based genetic screen for defects in light-triggered subcelluar translocation of the Drosophila photoreceptor channel TRPL. Fly. 2 (1), 36-46 (2008).
- Zheng, L., Carthew, R. W. Lola regulates cell fate by antagonizing Notch induction in the Drosophila eye. Mechanisms of Development. 125 (1-2), 18-29 (2008).
- Hibbard, K. L., O'Tousa, J. E. A role for the cytoplasmic DEAD box helicase Dbp21E2 in rhodopsin maturation and photoreceptor viability. Journal of Neurogenetics. 26 (2), 177-188 (2012).
- Huang, Y., Xie, J., Wang, T. A Fluorescence-based genetic screen to study retinal degeneration in Drosophila. PloS One. 10 (12), 0144925(2015).
- Zhao, H., Wang, J., Wang, T. The V-ATPase V1 subunit A1 is required for rhodopsin anterograde trafficking in Drosophila. Molecular Biology of the Cell. 29 (13), 1640-1651 (2018).
- Xiong, L., et al. ER complex proteins are required for rhodopsin biosynthesis and photoreceptor survival in Drosophila and mice. Cell Death and Differentiation. 27 (2), 646-661 (2020).
- Zhao, H., Wang, T. PE homeostasis rebalanced through mitochondria-ER lipid exchange prevents retinal degeneration in Drosophila. PLoS Genetics. 16 (10), 1009070(2020).
- Cerny, A. C., et al. The GTP- and phospholipid-binding protein TTD14 regulates trafficking of the TRPL ion channel in Drosophila photoreceptor cells. PLoS Genetics. 11 (10), 1005578(2015).
- Richter, D. Structural and functional analyses of TRP ion channels in the photoreceptor cells of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer, nat. University of Karlsruhe (TH). , Karlsruhe. (2007).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Developmental Biology. 351 (1), 128-134 (2011).
- Hardie, R. C., Liu, C. -H., Randall, A. S., Sengupta, S. In vivo tracking of phosphoinositides in Drosophila photoreceptors. Journal of Cell Science. 128 (23), 4328-4340 (2015).
- Chakrabarti, P., et al. A dPIP5K dependent pool of phosphatidylinositol 4,5 bisphosphate (PIP2) is required for G-protein coupled signal transduction in Drosophila photoreceptors. PLoS Genetics. 11 (1), 1004948(2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon