É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma abordagem baseada em espectrometria de massa para identificar fosfofamina fosfates e seus interacttores
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para o enriquecimento de fosfatrofames fosfoproteínas e suas proteínas interativas a partir de células e tecidos e sua identificação e quantificação por proteômica baseada em espectrometria de massa.
Resumo
A maioria dos processos celulares são regulados pela fosforilação dinâmica da proteína. Mais de três quartos das proteínas são fosfoiladas, e fosfoprotetases fosfoproteínas (PPPs) coordenam mais de 90% de toda a desfosforilação serina/threonina celular. A desregulamentação da fosforilação proteica tem sido implicada na fisiopatologia de várias doenças, incluindo câncer e neurodegeneração. Apesar de sua atividade generalizada, os mecanismos moleculares que controlam as PPPs e os controlados pelas PPPs são pouco caracterizados. Aqui, uma abordagem proteômica denominada contas inibidoras de fosfattase e espectrometria de massa (PIB-MS) é descrita para identificar e quantificar PPPs, suas modificações pós-translacionais e seus interagentes em apenas 12 h usando qualquer linha celular ou tecido. O PIB-MS utiliza um inibidor de PPP não seletivo, microcistina-LR (MCLR), imobilizado em contas de sepharose para capturar e enriquecer PPPs endógenos e suas proteínas associadas (denominada PPPome). Este método não requer a expressão exógena das versões marcadas de PPPs ou o uso de anticorpos específicos. O PIB-MS oferece uma maneira inovadora de estudar as PPPs evolutivamente conservadas e expandir nossa compreensão atual da sinalização de desfosforilação.
Introdução
A fosforilação proteica controla a maioria dos processos celulares, incluindo, mas não se limitando à resposta a danos no DNA, sinalização de fator de crescimento e a passagem pela mitose 1,2,3. Em células de mamíferos, a maioria das proteínas são fosfoiladas em um ou mais resíduos de soro, threonina ou tyrosina em algum momento, com fosfoserinas e fosfothreoninas compreendendo aproximadamente 98% de todos os locais de fosforilação 2,3. Embora as cinesases tenham sido extensivamente estudadas na sinalização celular, o papel das PPPs na regulação de processos celulares dinâmicos ainda está surgindo.
A dinâmica da fosforilação é controlada pela interação dinâmica entre quinases e fosfates. Nas células mamíferas, existem mais de 400 quinases proteicas que catalisam a fosforilação serina/threonina. Mais de 90% desses locais são desfosforilados por fosfoproteínas (PPPs), uma pequena família de enzimas que consiste em PP1, PP2A, PP2B, PP4-7, PPT e PPZ 2,3. PP1 e PP2A são responsáveis pela maioria da fosfoserina e fosfotoreonina desfosforilação dentro de uma célula 2,3,4. A notável diferença de número entre quinases e fosfattases e a falta de especificidade das subunidades catalíticas PPP in vitro levaram à crença de que as quinases são o principal determinante da fosforilação 2,3. No entanto, vários estudos mostraram fosfatidades para estabelecer especificidade de substrato através da formação de holoenzimas multimédicos 5,6,7,8,9. Por exemplo, o PP1 é um heterodimer que consiste em uma subunidade catalítica e, em um dado momento, uma das mais de 150 subunidades regulatórias 6,7,8. Por outro lado, PP2A é um heterotrimer que é formado por andaimes (A), um regulador (B) e uma subunidade catalítica (C) 2,3,9. Existem quatro famílias distintas de subunidades regulatórias PP2A (B55, B56, PR72 e striatina), cada uma com múltiplos genes, variantes de emenda e padrões de localização 2,3,9. A natureza multimérica das PPPs preenche a lacuna no número de quinases e subunidades catalíticas PPP. No entanto, cria desafios analíticos para o estudo da sinalização PPP. Para analisar de forma abrangente a sinalização PPP, é fundamental investigar as várias holoenzimias dentro de uma célula ou tecido. Grandes avanços foram feitos no estudo do kinome humano através do uso de contas inibidoras de quinase, denominadas contas inibidoras multiplex ou kinobeads, uma estratégia proteômica química onde os inibidores da quinase são imobilizados em contas e espectrometria de massa é usado para identificar quinases enriquecidas e seus interageres 10,11,12,13.
Estabelecemos uma abordagem semelhante para estudar biologia ppp. Esta técnica envolve a captura de afinidade de subunidades catalíticas PPP utilizando contas com um inibidor ppp imobilizado e não seletivo chamado microcistina-LR (MCLR) chamado de contas inibidoras de fosfatase (PIBs)14,15. Ao contrário de outros métodos que requerem a marcação endógena ou expressão de subunidades exógenas de PPP que poderiam alterar a atividade ou localização da proteína, o PIB-MS permite o enriquecimento de subunidades catatróticas PPP endógenas, suas subunidades regulatórias e andaimes associadas, e proteínas interativas (denominadas PPPome) a partir de células e tecidos em um determinado ponto de tempo ou sob condições específicas de tratamento. A MCLR inibe pp1, PP2A, PP4-6, PPT e PPZ em concentrações de nanomolar, tornando os PIBs altamente eficazes no enriquecimento para o PPPome16. Este método pode ser dimensionado para uso em qualquer material inicial de células para amostras clínicas. Aqui, descrevemos em detalhes o uso de PIBs e espectrometria de massa (PIB-MS) para capturar, identificar e quantificar eficientemente o PPPome endógeno e seus estados de modificação.

Figura 1: Resumo visual do protocolo PIB-MS. Em um experimento PIB-MS, as amostras podem ser obtidas de várias formas, desde células até tumores. A amostra é coletada, líseda e homogeneizada antes do enriquecimento de PPP. Para enriquecer para PPPs, o lysate é incubado com PIBs com ou sem um inibidor de PPP, como o MCLR. Os PIBs são então lavados, e as PPPs são elucidadas em condições de desnaturação. As amostras são preparadas para análise de espectrometria de massa pela remoção de detergentes através do enriquecimento de proteína SP3, digestão tripptica e dessalção. As amostras podem então ser rotuladas opcionalmente de TMT antes da análise de espectrometria de massa. Clique aqui para ver uma versão maior desta figura.
O PIB-MS envolve lise e esclarecimento de células ou tecidos, incubação do lysate com PIBs, elução e análise do elunato por meio de manchas ocidentais ou abordagens baseadas em espectrometria de massa (Figura 1). A adição de MCLR gratuito pode ser usada como um controle para distinguir aglutinantes PIB específicos de interagidores não específicos. Para a maioria das aplicações, uma abordagem sem rótulos pode ser usada para identificar diretamente proteínas em eluatos. Nos casos em que é necessária maior precisão na quantificação ou identificação de espécies de baixa abundância, o processamento posterior com rotulagem tandem mass tag (TMT) pode ser usado para aumentar a cobertura e diminuir a entrada.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: A geração de PIBs é feita conforme descrito por Moorhead et al., onde 1 mg de microcistina e cerca de 6 mL de sepharose são acoplado para gerar PIBs com capacidade de ligação de até 5 mg/mL17.
1. Preparação da amostra
NOTA: Uma quantidade inicial típica para PIB-MS é de 1 mg de proteína total por condição. Para este experimento, aproximadamente 2,5 x 106 células HeLa foram utilizadas para extrair 1 mg de proteína. Este cálculo deve ser realizado para cada linha celular ou tecido que está sendo utilizado em um experimento18. Se a amostra for limitada e 1 mg não puder ser obtida, a quantidade de entrada pode ser reduzida com uma pequena perda de detecção de subunidade ppp. Alternativamente, a rotulagem TMT pode ser empregada para permitir a mistura de todas as condições em uma amostra, aumentando a sensibilidade da detecção, como mostrado no Passo 9.
- Coletar amostras de tecido ou pelotas de células. Para pelotas celulares, colete células por centrifugação a 277 x g por 2 min à temperatura ambiente (RT), remova a mídia e lave as células com 5 mL de soro fisiológico tamponado de fosfato (PBS). As pelotas celulares podem ser armazenadas a -80 °C por vários meses.
- Prepare o tampão de lise (500 mM NaCl, 50 mM Tris-HCl pH 7.5, 0,5% Triton X-100 (vol/vol), 5 mM beta-glicerophosphoric ácido disodórico desodium pentahidrato de sal, 1:500 (vol/vol) inibidor de protease III) e mantenha no gelo. Faça o suficiente para lise e lave todas as amostras. Se começar com 1 mg de proteína por condição, faça cerca de 3 mL de tampão por amostra para lise e lava.
NOTA: O tampão de lise observado nesta etapa é uma solução de detergente suave, que pode não ser suficiente para solubilizar a membrana insolúvel ou proteínas associadas à citoesqueletal. Outros detergentes poderiam ser explorados para melhorar a solubilização. Uma série de concentrações de NaCl e Triton-X-100 foram testadas, e as concentrações acima foram consideradas ótimas para baixa origem e alta ligação subunita fosfatase. - Adicione o tampão de lise resfriado às amostras. Para lise de 1 mg de proteína, use 1 mL de tampão. Se a amostra estiver congelada, adicione o tampão de lise e deixe a amostra descongelar no gelo no buffer.
- Para as células, homogeneize as amostras através da sonificação, mantendo as células no gelo entre pulsos. Sonicar as amostras a 15% de amplitude com três pulsos de 15 s. Isso pode variar de acordo com o sonicator utilizado (ver Tabela de Materiais).
- Para tecidos, homogeneize as amostras primeiro usando um moedor de tecido Dounce para moer o tecido até liquefeito antes de sonicar como descrito na Etapa 1.3.1.
- Esclareça a amostra homogeneizada de detritos insolúveis por centrifugação a 21.130 x g por 15 min a 4 °C. Em seguida, sem perturbar as pelotas ou lipídios formados que foram coletados na lateral dos tubos, transfira os lises para novos tubos. Remova 100 μL da amostra de pré-enriquecimento do lise esclarecido se desejar e armazene a -20 °C. Certifique-se de manter os lises no gelo.
- Determine o teor total de proteínas em cada amostra realizando um ensaio de quantificação proteica, como um ensaio de ácido bicinchonínico (BCA), em uma pequena alíquota de cada amostra, de acordo com as instruções do fabricante. Certifique-se de que os lises são mantidos no gelo durante o ensaio BCA.
- Após a realização do ensaio BCA, transfira uma quantidade equivalente de proteína para novos tubos e dilua com tampão de lise para garantir que cada tubo tenha a mesma concentração de proteína (por exemplo, 1 mg/mL). Se o experimento exigir um controle inibidor de PPP, prepare duas alíquotas de cada amostra que tenham o mesmo teor de proteína e prossiga para a Etapa 1.7. Se não forem necessários controles inibidores de PPP, pule para o Passo 2. Certifique-se de que as amostras sejam mantidas no gelo.
NOTA: É fundamental que concentrações de proteínas iguais sejam utilizadas por condição em uma análise PIB-MS. Os controles inibidores de PPP são usados para distinguir aglutinantes específicos aos PIBs do fundo não específico. - Para o controle inibidor de PPP, trate uma amostra com MCLR livre (1 μM) e a outra com um volume igual de DMSO como controle. Vórtice as amostras suavemente e incuba-as no gelo por 15 minutos.
NOTA: O tratamento mclr dos lysates bloqueia a ligação de subunidades catalíticas ppp, mas não proteínas que se ligam não especificamente aos PIBs nas amostras. Tenha cuidado ao manusear a MCLR, pois ela é tóxica. Consulte as precauções de manuseio na Tabela de Materiais.
2. Preparação de PIBs
- Determine a quantidade de PIBs necessária para o experimento. Para 1 mg de proteína, podem ser obtidos 1-3 μg de PPPs e proteínas interativas; use pelo menos 10 μL de resina de PIBs sólido por amostra para minimizar a perda de contas. A capacidade de ligação dos PIBs é de 3-5 mg/mL14,17.
- Transfira a quantidade apropriada de PIBs para um tubo de 1,5 mL e lave-os 3x com 0,5 mL de tampão de lise suavemente vórtice e, em seguida, centrifugação a 376 x g para 30 s em RT entre as lavagens. Evite a pipetação das contas ao remover o tampão de lise entre as lavagens.
- Faça uma solução de 50% PIB/buffer (vol/vol) adicionando uma quantidade apropriada de tampão de lise aos PIBs lavados. Cano suavemente para cima e para baixo e gire a ponta da tubulação no chorume para resuspensar os PIBs.
- Transfira 20 μL do chorume para um novo tubo de 1,5 mL já contendo 0,5 mL de tampão de lise. O tampão de lise no tubo ajuda a expulsar as contas da ponta da tubulação. Faça isso até que haja tubos suficientes contendo uma quantidade igual de PIBs para cada amostra.
- Gire os tubos a 376 x g para 30 s em RT. Certifique-se de que todos os tubos contenham uma quantidade igual de resina de contas. Descarte qualquer supernasciente, deixando apenas resina sólida e um máximo de 50 μL de tampão de lise em cada tubo.
3. Incubação de PIBs com lises
- Transfira os lises do passo 1.6. ou Passo 1.7. para o tubo devidamente rotulado contendo PIBs a partir da etapa 2. Gire o lysate a 8 rpm com os PIBs por 1h a 4 °C.
4. Lavagem de PIBs
- Centrifugar os PIBs a 376 x g para 30 s a 4 °C para coletar as contas. Remova e descarte o supernascer, economizando uma alíquota de 100 μL para análise pós-enriquecimento, se desejar.
- Lave os PIBs 3x adicionando 0,5 mL de tampão de lise às contas, invertendo os tubos (vórtice não recomendado), coletando as contas por centrifugação a 376 x g por 30 s a 4 °C, e removendo o tampão de lise das contas assentadas, tomando cuidado para não interromper a pelota de contas.
5. Elução de PPPs dos PIBs
- Após a lavagem final, remova o máximo possível do tampão de lise sem que o tubo seja fornecido. Faça o buffer de eluição contendo 2% de SDS (vol/vol) e elute as PPPs dos PIBs adicionando volume suficiente de tampão de eluição para ser 4x-5x o volume de PIBs. Por exemplo, se forem utilizados 10 μL de PIBs, use 50 μL de buffer de eluição. Incubar os PIBs com o tampão de eluição a 65 °C por 1h para elutar as PPPs dos PIBs.
- Após a eluição, colete o eluato centrifugando os tubos a 376 x g por 30 s em RT e esbofiando o eluado em um tubo separado, tomando cuidado para não transferir nenhum PIBs. Use o eluato para análise de manchas ocidentais ou para análises de espectrometria de massa. Os eluatos podem ser armazenados a -20 °C por até vários meses.
- Para regenerar os PIBs para uso posterior, incubar as contas em 2% de SDS (vol/vol), girando a 8 rpm em RT por 1h. Lave-os 3x-5x em 25 mM Tris-HCl (pH 7.5) com rotação por 30 minutos por lavagem. Depois de todas as lavagens, armazene PIBs em 25 mM Tris-HCl (pH 7.5) tampão de armazenamento com azida de sódio (0,05% wt/vol).
- Analise os eluatos por manchas ocidentais ou espectrometria de massa. A análise de espectrometria de massa dos elunatos PIB é descrita abaixo.
6. Remoção de detergentes
NOTA: Várias abordagens podem ser usadas para remover detergente de amostras de elunato para análise de MS. Descobrimos que a preparação amostral (SP3), de um único pote, aprimorada em fases sólidas, descrita por Hughes et al., funciona bem19.
- Adicione 0,5 μL de contas SP3 a 50 μL de elunato a partir da etapa 5.2 acima. Use contas SP3 a uma proporção de 10:1 (μg:μg) ou pelo menos 0,5 μg/μL (a solução de estoque é de 50 μg/μL). Bata suavemente as contas e eluia.
- Adicione um volume de eluição de 100% de etanol à mistura de elunato de contas (por exemplo, se for utilizado 50 μL do eluato, utilize 50 μL de 100% de etanol). Incubar as amostras por 5 min em um termomixer definido para agitar a 1000 rpm a 24 °C.
- Para coletar todas as contas, coloque-as em um rack de tubo magnético. Uma vez que as contas coletam, descarte o supernasciente e lave as contas com 0,5 mL de 80% de etanol (vol/vol) 3x. Para lavar, resuspenque as contas por vórtice, colete as contas colocando-as no rack do tubo magnético e descarte o sobrenatante entre as lavagens.
- Lave as contas mais uma vez com 0,5 mL de acetonitrilo 100% (ACN) para remover todos os traços de etanol, removendo o máximo de ACN possível.
7. Digestão de proteínas
- Faça uma diluição de 1:100 de trippsina (concentração final de 0,004 μg/μL) em 166 mM HEPES (pH 8.5) e adicione 30 μL desta solução de trippsina a cada tubo com as contas, ressuscontínuas por vórtice.
- Incubar a mistura de contas sp3-trypsin no termomixer a 1000 rpm a 37 °C por 5h ou durante a noite a 30 °C. Coloque os tubos no rack magnético para coletar as contas e remova os digestivos para novos tubos.
- Para análise sem rótulos, sacie a reação adicionando ácido trifluoroacético de 20% (TFA) (vol/vol) a uma concentração final de 0,2% TFA (vol/vol). Verifique se o pH de cada amostra está entre 2-3 com um papel pH. Se não, adicione pequenas alíquotas adicionais de 20% de TFA até que isso seja alcançado. As amostras devem ser apropriadamente acidificadas antes da desalada. Continue até o passo 8.
- Para rotulagem TMT das amostras, não acidifice e continue até o Passo 9.
8. Desalando o digestor
- Prepare uma dica de estágio para cada amostra embalando uma ponta compatível com solvente de 200 μL de MS com resina C18, conforme descrito por Rappsilber et al.20. Use uma agulha de ponta sem corte para pressionar dois discos de material C18, garantindo que os discos permaneçam na agulha. Transfira os discos para a ponta compatível com solvente MS usando um êmbolo de arame fino para expulsar os discos da agulha.
NOTA: É fundamental usar dicas compatíveis com solventes de MS a partir deste ponto do protocolo, uma vez que outras pontas de tubulação podem lixiviar produtos químicos nas amostras que podem ser detectadas através de espectrometria de massa. - Equilibre cada ponta de estágio com 30 μL de 100% MeOH, depois com 30 μL de 60% meoH (vol/vol), seguido por 30 μL de 0,1% TFA (vol/vol). Empurre cada solução através da ponta do palco com uma seringa. Conecte uma ponta de pipeta ao final de uma seringa com uma película transparente para aumentar o contato entre a seringa e a ponta do palco, se necessário. Certifique-se de nunca deixar o material C18 dentro da ponta do palco para secar.
- Adicione o peptídeo acidificado digesto do passo 7.3 até a ponta do palco rotulado e empurre através da ponta do palco com uma seringa, novamente tomando cuidado para não deixar a ponta do palco ficar completamente seca.
- Lave cada amostra 2x com 30 μL de 0,1% TFA (vol/vol). Elute os peptídeos de cada ponta de etapa adicionando 30 μL de 60% MeOH (vol/vol) a cada ponta de palco e expulsando tudo da ponta com pressão de seringa em um tubo novo e rotulado. Este é o único passo onde o material C18 está completamente seco.
- Seque cada amostra por centrifugação a vácuo. Peptídeos secos podem ser armazenados a -20°C por vários meses. As amostras estão agora prontas para análise de espectrometria de massa sem rótulos. Use apenas metade da amostra para análise no espectrômetro de massa e a outra metade para reinjeção, se necessário. Certifique-se do uso de um método espectrômetro de massa adequado para análise.
9. Rotulagem TMT
NOTA: A rotulagem de etiquetas em massa tandem é usada para amostras multiplex para análise quantitativa. Um frasco de 0,8 mg de reagente TMT é suficiente para rotular até 0,8 mg de proteína21. Em um experimento de pulldown PIB a partir de 1 mg de proteína, 1-3 μg de subunidades de fosfoproteína são obtidas. O protocolo abaixo é ideal para até 10 μg de proteína.
- Reconstituir um frasco de 0,8 mg de reagente TMT em 80 μL de ACN anidro.
- Rotule cada amostra da Etapa 7.4 com um rótulo TMT diferente para até 18 canais. Certifique-se de observar qual rótulo TMT é adicionado a cada amostra. Adicione 2 μL do reagente TMT e 2 μL de ACN ao digestor de peptídeo, vórtice suavemente para misturar, centrífuga a 376 x g por 30 s em RT para coletar a amostra e incubar na RT por 1h para rotular a amostra.
- Para testar a eficiência de rotulagem TMT, faça uma amostra de verificação de etiqueta combinando 1 μL de cada reação de rotulagem em um tubo de 0,5 mL contendo 9 μL de água de grau LC-MS e 1 μL de hidroxilamina (vol/vol) para saciar a reação. Coloque as amostras rotuladas não saciadas restantes em um congelador de -80 °C. As amostras podem ser armazenadas por vários dias enquanto a eficiência de rotulagem é avaliada.
- Acidificar a amostra de verificação de etiqueta TMT adicionando 30 μL de 0,1% TFA (vol/vol). Verifique se o pH está entre 2-3. Caso não, adicione 20% de TFA (vol/vol) até que este pH seja alcançado. Desaltar a amostra de verificação de rótulo via inclinação de estágio, conforme descrito nas etapas 8.1.-8.5.
- Analise a amostra de verificação de etiqueta TMT no espectrômetro de massa para avaliar a eficiência da rotulagem. Filtre os resultados da pesquisa para uma taxa de descoberta falsa (FDR) de 1% no nível do peptídeo e determine a eficiência de rotulagem21,22.
- Peptídeos totalmente rotulados em TMT têm reagente TMT no N-terminus e em todas as lises. Quantifique as intensidades de íons de repórteres do TMT e compare sua soma total em todos os canais. A amostra é suficientemente rotulada quando >95% de todos os peptídeos são rotulados e as intensidades de soma de íons de repórter TMT são comparáveis
NOTA: Além da rotulagem incompleta, as diferenças nas intensidades de íons de repórter TMT somadas podem ser resultado de tubulações imprecisas da amostra de teste de 1 μL. Também poderia refletir uma verdadeira observação biológica, nesse caso todas as réplicas devem apresentar o mesmo comportamento. - Remova as amostras não saciadas do armazenamento de -80 °C e descongele-as. Se eles não estiverem totalmente rotulados, adicione 1 μL do reagente TMT apropriado à amostra conforme observado acima, incubar por 1h e repetir a verificação da etiqueta TMT. Se as amostras estiverem totalmente rotuladas, continue até a etapa 9.8.
- Quando totalmente rotulado, adicione 2 μL de 10% de hidroxilamina (vol/vol) às reações TMT para saciar a rotulagem. Incubar as amostras na RT por 15 minutos. Uma vez saciadas, as amostras podem ser armazenadas a -80 °C por vários meses.
- Combine todos os canais TMT extintos e adicione 2 μL de 20% TFA (vol/vol) para acidificar a reação. Verifique o pH da reação combinada, garantindo que esteja entre pH 2-3. Se não, adicione alíquotas pequenas de 20% de TFA até que o pH desejado seja atingido. Isso é fundamental para o desalting adequado.
- Remova a ACN por centrifugação de vácuo por 30 minutos e desalize a amostra conforme descrito abaixo.
10. Desalar a amostra combinada com etiqueta TMT
- Use uma placa de desalar SPE C18 com a capacidade proteica adequada (2 mg sorbent geralmente é suficiente para esta aplicação) para desalar o reagente TMT combinado. Equilibre o poço com 200 μL de 60% MeOH (vol/vol) e 200 μL de 10% MeOH/0,1% TFA (vol/vol).
- Carregue os peptídeos acidificados com tmt a partir da etapa 9.10. sobre a placa de desalting. Lave os poços amostrais 2x com 200 μL de 10% MeOH/0,1% TFA (vol/vol).
- Elute os peptídeos com 100 μL de 60% MeOH (vol/vol). Seque as amostras por centrifugação a vácuo. A amostra seca, com rótulo TMT, pode ser armazenada a -80 °C por vários meses.
- Para análise de espectrometria de massa da amostra rotulada TMT, injete metade da amostra e salve a outra metade se for necessária reinjeção.
NOTA: As quantidades de injeção na espectrometria de massa variam dependendo da coluna específica, configuração do instrumento e tipo de amostra.
11. Análise de dados
NOTA: Os métodos de filtragem e análise de dados variam e estão além do escopo deste protocolo, mas as seguintes notas sobre análise são incluídas para fornecer orientações específicas para o tipo de dados resultantes deste protocolo.
- Pesquise dados de espectrometria de massa bruta em um banco de dados proteome específico da espécie com base na origem das células da amostra ou tecido utilizado. Aqui, cometa foi usado como um algoritmo de busca23.
- Filtre os resultados de pesquisa com um FDR de 1%, ajustando os parâmetros específicos do algoritmo de pesquisa22. Para quantificação sem rótulos, use medições de área de pico MS1 para quantificar os dados. Para amostras rotuladas por TMT, use intensidades de íons de repórter derivados de MSn para quantificação. Para significância estatística, analise as amostras em triplicados biológicos.
- Para identificar as subunidades PPP e seus interagirres, certifique-se de que são comparadas amostras biológicas triplicadas de amostras inibidas por MCLR e tratadas com DMSO. Para comparar o PPPome em diferentes condições ou após o tratamento medicamentoso, garantir que triplicados biológicos de cada condição ou tratamento medicamentoso fossem gerados.
- Filtre os dados para que apenas proteínas com uma contagem total de peptídeos >1 em pelo menos duas das três amostras tratadas com controle DMSO estejam presentes. A competição MCLR nem sempre compete fora de todas as subunidades catalíticas. Além disso, algumas subunidades catalíticas PPP podem não especificamente grudar na resina de sepharose. Para explicar qualquer possibilidade ao filtrar proteínas que não se ligam especificamente à resina, remova proteínas com uma contagem total de peptídeos na condição tratada com MCLR maior do que a de qualquer subunidade catalítico PPP.
- Excluam contaminantes comuns, como queratina, colágeno, proteínas ribossômicas 40S e 60S, e ribonucleoproteínas nucleares heterogêneas que não são subunidades PPP, a partir da análise14.
- Importe dados filtrados em Perseus clicando em Upload de Matriz Genérica na seçãoCarga 24,25. O Log2 transforma os dados indo para o Basic > Transform, selecionando os dados e especificando a função de transformação, neste caso log2(x).
- Imputar valores perdidos de uma distribuição normal indo para a imputação > substituir valores ausentes da distribuição normal, selecionando os dados e especificando a largura (padrão 0.3) e a mudança de marcha para baixo (padrão 1.8) para o cálculo. Realize a normalização quântica indo para Normalizar > Normalização Quântica.
- Calcular as razões do log2 e os valores p do teste T do Aluno para as respectivas condições. Primeiro, anote os dados indo para a Anotação. Rows > Categóricos Linhas de Anotação. Realize o teste T indo a Testes > Testes de Duas Amostras e selecionando os grupos para comparar, o teste a ser realizado e o método para correção de teste de hipóteses múltiplas, conforme usado para truncação.
NOTA: Para identificação de novo , uma proteína é considerada uma proteína que interage por PPP se sua abundância for estatisticamente significante na condição tratada com MCLR versus DMSO, com uma alteraçãode log 2 vezes maior do que a troca mínima de dobra de qualquer subunidade PPP especificamente ligada.
Access restricted. Please log in or start a trial to view this content.
Resultados
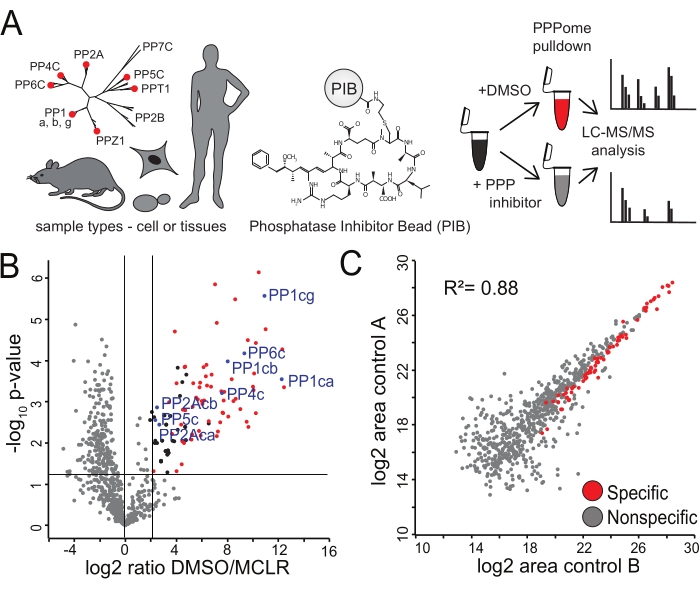
Figura 2: Identificação de aglutinantes específicos de PIBs. (A) Uma variedade de tipos de tecidos ou células pode ser analisada via PIB-MS. As células HeLa em triplicado biológico foram tratadas com DMSO ou com o INibidor de PPP MCLR, incubadas com PIBs, e analisadas via LC-MS/MS. (B) O ...
Access restricted. Please log in or start a trial to view this content.
Discussão
PIB-MS é uma abordagem química de proteômica usada para traçar o perfil quantitativo do PPPome de várias fontes de amostra em uma única análise. Muito trabalho tem sido feito usando contas inibidoras de quinase para estudar o kinome e como ele muda no câncer e outros estados da doença 10,11,12,13. No entanto, o estudo do PPPome fica para trás. Prevemos que essa abordagem seja capaz de...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar e nem conflitos de interesse.
Agradecimentos
A A.N.K. reconhece o suporte do NIH R33 CA225458 e R35 GM119455. Agradecemos aos laboratórios Kettenbach e Gerber por sua discussão útil.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Referências
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases--the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755(2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados