É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Infecção de Células Epiteliais Primárias Nasais Cultivadas em Interface Ar-Líquido para Caracterizar Interações Coronavírus-Hospedeiro Humano
Neste Artigo
Resumo
O epitélio nasal é o principal sítio de barreira encontrado por todos os patógenos respiratórios. Aqui, descrevemos métodos para usar células epiteliais nasais primárias cultivadas como culturas de interface ar-líquido (LPA) para caracterizar interações coronavírus-hospedeiro humano em um sistema fisiologicamente relevante.
Resumo
Três coronavírus humanos altamente patogênicos (HCoVs) - SARS-CoV (2002), MERS-CoV (2012) e SARS-CoV-2 (2019) - emergiram e causaram crises de saúde pública significativas nos últimos 20 anos. Quatro HCoVs adicionais causam uma parcela significativa de casos de resfriado comum a cada ano (HCoV-NL63, -229E, -OC43 e -HKU1), destacando a importância do estudo desses vírus em sistemas fisiologicamente relevantes. Os HCoVs entram no trato respiratório e estabelecem a infecção no epitélio nasal, o sítio primário encontrado por todos os patógenos respiratórios. Usamos um sistema primário de cultura epitelial nasal no qual amostras nasais derivadas do paciente são cultivadas em uma interface ar-líquido (LPA) para estudar as interações patógeno-hospedeiro neste importante sítio sentinela. Essas culturas recapitulam muitas características da via aérea in vivo , incluindo os tipos celulares presentes, a função ciliar e a produção de muco. Descrevemos métodos para caracterizar a replicação viral, o tropismo de células hospedeiras, a citotoxicidade induzida por vírus e a indução de imune inata em culturas nasais de LPA após infecção por HCoV, usando trabalhos recentes comparando HCoVs letal e sazonal como exemplo1. Uma maior compreensão das interações hospedeiro-patógeno no nariz tem o potencial de fornecer novos alvos para a terapêutica antiviral contra HCoVs e outros vírus respiratórios que provavelmente surgirão no futuro.
Introdução
Sete coronavírus humanos (HCoVs) foram identificados até o momento e causam uma série de doenças respiratórias2. Os HCoVs comuns ou sazonais (HCoV-NL63, -229E, -OC43 e -HKU1) são tipicamente associados à patologia do trato respiratório superior e causam cerca de 10%-30% dos casos de resfriado comum anualmente. Embora esse seja o fenótipo clínico típico associado ao HCoVs, esses vírus podem causar doença mais significativa do trato respiratório inferior em populações de risco, incluindo crianças, idosos e indivíduos imunocomprometidos 3,4. Três HCoVs patogênicos surgiram e causaram emergências de saúde pública significativas nos últimos 20 anos, incluindo síndrome respiratória aguda grave (SARS)-CoV, síndrome respiratória do Oriente Médio (MERS)-CoV e SARS-CoV-2. Os HCoVs letais estão associados a patologias mais graves do trato respiratório, o que é claramente ilustrado pela taxa de letalidade de >34% associada aos casos de MERS-CoV (894 mortes em mais de 2.500 casos desde seu surgimento em 2012)5,6. É importante notar que os HCoVs letais também causam uma série de doenças do trato respiratório, desde infecções assintomáticas até pneumonia letal, como visto com a pandemia COVID-19 em curso7.
Os HCoVs, como outros patógenos respiratórios, entram no trato respiratório e estabelecem uma infecção produtiva no epitélionasal8. Acredita-se que a disseminação para a via aérea inferior esteja associada à aspiração da cavidade oral/nasal para o pulmão, onde os HCoVs causam patologia mais significativa do trato respiratório inferior9,10,11. Assim, o nariz serve como o portal inicial para a entrada viral e é a barreira primária à infecção com sua robusta maquinaria de depuração mucociliar e mecanismos imunes inatos únicos destinados a prevenir a disseminação viral para as vias aéreasinferiores12,13. Por exemplo, foi relatado que células epiteliais nasais expressam níveis basais acima da média de interferons antivirais e genes estimulados por interferon, indicando que as células nasais podem ser preparadas para respostas precoces a vírus respiratórios14,15,16.
Utilizamos previamente células epiteliais nasais primárias derivadas do paciente cultivadas em uma interface ar-líquido (LPA) para modelar as interações HCoV-hospedeiro no nariz, onde as infecções por HCoV começam. As culturas nasais de LPA são permissivas tanto para HCoVs patogênicos (SARS-CoV-2 e MERS-CoV) quanto para HCoVs comuns (HCoV-NL63 e HCoV-229E) e oferecem várias vantagens sobre linhagens celulares epiteliais tradicionais das vias aéreas, como a A549 (uma linhagem celular de adenocarcinoma pulmonar)16,17. Após a diferenciação, as culturas nasais de LPA contêm uma população celular heterogênea e exibem muitas das funções esperadas do epitélio nasal in vivo, como a maquinaria de depuração mucociliar18. As células nasais também oferecem vantagens sobre os sistemas de cultura das vias aéreas inferiores (como as células epiteliais brônquicas humanas, HBECs), uma vez que a aquisição de células epiteliais nasais via escovação citológica é significativamente menos invasiva em comparação com o uso de técnicas como a broncoscopia para a obtenção de HBECs 19,20,21.
Este trabalho descreve métodos de utilização deste sistema de cultura de LPA nasal para caracterizar as interações HCoV-hospedeiro no epitélio nasal. Nós aplicamos estes métodos em trabalhos recentemente publicados para comparar SARS-CoV-2, MERS-CoV, HCoV-NL63, e HCoV-229E 1,16,17. Embora esses métodos e resultados representativos enfatizem o estudo dos HCoVs neste modelo de células nasais, o sistema é altamente adaptável a outros HCoVs, bem como a outros patógenos respiratórios. Além disso, esses métodos podem ser aplicados de forma mais ampla a outros sistemas de cultura de LPA, a fim de investigar a replicação viral e o tropismo celular, bem como a citotoxicidade e a indução imune inata após a infecção.
Protocolo
O uso de espécimes nasais foi aprovado pelo Comitê de Revisão Institucional da Universidade da Pensilvânia (protocolo # 800614) e pelo Comitê de Revisão Institucional da Filadélfia VA (protocolo # 00781).
1. Infecção de culturas nasais de LPA
NOTA: A aquisição de espécimes clínicos, bem como o crescimento e diferenciação de culturas nasais de LPA, está fora do escopo deste trabalho. Métodos específicos para a cultura de células epiteliais nasais primárias podem ser encontrados em trabalhos publicados recentemente utilizando essas culturas18,22,23. Os protocolos abaixo podem adicionalmente ser aplicados a culturas de LPA epitelial nasal comercialmente disponíveis, se desejado. Os protocolos e volumes detalhados abaixo são aplicáveis a insertos de transpoço de placa de 24 poços (6,5 mm de diâmetro, 0,33 cm de área de superfíciede membrana 2). Se usar culturas de LPA cultivadas em transpoços maiores (isto é, placas de 12 poços, 12 mm de diâmetro, 1,12 cm2 de área de superfície), ajuste os volumes proporcionalmente para refletir o tamanho do transpoço.
- Dia antes da infecção:
- Lavar culturas de LPA 3x com solução salina tamponada com fosfato (PBS) apicamente (adicionar ~200 μL de PBS aquecido, colocar em incubadora a 37 °C por 5 min, aspirar PBS e repetir).
- Substitua o meio basal (500 μL).
- Permitir que as culturas se equilibrem na temperatura em que as infecções serão conduzidas durante a noite (ou seja, se infectar a 33 °C, coloque as culturas em uma incubadora de 33 °C após as lavagens com PBS).
NOTA: HCoVs associados ao resfriado comum, como HCoV-229E e HCoV-NL63, são relatados para replicar de forma mais eficiente a 33 °C. Além disso, a temperatura do epitélio nasal in vivo é de 33 °C (isso difere da temperatura do pulmão, que é de 37 °C).
- Diluir o vírus conforme necessário no meio de águia modificado (DMEM) de Dulbecco livre de soro para atingir a multiplicidade desejada de infecção (MOI) em um volume total de inóculo de 50 μL.
NOTA: As infecções têm sido tipicamente conduzidas em MOI = 5 (MOI alto); no entanto, infecções em MOI = 0,5 (MOI baixo) também foram usadas para SARS-CoV-2 e HCoVs associado ao resfriado comum, e estes resultam em títulos virais de pico comparáveis, mas com cinética variável (qualquer MOI é aceitável). - Adicione inóculo apicamente e coloque as culturas de volta na incubadora por 1 h.
- Placas de rocha suavemente a cada 15 minutos durante a infecção (segurando a placa firmemente em ambas as mãos, balança para frente e para trás e de um lado para o outro para garantir a adsorção uniforme do inóculo viral).
- Após 1 h de incubação, aspirar o inóculo viral e lavar cada cultura infectada 3x com PBS para garantir a remoção do inóculo viral (para cada lavagem, adicionar 200 μL de PBS, incubar por 5 min e aspirar ou remover com uma pipeta).
- Se desejar, colete a terceira lavagem PBS para confirmar a remoção adequada do vírus de entrada.
- Substitua o meio basal por meio fresco nas LPA infectadas a cada 72 h durante a infecção.
2. Coleta de líquido de superfície apical (ASL) e titulação do vírus excretado
- Em momentos pré-determinados após a infecção, adicionar 200 μL de PBS à câmara apical de cada transwell infectado.
NOTA: Os pontos de tempo relevantes variam dependendo do HCoV de interesse e variam de 24 h a 192 h após a infecção (consulte a seção de resultados representativos para obter dados de replicação viral para vários HCoVs). - Pipetar PBS para cima e para baixo 5x para garantir a coleta máxima de vírus derramado apicamente, e coletar todo o volume em um tubo de microcentrífuga (esta é a amostra ASL).
NOTA: ASL inclui partículas virais derramadas, além de muco e outros produtos secretados apicamente de culturas de ALI. - Quantificar vírus infecciosos em ASL através de ensaio padrão de placa viral (diluir serialmente amostras contendo vírus para quantificar a concentração de partículas virais).
NOTA: Os tipos celulares e o período de incubação usados para o ensaio da placa dependerão do vírus usado: SARS-CoV-2 (células VeroE6); MERS-CoV (células VeroCCL81); HCoV-NL63 (células LLC-MK2); HCoV-229E (células Huh7). Detalhes sobre como conduzir ensaios de placa viral estão além do escopo deste manuscrito, mas foram detalhados anteriormente em uma publicação do Journal of Visualized Experiments (JoVE)24. - Se desejar, coletar o meio basal em vários momentos pós-infecção para confirmar a ausência de vírus liberados basalmente. Os HCoVs são tipicamente liberados apicamente das células epiteliais nasais, mas confirmam isso por meio do ensaio de placa do meio basal não diluído.
- Armazenar amostras de ASL a -80 °C se a quantificação por ensaio de placa não ocorrer no dia da coleta.
3. Quantificação do vírus intracelular
- Após a coleta da amostra de ASL, mova cada transwell para uma placa limpa de 24 poços pré-carregada com 500 μL de DMEM contendo 2% de soro fetal bovino (FBS) basalmente.
NOTA: DMEM com 2% FBS é usado para estabilizar o vírus durante ciclos subsequentes de congelamento-descongelamento. - Lave cada transwell 3x apicamente com PBS para garantir a remoção completa do vírus derramado apicamente.
- Após aspirar para remover a lavagem final com PBS, adicionar 100 μL de DMEM com SFB a 2% ao compartimento apical.
- Mova a placa que contém transwells com meios apical e basal para um congelador de -80 °C e complete três ciclos consecutivos de congelamento-descongelamento para lisar as células.
- Após o ciclo final de congelamento-descongelamento, coloque os meios apical (100 μL) e basal (500 μL) em um tubo limpo.
- Centrifugar a 500 × g durante 10 min a 4 °C para pellet de quaisquer detritos celulares.
- Colete o sobrenadante. Esta é a amostra de vírus intracelular para titulação via ensaio de placa padrão.
NOTA: A diluição de três vezes ocorre durante o processo de coleta em relação à coleta de ASL; As amostras de ASL são coletadas em 200 μL de PBS, enquanto a amostra de vírus intracelular é coletada em um volume total de 600 μL.
4. Medição da resistência elétrica transepitelial (TEER)
NOTA: Para a medição do TEA, deve-se usar PBS suplementado com cálcio e magnésio (PBS + Ca 2+/Mg2+). Um volt/ohmímetro epitelial ajustado para leitura em ohms é usado (veja a Tabela de Materiais).
- Limpar, equilibrar e deixar em branco o instrumento EVOM de acordo com as instruções do fabricante; usar um transwell "vazio" sem adição de células nasais para blanking. Registre a medição TEER em branco.
NOTA: Se estiver usando vários vírus, o instrumento EVOM deve ser limpo rigorosamente entre as condições para evitar contaminação cruzada (lavagens com etanol 70% seguidas de água deionizada são suficientes). - Mover cada transwell infectado para uma placa pré-marcada e limpa de 24 poços com 500 μL de PBS + Ca 2+/Mg2+ basalmente para lavar o meio basal residual das transwells.
- Adicionar 200 μL de PBS + Ca 2+/Mg2+ ao compartimento apical de cada transwell.
- Adicionar 1 mL de PBS + Ca 2+/Mg2+ à câmara de medição de Endohm-6.
- Mover cada transwell para a câmara de medição Endohm-6 e substituir a tampa da câmara de modo que o eletrodo apical descanse nos 200 μL de PBS no compartimento apical; o eletrodo basal é construído no fundo da câmara Endohm-6.
- Permita que a leitura EVOM se estabilize e registre a medição bruta do TEAR.
- Coletar amostra de ASL conforme descrito acima após a medição de TEER se a titulação for desejada (coletar os 200 μL de PBS + Ca 2+/Mg2+ que foi adicionado para a medição de TEA).
NOTA: A coleta de amostras de ASL pode induzir micro-rupturas na barreira epitelial que podem confundir as leituras de TEA, portanto, a ASL deve ser coletada após a medição de TEA. Além disso, o meio basal não deve ser trocado imediatamente antes das leituras ITER, pois isso também pode afetar os valores de ITER. - Para converter as leituras brutas do TEER em medidas finais em Ohms/cm2, subtraia o valor TEER em branco e multiplique esse valor pela área superficial da membrana do transpoço usando a equação (1):
TEER = [leitura TEER - valor TEER em branco] × (área de superfície da transwell) (1) - Para HCoVs, avaliar TEER a cada 24 h ou 48 h após a infecção; a cinética das alterações TEER geralmente varia entre os vírus e requer solução de problemas em vários pontos de tempo.
- Ao medir o TEER, inclua sempre culturas simuladas infectadas e avalie em cada momento (controle negativo).
NOTA: Para culturas simuladas, as medições TEER devem permanecer estáveis ou podem aumentar ligeiramente a partir da linha de base após a diferenciação das culturas ser concluída. A área de superfície dos suportes de membrana irá variar dependendo do tamanho e do fabricante dos transwells; As transwells de 24 poços normalmente têm uma área de superfície de 0,33 cm2.
5. Mensuração da citotoxicidade durante a infecção pelo ensaio de lactato desidrogenase (LDH)
NOTA: Neste trabalho, o conteúdo de LDH em amostras de ASL foi quantificado usando um kit de detecção de citotoxicidade disponível comercialmente. O sinal de LDH em meio basal foi frequentemente abaixo do limite de detecção e muitas vezes menos reprodutível do que o LDH quantificado em amostras de ASL de culturas infectadas com HCoV.
- Preparar controles adicionais necessários para o ensaio de LDH.
- Controle de antecedentes: usar PBS (usar PBS contendo cálcio e magnésio se amostras de ASL foram coletadas dessa forma).
- Controle positivo (valor teto): tratar as LPA apicamente com Triton X-100. Colete poços de controle Triton (normalmente use três culturas ALI para isso em cada ponto de tempo).
- Adicionar 200 μL de Triton X-100 a 2% em PBS diretamente ao compartimento apical do transwell.
- Incubar por 10-15 min para permitir que as células lisem completamente.
- Colete todo o volume como uma amostra do teto Triton.
- Controle negativo (baixo controle/liberação basal de LDH): coletar ASL de culturas de LPA infectadas simuladas derivadas do mesmo doador que culturas infectadas.
- Colete ASL simulado de controle negativo seguindo o procedimento de coleta de ASL acima.
NOTA: Os mesmos transwells podem ser usados para este controle negativo para todos os pontos de tempo, enquanto transwells frescos serão necessários para cada ponto de tempo para o controle Triton.
- Colete ASL simulado de controle negativo seguindo o procedimento de coleta de ASL acima.
- Para permitir a quantificação do vírus derramado, além das leituras de LDH de cada amostra ASL, carregue uma placa preta de fundo plano opticamente transparente de 96 poços da seguinte maneira:
- Diluir todas as amostras em PBS, utilizando 45 μL de amostra e 55 μL de PBS (100 μL de volume total).
- Trate o controle positivo Triton e simule amostras de controle de fundo da mesma maneira.
NOTA: Esta diluição permite o carregamento em triplicata de cada amostra experimental (45 μL x 3) e volume residual de ASL suficiente para titulação por ensaio de placa. - Carregue a placa de LDH em triplicata: teto Triton, controle de fundo simulado, controle PBS e amostras experimentais.
- Preparar a mistura de reação conforme indicado pelo fabricante (solução de corante + catalisador).
- Adicionar 100 μL de mistura de reação a cada poço e incubar a placa à temperatura ambiente por 20 min protegida da luz.
- Após 20 min, medir a absorbância a 492 nm.
- Calcular a porcentagem de citotoxicidade em relação ao valor teto de Triton usando a equação (2).
Citotoxicidade (%)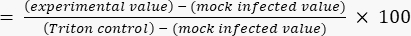 (2)
(2)
6. Preparo de culturas nasais de LPA para imagem de imunofluorescência (IF)
- Mova o transwell para uma placa fresca de 24 poços e lave 3x apicamente com PBS (colete a primeira lavagem PBS como ASL se titer; em seguida, execute duas lavagens PBS adicionais para remover o excesso de vírus derramado que pode prejudicar a qualidade do FI)
- Após a lavagem final com PBS, cobrir o transbem apical e basalmente em PFA a 4%.
- Incubar por 30 min para fixar em PFA a 4% e, em seguida, remover e lavar 3x com PBS.
- Excise o suporte de transwell que contém as células usando uma lâmina de barbear afiada ou tesoura.
- Para aumentar o número de alvos de FI para cada transwell, corte cada membrana ao meio e core cada metade com diferentes combinações de anticorpos.
- Permeabilizar com Triton X-100 a 0,2% em PBS por 10 min.
- Bloqueio com 10% de soro de burro normal e 1% de BSA em PBST (PBS + 0,2% Triton X-100) por 60 min à temperatura ambiente.
NOTA: Proteja as amostras da exposição à luz a partir deste passo em frente. - Incubar em solução primária de anticorpos durante a noite a 4 °C. Diluir todos os anticorpos 1:1.000 em tampão de bloqueio.
NOTA: Anticorpos representativos para a coloração de nucleocapsídeo HCoV, bem como marcadores de tipo celular epitelial e colorações citoesqueléticas, estão listados abaixo (consulte a Tabela de materiais para obter informações sobre o fabricante e números de catálogo).- Para a coloração do antígeno HCoV, use anticorpos direcionados ao nucleocapsídeo SARS-CoV-2, ao nucleocapsídeo MERS-CoV e ao nucleocapsídeo HCoV-NL63.
- Para identificar os tipos celulares epiteliais, utilizar anticorpos direcionados ao marcador de células caliciformes MUC5AC e ao marcador de células ciliadas tipo IV β-tubulina.
- Para marcadores citoesqueléticos, utilizar anticorpos direcionados contra faloidina (liga-se à F-actina) e marcador de adesão de células epiteliais: EpCAM (CD326).
- Incubar em solução de anticorpos secundários durante 60 minutos à temperatura ambiente; usar corantes de anticorpos secundários diluídos 1:1.000 em tampão de bloqueio.
- Após a cicatrização estar completa, transfira a membrana para uma lâmina de vidro com uma espátula, oriente o transwell com o lado apical em direção à lâmina e adicione a solução de montagem. Deixe descansar por 15-30 minutos antes de aplicar esmalte claro ao redor das bordas.
- Adquirir imagens utilizando microscópio confocal (passo eixo Z: 0,5 μm; varredura sequencial)1,16,17.
NOTA: Após fixação das culturas em PFA a 4% e lavagem com PBS, as culturas fixas podem ser armazenadas a 4 °C por semanas a meses antes da coloração e preparação para exames de imagem. Após coloração e montagem das membranas, as amostras podem ser armazenadas a longo prazo (>2 anos) a 4 °C no escuro.
7. Coleta de proteína intracelular para western immunoblotting ou RNA para análise de RT-qPCR
- Coletar ASL conforme descrito acima se quantificar títulos virais (protocolo seção 2).
- Mova o transwell para uma placa limpa de 24 poços, pois raspar a membrana pode levar à quebra da inserção.
- Para a análise de western blot, coletar lisado proteico total em 125 μL de tampão RIPA (50 mM Tris, pH 8, 150 mM NaCl, 0,5% desoxicolato, 0,1% SDS, 1% NP40) suplementado com inibidores de protease e inibidores de fosfatase.
- Adicionar 125 μL de tampão RIPA ao compartimento apical e incubar por 5-10 min.
- Raspe a membrana usando uma ponta de pipeta P200 para remover as células aderidas restantes e colete todo o volume (raspe vigorosamente em toda a superfície da membrana e, em seguida, pipete para cima e para baixo várias vezes para coletar toda a amostra).
- Incubar amostras de lisado proteico no gelo durante 10 min e, em seguida, centrifugar à velocidade máxima (20.000 × g) durante 10 min a 4 °C.
- Misturar o sobrenadante com tampão de amostra Laemmli 4x com β-mercaptoetanol (agente redutor) de acordo com os protocolos do fabricante.
- Ferver amostras de proteína a 95 °C por 5 min e, em seguida, executar usando protocolos tradicionais de western blotting 1,16,17.
- Para a coleta de RNA total, use um kit de extração de RNA disponível comercialmente de escolha.
NOTA: O compartimento apical das pastilhas transwell de 24 poços tem um volume máximo de ~200 μL; Assim, realizamos duas lavagens sequenciais em cada cultura para atingir o volume total recomendado. Os detalhes abaixo correspondem à coleta recomendada de amostras de RNA em um volume total de 350 μL. - Adicionar 200 μL de tampão de lise ao compartimento apical da transwell infectada.
- Deixe descansar por 5-10 min, raspe todas as células restantes da membrana usando uma ponta de pipeta e colete todo o volume em um tubo de microcentrífuga marcado.
- Adicionar 150 μL de tampão de lise adicional ao compartimento apical do transwell e pipetar para cima e para baixo antes de coletar no mesmo tubo de microcentrífuga.
- Extrair RNA de acordo com o protocolo do fabricante.
Resultados
Os números representativos são parcialmente adaptados a partir de dados que podem ser encontrados no manuscrito Otter et al.1. Culturas nasais de LPA derivadas de quatro ou seis doadores foram infectadas com um dos quatro HCoVs (SARS-CoV-2, MERS-CoV, HCoV-NL63 e HCoV-229E) de acordo com os protocolos descritos acima, e os títulos virais médios eliminados apicamente para cada vírus estão representados na Figura 1A. Enquanto todos esses quatro HCoVs se replicam pr...
Discussão
Os métodos aqui detalhados descrevem um sistema primário de cultura epitelial no qual células epiteliais nasais derivadas do paciente são cultivadas em uma interface ar-líquido e aplicadas ao estudo das interações HCoV-hospedeiro. Uma vez diferenciadas, essas culturas nasais de LPA recapitulam muitas características do epitélio nasal in vivo , incluindo uma população celular heterogênea com células ciliadas, caliciformes e basais representadas, bem como função mucociliar intacta com cílios e secr...
Divulgações
Susan Weiss faz parte dos Conselhos Consultivos Científicos da Ocugen. Noam A. Cohen é consultor da GSK, AstraZeneca, Novartis, Sanofi/Regeron e Oyster Point Pharmaceuticals e tem uma patente nos EUA, "Therapy and Diagnostics for Respiratory Infection" (10.881.698 B2, WO20913112865), e um acordo de licenciamento com a GeneOne Life Sciences.
Agradecimentos
Este estudo tem as seguintes fontes de financiamento: National Institutes of Health (NIH) R01AI 169537 (S.R.W. e N.A.C.), NIH R01AI 140442 (S.R.W.), VA Merit Review CX001717 (N.A.C.), VA Merit Review BX005432 (S.R.W. e N.A.C.), Penn Center for Research on Coronaviruses and other Emerging Pathogens (S.R.W.), Laffey-McHugh Foundation (S.R.W. e N.A.C.), T32 AI055400 (CJO), T32 AI007324 (AF).
Materiais
| Name | Company | Catalog Number | Comments |
| Alexa Fluor secondary antibodies (488, 594, 647) | Invitrogen | Various | |
| BSA (bovine serum albumin) | Sigma-Aldrich | A7906 | |
| cOmplete mini EDTA-free protease inhibitor | Roche | 11836170001 | |
| Cytotoxicity detection kit | Roche | 11644793001 | |
| DMEM (Dulbecco's Modified Eagle Media) | Gibco | 11965-084 | |
| DPBS (Dulbecco's Phosphate Buffered Saline) | Gibco | 14190136 | |
| DPBS + calcium + magnesium | Gibco | 14040-117 | |
| Endohm-6G measurement chamber | World Precision Instruments | ENDOHM-6G | |
| Epithelial cell adhesion marker (EpCAM; CD326) | eBiosciences | 14-9326-82 | |
| Epithelial Volt/Ohm (TEER) Meter (EVOM) | World Precision Instruments | 300523 | |
| FBS (Fetal Bovine Serum) | HyClone | SH30071.03 | |
| FV10-ASW software for imaging | Olympus | Version 4.02 | |
| HCoV-NL63 (Human coronavirus, NL63) | BEI Resources | NR-470 | |
| HCoV-NL63 nucleocapsid antibody | Sino Biological | 40641-V07E | |
| Hoescht stain | Thermo Fisher | H3570 | |
| Laemmli sample buffer (4x) | BIO-RAD | 1610747 | |
| LLC-MK2 cells | ATCC | CCL-7 | To titrate HCoV-NL63 |
| MERS-CoV (Human coronavirus, Middle East Respiratory Syndrome Coronavirus (MERS-CoV), EMC/2012) | BEI Resources | NR-44260 | |
| MERS-CoV nucleocapsid antibody | Sino Biological | 40068-MM10 | |
| MUC5AC antibody | Sigma-Aldrich | AMAB91539 | |
| Olympus Fluoview confocal microscope | Olympus | FV1000 | |
| Phalloidin-iFluor 647 stain | Abcam | ab176759 | |
| PhosStop easy pack (phosphatase inhibitors) | Roche | PHOSS-RO | |
| Plate reader | Perkin Elmer | HH34000000 | Any plate reader or ELISA reader is sufficient; must be able to read absorbance at 492 nm |
| RIPA buffer (50 mM Tris pH 8; 150 mM NaCl; 0.5% deoxycholate; 0.1% SDS; 1% NP40) | Thermo Fisher | 89990 | Can prep in-house or purchase |
| RNeasy Plus Kit | Qiagen | 74134 | |
| SARS-CoV-2 (SARS-Related Coronavirus 2, Isolate USA-WA1/2020) | BEI Resources | NR-52281 | |
| SARS-CoV-2 nucleocapsid antibody | Genetex | GTX135357 | |
| Triton-X 100 | Fisher Scientific | BP151100 | |
| Type IV β- tubulin antibody | Abcam | ab11315 | |
| VeroCCL81 cells | ATCC | CCL-81 | To titrate MERS-CoV |
| VeroE6 cells | ATCC | CRL-1586 | To titrate SARS-CoV-2 |
Referências
- Otter, C. J., et al. Infection of primary nasal epithelial cells differentiates among lethal and seasonal human coronaviruses. Proceedings of the National Academy of Sciences of the United States of America. 120 (15), 2218083120 (2023).
- Fehr, A., Perlman, S. Coronaviruses: An overview of their replication and pathogenesis. Methods in Molecular Biology. 1282, 1-23 (2015).
- Gaunt, E. R., Hardie, A., Claas, E. C. J., Simmonds, P., Templeton, K. E. Epidemiology and clinical presentations of the four human coronaviruses 229E, HKU1, NL63, and OC43 detected over 3 years using a novel multiplex real-time PCR method. Journal of Clinical Microbiology. 48 (8), 2940-2947 (2010).
- Kesheh, M. M., Hosseini, P., Soltani, S., Zandi, M. An overview on the seven pathogenic human coronaviruses. Reviews in Medical Virology. 32 (2), 2282 (2022).
- MERS-CoV Worldwide Overview. European Centre for Disease Prevention and Control Available from: https://www.ecdc.europa.eu/en/middle-east-respiratory-syndrome-coronavirus-mers-cov-situation-update (2022)
- Cao, Y., Liu, X., Xiong, L., Cai, K. Imaging and clinical features of patients with 2019 novel coronavirus SARS-CoV-2: A systematic review and meta-analysis. Journal of Medical Virology. 92 (9), 1449-1459 (2020).
- Vareille, M., Kieninger, E., Edwards, M. R., Regamey, N. The airway epithelium: Soldier in the fight against respiratory viruses. Clinical Microbiology Reviews. 24 (1), 210-229 (2011).
- Farzal, Z., et al. Comparative study of simulated nebulized and spray particle deposition in chronic rhinosinusitis patients. International Forum of Allergy and Rhinology. 9 (7), 746-758 (2019).
- Gaeckle, N. T., Pragman, A. A., Pendleton, K. M., Baldomero, A. K., Criner, G. J. The oral-lung axis: The impact of oral health on lung health. Respiratory Care. 65 (8), 1211-1220 (2020).
- Hou, Y., et al. SARS-CoV-2 reverse genetics reveals a variable infection gradient in the respiratory tract. Cell. 182, 429-446 (2020).
- Hariri, B. M., Cohen, N. A. New insights into upper airway innate immunity. American Journal of Rhinology and Allergy. 30 (5), 319-323 (2016).
- Hiemstra, P. S., McCray, P. B., Bals, R. The innate immune function of airway epithelial cells in inflammatory lung disease. European Respiratory Journal. 45 (4), 1150-1162 (2015).
- Hatton, C. F., et al. Delayed induction of type I and III interferons mediates nasal epithelial cell permissiveness to SARS-CoV-2. Nature Communications. 12 (1), 7092 (2021).
- Sungnak, W., et al. SARS-CoV-2 entry factors are highly expressed in nasal epithelial cells together with innate immune genes. Nature Medicine. 26 (5), 681-687 (2020).
- Li, Y., et al. SARS-CoV-2 induces double-stranded RNA-mediated innate immune responses in respiratory epithelial-derived cells and cardiomyocytes. Proceedings of the National Academy of Sciences of the United States of America. 118 (16), 2022643118 (2021).
- Comar, C. E., et al. MERS-CoV endoribonuclease and accessory proteins jointly evade host innate immunity during infection of lung and nasal epithelial cells. Proceedings of the National Academy of Sciences of the United States of America. 119 (21), 2123208119 (2022).
- Lee, R. J., et al. Bacterial D-amino acids suppress sinonasal innate immunity through sweet taste receptors in solitary chemosensory cells. Science Signaling. 10 (495), (2017).
- Brewington, J. J., et al. Brushed nasal epithelial cells are a surrogate for bronchial epithelial CFTR studies. JCI Insight. 3 (13), 99385 (2018).
- Comer, D. M., Elborn, J. S., Ennis, M. Comparison of nasal and bronchial epithelial cells obtained from patients with COPD. PLoS One. 7 (3), e32924 (2012).
- Vanders, R. L., Hsu, A., Gibson, P. G., Murphy, V. E., Wark, P. A. B. Nasal epithelial cells to assess in vitro immune responses to respiratory virus infection in pregnant women with asthma. Respiratory Research. 20 (1), 259 (2019).
- Lee, R. J., et al. Fungal aflatoxins reduce respiratory mucosal ciliary function. Scientific Reports. 6, 33221 (2016).
- Patel, N. N., et al. Fungal extracts stimulate solitary chemosensory cell expansion in noninvasive fungal rhinosinusitis. International Forum of Allergy and Rhinology. 9 (7), 730-737 (2019).
- Baer, A., Kehn-Hall, K. Viral concentration determination through plaque assays: Using traditional and novel overlay systems. Journal of Visualized Experiments. 93 (93), e52065 (2014).
- Robinot, R., et al. SARS-CoV-2 infection induces the dedifferentiation of multiciliated cells and impairs mucociliary clearance. Nature Communications. 12 (1), 4354 (2021).
- Whitsett, J. A. Airway epithelial differentiation and mucociliary clearance. Annals of the American Thoracic Society. 15, S143-S148 (2018).
- Gao, N., Raduka, A., Rezaee, F. Respiratory syncytial virus disrupts the airway epithelial barrier by decreasing cortactin and destabilizing F-actin. Journal of Cell Science. 135 (16), 259871 (2022).
- Schmidt, H., et al. IL-13 impairs tight junctions in airway epithelia. International Journal of Molecular Sciences. 20 (13), 3222 (2019).
- Huang, Z. Q., et al. Interleukin-13 alters tight junction proteins expression thereby compromising barrier function and dampens rhinovirus induced immune responses in nasal epithelium. Frontiers in Cell and Developmental Biology. 8, 572749 (2020).
- Saatian, B., et al. Interleukin-4 and interleukin-13 cause barrier dysfunction in human airway epithelial cells. Tissue Barriers. 1 (2), e24333 (2013).
- Coles, J. L., et al. A revised protocol for culture of airway epithelial cells as a diagnostic tool for primary ciliary dyskinesia. Journal of Clinical Medicine. 9 (11), 3753 (2020).
- Baldassi, D., Gabold, B., Merkel, O. M. Air−liquid interface cultures of the healthy and diseased human respiratory tract: Promises, challenges, and future directions. Advanced NanoBiomed Research. 1 (6), 2000111 (2021).
- Seibold, M. A. Interleukin-13 stimulation reveals the cellular and functional plasticity of the airway epithelium. Annals of the American Thoracic Society. 15, S98-S106 (2018).
- Morrison, C. B., et al. SARS-CoV-2 infection of airway cells causes intense viral and cell shedding, two spreading mechanisms affected by IL-13. Proceedings of the National Academy of Sciences of the United States of America. 119 (16), 2119680119 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados