JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
유도 성 T7 RNA 중합 효소 매개 멀티젠 발현 시스템, pMGX
요약
이 연구는 pMGX 플라스미드 시스템을 사용하여 대장균 에서 단일 플라스미드로부터 다중 유전자의 T7 매개 공동 - 발현을위한 방법을 기술한다.
초록
여러 단백질의 동시 발현은 합성 생물학, 단백질 - 단백질 복합체 연구, 생합성 경로 규명 및 활용에 점점 더 필수적입니다. 이 원고에서, 유도 성 T7 RNA 중합 효소의 조절하에 multigene 합성 오페론의 건설을위한 매우 효과적인 시스템의 사용이 설명된다. 이 시스템은 많은 유전자가 한 플라스미드에서 동시에 발현되도록합니다. 여기서, 암피실린 또는 카나마이신 내성 선택 마커 (A 및 K) 및 N 말단 헥사 히스티딘을 보유하거나 보유하지 않은 4 개의 관련된 벡터, pMGX-A, pMGX-hisA, pMGX-K 및 pMGX-hisK의 세트 태그 (his)가 공개됩니다. 이 벡터 시스템을 이용한 합성 오페론의 구축을위한 상세한 프로토콜은 대응 유전자와 함께 제공되어, 5 개의 유전자를 함유하는 pMGX- 기반 시스템이 용이하게 구축되어 대장균 에서 5 가지 암호화 된 단백질 모두를 생성하는데 사용될 수 있음을 보여준다. 이 시스템em과 프로토콜을 사용하여 연구자는 대장균 에서 복잡한 다중 구성 요소 모듈과 경로를 일상적으로 표현할 수 있습니다.
서문
여러 단백질의 동시 발현은 점점 더 필수적이며, 특히 합성 생물학 분야에서 여러 기능적 모듈이 표현되어야한다. 단백질 단백질 복합체를 연구 할 때, 발현과 기능은 흔히 동시 발현 2 , 3을 필요로 한다. 경로의 각 유전자가 4 , 5 , 6 , 7 , 8 로 표현되어야하는 생합성 경로를 특성화하고 활용하는데있다. 실험실 재조합 단백질 발현을위한 작업 말인 대장균 (Escherichia coli) 에서 특히 공동 발현을위한 많은 시스템이 개발되었다. 예를 들어, 선택성 마커가 다른 여러 플라스미드를 사용하여 다양한 다른 ex 압흔 벡터들 ( 10 , 11) . 다중 단백질 발현을위한 단일 플라스미드 시스템은 각 유전자의 발현을 조절하기 위해 다중 프로모터를 사용 하였다. 다수의 유전자가 단일 전사 물에 암호화되어있는 합성 오페론 2 , 13 ; 또는 어떤 경우에는 궁극적으로 단백질 분해 처리 된 폴리 펩타이드를 암호화하는 단일 유전자로 목적하는 목적 단백질을 생성한다.
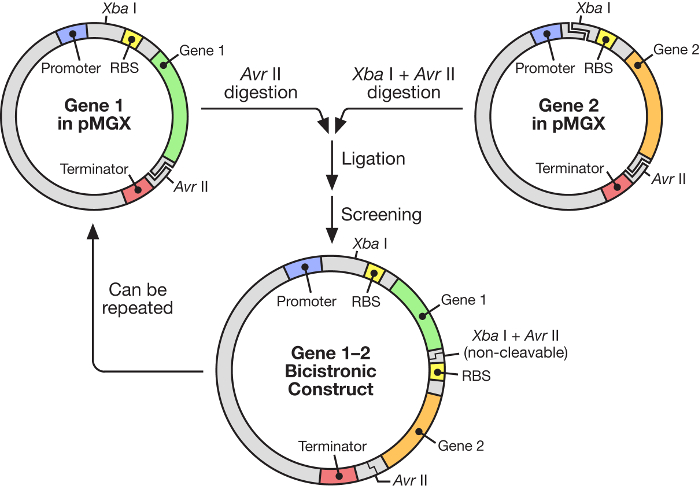
그림 1 : polycistronic vector의 구조를 보여주는 pMGX workflow. pMGX 시스템은 유도 성 T7 프로모터의 제어하에 합성 오페론을 제조하기위한 유연하고 사용하기 쉬운 전략을 제공한다.e.com/files/ftp_upload/55187/55187fig1large.jpg "target ="_ blank ">이 그림의 확대 버전을 보려면 여기를 클릭하십시오.
이 원고에서 유도 성 T7 RNA 중합 효소 ( 그림 1 )의 제어하에 multigene 합성 오페론의 건설을위한 매우 효과적인 시스템의 사용이 설명됩니다. 이 시스템은 많은 유전자가 한 플라스미드에서 동시에 발현되도록합니다. 이것은 원래 pKH22라고 불리는 플라스미드 시스템에 기반을두고 있으며, 여러 가지 다양한 응용 분야에서 성공적으로 사용되고 있습니다. 6 , 7 , 8 . 여기서,이 플라스미드 세트는 4 개의 관련된 벡터, 즉 C- 또는 N- 말단 태그가없는 발현 벡터 인 pMGX-A 및 암피실린 내성 마커를 포함하도록 확대된다. pMGX-hisA, N- 말단 헥사 히스티딘 태그 및 암피실린 내성 마커를 코딩하는 발현 벡터; pMGX-K, 발현 벡터 l임의의 C- 또는 N- 말단 태그 및 카나마이신 내성 마커를 수취하는 단계; 및 N 말단 헥사 히스티딘 태그 및 카나마이신 내성 마커를 코딩하는 발현 벡터 인 pMGX-hisK. 이 연구에서, pMGX 시스템, 특히 pMGX-A를 사용하여 5 개의 유전자를 포함하는 폴리 시스트 론 벡터를 생성하는 방법이 대장균 에서 각각의 개별 단백질의 성공적인 생산과 함께 입증되었다.
프로토콜
1. 관심 유전자 얻기
- 합성 유전자를 디자인하십시오.
- 대장균 발현을위한 유전자 서열을 최적화하십시오.
- 염기 서열 (AvrII, NdeI, EcoRI 및 XbaI)에서 문제가되는 제한 효소 부위를 제거합니다.
- 복제를위한 제한 사이트 통합; 5'-NdeI 사이트 및 3'-EcoRI 사이트가 권장된다. 필요한 경우 다른 사이트를 사용할 수 있습니다. 선택된 플라스미드의 다중 클로닝 영역을 참조하십시오 ( 그림 S1- S4 ). 원하는 경우 Western blot detection을 위해 5 '또는 3'encoding tag를 포함하십시오.
- 상업적으로 설계된 유전자를 주문하십시오.
- 제조 업체의 지시에 따라 무딘 복제 키트를 사용하여 무딘 벡터에 유전자를 무작위로 복제하십시오. 유전자를 증폭시키는 프라이머를 디자인한다 (단계 1.2.2로 진행). 또는 pMGX 플라스미드에 직접 클로닝하기 위해 추가로 5 '및 3'말단을 첨가하고 단계 2로 진행한다. 여기서, th대표적인 결과는 관심있는 유전자를 무딘 복제 한 결과입니다.
- 원하는 유전자를 증폭하십시오 (설계되고 최적화 된 합성 유전자로부터, 또는 원하는 유전자를 포함하는 주형 DNA로부터). 15 .
- 클로닝을위한 제한 사이트가있는 프라이머 디자인; 5'-NdeI 사이트 및 3'-EcoRI 사이트가 권장된다. 필요한 경우 다른 사이트를 사용할 수 있습니다. 선택된 플라스미드의 다중 클로닝 영역을 참조하십시오 ( 그림 S1- S4 ). 원하는 경우 Western blot detection을 위해 5 '또는 3'encoding tag를 포함 시키십시오.
- PCR은 원하는 유전자를 증폭 15 .
- 아가로 오스 겔 전기 영동에 의한 PCR 분석 16 .
- 비특이적 증폭이있는 경우 젤 추출을 통해 증폭 된 유전자를 제거하십시오. 그렇지 않으면 효소 정화 키트를 사용하십시오.
- 흡광도를 확인하여 분광 광도계를 사용하여 DNA를 정량화하십시오260 nm에서의 e; 용리 완충액이없는 blank 17 .
2. 다중 유전자 발현 시스템 벡터 pMGX 18 에 대한 관심 유전자 클로닝
- 제한은 얻은 관심 유전자와 원하는 벡터 인 pMGX를 NdeI 및 EcoRI로 소화시킨다.
참고 : 많은 양의 재 원형화 된 플라스미드를 얻은 경우, EcoRI을 첨가하기 전에 1 시간 동안 NdeI 반응을 진행시킨다.- 0.5-1.5 μg의 DNA와 각 효소 1 μL를 포함하는 40 μL 다이제스트 반응을 적절한 10x 완충액 4 μL와 함께 사용하십시오.
- NdeI 및 EcoRI 소화를 위해 EcoRI 완충액을 사용하고 먼저 NdeI 엔도 뉴 클레아 제를 첨가한다. 37 ° C에서 1 시간 동안 분해하십시오. 그런 다음 EcoRI 엔도 뉴 클레아 제를 첨가하고 다이제스트가 추가 시간 동안 진행되도록하십시오. NdeI는 DNA 말단에 가까운 절단에 민감하므로 EcoRI에 의한 초기 소화는 NdeI에 의한 효과적인 소화를 방해 할 수있다.
- 전기아가로 오스 젤에 제한 다이제스트를 ophorese (1 킬로바이트 유전자, 55 분 동안 110 V에서 0.7 % 아가로 오스 겔을 사용, 다른 유전자 크기에 대한 아가로 오스 겔의 비율을 선택하려면 참조 16 참조하십시오.
- 깨끗한 메스 또는 면도날을 사용하여 삽입 및 벡터 밴드를 잘라 내고 excised gel segment를 1.5 mL 튜브에 넣으십시오.
- 제조 업체의 프로토콜에 따라 젤 추출 키트를 사용하여 아가로 오스 겔에서 DNA를 추출합니다.
- 3 : 1 인서트 - 벡터 비율을 사용하여 관심있는 유전자를 pMGX 벡터에 삽입한다. 삽입물없이 소화가 된 pMGX의 음성 연결을 설정하십시오.
- 1 μL의 T4 DNA ligase, 0.15-0.5 μg의 벡터 DNA (~ 5 μL) 및 3 : 1 insert-to-vector ratio와 크기에 기초한 적절한 양의 insert를 포함하는 20 μL 연결 반응을 설정하십시오 삽입 된 유전자; pMGX 백본의 크기는 5,312 ~ 5,504 bp입니다. 모든 것을 포함하는 부정적인 반응 반응 포함 bu삽입되는 유전자. 음성 대조군 연결 및 벡터 삽입 삽입 반응에서 벡터 DNA의 양이 동등한 지 확인하십시오.
- ligation 반응을 XL1-Blue 화학적으로 유능한 대장균 세포로 연결하고 플라스미드 pMGX- yfg1 (관심 유전자 1 포함), pMGX- yfg2 (관심 유전자 2 포함) ... pMGX- yfgn (관심 유전자 n 함유). 무균 기술 (바이오 안전성 캐비닛 내부 또는 화염 아래)을 사용하십시오.
- 화학적으로 유능한 XL1-Blue E. coli 의 100 μL 분량을 얼음 위에서 5 분 동안 녹인 다음 5 μL의 연결 반응을 추가하십시오. 얼음 위에서 30 분 동안 품어 낸다.
- 42 ° C에서 보관 한 수욕에서 45 초 동안 세포를 가열 충격을 가한 다음 차가운 LB 200 μL를 넣으십시오. 얼음 위에서 2 분 동안 품어 낸다.
- 1 시간 동안 37 ° C, 220 RPM에서 세포를 흔들고, 다음을 포함하는 LB- 한천 플레이트에 플레이트 100 μL을 펼칩니다.priate 선택 마커 (암피실린 또는 카나마이신).
- 양성 형질 전환 체를 스크리닝하십시오.
- 음성 대조군 및 연결 플레이트의 식민지 수를 비교합니다. 1 : 2보다 큰 식민지 비율이 요구됩니다. 음성 대조군 플레이트에 많은 수의 콜로니가 있으면 단계 2.1로 돌아가서 메모를 검토합니다.
- 스크리닝 (1 n)을 위해 삽입 된 다른 유전자의 각 연결 반응으로부터 4-8 개의 개별 콜로니 (음성 대조군에 따라 : 라이 게이션 비)를 선택하고 4 mL LB 및 적절한 항생제 밤새 배양 / 개별 콜로니에 접종한다. 37 ° C 및 220 rpm으로 흔들어서 밤새 배양하십시오.
- 제조 업체의 프로토콜에 따라 플라스미드 DNA 분리 키트를 사용하여 플라스미드 DNA를 분리합니다.
- 적절한 150X 버퍼 2 μL와 150-500 NG DNA 및 각 효소 1 μL를 포함하는 20 μL NdeI + EcoRI 다이제스트 반응을 설정하십시오. 에서NdeI와 EcoRI를 동시에 추가하여 관심 유전자가 제한 효소 사이에 삽입되도록하십시오. 빈 pMGX 벡터로 부정적인 제어가 권장됩니다. 37 ℃에서 2 시간 동안 수욕상에서 소화시킨다.
3. 유전자 1, pMGX - yfg1을 포함하는 pMGX 벡터에 유전자 2 삽입
- 제한 효소는 AvrII 로 pMGX- yfg1 을 소화시키고 종아 장 인산 가수 분해 효소 (CIP)로 치료한다.
- 0.5-1.5 μg의 벡터 DNA (~ 5 ~ 10 μL의 분리 된 DNA)와 1 μL의 Avr II를 포함하는 40 μL 다이 제스트 반응물을 적절한 10x 완충액 4 μL와 함께 사용합니다. 37 ° C에서 1.5 시간 동안 다이제스트 한 다음 CIP 1.5 μL를 첨가합니다. 37 ℃에서 30 분간 더 방치한다.
- 제한 효소 는 AvrII 및 XbaI로 pMGX- yfg2 를 분해하여 관심 유전자를 유리시킨다.
- 0.5-1.5 μg의 DNA와 1 μ를 함유하는 40 μL 분해 반응을 사용하십시오; 4㎕의 적절한 10x 완충액을 갖는 각 효소의 L. 37 ° C에서 2 시간 동안 분해하십시오.
- 전기 영동 제한은 적절한 퍼센트 (0.7 %) 아가로 오스 겔에서 소화되고 깨끗한 메스 / 면도날을 사용하여 삽입 및 벡터 밴드를 제거합니다 (2.2-2.3 단계 참조).
- 제조 업체의 프로토콜에 따라 젤 추출 키트를 사용하여 DNA를 추출하고 DNA를 정량 17 .
- 3 : 1 인서트 - 벡터 비율을 사용하여 pMGX- yfg1에 Ligate 유전자 2를 삽입. 추가 삽입물이없는 pMGX- yfg1 의 음성 연결을 설정하십시오. 2.5 단계에서 위와 같이 설정하십시오.
- 단계 2.6에서 볼 수 있듯이 5 μL의 연결 반응을 XL1-Blue 화학적으로 유능한 대장균 세포로 연결하고 플라스미드 pMGX- yfg1,2 (관심 유전자 1 및 2 포함) 및 음성 pMGX- yfg1 대조 물을 연결 합니다.
- 음성 대조군 및 연결 플레이트의 식민지 수를 비교하십시오. 콜로니 카운트 비율 g1 : 2 이상이 필요합니다. 네거티브 컨트롤 플레이트에 많은 수의 콜로니가 있으면 단계 3.1로 돌아가서 CIP 처리를 검토하십시오.
- 라이 게이션 반응에서 4-8 개별 식민지 (부정적인 제어에 따라 : 결찰 비율)를 선택하고 개별 식민지 당 적절한 항생제 플러스 LB 4 ML을 접종하고 37 ° C와 220 RPM에서 하룻밤 자랍니다.
- 제조 업체의 프로토콜에 따라 플라스미드 DNA 절연 키트를 사용하여 플라스미드 DNA를 분리하고 DNA 17 정량.
- EcoRI로 pMGX- yfg1,2 의 제한 효소 절단을 수행하여 두 번째 유전자의 효과적인 삽입을 스크리닝 하십시오 .
- 150-500 ng의 DNA와 1 μL의 EcoRI 효소와 2 μL의 EcoRI 10x 완충액을 포함하는 20 μL 분해 반응을 사용하십시오. 37 ° C에서 2 시간 동안 분해하십시오.
- 적절한 퍼센트 아가 로스 겔에서 제한 다이제스트를 전기 영동한다; 해당하는 밴드를 찾는다.s를 유전자 2의 크기로 증가시킨다 (2.2 단계 참조). 유전자는 원하지 않는 역전 된 방향으로 벡터에 삽입 될 수 있습니다.
4. 유전자 1과 2, pMGX - yfg1,2를 포함하는 pMGX 벡터에 세 번째 유전자를 추가
- 제한은 AvrII 로 pMGX- yfg1,2 를 다이제스트하고 3.1 단계에서 볼 수 있듯이 CIP로 처리합니다.
- 제한 3.2 절에서 볼 수 있듯이 AvrII와 XbaI를 갖는 pMGX- yfg3 를 소화시킨다.
- 깨끗한 메스 / 면도날을 사용하여 적절한 퍼센트 아가로 오스 겔에서 제한 효소 절단 및 전기 용품 삽입 및 벡터 밴드의 전기 분해; 단계 2.2-2.3을 참조하십시오.
- 겔 추출 키트를 사용하여 아가로 오스 젤에서 DNA를 추출하고 DNA를 계량 17 .
- 3 : 1 삽입 대 벡터 비율을 사용하여 pMGX- yfg1,2 로 Ligate 유전자 3을 삽입하고 단계 3.5에서 볼 수 있듯이 추가 삽입없이 digested pMGX- yfg1,2 의 음성 연결을 설정합니다.
- 5 μL의 연결 반응을 XL로 전환1-Blue 화학적으로 유능한 대장균 세포, 단계 2.6에서 볼 수 있듯이 플라스미드 pMGX- yfg1,2,3 (관심 유전자 1, 2 및 3 포함) 및 음성 pMGX- yfg1,2 컨트롤을 연결했다.
- 음성 대조군 및 연결 플레이트의 식민지 수를 비교하십시오. 네거티브 컨트롤 플레이트에 많은 수의 콜로니가있는 경우 4.1 단계로 돌아가서 CIP 처리를 검토하십시오.
- 라이 게이션 반응에서 4 ~ 8 개의 개별 콜로니 (음성 대조군에 따라 결찰 비율에 따라)를 선택하고 개별 콜로니 당 적절한 항생제 + LB 4mL를 접종하십시오. 37 ° C와 220 rpm으로 밤새 자랍니다.
- 제조 업체의 프로토콜에 따라 플라스미드 DNA 분리 키트를 사용하여 플라스미드 DNA를 분리합니다.
- 단계 3.10에서 볼 수 있듯이 EcoRI로 pMGX - yfg1,2,3 의 제한 다이제스트를 수행하여 세 번째 유전자의 효과적인 삽입을 스크리닝 하십시오 .
- 적절한 퍼센트 아가 로스 겔에서 제한 다이제스트를 전기 영동한다; 보기유전자 2와 유전자 3의 크기에 상응하는 밴드 (2.2 단계 참조). 참고 : 유전자 3은 원하지 않는 반대 방향으로 벡터에 삽입 될 수 있습니다. 유전자 2와 3이 같은 크기라면, 다른 적당한 제한 다이제스트 사이트가 스크리닝을 위해 선택되어야한다.
참고 : 각 새 유전자에 대해 필요한만큼 반복하십시오.
5. Multigene 발현 시스템을 사용하여 관심있는 단백질 생산 및 Western Blotting에 의한 생산 평가
- 관심있는 모든 유전자를 포함하는 양성 클론을 BL21- (λDE3)과 같은 화학적으로 유능한 단백질 생산 대장균 으로 변형시킵니다.
- 화학적으로 유능한 BL21 - (λDE3) 대장균 의 100 μL 분량을 얼음에 5 분 동안 녹인 다음 1 μL의 양성 복제 된 플라스미드 DNA를 첨가하십시오. 얼음 위에서 30 분 동안 품어 낸다.
- 42 ° C로 유지 수조에서 45 초 동안 세포를 가열 충격을 가한 다음 차가운 LB 200 μL를 추가합니다. 얼음 위에서 2 분 동안 품어 낸다.
- 1 시간 동안 37 ° C와 220 RPM에서 세포를 흔들고 적절한 선택 마커 (암피실린 또는 카나마이신)가 들어있는 LB- 한천 플레이트에 플레이트 100 μL을 펴십시오.
- isopropyl-β-D-1- thiogalactopyranoside (IPTG) 유도로 단백질을 발현하십시오.
- B21 - (λDE3) 변형 플레이트에서 격리 된 식민지를 선택하고 적절한 항생제를 더한 LB 4 ML을 접종하십시오. 37 ° C 및 220 rpm으로 흔들어 밤새 성장.
- 하룻밤 배양 1 ML을 사용하여 적절한 항생제 문화 플러스 100 ML LB를 예방 접종.
- OD 600 이 0.6이되도록 220 rpm으로 흔들어 주면서 37 ° C에서 성장시킵니다.
- 100 μM IPTG로 배양하고 25 ° C 및 220 rpm에서 15 시간 동안 성장시킵니다.
- 문화 1 ML을 제거하고 1 분 13,000 XG에서 원심 분리기; 상등액을 버린다.
- 제조 업체의 지침에 따라 용해 솔루션을 사용하여 세포를 Lyse용해성 세포 용 해물의 웨스턴 블럿을 수행하여 모든 단백질이 성공적으로 생산되었는지 여부를 확인하십시오 19 .
결과
이 연구에서 목표는 단일 플라스미드에서 5 개의 단백질을 동시에 발현시키는 것이 었습니다. N- 또는 C- 말단 헥사 히스티딘 태그를 암호화하는 5 개 코돈 최적화 된 합성 유전자 단편은 상업적으로 구매되었다. 합성 유전자를 PCR에 의해 증폭시키고 개별적으로 PCR-blunt 벡터에 클로닝하고 서열을 결정 하였다. 폴리 시스 트론 플라스미드를 생성하기 위해, 관심있는 5 개의 ?...
토론
복합 유전자의 동시 발현은 특히 복잡하고 다중 유전자 대사 경로 3 , 4 , 5 를 특성화하고 재구성하는 데 점점 더 필수적입니다. pMGX 시스템은 대장균 루틴 6 , 7 , 8 에서 다중 유전자 동시 발현을 만들고 다양한 연구자가 접근 할 수 있도록한다. 이 연?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 연구는 캐나다의 자연 과학 및 공학 연구위원회 (Natural Science and Engineering Research Council of Canada)의 지원을 받았다.
자료
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50 x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm X 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
참고문헌
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -. J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998 (2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016)
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp Available from: https://www.jove.com/science-education/5065/the-western-blot (2016)
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유