Method Article
Purificación y análisis de un anticuerpo monoclonal de células de ovario de hámster chino utilizando un sistema automatizado de microbiorreactores
En este artículo
Resumen
Se ha descrito un protocolo detallado para la purificación y posterior análisis posterior de un anticuerpo monoclonal a partir del fluido de cultivo celular cosechado (HCCF) de microbiorreactores automatizados. También se presenta el uso de análisis para determinar los atributos de calidad críticos (CQA) y maximizar el volumen de muestra limitado para extraer información vital.
Resumen
Los anticuerpos monoclonales (mAbs) son uno de los productos biológicos más populares y bien caracterizados fabricados hoy en día. La mayoría de las células de ovario de hámster chino (CHO), las condiciones de cultivo y proceso deben optimizarse para maximizar los títulos de anticuerpos y lograr perfiles de calidad objetivo. Normalmente, esta optimización utiliza biorreactores automatizados a microescala (15 ml) para examinar varias condiciones de proceso en paralelo. Los criterios de optimización incluyen el rendimiento del cultivo y los atributos de calidad críticos (CQA) del producto de anticuerpos monoclonales (mAb), que pueden afectar su eficacia y seguridad. Las métricas de rendimiento del cultivo incluyen el crecimiento celular y el consumo de nutrientes, mientras que los CQA incluyen los perfiles de n-glicosilación y agregación del mAb, variantes de carga y peso molecular. Este protocolo detallado describe cómo purificar y posteriormente analizar las muestras de HCCF producidas por un sistema automatizado de microbiorreactores para obtener valiosas métricas de rendimiento y salidas. En primer lugar, se utiliza un método automatizado de cromatografía líquida de proteína sin proteínas (FPLC) para purificar el mAb de las muestras de cultivo celular cosechadas. Una vez concentrados, los perfiles glicanos son analizados por espectrometría de masas utilizando una plataforma específica (consulte la Tabla de Materiales). Los pesos moleculares de anticuerpos y los perfiles de agregación se determinan utilizando cromatografía de exclusión de tamaño-dispersión de luz angular múltiple (SEC-MALS), mientras que las variantes de carga se analizan utilizando electroforesis de zona capilar de microchip (mCZE). Además de las métricas de rendimiento del cultivo capturadas durante el proceso del biorreactor (es decir, viabilidad del cultivo, recuento celular y metabolitos comunes, como glutamina, glucosa, lactato y amoníaco), se analizan los medios gastados para identificar los nutrientes limitantes mejorar las estrategias de alimentación y el diseño general del proceso. Por lo tanto, también se describe un protocolo detallado para la cuantificación absoluta de aminoácidos por cromatografía líquida-espectrometría de masas (LC-MS) de medios gastados. Los métodos utilizados en este protocolo aprovechan las plataformas de alto rendimiento que son compatibles con un gran número de muestras de pequeño volumen.
Introducción
Las terapias con proteínas se utilizan para tratar una creciente variedad de condiciones médicas, incluyendo complicaciones de trasplante de tejido, trastornos autoinmunes y cánceres1. Desde 2004, la Administración de Alimentos y Medicamentos de los Estados Unidos (USFDA) ha documentado una proporción cada vez mayor de solicitudes de licencias biológicas (BLA) de todas las aprobaciones reguladas por el Centro de Evaluación e Investigación de Medicamentos (CDER), y los BLA representan más del 25% en 2014 y 20152.
Teniendo en cuenta este mercado en expansión, los fabricantes de productos biofarmacéuticos se enfrentan al desafío de entregar rápidamente más productos con una calidad constante. Los esfuerzos para aumentar el rendimiento de los productos se han centrado en la ingeniería celular CHO y el cribado de líneas de producción, aunque las mejoras más significativas se deben a los avances en la optimización de la estrategia de medios/alimentación y los controles ambientales del cultivo celular1, 3 , 4 , 5 durante el proceso de fabricación.
Dado que los mAbs se producen en un sistema biológico, puede haber variabilidad proteica inherente. La composición de anticuerpos puede alterarse después de la traducción, como la glicosilación o afectada por la degradación o las reacciones enzimáticas. Estas variaciones estructurales pueden provocar reacciones inmunitarias peligrosas o alterar la unión de anticuerpos, lo que a su vez puede reducir o eliminar la función terapéutica prevista5. Por lo tanto, los atributos de calidad crítica (CQA) de anticuerpos monoclonales - perfil De N-glicano, distribución de variantes de carga y el porcentaje de anticuerpos en forma monomérica - se supervisan y controlan regularmente como parte de un enfoque de Calidad por Diseño (QbD) durante el procesos de fabricación1,6. En un entorno de producción regulado, las proteínas terapéuticas deben cumplir los criterios de aceptación para obtener una licencia como medicamento comercial aprobado7. Los métodos presentados aquí serían típicamente parte del procesode caracterización de calidad para un anticuerpo 7,8,y cualquier científico de proteínas estará familiarizado con su uso.
En eltrabajo anterior 9, se ha descrito la aplicación y operación de microbiorreactores para el cribado de alto rendimiento de las condiciones de cultivo celular en el bioprocesamiento ascendente. El producto purificado obtenido a partir de las diferentes condiciones de los medios se somete a análisis de N-glicano utilizando LC-MS. Los patrones de glicosilación de proteínas terapéuticas pueden detectarse y caracterizarse utilizando las técnicas LC-MS10,11,y la la presencia de varias especies de glicanos se ha relacionado con parámetros de bioprocesos como la estrategia de alimentación, el pH y la temperatura12. El efecto de las diferentes condiciones de los medios en la calidad del producto, indicado por el porcentaje de la IgG resultante en forma monomérica, también se evalúa con Cromatografía de Exclusión de Tamaño- Dispersión de Luz Multi-Angle (SEC-MALS)13,14 , 15. El perfil de variante de carga representa una serie de modificaciones16 que podrían afectar a la función de un producto. La electroforesis de zona microcapilar (mCZE) es una técnica que ofrece un tiempo de análisis considerablemente más rápido en comparación con los métodos de cromatografía de intercambio de cationes tradicionales (CEX) y enfoque isoeléctrico capilar (cIEF) utilizados para el análisis de variantes de carga17 ,18. Se analizaron los medios de biorreactor gastados para realizar un seguimiento del consumo de aminoácidos durante la producción de proteínas en relación con los cambios en los atributos identificativos del anticuerpo19,20,21,22 , 23.
El análisis de proteínas nos permite identificar parámetros críticos del proceso (CCP) en función de las relaciones entre las entradas del proceso y los cambios en los CQA. Durante el desarrollo de bioprocesos, la identificación y medición de CPPs demuestra fundamentalmente el control del proceso y garantiza que el producto no ha cambiado, lo que es esencial en entornos de fabricación altamente regulados. En este artículo, se presentan técnicas analíticas para medir algunas de las características bioquímicas de la proteína más pertinente para los CQA del producto (perfil de Glicano, variantes de carga y homogeneidad de tamaño).
Protocolo
1. Purificación de anticuerpos
NOTA: El tampón de equilibrio para el anticuerpo interno es de 25 mM Tris, 100 mM NaCl, pH 7.5. El tampón de elución utilizado es de 0,1 M de ácido acético. Los tampones y la resina (Proteína A) dependen del anticuerpo específico purificado. El volumen de la columna es equivalente a la altura del lecho de la resina. La cantidad de fase móvil utilizada se determina en términos de volumen de columna.
- Inicialización del sistema de purificación
- Abra el software conectado al sistema de purificación. Mediante instrucciones manuales, equilibre la columna con el búfer de equilibrio a un caudal de 2 ml/min durante 40 minutos.
- En el colector de fracciones, coloque tubos cónicos de 15 ml para recoger eluato de anticuerpos purificados y tubos cónicos de 50 ml para recoger el flujo a través durante el lavado con alto contenido de sal. Asegúrese de que el colector de fracciones se restablezca a la posición inicial abriendo y cerrando el colector de fracciones antes del comienzo de la ejecución. El colector de fracciones se mantiene a 7 oC.
NOTA: El colector de fracciones se puede restablecer manualmente en la pestaña Colector de fracciones en Configuración, para tubos de 15 ml y 50 ml.
- Inyección de muestra
NOTA: El fluido de cultivo celular cosechado utilizado en los siguientes procedimientos se ha obtenido de células de ovario de hámster chino cultivadas en microbiorreactores automatizados9.- Agregue el fluido de cultivo de células de cosecha filtrada de 0,22 m a una jeringa vacía de 12 ml cuyo extremo de la boquilla esté tapado.
- Sosteniendo la jeringa con la boquilla hacia abajo, inserte el émbolo de la jeringa hasta que haya una pequeña porción del émbolo. Asegúrese de que el líquido no tenga fugas, gire la jeringa con la boquilla hacia arriba y retire la tapa.
- Todavía sosteniendo la jeringa con la boquilla hacia arriba, empuje el cilindro para disipar cualquier aire hasta que el fluido de cultivo celular esté en la punta de la boquilla. Inserte la boquilla de la jeringa en el puerto de inyección manual del sistema de purificación y gire para apretar.
- Empuje hacia abajo el émbolo hasta que se inyecte toda la muestra y sea visible en el bucle de muestra de gran volumen de 10 ml.
- Abra el archivo de método guardado. Guarde el archivo de resultados en la ubicación necesaria y especifique el nombre del archivo cuando se le solicite. Pulse ejecutar después de que la muestra se haya inyectado en el bucle de muestra de gran volumen.
- Ejecución del método de purificación
- Seleccione el método guardado y haga clic en Ejecutar cuando se lo solicite el software del instrumento (paso 1.2.5).
NOTA: El sistema está configurado para ejecutar los pasos siguientes. El usuario no necesita hacer nada mientras el instrumento se está ejecutando. - Equilibrar la columna con tres volúmenes de columna (CSV) de búfer de equilibrio a un caudal de 2 ml/min. Una vez que la columna se equilibra, el sistema, utilizando el bucle de muestra de gran volumen, inyectará la muestra en la columna a un caudal de 1 ml/min.
- Una caída en la señal UV a 280 nm indica que la muestra ha terminado de cargarse. Lave la columna con búfer de equilibrio a un caudal de 2 ml/min hasta que la señal UV caiga por debajo de 25 mAU.
- Utilice cuatro CV de Tris de 25 mM con 1 M de NaCl a pH 7,5 para realizar un lavado secundario de alta sal a un caudal de 2 ml/min. El colector de fracciones del sistema recogerá cualquier proteína/ADN que salga de la columna durante el lavado de sal en tubos de 50 ml.
- Aplique cinco CV de búfer de elución a un caudal de 1 ml/min para eluir el anticuerpo de la columna. Recoger el eluido en tubos de 15 ml basados en la señal UV; cuando la señal UV 280 está por encima de 35 mAU, comienza la recolección; la recolección termina cuando la señal cae por debajo de 50 mAU; esto se denomina corte máximo.
NOTA: El corte máximo garantiza la normalización de los perfiles de elución y para evitar el relave máximo de elución que puede contener agregados proteicos24. - Lave la columna con tres CV de búfer de equilibrio. La carrera termina después del paso de lavado.
- Después de la elución neutralizar inmediatamente la proteína purificada usando 1 M Tris base a un pH de 5,5 euros. Mida la concentración de proteínas utilizando un espectrofotómetro UV-Vis de microvolumen a 280 nm y 260 nm y guárdela a 4oC.
- Concentrar el anticuerpo purificado utilizando unidades centrífugas (paso 2). A continuación, someta el anticuerpo purificado al análisis de glicano utilizando LC-MS y después del análisis del perfil de agregación usando SEC-MALS (pasos 3 y 4).
NOTA: El anticuerpo purificado no debe congelarse sin un mayor intercambio de búferes, ya que los ciclos de congelación-descongelación frecuentes pueden causar agregación y precipitación.
- Seleccione el método guardado y haga clic en Ejecutar cuando se lo solicite el software del instrumento (paso 1.2.5).
2. Concentración de anticuerpos purificados
NOTA: El tampón Tris-acetato es ácido acético de 0,1 M neutralizado con 1 M Tris Base a un pH de 5,5 euros.
- Inserte filtros de 100 kDa en tubos de centrífuga.
- Lavar los filtros con 500 ml de agua destilada doble. Centrífuga durante 10 min a 14.000 x g a temperatura ambiente (RT). Repita este paso dos veces. Deseche el filtrado.
- Transfiera los filtros enjuagados a tubos de centrífuga frescos y agregue 500 ml de muestra a cada filtro. Centrífuga durante 10 min a 14.000 x g.
- Invierta el filtro en un tubo de giro fresco. Centrífuga durante 2 min a 1.000 x g para recoger la muestra concentrada.
- Determinar las concentraciones de la muestra utilizando un espectrofotómetro UV-Vis. Enblanco el espectrofotómetro utilizando una solución de tampón de tris-acetato. Utilice un coeficiente de extinción proteica de 1,37 mL•(mg•cm)-1 a 280 nm para una solución de IgG al 1% (% m/v).
- Utilice la muestra concentrada para preparar una solución de trabajo de 12,5 ml de 2 mg/ml para el análisis de glicano y 30 ml de solución de trabajo de 3,5 mg/ml para SEC-MALS.
NOTA: El protocolo se puede pausar aquí. Las muestras deben refrigerarse a 4 oC. A una concentración de 2 mg/ml, estas muestras deben ser estables durante al menos tres meses a 4 oC, mientras que las concentraciones más altas pueden precipitarse.
3. Análisis de Los N-glicanos utilizando espectroscopia de masas
- Etiquetado y aislamiento de N-Glycan
- Comience con concentraciones de anticuerpos de 2 mg/ml en un tampón adecuado, como fosfato sódico neutro, citrato o tampón HEPES. Preparar un estándar mAb intacto (como NIST mAb) a 2 mg/ml para procesar junto con las muestras experimentales para servir como un control positivo.
NOTA: Los anticuerpos deben estar en un tampón final que no contenga SDS y menos de 0,1 mM de nucleófilos (como Tris, TDT, glicina o histidina). SDS en el búfer de muestra debe eliminarse. Si los nucleófilos están en el tampón, entonces diluirlos o realizar un intercambio de tampón ya que interferirán con el kit. El protocolo general se proporciona con el kit de glicano. - Diluir 7,5 ml de los anticuerpos con 15,3 l de agua de grado LC-MS en tubos de 1 ml suministrados con el kit y luego desnaturalizar utilizando 6 ml de solución del 5% de un tensioactivo apto para enzimas y mS a 90 oC durante 3 min.
- Enfríe las muestras durante 3 minutos a temperatura ambiente (RT). A continuación, añadir 1,2 éL de PNGase F e incubar durante 5 min a 50 oC.
- Después de enfriar 3 min a RT, etiquete los N-glicanos colados añadiendo 12 l de reactivo de etiquetado fluorescente disuelto en dimetilformamida anhidra (DMF) y espere 5 min. Diluir la mezcla n-glicana etiquetada con 358 ol de acetonitrilo (ACN).
- Coloque una placa de cromatografía de interacción hidrófila (HILIC) en un colector de vacío con cuñas y bandeja de residuos. Utilice una pipeta multicanal para un gran número de muestras.
- Acondicionar los pozos con 200 ml de agua, donde se ajusta el vacío para que el líquido tome 15-30 s para pasar a través de la resina HILIC. Equilibrar con 200 l de ACN 85% antes de cargar la mezcla de glicano con etiqueta diluida a ACN (400 l), aplicando el vacío después de que cada nuevo líquido se añada a los pozos. Lavar la resina con 600 ml de ácido fórmico al 1% (FA)/90% de ACN dos veces.
- Sustituya la bandeja de residuos por tubos de recogida de 600 ml. Eluye los Nglicanos etiquetados con tampón de elución SPE (3 eluciones de 30 ol cada uno) en los tubos de recogida. Diluir las eluciones agrupadas con 310 ml de diluyente de muestra DMF/ACN. Pipetear las muestras en viales de toma de muestras automáticas para estar listos para el análisis de fluorescencia (FLR)-MS.
NOTA: Estas muestras son estables cuando se almacenan a -80 oC durante al menos 1 mes. Guarde la placa HILIC en su embalaje original, encintada cerrada y dentro de un desecador para su uso futuro.
- Comience con concentraciones de anticuerpos de 2 mg/ml en un tampón adecuado, como fosfato sódico neutro, citrato o tampón HEPES. Preparar un estándar mAb intacto (como NIST mAb) a 2 mg/ml para procesar junto con las muestras experimentales para servir como un control positivo.
- Análisis de LC-MS de N-glicanos etiquetados
- Analice las muestras de elución N-glicanas etiquetadas en un sistema de cromatografía líquida de ultra rendimiento (UPLC) acoplado a un detector de fluorescencia y espectrómetro de masas de tiempo de vuelo (Q-ToF) cuadrúpedos. Utilice una columna aprobada para la separación cromatográfica de los glicanos etiquetados y caliente a 60 oC durante las separaciones.
NOTA: La columna debe lavarse con 60% de acetonitrilo y 40% de H2O antes de su uso: 50 CV antes del primer uso o 20 CV si la columna se ha utilizado antes.- Utilice formato de amonio de 50 mM (AmF) (hecho con concentrado de fase móvil) y ACN 100% de grado LC-MS para las fases móviles. El AmF es sensible a los cambios de pH y se puede utilizar durante 1 mes después de la mezcla. Establezca el caudal inicial en 0,4 ml/min, con el gradiente LC que proporciona un aumento de AmF durante la fase de elución.
- Ajuste el detector FLR para medir a EX 265/EM 425 nm con una frecuencia de muestreo de 2 Hz. Ajuste el Q-ToF al modo de sensibilidad ion positiva MS1, con un rango de masa de 100-2.000 daltons (Da), un tiempo de escaneo de 0,25 segundos y adquisición continua de datos. Utilice la enkephalin de leucina (2 ng/L en 50% ACN/0.1% FA) para la referencia de masa interna, en el modo "No aplicar corrección".
NOTA: La corrección de referencia en masa interna se aplicará más adelante durante el procesamiento de datos. - Vuelva a colocar la escalera de dextran secuencialmente en 22,5 l de H2O, 25 ml de DMF y 52,5 l de ACN. Preparar alícuotas de 10 ml para su almacenamiento a -80 oC, ya que la escalera no es estable durante más de 24 h a temperaturas más altas (temperatura ambiente, 4 oC). La escalera de deshielo se degrada después de más de un ciclo de congelación de saw.
- Coloque las muestras en el tomamuestras automáticos a 10 oC. Cargue un vial de escalera de dextran junto con muestras, ya que la información de tiempo de retención de la escalera se utilizará para las asignaciones mientras que la información de masa utilizada para validar las identificaciones. Utilice inyecciones de 10 l para las muestras e inyecciones de 7,5 l para la escalera. Inyectar muestras en triplicado. Ejecute el método cargado.
- Analice las muestras de elución N-glicanas etiquetadas en un sistema de cromatografía líquida de ultra rendimiento (UPLC) acoplado a un detector de fluorescencia y espectrómetro de masas de tiempo de vuelo (Q-ToF) cuadrúpedos. Utilice una columna aprobada para la separación cromatográfica de los glicanos etiquetados y caliente a 60 oC durante las separaciones.
- Identificación de N-Glycan para datos LC-MS
- Realice el procesamiento de datos con un programa optimizado para datos de espectrometría de masas de fluorescencia de cromatografía de interacción hidrófila (HILIC-FLR-MS).
- Aplique correcciones internas de referencia de masa dentro del programa. Designe las inyecciones de escalera de desmedidas como "Estándar" en Información de muestra. En el método de análisis, establezca los tiempos de retención del compuesto de separación en los de los compuestos de escalera que se detectaron durante la ejecución.
- Para asegurarse de que se devolverá el % de área para los glicanos identificados, modifique el método de análisis: en la ficha Procesamiento, haga clic en Configuración de cuantificación - Calibrar y establezca "Tipo de ajuste de curva de calibración" en "Respuesta relativa (%)".
4. Análisis de la agregación de anticuerpos utilizando SEC-MALS
- Preparación de muestras
- Transfiera la proteína diluida de 3,5 mg/ml (paso 2) a un vial con un inserto de vidrio de 150 ml. Utilice una punta de carga de gel para pipetear en la campana inferior de la plaquita para evitar la introducción de burbujas.
- Tapar el vial con una tapa de septa y analizar inmediatamente. Conservar a 4oC si se analiza más tarde.
- Configuración y equilibrio SEC-MALS
NOTA: Analice la agregación en SEC-MALS configurado con una Cromatografía Líquida de Ultra Alta Presión (UHPLC) con un Detector MALS y un Detector de índice refractario controlado por el software MALS.- Configurar un archivo de método en el software UHPLC para el control del sistema UHPLC, ajustando el caudal a 0,4 ml/min con una fase móvil de 1x fosfato tamponado salino (PBS) (diluido de 10x), el volumen de inyección a 5 sL, la temperatura de la columna a 25 oC , y el detector de matriz de diodos (DAD) para supervisar 280 nm. Establezca el tiempo de ejecución en 20 min. Equilibrar el sistema durante al menos 4 horas antes de cualquier análisis de muestra.
- La interfaz entre los detectores UHPLC y Multi Angle Light Scattering - Refractive Index (MALS-RI) requiere el uso de la salida analógica en el DAD. Establezca la atenuación DAD en 1.000 mAU en el archivo de método DAD y la configuración AU/UV en 1 (instrumento UV>Canales>Canal 1).
- Encienda la lámpara DAD 30 minutos antes de comenzar el análisis y ajuste la longitud de onda a 280 nm. Al mismo tiempo, purgue la celda de referencia del índice de refracción (RI) durante 15 minutos o hasta que la línea base sea estable y, a continuación, cierre la celda de referencia.
- Configure la secuencia de software SEC-MALS, ajustando el tiempo de recogida a 12 min, el volumen de inyección a 5 l, el dn/dc a 0,185 ml/g, el coeficiente de extinción A280 si se ha determinado previamente experimentalmente o a 1,37 ml*(mg*cm) -1 , y la concentración del coeficiente de extinción A280 si se ha determinado previamente experimentalmente o a 1,37 ml*(mg*cm) -1 , y la concentración del coeficiente de extinción A280 si se ha determinado previamente experimentalmente o a 1,37 ml*(mg*cm) -1 , y la concentración del coeficiente de extinción A280 si se ha determinado previamente experimentalmente o a 1,37 ml*(mg*cm)-1, y la concentración del Muestra. Haga clic en Ejecutar y espere a que aparezca en la pantalla el cuadro de diálogo "esperando inyectar".
NOTA: El coeficiente de extinción A280 es específico de la proteína de interés y debe determinarse experimentalmente. - Configure una lista de muestra en el software UHPLC en el mismo orden que en el software MALS-RI y envíe.
NOTA: Es importante ejecutar una comprobación de idoneidad del sistema antes y después de una ejecución. Por lo general, la albúmina sérica bovina se utiliza para comprobar si hay un pico de ensanchamiento, una señal de que la columna sec puede necesitar limpieza o reemplazo. La misma inyección estándar de BSA se puede utilizar para especificar la alineación de la señal, el ensanchamiento de picos y la normalización del detector.
-
Análisis agregado con software MALS
- Haga clic en la pestaña marcada Procedimientos. Especifique el nivel mínimo de desprecio requerido; ninguno suele ser suficiente.
- Verifique que las líneas base se hayan dibujado correctamente y ajuste si es necesario, para los canales LS1, LS2, LS3, RI y UV. Establezca el área máxima de interés.
- Revise la distribución de masa molecular para confirmar que los picos llamados contienen partículas de tamaño similar.
5. Análisis de variantes de carga
- Preparación y etiquetado de la muestra
- Comience con 80 ml de una solución de anticuerpos de 3,5 mg/ml. Desalla rinde la muestra utilizando una columna de desalación de 0,5 ml (7k MWCO). Prepare la columna rompiendo primero el tapón inferior, luego aflojando el tapón superior y colocándolo en un tubo de microcentrífuga de 1,7 ml. Centrifugar la columna de dessalación durante 1 min a 1.500 x g.
NOTA: Marque el exterior de la columna con un punto para que se pueda colocar en la orientación original para los pasos siguientes. - Transfiera la columna a un nuevo tubo de microcentrífuga. Añadir los 80 s de proteína diluida a la parte superior de la columna. Alinee la columna con la orientación original. Centrífuga durante 2 min a 1.500 x g. Retire la muestra de la centrífuga, deseche la columna de desalación y mezcle bien la muestra.
NOTA: La desalación sólo es necesaria si la matriz de muestra contiene aminas primarias, excipientes que perturbarán la electroforesis de la muestra u otras sustancias incompatibles. - Diluir la muestra a una concentración final de 2 mg/ml en un volumen de 25 ml y añadir 5 l del tampón de etiquetado (ver Tabla de materiales: Kitde reactivos de variante de carga) en la placa de 96 pocillos. Preparar el reactivo de etiquetado diluyendo la cantidad necesaria de reactivo de etiquetado (ver Tabla de Materiales: Kitde Reactivo de Variante de Carga) 1:30 en dimetilformamida. Incubar la muestra durante 10 minutos a temperatura ambiente lejos de la luz.
NOTA: Es importante descongelar y luego utilizar inmediatamente este reactivo y utilizarlo dentro de los 10 minutos de la mezcla con DMF. - Después de la incubación, añadir 60 l de agua de grado reactivo y mezclar bien mediante pipeteo. Cubra la placa con un sello de placa y centrifugar la placa a 1.000 x g durante 1 min.
- Comience con 80 ml de una solución de anticuerpos de 3,5 mg/ml. Desalla rinde la muestra utilizando una columna de desalación de 0,5 ml (7k MWCO). Prepare la columna rompiendo primero el tapón inferior, luego aflojando el tapón superior y colocándolo en un tubo de microcentrífuga de 1,7 ml. Centrifugar la columna de dessalación durante 1 min a 1.500 x g.
- Preparación del chip de variante de carga
- Prepare el chip Charge Variant quitando la solución de almacenamiento y lavando los pozos 1, 3, 4, 7, 8 y 10 con agua. A continuación, sustituya el agua por el búfer de funcionamiento pH 7.2 (consulte Tabla de materiales: Kitde reactivos de variante de carga).
- Agregue 750 sl de pH 7.2 tampón en marcha al tubo tampón y coloque el tubo tampón en el punto indicado en la esquina superior izquierda de la bandeja de muestra. Ahora retire el sello de la placa de la placa de 96 pocillos, presione Descargar placa en la interfaz de usuario del instrumento e inserte la placa en la bandeja de muestra GXII.
NOTA: para este análisis se utilizaron búferes de pH 7.2. pH 5.6-7.2 tampones se pueden utilizar dependiendo de la proteína pI. Cuando se utilizan búferes de pH más bajos, pueden ser necesarios tiempos de ejecución de la muestra más largos. - Pulse el botón Descargar chip en la interfaz de usuario. Asegúrese de que los electrodos estén libres de partículas y, si no, limpie con un hisopo libre de pelusas. Al insertar el chip asegúrese de que la ventana en el centro de la viruta está libre de partículas o manchas. Si es necesario, límpielo con un paño suave sin pelusas.
NOTA: Cuando trabaje con chips de electroforesis capilar, retire el tampón por aspiración al vacío, seguido de la adición inmediata de la siguiente solución para evitar que los pozos se sequen. Para minimizar la introducción de burbujas, practique la técnica de pipeteo inverso. Al manipular el chip, tenga en cuenta el frágil capilar que se extiende desde la parte inferior del chip, asegurándose de que no se seque y no se rompa a través de la manipulación brusca. - Cierre la tapa de la cámara de viruta y seleccione el ensayo HT Protein Charge Variant. Haga clic en el botón Ejecutar. Siga las indicaciones para seleccionar los pozos de muestra, el tipo de placa, el tiempo de ensayo (68, 90 o 100 s) y el nombre de archivo. Haga clic en Inicio al final de las solicitudes.
- La limpieza de la viruta requiere el lavado de cada pozo 2veces con agua, seguido de la adición de búfer de almacenamiento (ver Tabla de materiales: Kitde reactivos de variante de carga). Una vez en el búfer de almacenamiento, reemplace el chip en el instrumento y, cuando se le solicite, seleccione el ensayo de carga de proteína HT. En la pantalla principal, seleccione Lavar en la interfaz de usuario. Una vez completado, retire el chip, limpie los electrodos con agua y un hisopo libre de pelusas, y guarde el chip a 4 oC.
- Análisis de variantes de carga
- Abra el software de análisis de instrumentos. Importe la ejecución yendo a Archivo>Importar archivode datos ... y haciendo clic en el archivo *.gxd deseado. Sólo el nombre se transferirá al software, por lo que cambiar el nombre de los pozos es ventajoso (Herramientas> Editorde nombres de muestra). Seleccione los archivos que desea exportar manteniendo pulsada la tecla Mayús mientras selecciona los archivos. Haga clic en Archivo>Exportar... y seleccione el cuadro Datos sin procesar y, a continuación, el cuadro Formato AIA.
- Abra la pestaña Examinar proyectos dentro del software de análisis. Haga clic en Base de datos>Importar datos... y seleccione el *exportado. CDF.
- Una vez importado, vaya a la pestaña Inyecciones, seleccione los archivos que desea analizar, haga clic con el botón derecho y vaya a Procesar... En la ventana que aparece, active la casilla de verificación situada junto a Procesar y seleccione el cuadro de radio "Usar método de procesamiento especificado" y el método de procesamiento deseado en el cuadro desplegable. En el cuadro desplegable inmediatamente debajo de la etiqueta "Cómo:" seleccione Calibrar y cuantificar. Una vez procesado, vaya a la pestaña Resultados y compruebe la integración de los cromatogramas.
NOTA: El método de procesamiento debe verificarse para cada método. Como punto de partida, los parámetros utilizados para el método de procesamiento actual se incluyen en el archivo complementario.
NOTA: La exportación de los datos se puede realizar en forma de informe o solo se puede exportar la cuantificación máxima. Estos se pueden hacer al mismo tiempo que el procesamiento o desde la ventana de resultados.
6. Análisis de aminoácidos
- Configuración de la curva estándar para la cuantificación absoluta de aminoácidos por LC-MS
- Preparar la mezcla de aminoácidos extendidos (EAA) disolviendo 59,45 mg de Asn, 59,00 mg de Hyp, 65,77 mg de Gln y 91,95 mg de Trp en 25 ml de 0,1 N HCl. La concentración final de cada aminoácido en la mezcla de EAA es de 18 nmol/L.
- Preparar la solución de stock estándar interno (ISTD) disolviendo 58,58 mg de Nva y 44,54 mg de Sar en 50 ml de HCl.
- Preparar los estándares completos de aminoácidos combinando la solución de aminoácidos que contiene Ala, Asp, Arg, Cys, Glu, Gly, His, Ile, Leu, Met, Phe, Pro, Ser, Thr, Trp, Tyr, Val a 1 nmol / L cada uno con la mezcla de eAA para concentraciones finales de aminoácidos de 900 , 225, 90, 22,5 y 9 pmol/L. Añadir el stock ISTD preparado a las normas de aminoácidos para una concentración final de 90 pmol/L o 900 pmol/L para crear estándares internos "bajos" y "altos" para utilizarlos como controles positivos para el método.
- Coloque las concentraciones de aminoácidos en viales de muestra en el muestreador automático de la UPLC. Generar una curva de calibración (9 a 900 pmol/L) en el software del instrumento basada en las concentraciones estándar de aminoácidos siguiendo las siguientes instrucciones.
- Utilice el Q-ToF en el modo de sensibilidad positiva de ionización por electrospray (ESI) acoplado a un UPLC para el análisis intacto de aminoácidos. Para la separación cromatográfica, utilice una columna de fase normal hecha para separaciones de aminoácidos. Preparar los siguientes tampones con reactivos de grado de espectrometría de masas: A - acetonitrilo + 0,1% ácido fórmico y formate de amonio B a 100 mM. Establezca el caudal DE LC en 0,6 ml/min y la temperatura de la columna en 40 oC.
- Utilice las siguientes condiciones de gradiente de 15 minutos para separaciones de aminoácidos: 14% B (0-3 min), 14-100% B (3-10 min), 100% B (10-13 min), 100-8% B (13-14 min), 8% B (14-15 min).
- Utilice las listas de columnas "Tipo de muestra" y "Conc A" en el programa de adquisición de EM para crear una curva de calibración para el análisis futuro de los medios de biorreactor crudo en el programa de cuantificación. Para que estas columnas aparezcan en el programa de adquisición, utilice el comando Personalizar visualización... al hacer clic con el botón derecho en la barra de menúsuperior superior.
- El tipo de muestra para los estándares de aminoácidos será "estándar", mientras que las muestras de medios serán "Analyte". Rellene la columna "Conc A" con las concentraciones numéricas de las normas en las unidades requeridas (por ejemplo: pmol/L).
- Ejecute las concentraciones estándar de aminoácidos preparados al menos dos veces. Valide que el instrumento UPLC y el espectrómetro de masas funcionan correctamente comprobando los picos ISTD.
- Utilice la opción "Editar método" en la aplicación de cuantificación para crear el método de cuantificación (*.mdb file). Defina todos los aminoácidos de interés en la aplicación de cuantificación, como el nombre del compuesto, el valor m/z y el tiempo de retención esperado. Cambie los parámetros de integración para el método aquí.
- Utilice el método de aminoácidos creado en las muestras estándar para crear la curva de calibración. Esta curva se puede exportar a un archivo *.cdb para su uso con los ejemplos de medios mediante el comando Exportar>Calibración....
- En la aplicación de cuantificación, guarde el diseño deseado en un archivo *.qlt para aplicarlo a conjuntos de datos futuros mediante "Guardar diseño como...". Nombre (nombre de inyección), Area y Conc son las columnas de salida más importantes.
- Análisis de aminoácidos de medios biorreactores crudos por LC-MS
- Los medios del biorreactor crudo centrífugo a 1.962 x g durante 5 minutos pasan a través de un filtro de 0,22 m.
- Realice un seguimiento con una limpieza de ácido perclórico para eliminar proteínas y partículas: mezcle el medio biorreactor filtrado con 0,4 N HClO4 en una proporción de 1:1 y centrífuga a 14.700 x g durante 5 min en RT. Recoger los medios clarificados en viales de automuestreador.
NOTA: Ajuste el volumen de inyección según sea necesario para que las concentraciones de aminoácidos se encuentren dentro del rango de calibración. Dependiendo del instrumento, el volumen de la inyección se puede ajustar entre 0,1 y 10 l. - Ejecute muestras de medios en triplicado por LC-MS. Utilice "Procesar muestras" en el programa de cuantificación junto con el método (*.mdb) y el archivo de calibración (*.cdb). El método y la curva de calibración se aplicarán automáticamente a las muestras de medios crudos mediante la aplicación de cuantificación una vez completadas todas las inyecciones.
- Para exportar datos para su análisis en otroprograma (como una hoja de cálculo), utilice el comando "Imprimir" y cree un archivo *.xps o *.pdf.
Resultados
El fluido de cultivo celular cosechado del biorreactor automatizado a microescala se purifica mediante cromatografía líquida de proteína rápida (FPLC), como se ve en la Figura 1 y los atributos de calidad críticos (CQA) de las proteínas purificadas se caracterizaron por varios métodos analíticos aguas abajo. Este es un beneficio clave del sistema automatizado de microbiorreactores; diferencias en los CQA se pueden evaluar rápidamente en una amplia gama de condiciones. Los datos de N-glicano de mAbs producidos por CHO que son procesados por espectrometría de masas deben aparecer como los cromatogramas mostrados en la Figura2. La figura representa una comparación entre dos cromatogramas que muestran que la mannariza 5 pico (M5) de una muestra es considerablemente menor. Si solo se observa una línea base ruidoso en lugar de picos, esto puede significar que la configuración de la cromatografía es defectuosa o que el procedimiento no es correcto. Con los controles, se puede simplificar la solución de problemas. En primer lugar, evaluar los picos de LAR desde la escalera de dextran; estos picos indican que el sistema cromatográfico funciona correctamente. A continuación, compare los picos obtenidos experimentalmente con los obtenidos de un estándar mAb intacto procesado. Si los picos del estándar son visibles, pero no se identifican picos de muestra, las muestras mAb no se procesaron correctamente. Esto puede deberse a la presencia de SDS o nucleófilos en el tampón que interfiere con el etiquetado y purificación de N-glicano.
SEC-MALS se puede utilizar para evaluar dos CQA más: el perfil de agregación y el peso molecular del anticuerpo. Un cromatograma REPRESENTATIVO SEC-MALS es comparable al que se muestra en la Figura3. La distribución de masa molecular y el peso molecular absoluto se determinaron utilizando el software requerido con un coeficiente de extinción de 1,37 ml*(mg*cm)-1 y un dn/dc de 0,185 ml/g. Como la llamada pico y la configuración de la línea de base en el software se realiza manualmente, los resultados pueden variar ligeramente de un usuario a uno. El peso molecular absoluto de la IgG1 monomérica de la Figura 3 es de 1,504 x 105 Da a 0,38% (azul) y el complejo de orden superior es de 7,799 x 105 Da a 3,0% (rojo). La polidispersidad de los agregados es mucho mayor que la del monómero, comolo indica la distribución de masa molar roja del pico 1 (Figura 3). La pequeña cantidad de muestra e importancia de la agregación como CQA hacen de esta técnica una herramienta analítica complementaria muy valiosa para el sistema automatizado de microbiorreactores.
El resultado de mCZE es un electroferograma, como en la Figura4, que muestra el perfil de variante de carga para un anticuerpo monoclonal. El perfil es una firma única para la proteína que se está investigando y es altamente sensible al pH de funcionamiento. También es visible un pico de tinte libre a la izquierda del perfil de la variante de carga. Al establecer un pH de funcionamiento, hay cierta discreción para el operador de equilibrar la resolución y la señal; además, el operador debe garantizar una buena separación del pico de tinte libre que migra a 30 s. La muestra se puede desalar después del etiquetado para eliminar este pico, aunque esto conduce a una pérdida significativa de señal. Una vez que se establece un pH de funcionamiento, se pueden comparar los perfiles de muestra. Aunque generalmente son consistentes, los cambios en la eficiencia del etiquetado o las diferencias en los excipientes pueden conducir a pequeñas diferencias en la migración de una muestra y el perfil de la variante de carga haciendo que los electroferogramas sean difíciles de comparar directamente. En su lugar, el método de comparación se basa generalmente en los porcentajes de especies básicas, principales y ácidas. En este caso, se pueden identificar diferencias relativas tan pequeñas como 1-2% utilizando mCZE.
El consumo de aminoácidos se puede controlar para determinar si el agotamiento está causando cambios en los CQA. Las lecturas de cromatogramas del espectrómetro de masas se pueden utilizar para evaluar la creación exitosa de una curva de calibración para la cuantificación absoluta de aminoácidos en muestras de medios biorreactores crudos. La Figura 5 muestra dos cromatogramas iónicos totales (TIC) y un cromatograma iónico extraído (XIC) como resultados representativos durante este proceso. En la Figura 5A, el TIC mostrado representa la señal de fondo del sistema de búfer como sólo se inyectó un espacio en blanco de agua. Figura 5 B representa un TIC representativo del estándar de aminoácidos donde, en comparación con el agua en blanco, se pueden observar pequeños picos que corresponden a las especies de aminoácidos individuales (como la lisina a 7,96 minutos). Para integrar el pico y facilitar la cuantificación del área pico (y por lo tanto la concentración), se utiliza el XIC donde sólo se muestra la señal de una "ventana de masa de cromatograma" definida. Dependiendo de la sensibilidad del instrumento y la calidad de la separación cromatográfica, la ventana de masa óptima tendrá que ser determinada por el usuario. En este ejemplo (Figura5C), se muestra el XIC de lisina (m/z a 147.1144) con una ventana de masa de 10 ppm donde la lisina en el aminoácido estándar eluye la columna a 8.03 minutos.
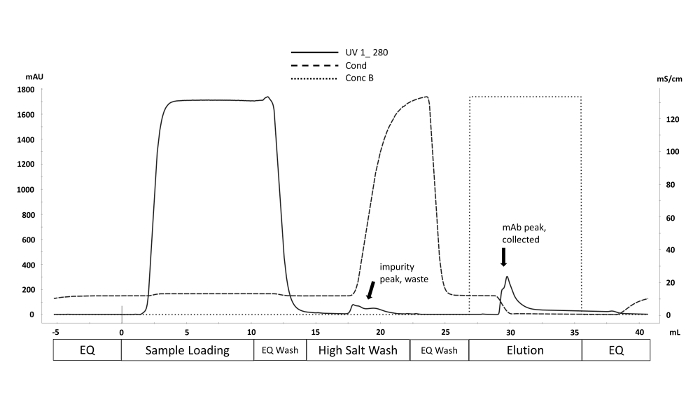
Figura 1 . Cromatografía representativa del esquema de purificación utilizando la técnica de Cromatografía Líquida de Proteína Rápida (FPLC). Las fases del método de purificación correspondientes al volumen (ml) se etiquetan a lo largo del eje X. La absorbancia UV a 280 nm (eje YAU, línea sólida) se controla a lo largo del ciclo de purificación. Las impurezas no específicamente unidas se desplazan por el aumento de la conductividad (eje y mS/cm, línea discontinua) durante el Lavado de Sal Alto. se muestra). Haga clic aquí para ver una versión más grande de esta figura.
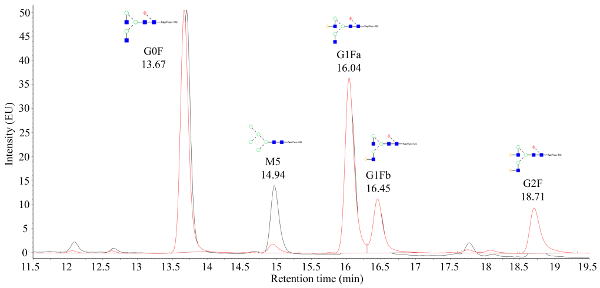
Figura 2. Cromatograma de fluorescencia representativo obtenido a partir de glicanos etiquetados verificados en masa. El eje x es tiempo de retención (minutos) mientras que el eje Y es la intensidad de la señal. El pico a 14,94 min representa el glicano Mannose 5 (M5), donde se puede observar una gran diferencia entre la intensidad de la señal M5 entre las dos muestras que están superpuestas. Haga clic aquí para ver una versión más grande de esta figura.
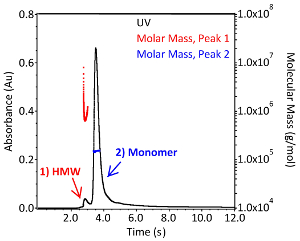
Figura 3. Distribución del peso molecular del anticuerpo monoclonal IgG1. Cromatografía de un anticuerpo monoclonal IgG1 intacto separado por cromatografía de exclusión de tamaño en 1x PBS (pH7.4). La absorbancia se monitorea a 280 nm (negro; eje izquierdo) y se utilizaron detectores de dispersión de luz e índice de refracción para calcular el peso molecular absoluto de cada pico (rojo y azul; eje derecho). Las especies de alto peso molecular están indicadas con el pico etiquetado como "HMW". Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 . Perfil de variante de carga de un anticuerpo monoclonal IgG1. Este electroferograma se genera en una plataforma mCZE. Un pico de tinte libre migra a 30 s y está bien separado del IgG1. Para la cuantificación, los picos se dividieron en especies básicas, principales y ácidas utilizando software de análisis de datos de instrumentos. La línea roja describe las áreas de pico integradas. Haga clic aquí para ver una versión más grande de esta figura.
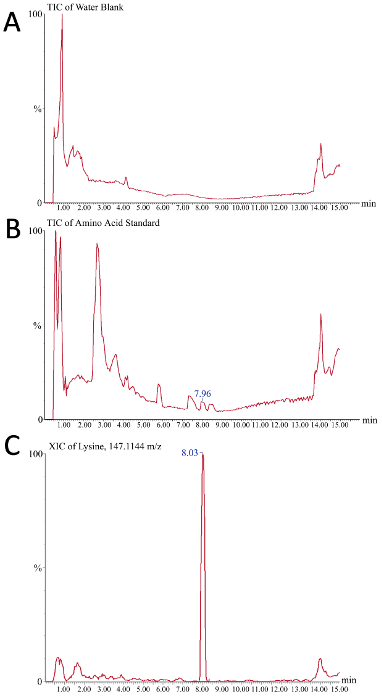
Figura 5. Resultados representativos de los cromatogramas iónicos para el análisis de aminoácidos basados en espectrometría de masas de medios biorreactores crudos. El eje X es el tiempo (minutos) mientras que el eje Y es la intensidad de la señal (A) Un espacio en blanco de agua sirve como el control negativo y revela la señal de fondo observada en el transcurso del gradiente de cromatografía líquida (B) El aminoácido de 225 pmol/L estándar se utiliza aquí como un control positivo, ya que los picos individuales observados en este cromatograma iónico total representan los diferentes aminoácidos de la mezcla estándar que se resuelve cromatográficamente (C) Un cromatograma iónico extraído representativo para m /z 147.1144, que es lisina. El pico de 7,96 min en B corresponde al pico 8,03 en Do de lisina. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
HCCF contiene escombros y partículas grandes que pueden obstruir y destruir la costosa instrumentación, por lo que se necesita una aclaración del cultivo antes de un procesamiento posterior. La centrifugación es generalmente el primer enfoque para separar las células y otras partículas insolubles de las proteínas seguidas de la filtración. Este HCCF filtrado se somete entonces a cromatografía líquida de proteína rápida (FPLC) para su purificación. La purificación de HCCF a partir de microbiorreactores automatizados para obtener el producto es un paso importante en el procesamiento posterior. Aquí, un sistema FPLC de sobremesa con una columna de proteína A se utiliza para obtener anticuerpos monoclonales del HCCF. Los análisis para procesos ascendentes pueden proporcionar información útil sobre el comportamiento celular y guiar el diseño de bioprocesos, lo que ayuda a obtener un producto de calidad coherente y confiable. Analytics también nos permite vincular atributos de calidad crítica (CQA) a procesos ascendentes y descendentes. Aquí se presentan cuatro ensayos que se utilizan comúnmente en la caracterización de anticuerpos monoclonales. Estas técnicas son robustas, fiables y fácilmente desplegables para el análisis de procesos y productos de una variedad de fuentes ascendentes que sólo se purifican parcialmente y todavía pueden contener niveles residuales de ADN y HCP.
Al limpiar muestras para análisis, se debe alcanzar un equilibrio importante entre la creación de una muestra lo suficientemente limpia para el análisis y la preservación de la variabilidad presente en el biorreactor. Los dos contaminantes más comunes que afectan al producto son el ADN y el HCP, que se pueden comprobar midiendo la absorbancia de la relación a 260/280 nm y a través de SDS-PAGE o éCE-SDS. Los ensayos presentados aquí no son sensibles a los bajos niveles de contenido de ADN. La pureza del producto es >95% pura, según lo determinado por el CE-SDS.
El análisis de variantes de carga con un sistema de electroforesis microcapilar proporciona un método de alto rendimiento para identificar variantes de carga, con chips y reactivos que son relativamente fáciles de implementar. La naturaleza de la técnica y la química del reactivo de etiquetado son sensibles a los excipientes y otras aminas primarias, lo que requiere un paso de desalación para la mayoría de las matrices de muestras. Por experiencia, los bajos niveles de ADN co-migran con el tinte libre de la reacción de etiquetado y no afectan la calidad de los resultados. Mientras que la variabilidad de la cuantificación de picos básicos, principales y ácidos es típicamente <1%, niveles más altos de ADN y otros contaminantes pueden aumentar la variabilidad del ensayo. Es extremadamente importante ser consistente con el etiquetado de proteínas y asegurar el uso rápido de DMF después de la eliminación de la botella y ser mezclado con el tinte. Las normas de lisina y/o histidina se recomiendan como controles de etiquetado. Con el tiempo y dependiendo de la calidad de la muestra, las virutas pueden ensuciar o perder el recubrimiento en los canales microfluídicos, lo que conduce a un mayor ruido, la presencia de picos fantasma y una mayor variación de muestra a muestra. Para identificar esta ocurrencia, se analizaron simultáneamente con las muestras los espacios en blanco y un estándar de idoneidad del sistema (es decir, NISTmAb) con las muestras a intervalos regulares. Cuando surgen problemas de chip, las virutas se pueden lavar con la solución de almacenamiento o reemplazarse.
Los métodos utilizados para el análisis de glicanos de glicoproteínas terapéuticas implican principalmente cromatografía líquida (LC) y/o espectrometría de masas (MS), con análisis de microarray de lectina ganando popularidad como tercera opción25. El método descrito en este documento utiliza LC y MS, que tiene beneficios y desventajas. Los métodos espectrométricos de masatienen la ventaja de la verificación masiva de los glicanos analizados, lo que no es posible con métodos basados en LC que utilizan una salida de detección fluorescente o microarrays de lectina. Este método utiliza LC y detección de fluorescencia para asignar identidades de glicano mediante la comparación de tiempo de retención con un estándar de escalera de dextran. El monitoreo de fluorescencia permite una mayor sensibilidad y cuantificación debido a la facilidad de su detección, donde la MUS por sí sola podría no ser capaz de cuantificar especies de baja abundancia debido a la baja eficiencia de ionización de los oligosacáridos. La información masiva de MS se utiliza para confirmar identidades de glicano, pero el software de procesamiento no utiliza la información de masas como criterios de asignación principal. Por lo tanto, sin cromatografía reproducible y picos fácilmente resueltas, este método puede sufrir con respecto a las asignaciones de glicanos. Afortunadamente, la información de masa puede ayudar con las asignaciones de glicano incluso en situaciones en las que la cromatografía es deficiente, como cambios en el tiempo de retención que dificultan las asignaciones de glicanoreproducibles. Si este método se utiliza sin EM, la cromatografía debe estar en el nivel más alto, ya que la información de masa no se puede utilizar para corregir la desviación del tiempo de residencia.
El método de análisis de aminoácidos descrito aquí utiliza LC-MS para la cuantificación rápida de aminoácidos no derivatizados en medios de cultivo celular crudo. Los métodos alternativos de análisis de aminoácidos requieren agentes de derivación de aminoácidos para permitir la detección UV26. El método LC-MS ofrece importantes ventajas sobre el método LC-UV: permite la identificación basada tanto en el tiempo de retención como en la masa iónial en comparación con el método LC-UV, que está limitado por la falta de caracterización de masa. Además, el método LC-MS ofrece ventajas de tiempo y reproducibilidad, ya que el método LC-UV requiere una reacción de derivación que consume mucho tiempo, que puede impartir variabilidad de la muestra27. Sin embargo, la inyección de medios de cultivo celular crudo en el método LC-MS puede causar efectos perjudiciales en la señal de LA MS debido a la suciedad del skimmer iónico. Una escalera de calibración se inyecta con frecuencia como una comprobación de idoneidad del sistema, y el orden de la muestra se aleatoriza para evitar sesgos en los datos.
El proceso de cultivo celular para la producción de anticuerpos utilizando microbiorreactores se describe previamente9. En este estudio, los protocolos detallados para los métodos de caracterización de anticuerpos monoclonales que maximizan los datos adquiridos a partir de volúmenes de muestra limitados están bien definidos. Las cantidades limitadas de líquido de cultivo celular cosechado a veces pueden restringir la información del producto adquirida y la selección de los procedimientos analíticos adecuados para obtener datos de calidad del producto es esencial. Los análisis son importantes para vincular los parámetros del proceso ascendente con los cambios en la calidad del producto. Aquí, se proporciona una guía para que los usuarios caractquen mAbs cuando trabajen con microbiorreactores.
Divulgaciones
Esta publicación refleja las opiniones del autor y no debe interpretarse como una declaración de las opiniones o políticas de la FDA.
Agradecimientos
Los autores quieren agradecer a Scott Lute por el apoyo analítico que brindó. El Programa de Ruta Crítica del CDER (CA #1-13) proporciona financiamiento interno parcial y apoyo a esta labor. Este proyecto es apoyado en parte por un nombramiento para el Programa de Participación en Pasantías/Investigación en la Oficina de Productos Biotecnológicos, Administración de Alimentos y Medicamentos de los Estados Unidos, administrado por el Instituto Oak Ridge para la Ciencia y la Educación a través de un acuerdo interinstitucional entre el Departamento de Energía de los Estados Unidos y la FDA.
Materiales
| Name | Company | Catalog Number | Comments |
| CHO DG44 Cell Line | Invitrogen | A1100001 | |
| Akta Avant 25 | General Electric Life Sciences | 28930842 | |
| Pro Sep vA Ultra Chromatography Resin | Millipore Sigma | 115115830 | Purification Stationary Phase |
| Omnifit 10cm Column | Diba Fluid Intelligence | 006EZ-06-10-AA | Housing for Stationary Phase |
| Tris Base | Fisher Scientific | BP154-1 | |
| Superloop 10 mL | GE Healthcare | 18-1113-81 | |
| µDawn Multi Angle Light Scattering Detector | Wyatt | WUDAWN-01 | |
| 0.22 µm Millex GV Filter Unit PVDF Membrane | Merck Millipore | SLGV033RB | |
| 10X Phosphate Buffered Saline | Corning | 46-013-CM | |
| 12 mL Syringe | Covidien | 8881512878 | |
| 1290 Infinity Binary Pump | Agilent Technologies | G4220A | |
| 1290 Infinity DAD | Agilent Technologies | G4212A | |
| 1290 Infinity Sampler | Agilent Technologies | G4226A | |
| 1290 Infinity Thermostat | Agilent Technologies | G1330B | |
| 1290 Infinity Thermostatted Column Compartment | Agilent Technologies | G1316C | |
| 15 mL Falcon tube | Corning Inc. | 352097 | |
| 150 uL Glass Inserts with Polymer Feet | Agilent Technologies | 5183-2088 | |
| 50 mL Falcon tube | Corning Inc. | 352070 | |
| 96-Well Plate | Bio-Rad | 127737 | |
| Acetic Acid | Sigma-Aldrich | 695072 | |
| Acetonitrile | Fisher Chemical | BPA996-4 | |
| ACQUITY I-Class UPLC BSM | Waters Corporation | 18601504612 | |
| ACQUITY I-Class UPLC Sample Manager | Waters Corporation | 186015000 | |
| ACQUITY UPLC FLR Detector | Waters Corporation | 176015029 | |
| Amicon Ultra-4 100 kDa centrifugal filters | Merck Millipore | UFC810096 | |
| Amino Acid Standard, 1 nmol/µL | Agilent Technologies | 5061-3330 | |
| Amino Acid Supplement | Agilent Technologies | 5062-2478 | |
| Ammonium Formate Solution - Glycan Analysis | Waters Corporation | 186007081 | |
| Blue Screw Caps with Septa | Agilent Technologies | 5182-0717 | |
| CD OptiCHO AGT Medium | Thermo Fisher Scientific | A1122205 | |
| Centrifuge Tubes | Eppendorf | 22363352 | |
| Charge Variant Chip | Perkin Elmer | 760435 | |
| Charge Variant Reagent Kit | Perkin Elmer | CLS760670 | |
| Chromatography Water (MS Grade) | Fisher Chemical | W6-4 | |
| Dimethylformamide | Thermo Scientific | 20673 | |
| Extraction Plate Manifold for Oasis 96-Well Plates | Waters Corporation | 186001831 | |
| Formic Acid | Fisher Chemical | A117-50 | |
| GlycoWorks RapiFlour-MS N-Glycan Starter Kit - 24 Sample | Waters Corporation | 176003712 | |
| GXII Buffer Tubes | E&K Scientific | 697075- NC | |
| GXII Detection Window Cleaning Cloth | VWR | 21912-046 | |
| GXII HT Touch | Perkin Elmer | CLS138160 | |
| GXII Ladder Tubes | Genemate | C-3258-1 | |
| GXII Lint-Free Swab | ITW Texwipe | TX758B | |
| Hydrochloric Acid | Fisher Scientific | A144-500 | |
| Intact mAb Mass Check Standard | Waters Corporation | 186006552 | |
| Intrada Amino Acid Column 150 x 2 mm | Imtakt | WAA25 | |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | Thermo Fisher Scientific | 840274100 | |
| Optilab UT-rEX Differential Refractive Index Detector | Wyatt | WTREX-11 | |
| Perchloric acid | Aldrich Chemistry | 311421 | |
| Pipet Tips with Microcapillary for Loading Gels | Labcon | 1034-960-008 | |
| Polypropylene 96-Well Microplate, F-bottom, Chimney-style, Black | Greiner Bio-One | 655209 | |
| RapiFlour-MS Dextran Calibration Ladder | Waters Corporation | 186007982 | |
| Screw Top Clear Vial 2mL | Agilent Technologies | 5182-0715 | |
| Sodium Chloride | Fisher Scientific | S271-1 | |
| Sodium Iodide | Sigma Aldrich | 383112 | |
| TSKgel UP-SW3000 4.6mm ID x 30 cm L | Tosoh Biosciences | 003449 | |
| UNIFI Scientific Information System | Waters Corporation | 667005138 | |
| Vacuum Manifold Shims | Waters Corporation | 186007986 | |
| Vacuum Pump | Waters Corporation | 725000604 | |
| Xevo G2 Q-ToF | Waters Corporation | 186005597 | |
| Zeba Spin Desalting Column, 0.5 mL | Thermo Scientific | 89883 |
Referencias
- . . Pharmaceutical cGMPs for the 21st Century: A Risk-Based Approach. , (2004).
- New Molecular Entity (NME) Drug and New Biologic Approvals. FDA Available from: https://www.dfa.gov/Drugs/DevelopmentApprovalProcess/HowDrugsareDevelopedandApproved/DrugandBiologicAprrovalReports/NDAandBLAApprovalReports/ucm373420.htm (2015)
- Foltz, I. N., Karow, M., Wasserman, S. M. Evolution and Emergence of Therapeutic Monoclonal Antibodies. Circulation. 127, 2222-2230 (2013).
- Kondragunta, B., Drew, J. L., Brorson, K. A., Moreira, A. R., Rao, G. Advances in clone selection using high-throughput bioreactors. Biotechnology Progress. 26 (4), 1095-1103 (2010).
- Hmiel, L., Brorson, K., Boyne, M. Post-translational structural modifications of immunoglobulin G and their effect on biological activity. Analytical & Bioanalytical Chemistry. 407 (1), 79-94 (2015).
- Rathore, A. S. Roadmap for implementation of quality by design (QbD) for biotechnology products. Trends in Biotechnology. 27 (9), 546-553 (2009).
- . International Council for Harminisation of Techinical Requirements for Pharmaceuticals for Human Use. ICH. , (1999).
- Berkowitz, S. A., Engen, J. R., Mazzeo, J. R., Jones, G. B. Analytical tools for characterizing biopharmaceuticals and the implications for biosimilars. Nature Reviews Drug Discovery. 11 (7), 527-540 (2012).
- Velugula-Yellela, S. R., et al. Use of high-throughput automated microbioreactor system for production of model IgG1 in CHO cells. Journal of Visualized Experiments. , (2018).
- Largy, E., Cantais, F., Van Vyncht, G., Beck, A., Delobel, A. Orthogonal liquid chromatography-mass spectrometry methods for the comprehensive characterization of therapeutic glycoproteins, from released glycans to intact protein level. Journal of Chromatography A. 1498, 128-146 (2017).
- Yang, J. -. M., et al. Investigation of the correlation between charge and glycosylation of IgG1 variants by liquid chromatography-mass spectrometry. Analytical Biochemistry. 448, 82-91 (2014).
- Agarabi, C. D., et al. Bioreactor Process Parameter Screening Utilizing a Plackett-Burman Design for a Model Monoclonal Antibody. Journal of Pharmaceutical Sciences. 104 (6), 1919-1928 (2015).
- Wen, J., Arakawa, T., Philo, J. S. Size-Exclusion Chromatography with On-Line Light-Scattering, Absorbance, and Refractive Index Detectors for Studying Proteins and Their Interactions. Analytical Biochemistry. 240 (2), 155-166 (1996).
- Veurink, M., Stella, C., Tabatabay, C., Pournaras, C. J., Gurny, R. Association of ranibizumab (Lucentis) or bevacizumab (Avastin) with dexamethasone and triamcinolone acetonide: An in vitro stability assessment. European Journal of Pharmaceutics and Biopharmaceutics. 78 (2), 271-277 (2011).
- Li, Y., Weiss, W. F., Roberts, C. J. Characterization of high-molecular-weight nonnative aggregates and aggregation kinetics by size exclusion chromatography with inline multi-angle laser light scattering. Journal of Pharmaceutical Sciences. 98 (11), 3997-4016 (2009).
- Espinosa-de la Garza, C. E., et al. Analysis of recombinant monoclonal antibodies by capillary zone electrophoresis. Electrophoresis. 34 (8), 1133-1140 (2013).
- Han, H., Livingston, E., Chen, X. High throughput profiling of charge heterogeneity in antibodies by microchip electrophoresis. Analytical Chemistry. 83 (21), 8184-8191 (2011).
- Wheeler, T. D., et al. Microchip zone electrophoresis for high-throughput analysis of monoclonal antibody charge variants. Analytical Chemistry. 86 (11), 5416-5424 (2014).
- Carrillo-Cocom, L., et al. Amino acid consumption in naive and recombinant CHO cell cultures: producers of a monoclonal antibody. Cytotechnology. 67 (5), 809-820 (2015).
- Chen, P., Harcum, S. W. Effects of amino acid additions on ammonium stressed CHO cells. Journal of Biotechnology. 117 (3), 277-286 (2005).
- Xing, Z., et al. Optimizing amino acid composition of CHO cell culture media for a fusion protein production. Process Biochemistry. 46 (7), 1423-1429 (2011).
- Fan, Y., et al. Amino acid and glucose metabolism in fed-batch CHO cell culture affects antibody production and glycosylation. Biotechnology and Bioengineering. 112 (3), 521-535 (2015).
- Read, E. K., et al. Fermentanomics informed amino acid supplementation of an antibody producing mammalian cell culture. Biotechnology Progress. 29 (3), 745-753 (2013).
- Mazzer, A. R., Perraud, X., Halley, J., O'Hara, J., Bracewell, D. G. Protein A chromatography increases monoclonal antibody aggregation rate during subsequent low pH virus inactivation hold. Journal of Chromatography. A. 1415, 83-90 (2015).
- Zhang, L., Luo, S., Zhang, B. Glycan analysis of therapeutic glycoproteins. MAbs. 8 (2), 205-215 (2016).
- Wahl, O., Holzgrabe, U. Amino acid analysis for pharmacopoeial purposes. Talanta. 154, 150-163 (2016).
- Le, A., Ng, A., Kwan, T., Cusmano-Ozog, K., Cowan, T. M. A rapid, sensitive method for quantitative analysis of underivatized amino acids by liquid chromatography-tandem mass spectrometry (LC-MS/MS). Journal of Chromatography B. 944, 166-174 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados