Method Article
Reinigung und Analyse eines monoklonalen Antikörpers aus chinesischen Hamster-Ovarialzellen mit einem automatisierten Mikrobioreaktorsystem
In diesem Artikel
Zusammenfassung
Ein detailliertes Protokoll zur Reinigung und anschließenden Analyse eines monoklonalen Antikörpers aus geernteter Zellkulturflüssigkeit (HCCF) automatisierter Mikrobioreaktoren wurde beschrieben. Die Verwendung von Analysen zur Bestimmung kritischer Qualitätsattribute (CQAs) und zur Maximierung des begrenzten Stichprobenvolumens zum Extrahieren wichtiger Informationen wird ebenfalls vorgestellt.
Zusammenfassung
Monoklonale Antikörper (mAbs) sind eines der beliebtesten und am besten charakterisierten biologischen Produkte, die heute hergestellt werden. Am häufigsten mit chinesischen Hamster-Ovarialzellen (CHO) Zellen hergestellt werden, Muss Kultur und Prozessbedingungen optimiert werden, um Antikörper-Titter zu maximieren und Zielqualitätsprofile zu erreichen. In der Regel verwendet diese Optimierung automatisierte Mikroskalen-Bioreaktoren (15 ml), um mehrere Prozessbedingungen parallel zu überprüfen. Zu den Optimierungskriterien gehören die Kulturleistung und die kritischen Qualitätsattribute (CQAs) des monoklonalen Antikörperprodukts (mAb), die sich auf seine Wirksamkeit und Sicherheit auswirken können. Kulturleistungsmetriken umfassen Zellwachstum und Nährstoffverbrauch, während die CQAs die N-Glykosylierungs- und Aggregationsprofile des mAb, Ladungsvarianten und Molekulargewicht umfassen. Dieses detaillierte Protokoll beschreibt, wie HCCF-Proben, die von einem automatisierten Mikrobioreaktorsystem erzeugt werden, gereinigt und anschließend analysiert werden, um wertvolle Leistungsmetriken und -ausgänge zu erhalten. Zunächst wird ein automatisiertes Protein-A-Methode zur schnellen Proteinflüssigkeitschromatographie (FPLC) verwendet, um das mAb aus geernteten Zellkulturproben zu reinigen. Einmal konzentriert, werden die Glykinprofile mittels Einer bestimmten Plattform durch Massenspektrometrie analysiert (siehe Materialtabelle). Antikörper-Molekulargewichte und Aggregationsprofile werden mittels Größenausschlusschromatographie-Multiple-Winkel-Lichtstreuung (SEC-MALS) bestimmt, während Ladungsvarianten mittels Mikrochip-Kapillarzonenelektrophorese (mCZE) analysiert werden. Zusätzlich zu den Kulturleistungsmetriken, die während des Bioreaktorprozesses erfasst wurden (d. h. Kulturlebensfähigkeit, Zellzahl und häufige Metaboliten wie Glutamin, Glukose, Laktat und Ammoniak), werden verbrauchte Medien analysiert, um verbesserung der Fütterungsstrategien und des gesamten Prozessdesigns. Daher wird auch ein detailliertes Protokoll zur absoluten Quantifizierung von Aminosäuren durch flüssige Chromatographie-Massenspektrometrie (LC-MS) verbrauchter Medien beschrieben. Die in diesem Protokoll verwendeten Methoden nutzen Plattformen mit hohem Durchsatz, die für eine große Anzahl von Samples mit kleinem Volumen kompatibel sind.
Einleitung
Protein-Therapeutika werden verwendet, um eine wachsende Vielfalt von Erkrankungen zu behandeln, einschließlich Gewebetransplantation Komplikationen, Autoimmunerkrankungen, und Krebs1. Seit 2004 dokumentiert die United States Food and Drug Administration (USFDA) einen steigenden Anteil an bioologischen Lizenzanträgen (BLAs) aller zulassungen, die vom Center for Drug Evaluation and Research (CDER) reguliert werden, wobei BLAs über 25 % der Zulassungen ausmachen. 2014 und 20152.
Angesichts dieses expandierenden Marktes sind biopharmazeutische Hersteller gefordert, schnell mehr Produkte mit gleichbleibender Qualität zu liefern. Die Bemühungen, die Produktausbeute zu steigern, konzentrierten sich auf CHO-Zelltechnik und Produktionslinien-Screening, obwohl die wichtigsten Verbesserungen auf Fortschritte bei der Optimierung der Medien-/Feed-Strategie und der Umgebungskontrollen für Zellenkulturen zurückzuführen sind1, 3 , 4 , 5 während des Herstellungsprozesses.
Da mAbs in einem biologischen System hergestellt werden, kann es eine inhärente Proteinvariabilität geben. Die Antikörperzusammensetzung kann posttranslational verändert werden, z. B. glykosyliert oder durch Abbau oder enzymatische Reaktionen beeinflusst werden. Diese strukturellen Variationen können gefährliche Immunreaktionen hervorrufen oder die Antikörperbindung verändern, was wiederum die beabsichtigte therapeutische Funktion reduzieren oder eliminieren kann5. Daher werden kritische Qualitätsattribute (CQAs) monoklonaler Antikörper - N-Glykanprofil, Ladungsvariantenverteilung und der Anteil der Antikörper in monomerer Form - regelmäßig im Rahmen eines Quality by Design (QbD)-Ansatzes während der Herstellungsprozesse1,6. In einem regulierten Produktionsumfeld müssen therapeutische Proteine die Akzeptanzkriterien erfüllen, um als zugelassenes kommerzielles Arzneimittel zugelassen zu werden7. Die hier vorgestellten Methoden wären in der Regel Teil des Qualitätscharakterisierungsprozesses für einen Antikörper7,8, und jeder Proteinwissenschaftler wird mit ihrer Verwendung vertraut sein.
In früheren Arbeiten9wurde die Anwendung und der Betrieb von Mikrobioreaktoren für das Hochdurchsatzscreening von Zellkulturbedingungen in der vorgelagerten Bioverarbeitung beschrieben. Das aus den unterschiedlichen Medienbedingungen gewonnene gereinigte Produkt wird einer N-Glykan-Analyse mit LC-MS unterzogen. Glykosylierungsmuster von therapeutischen Proteinen können mit den LC-MS-Techniken10,11und Das Vorhandensein verschiedener Glykanarten wurde mit Bioprozessparametern wie Futterstrategie, pH-Wert und Temperatur12in Verbindung gebracht. Die Auswirkungen der unterschiedlichen Medienbedingungen auf die Produktqualität, angegeben durch den Prozentsatz des resultierenden IgG in monomerer Form, werden auch mit Größenausschlusschromatographie- Multi-Angle Light Scattering (SEC-MALS)13,14 , 15. Das Ladungsvariantenprofil stellt eine Reihe von Modifikationen16 dar, die sich auf die Funktion eines Produkts auswirken könnten. Die Mikrokapillarzonenelektrophorese (mCZE) ist eine Technik, die eine wesentlich schnellere Analysezeit im Vergleich zu herkömmlichen Kationenaustausch(CEX)-Chromatographien und kapillaren isoelektrischen Fokussierungsmethoden (cIEF) bietet, die für die Chargenvariantenanalyse verwendet werden17 ,18. Verbrauchte Bioreaktormedien wurden analysiert, um den Aminosäureverbrauch während der Proteinproduktion zu verfolgen, da sie sich auf Veränderungen der identifizierenden Eigenschaften des Antikörpers beziehen19,20,21,22 , 23.
Proteinanalysen ermöglichen es uns, kritische Prozessparameter (CPPs) basierend auf den Beziehungen zwischen Prozesseingaben und Änderungen in CQAs zu identifizieren. Während der Bioprozessentwicklung demonstriert die Identifizierung und Messung von CPPs grundsätzlich die Prozesssteuerung und stellt sicher, dass sich das Produkt nicht verändert hat, was in stark regulierten Fertigungsumgebungen unerlässlich ist. In diesem Beitrag werden Analysetechniken zur Messung einiger biochemischer Eigenschaften des Proteins vorgestellt, das am relevantesten für Produkt-CQAs (N-Glykanprofil, Ladungsvarianten und Größenhomogenität) ist.
Protokoll
1. Reinigung von Antikörpern
HINWEIS: Der Ausgleichspuffer für den hauseigenen Antikörper beträgt 25 mM Tris, 100 mM NaCl, pH 7.5. Der verwendete Elutionspuffer beträgt 0,1 M Essigsäure. Die Puffer und Harze (Protein A) sind abhängig von dem spezifischen antikörper gereinigt. Das Säulenvolumen entspricht der Betthöhe des Harzes. Die Menge der verwendeten mobilen Phase wird anhand des Spaltenvolumens bestimmt.
- Initialisierung des Reinigungssystems
- Öffnen Sie die software, die an das Reinigungssystem angeschlossen ist. Mit manuellen Anweisungen die Säule mit dem Ausgleichspuffer mit einer Durchflussrate von 2 ml/min für 40 min ausdemieren. Beenden Sie den manuellen Lauf nach dem Ausgleich.
- Im Bruchkollektor 15 ml konische Rohre platzieren, um gereinigte Antikörper eluate und 50 ml konische Rohre zu sammeln, um durchfließend während der hohen Salzwäsche zu sammeln. Stellen Sie sicher, dass der Bruchkollektor auf die Startposition zurückgesetzt wird, indem Sie den Fraktionskollektor vor dem Beginn des Laufs öffnen und schließen. Der Bruchkollektor wird bei 7 °C gehalten.
HINWEIS: Der Fraktionskollektor kann manuell unter der Registerkarte Bruchsammler in den Einstellungenzurückgesetzt werden, sowohl für 15 ml als auch für 50 ml Rohre.
- Probeninjektion
HINWEIS: Die geerntete Zellkulturflüssigkeit, die in den folgenden Verfahren verwendet wird, wurde aus chinesischen Hamster-Ovarialzellen gewonnen, die in automatisierten Mikrobioreaktoren kultiviert wurden9.- Fügen Sie die 0,22 m gefilterte Erntezellkulturflüssigkeit zu einer leeren 12 ml Spritze hinzu, deren Düsenende gedeckelt ist.
- Halten Sie die Spritze mit der Düse nach unten, legen Sie den Spritzenkolben ein, bis ein kleiner Teil des Kolbens drin ist. Stellen Sie sicher, dass die Flüssigkeit nicht undicht ist, drehen Sie die Spritze mit der Nachoben-Düse und entfernen Sie die Kappe.
- Halten Sie die Spritze noch mit der Nachobendüse, drücken Sie den Zylinder, um Luft zu zerstreuen, bis die Zellkulturflüssigkeit an der Spitze der Düse ist. Setzen Sie die Spritzendüse in den manuellen Injektionsanschluss am Reinigungssystem ein und drehen Sie sie zum Anziehen.
- Drücken Sie auf den Kolben, bis die gesamte Probe injiziert ist und in der angeschlossenen 10 ml großen Probenschleife sichtbar ist.
- Öffnen Sie die gespeicherte Methodendatei. Speichern Sie die Ergebnisdatei am gewünschten Speicherort, und geben Sie den Dateinamen an, wenn Sie dazu aufgefordert werden. Hit-Lauf, nachdem die Probe in die große Volumen-Sample-Schleife injiziert wurde.
- Ausführen der Reinigungsmethode
- Wählen Sie die gespeicherte Methode aus, und klicken Sie auf Ausführen, wenn Sie von der Instrumentensoftware aufgefordert werden (Schritt 1.2.5).
HINWEIS: Das System ist so eingerichtet, dass die folgenden Schritte ausgeführt werden. Der Benutzer muss nichts tun, während das Gerät läuft. - Ausgleich der Spalte mit drei Spaltenvolumes (CVs) des Ausgleichspuffers mit einer Durchflussrate von 2 ml/min. Sobald die Spalte ausgeglichen ist, injiziert das System die Probe mit einer Durchflussrate von 1 ml/min in die Spalte.
- Ein Rückgang des UV-Signals bei 280 nm zeigt an, dass die Probe geladen ist. Waschen Sie die Säule mit Ausgleichspuffer mit einer Durchflussrate von 2 ml/min, bis das UV-Signal unter 25 mAU fällt.
- Verwenden Sie vier Lebensläufe mit 25 mM Tris mit 1 M NaCl bei pH 7,5, um eine sekundäre Hochsalzwäsche mit einer Durchflussrate von 2 ml/min durchzuführen. Der Systemfraktionssammler sammelt jedes Protein/DNA, das während der Salzwäsche in 50 ml-Rohren aus der Säule kommt.
- Wenden Sie fünf Lebensläufe mit einem Durchfluss von 1 ml/min auf, um den Antikörper aus der Säule zu löschen. Sammeln Sie das Eluat in 15 ml-Röhren auf Basis von UV-Signal; wenn das UV 280-Signal über 35 mAU liegt, beginnt die Sammlung; Sammlung endet, wenn das Signal unter 50 mAU fällt; dies wird Als Peak Cutting bezeichnet.
ANMERKUNG: Peak Cutting sorgt für eine Normalisierung der Elutionsprofile und um Elutionpeak Tailing zu vermeiden, die Proteinaggregate enthalten können24. - Waschen Sie die Spalte mit drei Lebensläufen des Ausgleichspuffers. Der Lauf endet nach dem Waschschritt.
- Nach der Elution neutralisieren Sie das gereinigte Protein sofort mit 1 M Tris-Basis auf einen pH-Wert von 5,5 .. Messen Sie die Proteinkonzentration mit einem Mikrovolumen UV-Vis Spektralphotometer bei 280 nm und 260 nm und lagern Sie bei 4 °C.
- Konzentrieren Sie den gereinigten Antikörper mit Zentrifugaleinheiten (Schritt 2). Dann unterziehen Sie den gereinigten Antikörper der Glykan-Analyse mit LC-MS und nach der Aggregationsprofilanalyse mit SEC-MALS (Schritte 3 & 4).
HINWEIS: Der gereinigte Antikörper sollte nicht ohne weiteren Pufferaustausch eingefroren werden, da häufige Frost-Tau-Zyklen Aggregation und Niederschlag verursachen können.
- Wählen Sie die gespeicherte Methode aus, und klicken Sie auf Ausführen, wenn Sie von der Instrumentensoftware aufgefordert werden (Schritt 1.2.5).
2. Konzentration von gereinigtem Antikörper
HINWEIS: Der Tris-Acetat-Puffer ist 0,1 M Essigsäure neutralisiert mit 1 M Tris Base auf einen pH-Wert von 5,5 .
- 100 kDa-Filter in Zentrifugenrohre einlegen.
- Waschen Sie die Filter mit 500 l doppeldestilliertem Wasser. Zentrifuge für 10 min bei 14.000 x g bei Raumtemperatur (RT). Wiederholen Sie diesen Schritt zweimal. Filtrat entsorgen.
- Übertragen Sie die geriderten Filter in frische Zentrifugenrohre und fügen Sie jedem Filter 500 l Probe hinzu. Zentrifuge für 10 min bei 14.000 x g.
- Invertieren Sie den Filter in ein frisches Spinrohr. Zentrifugieren Sie 2 min bei 1.000 x g, um die konzentrierte Probe zu sammeln.
- Bestimmen Sie die Probenkonzentrationen mit einem UV-Vis-Spektrophotometer. Leeren Sie das Spektralphotometer mit einer Lösung des Tris-Acetat-Puffers. Verwenden Sie einen Proteinextimtumkoeffizienten von 1,37 ml•(mg•cm)-1 bei 280 nm für eine 1% (% m/v) IgG-Lösung.
- Verwenden Sie die konzentrierte Probe, um 12,5 l mit 2 mg/ml Arbeitslösung für die Glykananalyse und 30 l 3,5 mg/ml Arbeitslösung für SEC-MALS vorzubereiten.
HINWEIS: Das Protokoll kann hier angehalten werden. Die Proben sollten bei 4 °C gekühlt werden. Bei einer Konzentration von 2 mg/ml sollten diese Proben mindestens drei Monate bei 4 °C stabil sein, während höhere Konzentrationen ausfallen könnten.
3. Analyse von N-Glykanen mittels Massenspektroskopie
- N-Glycan-Etikettierung und Isolierung
- Beginnen Sie mit Antikörperkonzentrationen von 2 mg/ml in einem geeigneten Puffer wie neutralem Natriumphosphat, Citrat oder HEPES-Puffer. Bereiten Sie einen intakten mAb-Standard (z. B. NIST mAb) mit 2 mg/ml vor, um neben den experimentellen Proben als Positivkontrolle zu arbeiten.
HINWEIS: Die Antikörper sollten sich in einem Endpuffer befinden, der keine SDS und weniger als 0,1 mM Nucleoophile (wie Tris, DTT, Glycin oder Histidin) enthält. SDS im Beispielpuffer müssen entfernt werden. Wenn sich Nucleophile im Puffer befinden, verdünnen Sie sie oder führen Sie einen Pufferaustausch durch, da sie das Kit stören. Das allgemeine Protokoll ist mit dem Glykan-Kit versehen. - Verdünnen Sie 7,5 l der Antikörper mit 15,3 l LC-MS-Wasser in 1 ml-Röhrchen, die mit dem Kit geliefert werden, und denaturieren Sie dann mit 6 l l 5%-Lösung eines enzymfreundlichen und MS-freundlichen Tensids bei 90 °C für 3 min.
- Kühlen Sie die Proben für 3 min auf Raumtemperatur (RT). Dann 1,2 l PNGase F hinzufügen und 5 min bei 50 °C brüten.
- Nach dem Abkühlen von 3 min auf RT die gespalteten N-Glykane mit 12 L fluoreszierendem Tagging-Reagenz in wasserfreiem Dimethylformamid (DMF) abstempeln und 5 min. warten. Verdünnen Sie die markierte N-Glykan-Mischung mit 358 l Acetonitril (ACN).
- Legen Sie eine hydrophile Interaktionschromatographie (HILIC) Platte in einen Vakuumkrümmer mit Shims und Abfallschale. Verwenden Sie eine Mehrkanalpipette für eine große Anzahl von Proben.
- Konditionieren Sie die Brunnen mit 200 l Wasser, wo das Vakuum so eingestellt wird, dass die Flüssigkeit 15-30 s dauert, um das HILIC-Harz zu passieren. Gleichgewicht mit 200 l 85 % ACN vor dem Laden des ACN-verdünnten glykanierten Glykangemischs (400 l), wobei das Vakuum nach dem Zuführen jeder neuen Flüssigkeit in die Brunnen aufgebracht wird. Waschen Sie das Harz zweimal mit 600 l 1% Ameisensäure (FA)/90% ACN.
- Ersetzen Sie die Abfallwanne durch 600 L Sammelrohre. Die beschrifteten N-Glykane mit SPE-Elutionspuffer (3 Eluionen zu je 30 L) in die Sammelröhrchen geben. Verdünnen Sie die gepoolten Eluionen mit 310 l DMF/ACN-Probenverdünnung. Pipette der Proben in Auto Sampler-Fläschchen, um für die Fluoreszenz-MS-Analyse bereit zu sein.
HINWEIS: Diese Proben sind stabil, wenn sie mindestens 1 Monat bei -80 °C gelagert werden. Bewahren Sie die HILIC-Platte in der Originalverpackung auf, verklebt geschlossen und in einem Trockenschrank für die zukünftige Verwendung.
- Beginnen Sie mit Antikörperkonzentrationen von 2 mg/ml in einem geeigneten Puffer wie neutralem Natriumphosphat, Citrat oder HEPES-Puffer. Bereiten Sie einen intakten mAb-Standard (z. B. NIST mAb) mit 2 mg/ml vor, um neben den experimentellen Proben als Positivkontrolle zu arbeiten.
- LC-MS-Analyse von markierten N-Glykanen
- Analysieren Sie die beschrifteten N-Glykan-Elutionsproben auf einem Ultra Performance Liquid Chromatography (UPLC)-System, das an einen Fluoreszenzdetektor und ein Quadrupol-Massenspektrometer (Q-ToF) gekoppelt ist. Verwenden Sie eine Säule, die für die chromatographische Trennung der beschrifteten Glykane und Wärme auf 60 °C während der Trennungen zugelassen ist.
HINWEIS: Die Säule muss vor der Verwendung mit 60 %Acetonitril und 40 % H2 O gespült werden: 50 Lebensläufe vor der ersten Verwendung oder 20 Lebensläufe, wenn die Säule zuvor verwendet wurde.- Verwenden Sie 50 mM Ammoniumformat (AmF) (hergestellt mit mobilem Phasenkonzentrat) und 100% LC-MS-Grade ACN für die mobilen Phasen. Das AmF ist empfindlich auf pH-Änderungen und ist 1 Monat nach dem Mischen nutzbar. Stellen Sie den Anfänglichen Durchfluss auf 0,4 ml/min ein, wobei der LC-Gradient während der Elutionsphase eine Erhöhung des AmF bietet.
- Stellen Sie den FLR-Detektor auf EX 265/EM 425 nm mit einer Abtastrate von 2 Hz ein. Stellen Sie den Q-ToF auf MS1-Positivionenempfindlichkeitsmodus ein, mit einem Massenbereich von 100-2.000 Daltons (Da), einer Scanzeit von 0,25 Sekunden und einer Kontinuumsdatenerfassung. Verwenden Sie Leucin-Enkephalin (2 ng/l in 50% ACN/0.1% FA) für die interne Massenreferenz im Modus "Korrektur nicht anwenden".
HINWEIS: Die interne Massenreferenzkorrektur wird später während der Datenverarbeitung angewendet. - Setzen Sie die Dextranleiter sequenziell in 22,5 l h2O, 25 l DMF und 52,5 l ACN wieder auf. Bereiten Sie 10 L Aliquots für die Lagerung bei -80 °C vor, da die Leiter bei höheren Temperaturen nicht für mehr als 24 h stabil ist (Raumtemperatur, 4 °C). Die Dextranleiter verschlechtert sich nach mehr als einem Frost-Tau-Zyklus.
- Legen Sie die Proben in den auf 10 °C eingestellten Auto-Sampler. Laden Sie eine Durchstechflasche mit Dextranleiter zusammen mit Proben, da die Aufbewahrungszeitinformationen der Leiter für Zuweisungen verwendet werden, während die Masseninformationen verwendet werden, um Identifikationen zu validieren. Verwenden Sie 10 L-Injektionen für die Proben und 7,5 L-Injektionen für die Leiter. Injektion von Proben in dreifacher Ausfertigung. Führen Sie die geladene Methode aus.
- Analysieren Sie die beschrifteten N-Glykan-Elutionsproben auf einem Ultra Performance Liquid Chromatography (UPLC)-System, das an einen Fluoreszenzdetektor und ein Quadrupol-Massenspektrometer (Q-ToF) gekoppelt ist. Verwenden Sie eine Säule, die für die chromatographische Trennung der beschrifteten Glykane und Wärme auf 60 °C während der Trennungen zugelassen ist.
- N-Glyglykan-Identifikation für LC-MS-Daten
- Führen Sie datenverarbeitung mit einem Programm durch, das für hydrophile Interaktionschromatographie-Fluoreszenz-Massenspektrometriedaten (HILIC-FLR-MS) optimiert ist.
- Wenden Sie interne Massenreferenzkorrekturen innerhalb des Programms an. Bestimmen Sie die Dextranleiterinjektionen in den Probeninformationenals "Standard". Legen Sie in der Analysemethodedie Retentionszeiten der Separation Compound auf die der Leiterverbindungen fest, die während des Laufs nachgewiesen wurden.
- Um sicherzustellen, dass Fläche % für identifizierte Glykane zurückgegeben wird, ändern Sie die Analysemethode: Klicken Sie unter der Registerkarte Verarbeitung auf Quantitationseinstellungen - Kalibrieren und setzen Sie "Kalibrierungskurvenanpassungstyp" auf "Relative Antwort (%)".
4. Analyse der Antikörperaggregation mit SEC-MALS
- Probenvorbereitung
- Übertragen Sie das 3,5 mg/ml (Schritt 2) verdünnte Protein in eine Durchstechflasche mit einem 150-L-Glaseinsatz. Verwenden Sie eine Gel-Ladespitze, um in die untere Glocke des Einsatzes zu pfeifen, um das Einbringen von Blasen zu vermeiden.
- Die Durchstechflasche mit einer Septa-Kappe kappen und sofort analysieren. Bei einer späteren Analyse bei 4 °C lagern.
- SEC-MALS Konfiguration und Ausgleich
HINWEIS: Analysieren Sie die Aggregation auf SEC-MALS, die mit einer Ultra-Hochdruck-Flüssigkeitschromatographie (UHPLC) konfiguriert ist, mit einem MALS-Detektor und einem Brechungsindexdetektor, der von der MALS-Software gesteuert wird.- Konfigurieren Sie eine Methodendatei in der UHPLC-Software zur Steuerung des UHPLC-Systems, die Denkrate auf 0,4 ml/min mit einer mobilen Phase von 1x Phosphatgepufferte Saline (PBS) (verdünnt von 10x), das Injektionsvolumen auf 5 l, die Säulentemperatur auf 25 °C und den Dioden-Array-Detektor (DAD) zur Überwachung von 280 nm. Stellen Sie die Laufzeit auf 20 min. Stellen Sie das System mindestens 4 h vor jeder Probenanalyse aus.
- Die Schnittstelle zwischen UHPLC- und Multi Angle Light Scattering - Refractive Index (MALS-RI)-Detektoren erfordert die Verwendung des analogen Ausgangs am DAD. Legen Sie die DAD-Dämpfung in der DAD-Methodendatei auf 1.000 mAU und die AU/UV-Einstellung auf 1 (UV-Instrument>Kanäle>Kanal 1) fest.
- Schalten Sie die DAD-Lampe 30 min vor Beginn der Analyse ein und stellen Sie die Wellenlänge auf 280 nm ein. Gleichzeitig bereinigen Sie die Referenzzelle Refractive Index (RI) für 15 min oder bis die Baseline stabil ist, und schließen Sie dann die Referenzzelle.
- Konfigurieren Sie die SEC-MALS-Softwaresequenz, setzen Sie die Entnahmezeit auf 12 min, das Injektionsvolumen auf 5 l, den dn/dc auf 0,185 mL/g, den A280-Aussterbekoeffizienten, wenn zuvor experimentell bestimmt oder auf 1,37 mL*(mg*cm)-1, und die Konzentration des probe. Klicken Sie auf Ausführen, und warten Sie, bis das Dialogfeld "Warten auf Injektion" auf dem Bildschirm angezeigt wird.
HINWEIS: Der A280-Aussterbekoeffizient ist spezifisch für das Protein von Interesse und sollte experimentell bestimmt werden. - Konfigurieren Sie eine Beispielliste in der UHPLC-Software in der gleichen Reihenfolge wie in der MALS-RI-Software und senden Sie sie ab.
HINWEIS: Es ist wichtig, eine Systemeignungsprüfung vor und nach einem Lauf durchzuführen. Rinderserumalbumin wird in der Regel verwendet, um nach einer Spitzenverbreiterung zu suchen, ein Zeichen dafür, dass die SEC-Säule möglicherweise gereinigt oder ersetzt werden muss. Die gleiche BSA-Standardinjektion kann verwendet werden, um Signalausrichtung, Peak-Broading und Detektornormalisierung zu spezifizieren.
-
Aggregierte Analyse mit MALS Software
- Klicken Sie auf die Registerkarte Mitdernzahlen. Geben Sie die erforderliche Mindestanzahl an Entbehrung an. keine ist in der Regel ausreichend.
- Stellen Sie sicher, dass die Basislinien korrekt gezeichnet wurden, und passen Sie ggf. für LS1-, LS2-, LS3-, RI- und UV-Kanäle an. Legen Sie den Spitzenbereich fest.
- Überprüfen Sie die molekulare Massenverteilung, um zu bestätigen, dass die genannten Spitzen Partikel ähnlicher Größe enthalten.
5. Chargenvariantenanalyse
- Probenvorbereitung und Etikettierung
- Beginnen Sie mit 80 l einer 3,5 mg/ml Antikörperlösung. Entsalzen Sie die Probe mit einer 0,5 ml Entsalzungssäule (7k MWCO). Bereiten Sie die Säule vor, indem Sie zuerst den unteren Stopfen abfangen, dann den oberen Stopfen lösen und in ein 1,7 ml Mikrozentrifugenrohr legen. Zentrifugieren Sie die Entsalzungssäule für 1 min bei 1.500 x g.
HINWEIS: Markieren Sie das Äußere der Säule mit einem Punkt, damit es in der ursprünglichen Ausrichtung für die nächsten Schritte platziert werden kann. - Übertragen Sie die Säule in ein neues Mikrozentrifugenrohr. Fügen Sie die 80 l des verdünnten Proteins an den oberen Rand der Säule. Richten Sie die Spalte an der ursprünglichen Ausrichtung aus. Zentrifuge für 2 min bei 1.500 x g. Entfernen Sie die Probe aus der Zentrifuge, verwerfen Sie die Entsalzungssäule und mischen Sie die Probe gut.
HINWEIS: Eine Entsalzung ist nur erforderlich, wenn die Probenmatrix primäre Aine, Hilfsstoffe, die die Probenelektrophorese stören, oder andere inkompatible Substanzen enthält. - Verdünnen Sie die Probe auf eine Endkonzentration von 2 mg/ml in einem Volumen von 25 l und fügen Sie 5 l des Etikettierpuffers (siehe Tabelle der Materialien: Charge Variant Reagent Kit) in die 96-Well-Platte. Bereiten Sie das Etikettierungsreagenz vor, indem Sie die erforderliche Menge an Etikettierungsreagenz verdünnen (siehe Tabelle der Materialien: Charge Variant Reagent Kit) 1:30 in Dimethylformamid. Inkubieren Sie die Probe für 10 min bei Raumtemperatur weg vom Licht.
HINWEIS: Es ist wichtig, aufzutauen und dann sofort dieses Reagenz zu verwenden und es innerhalb von 10 min nach dem Mischen mit DMF zu verwenden. - Nach der Inkubation 60 l Reagenzwasser hinzufügen und durch Pipettieren gut vermischen. Bedecken Sie die Platte mit einer Plattendichtung und zentrieren Sie die Platte bei 1.000 x g für 1 min.
- Beginnen Sie mit 80 l einer 3,5 mg/ml Antikörperlösung. Entsalzen Sie die Probe mit einer 0,5 ml Entsalzungssäule (7k MWCO). Bereiten Sie die Säule vor, indem Sie zuerst den unteren Stopfen abfangen, dann den oberen Stopfen lösen und in ein 1,7 ml Mikrozentrifugenrohr legen. Zentrifugieren Sie die Entsalzungssäule für 1 min bei 1.500 x g.
- Vorbereiten des Ladevariantenchips
- Bereiten Sie den Charge Variant Chip vor, indem Sie die Aufbewahrungslösung entfernen und die Brunnen 1, 3, 4, 7, 8 und 10 mit Wasser waschen. Ersetzen Sie dann das Wasser durch pH 7.2 Laufpuffer (siehe Tabelle der Materialien: Charge Variant Reagent Kit).
- Fügen Sie dem Pufferrohr 750 l pH 7,2-Laufpuffer hinzu und platzieren Sie das Pufferrohr an der angegebenen Stelle in der oberen linken Ecke des Probenfachs. Entfernen Sie nun die Plattendichtung von der 96-Well-Platte, drücken Sie die Entladeplatte auf der Benutzeroberfläche des Geräts und legen Sie die Platte in das GXII-Probenfach ein.
HINWEIS: für diese Analyse wurden pH-Puffer von 7,2 verwendet. je nach Protein-pI können pH-Puffer von 5,6-7,2 verwendet werden. Bei Verwendung niedrigerer pH-Puffer sind möglicherweise längere Ablaufzeiten erforderlich. - Drücken Sie die Taste Chip entladen auf der Benutzeroberfläche. Stellen Sie sicher, dass die Elektroden frei von Partikeln sind, und, falls nicht, mit einem fusselfreien Tupfer reinigen. Achten Sie beim Einsetzen des Chips darauf, dass das Fenster in der Mitte des Chips frei von Partikeln oder Flecken ist. Bei Bedarf mit einem fusselfreien weichen Tuch reinigen.
HINWEIS: Entfernen Sie bei der Arbeit mit Kapillarelektrophorese-Chips den Puffer durch Vakuumabsaugung, gefolgt von der sofortigen Zugabe der nächsten Lösung, um das Austrocknen von Brunnen zu verhindern. Um das Einbringen von Blasen zu minimieren, üben Sie Reverse Pipetting-Technik. Achten Sie beim Umgang mit dem Chip auf die zerbrechliche Kapillare, die sich von der Unterseite des Chips ausdehnt, und achten Sie darauf, dass er nicht austrocknet und nicht durch grobe Handhabung bricht. - Schließen Sie den Deckel an der Chipkammer und wählen Sie den HT Protein Charge Variant Assay aus. Klicken Sie auf die Schaltfläche Ausführen. Folgen Sie den Anweisungen, um die Probenbohrungen, den Plattentyp, die Testzeit (68, 90 oder 100 s) und den Dateinamen auszuwählen. Klicken Sie am Ende der Eingabeaufforderungen auf Start.
- Die Chipreinigung erfordert das Waschen jedes Brunnens 2x mit Wasser, gefolgt von der Zugabe von Storage Buffer (siehe Tabelle der Materialien: Charge Variant Reagent Kit). Sobald Sie sich im Speicherpuffer befinden, ersetzen Sie den Chip im Gerät und wählen Sie auf Aufforderung den HT Protein Charge Assay aus. Wählen Sie auf dem Hauptbildschirm auf der Benutzeroberfläche die Option Waschen aus. Nach Abschluss des Chips entfernen Sie den Chip, wischen Sie die Elektroden mit Wasser und einem fusselfreien Tupfer ab und lagern Sie den Chip bei 4 °C.
- Chargenvariantenanalyse
- Öffnen Sie die Instrumentenanalysesoftware. Importieren Sie die Ausführung, indem Sie zu Datei>Datendatei importieren... und klicken Sie auf die gewünschte *.gxd-Datei. Nur der Name wird auf Software übertragen, daher ist das Umbenennen der Brunnen vorteilhaft (Tools > Sample Name Editor). Wählen Sie die zu exportierenden Dateien aus, indem Sie die Verschiebung gedrückt halten, während Sie die Dateien auswählen. Klicken Sie auf Datei>Exportieren... und wählen Sie das Feld Rohdaten und dann das Feld AIA Format aus.
- Öffnen Sie die Registerkarte Projekte durchsuchen in der Analysesoftware. Klicken Sie auf Datenbank>Daten importieren... und wählen Sie das exportierte *aus. CDF-Dateien.
- Nach dem Import navigieren Sie zur Registerkarte Injektionen, wählen Sie die zu analysierenden Dateien aus, klicken Sie mit der rechten Maustaste und gehen Sie zu Prozess... Aktivieren Sie in dem angezeigten Fenster das Kontrollkästchen neben Prozess und das Radiofeld "Angegebene Verarbeitungsmethode verwenden" und die gewünschte Verarbeitungsmethode aus dem Dropdownfeld. Wählen Sie im Dropdown-Feld direkt darunter "How:" die Option Kalibrieren und Quantitieren aus. Navigieren Sie nach der Verarbeitung zur Registerkarte Ergebnisse, und überprüfen Sie die Integration der Chromatogramme.
HINWEIS: Die Verarbeitungsmethode muss für jede Methode überprüft werden. Als Ausgangspunkt sind die Parameter, die für die aktuelle Verarbeitungsmethode verwendet werden, in der Zusatzdatei enthalten.
ANMERKUNG: Der Export der Daten kann in Form eines Berichts erfolgen oder nur die Spitzenquantifizierung kann exportiert werden. Diese können zur gleichen Zeit wie die Verarbeitung oder aus dem Ergebnisfenster durchgeführt werden.
6. Aminosäureanalyse
- Einrichten der Standardkurve für die absolute Aminosäurequantifizierung durch LC-MS
- Bereiten Sie die erweiterte Aminosäure (EAA) Mischung durch Auflösen 59,45 mg Asn, 59,00 mg Hyp, 65,77 mg Gln und 91,95 mg Trp in 25 ml von 0,1 N HCl. Die Endkonzentration jeder Aminosäure im EAA-Gemisch beträgt 18 nmol/l.
- Bereiten Sie die interne Standard-Lagerlösung (ISTD) vor, indem Sie 58,58 mg Nva und 44,54 mg Sar in 50 ml HCl auflösen.
- Bereiten Sie die kompletten Aminosäurestandards vor, indem Sie die Aminosäure-Stammlösung mit Ala, Asp, Arg, Cys, Glu, Gly, His, Ile, Leu, Met, Phe, Pro, Ser, Thr, Trp, Tyr, Val bei je 1 nmol/L mit dem EAA-Gemisch für endgültige Aminosäurekonzentrationen von 900 , 225, 90, 22,5 und 9 pmol/l. Fügen Sie den vorbereiteten ISTD-Bestand zu den Aminosäurestandards für eine Endkonzentration von entweder 90 pmol/l oder 900 pmol/l hinzu, um "niedrige" und "hohe" interne Standards zu schaffen, die als Positivkontrollen für die Methode verwendet werden können.
- Legen Sie die Aminosäurekonzentrationen in Probenfläschchen in den Autosampler des UPLC. Generieren Sie in der Gerätesoftware eine Kalibrierkurve (9 bis 900 pmol/l) basierend auf den Aminosäurestandardkonzentrationen unter Verwendung der folgenden Anweisungen.
- Verwenden Sie den Q-ToF in der Elektrospray-Ionisation (ESI) positiven Empfindlichkeitsmodus gekoppelt an einen UPLC für intakte Aminosäureanalyse. Verwenden Sie für die chromatographische Trennung eine normale Phasensäule, die für Aminosäuretrennungen hergestellt ist. Bereiten Sie die folgenden Puffer mit Massenspektrometrie-Reagenzien vor: A = Acetonitril + 0,1% Ameisensäure und B = 100 mM Ammoniumformat. Stellen Sie den LC-Durchfluss auf 0,6 ml/min und die Säulentemperatur auf 40 °C ein.
- Verwenden Sie die folgenden 15-minütigen Gradientenbedingungen für Aminosäuretrennungen: 14% B (0-3 min), 14-100% B (3-10 min), 100% B (10-13 min), 100-8% B (13-14 min), 8% B (14-15 min).
- Verwenden Sie die Spalten "Sample Type" und "Conc A" im MS-Erfassungsprogramm, um eine Kalibrierkurve für die zukünftige Analyse von Rohbioreaktormedien im Quantifizierungsprogramm zu erstellen. Damit diese Spalten im Erfassungsprogramm angezeigt werden, verwenden Sie den Befehl Anzeige anpassen,... wenn Sie mit der rechten Maustaste auf die obere Menüleiste klicken.
- Der Probentyp für die Aminosäurestandards wird "Standard" sein, während die Medienproben "Analyt" sein werden. Füllen Sie die Spalte "Conc A" mit den numerischen Konzentrationen der Normen in den erforderlichen Einheiten (z. B. pmol/L) aus.
- Führen Sie die vorbereiteten Aminosäurestandardkonzentrationen mindestens zweimal aus. Überprüfen Sie, ob das UPLC-Instrument und das Massenspektrometer ordnungsgemäß funktionieren, indem Sie die ISTD-Spitzen überprüfen.
- Verwenden Sie die Option "Methode bearbeiten" in der Quantifizierungsanwendung, um die Quantifizierungsmethode (*.mdb-Datei) zu erstellen. Definieren Sie alle Aminosäuren, die für die Quantifizierungsanwendung von Interesse sind, wie z. B. den zusammengesetzten Namen, den m/z-Wert und die erwartete Retentionszeit. Ändern Sie hier die Integrationsparameter für die Methode.
- Verwenden Sie die erstellte Aminosäuremethode auf den Standardproben, um die Kalibrierkurve zu erstellen. Diese Kurve kann zur Verwendung mit den Medienbeispielen mit dem Befehl Exportieren>Kalibrierung... in eine *.cdb-Datei exportiert werden.
- Speichern Sie in der Quantifizierungsanwendung das gewünschte Layout in einer *.qlt-Datei, um es mit "Layout unter speichern..." auf zukünftige Datasets anzuwenden. Name (Einspritzname), Area und Conc sind die wichtigsten Ausgabespalten.
- Aminosäureanalyse von Rohbioreaktormedien durch LC-MS
- Zentrifugen-Rohbioreaktormedien bei 1.962 x g für 5 Minuten und durchqueren einen 0,22 m-Filter.
- Nacheiner einer Perchlorsäure-Reinigung, um Protein- und Partikel zu entfernen: Mischen Sie die gefilterten Bioreaktormedien mit 0,4 N HClO4 im Verhältnis 1:1 und Zentrifuge bei 14.700 x g für 5 min bei RT. Sammeln Sie die geklärten Medien in Autosampler-Durchstechflaschen.
HINWEIS: Passen Sie das Injektionsvolumen nach Bedarf an, damit die Aminosäurekonzentrationen innerhalb des Kalibrierbereichs liegen. Je nach Instrument kann das Einspritzvolumen zwischen 0,1 l und 10 l eingestellt werden. - Führen Sie Medienproben in dreifacher Richtung von LC-MS aus. Verwenden Sie "Prozessproben" unter dem Quantifizierungsprogramm zusammen mit der Methode (*.mdb) und der Kalibrierdatei (*.cdb). Die Methode und die Kalibrierkurve werden automatisch durch die Quantifizierungsanwendung auf die Rohmedienproben angewendet, sobald alle Injektionen abgeschlossen sind.
- Um Daten für die Analyse in einem anderen Programm (z. B. einer Kalkulationstabelle) zu exportieren, verwenden Sie den Befehl "Drucken" und erstellen Sie eine *.xps- oder *.pdf-Datei.
Ergebnisse
Die geerntete Zellkulturflüssigkeit aus dem automatisierten Mikro-Bioreaktor wird mit hilfe der schnellen Proteinflüssigkeitschromatographie (FPLC) gereinigt, wie in Abbildung 1 zu sehen ist, und die kritischen Qualitätsmerkmale (CQAs) der gereinigten Proteine zeichneten sich durch verschiedene nachgelagerte Analysemethoden. Dies ist ein wesentlicher Vorteil des automatisierten Mikrobioreaktorsystems; Unterschiede in CQAs können schnell über eine Vielzahl von Bedingungen hinweg beurteilt werden. N-Glykandaten von CHO-produzierten mAbs, die durch Massenspektrometrie verarbeitet werden, sollten wie die in Abbildung 2dargestellten Chromatogramme erscheinen. Die Abbildung zeigt einen Vergleich zwischen zwei Chromatogrammen, die zeigen, dass die Mannose 5 Peak (M5) aus einer Probe deutlich niedriger ist. Wenn nur eine laute Basislinie anstelle von Spitzen beobachtet wird, kann dies bedeuten, dass der Chromatographieaufbau fehlerhaft ist oder dass die Prozedur nicht erfolgreich ist. Mithilfe von Steuerelementen kann die Fehlerbehebung vereinfacht werden. Bewerten Sie zunächst die FLR-Spitzen von der Dextran-Leiter aus; diese Spitzen deuten darauf hin, dass das chromatographische System ordnungsgemäß funktioniert. Vergleichen Sie als Nächstes die experimentell erhaltenen Spitzen mit denen eines verarbeiteten intakten mAb-Standards. Wenn Spitzen aus dem Standard sichtbar sind, aber keine Probenspitzen identifiziert werden, wurden die mAb-Proben nicht korrekt verarbeitet. Dies kann auf SDS oder Nucleophil Anwesenheit im Puffer stören n-glykanische Etikettierung und Reinigung sein.
SEC-MALS kann verwendet werden, um zwei weitere CQAs zu bewerten: das Aggregationsprofil und das Molekulargewicht des Antikörpers. Ein repräsentatives SEC-MALS-Chromatogramm ist mit dem in Abbildung 3dargestellten vergleichbar. Die molekulare Massenverteilung und das absolute Molekulargewicht wurden mit der erforderlichen Software mit einem Aussterbekoeffizienten von 1,37 ml*(mg*cm)-1 und einem dn/dc von 0,185 ml/g bestimmt. Da Spitzenaufrufe und festlegen der Baseline in der Software manuell ausgeführt werden, können die Ergebnisse von Benutzer zu Benutzer leicht variieren. Das absolute Molekulargewicht des monomeren IgG1 aus Abbildung 3 beträgt 1,504 x 105 Da bei 0,38 % (blau) und der Komplex höherer Ordnung 7,799 x 105 Da bei 3,0 % (rot). Die Polydispersität der Aggregate ist viel größer als die des Monomers, wie die rote Molmassenverteilung von Peak 1 (Abbildung 3) zeigt. Die geringe Menge an Proben und die Bedeutung der Aggregation als CQA machen diese Technik zu einem sehr wertvollen ergänzenden Analysewerkzeug zum automatisierten Mikrobioreaktorsystem.
Das Ergebnis von mCZE ist ein Elektropherogramm, wie in Abbildung 4, das das Ladungsvariantenprofil für einen monoklonalen Antikörper zeigt. Das Profil ist eine einzigartige Signatur für das untersuchte Protein und ist hochempfindlich gegenüber dem betriebsbereiten pH-Wert. Ebenfalls sichtbar ist eine Freifärbungsspitze links vom Ladungsvariantenprofil. Bei der Festlegung eines Betriebs-pH-Werts gibt es für den Bediener einen gewissen Ermessensspielraum, um Auflösung und Signal auszugleichen; Darüber hinaus muss der Bediener eine gute Trennung von der Freifärbungsspitze gewährleisten, die bei 30 s wandert. Die Probe kann nach der Kennzeichnung entsalzt werden, um diesen Peak zu entfernen, obwohl dies zu einem erheblichen Signalverlust führt. Sobald ein Betriebs-pH-Wert festgestellt ist, können die Probenprofile verglichen werden. Obwohl im Allgemeinen konsistent, können Änderungen der Etikettierungseffizienz oder Unterschiede in den Hilfsstoffen zu geringfügigen Unterschieden bei der Migration einer Probe und dem Ladungsvariantenprofil führen, was elektropherogramme schwer direkt vergleichen lässt. Stattdessen basiert die Vergleichsmethode in der Regel auf den Prozentsätzen der Basis-, Haupt- und Saurierarten. In diesem Fall können mit mCZE relative Unterschiede von nur 1-2% identifiziert werden.
Der Aminosäureverbrauch kann überwacht werden, um festzustellen, ob die Erschöpfung Veränderungen der CQAs verursacht. Mit Chromatogrammauslesungen aus dem Massenspektrometer kann die erfolgreiche Erstellung einer Kalibrierkurve zur absoluten Quantifizierung von Aminosäuren in Rohbioreaktor-Medienproben ausgewertet werden. Abbildung 5 zeigt zwei Gesamtionenchromatogramme (TIC) und ein extrahiertes Ionenchromatogramm (XIC) als repräsentative Ergebnisse während dieses Prozesses. In Abbildung 5Azeigt das gezeigte TIC das Hintergrundsignal aus dem Puffersystem, da nur ein Wasserrohling injiziert wurde. Abbildung 5 B zeigt einen repräsentativen TIC des Aminosäurestandards, bei dem im Vergleich zum Wasserrohling kleine Spitzen beobachtet werden können, die den einzelnen Aminosäurearten entsprechen (z. B. Lysin bei 7,96 Minuten). Um den Peak zu integrieren und die Quantifizierung der Peak-Fläche (und damit der Konzentration) zu erleichtern, wird das XIC verwendet, wo nur das Signal aus einem definierten "Chromatogramm-Massenfenster" angezeigt wird. Je nach Empfindlichkeit des Instruments und Qualität der chromatographischen Trennung muss das optimale Massenfenster vom Anwender bestimmt werden. In diesem Beispiel (Abbildung 5C) wird der XIC von Lysin (m/z = 147.1144) mit einem Massenfenster von 10 ppm gezeigt, wo Lysin im Aminosäurestandard bei 8,03 Minuten aus der Säule ausgeht.
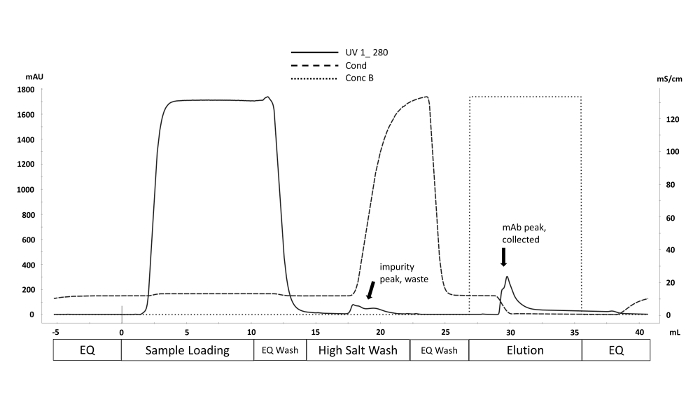
Abbildung 1 . Repräsentatives Chromatogramm des Reinigungsschemas mit der Fast Protein Liquid Chromatography (FPLC) Technik. Reinigungsmethodenphasen, die dem Volumen (mL) entsprechen, werden entlang der x-Achse beschriftet. Die UV-Absorption bei 280 nm (mAU y-Achse, volumenförmige Linie) wird während des gesamten Reinigungszyklus überwacht. Nicht spezifisch gebundene Verunreinigungen werden durch erhöhung der Leitfähigkeit (mS/cm y-Achse, gestrichelte Linie) während der Hochsalzwäsche verdrängt. Antikörper werden aus Protein-A-Säule mit der Einführung eines Elutionspuffers (Conc B, gepunktete Linie) eluiert, wenn der pH-Wert auf 4 (nicht angezeigt). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
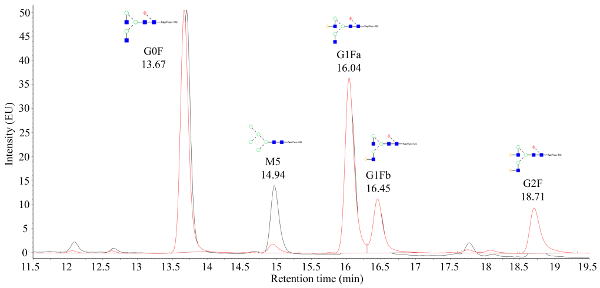
Abbildung 2. Ein repräsentatives Fluoreszenzchromatogramm, das aus getaggten Glykanen gewonnen wird, die massenverifiziert sind. Die x-Achse ist die Haltezeit (Minuten), während die y-Achse die Signalintensität ist. Der Peak bei 14,94 min stellt das Mannose 5 (M5) Glycan dar, wobei ein großer Unterschied zwischen der M5-Signalstärke zwischen den beiden überlagerten Proben beobachtet werden kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
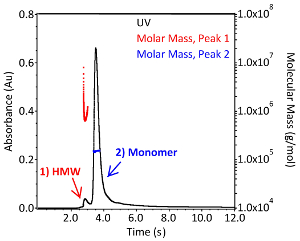
Abbildung 3. Molekulargewichtsverteilung von IgG1 monoklonalen Antikörpern. Chromatogramm eines intakten monoklonalen IgG1-Antikörpers, getrennt durch Größenausschlusschromatographie in 1x PBS (pH7.4). Die Absorption wird bei 280 nm (schwarz; linke Achse) überwacht und Lichtstreuungs- und Brechungsindexdetektoren wurden verwendet, um das absolute Molekulargewicht jedes Peaks (rot und blau; rechte Achse) zu berechnen. Hochmolekulare Arten werden mit der Spitze mit der Bezeichnung "HMW" angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4 . Chargenvariantenprofil eines monoklonalen IgG1-Antikörpers. Dieses Elektropherogramm wird auf einer mCZE-Plattform erzeugt. Ein Freifärbespitzenwanderwert wandert bei 30 s und ist gut vom IgG1 getrennt. Für die Quantifizierung wurden die Spitzen mithilfe von Instrumentendatenanalysesoftware in Basis-, Haupt- und saure Arten aufgeteilt. Die rote Linie umreißt die integrierten Spitzenbereiche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
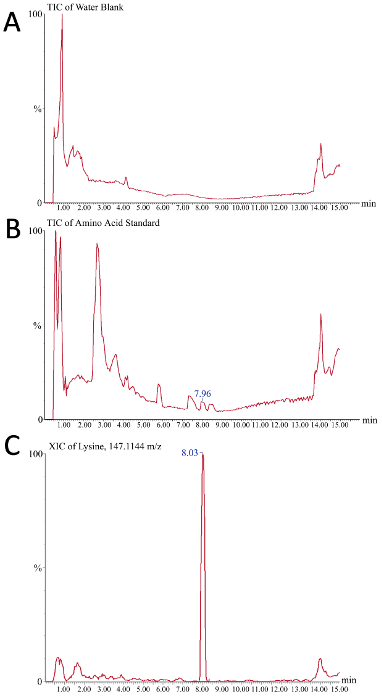
Abbildung 5. Repräsentative Ergebnisse der Ionenchromatogramme für die Massenspektrometrie-basierte Aminosäureanalyse von Rohbioreaktormedien. Die x-Achse ist Zeit (Minuten), während die y-Achse Signalintensität ist (A) Ein Wasserrohling dient als Negativkontrolle und zeigt das Hintergrundsignal an, das im Laufe des Flüssigchromatographie-Gradienten beobachtet wird (B) Die 225 pmol/L-Aminosäure Standard wird hier als Positivkontrolle verwendet, da die einzelnen Spitzen, die in diesem Gesamtionenchromatogramm beobachtet werden, die verschiedenen Aminosäuren des Standardmixes darstellen, der chromatographisch aufgelöst wird (C) Ein repräsentativ extrahiertes Ionenchromatogramm für m /z 147.1144, das ist Lysin. Der 7,96 min Spitzenwert in B entspricht dem 8,03-Spitzenwert in C von Lysin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
HCCF enthält Schmutz und große Partikel, die kostspielige Instrumente verstopfen und zerstören können, so dass eine Kulturklärung vor der weiteren Weiterverarbeitung erforderlich ist. Zentrifugation ist im Allgemeinen der erste Ansatz, um Zellen und andere unlösliche Partikel von Proteinen zu trennen, gefolgt von Filtration. Dieser gefilterte HCCF wird dann zur Reinigung der Fast Protein Liquid Chromatography (FPLC) unterzogen. Die Reinigung von HCCF aus automatisierten Mikrobioreaktoren zur Gewinnung des Produkts ist ein wichtiger Schritt in der Weiterverarbeitung. Hier bei der Gewinnung monoklonaler Antikörper aus dem HCCF wird ein Tisch-FPLC-System mit einer Protein-A-Säule verwendet. Analysen für upstreame Prozesse können nützliche Einblicke in das Zellverhalten bieten und das Bioprozessdesign leiten und so zu einem konsistenten und zuverlässigen Qualitätsprodukt beitragen. Analytics ermöglicht es uns auch, Kritische Qualitätsattribute (CQAs) mit vor- und nachgelagerten Prozessen zu verknüpfen. Hier werden vier Assays vorgestellt, die häufig bei der Charakterisierung monoklonaler Antikörper verwendet werden. Diese Techniken sind robust, zuverlässig und für Prozess- und Produktanalysen aus einer Vielzahl von vorgelagerten Quellen, die nur teilweise gereinigt werden und noch Restmengen an DNA und HCP enthalten können, leicht einsetzbar.
Bei der Reinigung von Proben für Analysen muss ein wichtiges Gleichgewicht zwischen der Erstellung einer Probe, die für die Analyse ausreichend sauber ist, und der Erhaltung der Im Bioreaktor vorhandenen Variabilität gefunden werden. Die beiden häufigsten Kontaminanten, die sich auf das Produkt auswirken, sind DNA und HCP, die durch Messung der Verhältnisabsorption bei 260/280 nm und durch SDS-PAGE oder CE-SDS überprüft werden können. Die hier vorgestellten Assays reagieren nicht empfindlich auf niedrige DNA-Gehalte. Die Reinheit des Produktes ist >95% rein, wie durch die Definition von CE-SDS bestimmt.
Die Chargenvariantenanalyse mit einem mikrokapillaren Elektrophoresesystem bietet eine Methode mit hohem Durchsatz, um Ladungsvarianten mit Chips und Reagenzien zu identifizieren, die relativ einfach zu implementieren sind. Die Art der Technik und die Chemie des Etikettierungsreagenzsindsind sind sowohl empfindlich gegenüber Hilfsstoffen als auch anderen primären Aminen, was für die meisten Probenmatrixen einen Entsalzungsschritt erfordert. Erfahrungsgemäß wandern niedrige DNA-Werte mit dem Freifarbstoff aus der Etikettierungsreaktion und beeinflussen nicht die Qualität der Ergebnisse. Während die Variabilität der Grund-, Haupt- und Saurier-Spitzenquantifizierung in der Regel <1% beträgt, können höhere DNA-Spiegel und andere Verunreinigungen die Variabilität des Tests erhöhen. Es ist äußerst wichtig, mit der Proteinkennzeichnung konsistent zu sein und die schnelle Verwendung von DMF nach dem Entfernen aus der Flasche und dem Mix mit dem Farbstoff sicherzustellen. Lysin- und/oder Histidin-Standards werden als Etikettierkontrollen empfohlen. Im Laufe der Zeit und abhängig von der Probenqualität können Chips die Beschichtung auf den Mikrofluidienkanälen verunreinigen oder verlieren, was zu größerem Rauschen, dem Vorhandensein von Geisterspitzen und einer größeren Variation von Probe zu Probe führt. Um dieses Vorkommen zu identifizieren, wurden Inlädiele und ein Systemeignungsstandard (z.B. NISTmAb) in regelmäßigen Abständen gleichzeitig mit den Proben analysiert. Wenn Chipprobleme auftreten, können die Chips mit der Speicherlösung gewaschen oder ersetzt werden.
Die Methoden für die Glykananalyse von therapeutischen Glykoproteinen betreffen in erster Linie flüssige Chromatographie (LC) und/oder Massenspektrometrie (MS), wobei die Lectin-Mikroarray-Analyse als dritte Option an Popularität gewinnt25. Die in diesem Dokument beschriebene Methode verwendet sowohl LC als auch MS, was Vor- und Nachteile hat. Massenspektrometrische Methoden haben den Vorteil der Massenverifizierung der analysierten Glykane, was mit LC-basierten Methoden mit einem fluoreszierenden Detektionsausgang oder Lektin-Mikroarrays nicht möglich ist. Bei dieser Methode werden LC- und Fluoreszenzdetektionen verwendet, um Glykanidentitäten mithilfe eines Retentionszeitvergleichs einem Dextranleiterstandard zuzuweisen. Die Fluoreszenzüberwachung ermöglicht eine erhöhte Empfindlichkeit und Quantifizierung aufgrund der einfachen Detektion, bei der MS allein aufgrund der schlechten Ionisationseffizienz von Oligosacchariden möglicherweise nicht in der Lage ist, Arten mit geringer Häufigkeit zu quantifizieren. Die Masseninformationen von MS werden verwendet, um glykanische Identitäten zu bestätigen, aber die Verarbeitungssoftware verwendet keine Masseninformationen als primäre Zuweisungskriterien. Daher kann diese Methode ohne reproduzierbare Chromatographie und leicht lösbare Spitzen bei Glykan-Zuweisungen leiden. Glücklicherweise kann die Masseninformation bei Glykan-Zuweisungen helfen, selbst in Situationen, in denen die Chromatographie unterdurchschnittlich ist, wie z. B. Verschiebungen der Retentionszeit, die reproduzierbare Glykan-Aufgaben behindern. Wenn diese Methode ohne MS verwendet wird, muss die Chromatographie auf der höchsten Ebene sein, da Masseninformationen nicht verwendet werden können, um die Verweilzeitdrift zu korrigieren.
Die hier beschriebene Aminosäureanalysemethode nutzt LC-MS für die schnelle Quantifizierung unteriatisierter Aminosäuren in Rohzellkulturmedien. Alternative Aminosäureanalysemethoden erfordern Aminosäure-Derivatisierungsmittel, um den UV-Nachweis zu ermöglichen26. Die LC-MS-Methode bietet wichtige Vorteile gegenüber der LC-UV-Methode: Sie ermöglicht die Identifikation sowohl auf Retentionszeit als auch auf Ionenmasse im Gegensatz zur LC-UV-Methode, die durch einen Mangel an Massencharakterisierung begrenzt wird. Darüber hinaus bietet die LC-MS-Methode Zeit- und Reproduzierbarkeitsvorteile, da das LC-UV-Verfahren eine zeitaufwändige Ableitungsreaktion erfordert, die Probenvariabilität27vermitteln kann. Die Injektion von Rohzellkulturmedien in die LC-MS-Methode kann jedoch schädliche Auswirkungen auf das MS-Signal aufgrund von Ionenabschäum-Fouling haben. Eine Kalibrierleiter wird häufig als Systemeignungsprüfung injiziert, und die Stichprobenreihenfolge wird randomisiert, um Verzerrungen in den Daten zu verhindern.
Das Zellkulturverfahren zur Antikörperproduktion mit Mikrobioreaktoren wurde zuvor beschrieben9. In dieser Studie sind detaillierte Protokolle für monoklonale Antikörpercharakterisierungsmethoden, die Daten maximieren, die aus begrenzten Probenvolumina gewonnen wurden, klar definiert. Begrenzte Mengen an geernteter Zellkulturflüssigkeit können manchmal die erfassten Produktinformationen einschränken, und die Auswahl der richtigen Analyseverfahren zur Gewinnung von Produktqualitätsdaten ist unerlässlich. Analysen sind wichtig, um vorgelagerte Prozessparameter mit den Änderungen der Produktqualität zu verknüpfen. Hier wird eine Richtlinie für Den Anwender zur Charakterisierung von mAbs bei der Arbeit mit Mikrobioreaktoren bereitgestellt.
Offenlegungen
Diese Veröffentlichung spiegelt die Ansichten des Autors wider und sollte nicht so ausgelegt werden, dass sie die Ansichten oder Richtlinien der FDA widerspiegelt.
Danksagungen
Die Autoren danken Scott Lute für die analytische Unterstützung, die er geleistet hat. Die teilweise interne Finanzierung und Unterstützung dieser Arbeiten erfolgt durch das CDER Critical Path Program (CA #1-13). Dieses Projekt wird zum Teil durch einen Termin für das Praktikums-/Forschungsbeteiligungsprogramm beim Office of Biotechnology Products, U.S. Food and Drug Administration, unterstützt, das vom Oak Ridge Institute for Science and Education zwischen dem US-Energieministerium und der FDA.
Materialien
| Name | Company | Catalog Number | Comments |
| CHO DG44 Cell Line | Invitrogen | A1100001 | |
| Akta Avant 25 | General Electric Life Sciences | 28930842 | |
| Pro Sep vA Ultra Chromatography Resin | Millipore Sigma | 115115830 | Purification Stationary Phase |
| Omnifit 10cm Column | Diba Fluid Intelligence | 006EZ-06-10-AA | Housing for Stationary Phase |
| Tris Base | Fisher Scientific | BP154-1 | |
| Superloop 10 mL | GE Healthcare | 18-1113-81 | |
| µDawn Multi Angle Light Scattering Detector | Wyatt | WUDAWN-01 | |
| 0.22 µm Millex GV Filter Unit PVDF Membrane | Merck Millipore | SLGV033RB | |
| 10X Phosphate Buffered Saline | Corning | 46-013-CM | |
| 12 mL Syringe | Covidien | 8881512878 | |
| 1290 Infinity Binary Pump | Agilent Technologies | G4220A | |
| 1290 Infinity DAD | Agilent Technologies | G4212A | |
| 1290 Infinity Sampler | Agilent Technologies | G4226A | |
| 1290 Infinity Thermostat | Agilent Technologies | G1330B | |
| 1290 Infinity Thermostatted Column Compartment | Agilent Technologies | G1316C | |
| 15 mL Falcon tube | Corning Inc. | 352097 | |
| 150 uL Glass Inserts with Polymer Feet | Agilent Technologies | 5183-2088 | |
| 50 mL Falcon tube | Corning Inc. | 352070 | |
| 96-Well Plate | Bio-Rad | 127737 | |
| Acetic Acid | Sigma-Aldrich | 695072 | |
| Acetonitrile | Fisher Chemical | BPA996-4 | |
| ACQUITY I-Class UPLC BSM | Waters Corporation | 18601504612 | |
| ACQUITY I-Class UPLC Sample Manager | Waters Corporation | 186015000 | |
| ACQUITY UPLC FLR Detector | Waters Corporation | 176015029 | |
| Amicon Ultra-4 100 kDa centrifugal filters | Merck Millipore | UFC810096 | |
| Amino Acid Standard, 1 nmol/µL | Agilent Technologies | 5061-3330 | |
| Amino Acid Supplement | Agilent Technologies | 5062-2478 | |
| Ammonium Formate Solution - Glycan Analysis | Waters Corporation | 186007081 | |
| Blue Screw Caps with Septa | Agilent Technologies | 5182-0717 | |
| CD OptiCHO AGT Medium | Thermo Fisher Scientific | A1122205 | |
| Centrifuge Tubes | Eppendorf | 22363352 | |
| Charge Variant Chip | Perkin Elmer | 760435 | |
| Charge Variant Reagent Kit | Perkin Elmer | CLS760670 | |
| Chromatography Water (MS Grade) | Fisher Chemical | W6-4 | |
| Dimethylformamide | Thermo Scientific | 20673 | |
| Extraction Plate Manifold for Oasis 96-Well Plates | Waters Corporation | 186001831 | |
| Formic Acid | Fisher Chemical | A117-50 | |
| GlycoWorks RapiFlour-MS N-Glycan Starter Kit - 24 Sample | Waters Corporation | 176003712 | |
| GXII Buffer Tubes | E&K Scientific | 697075- NC | |
| GXII Detection Window Cleaning Cloth | VWR | 21912-046 | |
| GXII HT Touch | Perkin Elmer | CLS138160 | |
| GXII Ladder Tubes | Genemate | C-3258-1 | |
| GXII Lint-Free Swab | ITW Texwipe | TX758B | |
| Hydrochloric Acid | Fisher Scientific | A144-500 | |
| Intact mAb Mass Check Standard | Waters Corporation | 186006552 | |
| Intrada Amino Acid Column 150 x 2 mm | Imtakt | WAA25 | |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | Thermo Fisher Scientific | 840274100 | |
| Optilab UT-rEX Differential Refractive Index Detector | Wyatt | WTREX-11 | |
| Perchloric acid | Aldrich Chemistry | 311421 | |
| Pipet Tips with Microcapillary for Loading Gels | Labcon | 1034-960-008 | |
| Polypropylene 96-Well Microplate, F-bottom, Chimney-style, Black | Greiner Bio-One | 655209 | |
| RapiFlour-MS Dextran Calibration Ladder | Waters Corporation | 186007982 | |
| Screw Top Clear Vial 2mL | Agilent Technologies | 5182-0715 | |
| Sodium Chloride | Fisher Scientific | S271-1 | |
| Sodium Iodide | Sigma Aldrich | 383112 | |
| TSKgel UP-SW3000 4.6mm ID x 30 cm L | Tosoh Biosciences | 003449 | |
| UNIFI Scientific Information System | Waters Corporation | 667005138 | |
| Vacuum Manifold Shims | Waters Corporation | 186007986 | |
| Vacuum Pump | Waters Corporation | 725000604 | |
| Xevo G2 Q-ToF | Waters Corporation | 186005597 | |
| Zeba Spin Desalting Column, 0.5 mL | Thermo Scientific | 89883 |
Referenzen
- . . Pharmaceutical cGMPs for the 21st Century: A Risk-Based Approach. , (2004).
- New Molecular Entity (NME) Drug and New Biologic Approvals. FDA Available from: https://www.dfa.gov/Drugs/DevelopmentApprovalProcess/HowDrugsareDevelopedandApproved/DrugandBiologicAprrovalReports/NDAandBLAApprovalReports/ucm373420.htm (2015)
- Foltz, I. N., Karow, M., Wasserman, S. M. Evolution and Emergence of Therapeutic Monoclonal Antibodies. Circulation. 127, 2222-2230 (2013).
- Kondragunta, B., Drew, J. L., Brorson, K. A., Moreira, A. R., Rao, G. Advances in clone selection using high-throughput bioreactors. Biotechnology Progress. 26 (4), 1095-1103 (2010).
- Hmiel, L., Brorson, K., Boyne, M. Post-translational structural modifications of immunoglobulin G and their effect on biological activity. Analytical & Bioanalytical Chemistry. 407 (1), 79-94 (2015).
- Rathore, A. S. Roadmap for implementation of quality by design (QbD) for biotechnology products. Trends in Biotechnology. 27 (9), 546-553 (2009).
- . International Council for Harminisation of Techinical Requirements for Pharmaceuticals for Human Use. ICH. , (1999).
- Berkowitz, S. A., Engen, J. R., Mazzeo, J. R., Jones, G. B. Analytical tools for characterizing biopharmaceuticals and the implications for biosimilars. Nature Reviews Drug Discovery. 11 (7), 527-540 (2012).
- Velugula-Yellela, S. R., et al. Use of high-throughput automated microbioreactor system for production of model IgG1 in CHO cells. Journal of Visualized Experiments. , (2018).
- Largy, E., Cantais, F., Van Vyncht, G., Beck, A., Delobel, A. Orthogonal liquid chromatography-mass spectrometry methods for the comprehensive characterization of therapeutic glycoproteins, from released glycans to intact protein level. Journal of Chromatography A. 1498, 128-146 (2017).
- Yang, J. -. M., et al. Investigation of the correlation between charge and glycosylation of IgG1 variants by liquid chromatography-mass spectrometry. Analytical Biochemistry. 448, 82-91 (2014).
- Agarabi, C. D., et al. Bioreactor Process Parameter Screening Utilizing a Plackett-Burman Design for a Model Monoclonal Antibody. Journal of Pharmaceutical Sciences. 104 (6), 1919-1928 (2015).
- Wen, J., Arakawa, T., Philo, J. S. Size-Exclusion Chromatography with On-Line Light-Scattering, Absorbance, and Refractive Index Detectors for Studying Proteins and Their Interactions. Analytical Biochemistry. 240 (2), 155-166 (1996).
- Veurink, M., Stella, C., Tabatabay, C., Pournaras, C. J., Gurny, R. Association of ranibizumab (Lucentis) or bevacizumab (Avastin) with dexamethasone and triamcinolone acetonide: An in vitro stability assessment. European Journal of Pharmaceutics and Biopharmaceutics. 78 (2), 271-277 (2011).
- Li, Y., Weiss, W. F., Roberts, C. J. Characterization of high-molecular-weight nonnative aggregates and aggregation kinetics by size exclusion chromatography with inline multi-angle laser light scattering. Journal of Pharmaceutical Sciences. 98 (11), 3997-4016 (2009).
- Espinosa-de la Garza, C. E., et al. Analysis of recombinant monoclonal antibodies by capillary zone electrophoresis. Electrophoresis. 34 (8), 1133-1140 (2013).
- Han, H., Livingston, E., Chen, X. High throughput profiling of charge heterogeneity in antibodies by microchip electrophoresis. Analytical Chemistry. 83 (21), 8184-8191 (2011).
- Wheeler, T. D., et al. Microchip zone electrophoresis for high-throughput analysis of monoclonal antibody charge variants. Analytical Chemistry. 86 (11), 5416-5424 (2014).
- Carrillo-Cocom, L., et al. Amino acid consumption in naive and recombinant CHO cell cultures: producers of a monoclonal antibody. Cytotechnology. 67 (5), 809-820 (2015).
- Chen, P., Harcum, S. W. Effects of amino acid additions on ammonium stressed CHO cells. Journal of Biotechnology. 117 (3), 277-286 (2005).
- Xing, Z., et al. Optimizing amino acid composition of CHO cell culture media for a fusion protein production. Process Biochemistry. 46 (7), 1423-1429 (2011).
- Fan, Y., et al. Amino acid and glucose metabolism in fed-batch CHO cell culture affects antibody production and glycosylation. Biotechnology and Bioengineering. 112 (3), 521-535 (2015).
- Read, E. K., et al. Fermentanomics informed amino acid supplementation of an antibody producing mammalian cell culture. Biotechnology Progress. 29 (3), 745-753 (2013).
- Mazzer, A. R., Perraud, X., Halley, J., O'Hara, J., Bracewell, D. G. Protein A chromatography increases monoclonal antibody aggregation rate during subsequent low pH virus inactivation hold. Journal of Chromatography. A. 1415, 83-90 (2015).
- Zhang, L., Luo, S., Zhang, B. Glycan analysis of therapeutic glycoproteins. MAbs. 8 (2), 205-215 (2016).
- Wahl, O., Holzgrabe, U. Amino acid analysis for pharmacopoeial purposes. Talanta. 154, 150-163 (2016).
- Le, A., Ng, A., Kwan, T., Cusmano-Ozog, K., Cowan, T. M. A rapid, sensitive method for quantitative analysis of underivatized amino acids by liquid chromatography-tandem mass spectrometry (LC-MS/MS). Journal of Chromatography B. 944, 166-174 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten