このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
エチルメタンスルホン酸変異体による小粒作物におけるゲノム中の標的誘導局所病変(TILLING)集団の開発
要約
記載は、エチルメタンスルホネート(EMS)をミュータゲンとして使用する小さな穀物作物における標的誘導局所病変(TILLING)集団を開発するためのプロトコルである。また、Cel-1アッセイを用いて変異検出するためのプロトコルも提供される。
要約
標的化された局所病変INゲノム(TILLING)は、化学的変異と標的遺伝子の配列変動の検出を含む強力な逆遺伝学ツールです。TILLINGは、特に変換ベースのアプローチが重大な制限を持つ小さな粒において、遺伝子検証のための非常に貴重な機能ゲノミクスツールです。堅牢な変異型集団を開発することは、TILLINGベースの遺伝子検証研究の効率性を決定する鍵となります。全体的な突然変異頻度が低いTILLING集団は、望ましい変異を見つけるために非現実的に大きな集団をスクリーニングする必要があることを示し、一方、高い変異原濃度は集団の死亡率が高く、不十分につながる変異型個体の数。効果的な集団が開発されると、対象遺伝子の変異を検出する複数の方法があり、プラットフォームの選択は、リソースの実験規模と可用性に依存します。変異体同定のためのCel-1アッセイおよびアガロースゲルベースのアプローチは便利で、再生可能で、資源集約的なプラットホームである。それは簡単で、計算上の知識を必要としないという点で有利であり、基本的な実験室装置を用いて少数の遺伝子の検証のために特に適している。本記事では、投与曲線の調製、変異集団の変異形成および維持、およびPCRベースのCel-1アッセイを用いた変異型集団のスクリーニングを含む、良好なTILLING集団の開発方法を説明する。.
概要
ゲノムの点の突然変異は、研究者にとって多くの有用な目的を果たすことができます。その性質と位置に応じて、これらの突然変異は、目的のタンパク質の遺伝子または異なるドメインに機能を割り当てるために使用することができます。一方、新規な遺伝的変異の源として、表現型スクリーンを用いて所望の形質に有用な変異を選択し、さらに作物改善に使用することができる。TILLINGは、化学的変異および標的遺伝子の配列変動の検出を含む強力な逆遺伝学ツールです。アラビドプシス1とショウジョウフィリアメラノガスター2で最初に開発され、TILLING集団は、ヘキサプロイドパン小麦(トリチカム・アエスチバム)3などの多くの小さな穀物作物で開発され、利用されています。大麦(大群)4, テトラプロイドデュラム小麦 (T. ディコカイドデュラム)5, ジプロイド小麦 (T. モノコカム)6と小麦エージロップスタウスキイの"D" ゲノム前駆者 7.これらのリソースは、アバイオティクスおよび生物学的ストレス耐性8を調節し、開花時間9を調節し、栄養的に優れた作物品種5を開発する遺伝子の役割を検証するために使用されてきた。
TILLINGは、エチルメタンスルホン酸エチル、アジドナトリウム、N-メチル-N-ニトロスール(MNU)、メチルメタンスルホネート(MMS)などのアルキル化変異剤剤の使用と共に、いくつかの理由から他の逆遺伝学ツールよりも優れています。第1に、変異は、実質的に任意の種または種々の植物10に対して行うことができ、小粒11の場合には特に困難である形質転換のボトルネックとは無関係である。第二に、他の遺伝子検証アプローチによって得られるノックアウト変異を生成することに加えて、様々な誤解およびスプライシング変異を誘導することができ、これは目的とするタンパク質の個々のドメインの機能を識別することができる12。さらに、TILLINGはゲノム全体に変異の不滅のコレクションを生成します。したがって、単一の集団を複数の遺伝子の機能的検証に使用できます。対照的に、他の逆遺伝学ツールは、研究13下の遺伝子のみに固有のリソースを生成する。TILLINGを通じて同定された有用な突然変異は、繁殖目的で展開することができ、多くの国で非トランスジェニック分類が依然として不確実である遺伝子編集とは異なり、規制の対象とならない。これは、国際的に取引されている小さな穀物に特に関連します14.
TILLINGは、シンプルで効率的な遺伝子検証戦略であり、目的の遺伝子を調るために変異型集団を開発する必要があります。効果的な変異型集団の開発は、TILLINGベースの遺伝子検証研究の効率を決定する鍵となります。全体的な突然変異頻度が低いTILLING集団は、望ましい変異に対して実質的に大きな集団をスクリーニングする必要があることを示し、一方、高い変異原濃度は集団の死亡率が高く、その数が不十分であることを示している。変異型個体。良好な集団が発達すると、対象遺伝子の変異を検出する複数の方法があり、プラットフォームの選択は、リソースの実験規模と可用性に依存します。全ゲノムシーケンシングおよびエキソメシーケンシングは、小さなゲノム15、16を有する植物におけるTILLING集団における全変異を特徴付けるために使用されてきた。2つのTILLING集団のエキソメ配列決定は、パンおよびデュラム小麦で行われており、望ましい変異を同定し、関心のある変異線を注文するために一般に利用可能である17。これは、望ましい突然変異の可用性の面で大きな公共リソースです。しかし、遺伝子検証研究では、野生型の線は目的の候補遺伝子を持つべきである。残念ながら、別の背景にあるいくつかの候補遺伝子の逆遺伝学ベースの検証のために、TILLING集団全体のエキソメを配列することは依然としてコストがかかります。アンプリコンシーケンシングとCel-1ベースのアッセイは、小麦の標的集団における変異の検出に使用されており、Cel-1アッセイはより単純で、計算知識を必要としないため、特に基本的な少数の遺伝子の検証に適しています。ラボ機器6,18.
本記事では、投与曲線の調製、変異集団の変異形成および維持、およびPCRベースのCel-1アッセイを用いた変異型集団のスクリーニングを含む、良好なTILLING集団の開発のための方法を説明する。.このプロトコルは、トリチカム・アエスチダム、トリチカム・モノッコカム6、大麦、エージロップス・タウチイ7、およびいくつかの変異集団の開発と利用において既に成功している。他。これらの方法の明示的な詳細と、研究者が選択した小さな穀物植物の変異原としてEMSを使用して、TILLING集団を開発するのに役立つ有用なヒントが含まれています。
プロトコル
1. 効果的な変異形成のための投与曲線の調製
- 蒸留水の50 mLを含む6つの250 mLガラスフラスコ(各フラスコで100個)に関心のある遺伝子型で100種を浸します。種子による浸漬のために室温(RT)で8時間100rpmで振る。
- ヒュームフードでは、0.4%、0.6%、0.8%、1.0%、および1.2%(w/v)エチルメタンスルホン酸(EMS)溶液を0.167、0.249、0.331、0.415、および0.498 mLのEMSをそれぞれ溶解して調用する。
注:EMSは1.206 g/mLの密度のRTで液体である。
注意:EMSを取り扱う間、適切な個人用保護具(PPE)を使用してください。 - 5つのフラスコから水を取り出し、浸漬種子を含む各フラスコに50 mLのEMS溶液を加え、0.0%、0.4%、0.6%、0.8%、1.0%、1.2%のEMS溶液で6種類の処理が行われます。75 rpmおよびRTで16時間のフラスコを振る。
- EMS溶液をデカントし、チーズクロスを使用して処理ごとに別々に処理された種子を収集します。使用したEMS溶液を1体のEMS不活性化溶液(0.1M NaOH、20%w/v Na2 S2O3)を24時間添加して不活性化します。また、汚染されたフラスコとピペットの先端をEMS不活性化液で24時間治療します。
- EMS処理された種子を水道水の下で2時間洗浄し、各種子をポッティング土壌を含むルートトレーナーに個別に移植します。
- 16時間の光期間の下で20-25 °Cで植物を成長させる。
- 移植の15日後の植物の生存に関するデータを記録する。各治療の生存率を計算するには、次の式を使用します。
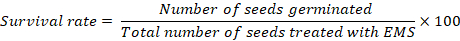
注:コントロールの発芽率が100%未満の場合、コントロールで発芽できなかった種子の数を減算した後、すべての治療における正確な生存率を計算する必要があります。40%~60%の生存率は、効果的な変異形成のために望ましい。40%-60%の望ましい致死率を達成するまで、処理された種子の生存に応じて修飾濃度で投与量最適化の第2ラウンドを実行する必要があるかもしれません。
2. 変異集団の変異形成と維持
- 300 mLの蒸留水を含む5つの1,000 mLフラスコに、少なくとも3,000種子を均等に分割(各600種子)の最終バッチを浸します。RTの下で100 rpmで8時間振ります。
注:望ましい集団の最終的な大きさは、遺伝子型の変異頻度とプロイディレベルに依存しますが、ヘキサプロイドには少なくとも3,000種、テトラプロイドには4,000種、ジプロイドには7,000種以上を使用することをお勧めします。 - ヒュームフードで、蒸留水中に最適化されたEMS濃度溶液の1,500 mLを調出します。
- フラスコから水をデカントし、600個の浸漬種子を含む各フラスコにEMS溶液の最適濃度の300mlを追加します。75 rpmとRTで16時間フラスコを振ります。
- EMSをデカントし、チーズ生地で処理された種子を収集します。手順 1.4 で行うように、EMS 不活性化溶液を使用して EMS 溶液および処理容器を不活性化します。
- EMS処理種子を水道水の下で2時間洗浄し、各EMS処理M1種子を個別にルートトレーナーに移植します。
- M1植物(M0種子由来)を16時間の光期間下で20~25°Cで成長させる。
注:関心のある遺伝子型が冬型の成長習慣を有する場合は、2つの葉の段階で6週間4°Cで苗をバーナライズする必要があるかもしれません。 - M1植物を自己受粉させ、肥沃なM1植物ごとに別々にM2種子を収穫する。
注:潜在的な横断の可能性を避けるために、麻酔の前に受粉袋でM1植物のスパイクをカバーします。 - 遺伝的冗長性を避けるために、各M1植物から単一のM2種子を植えます。
- ラック96ウェルマイクロチューブの1.1 mLの2葉段階でM2植物から組織を収集します。各植物から約80mmの葉組織を収集し、各サンプルのIDを組織回収計画に記録します。
- 凍結乾燥剤を用いて葉組織を凍結乾燥させ、-80°Cに保存する。
- M2植物を16時間の光期間で20~25°Cに維持します。
- 一定の間隔でM2植物の変異型に関するデータを記録する。期待される発薬型は、アルビノ、塩素、草芽、多変性、部分的に肥沃、滅菌などである。
- M2植物が自己受精し、成熟することを許可します。M2植物のM3種子を別々に収穫して保存する(図1)。
注:M種子は、逆遺伝学研究における表現型の検証に使用されます。したがって、Mは慎重にカタログ化され、涼しく乾燥した条件下で保存する必要があります。または、フィールドでシードを増やす必要がある場合は、各 M 植物に対してヘッド行を植え、各行の M シードを刈り取り、TILLING 母集団リソースとして個別に保存することもできます。
3. 変異体の遺伝的特徴付けのためのCel-1アッセイ
- メーカーの推奨に従って、DNA精製システムを用いた植物DNA抽出キットを用いてM2の葉組織からDNAを抽出する(材料の表を参照)。
- 分光光度計を用いてDNAを定量し、96ウェルブロックでヌクレアーゼフリー水でDNA濃度を25ng/μLに正規化します。
注:低品質のDNA(塗りつぶされたDNA)はプールされたサンプルに偽陰性をもたらす可能性があるため、ゲル上でDNAを実行してDNAの品質を確認することが重要です。 - 各サンプルの行と列のアイデンティティを維持しながら、4つの96ウェルブロックのDNAを1つのプレートに組み合わせることで、4倍のDNAプールを作成します。各サンプルから50 μLのDNAをプールプレートに加え、各プールプレートに4つの異なる96ウェルブロックから合計200 μLのDNAが含まれるようにします。
- プールプレート行列の形式でプールされたDNAの同一性をカタログ化します(例えば、プール1 A1 = Box1A1 + Box2A1 + Box3A1 + Box4A1)。
- ゲノム特異的プライマー(GSP)ページを使用して目的の遺伝子のためのゲノム特異的プライマー[A1]を設計し、ポリプロイド種のデフォルト設定で、https://probes.pw.usda.gov/GSP>ディプロイドの場合は、既定の設定を使用してプライマーを設計する場合は、プライマー 3 を使用します。必要に応じて、目的の遺伝子のコード領域全体をカバーするように、複数のプライマーを設計する。
注:最新のIWGSCアセンブリは、URGI BLASTツールを使用して、小麦に関心のある遺伝子の配列を得るために使用することができます。最適アンプリコンの長さは800~1,500bpの範囲である表1は、ヘキサプロイド小麦中のワックス遺伝子のプライマーの例を示す。 - プールされたDNA上の遺伝子特異的プライマーのPCRを次のように実行します。
- PCRバッファーの5 μL、4 μLのフォワードおよびリバースプライマーの2 μL(各)、0.1 μLのDNAポリメラーゼ(材料の表を参照)、5 μLのプールされたDNAテンプレートを加え、ヌクレアーゼフリー水を使用して体積を25μLに増やします。
- 次のように熱サイクラーでPCR反応を実行するには、タッチダウンプロファイルを使用して、1分間95°C、1分間95°Cの7サイクル、1サイクルあたり1°Cの温度低下で1分間67°C~60°C、2分間72°CのPCR反応を実行します。続いて、95°Cの30サイクルを1分間、60°Cを1分間、72°Cを2分間、72°Cで7分間の最終延長を行った。
注:上記のPCRプロファイルは、プライマー3デフォルト設定を使用して設計されたプライマーのほとんどのための小麦DNAテンプレート上で動作します。非特異的増幅の場合、PCRプロファイルは次のステップに進む前に厳格にする必要があります。
- プロファイルを使用してPCR産物を熱サイクラーでインキュベートすることにより、不一致のDNA間でヘテロドプレックスを生成する:95°C、1sの95°C、1サイクルあたり2°Cの温度低下で1分間95°C、85°C~25°Cの60サイクルサイクルあたり1°Cの減少。
- ヘテロドプレックスPCR製品に自家製Cel-1エンドヌクレアーゼを2.5μL加え、45°Cで45分間インキュベートします。0.5 M EDTA(pH 8.0)の2.5 μLを加えてCel-1反応を終了します。
注:Cel-1エンドヌクレアーゼは、Till et al. al.19によって行われるプロトコルを使用して新鮮なセロリの茎から抽出することができるそれは、以前に特徴付けられた変異体を使用してテストすることができるCel-1エンドヌクレアーゼの活性と最適量をテストすることが非常に重要です。または市販の変異検出キット。 - Cel-1処理製品を100Vで3.0%のアガロースゲルで2.5時間実行します。より小さく、ユニークな切断バンドを含むウェルは、全長の汚れたバンドに加えて、変異型DNAサンプルを含む。
- デンコンボリュート変異型プール。
- ステップ 3.9 で特定された変異プールを構成する個々の M2 DNA サンプルについては、手順 3.6、3.7、3.8、および 3.9 に従います。
- 変異体のザイゴシティを決定するには、個々のM2 DNAに対して2つのPCR(ステップ3.6に記載)を実行し、最初の反応はM2 DNAの2.5μLと野生型DNAの2.5μLを含み、2番目の反応はM2 DNAの5μLのみを含む。突然変異がヘテロジゴウスである場合、追加の割り込みバンドが両方の反応に存在します。一方、変異体がホモ接合体である場合にのみ、追加の切断バンドが最初の反応で見つかります。
- 突然変異の性質を特定するには、製造元の指示に従って Sanger シーケンシング プラットフォームを使用して確認された変異体の PCR 製品をシーケンスします。
4. 突然変異頻度の計算
注: TILLING 母集団の突然変異頻度は、その集団の個体で 1 つの突然変異が発生する平均物理距離を指します。例えば、TILLING母集団における1/35kbの突然変異頻度は、その集団の平均的な個体がゲノム内の35kbごとに1変異を有することを意味する。
- TILLING 母集団の突然変異頻度を決定するには、スクリーニングされた塩基の総数を計算します。
- スクリーニングされたベースの合計数を計算するには、PCR 製品サイズにスクリーニングされた個人の合計数を乗算します。
- スクリーニングされた塩基の総数を、次の式を用いて観察された固有の変異の数で除算すると、与えられたTILLING母集団に1個の変異を有する物理領域が得られます。
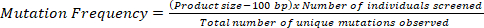
注:アガロースゲルベースのプラットフォームに基づいて両端で50 bpの解像度の制限を考慮するには、計算の製品サイズから100 bpを引きます。
結果
図2は、ヘキサプロイドパン小麦品種ジャガー、ジプロイド小麦トリチカムモノコカム6、および小麦エージロップスタウシイ7のゲノムドナーの投与曲線を示す。所望の50%の生存率に対するEMS用量は、それぞれT.モノコカム、Ae.タウシイ、およびT.aestivumについて約0.25%、0.6%およ?...
ディスカッション
TILLINGは遺伝子検証のための非常に貴重な逆遺伝学ツールであり、特に変換ベースのアプローチが深刻なボトルネックを持つ小さな穀物に対して11.高い変異頻度で変異化集団を開発することは、機能的ゲノミクス研究を行う上で重要なステップの1つである。堅牢なTILLING集団を開発する上で最も重要なステップは、EMSの最適濃度を決定することです。M1における40%〜60%...
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
この研究は、USDA国立食糧農業研究所、ハッチプロジェクト1016879、およびMAESグラント第2956952号を介してメリーランド農業実験ステーションによって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 96 well 1.1 ml microtubes in microracks | National Scientific | TN0946-08R | For collecting leaf tissues |
| Agarose I biotechnology grade | VWR | 0710-500G | |
| Biosprint 96 DNA Plant Kit | Qiagen | 941558 | Kit for DNA extraction |
| Cel-1 endonuclease | Extracted as described by Till et al 2006 | Single strand specific endonuclease | |
| Centrifuge 5430 R | Eppendorf | ||
| Ethyl methanesulfonate | Sigma Aldrich | M-0880-25G | EMS, Chemical mutagen |
| Freeze Dry/Shell freeze system | Labconco | For lyophilization of leaf tissue | |
| Kingfisher Flex purification system | Thermo fisher scientific | 5400610 | High throughput DNA extraction robot |
| My Taq DNA Polymerase | Bioline | BIO-21107 | |
| Nuclease free water | Sigma aldrich | W4502-1L | |
| NuGenius gel imaging system | Syngene | ||
| Orbit Environ-shaker | Lab-line | ||
| SPECTROstar Nano | BMG LABTECH | Nano drop for DNA quantification | |
| T100 Thermal cycler | BIO-RAD | 1861096 |
参考文献
- McCallum, C. M., Comai, L., Greene, E. A., Henikoff, S. Targeted screening for induced mutations. Nature Biotechnology. 18 (4), 455-457 (2000).
- Bentley, A., MacLennan, B., Calvo, J., Dearolf, C. R. Targeted Recovery of Mutations in Drosophila. Genetics. 156 (3), 1169-1173 (2000).
- Tsai, H., et al. Discovery of Rare Mutations in Populations: TILLING by Sequencing. Plant Physiology. 156 (3), 1257-1268 (2011).
- Caldwell, D. G., et al. A structured mutant population for forward and reverse genetics in Barley (Hordeum vulgare L.). The Plant Journal. 40 (1), 143-150 (2004).
- Hazard, B., et al. Induced Mutations in the Starch Branching Enzyme II ( SBEII ) Genes Increase Amylose and Resistant Starch Content in Durum Wheat. Crop Science. 52 (4), 1754-1766 (2012).
- Rawat, N., et al. A diploid wheat TILLING resource for wheat functional genomics. BMC Plant Biology. 12, 205 (2012).
- Rawat, N., et al. TILL-D: An Aegilops tauschii TILLING Resource for Wheat Improvement. Frontiers in Plant Science. 9, (2018).
- Rawat, N., et al. Wheat Fhb1 encodes a chimeric lectin with agglutinin domains and a pore-forming toxin-like domain conferring resistance to Fusarium head blight. Nature Genetics. 48 (12), 1576-1580 (2016).
- Kippes, N., Chen, A., Zhang, X., Lukaszewski, A. J., Dubcovsky, J. Development and characterization of a spring hexaploid wheat line with no functional VRN2 genes. Theoretical and Applied Genetics. 129 (7), 1417-1428 (2016).
- Greene, E. A., et al. Spectrum of Chemically Induced Mutations From a Large-Scale Reverse-Genetic Screen in Arabidopsis. Genetics. 164 (2), 731-740 (2003).
- Harwood, W. A. Advances and remaining challenges in the transformation of barley and wheat. Journal of Experimental Botany. 63 (5), 1791-1798 (2012).
- Henikoff, S., Comai, L. Single-Nucleotide Mutations for Plant Functional Genomics. Annual Review of Plant Biology. 54 (1), 375-401 (2003).
- Uauy, C., et al. A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat. BMC Plant Biology. 9 (1), 115 (2009).
- Uauy, C., Wulff, B. B. H., Dubcovsky, J. Combining Traditional Mutagenesis with New High-Throughput Sequencing and Genome Editing to Reveal Hidden Variation in Polyploid Wheat. Annual Review of Genetics. 51 (1), 435-454 (2017).
- Li, G., et al. The Sequences of 1504 Mutants in the Model Rice Variety Kitaake Facilitate Rapid Functional Genomic Studies. The Plant Cell. 29 (6), 1218-1231 (2017).
- Jiao, Y., et al. A Sorghum Mutant Resource as an Efficient Platform for Gene Discovery in Grasses. The Plant Cell. 28 (7), 1551-1562 (2016).
- Krasileva, K. V., et al. Uncovering hidden variation in polyploid wheat. Proceedings of the National Academy of Sciences. , 201619268 (2017).
- Dong, C., Dalton-Morgan, J., Vincent, K., Sharp, P. A Modified TILLING Method for Wheat Breeding. The Plant Genome. 2 (1), 39-47 (2009).
- Till, B. J., Zerr, T., Comai, L., Henikoff, S. A protocol for TILLING and Ecotilling in plants and animals. Nature Protocols. 1 (5), 2465-2477 (2006).
- Wu, J. -. L., et al. Chemical- and Irradiation-induced Mutants of Indica Rice IR64 for Forward and Reverse Genetics. Plant Molecular Biology. 59 (1), 85-97 (2005).
- Feldman, M., Levy, A. A. Genome Evolution Due to Allopolyploidization in Wheat. Genetics. 192 (3), 763-774 (2012).
- Comai, L. The advantages and disadvantages of being polyploid. Nature Reviews Genetics. 6 (11), 836-846 (2005).
- Guo, H., et al. Development of a High-Efficient Mutation Resource with Phenotypic Variation in Hexaploid Winter Wheat and Identification of Novel Alleles in the TaAGP.L-B1 Gene. Frontiers in Plant Science. 8, (2017).
- Rakszegi, M., et al. Diversity of agronomic and morphological traits in a mutant population of bread wheat studied in the Healthgrain program. Euphytica. 174 (3), 409-421 (2010).
- Tsai, H., Ngo, K., Lieberman, M., Missirian, V., Comai, L. Tilling by Sequencing. Plant Functional Genomics: Methods and Protocols. , 359-380 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved