Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Усиление парамагнитной релаксации для обнаружения и характеристики самоассоциаций внутренне неупорядоченных белков
В этой статье
Резюме
Представлен протокол применения ЯМР-спектроскопии с усилением парамагнитной релаксации для выявления слабых и переходных меж- и внутримолекулярных взаимодействий в внутренне неупорядоченных белках.
Аннотация
Внутренне неупорядоченные белки и внутренне неупорядоченные участки внутри белков составляют большую и функционально значимую часть протеома человека. Высокая гибкость этих последовательностей позволяет им формировать слабые, дальние и переходные взаимодействия с различными биомолекулярными партнерами. Специфические, но низкоаффинные взаимодействия способствуют беспорядочному связыванию и позволяют одному внутренне неупорядоченному сегменту взаимодействовать с множеством целевых сайтов. Из-за переходного характера этих взаимодействий их может быть трудно охарактеризовать методами структурной биологии, которые полагаются на белки для формирования единой, преобладающей конформации. ЯМР с усилением парамагнитной релаксации является полезным инструментом для идентификации и определения структурных основ слабых и переходных взаимодействий. Описан подробный протокол использования усиления парамагнитной релаксации для характеристики малонаселенных комплексов встреч, которые образуются между внутренне неупорядоченными белками и их белком, нуклеиновой кислотой или другими биомолекулярными партнерами.
Введение
Внутреннее расстройство (ID) описывает белки (IDP) или области внутри белков (IDR), которые не спонтанно сворачиваются в стабильные вторичные или третичные структуры, но являются биологически активными. Как правило, функция IDP/IDR заключается в содействии специфическим, но обратимым взаимодействиям с биомолекулами в физиологическихусловиях1. Таким образом, IDP и IDR участвуют в ряде клеточных функций, включая рекрутирование, организацию и стабилизацию мультибелковых комплексов, например, сборку и активность сплайсосомы2, рекрутирование и организацию компонентов в сайтах повреждения ДНК3, организацию и стабилизацию рекрутирования транскрипционных комплексов4 или хроматинового ремоделера BAF5. Кроме того, IDP обнаруживаются в сигнальных нексусах, где их беспорядочные связи с различными партнерами по связыванию позволяют им опосредуть передачу информации через клеточные белковыесети. Недавние работы также выявили склонность областей IDR к самоассоциированию, образуя биомолекулярные конденсаты в процессе разделения фаз жидкость-жидкость7. В настоящее время считается, что многие из вышеупомянутых функций, связанных с ИД, также включают в себя некоторый аспект образования конденсата8. Несмотря на важность ИД для сборки, стабилизации, скаффолдинга и передачи сигналов биомолекулярных комплексов, атомные детали их специфических взаимодействий трудно идентифицировать, поскольку IDP и IDR обычно не поддаются структурным исследованиям с использованием рентгеновской кристаллографии или криогенной электронной микроскопии.
Ядерный магнитный резонанс (ЯМР) является идеальным методом для исследования ИД, поскольку он не зависит от наличия жестких или однородных структурных ансамблей, а сообщает о непосредственном локальном окружении отдельных ядер. Резонансная частота, или химический сдвиг, ядра в данной молекуле зависит от слабых магнитных полей, индуцированных локальным распределением электронов, которое, в свою очередь, зависит от длины связи, углов, близости других ядер, взаимодействия с партнерами по связыванию идругих факторов. Таким образом, каждое ядро действует как уникальный, сайт-специфичный структурный зонд, чувствительный к изменениям в своей локальной химической среде. Несмотря на эти преимущества, ЯМР является массовым методом, и наблюдаемый химический сдвиг является средним значением для всех сред, отобранных конкретным ядром. Был разработан ряд методов ЯМР, многие из которых описаны в этом выпуске, для восстановления структурной, динамической и кинетической информации о высокоэнергетических, малонаселенных биомолекулярных конформациях, содержащихся в усредненном химическом сдвиге10,11. Несмотря на то, что эти состояния временно заселены, их идентификация и количественная оценка важны для определения деталей функциональных механизмов12. Например, в случае IDP и IDR конформационный ансамбль может быть смещен в сторону преимущественного отбора конформаций, продуктивных для формирования комплексов встреч с физиологическими партнерами по связыванию. Обнаружение этих состояний, а также идентификация меж- и внутримолекулярных взаимодействий и динамики, специфичных для остатков, важны для определения основных структурных механизмов функционирования белков и образования комплексов.
Описан протокол использования ЯМР с усилением парамагнитной релаксации (PRE) для исследования переходных, малонаселенных состояний, важных для формирования IDP/IDR-опосредованных биомолекулярных комплексов13. Этот подход полезен для изучения транзиторных белок-белковых взаимодействий, таких как те, которые способствуют сборке амилоидных фибрилл из α-синуклеина 14,15 или самоассоциации FUS16, а также для характеристики специфических белок-белковых взаимодействий, таких как между сигнальными белками17. Представлен пример самоассоциативного IDP, в котором специфические меж- и внутримолекулярные взаимодействия приводят к преимущественно компактным состояниям, а также сайт-специфичным взаимодействиям, которые управляют самоассоциацией.
PRE возникает в результате магнитного дипольного взаимодействия ядра с парамагнитным центром с изотропным g-тензором, обычно поставляемым в виде неспаренного электрона на нитроксидной группе или в виде парамагнитного атома металла18 (рис. 1). В то время как атомы с анизотропными g-тензорами также производят эффект PRE, анализ этих систем более сложен из-за искажающих эффектов, вносимых псевдоконтактными сдвигами (PCS) или остаточной дипольной связью (RDC)13,19. Сила взаимодействия между ядром и парамагнитным центром зависит от расстояния <r-6> между ними. Это взаимодействие приводит к увеличению скоростей ядерной релаксации, что приводит к обнаруживаемому уширению линии даже для дальних взаимодействий (~10-35 Å), поскольку магнитный момент неспаренного электрона очень силен20,21. Детектирование переходных состояний с помощью PRE возможно при соблюдении следующих двух условий; (1) переходное взаимодействие происходит в быстром обмене на временной шкале ЯМР (наблюдаемый химический сдвиг представляет собой средневзвешенное по популяции значение состояний обмена); и (2) расстояние между ядрами и парамагнитным центром короче в переходно-населенном состоянии, чем в основном состоянии11. Поперечная ПЭ обозначается Γ2 и для практических целей вычисляется по разности скоростей поперечной релаксации в 1Н между образцом, содержащим парамагнитный центр, и диамагнитным регулятором. Для углубленного рассмотрения теории PRE и связанных с ней псевдоконтактных сдвигов в режимах быстрого и медленного обмена читатель может обратиться к подробным обзорам Клора и его коллег13,22. Здесь рассматривается только ситуация, когда 1H N-Γ2 находится в режиме быстрого обмена, где из-за r-6-зависимости PRE наблюдаемая скорость релаксации связана как с расстоянием, на которое парамагнитный центр приближается к ядру, так и с количеством времени, которое он проводит в этой конформации. Таким образом, переходные конформации, не предполагающие близкого сближения, приводят к малому PRE, в то время как более тесные взаимодействия, даже если они кратковременны, приведут к большему PRE.
Для IDP PRE используется для измерения и дифференциации взаимодействий, происходящих внутри одной молекулы (внутримолекулярное) и между отдельными молекулами (межмолекулярное). Прикрепив парамагнитный центр к видимому (например, 15N-меченному) или невидимому (например, природному изобилию 14N) белку, можно определить источник (меж- или внутримолекулярный) PRE (рис. 2). Сайт-направленный мутагенез, при котором вводится остаток цистеина, является удобным подходом для присоединения парамагнитного центра (спин-метки) к белку23. Несколько типов молекул были предложены для использования в качестве спиновых меток, в том числе хелатирующие (на основе ЭДТА) и свободнорадикальные (на основе нитроксида)24. Были описаны различные спиновые метки нитроксида, которые доступны с различными химически активными химическими веществами, такими как метанетиосульфонат, малеимид и йодоацетамид25,26 (рис. 1). Гибкость, присущая метке или линкеру, может быть проблематичной для некоторых анализов, и в этих ситуациях были предложены различные стратегии для ограничения движения метки, такие как добавление громоздких химических групп или использование второго линкера для привязки метки к белку (прикрепление двух сайтов)27. 28. См. Кроме того, коммерчески доступные метки могут содержать диастереомерные белки, но, как правило, это не способствует наблюдаемому PRE29. Описано использование 3-Малеймидо-ПРОКСИЛА, присоединенного к свободному цистеину с помощью химии малеимида, поскольку он легко доступен, экономически эффективен, необратим, а восстановитель трис(2-карбоксиэтил)фосфин (ТЦЭП) может сохраняться в растворе на протяжении всей реакции мечения. Поскольку 3-Малеймидо-ПРОКСИЛ имеет изотропный g-тензор, ПКС или РДК не индуцируются, и одни и те же назначения химического сдвига могут быть использованы как для парамагнитных, так и для диамагнитных образцов13.
1HN-T 2 измеряется с использованием стратегии двух временных точек (T a, Tb), которая, как было показано ранее, столь же точна, как и сбор полного ряда эволюции, состоящего из 8-12 временных точек30. Первая временная точка (T a) устанавливается как можно ближе к нулю, а оптимальная длина второй временной точки зависит от величины наибольшего ожидаемого PRE для данного образца и может быть оценена по формуле: Tb ~ 1,15/(R 2,dia + Γ 2), где R 2,dia представляет R 2 диамагнитного образца13. Если величина наибольших PRE неизвестна, установка Tb в ~ 1 раз больше 1H T2 белка является хорошей начальной оценкой и в дальнейшем оптимизируется путем корректировки T2 для улучшения соотношения сигнал/шум. Эта двухточечная стратегия измерения значительно сокращает экспериментальное время, необходимое для измерения PRE, и дает время для большего усреднения сигнала, особенно с учетом того, что для минимизации эффектов неспецифических контактов между молекулами используются относительно разбавленные образцы. Последовательность импульсов на основе HSQC используется для измерения 1H N-T2 и была подробно описана в другом месте30. Для повышения чувствительности жесткие импульсы прямой и обратной передач INEPT могут быть заменены фигурными импульсами; в качестве альтернативы последовательность легко преобразуется в считывание на основе TROSY31. Поскольку IDP, как правило, имеют гораздо более длительную скорость поперечной релаксации, что приводит к более узкой ширине линии (из-за присущего им беспорядка), чем глобулярные белки аналогичного размера, длительное время регистрации в косвенном измерении может быть использовано для улучшения спектрального разрешения и смягчения ограничения дисперсии химического сдвига, присущего IDP.
PRE является полезным инструментом для изучения белок-белковых и белок-нуклеиновых кислот, особенно переходных или малонаселенных. Представлен подробный протокол подготовки образца ЯМР, пригодного для измерения ПРЭ, включая этапы очистки белка, сайт-ориентированного спинового мечения, настройки и калибровки импульсной программы, обработки и интерпретации данных ЯМР. В работе отмечаются важные экспериментальные соображения, которые могут повлиять на качество данных и исход эксперимента, включая концентрацию образца, выбор спиновой метки и удаление парамагнитных компонентов.
протокол
Общие требования к протоколу: оборудование для очистки белков, УФ-ВИД спектрометр, высокопольный ЯМР-спектрометр и операционное программное обеспечение, в том числе программное обеспечение для анализа постобработки; NMRPipe32, Sparky 33 (или CCPN Analysis 34, или NMRViewJ35).
1. Рекомбинантная экспрессия и очистка белка для PRE-измерений
- Спроектируйте конструкцию экспрессии для интересующего белка таким образом, чтобы присутствовал один остаток цистеина. Для введения свободного цистеина в различных позициях в интересующем белке потребуются множественные мутации36.
- Экспрессируют и очищают образец с естественным содержанием (14N) или 15N-меченым образцом интересующего белка, используя установленный протокол37.
ПРИМЕЧАНИЕ: Системы экспрессии E. coli обеспечивают экономически эффективный и надежный метод экспрессии рекомбинантных белков, поскольку изотопное обогащение 15N является минимальным требованием для биомолекулярной гетероядерной ЯМР-спектроскопии. Типичными этапами являются экспрессия в минимальной среде, хроматографическая очистка и удаление метки аффинной очистки. Этот протокол предполагает, что установлен надежный протокол экспрессии и очистки, который может производить достаточное количество белка подходящего качества для ЯМР-исследований.- Поддерживайте 1 мМ восстановителя (DTT или TCEP) в буферах на всех этапах очистки, чтобы предотвратить реакцию свободного цистеина и образование межмолекулярных дисульфидных связей для IDP.
ПРИМЕЧАНИЕ: Некоторые системы могут быть более толерантными и менее склонными к агрегации невосстановительным условиям в зависимости от конкретных характеристик белка, а также температуры, рН и буферной системы, выбранной для очистки38. - Прежде чем продолжить, удалите аффинные метки, используемые для очистки, поскольку они могут неспецифически взаимодействовать с белком непредсказуемым образом или, возможно, содержать реактивные остатки цистеина, которые могут непреднамеренно служить непреднамеренным местом прикрепления.
- Подготовьте меченый 15Н эталонный образец без мутаций цистеина, смешанный с растворимой версией спин-метки для оценки вклада растворителей PRE.
- Поддерживайте 1 мМ восстановителя (DTT или TCEP) в буферах на всех этапах очистки, чтобы предотвратить реакцию свободного цистеина и образование межмолекулярных дисульфидных связей для IDP.
2. Конъюгация спиновой метки нитроксида 3-Малеймидо-ПРОКСИЛ
- Хранят или обменивают очищенный белок в дегазированный буфер, содержащий 50 мМ Tris pH 7 и 1 мМ TCEP; буфер также может содержать до 8 М мочевины, если это необходимо для улучшения растворимости белка.
В качестве альтернативы можно быстро разбавить раствор белкового сырья по меньшей мере до 10 объемных эквивалентов дегазированного 50 мМ Tris pH 7 и 1 мМ буфера TCEP. Убедитесь, что концентрация белка перед добавлением спин-метки составляет не менее 100 мкМ. - Добавьте 3-Малеймидо-проксил из исходного раствора к 20-кратному молярному избытку интересующего белка. Образец защищают от света и кислорода и инкубируют в течение ночи при комнатной температуре или 4 °C; Легкое покачивание или нутация могут повысить эффективность маркировки.
- Готовят исходные растворы спин-метки, растворяя порошок 3-Малеймидо проксила в 95% этаноле. Аликвоты сырья можно хранить при температуре -80 °C менее 6 месяцев.
- Критический этап: Удалите непрореагировавшую свободную спин-метку, чтобы предотвратить появление неспецифических растворителей PRE. Это достигается с помощью гель-фильтрации или (предпочтительно) обширного диализа образца белка. На этом этапе белок также вводится в буфер, подходящий для ЯМР.
ПРИМЕЧАНИЕ: Восстановители должны быть приготовлены свежими, и следует учитывать совместимость между буферными компонентами; например, TCEP быстро разлагается в буферах на основе фосфатов, и этой комбинации следует избегать39. - Обработайте все буферы, используемые с этого этапа, хелатирующей смолой, селективной для двухвалентных и переходных металлов, для удаления парамагнитных ионов или погасителей спин-меток. Если белок не может быть сохранен в буфере ЯМР, концентрируйте белок, который будет быстро разбавлен в буфере, подходящем для ЯМР.
- Контроль эффективности встраивания спин-этикеток.
- Используйте реактив Эллмана (5,5-дитио-бис-(2-нитробензойная кислота) для количественного определения свободных сульфгидрильных групп в растворе40.
ПРИМЕЧАНИЕ: Подробные протоколы можно получить у производителя. Для этих целей важно определить включение спин-метки, концентрация свободных сульфгидрильных групп сравнивается с общей концентрацией белка. Процент свободных сульфгидрильных групп — это процент молекул, к которым не прикреплена нитроксидная спиновая метка. - Отслеживайте интенсивность пика, соответствующего меченому остатку цистеина, чтобы судить о встраивании спин-метки в интересующий белок.
ПРИМЕЧАНИЕ: Это быстрый и эффективный подход к определению степени спинового мечения белка. Полное включение спин-метки приведет к исчезновению пика из спектра. При плохой дисперсии, характерной для IDP, пик, соответствующий мутантному остатку цистеина, не всегда может быть легко идентифицирован, поэтому рекомендуется использовать реагент Эллмана (шаг 2.6.1).
- Используйте реактив Эллмана (5,5-дитио-бис-(2-нитробензойная кислота) для количественного определения свободных сульфгидрильных групп в растворе40.
3. Подготовьте образец ЯМР для измерения внутри- или межмолекулярного PRE
- Подготовка образца для измерения внутримолекулярного PRE
- В буфере, пригодном для ЯМР, получают изотопно обогащенный 15Н, спин-меченый белок до концентрации не менее 100 мкМ, но не более 300 мкМ. Общий объем образца (включаяD2O) составляет 500 - 550 мкл.
ПРИМЕЧАНИЕ: К распространенным буферам для ЯМР относятся фосфаты, ацетат, (би)карбонат и TRIS. Также могут подойти буферы Good, такие как MES, HEPES. Будьте осторожны при выборе буферов, чтобы не допустить перекрестной реактивности с другими компонентами решения. - Убедитесь, что pH ~7,2 или ниже, чтобы свести к минимуму последствия амидного протонного обмена с водой. Концентрация соли должна быть как можно ниже (обычно менее 150 мМ), хотя основное внимание уделяется поддержанию стабильности белка.
ПРИМЕЧАНИЕ: Подходы к проведению экспериментов с ЯМР в условиях высокой содержания соли описаны в другом месте41.
- В буфере, пригодном для ЯМР, получают изотопно обогащенный 15Н, спин-меченый белок до концентрации не менее 100 мкМ, но не более 300 мкМ. Общий объем образца (включаяD2O) составляет 500 - 550 мкл.
- Подготовка образца для измерения межмолекулярного PRE
- Выполните этот шаг или шаг 3.1; Они не выполняются одновременно. Приготовьте 14N натурального изобилия, спин-меченого белка в выбранном буфере ЯМР.
- Подготавливают образец белка, смешивая изотопно обогащенный 15N немеченый спин-меченый белок с 1%-50% 14 Н натурального изобилия 14Н спин-меченого белка так, чтобы конечная концентрация была идентична образцу, приготовленному в 3.1.1. Общий объем образца (включаяD2O) составляет 500 - 550 мкл.
- Эмпирически оптимизировать соотношение белков 15N и 14N для каждого исследуемого белка. Соотношение 1%, 5% и 20% белка, меченного 14N-спином, является хорошей отправной точкой.
ПРИМЕЧАНИЕ: Накопление PRE в зависимости от добавленного белка, меченного 14N-спином, указывает на специфический эффект; наблюдаемый PRE является специфичным для выборки, поскольку он зависит от расстояния и популяции (как обсуждалось выше), и, следовательно, потребуются более высокие соотношения белка, меченного 14N-спином, если взаимодействие является особенно переходным17.
- Перенесите образец ЯМР (интра- или межмолекулярный) в 5-миллиметровую ЯМР-трубку, подходящую для использования в высокопольных магнитах, с помощью стеклянной пипетки или микропипетки с длинным стержнем (9 дюймов). Убедитесь, что все образцы ЯМР содержат 5%-10%D2O, чтобы облегчить блокировку поля.
ПРИМЕЧАНИЕ: Пробирки для ЯМР, в которых используются полимерные пробки для уменьшения необходимого объема образца, не рекомендуются для измерений PRE из-за трудностей, связанных с эффективным прокладкой образца.
4. Настройте ЯМР-спектрометр и проведите эксперимент с определенными параметрами
- Соблюдайте особую осторожность при работе со сверхпроводящими высокопольными ЯМР-спектрометрами.
ПРИМЕЧАНИЕ: Опасности включают травмы из-за внезапного ускорения металлических предметов по направлению к магниту, помех имплантированным медицинским устройствам и удушье из-за внезапного высвобождения газа N 2 и He2 в случае затухания магнита. Следующие шаги предполагают, что читатель прошел необходимую подготовку, осведомлен об этих и других местных опасностях и получил разрешение от руководителя объекта на работу со спектрометром ЯМР. Если вы сомневаетесь в шаге или инструкции, проконсультируйтесь с менеджером объекта или опытным пользователем, чтобы предотвратить возможные травмы или повреждение спектрометра. - Следующие шаги предполагают наличие коммерческого ЯМР-спектрометра, работающего под управлением современной версии программного обеспечения для управления сбором данных. Скачайте файлы программы pulse и параметров и разместите их в соответствующих каталогах.
ПРИМЕЧАНИЕ: Импульсная программа и набор параметров, подходящие для использования со спектрометром Bruker и TopSpin (3.2 или более поздней версии), предоставляются по запросу авторов.- Критический шаг: предполагается знакомство с установкой неродных импульсных программ ЯМР; При необходимости проконсультируйтесь с управляющим объектом или опытным пользователем.
- Поместите образец в магнит, зафиксируйте сигнал 2H с помощью команды Lock, настройте и согласуйте канал 1H в соответствии с протоколами оборудования (точная процедура будет зависеть от того, оснащен ли датчик модулем дистанционной настройки и согласования).
- Отрегулируйте регулировочные шайбы с помощью подпрограммы topshim, чтобы оптимизировать подавление сигнала растворителя.
- Откалибруйте импульсы 1Н и 15Н 90° стандартными методами.
- Откалибруйте импульс 1H с помощью программы popt (сначала используйте pulsecal для оценки длины импульса).
- Откалибруйте импульс 15Н по стандартному образцу; Убедитесь, что это значение было откалибровано недавно, обсудив это с техническим директором или опытным пользователем.
- В качестве альтернативы можно откалибровать импульс 15Н на образце, изменяя один из 90° импульсов эксперимента HMQC до тех пор, пока не будет получен нулевой сигнал.
- Определите правильное затухание для сформированных импульсов с помощью функции инструмента формы (stdisp).
- Откройте соответствующий файл фигуры pulse, нажав на значок папки. Сформированные импульсы находятся в разделе параметров импульсов ACQUPARS.
- Загрузите файл определения импульса и нажмите Analyze Waveform > Integrate Shape. Введите откалиброванный жесткий импульс 1H 90°, желаемую длину импульса и вращение (90° или 180°).
- Рассчитайте уровень мощности сформированного импульса, добавив изменение уровня мощности к затуханию для калиброванного импульса 90°.
- Запись стандартного 1H, 15N HSQC (hsqcetfpf3gpsi) для оптимизации ширины развертки, несущей частоты и проверки водяного подавления25.
- Отрегулируйте ширину сдвига и количество косвенных приращений размеров с помощью команд sw и td или непосредственно в соответствующих диалоговых окнах. Как правило, для сбора PRE спектральные ширины выбираются таким образом, чтобы спектр не был свернут.
5. Постановка эксперимента 1H N-T2
- Откалибруйте сформированные импульсы, как описано выше (4.4.5-4.5.7). Сформированные файлы параметров импульсов для эксперимента PRE: Eburp2.1000 (импульс 90°), Reburp.1000 и Iburp2.1000. Введите калиброванную длительность импульса в разделе параметров импульсов на вкладке ACQUPARS .
- В этом эксперименте измеряется 1HN-T 2 с использованием подхода30 с двумя точками задержки по времени.
- Установите временные задержки, отредактировав файл vdlist, первая задержка (Ta) устанавливается равной 0,01 мс.
- Выберите вторую задержку, (T b), используя отношение к ожидаемому максимальному PRE (T b ~ 1,15/(R 2,dia + Γ 2), где R 2,dia представляет R2 диамагнитного образца13. Без предварительного знания величины вклада PRE в наблюдаемую релаксацию, хорошей отправной точкой является установка T b в ~1x 1H T 2.
- Затем определите подходящее значение, сравнив первые приращения (обработанные командой efp) спектров T a и T b и настроив T b таким образом, чтобы сигнал затухал до 40%-50% от его начального значения.
ПРИМЕЧАНИЕ: Этот подход оптимизирует спектральное соотношение сигнал/шум, что необходимо для образцов, которые не могут быть высококонцентрированными (< 50 мкМ). Подходящие значенияTb зависят от образца, но обычно колеблются от 8 до 40 мс для белка среднего размера.
- Определите количество сложных точек для записи и количество сканирований для достаточного усреднения сигнала. Поскольку IDP имеют более длительное содержание 15N T2, чем свернутые белки сопоставимого размера, можно использовать более длительное время поглощения в косвенном измерении.
ПРИМЕЧАНИЕ: Это значение зависит от конкретных характеристик белка, но может быть приблизительно оценено по 15N T 2 и оптимизировано путем мониторинга затухания сигнала в FID. Для прямого измерения для большинства образцов достаточно 1024* комплексных точек (ширина развертки 13 ppm, время сбора 112,6 мс). - Используйте команду expt для вычисления времени эксперимента, а затем запустите эксперимент с помощью команды zg.
6. Изготовьте диамагнитную пробу, восстановив спин-метку аскорбиновой кислотой
- Растворите аскорбат натрия в буфере ЯМР и отрегулируйте pH в соответствии с исходным буфером ЯМР.
- Рассчитайте концентрацию аскорбата натрия таким образом, чтобы можно было добавить 10-кратный молярный избыток аскорбата над концентрацией спин-метки при наименьшем изменении объема пробы. Например, для образца белка размером 100 мкМ подходит запас аскорбата в 100 мМ. Для уменьшения спин-метки потребуется добавить 5,5 мкл исходного раствора аскорбиновой кислоты, что составляет всего 1 % от общего объема образца.
- Добавьте необходимое количество аскорбиновой кислоты в пробирку для ЯМР, поместив каплю ниже края пробирки, закройте пробирку, осторожно переверните пробирку для смешивания, а затем отжмите при 200-400 x g в течение 10-20 с в центрифуге с ручным приводом, чтобы образец осел на дне пробирки.
- Оберните ЯМР-пробирку фольгой, чтобы защитить от света, и дайте реакции протекать не менее 3 часов.
- Запишите 1H N-T2 на диамагнитном образце, используя те же параметры, что и парамагнитный образец.
- Выполните повторную калибровку импульсов. Однако они не должны были измениться по сравнению с парамагнитными измерениями; Если они значительно отличаются (> разницей в 0,5 мкс), учитывайте качество образца (например, разложение, осаждение).
- Убедитесь, что все параметры захвата, включая заданные задержки релаксации (vdlist), количество фиктивных сканирований, количество собранных сканов, количество собранных сложных точек, время захвата, ширину развертки и несущие частоты, остаются одинаковыми для диамагнитных и парамагнитных образцов.
7. Процессные парамагнитные и диамагнитные спектры
- Скопируйте данные на локальный компьютер или рабочую станцию, на которой установлены и настроены NMRPipe и Sparky. Создайте папку с именем proc в каталоге данных эксперимента, содержащем файл ser.
- Скопируйте скрипты NMRPipe fid.com, p3d.com и nmrproc.com в proc (скрипты обработки доступны по запросу авторов).
- Используйте скрипт fid.com для преобразования формата данных Bruker (ser) в формат NMRPipe.
- Используйте скрипт p3D.com для разбиения псевдо3D-плоскостей на отдельные спектры.
- Используйте сценарий nmrproc.com для чтения выходных данных сценария fid.com, применения подавления растворителей, оконной функции, добавления нулей к исходным данным (заполнение нулями), применения фазовой коррекции, выполнения преобразования Фурье, обрезки данных для отображения и записи обработанных данных на диск. Сценарий будет выводить по одному файлу для каждой записанной задержки релаксации (T a и Tb).
ПРИМЕЧАНИЕ: Каждый из этих сценариев настраивается для оптимизации обработки в соответствии с конкретными деталями каждого эксперимента. Учебные пособия и примеры наборов данных включены в дистрибутив NMRPipe, доступный на веб-сайте NMRPipe32. NMRDraw может быть использован для спектрального просмотра во время обработки (например, для установки соответствующих фазовых углов и т.д.). Параметры, доступные для команд NMRPipe, можно просмотреть с помощью команды nmrPipe -help.
8. Передача резонансных назначений и извлечение высот пиков
- Измените информацию заголовка файла для каждого файла спектра (T a, Tb как для парамагнитных, так и для диамагнитных образцов) с помощью команды sethdr [filename] -ndim 2.
- Используйте Sparky для извлечения пиковых высот33, выполнив шаги 8.3-8.5. Другие программные пакеты, в том числе NMRPipe (NMRDraw)32, CCPN Analysis 34 и NMRViewJ35, также подходят.
- Прочтите спектральные файлы в Sparky. На этом этапе набор данных будет состоять из одного спектра для каждой временной точки спектра (T a, Tb) как для парамагнитных, так и для диамагнитных образцов, измеренных для каждого положения спиновой метки в белке.
- Используйте Sparky для выбора пиков (команда: F8, затем щелкните и перетащите) и переноса назначений с помощью инструмента «Перенести список пиков» из списка пиков.
ПРИМЕЧАНИЕ: Резонансные присвоения интересующего белка необходимы для последовательно-специфической интерпретации наблюдаемых PREs36.- Задайте контуры как в парамагнитном, так и в диамагнитном спектрах на одном уровне. Убедитесь, что контуры настроены таким образом, чтобы спектры, собранные после временной задержки, не исключали пики, но были достаточно высокими, чтобы спектры Ta не были чрезмерно зашумленными.
- Сохраните новые списки пиков для каждого спектра и включите измеренную пиковую интенсивность и вычисленное Sparky отношение сигнал/шум (команда: lt, чтобы открыть список пиков, нажмите параметры, чтобы включить столбцы интенсивности и SNR, команда: сохранить).
9. Извлеките 1ч N-T2 нормы на каждый остаток и рассчитайте PRE
- Импортируйте списки пиков в программное обеспечение для работы с электронными таблицами или предпочитаемый язык программирования, например Python.
ПРИМЕЧАНИЕ: Для каждой позиции спин-метки на белке набор данных будет состоять из четырех списков пиков с соответствующими пиковыми интенсивностями, по одному из Ta и Tb как для парамагнитных, так и для диамагнитных экспериментов. - Рассчитайте 1HN R2 как для парамагнитного, так и для диамагнитного образцов, используя уравнение:
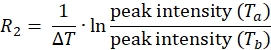
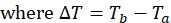
- Используйте приведенное выше уравнение для определения скорости релаксации для каждого остатка для парамагнитных и диамагнитных образцов.
- Определите норму 1HN-Γ 2 для каждого остатка, используя уравнение:
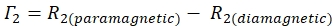
- Используйте вычисленное отношение сигнал/шум (SNR) Sparky, чтобы вычислить неопределенность пиковой высоты для каждого остатка.
- Распространите ошибку с помощью уравнения:

- Постройте график 1HN-Γ 2 в зависимости от остаточного числа с использованием диаграммы рассеяния с учетом погрешности, рассчитанной в 9.6.
Результаты
Внутримолекулярные 1H N-Γ2 PRE регистрировали на самоассоциирующем, внутренне неупорядоченном фрагменте (остатки 171-264), полученном из домена низкой сложности РНК-связывающего белка EWSR142 (рис. 3). Ожидается, что остатки, находящиеся в непос?...
Обсуждение
Представлен метод характеристики переходных взаимодействий, существующих в малых популяциях между внутренне неупорядоченными белками и различными партнерами связывания с использованием PRE. В показанном примере белок является самоассоциативным, и, таким образом, PRE может возникать в ...
Раскрытие информации
Все авторы прочитали и одобрили рукопись. Конфликт интересов не декларируется.
Благодарности
Мы благодарим докторов Джинфа Ин и Кристин Кано за полезные дискуссии и техническую помощь. DSL является стипендиатом St. Baldrick's Scholar и выражает признательность за поддержку Фонда St. Baldrick's Foundation (634706). Эта работа была частично поддержана Фондом Уэлча (AQ-2001-20190330) для DSL, Фондом Макса и Минни Томерлин Фолькер (грант Фонда Волкера для молодых исследователей для DSL), стартовыми фондами UTHSA для DSL и стипендией Грихи для выпускников в области детского здоровья для CNJ. Эта работа основана на исследованиях, проведенных в Центре структурной биологии, входящем в состав Институциональных исследовательских центров Научного центра здоровья Техасского университета в Сан-Антонио, при поддержке Офиса вице-президента по исследованиям и Общего ресурса по разработке лекарств и структурной биологии Онкологического центра Мэйса (NIH P30 CA054174).
Материалы
| Name | Company | Catalog Number | Comments |
| 0.45 µm and 0.22 µm syringe filters | Millipore Sigma | SLHVM33RS SLGVR33RS | Filter lysate before first purification step and before size exclusion chromatography. |
| 100 mm Petri Dish | Fisher | FB0875713 | Agar plates for bacterial transformation. |
| 14N Ammonium chloride | Sigma Aldrich | 576794 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 15N Ammonium chloride | Sigma Aldrich | 299251 | Use of 15N in M9 medium will produce an NMR visible protein, 14N will produces an NMR invisible protein |
| 3 L Fernbach baffled flask | Corning | 431523 | Bacterial expression culture |
| 3-Maleimido-Proxyl | Sigma Aldrich | 253375 | Nitroxide spin label |
| 50 mL conical centrifuge tubes | Thermo Fisher | 14-432-22 | Solution/protein storage |
| Amicon centrifugal filter | Millipore Sigma | UFC900308 | Protein concentration |
| Ampicillan | Sigma Aldrich | A5354 | Antibiotic for a selective marker, exact choice depends on the expression construct plasmid |
| Analytical balance | Oahus | 30061978 | Explorer Pro, for weighing reagents |
| Ascorbic acid | Sigma Aldrich | AX1775 | Reduces nitroxide spin label |
| Autoclave | Sterilize glassware and culture media | ||
| Calcium chloride | Sigma Aldrich | C4901 | M9 media component |
| Centrifuge bottles | Thermo Fisher | 010-1459 | Harvest E. coli cells after recombinant protein expression |
| Centrifuge, hand-crank | Thomas Scientific | 0241C68 | Boekel hand-driven, low-speed centrifuge with 15 mL buckets that can accommodate NMR tubes |
| Chelex 100 | Sigma Aldrich | C7901 | Remove contaminating paramagnetic compounds from buffer solutions |
| Computer workstation | Linux or Mac OS compatable with NMR data processing and analysis software packages such as NMRPipe and Sparky | ||
| Deuterium oxide | Sigma Aldrich | 151882 | Needed for NMR lock signal |
| Dextrose | Sigma Aldrich | D9434 | M9 media component |
| Dibasic Sodium Phosphate | Sigma Aldrich | S5136 | M9 media component |
| Ellman's reagent (5,5-dithio-bis-(2-nitrobenzoic acid) | Thermo Fisher | 22582 | Quantification of free cystiene residues |
| High speed centrifuge tubes | Thermo Fisher | 3114-0050 | Used to clear bacterial lysate. |
| High-field NMR instrument (600 - 800 MHz) | Bruker | Equiped with a multichannel cryogenic probe and temperature control | |
| IMAC column, HisTrap FF | Cytvia | 17528601 | Initial fractionation of crude bacterial lysate |
| Isopropyl B-D-thiogalactoside (IPTG) | Sigma Aldrich | I6758 | Induces protein expression for genes under control of lac operator |
| LB agar | Thermo Fisher | 22700025 | Items are used for transforming E. coli to express protein of interest, substitions for any of these items with like products is acceptable. |
| LB broth | Thermo Fisher | 12780052 | |
| Low-pressure chromatography system | Bio-Rad | 7318300 | BioRad BioLogic is used for low-pressure chomatograph such as running IMAC columns |
| Magnesium sulfate | Sigma Aldrich | M7506 | M9 media component |
| Medium pressure chromatography system | Bio-Rad | 7880007 | BioRad NGC equipped with a multi-wavelength detector, pH and conductivity monitors, and automatic fraction collector |
| MEM vitamin solution | Sigma Aldrich | M6895 | M9 media component |
| Microfluidizer | Avestin | EmulsiFlex-C3 | Provides rapid and efficient bacterial cell lysis |
| Micropipettes | Thermo Fisher | Calibrated set of micropippetters with properly fitting disposable tips (available from multiple manufacturers e.g. Eppendorf) | |
| Monobasic potassium phosphate | Sigma Aldrich | 1551139 | M9 media component |
| NMR pipettes | Sigma Aldrich | 255688 | To remove sample from NMR tube |
| NMR sample tube | NewEra | NE-SL5 | Suitable for high-field NMR spectrometers |
| Preparative Centrifuge | Beckman Coulter | Avanti J-HC | Harvest E. coli cells after recombinant protein expression |
| Round bottom polystyrene centrifuge tubes | Corning | 352057 | Clear bacterial lysate |
| Shaking incubator | Eppendorf | S44I200005 | Temperature controlled growth of E. coli starter and expression cultures |
| Sodium chloride | Sigma Aldrich | S5886 | M9 media component |
| Sonicating water bath and vacuum source | Thomas Scientific | Used to degas buffer solutions | |
| Sonicator | Thermo Fisher | FB505110 | Used for bacterial cell lysis or shearing bacterial DNA |
| Spectrophotometer | Implen | OD600 Diluphotometer | Monitor growth of E.coli protein expression cultures |
| Superdex 200 16/600 size exculsion colum | Cytvia | 28989333 | Final protein purification step |
| Topspin software, version 3.2 or later | Bruker | Operating software for the NMR instrument | |
| Transformation competent E. coli cells | Thermo Fisher | C600003 | One Shot BL21 Star (DE3) chemically competent E. coli, other strains may be compatable |
| Tris(2-carboxyethyl)phosphine (TCEP) | ThermoFisher | 20490 | Reducing agent compatable with some sulfhydryl-reactive conjugations |
| UV-Vis spectrophotometer | Implen | NP80 | Measure protein concentration. |
| Water bath, temperature controlled | ThermoFisher | FSGPD25 | For heat shock step of bacterial transformation |
| Yeast extract | Sigma Aldrich | Y1625 | For supplementing M9 media if required |
Ссылки
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nature Reviews: Molecular Cell Biology. 6 (3), 197-208 (2005).
- Korneta, I., Bujnicki, J. M. Intrinsic disorder in the human spliceosomal proteome. PLoS Computational Biology. 8 (8), 1002641 (2012).
- Frege, T., Uversky, V. N. Intrinsically disordered proteins in the nucleus of human cells. Biochemistry and Biophysics Reports. 1, 33-51 (2015).
- Liu, J., et al. Intrinsic disorder in transcription factors. Biochemistry. 45 (22), 6873-6888 (2006).
- El Hadidy, N., Uversky, V. N. Intrinsic disorder of the BAF complex: Roles in chromatin remodeling and disease development. International Journal of Molecular Sciences. 20 (21), (2019).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nature Reviews: Molecular Cell Biology. 16 (1), 18-29 (2015).
- Brangwynne, C. P. Phase transitions and size scaling of membrane-less organelles. Journal of Cell Biology. 203 (6), 875-881 (2013).
- Shin, Y., Brangwynne, C. P. Liquid phase condensation in cell physiology and disease. Science. 357 (6357), (2017).
- Cavanagh, J. . Protein NMR spectroscopy : principles and practice. 1st edition. , (2018).
- Sekhar, A., Kay, L. E. NMR paves the way for atomic level descriptions of sparsely populated, transiently formed biomolecular conformers. Proceedings of the National Academy of Sciences of the United States of America. 110 (32), 12867-12874 (2013).
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Alderson, T. R., Kay, L. E. NMR spectroscopy captures the essential role of dynamics in regulating biomolecular function. Cell. 184 (3), 577-595 (2021).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chemical Reviews. 109 (9), 4108-4139 (2009).
- Wu, K. P., Baum, J. Detection of transient interchain interactions in the intrinsically disordered protein alpha-synuclein by NMR paramagnetic relaxation enhancement. Journal of the American Chemical Society. 132 (16), 5546-5547 (2010).
- Janowska, M. K., Wu, K. P., Baum, J. Unveiling transient protein-protein interactions that modulate inhibition of alpha-synuclein aggregation by beta-synuclein, a pre-synaptic protein that co-localizes with alpha-synuclein. Scientific Reports. 5, 15164 (2015).
- Murthy, A. C., et al. Molecular interactions underlying liquid-liquid phase separation of the FUS low-complexity domain. Nature Structural & Molecular Biology. 26 (7), 637-648 (2019).
- Fawzi, N. L., Doucleff, M., Suh, J. Y., Clore, G. M. Mechanistic details of a protein-protein association pathway revealed by paramagnetic relaxation enhancement titration measurements. Proceedings of the National Academy of Sciences of the United States of America. 107 (4), 1379-1384 (2010).
- Griffith, O. H., Waggoner, A. S. Nitroxide free radicals: spin labels for probing biomolecular structure. Accounts of Chemical Research. 2 (2), 17-24 (1969).
- Bertini, I., Luchinat, C., Parigi, G., Ravera, E. . NMR of Paramagnetic Macromolecules, Applications to Metallobiomolecules and Models. 2 edn. , (2016).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Physical Review. 73 (7), 679-712 (1948).
- Solomon, I. Relaxation processes in a system of two spins. Physical Review. 99 (2), 559 (1955).
- Clore, G. M. Practical aspects of paramagnetic relaxation enhancement in biological macromolecules. Methods in Enzymology. 564, 485-497 (2015).
- Klare, J. P. Site-directed spin labeling EPR spectroscopy in protein research. Biological Chemistry. 394 (10), 1281-1300 (2013).
- Clore, G. M., Tang, C., Iwahara, J. Elucidating transient macromolecular interactions using paramagnetic relaxation enhancement. Current Opinion in Structural Biology. 17 (5), 603-616 (2007).
- Melanson, M., Sood, A., Torok, F., Torok, M. Introduction to spin label electron paramagnetic resonance spectroscopy of proteins. Biochemistry and Molecular Biology Education. 41 (3), 156-162 (2013).
- Czogalla, A., Pieciul, A., Jezierski, A., Sikorski, A. F. Attaching a spin to a protein -- site-directed spin labeling in structural biology. Acta Biochimica Polonica. 54 (2), 235-244 (2007).
- Lindfors, H. E., de Koning, P. E., Drijfhout, J. W., Venezia, B., Ubbink, M. Mobility of TOAC spin-labelled peptides binding to the Src SH3 domain studied by paramagnetic NMR. Journal of Biomolecular NMR. 41 (3), 157-167 (2008).
- Fawzi, N. L., et al. A rigid disulfide-linked nitroxide side chain simplifies the quantitative analysis of PRE data. Journal of Biomolecular NMR. 51 (1-2), 105-114 (2011).
- Bleicken, S., et al. gem-Diethyl pyrroline nitroxide spin labels: Synthesis, EPR characterization, rotamer libraries and biocompatibility. ChemistryOpen. 8 (8), 1035 (2019).
- Iwahara, J., Tang, C., Clore, G. M. Practical aspects of 1H transverse paramagnetic relaxation enhancement measurements on macromolecules. Journal of Magnetic Resonance. 184, 185-195 (2007).
- Venditti, V., Fawzi, N. L. Probing the atomic structure of transient protein contacts by paramagnetic relaxation enhancement solution NMR. Methods in Molecular Biology. 1688, 243-255 (2018).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. Journal of Biomolecular NMR. 6 (3), 277-293 (1995).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Vranken, W. F., et al. The CCPN data model for NMR spectroscopy: development of a software pipeline. Proteins. 59 (4), 687-696 (2005).
- Johnson, B. A. Using NMRView to visualize and analyze the NMR spectra of macromolecules. Methods in Molecular Biology. 278, 313-352 (2004).
- Sjodt, M., Clubb, R. T. Nitroxide labeling of proteins and the determination of paramagnetic relaxation derived distance restraints for NMR studies. Bio-Protocol. 7 (7), (2017).
- Zhang, H., van Ingen, H. Isotope-labeling strategies for solution NMR studies of macromolecular assemblies. Current Opinion in Structural Biology. 38, 75-82 (2016).
- Rabdano, S. O., et al. Onset of disorder and protein aggregation due to oxidation-induced intermolecular disulfide bonds: case study of RRM2 domain from TDP-43. Scientific Reports. 7 (1), 11161 (2017).
- Burns, J. A., Butler, J. C., Moran, J., Whitesides, G. M. Selective reduction of disulfides by tris(2-carboxyethyl)phosphine. Journal of Organic Chemistry. 56 (8), 2648-2650 (1991).
- Ellman, G. L. Tissue sulfhydryl groups. Archives of Biochemistry and Biophysics. 82 (1), 70-77 (1959).
- Binbuga, B., Boroujerdi, A. F., Young, J. K. Structure in an extreme environment: NMR at high salt. Protein Science. 16 (8), 1783-1787 (2007).
- Schwartz, J. C., Cech, T. R., Parker, R. R. Biochemical properties and biological functions of FET proteins. Annual Review of Biochemistry. 84, 355-379 (2015).
- Nabuurs, S. M., de Kort, B. J., Westphal, A. H., van Mierlo, C. P. Non-native hydrophobic interactions detected in unfolded apoflavodoxin by paramagnetic relaxation enhancement. European Biophysics Journal. 39 (4), 689-698 (2010).
- Wiedemann, C., Kumar, A., Lang, A., Ohlenschlager, O. Cysteines and disulfide bonds as structure-forming units: Insights from different domains of life and the potential for characterization by NMR. Frontiers in Chemistry. 8, 280 (2020).
- Wommack, A. J., et al. NMR solution structure and condition-dependent oligomerization of the antimicrobial peptide human defensin 5. Biochemistry. 51 (48), 9624-9637 (2012).
- Taylor, A. M., et al. Detailed characterization of cysteine-less P-glycoprotein reveals subtle pharmacological differences in function from wild-type protein. British Journal of Pharmacology. 134 (8), 1609-1618 (2001).
- Hu, K., Doucleff, M., Clore, G. M. Using multiple quantum coherence to increase the 15N resolution in a three-dimensional TROSY HNCO experiment for accurate PRE and RDC measurements. Journal of Magnetic Resonance. 200 (2), 173-177 (2009).
- Anthis, N. J., Doucleff, M., Clore, G. M. Transient, sparsely populated compact states of apo and calcium-loaded calmodulin probed by paramagnetic relaxation enhancement: interplay of conformational selection and induced fit. Journal of the American Chemical Society. 133 (46), 18966-18974 (2011).
- Battiste, J. L., Wagner, G. Utilization of site-directed spin labeling and high-resolution heteronuclear nuclear magnetic resonance for global fold determination of large proteins with limited nuclear overhauser effect data. Biochemistry. 39 (18), 5355-5365 (2000).
- Donaldson, L. W., et al. Structural characterization of proteins with an attached ATCUN motif by paramagnetic relaxation enhancement NMR spectroscopy. Journal of the American Chemical Society. 123 (40), 9843-9847 (2001).
- Gaponenko, V., et al. Protein global fold determination using site-directed spin and isotope labeling. Protein Science. 9 (2), 302-309 (2000).
- Trindade, I. B., Invernici, M., Cantini, F., Louro, R. O., Piccioli, M. PRE-driven protein NMR structures: an alternative approach in highly paramagnetic systems. FEBS Journal. 288 (9), 3010-3023 (2021).
- Nitsche, C., Otting, G. Pseudocontact shifts in biomolecular NMR using paramagnetic metal tags. Progress in Nuclear Magnetic Resonance Spectroscopy. 98-99, 20-49 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены